李秀, 杨海涛, 王泽方


天津大学生命科学学院, 天津 300072
收稿日期:2019-02-28;修回日期:2019-03-28;网络出版日期:2019-04-09
基金项目:国家自然科学基金(31528006,81601593);国家重点基础研究发展计划(973计划)(2015CB859800,2014CB542800);国家重点研发计划(2016YFD0500300)
*通信作者:王泽方, Tel:+86-22-27403906, E-mail:zefangwang@tju.edu.cn.
摘要:聚对苯二甲酸乙二醇酯(Polyethylene terephthalate,PET)因其良好的耐用性和可塑性,已在世界范围内的工业领域和日常生活中得到广泛应用。目前自然环境中大量PET使用废弃物的积累和迁移给全球生态系统带来了严重负担,因此PET的降解问题已成为全球性的热点问题。微生物酶降解法目前被认为是一种理想绿色PET降解方法,有希望应用于大规模降解PET废弃物降解处理。传统的PET降解酶主要包括脂肪酶、酯酶和角质酶等,但这些酶的PET降解活性相对不高。近期科学家从Ideonella sakaiensis细菌中分离了一种新型水解酶PETase,能够特异性高效降解PET。本文从结构生物学角度对多种PET降解酶进行梳理,重点总结了新近发现的PETase催化机制,为发展改造更有效的PET降解酶提供理论依据。
关键词:聚对苯二甲酸乙二醇酯PET降解酶结构研究降解机制
Advance in polyethylene terephthalate degrading enzyme
Xiu Li, Haitao Yang, Zefang Wang


School of Life Sciences, Tianjin University, Tianjin 300072, China
Received: 28 February 2019; Revised: 28 March 2019; Published online: 9 April 2019
*Corresponding author: Zefang Wang, Tel: +86-22-27403906; E-mail:zefangwang@tju.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (31528006 and 81601593), the National Key Basic Research Program of China (973 Program) (2015CB859800 and 2014CB542800) and by the National Key Research Program of China (2016YFD0500300)
Abstract: Polyethylene terephthalate is one of the most used synthetic polymers in industrial fields and daily life due to its excellent durability and plasticity. In the meantime, a large amount of PET waste has caused severe environmental problems, so that researchers are paying more and more attention to the degradability of PET waste. Among all of the methods of plastic degradation, the means of biodegradation is considered to be the most environmental-friendly treatment at present and it will be efficient for large-scale degradation of PET waste. Previous research has indicated that PET degrading enzymes mainly include lipases, esterases and cutinases, but they exhibit poor PET degradation activity. A new PETase discovered recently, which is isolated from Ideonella sakaiensis, has shown high efficacy and specificity to degradation of PET materials. Here, we summarize the progress on structural research of PET degrading enzymes and describe the catalytic mechanism of PETase, to help modify more effective PET degrading enzymes.
Keywords: polyethylene terephthalatePET degrading enzymesstructural researchdegradation mechanism
塑料制品因具有良好的耐热性、电绝缘性、生物安全性等特点,被大量用于纺织制造、包装材料等众多领域中。据研究人员统计,全球每年约有1亿吨塑料制品产生,并呈逐年上升趋势。大部分塑料最终都成为垃圾,造成了严重的环境污染问题,引起了社会的极大关注。因此,如何实现将塑料有效回收降解越来越受到科学家们的重视。
聚对苯二甲酸乙二醇酯(PET),是一种最常见的塑料,是由对苯二甲酸(TPA)和乙二醇(EG)缩聚而成的高分子化合物,通常以无定型和半结晶的形式存在[1]。通常情况下,PET降解的方法主要分为物理法、化学法和生物法[2-4]。物理法首先将回收的PET塑料进行分类,通过机械处理成碎片后,进一步洗涤与干燥处理,再利用加热等工艺进行再次制造成粒。虽然物理回收得到很广泛的应用,但是这种方法往往会发生PET分子链断裂、分子量减少以及无法去除其中的杂质成分等情况,这大大降低了PET的使用性能。化学法主要包括糖解、醇解、水解、氨解等,这些方法都是建立在高温或极端化学试剂的基础上,将PET高聚物进行解聚反应,从而产生部分低聚物和单体,但是这些方法降解成本高、容易产生额外的环境污染物,这大大限制了化学法降解PET的应用。
随着生物技术的发展,生物降解PET具有很大的应用前景。与化学降解法相比,生物法不仅大大降低了降解成本,而且不产生额外的污染物。生物法主要通过微生物作用将PET大分子聚合物降解。由于PET大分子无法进入微生物体内,微生物需要通过分泌一些胞外降解酶,将PET聚合物降解成小分子量的水溶性小分子,这些水溶性小分子再被吸收到微生物体内,通过体内酶的进一步消化,最终水解成水、二氧化碳等物质[5] (图 1)。由此看来,这些胞外降解酶的作用是生物法的限速步骤,只有PET大分子进一步被水解成水溶性小分子后,微生物才能发挥彻底降解作用。
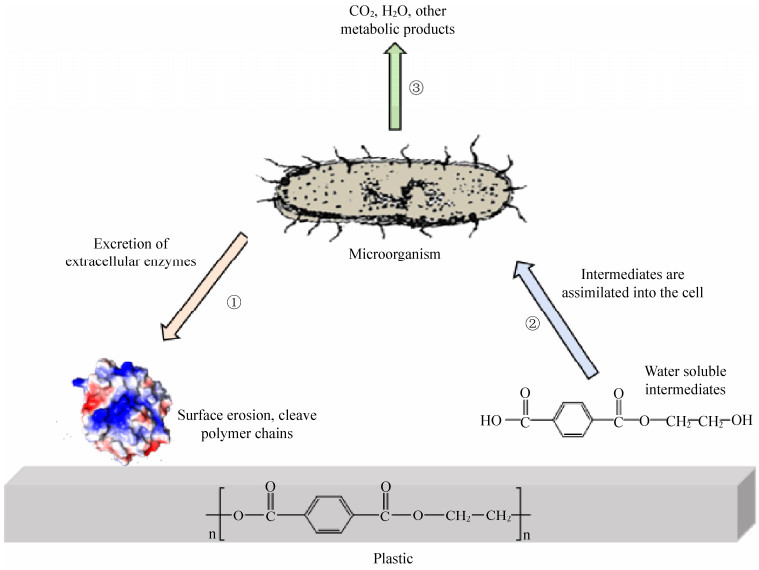 |
| 图 1 PET塑料的生物降解机理[5] Figure 1 Mechanism of PET plastic biodegradation[5]. |
| 图选项 |
研究人员从这些微生物中分离鉴定出在体外发挥降解作用的功能酶,研究发现这些降解酶可以将PET水解成对苯二甲酸乙二醇酯(MHET)、TPA、EG等原料成分。这些降解酶的作用不仅解决了PET塑料的污染问题,而且可以进一步回收原料成分,更加绿色环保,因此微生物酶降解法具有很大的应用前景。到目前为止,研究人员报道了多种具有PET降解能力的微生物酶,但是这些降解酶存在降解活性低、稳定性差等问题,还无法进行大规模应用。本文拟对PET降解酶的最新研究进展进行总结,旨在为改造出更高效的PET降解酶提供一定的参考依据。
1 PET降解酶的发展状况 PET作为合成芳香族聚酯,自然界不存在天然的微生物酶能够特异性降解PET。由于PET内部是由酯键连接而成的,研究人员利用酯酶和脂肪酶来水解PET中的酯键。后来的研究发现,酯酶只能改善PET膜或纤维的表面亲水性[6];而脂肪酶具有“盖子”结构覆盖了酶的催化中心,阻碍了酶与PET底物的有效催化,表现出较低的水解活性[7-8]。于是,研究人员利用环境微生物来降解PET材料。堆肥中因为存在多种微生物能够降解角质、纤维素等物质,逐渐成为研究人员筛选降解菌株的来源。
在过去的研究中,研究人员以PET或其模拟物作为底物,在堆肥中进行菌株筛选,分离出多种具有降解活性的微生物,包括腐皮镰孢菌(Fusarium solani)[9-10]、特异腐质霉(Humicola Insolens)[11]等真菌和嗜热子囊菌(Thermobifida fusca)[12-15]、绿色糖单孢菌(Saccharomonospora viridis)[16]等放线菌。这些菌株虽然具有一定的降解活性,但是降解效率却很低。为了进一步研究PET的生物降解,研究人员尝试从这些降解菌株中分离出具有降解活性的微生物酶。在这些微生物酶中,由于角质酶的催化中心不存在“盖子”结构,有助于酶识别PET聚合物底物,因此角质酶及其同系物相较其他酶表现出较高的降解能力(表 1)。
表 1. 传统PET降解酶的降解能力 Table 1. Application of microbial polyester hydrolases in PET degradation
| Enzyme | Functions | Source | Material | T/℃ | Time | Weight loss/% | Reference |
| HiC | Specific hydrolyze cutins, also hydrolyze C4/C6/C8/C10 pNP-aliphatic esters, aliphatic-aromatic copolyesters (PET、PCL (Poly(ε-caprolactone)) | Humicola Insolens | Low- Crystallinity (7%) PET films | 70 | 96 h | 97±3 | [11] |
| TfH | Hydrolyze cutins, C2 pNP-aliphatic esters, aliphatic-aromatic copolyesters (PET, dibutyl adipate) | Thermobifida fusa DSM43793 | Melt pressing of a beverage bottle | 55 | 3 weeks | ≈50 | [13] |
| TfH | Thermobifida fusa DSM43793 | Low crystalline PET film | 65 | 48 h | 12.9±1.2 | [15] | |
| TfCut2 | Hydrolyze cutins, C2/ C4 pNP-aliphatic esters、aliphatic-aromatic copolyesters (PET) | Thermobifida fusa KW3 | Low crystalline PET film | 65 | 48 h | 12.6±0.2 | [15] |
| Variants of TfCut2 | Thermobifida fusa KW3 | Low crystalline PET film | 70 | 48 h | 25.0±0.8 | [14] | |
| LC-cutinase (LCC) | Hydrolyze cutins, C2/C4/C6/C8/C12/C14/C16pNP-aliphatic esters, aliphatic-aromatic copolyesters (PET, PCL) | Metagenome from leaf-branch compost | PET fims | 50–70 | 24 h | ≈22 | [17] |
| Variants of Cut190 | Hydrolyze cutins, C2/C4/C6/C8/C10 pNP-aliphatic esters, aliphatic-aromatic copolyesters (PET, PBSA) | Saccharomonospora viridis AHK190 | PET-GF films | 63 | 3 days | 13.5±0.5 | [18] |
| PET-S films | 63 | 3 days | 27.0±1.0 | [18] |
表选项
来源于Thermobifida fusa DSM43793菌株的TfH可以催化角质、不溶性甘油酸酯和可溶性酯的水解,对短链脂肪酸具有更高的水解活性[19-20]。Müller等[13]发现TfH能够在55 ℃的条件下降解10%结晶度的PET膜,然而这种降解反应时间长达3周且需要每周更换酶溶液,最终使PET膜的重量损失了50%。Then等[15]通过向TfH中添加Ca2+、Mg2+能够提高酶的热稳定性,使酶能够在65 ℃反应48 h,把低结晶度PET膜降解约13%。来自Thermobifida fusa KW3菌株的TfCut2角质酶与TfH角质酶的序列相似性达到90%以上,研究发现TfCut2能够降解角质、短链脂肪酸,并对PET表现出一定的降解活性[21-22]。随后通过氨基酸突变实验来提高TfCut2的热稳定性,使酶能够在70 ℃降解低结晶度PET膜,一定程度上提高了酶的降解活性[14]。
Fusako等[18]从Saccharomonospora viridis AHK190菌株中分离纯化出具有降解活性的Cut190角质酶,实验结果表明通过氨基酸突变实验提高了酶的热稳定性,该酶在63 ℃反应3 d使低结晶度PET膜减少约27%,相对于野生型酶,突变型酶的活性提高了一倍。来源于特异腐质霉(Humicola insolens)的HiC角质酶是迄今为止酶活性最高且具有热稳定性的真菌聚酯水解酶,HiC能够特异性水解角质,并对长链脂肪酸表现出较高的催化活性[23]。后来的研究发现,HiC可以在70 ℃下反应96 h后,几乎完全水解低结晶(7%) PET膜[11]。
虽然这些传统PET降解酶对无定型态或结晶度低的PET材料具有较高的降解能力,然而却无法降解高结晶度的PET材料[24],其原因可能是大多数的PET降解酶对其不具有底物专一性,没有合适的酶催化中心来容纳高结晶度的PET长链分子[25]。针对这些传统PET降解酶的研究现状,研究人员通过提高降解温度来提高PET分子长链的摆动性,从而增加PET大分子长链和酶活中心配对的概率来提高降解效率[26]。虽然目前已经有耐高温的PET降解酶被筛选或者改造出来,但通过这种方法来提高反应酶活性的效果也十分有限[27]。而且这种通过增加降解温度来提高酶降解效率的方式需要环境升温的措施不利于酶的实际应用。更多的研究是通过改造降解酶的催化中心以促进酶与PET底物的结合[28-29],如通过扩大底物结合口袋有利于PET大分子进入酶的催化中心,增加底物结合口袋的疏水性有利于酶与疏水性PET的吸附结合。虽然研究人员对这些PET降解酶进行了改造研究,但是这些酶仍存在降解活性低的问题,仅有法国Carbios公司报道了关于酶法工艺技术将PET完全降解成TPA和EG原料成分的应用[30]。
值得注意的是,日本团队近期发现一种能“吃”PET塑料的超级细菌Ideonella sakaiensis 201-F6,该菌株能够在30 ℃的条件下反应6周后,使低结晶度PET膜完全降解。随后研究人员从该菌株鉴定出在体外发挥降解作用的水解酶,并命名为PETase[31]。研究表明,PETase与低结晶度PET膜在30 ℃的条件下反应18 h后,检测产物几乎为MHET,只有少量的TPA和对苯二甲酸乙二醇二酯(BHET)产物。研究人员将这种菌与传统PET降解酶相比,PETase显示出以下特征:(1) PETase对PET具有较高的特异性,不降解其他聚酯类塑料;(2) PETase对低结晶度的PET膜的降解效率是传统PET降解酶效率的100倍左右;(3) PETase对高结晶度的PET塑料瓶的降解效率是传统PET降解酶效率的20倍左右;(4) PETase能够在室温及pH为7.0的生理条件下发挥降解作用,与传统PET降解酶在高温条件下才能发挥降解作用形成了鲜明的对比。这一发现,也让研究人员看到了生物酶法降解PET的远大前景。然而,虽然PETase已表现出较高的PET降解能力,但如果想真正实现工业化应用,还需要进一步提高它的酶活性。
2 PET降解酶的机制研究 PET降解酶首先需要吸附到PET表面才能发挥降解功能[5, 32]。然而,PET聚合物具有很强的疏水性,不利于降解酶与PET聚合物的有效吸附[9]。研究人员发现,PET降解酶自身并不存在负责底物吸附的特异性结合域,降解酶对PET表面的初始吸附可能只是由催化中心附近的疏水区域介导的[22, 33-34]。关于PET降解酶是如何发挥降解作用的分子机制仍不清晰。研究人员尝试借助结构生物学手段来研究降解酶与PET复合物的结合状态,进而阐释PET降解酶发挥催化作用的分子机制。
Roth等[35]最初解析了TfCut2的晶体三维结构(图 2-A),该酶采用典型α/β水解酶折叠方式,结构中心由9个平行的β折叠组成,多个α螺旋分布在β折叠的两侧,保守的催化三联体(Ser130-Asp176-His208)位于酶表面的凹槽中。TfCut2与抑制剂苯甲基磺酰氟(PMSF)的复合物晶体结构表明[35],Met131和Tyr60的主链氮原子与聚酯底物的羰基O可以形成氧负离子洞,Ile178、Tyr60为底物提供疏水作用力。基于TfCut2与PMSF的结合方式,研究人员将2PET共价对接到酶的催化中心,发现2PET无法在构象不改变的情况下进入酶的活性中心,这说明了TfCut2对PET底物不具有特异性。TfCut2在较高的温度下降解PET,是由于PET分子链发生一定的构象改变,进入酶的活性中心,催化三联体进而发挥水解作用。这也是传统PET降解酶无法降解高结晶度的PET材料的原因。
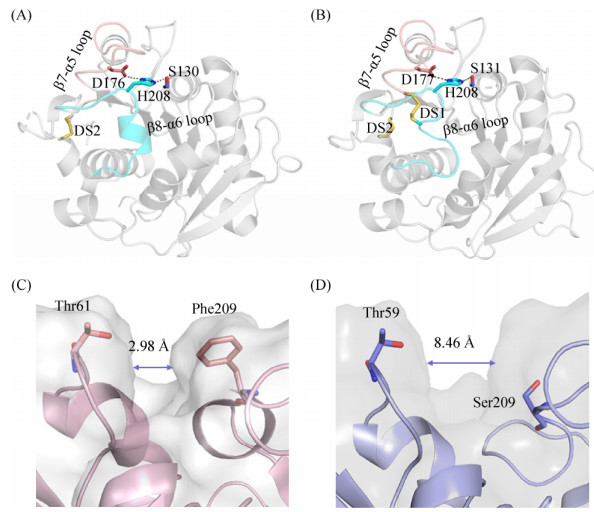 |
| 图 2 PET降解酶的三维结构[35-37] Figure 2 Overall structures of PET-degrading enzymes[35-37]. A: Cartoon representation of the TfCut2 structure (PDB ID: 4CG1); B: Comparative structure of the PETase (PDB ID: 5XG0); C: Narrower cleft of the TfCut2 is shown between Thr61 and Phe209; D: Wide cleft of the TfCut2 between Thr59 and Ser209 in equivalent positions. |
| 图选项 |
随后,由于特异性较高的PETase被发现,多个研究团队展开了对该酶的结构研究,并获得了PETase及其复合物的三维结构[36-40] (图 2-B)。PETase的晶体结构显示,也呈现出典型α/β水解酶折叠方式,但是它和TfCut2的结构仍存在一定差异:(1) PETase分子内部形成两个分子内二硫键(DS1和DS2),而TfCut2只有一对二硫键(图 2-A、2-B)。二者保守的DS2连接C末端螺旋的最后一个Loop环,由于DS2远离酶活中心,只影响酶的热稳定性。而PETase中Cys174和Cys210形成一对位于活性口袋附近额外的二硫键DS1,连接着含有催化酸的β7-α5 loop与含有催化碱的β8-α6 loop,为活性口袋提供了一定的刚性。(2) PETase中的β8-α6 loop环比其他降解酶多3个残基(图 2-A、2-B),这个延伸的loop环延伸底物结合口袋的裂缝,增加大分子底物与酶的结合空间,因此可以说这个延伸的loop环也可能会产生一个结合PET的亚位点。(3) PETase活性口袋附近209位是较小的Ser残基,而TfCut2、LCC对应位置的氨基酸为较大Phe残基。其中PETase中Thr59和Ser209形成的口袋宽度是8.46 ?,而TfCut2中Thr61和Phe209形成口袋宽度是2.98 ?,PETase更宽的口袋更有利于PET大分子进入催化中心[37] (图 2-C、2-D)。(4) PETase的W156出现了摆动现象。Han等[36]指出,PETase第185位的Ser取代了传统PET降解酶中的His,这个较小的丝氨酸残基,允许Trp156存在多种构象,而传统PET降解酶由于组氨酸和色氨酸侧链的堆积使Trp156只呈现一种构象。PETase的构象变化增大了酶的底物结合口袋,容许PET大分子进入酶的活性中心。这些区别解释了PETase比传统PET酶的底物特异性更高的原因。
另外,Han等在解析PETase与底物类似物对苯二甲酸单酯(HEMT)、产物类似物对硝基苯酚(pNP)的复合物结构的基础上(图 3-A、3-B),推测出了PETase水解PET底物可能的催化机制[36]。当PETase与PET底物结合时,靠近第一个苯环的羰基被引向酶的催化中心,Ser131羟基对酯键羰基C发生亲核进攻,形成瞬时酰基-酶中间体复合物,其中Met132和Tyr158与酯键羰基O形成氧洞结构从而稳定酰基-酶中间体复合物,Trp156的吲哚环向PET的TPA部分提供一定的T堆积力;接着,水分子对形成的酰基-酶中间体复合物进行第二次亲核攻击,致使酯键断裂;最后,剩余的苯甲酸基团形成较宽的平面,容易与Trp156的吲哚环形成更强的π堆积相互作用。因此,产物被旋转拉开,最终从活性中心释放。Joo等[37]在解析PETase三维结构的基础上,利用共价对接计算方法2-HE(MHET)4对接到PETase的催化中心来推测酶与底物的结合方式,以及可能的PETase的水解机制,和Han等推测的催化机制是完全吻合的。
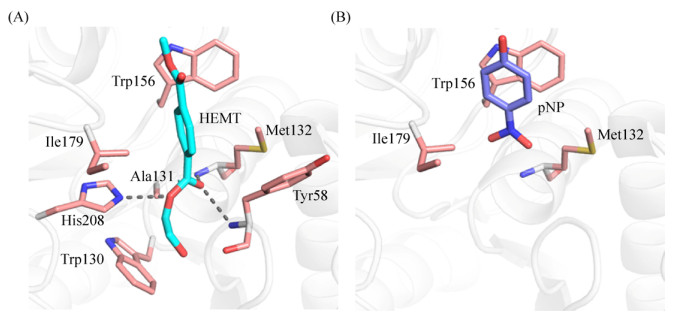 |
| 图 3 PETase的复合物结构[36] Figure 3 Complex structure and catalytic reaction of PETase[36]. Active site interaction networks of PETase are presented for the complexes with (A) HEMT (PDB ID: 5XH3) and (B) pNP (PDB ID: 5XH2). |
| 图选项 |
3 PET降解酶的工程改造 自然界存在的PET降解酶往往具有稳定性差、活性不高、表达量低等现象,这限制了PET降解酶的大规模工业应用。为了进一步提高PET降解酶的效率,研究人员尝试利用理性设计和定向诱变等蛋白质工程方法,对PET降解酶进行改造来增强酶活性、特异性和稳定性等。
3.1 改造酶底物结合口袋 酶与底物的充分接触是实现有效水解的重要因素[32]。来源于腐皮镰孢菌(Fusarium solani)的FsC角质酶能够水解角质和短链脂肪族聚酯,后来研究人员发现该酶对PET表现出较低的水解活性[10]。基于PET降解酶的三维结构分析,Araujo等[9]首次利用较小的丙氨酸代替较大氨基酸的方式,对FsC角质酶的活性中心附近氨基酸进行定向突变以扩大底物的结合口袋。相对于野生型角质酶,突变体Leu182Ala和Leu81Ala对PET纤维的水解活性分别提高4倍和5倍,这一定程度上改善了酶的活性。Silva等[41]对来源于嗜热子囊菌(Thermobifida fusa WSH03-11)的Tfu_0883角质酶的底物结合口袋进行改造,通过设计双突变Gln132Ala/Thr101Ala来增加口袋的疏水性,双突变体Gln132Ala/Thr101Ala对PET纤维的降解活性提高1.6倍,说明增加酶底物结合口袋的疏水性有助于提高酶的活性。值得一提的是,本人所在课题组在解析了PETase三维结构的基础上,对酶底物口袋进行定点突变改造,获得的突变体对PET的降解活性增加了2.3倍[42]。这些研究均表明,通过增加角质酶底物结合口袋的疏水性和增大底物口袋的结合空间,在一定程度上能够提高酶的催化能力。
3.2 增强酶稳定性 有些PET需要在高温下进行降解,当反应温度接近PET的玻璃化转变温度(约70 ℃)时,PET的无定型区更容易被酶攻击进而降解[11, 43],因此增强酶的热稳定性也是非常必要的。研究人员发现通过向来源于细菌的聚酯水解酶中添加Ca2+、Mg2+等二价金属离子,可以增强酶的热稳定性,使酶能够在70 ℃具有较高的水解活性[15, 18, 44]。Then等[14]对来自嗜热子囊菌(Thermobifida fusca KW3)的聚酯水解酶TfCut2突变体改造的基础上,通过增加酶中的盐桥或二硫桥来提高酶的热稳定性,使酶能够在70 ℃有效地降解无定形PET膜。这些研究表明,通过增加酶的热稳定性,能够使酶在较高温度下降解PET,这一定程度上提高了酶对PET的降解能力。
除此以外,由于PET降解酶表面大多是亲水性基团,只有少数疏水性氨基酸暴露在蛋白的表面,因此亲水性的PET降解酶无法有效地低吸附到疏水性PET的表面。在PET降解酶与疏水性PET表面靠近时,强烈的疏水作用力可能引起酶的构象变化而导致降解酶的活性降低甚至完全丧失。研究人员发现可以通过蛋白质工程的手段将更多的疏水性氨基酸残基暴露在PET降解酶催化中心旁边来增加酶的吸附,也有研究将PET降解酶进行截短,来增加暴露在蛋白表面的疏水氨基酸残基来提高酶的吸附[45-46]。这些方法虽然提高了PET酶吸附能力,但大多数情况下也降低PET酶活性。后来研究发现,疏水蛋白可以帮助PET吸附到PET表面,通过构建疏水蛋白与PET降解酶的融合表达载体,可以大大提高PET降解酶的降解能力[47-48]。
3.3 减少酶的产物抑制 PET降解酶在降解PET的过程中释放出对苯二甲酸双酯(BHET)、MHET、TPA和EG等物质[49-50]。有研究发现,MHET会抑制TfCut2降解酶的活性[51]。TfCut2对MHET有较高亲和力,使MHET竞争性结合TfCut2,这在一定程度上限制了TfCut2对PET的降解活性。Barth等[52]利用超滤膜反应器有效地降低了MHET等中间产物的抑制作用。另外,研究人员利用来源于南极假丝酵母(Candida antarctica)的脂肪酶CalB来降解MHET,与HiC组成的双酶体系,使酶的降解能力提高了7.7倍[53]。Wei等[29]研究TfCut2的底物口袋的氨基酸,设计的G62A突变体,使TfCut2对MHET的结合力减少了5.5倍,从而使酶对PET的降解能力增加了2.7倍。
3.4 增加降解酶与PET的有效接触面积 水溶性PET降解酶无法渗透到疏水性PET聚合物中,而是先附着在PET聚合物表面再发挥降解作用,因此可通过增加降解酶与PET聚合物的可接触面积来提高酶的降解效率。Wei等[32]将PET材料处理成直径为100 nm的PET颗粒,增加了TfCut2与PET颗粒的吸附能力,从而显著提高了TfCut2的降解效率。来源于嗜热纤维菌(Thermobifida cellulosilytica)的Thc_cut1角质酶能够降解角质、PET等物质[22],Pellis等[54]利用Thc_cut1角质酶来降解PET粉末和PET薄膜。研究表明,在相同的反应条件下Thc_cut1角质酶对PET粉末表现出更高的降解活性。随后,Gamerith等[55]将PET饮料瓶处理成直径为0.25–0.5 mm的PET颗粒,大大增加了Thc_cut1对PET颗粒的降解效率。这些研究表明,通过增加降解酶与PET底物的有效接触面积,一定程度上提高酶的降解效率。
4 展望 目前为止,虽然发现了多种降解酶对PET有一定的降解能力,但是这些降解酶实际应用到工业降解仍需要极大地提高酶活力。研究人员尝试增加酶的降解活性和增加酶的吸附能力,但是这些研究方法都是在单独研究降解酶吸附或酶解过程,并没有将吸附和酶解过程有机地统一起来。本实验室最近的实验结果表明疏水蛋白的自组装功能可以控制PETase在PET表面的吸附行为,同时利用大肠杆菌和毕赤酵母表面共展示HFBI-PETase融合酶,可极大提高酶的催化效率[56-57]。这一研究为今后发展改造更有效的PET降解酶提供了新的思路和方法。
References
| [1] | Marten E, Müller RJ, Deckwer WD. Studies on the enzymatic hydrolysis of polyesters. Ⅱ. Aliphatic-aromatic copolyesters. Polymer Degradation and Stability, 2005, 88(3): 371-381. DOI:10.1016/j.polymdegradstab.2004.12.001 |
| [2] | 杨融.废弃PET瓶的循环使用研究及其在聚氨酯中的应用.西安理工大学硕士学位论文, 2018. http://cdmd.cnki.com.cn/Article/CDMD-10700-1018835608.htm |
| [3] | Singh N, Hui D, Singh R, Ahuja IPS, Feo L, Fraternali F. Recycling of plastic solid waste: a state of art review and future applications. Composites Part B: Engineering, 2017, 115: 409-422. DOI:10.1016/j.compositesb.2016.09.013 |
| [4] | Wei R, Zimmermann W. Biocatalysis as a green route for recycling the recalcitrant plastic polyethylene terephthalate. Microbial Biotechnology, 2017, 10(6): 1302-1307. DOI:10.1111/1751-7915.12714 |
| [5] | Mueller RJ. Biological degradation of synthetic polyesters—Enzymes as potential catalysts for polyester recycling. Process Biochemistry, 2006, 41(10): 2124-2128. DOI:10.1016/j.procbio.2006.05.018 |
| [6] | Alisch M, Feuerhack A, Müller H, Mensak B, Andreaus J, Zimmermann W. Biocatalytic modification of polyethylene terephthalate fibres by esterases from actinomycete isolates. Biocatalysis and Biotransformation, 2004, 22(5/6): 347-351. |
| [7] | Guebitz GM, Cavaco-Paulo A. Enzymes go big: surface hydrolysis and functionalisation of synthetic polymers. Trends in Biotechnology, 2008, 26(1): 32-38. |
| [8] | Eberl A, Heumann S, Brückner T, Araujo R, Cavaco-Paulo A, Kaufmann F, Kroutil W, Guebitz GM. Enzymatic surface hydrolysis of poly (ethylene terephthalate) and bis (benzoyloxyethyl) terephthalate by lipase and cutinase in the presence of surface active molecules. Journal of Biotechnology, 2009, 143(3): 207-212. |
| [9] | Araújo R, Silva C, O'Neill A, Micaelo N, Guebitz G, Soares CM, Casal M, Cavaco-Paulo A. Tailoring cutinase activity towards polyethylene terephthalate and polyamide 6, 6 fibers. Journal of Biotechnology, 2007, 128(4): 849-857. |
| [10] | Nimchua T, Eveleigh DE, Sangwatanaroj U, Punnapayak H. Screening of tropical fungi producing polyethylene terephthalate-hydrolyzing enzyme for fabric modification. Journal of Industrial Microbiology & Biotechnology, 2008, 35(8): 843-850. |
| [11] | Ronkvist ?M, Xie WC, Lu WH, Gross RA. Cutinase-catalyzed hydrolysis of poly (ethylene terephthalate). Macromolecules, 2009, 42(14): 5128-5138. DOI:10.1021/ma9005318 |
| [12] | Kleeberg I, Hetz C, Kroppenstedt RM, Müller RJ, Deckwer WD. Biodegradation of aliphatic-aromatic copolyesters by Thermomonospora fusca and other thermophilic compost isolates. Applied and Environmental Microbiology, 1998, 64(5): 1731-1735. |
| [13] | Müller RJ, Schrader H, Profe J, Dresler K, Deckwer WD. Enzymatic degradation of poly (ethylene terephthalate): rapid hydrolyse using a hydrolase from T. fusca. Macromolecular Rapid Communications, 2005, 26(17): 1400-1405. DOI:10.1002/marc.200500410 |
| [14] | Then J, Wei R, Oeser T, Gerdts A, Schmidt J, Barth M, Zimmermann W. A disulfide bridge in the calcium binding site of a polyester hydrolase increases its thermal stability and activity against polyethylene terephthalate. FEBS Open Bio, 2016, 6(5): 425-432. DOI:10.1002/2211-5463.12053 |
| [15] | Then J, Wei R, Oeser T, Barth M, Belisário-Ferrari MR, Schmidt J, Zimmermann W. Ca2+ and Mg2+ binding site engineering increases the degradation of polyethylene terephthalate films by polyester hydrolases from Thermobifida fusca. Biotechnology Journal, 2015, 10(4): 592-598. |
| [16] | Hu XP, Osaki S, Hayashi M, Kaku M, Katuen S, Kobayashi H, Kawai F. Degradation of a terephthalate-containing polyester by thermophilic actinomycetes and Bacillus species derived from composts. Journal of Polymers and the Environment, 2008, 16(2): 103-108. DOI:10.1007/s10924-008-0088-5 |
| [17] | Sulaiman S, You DJ, Kanaya E, Koga Y, Kanaya S. Crystal structure and thermodynamic and kinetic stability of metagenome-derived LC-cutinase. Biochemistry, 2014, 53(11): 1858-1869. DOI:10.1021/bi401561p |
| [18] | Kawai F, Oda M, Tamashiro T, Waku T, Tanaka N, Yamamoto M, Mizushima H, Miyakawa T, Tanokura M. A novel Ca2+-activated, thermostabilized polyesterase capable of hydrolyzing polyethylene terephthalate from Saccharomonospora viridis AHK190. Applied Microbiology and Biotechnology, 2014, 98(24): 10053-10064. DOI:10.1007/s00253-014-5860-y |
| [19] | Gouda MK, Kleeberg I, van den Heuvel J, Müller RJ, Deckwer WD. Production of a polyester degrading extracellular hydrolase from Thermomonospora fusca. Biotechnology Progress, 2002, 18(5): 927-934. DOI:10.1021/bp020048b |
| [20] | Kleeberg I, Welzel K, VandenHeuvel J, Müller RJ, Deckwer WD. Characterization of a new extracellular hydrolase from Thermobifida fusca degrading aliphatic-aromatic copolyesters. Biomacromolecules, 2005, 6(1): 262-270. DOI:10.1021/bm049582t |
| [21] | Chen S, Su LQ, Billig S, Zimmermann W, Chen J, Wu J. Biochemical characterization of the cutinases from Thermobifida fusca. Journal of Molecular Catalysis B: Enzymatic, 2010, 63(3/4): 121-127. |
| [22] | Acero EH, Ribitsch D, Steinkellner G, Gruber K, Greimel K, Eiteljoerg I, Trotscha E, Wei R, Zimmermann W, Zinn M, Cavaco-Paulo A, Freddi G, Schwab H, Guebitz G. Enzymatic surface hydrolysis of PET: effect of structural diversity on kinetic properties of cutinases from Thermobifida. Macromolecules, 2011, 44(12): 4632-4640. DOI:10.1021/ma200949p |
| [23] | Feder D. Humicola insolens cutinase; a novel catalyst for polymer synthesis reactions. Doctor Dissertation of Polytechnic Institute of New York University, 2013. |
| [24] | Wei R, Zimmermann W. Microbial enzymes for the recycling of recalcitrant petroleum-based plastics: how far are we?. Microbial Biotechnology, 2017, 10(6): 1308-1322. DOI:10.1111/1751-7915.12710 |
| [25] | Narancic T, O'Connor KE. Microbial biotechnology addressing the plastic waste disaster. Microbial Biotechnology, 2017, 10(5): 1232-1235. DOI:10.1111/1751-7915.12775 |
| [26] | Lu XB, Liu Y, Zhou H. Learning nature: recyclable monomers and polymers. Chemistry-A European Journal, 2018, 24(44): 11255-11266. DOI:10.1002/chem.201704461 |
| [27] | Gamerith C, Vastano M, Ghorbanpour SM, Zitzenbacher S, Ribitsch D, Zumstein MT, Sander M, Acero EH, Pellis A, Guebitz GM. Enzymatic degradation of aromatic and aliphatic polyesters by P. pastoris expressed cutinase 1 from Thermobifida cellulosilytica. Frontiers in Microbiology, 2017, 8: 938. DOI:10.3389/fmicb.2017.00938 |
| [28] | Pellis A, Haernvall K, Pichler CM, Ghazaryan G, Breinbauer R, Guebitz GM. Enzymatic hydrolysis of poly (ethylene furanoate). Journal of Biotechnology, 2016, 235: 47-53. DOI:10.1016/j.jbiotec.2016.02.006 |
| [29] | Wei R, Oeser T, Schmidt J, Meier R, Barth M, Then J, Zimmermann W. Engineered bacterial polyester hydrolases efficiently degrade polyethylene terephthalate due to relieved product inhibition. Biotechnology and Bioengineering, 2016, 113(8): 1658-1665. DOI:10.1002/bit.25941 |
| [30] | Qian BZ. Carbios develops bioenzymatic recycling of PET technology. China Synthetic Fiber Industry, 2016, 39(2): 38. (in Chinese) 钱伯章. Carbios开发生物酶法循环回收PET技术. 合成纤维工业, 2016, 39(2): 38. |
| [31] | Yoshida S, Hiraga K, Takehana T, Taniguchi I, Yamaji H, Maeda Y, Toyohara K, Miyamoto K, Kimura Y, Oda K. A bacterium that degrades and assimilates poly(ethylene terephthalate). Science, 2016, 351(6278): 1196-1199. DOI:10.1126/science.aad6359 |
| [32] | Wei R, Oeser T, Barth M, Weigl N, Lübs A, Schulz-Siegmund M, Hacker M, Zimmermann W. Turbidimetric analysis of the enzymatic hydrolysis of polyethylene terephthalate nanoparticles. Journal of Molecular Catalysis B: Enzymatic, 2014, 103: 72-78. DOI:10.1016/j.molcatb.2013.08.010 |
| [33] | Chen S, Su LQ, Chen J, Wu J. Cutinase: characteristics, preparation, and application. Biotechnology Advances, 2013, 31(8): 1754-1767. DOI:10.1016/j.biotechadv.2013.09.005 |
| [34] | Wei R, Oeser T, Zimmermann W. Synthetic polyester-hydrolyzing enzymes from thermophilic actinomycetes. Advances in Applied Microbiology, 2014, 89: 267-305. DOI:10.1016/B978-0-12-800259-9.00007-X |
| [35] | Roth C, Wei R, Oeser T, Then J, Follner C, Zimmermann W, Str ter N. Structural and functional studies on a thermostable polyethylene terephthalate degrading hydrolase from Thermobifida fusca. Applied Microbiology and Biotechnology, 2014, 98(18): 7815-7823. DOI:10.1007/s00253-014-5672-0 |
| [36] | Han X, Liu WD, Huang JW, Ma JT, Zheng YY, Ko TP, Xu LM, Cheng YS, Chen CC, Guo RT. Structural insight into catalytic mechanism of PET hydrolase. Nature Communications, 2017, 8(1): 2106. DOI:10.1038/s41467-017-02255-z |
| [37] | Joo S, Cho IJ, Seo H, Son HF, Sagong HY, Shin TJ, Choi SY, Lee SY, Kim KJ. Structural insight into molecular mechanism of poly (ethylene terephthalate) degradation. Nature Communications, 2018, 9(1): 382. DOI:10.1038/s41467-018-02881-1 |
| [38] | Austin HP, Allen MD, Donohoe BS, Rorrer NA, Kearns FL, Silveira RL, Pollard BC, Dominick G, Duman R, El Omari K, Mykhaylyk V, Wagner A, Michener WE, Amore A, Skaf MS, Crowley MF, Thorne AW, Johnson CW, Lee Woodcock H, McGeehan JE, Beckham GT. Characterization and engineering of a plastic-degrading aromatic polyesterase. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(19): E4350-E4357. DOI:10.1073/pnas.1718804115 |
| [39] | Danso D, Schmeisser C, Chow J, Zimmermann W, Wei R, Leggewie C, Li XZ, Hazen T, Streit WR. New insights into the function and global distribution of polyethylene terephthalate (PET)-degrading bacteria and enzymes in marine and terrestrial metagenomes. Applied and Environmental Microbiology, 2018, 84(8): e02773-17. |
| [40] | Fecker T, Galaz-Davison P, Engelberger F, Narui Y, Sotomayor M, Parra LP, Ramirez-Sarmiento CA. Active site flexibility as a hallmark for efficient PET degradation by I. sakaiensis PETase. Biophysical Journal, 2018, 114(6): 1302-1312. DOI:10.1016/j.bpj.2018.02.005 |
| [41] | Silva C, Da S, Silva N, Matamá T, Araújo R, Martins M, Chen S, Chen J, Wu J, Casal M, Cavaco-Paulo A. Engineered Thermobifida fusca cutinase with increased activity on polyester substrates. Biotechnology Journal, 2011, 6(10): 1230-1239. DOI:10.1002/biot.201000391 |
| [42] | 王泽方, 杨海涛, 段胤凯, 蒋林芮, 陈卓芝, 牟钟毓, 刘曼琳, 胡睿行. PET分解酶突变体、编码基因及其应用.中国: CN201610912532.1. 2016-10-20. |
| [43] | Alves NM, Mano JF, Balaguer E, Meseguer Due?as JM, Gómez Ribelles JG. Glass transition and structural relaxation in semi-crystalline poly (ethylene terephthalate): a DSC study. Polymer, 2002, 43(15): 4111-4122. DOI:10.1016/S0032-3861(02)00236-7 |
| [44] | Thumarat U, Nakamura R, Kawabata T, Suzuki H, Kawai F. Biochemical and genetic analysis of a cutinase-type polyesterase from a thermophilic Thermobifida alba AHK119. Applied Microbiology and Biotechnology, 2012, 95(2): 419-430. DOI:10.1007/s00253-011-3781-6 |
| [45] | Biundo A, Ribitsch D, Steinkellner G, Gruber K, Guebitz GM. Polyester hydrolysis is enhanced by a truncated esterase: less is more. Biotechnology Journal, 2016, 12(8). DOI:10.1002/biot.201600450 |
| [46] | Kawabata T, Oda M, Kawai F. Mutational analysis of cutinase-like enzyme, Cut190, based on the 3D docking structure with model compounds of polyethylene terephthalate. Journal of Bioscience and Bioengineering, 2017, 124(1): 28-35. |
| [47] | Ribitsch D, Acero EH, Przylucka A, Zitzenbacher S, Marold A, Gamerith C, Tschelie?nig R, Jungbauer A, Rennhofer H, Lichtenegger H, Amenitsch H, Bonazza K, Kubicek CP, Druzhinina IS, Guebitz GM. Enhanced cutinase-catalyzed hydrolysis of polyethylene terephthalate by covalent fusion to hydrophobins. Applied and Environmental Microbiology, 2015, 81(11): 3586-3592. DOI:10.1128/AEM.04111-14 |
| [48] | Ribitsch D, Yebra AO, Zitzenbacher S, Wu J, Nowitsch S, Steinkellner G, Greimel K, Doliska A, Oberdorfer G, Gruber CC, Gruber K, Schwab H, Stana-Kleinschek K, Acero EH, Guebitz GM. Fusion of binding domains to Thermobifida cellulosilytica cutinase to tune sorption characteristics and enhancing PET hydrolysis. Biomacromolecules, 2013, 14(6): 1769-1776. DOI:10.1021/bm400140u |
| [49] | Vertommen MAME, Nierstrasz VA, van der Veer M, Warmoeskerken MMCG. Enzymatic surface modification of poly (ethylene terephthalate). Journal of Biotechnology, 2005, 120(4): 376-386. DOI:10.1016/j.jbiotec.2005.06.015 |
| [50] | Wei R, Oeser T, Billig S, Zimmermann W. A high-throughput assay for enzymatic polyester hydrolysis activity by fluorimetric detection. Biotechnology Journal, 2012, 7(12): 1517-1521. DOI:10.1002/biot.201200119 |
| [51] | Barth M, Oeser T, Wei R, Then J, Schmidt J, Zimmermann W. Effect of hydrolysis products on the enzymatic degradation of polyethylene terephthalate nanoparticles by a polyester hydrolase from Thermobifida fusca. Biochemical Engineering Journal, 2015, 93: 222-228. DOI:10.1016/j.bej.2014.10.012 |
| [52] | Barth M, Wei R, Oeser T, Then J, Schmidt J, Wohlgemuth F, Zimmermann W. Enzymatic hydrolysis of polyethylene terephthalate films in an ultrafiltration membrane reactor. Journal of Membrane Science, 2015, 494: 182-187. DOI:10.1016/j.memsci.2015.07.030 |
| [53] | Carniel A, Valoni é, Junior JN, da Concei??o Gomes A, de Castro AM. Lipase from Candida antarctica (CALB) and cutinase from Humicola insolens act synergistically for PET hydrolysis to terephthalic acid. Process Biochemistry, 2017, 59: 84-90. DOI:10.1016/j.procbio.2016.07.023 |
| [54] | Pellis A, Gamerith C, Ghazaryan G, Ortner A, Acero EH, Guebitz GM. Ultrasound-enhanced enzymatic hydrolysis of poly (ethylene terephthalate). Bioresource Technology, 2016, 218: 1298-1302. DOI:10.1016/j.biortech.2016.07.106 |
| [55] | Gamerith C, Zartl B, Pellis A, Guillamot F, Marty A, Acero EH, Guebitz GM. Enzymatic recovery of polyester building blocks from polymer blends. Process Biochemistry, 2017, 59: 58-64. DOI:10.1016/j.procbio.2017.01.004 |
| [56] | 王泽方, 杨海涛, 王雪, 童善惟, 陈卓芝, 程莹莹, 侯宇佳.细胞表面展示PET分解酶的重组毕赤酵母及构建与应用.中国: CN201610912589.1. 2016-10-20. |
| [57] | 王泽方, 杨海涛, 陈卓芝, 程莹莹, 王雪, 童善惟, 侯宇佳.细胞表面共展示PET分解酶和疏水蛋白的重组毕赤酵母.中国: CN201610912165.5. 2016-10-20. |
