杨燕, 罗林根, 徐妙, 夏立秋


湖南师范大学生命科学学院, 微生物分子生物学国家重点实验室培育基地, 湖南长沙 410081
收稿日期: 2015-06-24; 修回日期: 2015-09-07; 网络出版日期: 2015-09-29
基金项目: 国家"863"计划(2011AA10A203);国家"973"计划(2012CB722301);国家自然科学基金(31070006);湖南省2011协同创新中心项目(20134486);湖南省教育厅项目(10CY013)
通信作者: 夏立秋,Tel: +86-731-88872905;E-mail: xialq@hunnu.edu.cn
摘要: [目的] 构建亮氨酰氨肽酶基因(pepA)被阻断的刺糖多孢菌工程菌株,并鉴定该基因对刺糖多孢菌菌丝形态、生物量、菌体全蛋白表达水平及产多杀菌素能力的影响,探究该基因调控多杀菌素合成的可能机制。[方法] 利用PCR扩增刺糖多孢菌中的pepA基因同源片段,经酶切连接技术构建敲除载体pOJ260-pepA;通过接合转移和单交换同源重组将该载体整合至刺糖多孢菌染色体中,获得工程菌株S. sp-△pepA;利用培养特征、形态学、高效液相色谱、SDS-PAGE等方法对菌株进行研究分析。[结果] 工程菌株S. sp-△pepA菌丝片段化程度加剧,生长态势被延缓且生物量降低,但有效促进了多杀菌素的生物合成。阻断亮氨酰胺肽酶基因的表达使刺糖多孢菌菌体全蛋白表达情况发生明显改变,找到表达水平显著上调的差异蛋白核糖体蛋白亚基和醛基脱氢酶,核糖体蛋白亚基通过影响蛋白质代谢对菌体生长产生影响;醛基脱氢酶则可与乙醇脱氢酶、乙酰辅酶A的合成酶相互作用影响辅酶A合成,而辅酶A是合成多杀菌素的重要底物。[结论] 在刺糖多孢菌合成多杀菌素的次级代谢过程中,pepA基因作为负调控因子发挥作用。
关键词: 刺糖多孢菌亮氨酰氨肽酶基因阻断多杀菌素接合转移
Disruption of leucyl aminopeptidase gene affects phenotypes and second metabolite production of Saccharopolyspora spinosa
Yan Yang, Lingen Luo, Miao Xu,, Liqiu Xia


Hunan Provincial Key Laboratory of Microbial Molecular Biology-State Key Laboratory Breeding Base of Microbial Molecular Biology, College of Life Science, Hunan Normal University, Changsha 410081, Hunan Province, China
Received: 24 June 2015; Revised: 7 September 2015; Published online: 29 September 2015
Fountion Item: Supported by the National High-tech R&D Program (2011AA10A203), the National Basic Research Program of China(2012CB722301), the National Natural Science Foundation of China (31070006), the Project of Hunan Provincial CollaborativeInnovation Center (20134486) and by the Key Project of Hunan Provincial Education Department (10CY013)
Corresponding author. Liqiu Xia,Tel: +86-731-88872905;E-mail: xialq@hunnu.edu.cn
Abstract:[Objective] In order to investigate effects of leucyl aminopeptidase on mycelia morphology, growth rate, spinosad yield and protein expression in Saccharopolyspora spinosa by disrupting its encoding gene pepA and analyzing the characteristics of engineered S. spinosa. [Methods] The pepA gene of S. spinosa was amplified based on the conserved sequence and cloned into Escherichia coli-Streptomyces shuttle vector pOJ260 to generate pOJ260-pepA, which was transformed into S. spinosa by conjugation. Mycelium observation, SDS-PAGE and HPLC were used to analyze the engineered strain. [Results] Mycelia in S. sp-ΔpepA displayed a much higher degree of fragmentation and fewer branches compared to that of parental strain. Meanwhile, the growth rate of S. sp-ΔpepA was retarded and its biomass was reduced. Shake-flask fermentation demonstrated that spinosad yield increased by 122% in S. sp-ΔpepA strain compared to that of parental strain. SDS-PAGE analysis showed that protein expression profile of the engineered strain significantly changed. [Conclusion] The pepA gene negatively regulates the biosynthesis of spinosad and disruption of pepA gene could affect the mycelial morphology and growth of S. spinosa.
Key words: Saccharopolyspora spinosapepA genedisruptionspinosadconjugational transfer
亮氨酰氨肽酶(PepA; EC 3.4.11.1)是氨肽酶M17超家族中的一员,该家族成员均具有从蛋白质、多肽的N端将亮氨酸残基切割下来的作用[1]。该家族氨肽酶在生物界普遍存在,其功能结构的研究仅在牛、大肠杆菌、土豆中开展过[2, 3, 4]。已有研究证明大肠杆菌的PepA具有DNA结合活性,能与多种启动子序列结合[5],对pepA蛋白六聚体X射线三维结构分析发现其具有一个凹槽结构,该结构被认为是DNA结合位点[3]。Song等在天蓝色链霉菌中将亮氨酰氨肽酶基因(pepA)敲除发现缺失突变株产孢子能力和抗生素合成能力明显提高,双向电泳和RT-PCR检测发现ftsZ,ssgA和actII-ORF4的蛋白表达和转录水平也有较大幅度提高,这表明天蓝色链霉菌中的pepA在抗生素合成过程中作为负调控因子发挥作用[6]。
由刺糖多孢菌合成的次级代谢产物多杀菌素具有新颖独特的作用机理,能有效控制鳞翅目Lepidoptera、双翅目Diptera和缨翅目Thysanoptera 等害虫,同时对鞘翅目Coleoptera、直翅目Orthoptera、膜翅目Hymenoptera等某些特定种类的害虫也有一定的毒杀作用[7]。多杀菌素主要靶标是烟碱型乙酰胆碱受体(nAChRs)和γ-氨基丁酸受体(GABARs),但其烟碱受体作用机理不同于现有烟碱类杀虫剂诸如吡虫啉等,与γ-氨基丁酸受体的作用机理亦不同于现有的阿维菌素类、氟虫腈类杀虫剂。它主要通过激活相关害虫中央神经系统的神经细胞而引起非功能性的肌肉收缩和震颤,使中央神经系统广泛超活化以致死亡[8]。多杀菌素独特的杀虫机理表现出生物农药的安全性和化学农药的快速性。野生型刺糖多孢菌的多杀菌素生物合成含量较低、发酵周期很长、生产成本较高是限制多杀菌素在作物生产应用上的主要原因。本研究首次从刺糖多孢菌基因组中扩增了pepA基因中间同源序列,亚克隆到载体pOJ260[9] 中,再通过单交换同源重组将其整合到刺糖多孢菌的染色体上,以期通过阻断负调控基因的表达,影响刺糖多孢菌中次级代谢产物的产生,从而促进多杀菌素的生物合成。
1 材料和方法 1.1 菌株和质粒本研究所用菌株、质粒与引物见表 1。
表 1. 菌株、质粒和引物Table 1. Strains, plasmids and primers used in this study
| Strains/Plasmids/Primers | Relative description | Source |
| Restriction enzyme sites were underlined. | ||
| Strains | ||
| E. coli GB2005 | Host for general cloning | This lab |
| E. coli S17-1 | Donor strain for conjugation | This lab |
| S. spinosa | Spinosad producing strain | This lab |
| S. sp–△pepA | S. spinosa harboring pOJ260-pepA | This study |
| Plasmids | ||
| pMD-18T | E. coli cloning vector,containing AmpR and LacZ | TaKaRa |
| pMD-pepA | pMD-18T harboring partial pepA | This study |
| pOJ260 | E. coli-Streptomyces shuttle expression vector | This lab |
| pOJ260-pepA | pOJ260 harboring partial pepA | This study |
| Primers | Sequence (5'→3') | |
| pepA -F | CCAAGCTTGGCGCCGCGTACGACGGAGACC | This study |
| pepA -R | CCGATATCGG GCCGCCCTTCGGGGTGTAGC | This study |
| AM-F | ATCTACGTCTGTCGAGAAGT | This study |
| AM-R | ATGCCGCTGCCGGAGGAGCT | This study |
| AN-F | GCCGACCACGAAGTCCACCT | This study |
| AN-R | TCTTTATAGTCCTGTCGGGT | This study |
| aac(3)IV-F | GTCCAATACGAATGGCGAAAAGC | This study |
| aac(3)IV-R | ATAACATTCTTCGCATCCCGCC | This study |
表选项
1.2 培养基成分与菌体培养条件大肠杆菌培养采用LB液体或固体培养基(含Tryptone 1%,Yeast extract 0.5%,NaCl 1%,固体培养基则加入1.5%琼脂粉),培养温度为37 ℃;刺糖多孢菌种子活化培养基采用CSM培养基(含TSB 3%,Yeast extract 0.3%,MgSO4·7H2O 0.2%,Glucose 0.5%,Maltose 0.4%),转接采用胰蛋白酶大豆肉汤培养基(含TSB 3%)[10],刺糖多孢菌形态观察采用BHI (BHI 3.7%,琼脂粉1.3%)、R6 (含蔗糖 20%,Dextrin 1%,Casamino acids 0.1%,K2SO4 0.01%,FeSO4·7H2O 0.01%,MnCl2·4H2O 0.01‰,MgSO4·7H2O 0.05‰,ZnSO4·7H2O 0.01‰,BHI 2.6%,MOPS 0.01 mol/L,CaCl2 0.048 mol/L,L-谷氨酸 0.065 mol/L,琼脂粉2%)和ISP-2 (含Glucose 0.04%,Maltose 1%,Yeast extract 0.4%,琼脂粉2%)固体培养基,培养温度为30 ℃。
1.3 工具酶、试剂、抗生素及使用浓度LA Taq DNA 聚合酶、Primer Star DNA聚合酶、限制性内切酶、T4 DNA连接酶等购自大连宝生物工程有限公司。细菌基因组DNA提取试剂盒、DNA片段和PCR产物回收试剂盒、质粒小量提取试剂盒购自北京Bioteke公司。PCR引物合成及测序由生工公司完成。抗生素均购自Sigma公司,工作浓度分别为:阿伯拉霉素(Apramycin),用于大肠杆菌的筛选浓度为20 mg/L,用于刺糖多孢菌的筛选浓度为50 mg/L;氨苄青霉(ampicillin,Amp)用于固体培养基的浓度为50 mg/L,用于液体培养基的浓度为100 mg/L;萘啶酮酸(nalidixic acid,NA) 25 mg/L;链霉素(streptomycin,Str) 100 mg/L;三甲氧苄氨嘧啶(trimethoprim,TMP) 100 mg/L。
1.4 pepA同源基因的确定以天蓝色链霉菌中pepA基因核苷酸序列为标准,利用BLAST在线搜索刺糖多孢菌中该基因同源片段,而后利用Clustal X比对2个基因编码蛋白pepA的相似性[11]。
1.5 敲除载体pOJ260-pepA的构建1.5.1 pepA基因片段的扩增及回收测序:利用细菌基因组DNA提取试剂盒按使用说明书操作提取刺糖多孢菌基因组,以该基因组为模板,通过Primer Star DNA聚合酶扩增约1400 bp的pepA基因片段。反应体系20 μL:DNA模板(313 ng/μL) 1 μL,Primer Star DNA酶(2.5 U/μL) 0.1 μL,2×GC buffer 10 μL,dNTPs (10 mmol/L each) 2 μL,上、下游引物(10 μmol/L,表 1)各0.5 μL,最后加灭菌ddH2O补足;PCR反应程序如下:94 ℃ 5 min;94 ℃ 30 s,61 ℃ 30 s,72 ℃ 105 s,30个循环;72 ℃ 10 min,20 ℃终止PCR反应。该PCR产物经DNA片段和PCR产物回收试剂盒按使用说明书操作回收纯化pepA基因片段后,与pMD-18T载体连接;热转化大肠杆菌[12],用LB固体培养基过夜培养后,挑取单克隆在LB液体培养中扩大培养后利用质粒小量提取试剂盒提质粒;所得质粒经Hind Ⅲ和EcoR Ⅴ酶切鉴定后送上海生工测序,测序结果经NCBI BLAST Network进行比对分析。
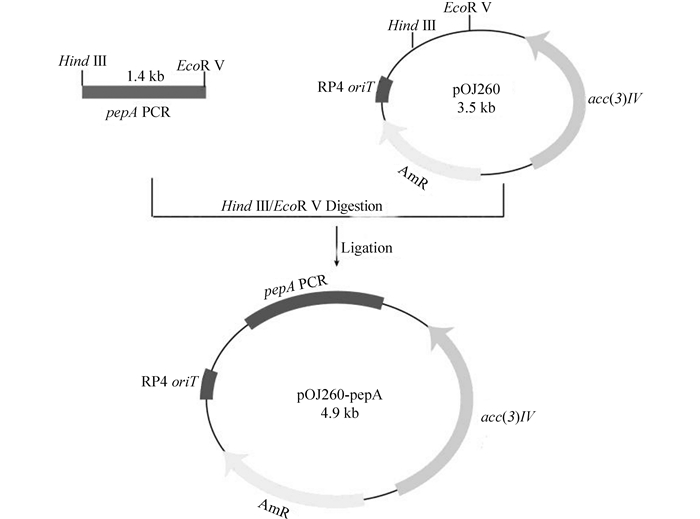
1.5.2 pOJ260-pepA的构建:测序正确后的pMD-pepA质粒经Hind Ⅲ和EcoR Ⅴ酶切后回收pepA基因片段,而后用T4 DNA连接酶将该回收片段与经过同样双酶切的pOJ260载体片段过夜连接[12],得到敲除载体pOJ260-pepA(图 1)。
 |
| 图 1. pepA基因编码蛋白同源性比对 Figure 1. Homology analysis of amino acid residue sequence coded by pepA. |
| 图选项 |
1.6 工程菌株S. sp-△pepA的构建及pOJ260-pepA插入位点的鉴定将重组质粒pOJ260-pepA电转[12]入供体菌E. coli S17-1,经阿伯拉霉素、链霉素、三甲氧苄氨嘧啶三抗筛选阳性转化子,提取质粒并用Hind Ⅲ和ClaⅠ进行酶切鉴定,获得转化子S17-1/ pOJ260-pepA。通过接合转移[13, 14]将重组质粒导入野生型刺糖多孢菌中,筛选具有阿伯拉霉素抗性的菌落,并用引物对aac(3)IV-F/aac(3)IV-R(表 1)进行菌落PCR扩增阿伯拉霉素抗性基因片段以鉴定正确转化子,利用LA Taq DNA 聚合酶(5 U/μL)20 μL体系(同上)扩增阿伯拉霉素抗性基因程序如下:94 ℃ 5 min;94 ℃ 30 s,61 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min,20 ℃终止PCR反应。将基因组上整合了pOJ260-pepA的工程菌株命名为S. sp-△pepA。随后提取工程菌株基因组,用引物AM-F/AM-R及AN-F/AN-R(表 1)扩增插入片段和刺糖多孢菌基因组上下游片段鉴定插入位点。PCR反应程序如下:94 ℃ 5 min;94 ℃ 30 s,63 ℃ 30 s,72 ℃ 45 s,30个循环;72 ℃ 10 min,20 ℃终止PCR反应。
1.7 pepA基因阻断表达对刺糖多孢菌生长及菌株形态的影响菌体活化:将200 μL菌种保藏液接种于装量为20 mL CSM液体培养基的250 mL摇瓶中,30 ℃、160 r/min培养2 d。刺糖多孢菌液体培养基中生长曲线的测定采用光密度法:按1%接种量接种活化好的菌种于装量为20 mL TSB液体培养基的250 mL摇瓶中,30 ℃、160 r/min培养5 d,分别定期取样测定光密度值OD600,试验重复3次,取平均值制作生长曲线。将活化后的40 μL原始菌和工程菌菌液分别涂布于BHI、R6和ISP-2固体培养基上[15],30 ℃倒置培养72 h,观察不同平板上菌落生长情况。接种等量的活化后的原始菌与工程菌菌液至20 mL TSB培养基,165 r/min振荡培养2 d,利用光学显微镜(Olympus BH-2型显微镜,日本Olympus公司)观察其菌体形态[14]。
1.8 工程菌株S. sp-△pepA多杀菌素产量变化检测分析分别取相同发酵条件下工程菌株S. sp-△pepA和原始菌株S. spinosa的发酵液500 μL,与等体积的丙酮混匀,室温振荡1-2 h,避光,9000 r/min离心10 min,吸取上清,经0.22 μm滤膜过滤,滤液经安捷伦1290液相色谱仪检测分析多杀菌素含量,色谱柱为SB-C18,4.6 mm×150 mm,5-Micron,流动相:甲醇:乙腈:2%醋酸铵(V/V/V)= 45:45:10,流速0.5 mL/min,紫外检测波长250 nm,进样体积5 μL,通过HPLC外标法计算发酵液中多杀菌素的含量。
1.9 pepA基因阻断表达对刺糖多孢菌菌体全蛋白的影响刺糖多孢菌菌体全蛋白的提取采用超生破碎细胞法:按1%接种量接种活化好的菌种于装量为20 mL TSB液体培养基(含 TSB 3%)的250 mL摇瓶中,30 ℃、160 r/min培养3 d,分别取不同时期菌体进行超生破碎(JY92-Ⅱ型超声波细胞粉碎机,宁波新芝生物科技公司),提取菌体全蛋白。得到的菌体全蛋白利用Bradford法定量后进行SDS-PAGE检测其蛋白表达差异[16, 17]。
1.10 差异蛋白胶内酶解及1D-LC-MS/MS鉴定挖取SDS-PAGE胶上的差异蛋白条带进行酶解[17],酶解后蛋白溶液利用Thermo Scientific LTQ XL型号的二维液相色谱电喷雾串联质谱仪上进行分离和鉴定,整个过程受Xaclbiur数据系统的控制[17]。
1.11 工程菌株S. sp-△pepA遗传稳定性检测将S. sp-△pepA菌株的菌种保藏液用不加抗生素的CSM液体培养基活化48 h,梯度稀释后涂布于无抗生素的BHI平板,30 ℃培养5 d;挑取50个单菌落转接于含阿伯拉霉素(50 mg/L)的BHI平板上,30 ℃培养3-4 d,计数抗性克隆数;并从中随机挑取20个单菌落,于无抗的CSM活化48 h后做菌液PCR扩增检测aac(3)IV片段。
2 结果和分析 2.1 pepA同源基因的确定以天蓝色链霉菌中pepA基因(sco2179)核苷酸序列为标准,利用BLAST在线搜索刺糖多孢菌中该基因同源片段,而后利用Clustal X比对2个基因编码蛋白pepA的相似性(图 2),通过比对可知,两者核苷酸相似性达74%,蛋白同源性达50%。
 |
| 图 2. pepA基因编码蛋白同源性比对 Figure 2. Homology analysis of amino acid residue sequence coded by pepA. |
| 图选项 |
2.2 敲除载体pOJ260-pepA的构建及鉴定以刺糖多孢菌基因组为模板,利用引物pepA-F和pepA-R,PCR扩增pepA基因中间片段。PCR产物经Hind III/EcoR Ⅴ双酶切及回收后,与同样经过双酶切及回收的pOJ260载体片段连接,转化大肠杆菌,抽提转化子质粒,酶切鉴定,得到敲除载体pOJ260-pepA(图 3)。该载体带有大肠杆菌pUC复制子,接合转移起始位点(oriT),阿伯拉霉素抗性基因(aac(3)IV,可以作为E. coli和S. spinosa的筛选标记)。
 |
| 图 3. pOJ260-pepA的构建 Figure 3. Constraction of pOJ260-pepA. A: PCR amplification of the partial pepA gene. M: DNA marker; 1: the partial length of pepA gene. B: Endonuclease digestion assay of pOJ260-pepA. M: DNA marker; 1: digestion of Hind Ⅲ; 2: digestion of Hind Ⅲ and Cla Ⅰ. |
| 图选项 |
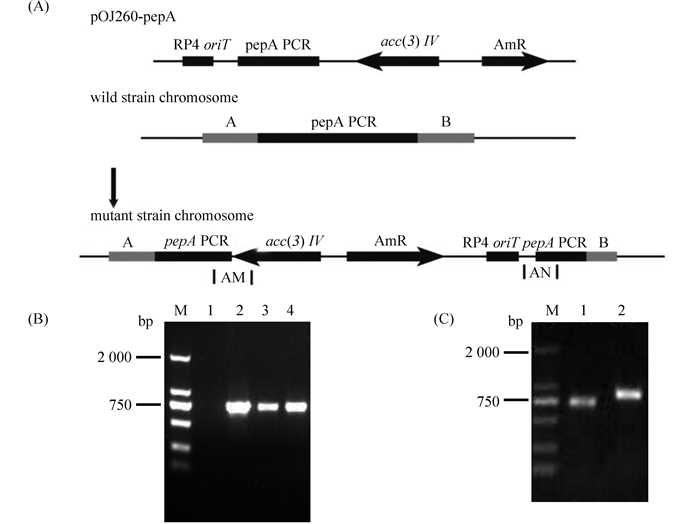
2.3 接合转移构建工程菌株S. sp-△pepA及功能质粒插入位点鉴定将R6平板上长出的接合子转接至含阿伯拉霉素(50 mg/L)的BHI平板上,转接2次,再挑取单菌落至TSB液体培养基,提取基因组为模板进行PCR鉴定。以工程菌S. sp-△pepA基因组为模板,分别以aac(3)IV-F/aac(3)IV-R 引物对PCR 扩增773 bp阿伯拉霉素抗性基因片段,结果表明(图 4-B) pepA基因中间片段已成功的整合至刺糖多孢菌染色体上。另外,以工程菌株S. sp-△pepA基因组为模板,用AM-F/AM-R和AN-F/AN-R分别扩增上游片段AM(700 bp)、下游片段AN(920 bp),结果表明(图 4-C)阻断质粒插入到刺糖多孢菌染色体上的位置与预想一致。
 |
| 图 4. S. sp-△pepA的PCR鉴定 Figure 4. Identification of S. sp-△pepA. A: Diagram of the single-crosscover homologous recombination. B: PCR amplification of partial aac(3)IV gene. M: DNA marker; lane 1: negative control, using the chromosome of S. spinosa as template; lane 2: positive control, using pOJ260-pepA as template; lane 3, 4: experimental group, using the chromosome of S. sp-△pepA as template. C: PCR confirmation of integration site. M: DNA marker; lane 1: downstream segment AN; lane 2: upstream segment AM. |
| 图选项 |
2.4 pepA基因的阻断表达对刺糖多孢菌生长与多杀菌素生物合成的影响观察了工程菌株S. sp-△pepA及原始菌株S. spinosa在不同培养基中的生长发育变化与形态差异。在BHI培养基、ISP-2培养基和R6培养基上培养72 h后,原始菌株已产生较多白色孢子,而工程菌株只产生很少量的孢子,其孢子萌发及形成速率较原始菌株均明显延迟(图 5-A);尤其是将等量的工程菌与原始菌活化菌液涂布于无抗BHI板培养,培养84 h时,原始菌可观察到大片白色孢子,而工程菌仅有稀薄的一层孢子(图 5-B)。TSB 液体培养基中培养48 h后,采用光学显微镜观察2菌株的菌体形态,结果显示原始菌株菌丝细长且分枝多,而工程菌株菌丝较粗短、分枝少且片段化程度高,此外,原始菌结团较工程菌严重(图 6)。
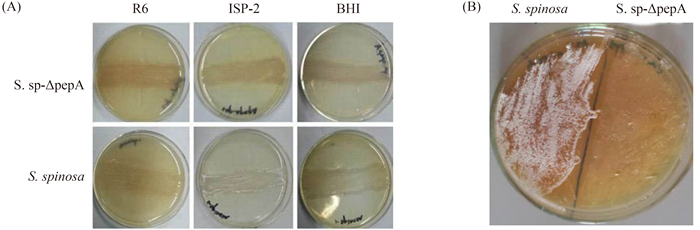 |
| 图 5. 工程菌株S. sp-△pepA与原始菌株S. spinosa的菌落形态比较 Figure 5. Phenotypes of S. sp-△pepA and S. spinosa. A: Phenotypes of S. sp-△pepA and S. spinosa (72 h) in different media; B: Phenotypes of S. sp-△pepA and S. spinosa (84 h) in BHI media. |
| 图选项 |
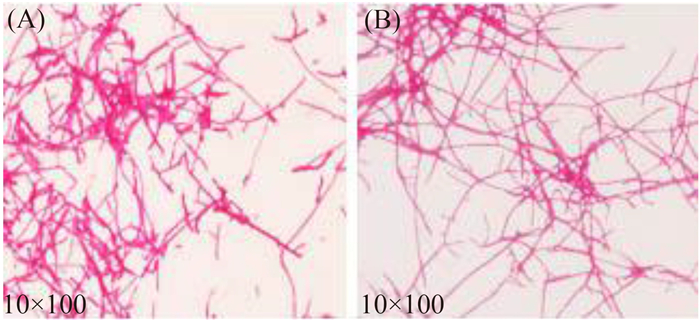 |
| 图 6. 光学显微镜观察工程菌株(A)与原始菌株(B)培养48 h的菌丝形态 Figure 6. Mycelium observation of S. sp-△pepA (A) and S. spinosa (B). |
| 图选项 |
在TSB液体培养基中原始菌株S. spinosa培养24 h后进入对数生长期,48-60 h间又有1个继续增长期,生长过程中无明显稳定期存在。工程菌S. sp-△pepA也是生长24 h进入对数生长期,但工程菌第二生长期菌体生物量无明显增加,而在64-72h菌体生长增强,生物量显著提高;然而工程菌细胞密度一直低于原始菌株,尤其是两者最高生物量相差较大(图 7)。
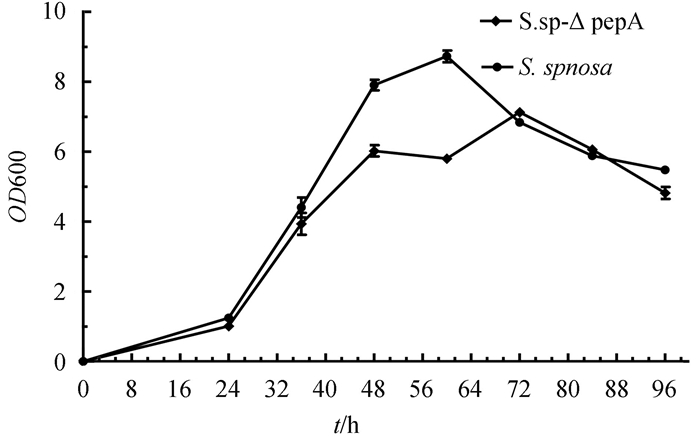 |
| 图 7. 工程菌株S. sp-△pepA与原始菌株S. spinosa的生长曲线比较 Figure 7. Growth curve of S. sp-△pepA and S. spinosa. |
| 图选项 |
HPLC检测结果显示,与原始菌株相比,发酵培养基中工程菌株多杀菌素A+D的产量达137.4 mg/L,相较原始菌的112.5 mg/L提高到122.1%(图 8),说明pepA基因的阻断在一定程度上促进了刺糖多孢菌中多杀菌素的生物合成。
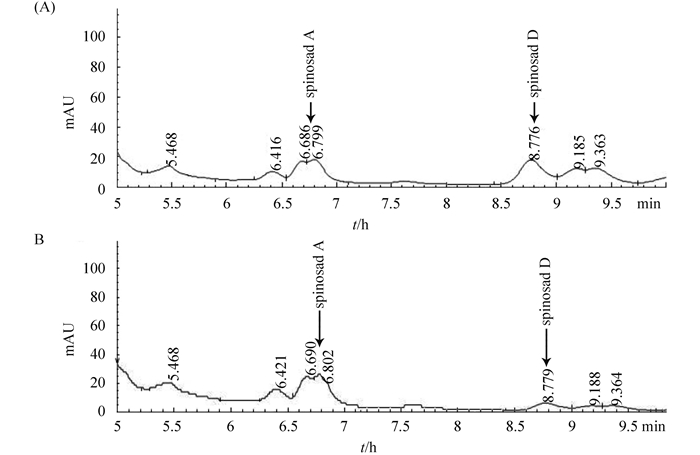 |
| 图 8. HPLC检测原始菌株S. spinosa (A)与工程菌株S. sp-△pepA (B)的多杀菌素产量 Figure 8. HPLC analysis of spinosyn production by S. spinosa (A) and S. sp-△pepA (B). |
| 图选项 |
2.5 工程菌株菌体全蛋白分析通过SDS-PAGE分析工程菌株与原始菌株菌体全蛋白,发现工程菌株全蛋白条带减少在97.2-200.0 kDa区域,推测该区域应该包含pepA基因编码蛋白;另外,工程菌株中某些蛋白表达较原始菌株弱且明显少了蛋白条带A,蛋白条带B、C表达明显增强(图 9)。
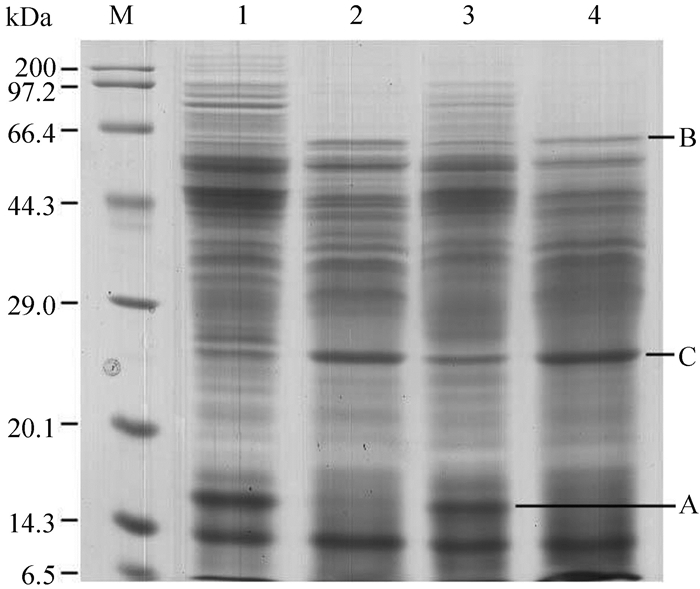 |
| 图 9. 工程菌株与原始菌株全蛋白SDS-PAGE检测 Figure 9. SDS-PAGE gel analysis of total proteins. M: protein marker; lane 1, 3: samples from 60 h and 72 h S. spinosa cells, respectively; lane 2, 4: samples from 60 and 72 h S. sp-△pepA cells, respectively. |
| 图选项 |
2.6 差异蛋白分析将SDS-PAGE检测到的差异蛋白条带A、B、C胶内酶解后通过1D-LC-MS/MS鉴定,发现它们分别为30S核糖体蛋白亚基S11(RpsK)、醛基脱氢酶(AldH)和30S核糖体蛋白亚基S4(RpsD)(图 10,表 2)。利用在线蛋白分析软件Uniprot (www. uniprot.org)和STRING(string.embl.de)对其功能进行分析得到AldH具有NAD(醛脱氢酶)活性,可催化醛基转化为羧基,还可与乙醛脱氢酶、乙酰辅酶A合成酶相互作用,从而影响辅酶A的合成;辅酶A是合成多杀菌素前体糖苷配基的重要底物。工程菌株S. sp-△pepA多杀菌素产量较原始菌株有一定提高(图 8)可能与该酶过表达有关。RpsK、RpsD两者均为30S核糖体蛋白亚基,它们作用于核糖体蛋白组装,从而影响蛋白翻译,对菌体初级代谢产生一定影响。工程菌株S.sp-△pepA中RpsK表达被抑制,因而其生物量较原始菌株显著下降,尤其是当两者生长状态达到最佳时蛋白表达被抑制的影响也最显著(图 7,图 9)。
 |
| 图 10. 差异蛋白的部分特异性肽段 Figure 10. Special peptides of identified proteins. A: RpsK; B: AldH; C: RpsD. |
| 图选项 |
表 2. 差异蛋白LC-MS/MS分析Table 2. Proteins identified from SDS-PAGE gel analysis
| Band numbers | ID | Protein description | Theoretical MW/kDa | Possible function |
| A | A4FPJ4 | 30S ribosomal protein S11 (RpsK) | 14.3 | Binding RNA Binding rRNA |
| B | A4FGH6 | Aldehyde dehydrogenase (AldH) | 55.8 | Aldehyde dehydrogenase (NAD) activity an aldehyde + NAD+ + H2O = an acid + NADH + H+ |
| C | A4FPJ3 | 30S ribosomal protein S4 (RpsD) | 23.2 | Binding RNA Binding rRNA |
表选项
3 讨论刺糖多孢菌多杀菌素含量极低促使人们对其进行菌种改良和发酵工艺优化以提高其产量。已有对刺糖多孢菌利用传统的理化诱变结合高通量筛选[18],原生质体融合及再生[19],多杀菌素生物合成相关基因加倍等方法对其进行菌种改良的研究报道。近年来,利用基因工程技术加倍与过量表达放线菌中与次级代谢相关的正调控功能基因,成为了提高相关抗生素产量的有效途径,但对于阻断刺糖多孢菌负调控因子的表达以提高多杀菌素产量的相关报道少见。
许多调控子全局掌控次级代谢,其调控机制也慢慢被解析。与转录和翻译过程相关的基础元件也能直接或间接影响次级代谢,但研究其具体机制尚存在瓶颈,因而可以通过观察菌体抗生素合成及孢子分化的变化来监测基础元件调控次级代谢的流程。已有研究表明大肠杆菌的pepA具有DNA结合活性,能与多种启动子序列结合[5],但天蓝色链霉菌的同种蛋白没有亮氨酰氨肽酶活性却能影响其抗生素ACT、RED的合成[20]。本研究利用生物信息学软件BLAST和Cluster X比对刺糖多孢菌全基因组和天蓝色链霉菌的亮氨酰氨肽酶基因(SCO2179),发现刺糖多孢菌也有该蛋白且序列高度同源(图 2);以此为基础将刺糖多孢菌中pepA基因中间片段整合至刺糖多孢菌染色体上通过单交换同源重组阻断其表达得到工程菌株S. sp-△pepA,其生物量和产孢子能力明显低于原始菌株S. spinosa(图 5,图 6),且菌丝较粗短,结团现象得到缓解(图 7);而且其多杀菌素的产量较原始菌株有较大提高(图 8)。pepA具有DNA结合活性,能与多种启动子序列结合[5],从而影响启动子下的调控基因表达水平。pepA与氨甲酰磷酸合酶操纵子(carAB)结合形成的复合体PepA-carAB能降低胺甲酰磷酸合酶的磷酸化水平以减弱其转录翻译水平[21]。阻断pepA还能显著提高细胞分裂激活因子(SsgA)的转录水平[6],SsgA只存在于形态复杂的放线菌中[22],能直接激活产孢专一的细胞分裂,并且过量表达能引起天蓝色链霉菌菌丝形态发生较大改变,使营养菌丝的宽度显著增加,并且形成大量被异常增厚膈膜分开的类孢子室[23],从而影响菌丝发育。放线菌发酵产生次生代谢的产量与发酵成本受到其丝状菌丝形态的严重影响,液体培养中链霉菌的菌丝形态主要由细胞分裂激活因子ssgA的表达水平决定,SsgA表达量越高,营养菌丝胞间膈膜的形成频率越高,菌丝的片段化程度也越高[24]。
比对生长曲线发现工程菌株在整个生长周期中的细胞密度均低于原始菌株,这可能是因为pepA被阻断后某些受该调控子正调控的基因表达受到阻遏,比对菌体全蛋白SDS-PAGE(图 9)也能发现工程菌株蛋白条带明显减少。在BHI和ISP-2培养基上,与原始菌株相比,其工程菌株孢子萌发及孢子形成延迟,但最终仍然能够正常产生孢子,这可能也与功能菌株功能不足有关;在营养丰富的R6培养基上,两者生长速率及菌落形态无明显差异,推测与培养基组分不同有关。另外,经光学显微镜观察工程菌株菌丝较细短,分支多,有利于菌株发酵时的溶氧,对多杀菌素的生物合成起促进作用。
LC-MS/MS鉴定到的差异蛋白分别为30S核糖体蛋白亚基S11(RpsK)、醛基脱氢酶(AldH)和30S核糖体蛋白亚基S4(RpsD)。对其功能分析可知具有醛脱氢酶活性可催化醛基转化为羧基,而RpsK、RpsD两者均为30S核糖体蛋白亚基。AldH与乙醛脱氢酶、乙酰辅酶A合成酶相互作用,从而影响辅酶A的合成;辅酶A是合成多杀菌素前体糖苷配基的重要底物。工程菌株S.sp-△pepA多杀菌素产量较原始菌株有一定提高(图 8)可能与该酶过表达有关。RpsK、RpsD它们作用于核糖体蛋白组装,从而影响蛋白翻译,对菌体初级代谢产生一定影响。工程菌株S. sp-△pepA中RpsK表达被抑制,因而其生物量较原始菌株显著下降,尤其是当两者生长状态达到最佳时蛋白表达被抑制的影响也最显著(图 7,图 9)。
参考文献
| [1] | Kim H, Lipscomb WN. Structure and mechanism of bovine lens leucine aminopeptidase. Advances in Enzymology and Related Areas of Molecular Biology, 1994, 68:153-213. |
| [2] | Matsui M, Fowler JH, Walling LL. Leucine aminopeptidases:diversity in structure and function. Biological Chemistry, 2006, 387(12):1535-1544. |
| [3] | Sträter N, Sherrat DJ, Collons SD. X-ray structure of aminopeptidase A from Escherichia coli and a model for the nucleoprotein complex in Xer-site specific recombination. The EMBO Journal, 1999, 18(16):4513-4522. |
| [4] | Tu CJ, Park SY, Walling LL. Isolation and characterization of the neutral leucine aminopeptidase (LapN) of tomato. Plant Physiology, 2003, 132(1):243-255. |
| [5] | Devroede N, Huysveld N, Charlier D. Mutational analysis of intervening sequences connecting the binding sites for integration host factor, PepA, PurR, and RNA polymerase in the control region of the Escherichia coli carAB operon, encoding carbamoylphosphate synthase. Journal of Bacteriology, 2006, 188(9):3236-3245. |
| [6] | Song E, Rajesh T, Lee BR, Kim EJ, Jeon JM, Park SH, Park HY, Choi KY, Kim YG, Yanq YH, Kim BG. Deletion of an architectural unit, leucyl aminopeptidase (SCO2179), in Streptomyces coelicolor increases actinorhodin production and sporulation. Applied Microbiology and Biotechnology, 2013, 97(15):6823-6833. |
| [7] | Kirst HA, Michel KH, Martin JW, Creemer LC, Chio EH, Yao RC, Nakatsukasa WM, Boeck LD, Occolowitz JL, Paschal JW, Deeter JB, Jones ND, Thompson GD. A83543A-D, unique fermentation-derived tetracyclic macrolides. Tetrahedron Letters, 1991, 32(37):4839-4842. |
| [8] | Sparks TC, Crouse GD, Durst G. Natural products as insecticides:the biology, biochemistry and quantitative structure-activity relationships of spinosyns and spinosoids. Pest Management Science, 2001, 57(10):896-905. |
| [9] | Bierman M, Logan R, O'Brien K, Seno ET, Rao RN, Schoner BE. Plasmid cloning vectors for the conjugal transfer of DNA from Escherichia coli to Streptomyces spp.. Gene, 1992, 116(1):43-49. |
| [10] | Pan HX, Li JA, He NJ, Chen JY, Zhou YM, Shao L, Chen DJ. Improvement of spinosad production by overexpression of gtt and gdh controlled by promoter PermE in Saccharopolyspora spinosa SIPI-A2090. Biotechnology Letters, 2011, 33(4):733-739. |
| [11] | Pan YY, Lu C, Dong HL, Yu LJ, Liu G, Tan HR. Disruption of rimP-SC, encoding a ribosome assembly cofactor, markedly enhances the production of several antibiotics in Streptomyces coelicolor. Microbial Cell Factories, 2013, 12(1):65-81. |
| [12] | Maniatis T, Fritsch EF, Sambrook J. Molecular cloning:a laboratory manual. New York:Cold Spring Harbor Laboratory Press, 1982. |
| [13] | Matsushima P, Brouqhton MC, Turner JR, Baltz RH. Conjugal transfer of cosmid DNA from Escherichia coli to Saccharopolyspora spinosa:effects of chromosomal insertions on macrolide A83543 production. Gene, 1994, 146(1):39-45. |
| [14] | Kieser T, Bibb MJ, Buttner MJ, Chater KF, Hopwood DA. Practical Streptomyces Genetics. Norwich, UK:The John Innes Foundation, 2000, 161-211. |
| [15] | Swiercz JP, Nanji T, Gloyd M, Guarné A, Elliot MA. A novel nucleoid-associated protein specific to the actinobacteria. Nucleic Acids Research, 2013, 41(7):4171-4184. |
| [16] | Luo YS, Ding XZ, Xia LQ, Huang F, Li WP, Huang SY, Tang Y, Sun YJ. Comparative proteomic analysis of Saccharopolyspora spinosa SP06081 and PR2 strains reveals the differentially expressed proteins correlated with the increase of spinosad yield. Proteome Science, 2011, 9:40. |
| [17] | Yang Q, Ding XZ, Liu XM, Liu S, Sun YJ, Yu ZQ, Hu SB, Rang J, He H, Xia LQ. Differential proteomic profiling reveals regulatory proteins and novel links between primary metabolism and spinosad production in Saccharopolyspora spinosa. Microbial Cell Factories, 2014, 13(1):27-43. |
| [18] | Li Y, Chang C, Yang KQ. Biosynthetic pathway and synthetic strategy of spinosad-a review. Acta Microbiologica Sinica, 2011, 51(11):1431-1439. (in Chinese) 李月, 常城, 杨克迁. 多杀菌素生物合成途径及改造策略. 微生物学报2011, 51(11):1431-1439. |
| [19] | Wang C, Zhang XL, Chen Z, Wen Y, Song Y. Strain construction for enhanced production of spinosad via intergeneric protoplast fusion. Canadian Journal of Microbiology, 2009, 55(9):1070-1075. |
| [20] | Yang YH, Song E, Willemse J, Park SH, Kim WS, Kim EJ, Lee BR, Kim JN, van Wezel GP, Kim BG. A novel function of Streptomyces integration host factor (sIHF) in the control of antibiotic production and sporulation in Streptomyces coelicolor. Antonie Van Leeuwenhoek, 2012, 101(3):479-492. |
| [21] | Le Minh PN, Devroede N, Massant J, Maes D, Charlier D. Insights into the architecture and stoichiometry of Escherichia coli PepA·DNA complexes involved in transcriptional control and site-specific DNA recombination by atomic force microscopy. Nucleic Acids Research, 2009, 37(5):1463-1476. |
| [22] | Noens EEE, Mersinias V, Traag BA, Smith CP, Koerten HK, van Wezel GP. SsgA-like proteins determine the fate of peptidoglycan during sporulation of Streptomyces coelicolor. Molecular Microbiology, 2005, 58(4):929-944. |
| [23] | van Wezel GP, van der Meulen J, Kawamoto S, Luiten RGM, Koerten HK, Kraal B. SsgA is essential for sporulation of Streptomyces coelicolor A3(2) and affects hyphal development by stimulating septum formation. Journal of Bacteriology, 2000, 182(20):5653-5662. |
| [24] | van Wezel GP, van der Meulen J, Taal E, Koerten H, Kraal B. Effects of increased and deregulated expression of cell division genes on the morphology and on antibiotic production of streptomycetes. Antonie van Leeuwenhoek, 2000, 78(3/4):269-276. |
