侯婷婷1, 2, 钟志平1, 2, 刘缨1, 刘志培1


1. 中国科学院微生物研究所, 微生物资源前期开发国家重点实验室, 北京 100101;
2. 中国科学院大学生命科学院, 北京 100049
收稿日期: 2015-05-21; 修回日期: 2015-06-24; 网络出版日期: 2015-07-05
基金项目: 山东省自主创新工程项目(2014ZZCX06204);中国科学院创新工程项目(KZCX2-EW-Q212)
通信作者: 刘志培, Tel: +86-10-64806081; E-mail: liuzhp@sun.im.ac.cn
摘要: [目的] 为揭示工厂化循环水青石斑鱼养殖水体的细菌群落特征,比较患病养殖池与健康养殖池的细菌群落结构差异,探讨细菌群落结构与青石斑鱼病害相关的相关性。[方法] 采用Illumina Miseq高通量测序方法,分析比较了患病和健康养殖水体细菌群落结构、α-多样性指数(包括多度、均一度和系统发育多样性);并结合传统方法从患病青石斑鱼病灶部位分离疑似病原菌。[结果] 患病和健康养殖水体中细菌群落的α-多样性并无太大差异,但主坐标分析与热图样本聚类分析表明细菌群落结构明显不同。二者的优势细菌门均为Proteobacteria、Verrucomicrobia和Bacteroidetes,但它们的相对丰度差异显著。患病养殖水体中α-Proteobacteria (25.07%)和γ-Protebacteria (22.74%)丰度相当,而健康养殖水体中γ-Protebacteria (40.49%)显著高于α-Proteobacteria (10.87%)。患病水体的Verrucomicrobia丰度(26.4%)远高于健康水体(10.9%);而Bacteroidetes的相对丰度则相反(12.3% vs 20.9%)。主要的差异类群包括α-Proteobacteria的Rhodobacteraceae和Rhodospirillaceae,γ-Proteobacteria的Alteromonadaceae、HTCC2188和Oceanospirillaceae,Verrucomicrobia的Verrucomicrobiaceae和Bacteroidetes的Cryomorphaceae。更表现在核心微生物类群的差异,健康养殖池水体以Glaciecola、HTCC、Sediminicola、Prevotella等对于养殖动物有益或无害的属为核心微生物;而患病养殖池水体则以Vibrio、Rubritalea、Oleibacter等病原菌或对养殖动物不利的属为核心微生物。从患病青石斑鱼的皮肤、肝脏和脾脏共分离得到弧菌20株,Acinetobacter haemolyticus 1株。[结论] 患病的青石斑鱼循环水养殖水体中的细菌群落明显不同于健康养殖水体,特别是核心微生物的差异,其以Vibrio等病原菌或对养殖动物不利的属为主。该结果为青石斑鱼循环水养殖系统的管理、病害的诊断和监测提供理论与实验基础。
关键词: 青石斑鱼循环水养殖系统养殖水体细菌群落核心微生物
Bacterial community characterization of rearing water of marine recirculating aquaculture systems for yellow grouper (Epinephelus awoara)
Tingting Hou1, 2, Zhiping Zhong1, 2, Ying Liu1, Zhipei Liu1


1. State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
2. College of Life Sciences, University of Chinese Academy of Sciences, Beijing 100049, China
Received: 21 May 2015; Revised: 24 June 2015; Published online: 5 July 2015
Foundation Item: Supported by the Innovation Program of Shandong Province (2014ZZCX06204) and by the Innovation Program of Chinese Academy of Sciences (KZCX2-EW-Q212)
Corresponding author: Zhipei Liu, Tel: +86-10-64806081; E-mail: liuzhp@sun.im.ac.cn
Abstract:[Objective] This study was aimed to reveal the bacterial community characteristics of rearing water of marine recirculating aquaculture system for yellow grouper (Epinephelus awoara), and compare the differences between bacterial community structure of healthy rearing water and sick rearing water in order to elucidate the relationship between bacterial community and fish disease. [Methods] The next generation sequencing method was used, and the bacterial community structure and α-diversity indices (species richness, species evenness and phylogenetic diversity) between the rearing water of healthy and diseased groups were studied and compared. In addition, the traditional cultivation method was used to isolate suspected pathogens from the niduses of diseased yellow groupers. [Results] There was no significant difference between the α-diversities of healthy and diseased rearing water bodies, however, the results of principal coordinates analysis (PCoA) and the sample clustering of heatmap showed that the bacterial communities of healthy and diseased rearing water bodies were quite different. Although phyla Proteobacteria, Verrucomicrobia and Bacteroidetes were all the predominant ones in both communities, their relative abundance varied greatly. In diseased community, the relative abundances of α-Proteobacteria (25.07%) and γ-Protebacteria (22.74%) were similar, whereas the proportion of γ-Protebacteria (40.49%) was much higher than α-Proteobacteria (10.87%) in the healthy community. The differences of Verrucomicrobia and Bacteroidetes between the healthy and the diseased rearing water were also significant with relative abundances of 10.9% and 26.4%, and 20.9% and 12.3%, respectively. The significantly different families were Rhodobacteraceae and Rhodospirillaceae within class α-Proteobacteria; Alteromonadaceae and HTCC2188 within class γ-Protebacteria; Verrucomicrobiaceae within Verrucomicrobia; Cryomorphaceae within Bacteroidetes. The healthy and the diseased communities owned specific core microbes themselves. Glaciecola, HTCC, Sediminicola and Prevotella were the core genera in healthy rearing water, and Vibrio, Rubritalea and Oleibacter in diseased rearing water. Twenty strains of Vibrio spp. and one of Acinetobacter haemolyticus were isolated from skin, liver and spleen of diseased yellow grouper. [Conclusion] The shift of bacterial community structure and relative abundance of rearing water will help monitor the healthy status of recirculating aquaculture system. Our study provides theory and experimental basis to diagnosis and monitor of Vibrio disease for yellow grouper recirculating aquaculture system.
Key words: Yellow grouper (Epinephelus awoara)recirculating aquaculture system (RAS)rearing waterbacterial communitycore microbes
渔业是我国的重要产业,在国民经济中占有很大的比重。自2002年起,我国成为水产品最大的出口国[1]。由于传统渔业开放或半开放的养殖模式,导致其对水体的利用率低、养殖条件难以控制、产量低、食品安全难以保障、易对环境造成污染[2]。相比之下,循环水养殖(Recirculating Aquaculture System,RAS)技术凭借其对生产过程中参数的有效控制和高效的废水处理和循环利用[3],已经发展成密集型、高投入、高产出、高效率、可持续的养殖模式。循环水养殖不对土地、气候、水源过多依赖,因此它正逐渐地接管传统养殖技术,并且已被世界多国应用于生产高经济价值的鱼类[4, 5]。青石斑鱼(Epinephelus awoara)是我国重要的海洋养殖品种,但是利用循环水养殖技术进行大规模生产还比较少见。
养殖池中微生物群落与养殖动物直接接触,鱼类与微生物的互作只发生在养殖池中,其健康状况、病害的发生与流行与养殖池中的微生物群落息息相关。循环水养殖系统是个封闭的系统,引入外界水源不到3%,虽可大大减少了引入外来病原菌的机率。但由于其是个循环的系统,一旦某个养殖池存在病原菌时,将可能引发大规模病害的爆发,甚至导致整个养殖厂的崩溃,造成严重的经济损失。因此,非常有必要了解循环水养殖水体中的微生物群落特征,比较健康养殖水体和患病水体之间微生物群落的差异,探讨微生物群落结构与养殖动物健康状况之间的关系。然而,迄今为止极少见到有关方面的研究报道。
本研究以青石斑鱼海水循环水养殖系统为对象,采用illumina Miseq高通量测序技术,检测、比较健康养殖系统与患病养殖系统的养殖水体的细菌群落特征与差异,并从患病青石斑鱼病灶部位分离培养细菌,初步确定引发病害的疑似病原菌。以期为青石斑鱼的循环水养殖系统的管理、病害防治和构建循环水养殖的病害预警系统和控制策略提供依据。
1 材料和方法 1.1 青石斑鱼循环水养殖系统本研究依托天津海发珍品实业发展有限公司,以该公司的工厂化青石斑鱼循环海水养殖系统为研究对象。每套养殖系统包括10个养殖池,养殖池排水经水循环系统、水质净化处理与消毒后循环到养殖池。养殖池规格为36 m2/36 m3,养殖密度为25 kg/m3。养殖水体pH控制在7.6-7.7,溶氧(Dissolved Oxygen,DO)控制在8-10 mg/L,盐度为2.5% (W/V),水温控制在28-29 °C。2013年10月起,其中一套循环水养殖系统出现了严重的病害,病症为拒食、游动缓慢、表皮溃烂、肝脏充血肿大,造成了青石斑鱼的大量死亡,死亡率最高达到了5%/d。
1.1.1 养殖水体采样:于2013年11月11日分别随机从3个患病养殖池和3个健康养殖池(Q表示患病池样品,S表示健康池样品)采集养殖水体样品(0.5 L)各1个,共6个样品。所有水样保存于4 °C并在12 h内运回实验室,并及时用直径为0.22 μm的聚醚砜膜(津腾,中国)抽滤。将带有水体宏基因组的滤膜保存于-80 °C,用于宏基因组DNA的提取。
1.1.2 患病青石斑鱼样品:于2013年11月11日,将3条患病青石斑鱼置于充氧的海水中,带回实验室,用于病灶部位细菌的分离。
1.2 水样宏基因组提取和PCR扩增子焦磷酸测序水体宏基组的提取采用试剂盒(E.Z.N.A Water DNA Extraction kit,Omega Bio-Tek,USA),具体操作参照试剂盒说明书。
对16S rRNA基因V4高变区进行PCR扩增,所用引物为细菌特异性引物515F (5′-GTGCCAGCM GCCGCGGTAA-3′)和806R (5′-GGACTACHVGG GTWTCTAAT-3′)[6]。PCR扩增子焦磷酸测序应用Illumina Miseq平台(250 bp paired-end方法)[6, 7]。对于每一个样品都进行2次平行测序。简单地说,Barcode和Illumina adaptors与PCR引物融合产生带有这二者的扩增子产物,然后每个PCR产物等量混合建库,并测序。测序由诺和致源生物信息公司(北京)完成。
1.3 序列分析1.3.1 高通量序列处理:Illumina Miseq产生的数据用QIIME (version 1.8.0-amd64.vdi)[8]分析,除了特别指明外所有参数均采用默认参数。对刚下机的低质量数据(Raw reads)运用以下参数进行质量过滤得到Clean Tags:片段拼接错配率<0.025%、引物序列错配率<3%、Barcode错配率为0但是允许一个末端碱基缺失。然后运用UPARSE流程[9]对Clean Tags按照97%相似度聚类成OTU (Operational Taxonomic Unit),最终运用BMP脚本[10]生成OTU表格(OTU table)。Singletons通过参数-minisize 2去除。UPARSE流程应用UCHIME算法[11]能够准确快速地检测嵌合体。用RDP数据库[12]进行物种注释。OTU table用Qiime脚本生成。在接下来的统计分析中,古菌、线粒体和叶绿体的16S rRNA基因序列已去除。
1.3.2 统计分析:为了排除序列差异所引入的差异,所有样本的序列都均一化到17052条序列(Min=17052,Max=72541,Mean=42843,SD=20810)。统计每个样品中各分类群在各分类水平上的相对丰度,并用柱状图进行可视化。用Chao1[13]、Shannon[14]和PD[15]指数分别估测微生物群落的物种多度(species richness)、物种均一度(species evenness)和系统发育多样性(phylogenetic diversity)。运用One-way ANOVA检验不同健康状态养殖水体α-多样性差异的显著性。能够反映系统发育信息的weighted unifrac和 unweighted unifrac距离[16]根据稀释后的OTU table生成。Unifrac矩阵的主坐标分析(Principal Coordinates Analysis,PCoA)用于说明不同健康状态养殖池之间微生物群落的差异(β-多样性)。核心微生物群(core microbiome)是人类微生物组(human microbiome)研究中的一个重要概念,指两个或多个与某一特定环境相关的微生物集群(microbial assemblages)中所共有的成员[17]。分析中,可以通过计算某一环境条件下所有样品中均存在的OTU找出该环境的核心微生物菌群[18]。采用热图(heatmap)对核心微生物群在各分类水平进行可视化展示,热图中各样品的丰度经“标准化”处理,即centered和scaled处理。
1.4 病灶部位菌株分离和鉴定用无菌剪刀、镊子解剖患病青石斑鱼,用接种环分别挑取皮肤病灶部位、肝组织、脾组织划线于2216E和TCBS固体平板,30 °C倒置培养2 d,挑取表型有差异的单个菌落划线转接于2216E平板,直到纯化。纯化后的细菌用2216E甘油管(W/V=25%)于-80 °C长期保存。
分离纯化的纯培养物,挑取单菌落,利用27F和1492R通用引物[19]对细菌16S rRNA基因进行扩增。PCR条件为:94 °C 5 min;94 °C 1 min,55 °C 1 min,72 °C 1.5 min,30个循环;72 °C 10 min。将获得的PCR产物直接送至商业的测序公司进行测序。得到的核酸序列与NCBI中的GenBank数据库进行BLAST比对(http://www.ncbi. nlm.nih.gov/blast/Blast.cgi),根据比对结果查找与该菌株序列相似性较高的菌株,初步确定该菌株分类地位。
2 结果和分析 2.1 高通量序列数据6个样品经过质量过滤、嵌合体(Chimeric)移除,总共收集到268970条序列,并聚类于1629个OTU,其中5个OTU (775条序列)属于古细菌,1个OTU (2条序列)属于线粒体,2个OTU (4条序列)属于叶绿体,此外,505个OTU (共8473条序列)在域的水平未被归类,属于未知序列。在下游的分析中只保留了未被分类的序列和细菌的序列。
2.2 患病和健康养殖水体细菌群落特征2.2.1 患病和健康养殖水体细菌群落α-多样性:经过均一化之后,共得到102312条序列用于进一步的统计分析。经16S rRNA基因序列97%相似性聚类,对各样品OTU数目和表征微生物群落物种多度、物种均一度和系统发育多样性的α-多样性指标总结如表1。结果表明,各样品OTU数接近;患病和健康青石斑鱼养殖水体细菌群落Chao1指数分别为614.62±130.90和627.52±183.89,Shannon指数分别为5.08±0.26和5.09±0.17,PD距离分别为37.46±2.59和42.37±5.28。经One-way ANOVA分析,患病和健康养殖水体中上述α-多样性指数均无显著性差异(P>0.05)。
表 1. 患病和健康养殖水体细菌群落α-多样性Table 1. α-diversity of bacterial communities of rearing water bodies of sick and healthy ponds
| Samples * | 97% OTU | α-diversity indices | ||
| Chao 1 | Shannon | PD | ||
| Q12-8 | 545 | 474.59 | 5.11 | 37.16 |
| Q12-9 | 423 | 635.38 | 4.82 | 35.03 |
| Q12-12 | 483 | 733.90 | 5.33 | 40.18 |
| S5-6 | 421 | 792.53 | 5.27 | 46.31 |
| S6-1 | 407 | 660.75 | 5.06 | 44.44 |
| S6-4 | 521 | 429.28 | 4.94 | 36.37 |
| *Q and S represent rearing water samples collected from sick and healthy tanks, respectively. | ||||
表选项
2.2.2 细菌群落结构和相对丰度:基于unifrac距离的主坐标分析能够揭示两个细菌群落之间相似性,两者之间的unifrac距离越小,它们二者之间所共有的微生物遗传世系就越多。Weighted主坐标分析在样品聚类时考虑了物种的丰度,而unweighted主坐标分析仅考虑物种的存在与否。Weighted和unweighted主坐标分析都表明水样按照青石斑鱼的健康状态沿着PC1轴聚类(图1),也就是说,两个聚类群分别由来自健康和患病养殖池的样品组成。Weighted 和unweighted unifrac主坐标分析,前两个主坐标的累计方差贡献率分别高达94.21%和46.34%。尤其weighted unifrac主坐标分析,第一主坐标PC1和第二主坐标PC2分别占总变异来源的76.79%和17.42%。主坐标分析结果表明患病青石斑鱼养殖水体和健康青石斑鱼养殖水体细菌群落之间同一类群的丰度(图1-A)和群落成员的种类(图1-B)均存在明显差异。
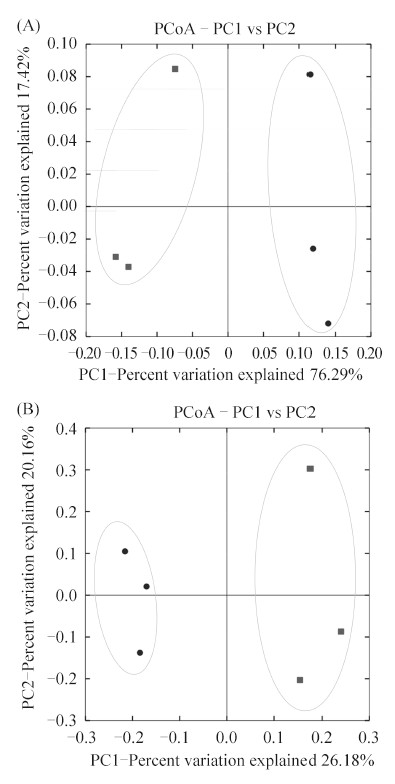 |
| 图 1. 基于weighted unifrac距离(A)和unweighted unifrac距离(B)的主坐标分析 Figure 1. Principal coordinates analysis of weighted unifrac distance (A) and unweighted unifrac distance (B). Square symbols represent samples from healthy tanks and circle symbols represent samples from sick tanks. |
| 图选项 |
通过统计各分类水平细菌群落信息,能够得知患病和健康养殖水体细菌群落之间具体的差异。图2为患病和健康养殖水体细菌群落在纲水平的相对丰度(图2-A)和各分类水平优势类群(图2-B)。图2的结果表明,循环水养殖水体中细菌群落的构成比较单一,在任意分类水平都是个别几种类群占据绝对的优势地位,其余大部分类群构成很小的比例,如在患病和健康养殖水体中Proteobacteria、Verrucomicrobia和Bacteroidetes三个门均占据细菌总量的85%以上(图2-A)。图2-B表明,在各分类水平上,2个细菌群落的组成比较相似,但是同一分类水平的类群的相对丰度明显不同。对于患病养殖池水体,优势门Proteobacteria、 Verrucomicrobia和Bacteroidetes的相对丰度分别为49.0%、26.4%和12.3%,优势纲Verrucomicrobiae、 α-Proteobacteria、γ-Protebacteria和Flavobacteriia的相对丰度分别为26.38%、25.07%、22.74%和9.05%,优势目Verrucomicrobiales、 Rhodobacterales、Vibrionales和 Flavobacteriia的相对丰度分别为26.4%、23.8%、15.7%和9.0%;健康养殖池水体中,优势门Proteobacteria、Bacteroidetes和Verrucomicrobia的相对丰度分别为52.5%、20.9%和10.9%,优势纲γ-Protebacteria、Flavobacteriia、Verrucomicrobiae和α-Proteobacteria的相对丰度分别为 40.5%、17.6%、10.9%和10.9%,优势目Alteromonadales、Flavobacteriia、Vibrionales、Verrucomicrobiales和Rhodobacterales的相对丰度分别为19.1%、17.6%、16.2%、10.9%和9.7%。此外,在健康养殖水体中,腈基降解菌纲(Nitriliruptoria)的相对丰度也比较高,为9.08%;而在患病养殖水体中未知菌群(4.32 %)比较高(图2-A)。
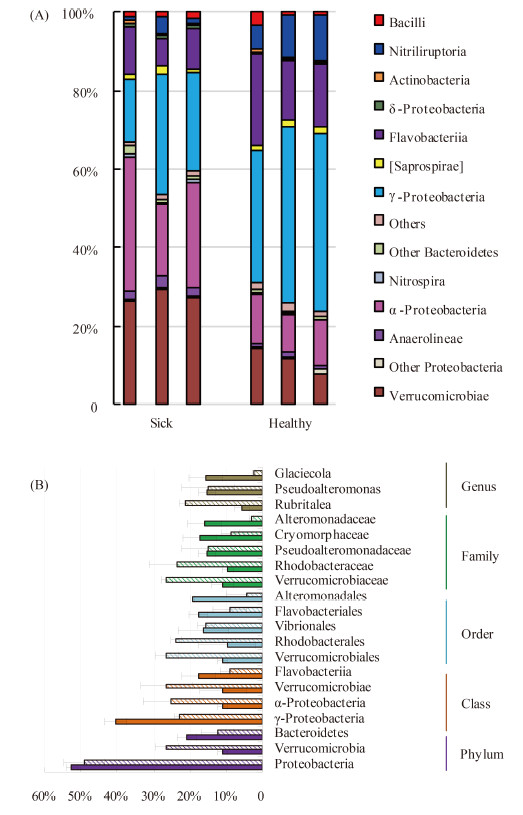 |
| 图 2. 患病和健康养殖水体细菌纲水平相对丰度(A)和各分类水平优势分类群(B) Figure 2. Relative abundance at class level (A) and the dominant taxa at each taxonomic level (B) of bacterial communities of rearing water of healthy and sick tanks. In (A), minor phyla totally accounting < 2% were shown together as ‘Others’; In (B), lined columns are samples from sick tanks; solid columns are samples from healthy tanks. Error bars represent a single standard deviation. |
| 图选项 |
2.2.3 细菌群落的核心微生物:本研究的核心微生物分析表明,患病池和健康池养殖水体细菌群落的核心OTU数分别为214和235。图3为基于核心微生物群在属水平丰度的热图,该图的结果表明,核心微生物群根据进化关系聚类为两大分支——健康池分支(分支I)和患病池分支(分支II),说明这2个细菌群落的核心微生物群明显不同。健康养殖池(分支Ⅰ)的核心属主要包括Glaciecola、HTCC、Sediminicola、Prevotella、Roseivirga、Psychrobacter、Rhodoferax、Fluviicola、Marinomonas、Olleya、Thalassomonas和Sporosarcina等海水中常见的细菌类群,为非病原菌或条件致病菌;而患病养殖(分支Ⅱ)的核心属主要包括Vibrio、Rubritalea、Oleibacter、Nitrospira、Bacillus、Owenweeksia、Reinekea、nsmpVI18、Persicirhabdus、Loktanella、Hyphomonas、Sphingobacterium、Lishizenia、Bacteriovorax、
Congregibacter、Lewinella、Mogibacterium和Pseudomonas等,特别是Vibrio、Owenweeksia和Pseudomonas等属于鱼类的病原菌或条件致病菌。
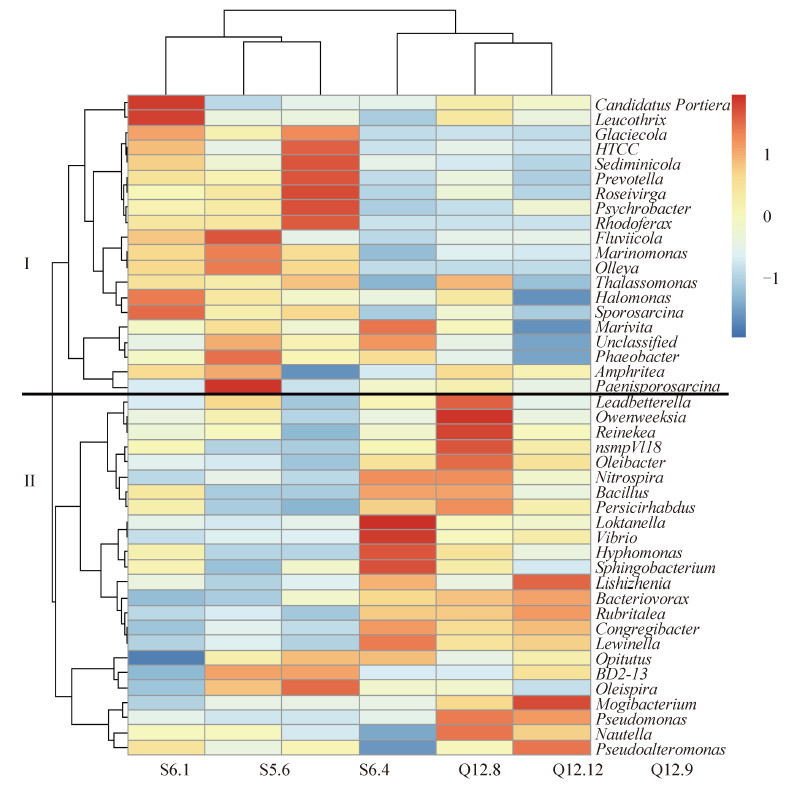 |
| 图 3. 核心微生物群在属水平热图 Figure 3. Heatmap of core microbiome at geuns level |
| 图选项 |
2.3 病鱼疑似病原菌的分离与分析从病灶部位分离细菌经16S rRNA基因鉴定结果列于表2。结果表明,共分离得到21株纯菌株,其中20株为弧菌(Vibrio),共6个种,包括对人和水生动物致病性都比较强的V. harveyi;1株溶血不动杆菌(Acinetobacter haemolyticus),分离自肝脏。采用MA和TCBS培养基均分离得到5个种,其中用MA分离到12株(4种弧菌,1株溶血不动杆菌),而用TCBS分离得到9株(5种弧菌)。两种培养基均能够从皮肤、肝脏、脾脏三种病灶部位分离得到优势菌株,TCBS培养基表现出对弧菌的偏好性,而MA则可以分离得到除弧菌以外的疑似病原菌。弧菌具有多种毒力因子[20, 21],可以引起多种海洋动物包括皮肤溃烂在内的多种疾病[22, 23]。溶血不动杆菌能够定殖于人肠道并产生志贺毒素[24],从而导致血性腹泻。因此所分离的21株均可能为患病养殖池青石斑鱼病害的病原菌。
表 2. 病灶部位分离细菌总结Table 2. A summary of suspected pathogens isolated from sick fishes
| Phylogenetic types | MA (No.) | TCBS (No.) | Sources |
| Vibrio sinaloensis | 4 | 1 | Skin, Liver, Spleen |
| Vibrio harveyi | 5 | 2 | Skin, Liver, Spleen |
| Vibrio owensii | 0 | 2 | Skin |
| Vibrio alfacsensis | 0 | 2 | Skin |
| Vibrio alqoinfesta | 1 | 2 | Skin, Spleen |
| Vibrio fortis | 1 | 0 | Liver |
| Acinetobacter haemolyticus | 1 | 0 | Liver |
表选项
3 讨论养殖水体中微生物对鱼类的健康至关重要,微生物的群落结构指示着鱼类健康状况的变化。本研究采用高通量测序技术深度测序(17052条序列/样),评估了青石斑鱼海水循环水养殖系统患病养殖水体和健康养殖水体中细菌群落的特征,比较了二者细菌多样性和群落结构的差异。患病和健康养殖水体细菌群落的α-多样性并无明显差异(表1),但是二者的群落结构却明显不同(图1、2、3)。健康和患病养殖池水体中Proteobacteria的丰度都是最高的,但是在健康养殖水体中占据优势的是α-Proteobacteria,而在患病养殖水体中占据优势的是α-Proteobacteria。据文献报道,α-Proteobacteria是循环水健康养殖池的最优势Proteobacteria类群[25],本研究中的健康养殖池的结果与其一致。此外,也有研究表明,α-Proteobacteria类群也是健康青石斑鱼以及其他鱼类的皮肤、腮、肠道等的优势类群[26, 27, 28, 29]。因此,α-Proteobacteria类群的丰度可以很好地反映养殖水体以及养殖动物的健康状况,也说明了该类群微生物对于维持养殖系统的稳定运行的重要性。
Verrucomicrobia门细菌广泛分布于海洋环境中,约占水体细菌群落总数的2%[30]。然而在本研究中,患病和健康养殖池水体中该类群的平均相对丰度分别达到了26.4%和10.9% (图2),远远高于自然海水环境,其最主要成员为Rubritalea属。Verrucomicrobia门的细菌绝大多数属于未培养的微生物,其功能还鲜为人知[31]。迄今为止所报道的6株Rubritalea属的细菌有5株分离自海洋动物[32, 33, 34, 35, 36],表明该属成员很可能与动物之间存在互作关系,与动物的健康状况密切相关。此外,本研究中高比例的Verrucomicrobia门的结果,一方面可能由于循环水养殖系统的正向选择;另一方面由于之前研究所用的PCR引物的偏好性从而低估环境中Verrucomicrobia的丰度[37],而本研究中所采用的引物515f/806r恰能将偏好性降到最低[38]。
Bacteroidetes是海洋环境重要的微生物类群,是溶解性有机物(Dissolved Organic Matter,DOM) 的主要消费者[39]。在不同健康状态下养殖水体中该类群细菌的含量也存在明显差异(图2)。健康养殖池水体中的Flavobacteria纲,以及该纲成员Cryomorphaceae科的相对丰度都约是患病养殖池水体中的2倍(图2-B)。Flavobacteria细菌是海水中的高分子量有机物的主要降解者[40],Cryomorphaceae的很多成员在海洋环境的碳和能量循环中起着不可或缺的作用[41]。健康和患病养殖水体中这些菌群的变化可能能够反应两种状态下水质的不同而造成环境压力的不同,高丰度的有机物降解菌群使得养殖水体中的有机物降解速率增快,鱼所面临的环境压力则要小得多。
核心微生物,特别是核心物种或核心OTU,以及由核心微生物组成的核心微生物菌群(core microbiome)是某一微生物群落的特征微生物类群,对于群落的功能起着关键作用[18]。本研究中的健康养殖池和患病养殖池的核心微生物群(属水平)明显不同(图3),特别是患病养殖池水体中(图3分支II)核心微生物群包含一些已知的病原菌或条件致病菌,如Vibrio、Owenweeksia、Pseudomonas属等;而这些属都不是健康养殖池水体(图3分支I)的核心微生物。此外,从患病青石斑鱼病灶部位分离的菌株绝大部分属于弧菌(表2)。弧菌具有多种毒力因子,如胞外产物、脂多糖、外毒素等[20, 21],可引起多种海洋动物包括皮肤溃烂在内的多种病害[22, 23],正是患病鱼的症状。由此可以初步推断患病循环水养殖池的病害是由弧菌导致。
通过对患病和健康养殖水体微生物群落的比较,发现了养殖动物的不同健康状态存在不同的微生物群落结构。该结果为循环水养殖系统病害防治系统的预警提供了有利的参考。核心微生物群的动态变化或许可以为弧菌病害的监测提供参考。此外,α-Proteobacteria作为青石斑鱼土著微生物中重要的部分,或许可以通过检测它的改变达到一定的诊断目的。
致谢
感谢天津海发珍品有限公司提供研究对象和实验的原始材料,感谢中国科学院微生物研究所伍林军和诺禾致源生物信息技术有限公司刘文彬先生在数据分析中所提供的帮助。
参考文献
| [1] | Fisheries and aquaculture department. The state of world fisheries and aquaculture. Rome: FAO, 2012. |
| [2] | Yang J, He ZG. Discussions on the current situation of aquaculture and its prospects. China Science & Technology Panorama Magazine, 2012, (5): 248. (in Chinese)杨军, 何志刚. 关于我国水产养殖现状及其前景的探讨. 中国科技纵横, 2012, (5): 248. |
| [3] | Bovendeur J, Eding EH, Henken AM. Design and performance of a water recirculation system for high-density culture of the African catfish, Clarias gariepinus (Burchell 1822). Aquaculture, 1987, 63(1/4): 329-353. |
| [4] | Martins P, Cleary DFR, Pires ACC, Rodrigues AM, Quintino V, Calado R, Gomes NCM. Molecular analysis of bacterial communities and detection of potential pathogens in a recirculating aquaculture system for Scophthalmus maximus and Solea senegalensis. PLoS One, 2013, 8(11): e80847. |
| [5] | Wold PA, Holan AB, Øie G, Attramadal K, Bakke I, Vadstein O, Leiknes TO. Effects of membrane filtration on bacterial number and microbial diversity in marine recirculating aquaculture system (RAS) for Atlantic cod (Gadus morhua L.) production. Aquaculture, 2014, 422-423: 69-77. |
| [6] | Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Lozupone CA, Turnbaugh PJ, Fierer N, Knight R. Global patterns of 16S rRNA diversity at a depth of millions of sequences per sample. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(Suppl 1): 4516-4522. |
| [7] | Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Huntley J, Fierer N, Owens SM, Betley J, Fraser L, Bauer M, Gormley N, Gilbert JA, Smith G, Knight R. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. The ISME Journal, 2012, 6(8): 1621-1624. |
| [8] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Peña AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Tumbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. |
| [9] | Edgar RC. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. |
| [10] | Pylro VS, Roesch LFW, Ortega JM, do Amaral AM, Tótola MR, Hirsch PR, Rosado AS, Góes-Neto A, da Costa da Silva AL, Rosa CA, Morais DK, Andreote FD, Duarte GF, de Melo IS, Seldin L, Lambais MR, Hungria M, Peixoto RS, Kruger RH, Tsai SM, Azevedo V. The Brazilian Microbiome Project Organization Committee. Brazilian Microbiome Project: revealing the unexplored microbial diversity--challenges and prospects. Microbial Ecology, 2014, 67(2): 237-241. |
| [11] | Edgar RC, Haas BJ, Clemente JC, Quince C, Knight R. UCHIME improves sensitivity and speed of chimera detection. Bioinformatics, 2011, 27(16): 2194-2200. |
| [12] | Wang Q, Garrity GM, Tiedje JM, Cole JR. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Applied and Environmental Microbiology, 2007, 73(16): 5261-5267. |
| [13] | Colwell RK, Coddington JA. Estimating Terrestrial Biodiversity through Extrapolation. Philosophical Transactions of the Royal Society of London. Series B: Biological Sciences, 1994, 345(1311): 101-118. |
| [14] | Magurran AE. Ecological diversity and its measurement. Princeton: Princeton University Press, 1988. |
| [15] | Lozupone CA, Knight R. Species divergence and the measurement of microbial diversity. FEMS Microbiology Reviews, 2008, 32(4): 557-578. |
| [16] | Lozupone C, Knight R. UniFrac: a new phylogenetic method for comparing microbial communities. Applied and Environmental Microbiology, 2005, 71(12): 8228-8235. |
| [17] | Turnbaugh PJ, Ley RE, Hamady M, Fraser-Liggett CM, Knight R, Gordon JI. The Human Microbiome Project. Nature, 2007, 449(7164): 804-810. |
| [18] | Shade A, Handelsman J. Beyond the Venn diagram: the hunt for a core microbiome. Environmental Microbiology, 2012, 14(1): 4-12. |
| [19] | Stackebrandt E, Goodfellow M. Nucleic acid techniques in bacterial systematics. New York: A Wiley-Interscience Publication, 1991. |
| [20] | Higgins DA, Pomianek ME, Kraml CM, Taylor RK, Semmelhack MF, Bassler BL. The major Vibrio cholerae autoinducer and its role in virulence factor production. Nature, 2007, 450(7171): 883-886. |
| [21] | Zhang L L, Orth K. Virulence determinants for Vibrio parahaemolyticus infection. Current Opinion in Microbiology, 2013, 16(1): 70-77. |
| [22] | Zheng TL, Wang GL, Jin S. Prevention and cure of vibriosis in aquatic animals: a review. Journal of Oceanography in Taiwan Strait, 2002, 21(3): 372-378. (in Chinese)郑天伦, 王国良, 金珊. 海水养殖动物弧菌病防治的研究进展. 台湾海峡, 2002, 21(3): 372-378. |
| [23] | Chen Q, Yan QP, Ma S. Progress on pathogenicity research of Vibrio alginolyticus. Marine Sciences, 2006, 30(8): 83-89. (in Chinese)陈强, 鄢庆枇, 马甡. 溶藻弧菌致病性研究进展. 海洋科学, 2006, 30(8): 83-89. |
| [24] | Grotiuz G, Sirok A, Gadea P, Varela G, Schelotto F. Shiga toxin 2-producing Acinetobacter haemolyticus associated with a case of bloody diarrhea. Journal of Clinical Microbiology, 2006, 44(10): 3838-3841. |
| [25] | Lahav O, Massada IB, Yackoubov D, Zelikson R, Mozes N, Tal Y, Tarre S. Quantification of anammox activity in a denitrification reactor for a recirculating aquaculture system. Aquaculture, 2009, 288(1/2): 76-82. |
| [26] | Feng JB, Hu CQ, Luo P, Zhang LP, Chen C. Microbiota of yellow grouper (Epinephelus awoora Temminck & Schlegel, 1842) fed two different diets. Aquaculture Research, 2010, 41(12): 1778-1790. |
| [27] | Roeselers G, Mittge EK, Stephens WZ, Parichy DM, Cavanaugh CM, Guillemin K, Rawls JF. Evidence for a core gut microbiota in the zebrafish. The ISME Journal, 2011, 5(10): 1595-1608. |
| [28] | Shiina A, Itoi S, Washio S, Sugita H. Molecular identification of intestinal microflora in Takifugu niphobles. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2006, 1(1): 128-132. |
| [29] | Yang GM, Bao BL, Peatman E, Li HR, Huang LB, Ren DM. Analysis of the composition of the bacterial community in puffer fish Takifugu obscurus. Aquaculture, 2007, 262(2/4): 183-191. |
| [30] | Freitas S, Hatosy S, Fuhrman JA, Huse SM, Welch DBM, Sogin ML, Martiny AC. Global distribution and diversity of marine Verrucomicrobia. The ISME Journal, 2012, 6(8): 1499-1505. |
| [31] | Stevenson BS, Eichorst SA, Wertz JT, Schmidt TM, Breznak JA. New strategies for cultivation and detection of previously uncultured microbes. Applied and Environmental Microbiology, 2004, 70(8): 4748-4755. |
| [32] | Kasai H, Katsuta A, Sekiguchi H, Matsuda S, Adachi K, Shindo K, Yoon J, Yokota A, Shizuri Y. Rubritalea squalenifaciens sp. nov., a squalene-producing marine bacterium belonging to subdivision 1 of the phylum 'Verrucomicrobia'. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 7): 1630-1634. |
| [33] | Scheuermayer M, Gulder TA, Bringmann G, Hentschel U. Rubritalea marina gen. nov., sp. nov., a marine representative of the phylum 'Verrucomicrobia', isolated from a sponge (Porifera). International Journal of Systematic and Evolutionary Microbiology, 2006, 56(Pt 9): 2119-2124. |
| [34] | Yoon J, Matsuda S, Adachi K, Kasai H, Yokota A. Rubritalea halochordaticola sp. nov., a carotenoid-producing verrucomicrobial species isolated from a marine chordate. International Journal of Systematic and Evolutionary Microbiology, 2011, 61(Pt 7): 1515-1520. |
| [35] | Yoon J, Matsuo Y, Matsuda S, Adachi K, Kasai H, Yokota A. Rubritalea sabuli sp. nov., a carotenoid- and squalene-producing member of the family Verrucomicrobiaceae, isolated from marine sediment. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(Pt 4): 992-997. |
| [36] | Yoon J, Matsuo Y, Matsuda S, Adachi K, Kasai H, Yokota A. Rubritalea spongiae sp. nov. and Rubritalea tangerina sp. nov., two carotenoid- and squalene-producing marine bacteria of the family Verrucomicrobiaceae within the phylum 'Verrucomicrobia', isolated from marine animals. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 10): 2337-2343. |
| [37] | Bergmann GT, Bates ST, Eilers KG, Lauber CL, Caporaso JG, Walters WA, Knight R, Fierer N. The under-recognized dominance of Verrucomicrobia in soil bacterial communities. Soil Biology & Biochemistry, 2011, 43(7): 1450-1455. |
| [38] | Liu ZZ, Lozupone C, Hamady M, Bushman FD, Knight R. Short pyrosequencing reads suffice for accurate microbial community analysis. Nucleic Acids Research, 2007, 35(18): e120. |
| [39] | Cottrell MT, Kirchman DL. Natural assemblages of marine proteobacteria and members of the Cytophaga-Flavobacter cluster consuming low- and high-molecular-weight dissolved organic matter. Applied and Environmental Microbiology, 2000, 66(4): 1692-1697. |
| [40] | Kirchman DL. The ecology of Cytophaga-Flavobacteria in aquatic environments. FEMS Microbiology Ecology, 2002, 39(2): 91-100. |
| [41] | Nikrad MP, Cottrell MT, Kirchman DL. Uptake of dissolved organic carbon by gammaproteobacterial subgroups in coastal waters of the West Antarctic Peninsula. Applied and Environmental Microbiology, 2014, 80(11): 3362-3368. |
