彭忠1, 梁婉1,2, 吴斌1


1.华中农业大学动物医学院, 农业微生物学国家重点实验室, 生猪健康养殖协同创新中心, 湖北 武汉 430070;
2.华中农业大学动物科学技术学院, 农业动物遗传育种与繁殖教育部重点实验室, 生猪健康养殖协同创新中心, 湖北 武汉 430070
收稿日期:2016-01-03;修回日期:2016-03-08;网络出版日期:2016-03-24
基金项目:公益性行业(农业)科研专项(201403054);湖北省科技支撑计划项目(2014BBB10,2013BBB14)
*通信作者:吴斌,Tel:+86-27-87287201;Fax:+86-27-87282608;E-mail:wub@mail.hzau.edu.cn
摘要: 多杀性巴氏杆菌是一种能感染多种动物甚至是人的重要革兰氏阴性病原菌。目前临床上用于多杀性巴氏杆菌诊断的分型方法主要包括血清学分型方法和分子分型方法。其中血清学分型方法主要基于免疫学实验技术建立,操作过程繁琐,技术要求高,工作量大,不适用于临床上大规模快速开展多杀性巴氏杆菌流行病学调查的需要;而基于分子生物学手段建立的分子分型方法相对于传统的血清学分型方法而言具有快速、简单、灵敏、灵活等特点,特别是某些分子分型方法与传统的分型方法形成了较为精确的对应关系,因而在临床上得到了广泛的应用。目前适用于临床上开展多杀性巴氏杆菌分离鉴定的分子分型方法主要包括多重PCR方法及多位点序列分型法(MLST),其中多重PCR方法又包括基于荚膜编码区及脂多糖外核编码簇建立的PCR方法。本文将重点就这3种常用的多杀性巴氏杆菌分子分型方法进行综述,介绍其建立原理、实现手段以及各自的优缺点,为临床上开展多杀性巴氏杆菌的流行病学调查特别是分子流行病学调查提供参考。
关键词: 多杀性巴氏杆菌 分子分型方法 多重PCR 多位点序列分型
Molecular typing methods for Pasteurella multocida-A review
Peng Zhong1, Liang Wan1,2, Wu Bin1


1.State Key Laboratory of Agricultural Microbiology, the Cooperative Innovation Center for Sustainable Pig Production, College of Veterinary Medicine, Huazhong Agricultural University, Wuhan 430070, Hubei Province, China;
2.Key Laboratory of Agricultural Animal Genetics, Breeding and Reproduction, Ministry of Education, the Cooperative Innovation Center for Sustainable Pig Production, College of Animal Science and Technology, Huazhong Agricultural University, Wuhan 430070, Hubei Province, China
Received 03 January 2016; Revised 08 March 2016; Published online 24 March 2016
*Corresponding author: Tel:+86-27-87287201;Fax:+86-27-87282608;E-mail:wub@mail.hzau.edu.cn
Supported by the Special Public Welfare Industry (Agriculture) Research Program Grant (201403054) and by the Hubei Provincial Sci-Tech Support Plan Grants (2014BBB10, 2013BBB14)
Abstract: Pasteurella multocida is an important gram-negative pathogenic bacterium that could infect wide ranges of animals. Humans could also be infected by P. multocida via animal bite or scratching. Current typing methods for P. multocida include serological typing methods and molecular typing methods. Of them, serological typing methods are based on immunological assays, which are too complicated for clinical bacteriological studies. However, the molecular methods including multiple PCRs and multilocus sequence typing (MLST) methods are more suitable for bacteriological studies of P. multocida in clinic, with their simple operation, high efficiency and accurate detection compared to the traditional serological typing methods, they are therefore widely used. In the current review, we briefly describe the molecular typing methods for P. multocida. Our aim is to provide a knowledge-foundation for clinical bacteriological investigation especially the molecular investigation for P. multocida.
Key words: Pasteurella multocida molecular typing methods multiple PCRs multilocus sequence typing
多杀性巴氏杆菌是一种重要的人兽共患病原菌,能感染多种家畜、家禽、宠物、野生动物和人,主要导致感染者发生呼吸系统疾病、局灶性感染及出血性败血症。多杀性巴氏杆菌不同菌株造成感染的临床表现不同,其导致的常见疾病包括禽霍乱、反刍动物的出血性败血症及肺炎、猪萎缩性鼻炎和巴氏杆菌肺炎、兔鼻瘘和出血性败血症及人的皮肤坏死、脑膜炎、蜂窝组织炎、脓肿、腹膜炎等[1-2],这些疾病的爆发和流行严重威胁动物及人的健康安全。
多杀性巴氏杆菌的分型方法主要包括血清学分型方法和分子分型方法。最常用的血清学分型方法包括Carter分型方法和Heddleston分型方法,即分别依据荚膜抗原的不同和脂多糖抗原的不同将多杀性巴氏杆菌分为5种血清型(A、B、D、E、F)[3]和16种血清型(1-16)[4],它们建立的技术基础分别为间接血凝试验(Passive hemagglutination,PHA)[3]和琼脂扩散沉淀试验(Gel diffusion precipitin tests,GDP)[4]。多杀性巴氏杆菌血清学分型方法的建立对于其流行病学调查的开展及致病规律的研究具有十分重要的意义,很多报道均指出多杀性巴氏杆菌的血清型与其导致的疾病存在一定的联系(表 1)[1, 5]。然而,该方法过于繁琐,同时对于抗血清的浓度、滴度和特异性均具有较高的要求,实现工艺较为复杂,技术难度较大,不适合于临床上大规模快速地开展多杀性巴氏杆菌流行病学调查。
表 1. 多杀性巴氏杆菌血清型与巴氏杆菌病之间的关系* Table 1. General relationships between disease syndromes and Pasteurella multocida capsular serogroups*
| Diseases | Serogroups | Species affected | Virulence factors |
| Animal bite-associated sepsis | Usually A, occasionally D | Any, but most commonly humans, cats and dogs | Not established |
| Haemorrhagic septicaemia | B, E | Cattle, buffalo, pigs, goats, deer, camels, rarely horses | Capsule, fimbriae, filamentous, haemagglutinin |
| Avian cholera | A, F, very rarely D | Chickens, turkeys, ducks, wild birds | Capsule, LPS, iron acquisition proteins, fimbriae, filamentous haemagglutinin, sialic acid uptake |
| Atrophic rhinitis | D, rarely A | Pigs, rabbits | Pasteurella multocida toxin (PMT) |
| Snuffles | A, D | Rabbits | Not established |
| Enzootic pneumonia, shipping fever | A, D, rarely F | Cattle, sheep and pigs | Not established. Co-infection with other bacterial or viral species important |
| *: cited from reference [1]. | |||
表选项
随着分子生物学技术的快速发展,特别是PCR技术及基因测序技术的日益普及,基于分子生物学技术手段建立了多种多杀性巴氏杆菌的分子分型方法,主要包括多重PCR方法和多位点序列分型法(MLST),它们在多杀性巴氏杆菌的临床诊断中得到了较为广泛的应用。相对于传统的血清学分型方法而言,这些方法具有快速、简单、灵敏、灵活等特点,特别是其中基于多杀性巴氏杆菌荚膜编码基因簇和脂多糖外核编码基因簇建立的多重PCR方法与传统的Carter和Heddleston分型方法形成了较为精确的对应关系,这在一定程度上又使得临床上快速界定多杀性巴氏杆菌的血清型成为可能。本文将重点就多杀性巴氏杆菌的主要分子分型方法进行综述,为临床上快速开展多杀性巴氏杆菌流行病学调查特别是分子流行病学调查提供参考。
1 基于荚膜编码区的多重PCR分型法 荚膜(Capsule)是多杀性巴氏杆菌表面的主要组分之一,也是其重要的毒力因子之一,在多杀性巴氏杆菌抗吞噬的过程中发挥着十分重要的作用[6]。多杀性巴氏杆菌血清学分型方法中的Carter分型系统正是以荚膜作为主要研究对象而建立的[3]。针对不同Carter血清型多杀性巴氏杆菌的全基因组序列分析发现,多杀性巴氏杆菌中负责荚膜合成的基因在其基因组中成簇存在 [7-8];并且整个荚膜编码基因簇按照功能的不同可被分为3个不同的区域,其中区域一包含4个基因(hexABCD或cexABCD),与荚膜多糖的输出有关(capsule export);区域三由2个基因组成(phyBA或lipBA),一般与荚膜多糖的磷脂替换有关(phospholipid substitution)[9],这2个区域在不同血清型的多杀性巴氏杆菌较为保守(图 1)。区域二与荚膜多糖的形成有关(Capsule biosynthesis)[9],该区域在不同血清型的多杀性巴氏杆菌存在一定的差异,总体而言可以分为2大类:A型、D型和F型归为一类,其区域二中含有4个基因(A型:hyaEDCB;B型:dcbEFCB;F型:fcbE DCB),其中hyaB (dcbB/fcbB)和hyaC (dcbC/fcbC)在不同荚膜血清型的菌株比较保守,hyaE (dcbE/fcbE)在不同血清型的菌株间也存在一定的同源性,然而hyaD(dcbF/fcbD)在不同血清型的菌株间同源性较低(图 1);B型和E型菌株间区域二的相似度较高,均含有9个编码基因(B型:bcbABCDEFGHI;E型:ecbABJKDEFGI),其中,E型荚膜多糖合成基因ecbI、ecbB 及ecbA的编码产物分别与B型荚膜多糖合成基因bcbI、bcbB及bcbA的编码产物存在较高的同源性(匹配度 > 87%,相似度 > 91%),而ecbDEFG与bcbDEFG之间也存在一定的同源性(匹配度介于44%-89%之间),此外,ecbK编码产物的C段序列与bcbC的编码产物的相似度在55%左右,只有ecbJ在B型荚膜多糖中未见具有匹配度的对应基因[9]。因此,在所有与荚膜形成有关的基因中,hyaD、bcbD、dcbF、ecbJ、fcbD等基因表现出了相应的荚膜型特异性。基于此,Townsend等(2001)利用这些基因不同位点的序列设计引物(表 2),建立了一套利用PCR技术快速鉴定多杀性巴氏杆菌荚膜血清型的方法[9]。该方法具有快速、简便、准确等特点,并且能够准确地与传统的Carter分型方法形成对应关系,已经取代了Carter方法成为多杀性巴氏杆菌的临床检测中应用最为广泛的分型方法。我们利用该方法分别于2003 -2007年以及2014年间来自我国多个规模化猪场患有呼吸系统疾病的猪多组织器官样品进行了多杀性巴氏杆菌的分离与鉴定,揭示了我国猪群中流行的多杀性巴氏杆菌主要为A型菌株和D型菌株[10-11];并且还首次报道了F型多杀性巴氏杆菌在我国猪群中的存在[11]。然而,该方法也存在一定的不足,例如我们在对这些样品进行多杀性巴氏杆菌的分离与鉴定的过程中发现部分菌株的荚膜血清型无法通过该多重PCR方法进行界定,这一类多杀性巴氏杆菌被统称为“未定型菌株”(untypable strain)[10]。未定型多杀性巴氏杆菌在国外的类似工作中也有过报道[12-13],这一类菌株在临床上多与胸膜肺炎放线杆菌、副猪嗜血杆菌等其它病原体形成混合感染,常与猪肺炎的形成有关。我们推测,“未定型菌株”或许与当前依然缺乏合适的界定方法有关[10]。
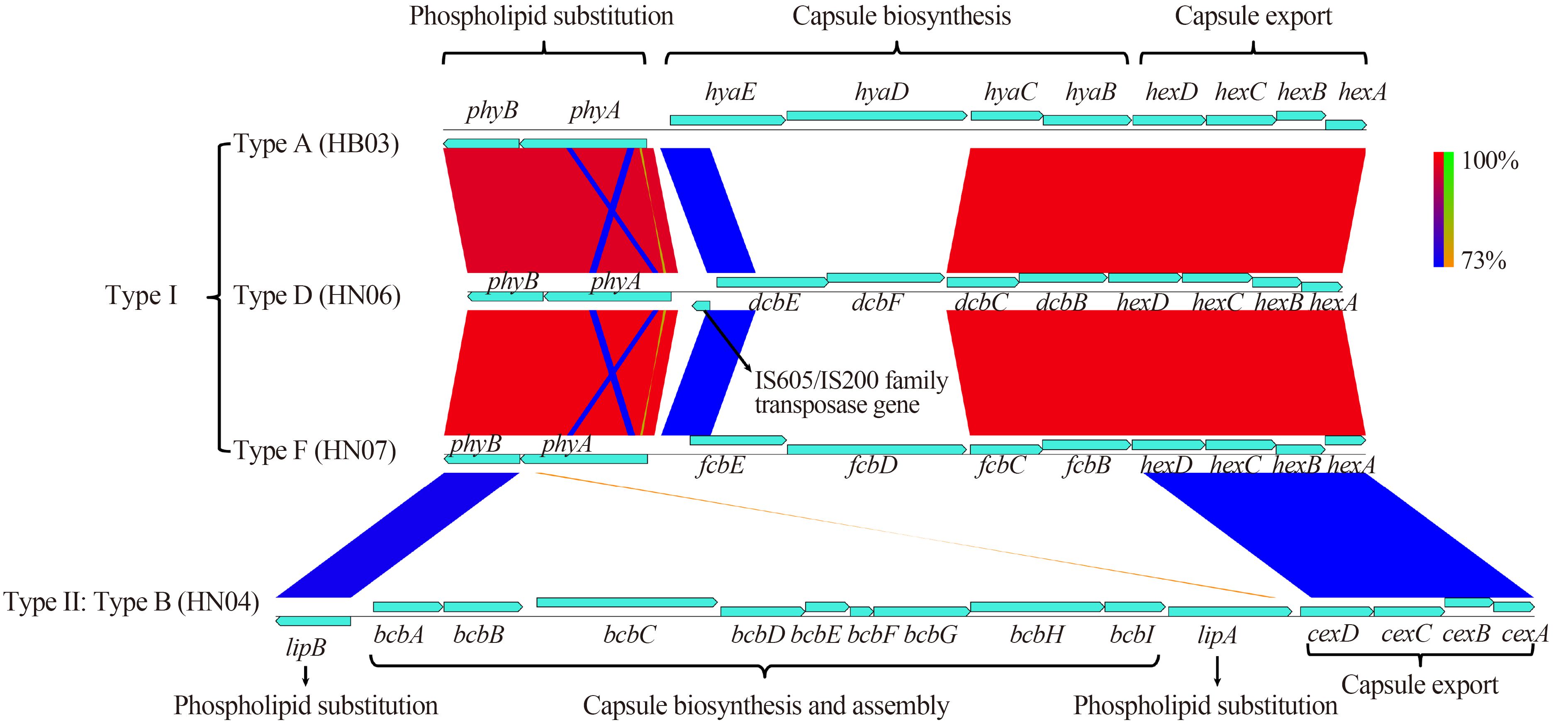 |
| 图 1. 不同血清型多杀性巴氏杆菌荚膜合成基因簇比较基因组学分析 Figure 1. Comparative analysis of capsule synthetic loci of Pasteurella multocida serogroups |
| 图选项 |
表 2. 用于检测多杀性巴氏杆菌荚膜血清型的引物* Table 2. Primers used for determination of Pasteurella multocida capsular serogroups*
| Serogroup | Target gene | Sequence (5′→3′) | Product/bp | Position |
| A | hyaD-hyaC | TGCCAAAATCGCAGTCAG | 1044 | 8846–8863 |
| TTGCCATCATTGTCAGTG | 9890–9873 | |||
| B | bcbD | CATTTATCCAAGCTCCACC | 760 | 13621–13603 |
| GCCCGAGAGTTTCAATCC | 12863–12880 | |||
| D | dcbF | TTACAAAAGAAAGACTAGGAGCCC | 657 | 3142–3165 |
| CATCTACCCACTCAACCATATCAG | 3789–3766 | |||
| E | ecbJ | TCCGCAGAAAATTATTGACTC | 511 | 4387–4408 |
| GCTTGCTGCTTGATTTTGTC | 4899–4881 | |||
| F | fcbD | AATCGGAGAACGCAGAAATCAG | 851 | 2881–2896 |
| TTCCGCCGTCAATTACTCTG | 3733–3714 | |||
| *: cited from reference [9] | ||||
表选项
2 基于脂多糖外核编码基因簇的PCR分型法 脂多糖(Lipopolysaccharide,LPS)是多杀性巴氏杆菌表面的另外一种重要组成成分,也是其主要毒力因子之一[6]。此外,LPS还是多杀性巴氏杆菌血清分型方法的重要参考依据之一;Heddleston分型系统正是基于LPS将多杀性巴氏杆菌分为16种血清型(1-16)[4]。与绝大多数细菌LPS分子不同的是,多杀性巴氏杆菌产生的LPS不含O抗原,其LPS分子主要包括内核和外核两部分,其中内核主要由类脂A(lipid A,也称内毒素)与内核低聚糖组成,而外核的主要成分也是低聚糖[15]。绝大多数多杀性巴氏杆菌在通常情况下能同时产生2种不同糖型的LPS(A型和B型)[15]。就不同糖型的LPS而言,其外核多糖的结构通常较为保守,而内核多糖的结构却存在一定的差异性,这种差异性主要体现在B型LPS的内核包含1个lipidA及2个3-脱氧-d-甘露辛酮糖酸(Kdo)残基,而A型LPS的内核中与lipidA相连的则是1个磷酸化的Kdo残基[15]。负责内核多糖编码和装配的基因通常分散于多杀性巴氏杆菌基因组的不同位点,并且绝大多数内核编码及装配基因均较为保守。不同糖型的LPS外核结构较为固定,主要由不同数目的葡萄糖(Glc)、半乳糖(Gal)、乙酰氨基葡萄糖(GlcNAc)以及庚糖(Hep)按照一定的比例通过一系列的转移酶连接组成[15]。与LPS内核装配基因在基因组分布较为分散不同的是,与LPS外核合成和装配有关的基因在多杀性巴氏杆菌基因组上的分布则较为集中,均位于两个保守的、与LPS合成无关的基因priA和fpg之间[7-8, 14]。
Harper等针对Heddleston 1、2、3、4、5、8、9、10、11、12、13、14和15型多杀性巴氏杆菌的LPS进行分析发现,这些菌株LPS的结构差异性主要体现在其外核结构上[16-21]。在基因水平上表现为负责编码LPS外核糖链装配所需酶的基因数目及同源性存在较大的差异;进一步研究发现,多杀性巴氏杆菌的Heddleston血清型与其外核编码基因簇存在一定的对应关系,并且部分不同Heddleston血清型多杀性巴氏杆菌共用一套结构和功能相似的外核装配基因系统(图 2)。根据不同Heddleston血清型多杀性巴氏杆菌外核装配所需的基因数目及同源性的不同可以将其LPS外核多糖分为8个不同的基因型(L1-L8)[22]。多杀性巴氏杆菌LPS外核编码基因簇的这些差异性和相似性,为多杀性巴氏杆菌新型LPS分型系统的建立奠定了理论基础。
 |
| 图 2. 不同 Heddleston 血清型的多杀性巴氏杆菌 LPS 外核亲缘关系及结构示意图 Figure 2. Phylogenetic analysis and genetic structure of Pasteurella multocida LPS outer core loci with different Heddleston serotypes. Genome sequences of Pasteurella multocida strains HB03 (type A), HN06 (type D) and HN07 (type F) were submitted to NCBI under accession numbers CP003328 [14], CP003313 [8]; CP007040. Whole genome sequencing of Pasteurella multocida HN04 (type B) was also completed but its genome was uncommitted. The map was generated via Easyfig 2.1. |
| 图选项 |
根据已有的报道[16-18, 20-21],在多杀性巴氏杆菌Heddleston 1、2、3、4、5、8、10、11、12、13、14及15型菌株的外核编码簇中均存在一个高度保守的基因hptE,该基因与fpg相邻,其所编码的庚糖基转移酶(HptE)对于多杀性巴氏杆菌LPS内核多糖和外核多糖之间的相互连接是必需的。由于多杀性巴氏杆菌LPS的外核编码簇位于2个保守的基因priA和fpg之间,并且不同Heddleston血清型菌株外核编码簇的长短不一(图 2),因此,可以分别基于priA和hptE作为起始和终止位点设计引物建立PCR方法扩增这2个基因的中间区域,从而实现对上述Heddleston血清型的多杀性巴氏杆菌进行快速诊断的目的。正是基于这一原理,Tsai等建立了PCR-限制性片段长度多态性(PCR-RFLP)方法,并且利用该方法对禽霍乱多杀性巴氏杆菌分离株进行了分型,得到了较为稳定的结果[23]。然而,由于hptE基因只存在于L1 (对应Heddleston 1和14型菌株),L2 (对应Heddleston 2和5型菌株),L3 (对应Heddleston 3和4型菌株),L6 (对应Heddleston 10、11、12和15型菌株)与L7 (对应Heddleston 8和13型菌株)型多杀性巴氏杆菌中(图 2),因此利用该分型方法只能对16种Heddleston血清型中的12种进行快速诊断,无法诊断Heddleston 6、7、9、16型菌株。在此背景下,Harper等选取了外核编码簇上不同的基因位点进行引物设计(图 2,表 3)建立起了一种能够对全部8种LPS基因型进行鉴定的多重PCR方法,并且该方法与质谱分析结果的匹配度非常好,具有较高的可靠性[22],由于这8种基因型与16种Heddleston血清型具有一定的对应关系,因此该方法能够对全部16种Heddleston血清型的多杀性巴氏杆菌进行快速诊断,但是该方法所得出的结果不能与经典的Heddleston分型方法形成精确的一一对应关系,加之由于该方法报道的时间较晚,临床上还未见利用该方法开展多杀性巴氏杆菌流行病学调查的报道。即便如此,该方法对于临床上开展多杀性巴氏杆菌的分子流行病学调查依然具有十分重要的价值。
表 3. 用于检测多杀性巴氏杆菌脂多糖基因型的引物* Table 3. Primers for determination of Pasteurella multocida LPS genotypes*
| Genotype | Sequence (5′→3′) | Products/bp |
| L1 | ACATTCCAGATAATACACCCG | 1307 |
| ATTGGAGCACCTAGTAACCC | ||
| L2 | CTTAAAGTAACACTCGCTATTGC | 810 |
| TTTGATTTCCCTTGGGATAGC | ||
| L3 | TGCAGGCGAGAGTTGATAAACCATC | 474 |
| CAAAGATTGGTTCCAAATCTGAATGGA | ||
| L4 | TTTCCATAGATTAGCAATGCCG | 550 |
| CTTTATTTGGTCTTTATATATACC | ||
| L5 | AGATTGCATGGCGAAATGGC | 1175 |
| CAATCCTCGTAAGACCCCC | ||
| L6 | TCTTTATAATTATACTCTCCCAAGG | 668 |
| AATGAAGGTTTAAAAGAGATAGCTGGAG | ||
| L7 | CCTATATTTATATCTCCTCCCC | 931 |
| CTAATATATAAACCATCCAACGC | ||
| L8 | GAGAGTTACAAAAATGATCGGC | 255 |
| TCCTGGTTCATATATAGGTAGG | ||
| *: cited from reference [1]. | ||
表选项
3 基于管家基因的多位点序列分型法(MLST) 多位点序列分型法(Multilocus sequence typing,MLST)是一种基于核酸序列测定对细菌进行分型的方法。其基本原理是基于一定数目的管家基因(一般是7个)的序列组合从而确定目标细菌的序列型(Sequence type,ST)[24]。基于特定的引物确定好管家基因的序列后提交至专门的MLST在线数据库进行比对后即可快速地确定靶细菌的ST型。目前用于界定多杀性巴氏杆菌ST型的在线数据库主要有2个(参见Pasteurella multocida MLST Databases网站:http://pubmlst.org/pmultocida/):Multiple host MLST 数据库由英国格拉斯哥大学(University of Glasgow)开发,该数据库可以界定不同宿主(包括牛、羊、猪、禽)来源的多杀性巴氏杆菌的ST型[25],目前应用较为广泛;RIRDC MLST 数据库则由澳大利亚动物研究所(Animal Research Institute)开发,主要是针对禽源多杀性巴氏杆菌进行ST型界定[26]。除此之外,这2个数据库所匹配的部分管家基因也存在一定的差异。Multiple host MLST数据库所匹配的7个管家基因为adk(腺苷酸环化酶基因)、aroA(5-烯醇丙酮酰莽草酸-3-磷酸合成酶基因)、deoD(嘌呤核苷酸磷酸化酶基因)、gdhA(谷氨酸脱氢酶基因)、g6pd(葡萄糖-6磷酸脱氢酶基因)、mdh(苹果酸脱氢酶基因)、pgi(磷酸葡萄糖异构酶基因)(相应的扩增及测序引物序列参见Pasteurella multocida MLST Databases网站),而RIRDC MLST 数据库所匹配的7个管家基因为adk、est(酯酶基因)、pmi(6-磷酸-甘露糖异构酶基因)、zwf(即g6pd)、mdh、gdhA、pgi(相应的扩增及测序引物序列参见Pasteurella multocida MLST Databases网站)。这些管家基因均为保障多杀性巴氏杆菌生存所必需的代谢相关基因。对于任意一株ST型未知的多杀性巴氏杆菌,只需要根据Pasteurella multocida MLST Databases上所给定的引物序列及PCR扩增体系和条件对与数据库所匹配的上述管家基因进行扩增和测序,然后将序列提交至对应的数据库进行检索即可快速界定该菌株的序列型。
MLST技术相对于基于多杀性巴氏杆菌荚膜编码簇或LPS外核编码簇所建立的PCR分型技术更加方便快捷,但是其所需的成本太高。加之,由于受到传统的Carter分型系统和Heddleston分型系统的影响,因此MLST技术在当前针对多杀性巴氏杆菌的临床研究中并不常用。Sarangi等利用该方法对在印度流行的多杀性巴氏杆菌展开了分子流行病学调查,揭示了在印度流行的多杀性巴氏杆菌的10种不同ST型,其中ST 277、 ST 280、ST 281、ST 282是首次被发现[27]。Petersen等则针对55株与出血性败血症相关的多杀性巴氏杆菌进行MLST分型发现与该病相关的菌株主要为ST122(50株)[28]。Wang等针对我国江苏地区的禽源多杀性巴氏杆菌流行菌株开展了MLST分型调查发现与禽霍乱相关的40株多杀性巴氏杆菌全部属于ST129[29]。Petersen和Wang等的研究均说明多杀性巴氏杆菌的序列型与疾病之间也可能存在一定的相关性。因此,该方法对于多杀性巴氏杆菌的临床流行研究对于了解该病原体与疾病的关系同样具有十分重要的意义。
4 展望 多杀性巴氏杆菌是临床上危害比较严重的一种病原菌,其感染宿主十分广泛,由其感染导致的疾病种类及临床表现也多种多样。很多已有的研究均表明,多杀性巴氏杆的血清型或序列型与其致病性存在一定的联系。因此,临床上针对多杀性巴氏杆菌分离株快速开展分型鉴定对于了解其感染和致病规律,建立细菌流行病学资料库具有十分重要的意义。在临床上开展多杀性巴氏杆菌血清学分型方法操作繁琐、技术难度大的背景下,基于分子生物学手段建立的分子分型方法为临床上快速开展多杀性巴氏杆菌的分型提供了一条更加便捷的途径,它们的应用极大地提高了临床应用过程中多杀性巴氏杆菌的分型效率。虽然在现有的技术层面下,多重PCR方法和MLST分型方法依然存在诸如无法界定“未定型多杀性巴氏杆菌”和成本过高等不足的地方,但是分子分型方法相对于传统的血清学分型方法而言依然具有十分明显的优势,这类分型方法特别是基于荚膜编码区建立的多重PCR分型法已经成为当前临床上开展多杀性巴氏杆菌分离鉴定工作最为常用的方法,而多位点序列分型法(MLST)在临床上也得到了广泛的应用。随着基于脂多糖外核编码基因簇所建立的PCR分型方法的提出,该方法也将逐步地广泛应用。
参考文献
| [1] | Wilkie IW, Harper M, Boyce JD, Adler B. Pasteurella multocida: diseases and pathogenesis//Aktories K, Orth JHC, Adler B. Pasteurella multocida. Berlin Heidelberg: Springer, 2012: 1-22. |
| [2] | Wilson BA, Ho M. Pasteurella multocida: from zoonosis to cellular microbiology.Clinical Microbiology Reviews, 2013, 26(3): 631–655DOI:10.1128/CMR.00024-13. |
| [3] | Carter GR. Studies on Pasteurella multocida. I. A hemagglutination test for the identification of serological types.American Journal of Veterinary Research, 1955, 16(60): 481–484. |
| [4] | Heddleston KL, Gallagher JE, Rebers PA. Fowl cholera: gel diffusion precipitin test for serotyping Pasteurella multocida from avian species.Avian Diseases, 1972, 16(4): 925–936DOI:10.2307/1588773. |
| [5] | Kubatzky KF. Pasteurella multocida and immune cells//Aktories K, Orth JHC, Adler B. Pasteurella multocida. Berlin Heidelberg: Springer, 2012: 53-72. |
| [6] | Harper M, Boyce JD, Adler B. Pasteurella multocida pathogenesis: 125 years after Pasteur.FEMS Microbiology Letters, 2006, 265(1): 1–10DOI:10.1111/fml.2006.265.issue-1. |
| [7] | Peng Z, Liang W, Liu WJ, Wu B, Tang B, Tan C, Zhou R, Chen HC. Genomic characterization of Pasteurella multocida HB01, a serotype A bovine isolate from China.Gene, 2016, 581(1): 85–93DOI:10.1016/j.gene.2016.01.041. |
| [8] | Liu WJ, Yang MJ, Xu ZF, Zheng HJ, Liang W, Zhou R, Wu B, Chen HC. Complete genome sequence of Pasteurella multocida HN06, a toxigenic strain of serogroup D.Journal of Bacteriology, 2012, 194(12): 3292–3293DOI:10.1128/JB.00215-12. |
| [9] | Townsend KM, Boyce JD, Chung JY, Frost AJ, Adler B. Genetic organization of Pasteurella multocida cap loci and development of a multiplex capsular PCR typing system.Journal of Clinical Microbiology, 2001, 39(3): 924–929DOI:10.1128/JCM.39.3.924-929.2001. |
| [10] | Tang XB, Zhao ZQ, Hu JY, Wu B, Cai XW, He QG, Chen HC. Isolation, antimicrobial resistance, and virulence genes of Pasteurella multocida strains from swine in China.Journal of Clinical Microbiology, 2009, 47(4): 951–958DOI:10.1128/JCM.02029-08. |
| [11] | Peng Z, Wang G, Wu GJ, Wang HN, Guo L, Tang XB, Wu B, Chen HC. Isolation and identification of Pasteurella multocida from swine.Chinese Veterinary Science, 2015, 45(9): 943–951(in Chinese).彭忠, 王庚, 吴广敬, 王豪男, 郭龙, 汤细彪, 吴斌, 陈焕春. 猪源多杀性巴氏杆菌的分离与鉴定.中国兽医科学, 2015, 45(9): 943–951. |
| [12] | Choi C, Kim B, Cho WS, Kim J, Kwon D, Cheon DS, Chae C. Capsular serotype, toxA gene, and antimicrobial susceptibility profiles of Pasteurella multocida isolated from pigs with pneumonia in Korea.Veterinary Record, 2001, 149(7): 210–212DOI:10.1136/vr.149.7.210. |
| [13] | Davies RL, MacCorquodale R, Baillie S, Caffrey B. Characterization and comparison of Pasteurella multocida strains associated with porcine pneumonia and atrophic rhinitis.Journal of Medical Microbiology, 2003, 52(1): 59–67DOI:10.1099/jmm.0.05019-0. |
| [14] | Liang WL, Peng Z, Liu WJ, Xu ZF, Chen HC, Wu B. Complete genome sequence of P. multocida serotype A strain HB03, isolated from swine//Proceedings of the 23rd IPVS congress. Cancun, Mexico: IPVS, 2014,2: 30. |
| [15] | Harper M, Cox AD, Adler B, Boyce JD. Pasteurella multocida lipopolysaccharide: the long and the short of it.Veterinary Microbiology, 2011, 153(1/2): 109–115. |
| [16] | Harper M, Michael FS, John M, Vinogradov E, Adler B, Boyce JD, Cox AD. Pasteurella multocida Heddleston serovars 1 and 14 express different lipopolysaccharide structures but share the same lipopolysaccharide biosynthesis outer core locus.Veterinary Microbiology, 2011, 150(3/4): 289–296. |
| [17] | Michael FS, Harper M, Parnas H, John M, Stupak J, Vinogradov E, Adler B, Boyce JD, Cox AD. Structural and genetic basis for the serological differentiation of Pasteurella multocida Heddleston serotypes 2 and 5.Journal of Bacteriology, 2009, 191(22): 6950–6959DOI:10.1128/JB.00787-09. |
| [18] | Harper M, Michael FS, Vinogradov E, John M, Steen JA, van Dorsten L, Boyce JD, Adler B, Cox AD. Structure and biosynthetic locus of the lipopolysaccharide outer core produced by Pasteurella multocida serovars 8 and 13 and the identification of a novel phospho-glycero moiety.Glycobiology, 2013, 23(3): 286–294DOI:10.1093/glycob/cws154. |
| [19] | Harper M, Michael FS, Vinogradov E, John M, Boyce JD, Adler B, Cox AD. Characterization of the lipopolysaccharide from Pasteurella multocida Heddleston serovar 9: identification of a proposed bi-functional dTDP-3-acetamido-3,6-dideoxy-α-d-glucose biosynthesis enzyme.Glycobiology, 2012, 22(3): 332–344DOI:10.1093/glycob/cwr147. |
| [20] | Harper M, Michael FS, John M, Vinogradov E, Steen JA, van Dorsten L, Steen JA, Turni C, Blackall PJ, Adler B, Cox A, Boyce JD. Pasteurella multocida Heddleston serovar 3 and 4 strains share a common lipopolysaccharide biosynthesis locus but display both inter- and intrastrain lipopolysaccharide heterogeneity.Journal of Bacteriology, 2013, 195(21): 4854–4864DOI:10.1128/JB.00779-13. |
| [21] | Harper M, Michael FS, John M, Steen J, van Dorsten L, Parnas H, Vinogradov E, Adler B, Cox AD, Boyce JD. Structural analysis of lipopolysaccharide produced by Heddleston serovars 10, 11, 12 and 15 and the identification of a new Pasteurella multocida lipopolysaccharide outer core biosynthesis locus, L6.Glycobiology, 2014, 24(7): 649–659DOI:10.1093/glycob/cwu030. |
| [22] | Harper M, John M, Turni C, Edmunds M, St Michael F, Adler B, Blackall PJ, Cox AD, Boyce JD. Development of a rapid multiplex PCR to genotype Pasteurella multocida strains using the lipopolysaccharide outer core biosynthesis locus.Journal of Clinical Microbiology, 2014DOI:10.1128/JCM.02824-14. |
| [23] | Tsai YC, Shien JH, Wu JR, Shieh HK, Chang PC. Polymerase chain reaction-restriction fragment length polymorphism analysis of the genes involved in the biosynthesis of the lipopolysaccharide of Pasteurella multocida.Journal of Veterinary Diagnostic Investigation, 2011, 23(3): 543–546DOI:10.1177/1040638711404145. |
| [24] | Matsumura Y. Multilocus sequence typing (MLST) analysis.The Japanese Journal of Clinical Pathology, 2013, 61(12): 1116–1122. |
| [25] | Jolley KA, Maiden MCJ. BIGSdb: Scalable analysis of bacterial genome variation at the population level.BMC Bioinformatics, 2010, 11(1): 595DOI:10.1186/1471-2105-11-595. |
| [26] | Jolley KA, Chan MS, Maiden MCJ. mlstdbNet-distributed multi-locus sequence typing (MLST) databases.BMC Bioinformatics, 2004, 5(1): 86DOI:10.1186/1471-2105-5-86. |
| [27] | Sarangi LN, Thomas P, Gupta SK, Kumar S, Viswas KN, Singh VP. Molecular epidemiology of Pasteurella multocida circulating in India by multilocus sequence typing.Transboundary and Emerging Diseases, 2016, 63(2): e286–e292DOI:10.1111/tbed.2016.63.issue-2. |
| [28] | Petersen A, Bisgaard M, Townsend K, Christensen H. MLST typing of Pasteurella multocida associated with haemorrhagic septicaemia and development of a real-time PCR specific for haemorrhagic septicaemia associated isolates.Veterinary Microbiology, 2014, 170(3/4): 335–41. |
| [29] | Wang YH, Zhu J, Lu CP, Wu B, Liu DD, Hang W, Liu HM, Liu XF. Evidence of circulation of an epidemic strain of Pasteurella multocida in Jiangsu, China by multi-locus sequence typing (MLST).Infection, Genetics and Evolution, 2013, 20: 34–38DOI:10.1016/j.meegid.2013.07.027. |
