杨绍梅1, 郭磊2, 班睿1

 , 谢希贤2
, 谢希贤21. 天津大学化工学院, 系统生物工程教育部重点实验室, 天津 300072;
2. 天津科技大学生物工程学院, 代谢控制发酵技术国家地方联合工程实验室, 天津 300457
收稿日期: 2015-04-22; 修回日期: 2015-04-22
资助课题: 国家"863计划"项目(2012AA02A701)
通讯作者: 班睿,Tel/Fax:+86-22-87402179;E-mail:banrui@tju.edu.cn
摘要: 【目的】探究磷酸核糖焦磷酸(PRPP)合成酶(prs)和氨甲酰磷酸合成酶(pyrAA/pyrAB)的点突变,以及异源5'-核苷酸酶(sdt1)的过表达,对枯草芽孢杆菌尿苷生物合成的影响。【方法】依据推断的变构位点,分别在prs基因和pyrAB基因编码序列中引入点突变;将点突变的prs基因在染色体xylR位点整合表达,pyrAB基因则在染色体原位被修饰; sdt1基因在染色体sacB位点整合过表达。通过对重组菌摇瓶发酵液中尿苷、胞苷和尿嘧啶的分析,表征相关基因修饰对尿苷合成的影响。【结果】在PRPP合成酶中引入Asn120Ser、Leu135Ile和Glu52Gly或Val312Ala点突变,分别导致尿苷积累量提高67%和96%。进一步在氨甲酰磷酸合成酶中引入Ser948Phe、Thr977Ala和Lys993Ile点突变,导致尿苷积累量又增加了182%,达到6.97 g/L。在此基础上,过表达异源5'-核苷酸酶,导致尿苷产量增加17%,达到8.16 g/L。【结论】PRPP合成酶和氨甲酰磷酸合成酶的酶活或反馈抑制调节机制,是限制尿苷过量合成的重要因素。PRPP合成酶的Asn120Ser和Leu135Ile点突变,以及氨甲酰磷酸合成酶的Ser948Phe、Thr977Ala和Lys993Ile点突变,能够显著促进尿苷合成。PRPP合成酶附加的Glu52Gly或Val312Ala点突变,有利于尿苷合成。异源的嘧啶专一性5'-核苷酸酶的引入,也对尿苷的合成有明显的促进作用。
关键词: PRPP合成酶氨甲酰磷酸合成酶5'-核苷酸酶尿苷合成枯草芽孢杆菌
Effect of key-gene modification on uridine biosynthesis in Bacillus subtilis
Shaomei Yang1, Lei Guo2, Rui Ban1

 , Xixian Xie2
, Xixian Xie21. Key Laboratory of Systems Biotechnology, Ministry of Education, School of Chemical Engineering and Technology, Tianjin University, Tianjin 300072, China;
2. National and Local United Engineering Laboratory of Metabolic Control Fermentation Technology, College of Biotechnology, Tianjin University of Science and Technology, Tianjin 300457, China
Abstract:[Objective] We studied several crucial factors influencing the uridine biosynthesis in Bacillus subtilis, including mutations of phosphoribosylpyrophosphate synthetase(PRPP synthetase)(prs) and carbamyl phosphate synthetase(pyrAA/pyrAB), and overexpression of heterologous 5'-nucleotidase(sdt1).[Methods] According to the inferred allosteric sites, we introduced point mutation into coding sequences of prs and pyrAB. The mutated prs gene was integratedly expressed in the xylR locus of the chromosome and the pyrAB gene was modified in-situ. The sdt1 gene was overexpressed in the saB locus of the chromosome. The effect of the genetic modification on uridine biosynthesis was characterized by the analysis of uridine, cytidine and uracil in the fermentation broth.[Results] The mutations of Asn120Ser, Leu135Ile, Glu52Gly or Val312Ala on PRPP synthase resulted in an increase of uridine production by 67% and 96%, respectively. The mutations of Ser948Phe, Thr977Ala and Lys993Ile on carbamyl phosphate synthase resulted in a 182% increase of uridine yield to 6.97 g/L. The overexpression of heterologous 5'-nucleotidase resulted in a 17% increase of uridine yield to 8.16 g/L.[Conclusion] The activity and regulation mechanism of PRPP synthase and carbamyl phosphate synthase was an important factor to limit the excessive synthesis of uridine. Asn120Ser and Leu135Ile mutations of PRPP synthase and Ser948Phe, Thr977Ala and Lys993Ile mutations of carbamyl phosphate synthase will facilitate the biosynthesis of uridine. The additional Glu52Gly and Val312Ala mutations of PRPP synthase were beneficial for uridine biosynthesis. The reaction from UMP to uridine also limited the biosynthesis of uridine in B. subtilis.
Key words: PRPP synthetasecarbamyl phosphate synthetase5'-nucleotidaseuridine biosynthesisBacillus subtilis
尿苷(Uridine)化学名为1-β-D-呋喃核糖苷尿嘧啶,作为合成某些抗病毒或抗肿瘤药物的原料,在制药工业中有一定的需求[1]。枯草芽孢杆菌(Bacillus subtilis)具有嘧啶核苷酸从头合成途径,以及以尿苷单磷酸(UMP)为前体物的尿苷生物合成途径[2](图1)。但野生型B. subtilis在一般培养条件下,发酵液中不能积累可检测量的尿苷。Yuta-ka Tsunemi等采用诱变筛选尿嘧啶结构类似物抗性突变株的育种方法,成功地获得了过量合成尿苷的B. subtilis突变株[3, 4]。但国内研究者采用相同育种方法筛选出的抗性突变株,尿苷产量远低于国外文献报道水平[5]。基因工程技术已经应用于尿苷生产菌株的遗传育种,朱晖等采用基因操作方法,通过缺失pyr操纵子转录调节蛋白编码基因(pyrR)、嘧啶核苷磷酸化酶基因(pdp)和嘧啶透过酶基因(nupC),过表达磷酸核糖焦磷酸合成酶基因(prs),构建了产尿苷重组菌株B. subtilis TD231-1,尿苷产量为1.48 g/L[6]。
 |
| 图1 枯草芽孢杆菌内尿苷生物合成路径 Figure 1 The uridine biosynthesis pathway of B. subtilis. |
| 图选项 |
磷酸核糖焦磷酸(PRPP)是UMP从头合成途径的主要前体物,其胞内水平直接影响UMP的合成。因此,PRPP合成酶(EC 2.7.6.1)很可能是影响尿苷生物合成的关键酶。PRPP合成酶被prs基因编码,其表达调控机制不明[7]。PRPP合成酶是变构酶,其活性受胞内ADP或GDP水平的反馈抑制[8, 9]。有关B. subtilis的PRPP合成酶反馈抑制的变构调节位点(allosteric site),目前还没有明确的实验信息。但对人PRPP合成酶(PRS1)[10, 11],以及解淀粉芽孢杆菌(Bacillus amyloliquefaciens)PRPP合成酶的研究[12],为推断B. subtilis的PRPP合成酶具体的变构位点提供了参考信息。
氨甲酰磷酸合成酶(EC 6.3.5.5)催化UMP从头合成途径的第一步反应,推测也是影响UMP及尿苷合成的关键酶。B. subtilis的氨甲酰磷酸合成酶由pyr操纵子中的pyrAA/pyrAB基因编码,pyr操纵子的表达受UMP反馈阻遏,调节蛋白为PyrR[13]。同时,氨甲酰磷酸合成酶也是变构酶,其催化亚基由pyrAA编码,调节亚基由pyrAB编码,而其活性受UMP的反馈抑制[14],但具体的变构位点也不明确。对E. coli氨甲酰磷酸合成酶[15, 16, 17],以及对B. amyloliquefaciens氨甲酰磷酸合成酶点突变的研究[18],为推断B. subtilis氨甲酰磷酸合成酶的变构调节位点,提供了有价值的参考依据。
UMP的5′-磷酸基团水解生成尿苷,这是尿苷合成的最后一步反应。但是,在B. subtilis中,尚未发现催化这步反应的专一性5′-核苷酸酶,推测可能是由非专一性的磷酸酶催化[4]。酿酒酵母(Saccharomyces cerevisiae)中的SDT1蛋白,是嘧啶核苷酸专一性的5′-核苷酸酶,过量表达SDT1可导致菌株对嘧啶结构类似物的敏感性降低[19, 20]。将SDT1引入B. subtilis,以弥补其缺乏专一性5′-核苷酸酶的缺陷,可能会有利于尿苷的生物合成。
本研究以产尿苷的重组B. subtilis TD131为出发菌,对其prs和pyrAA/pyrAB基因进行了修饰,过表达了异源的嘧啶核苷酸专一性5′-核苷酸酶,考察了PRPP合成酶和氨甲酰磷酸合成酶点突变的效应,以及过表达5′-核苷酸酶对尿苷生物合成的影响;并对影响尿苷合成的主要代谢因素进行了分析。
1 材料和方法1.1 材料1.1.1 菌株与质粒:表1为本实验所使用的菌株和质粒。
表1 实验所用的菌株和质粒Table 1 The strains and plasmids uesd in the study
| Strains and plasmids | Genotype | Source |
| B. subtilis TD131 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC | stored in this lab |
| B. subtilis TD231-1m | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prs-DN*-cat-araR | stored in this lab |
| B. subtilis TD231-1 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prs | stored in this lab |
| B. subtilis TD232-1 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnl | this study |
| B. subtilis TD232-2 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnld | this study |
| B. subtilis TD232-3 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnle | this study |
| B. subtilis TD232-4 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnlv | this study |
| B. subtilis TD234m | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnle, ΔpyrAB::cat | this study |
| B. subtilis TD234-8 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnle, pyrABttkn | this study |
| B. subtilis TD234-9 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnle, pyrABttk | this study |
| B. subtilis TD234-10 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnle, pyrABttkm | this study |
| B. subtilis TD246 | trpC2, ΔaraR::Para-neo, Δhom, Δcdd, ΔpyrR, Δpdp-nupC, ΔxylR::PAE-prsnle, pyrABttk, ΔsacB::Pcdd-sdt1 | This study |
| B. subtilis LXZm | trpC2, ΔaraR::Para-neo, ΔsacB::Pcdd-ribA-DN*-cat-araR | stored in this lab |
| S. cerevisiae G5 | W303-1A, Δtpk1, Δtpk2, Δtpk3, Δyak1, Δpde1 | stored in this lab |
| pC194 | Cmr | stored in this lab |
| Cmr: chloromycetin resistance; DN*: the fragment used for homologous recombination. | ||
表选项
1.1.2 主要培养基:(1) LB培养基(g/L):蛋白胨10,NaCl 10,酵母抽提物5,pH 7.5,用于B. subtilis的一般培养,固体培养基加入15 g的琼脂A,需要时加入新霉素15 μg/mL,或氯霉素6 μg/mL。(2)基本培养基(g/L):葡萄糖5.0,(NH4)2SO4 2.0,柠檬酸钠1.0,MgSO4·7H2O 0.2,K2HPO4 4.0,KH2PO4 6.0,酸水解酪素0.4,L-色氨酸0.05,pH 7.2,需要时补加尿嘧啶50 μg/mL。(3)种子培养基(g/L):葡萄糖20.0,豆粕水解液20 mL,酵母粉10.0,NaCl 2.5,MgSO4·7H2O 1.0,KH2PO4 1.0,谷氨酸钠5.0,苏氨酸/甲硫氨酸/异亮氨酸0.03,pH 6.7–7.0。(4)发酵培养基(g/L):葡萄糖80,豆粕水解液30 mL,酵母粉15,玉米浆20 mL,(NH4)2SO4 15,MgSO4·7H2O 8.0,KH2PO4 2.5,谷氨酸钠15,苏氨酸/甲硫氨酸/异亮氨酸0.03,VB3,5,12 0.1 mg,pH 6.7–7.0。
1.1.3 主要试剂:Taq酶购自北京索莱宝科技有限公司;Hifi DNA聚合酶购自北京全式金生物技术有限公司;dNTPs购自Thermo Scientific公司;DNA序列合成和测序由北京奥科鼎盛生物科技有限公司完成;尿苷和胞苷标准品购自Sigma公司;其余生化试剂为进口或国产分析纯试剂。
1.2 DNA操作所有DNA片段都来自PCR扩增或合成;用于转化的DNA片段均采用重叠PCR方法拼接形成[21]。B. subtilis感受态细胞的制备及转化采用Spizizen方法[22, 23]。表2为本实验所用的PCR引物及合成序列。
表2 引物及合成序列Table 2 Primers and synthetic sequences
| Primers | Primer sequence (5′→ 3′) |
| prs-U1 | CAACAGAGCCAAGCAGAC |
| prs-U2 | CTTGTAGACTGGATGATGTAAC |
| prs-D1 | AACTGGCTGACCGACTAA |
| prs-D2 | AGGATGCGATTCAATTATGC |
| prs-1 | GTTACATCATCCAGTCTACAAG |
| prs-2 | TTAGTCGGTCAGCCAGTT |
| prs-CX1 | TCCGCCGATTACTTCTTG |
| prs-CX2 | CCATGTCACTATTGCTTCAG |
| pyrAB-U1 | GTGTGATGGTGCTTGGTT |
| pyrAB-U2 | CGTAGTTCGGGATTTGAATAC |
| pyrAB-D1 | GGAATAATCGGTCAGGATGG |
| pyrAB-D2 | GGATGTCTCACTTTCGTTTG |
| pyrAB-M1 | GTATTCAAATCCCGAACTACG |
| pyrAB-M2 | CCATCCTGACCGATTATTCC |
| pyrAB-C1 | CGCTACGCTCAAATCCTT |
| pyrAB-C2 | GGCAGGTTAGTGACATTAGA |
| pyrAB-U2q | TAAAGGATTTGAGCGTAGCGAGAGACTGAGGCGGATAA |
| pyrAB-D1q | TTCTAATGTCACTAACCTGCCTGGTGTTGCCTGCCTAA |
| pyrAB-CX1 | AGAGGAACTGCTTCATTACA |
| pyrAB-CX2 | GCTGGTTAGAACATACTGTC |
| sdt-U1 | CTCCTCCAGCAAGATGATT |
| sdt-Cdd2 | GTGTAAATTCCTCCCTTACCT |
| sdt1q | AGGTAAGGGAGGAATTTACACATGACTGTTGAATATACTGC |
| sdt2 | AATTGCCTTGTCTGGTTTG |
| sdt-D1q | CAAACCAGACAAGGCAATTCAGAGGATTCTACGCAGAC |
| sdt-G2 | GGAGTCAGTGAACAGGTAC |
| sdt-CX1 | GACAAACATCACGCTCTTG |
| sdt-CX2 | CGTAGAATCCTCTGAATTGC |
| Fragments | Synthetic sequence (5′→ 3′) |
| P′ | GTTACATCATCCAGTCTACAAGTGACCCCGTTAACGAGCATATTATGGAACTGCTGATTATGGTAGATGC GTTAAAACGCGCTTCTGCAAAAACGATTAACATTGTTATTCCTTATTACGGTTATGCGCGTCAAGACAGA AAAGCAAGATCCCGTGAGCCAATCACAGCTAAACTTTTCGCTAGCCTGCTTGAAACAGCCGGTGCGACTC GTGTGATTGCACTTGACATACATGCGCCGCAAATTCAAGGATTCTTTGATATACCGATTGACCACTTAATG GGTGTTCCGATTTTAGGAGAATATTTTGAAGGCAAAAATCTTGAAGATATCGTCATTGTTTCACCAGACCA TGGCGGTGTGACACGTGCCCGCAAACTGGCTGACCGACTAA |
| M | GTATTCAAATCCCGAACTACGGTTCCGTGCTTTTATTCGTAGCTGATAAGGACAAAGAAGAAGGGCTTGC CATTGCTAAGCGGTTCCACGCGATCGGCTACAACATTTTAGCGACGGAAGGAGCGGCAGGCTACCTGAA AGAAGCTTCCATTCCAGCGAAGGTCGTCGGAATAATCGGTCAGGATGG |
| Bold bases indicate site-directed mutagensis base. | |
表选项
1.3 prs基因的点突变与过表达首先合成一段prs基因的编码序列P′,在其中引入预定的3个碱基突变。然后通过重叠PCR方法,将P′片段与prs基因的其它序列、上游同源臂U、下游同源臂G、选择标记盒CR和内部同源重组片段D,拼接为UPDCRG片段。在UPDCRG片段中,prs基因仅保留编码序列,其启动子被来自噬菌体φ29的A1启动子取代,其前导区被B. sub-tilis aprE基因的前导区替换。最后采用无标记基因修饰方法[24],在受体菌染色体xylR位点,整合点突变的prs基因。即用UPDCRG片段转化受体菌感受态细胞,用氯霉素抗性平板筛选转化子;选择有氯霉素抗性、无新霉素抗性的菌落转接LB培养基,培养4 h,弹出反选择标记盒。上述培养液适当稀释后涂布新霉素抗性平板,筛选具有新霉素抗性而失去氯霉素抗性的转化子,最终经PCR 验证和DNA测序确认。
1.4 pyrAB基因的原位修饰首先通过重叠PCR方法,拼接一段由上游同源臂U、氯霉素抗性基因和下游同源臂D构成的DNA片段。然后转化受体菌,通过同源重组,在染色体pyr操纵子中的pyrAB基因位点,插入氯霉素抗性基因,并替换掉部分pyrAB基因编码序列;通过筛选氯霉素抗性菌株,获得尿嘧啶缺陷型转化子。合成一段pyrAB基因的编码序列M,在其中引入预定的5个碱基突变;再将M片段与U′、D′拼接成U′MD′片段,转化尿嘧啶缺陷株,通过同源重组,M片段将替换掉氯霉素抗性基因;用基本培养基平板筛选转化子,得到原养型的重组菌。
1.5 sdt1基因的过表达选择B. subtilis染色体的sacB位点过表达sdt1基因。首先从S. cerevisiae染色体上扩增sdt1基因的编码序列S片段,从B. subtilis LXZm菌株染色体上扩增出含有上游同源臂U、启动子Pcdd及前导区的UPcdd片段,以及含有下游同源臂G、选择标记盒CR和内部同源重组片段D的DCRG片段;然后通过重叠PCR方法,拼接成UPcdd-SDCRG片段。用UPcdd-SDCRG片段转化受体菌感受态细胞,采用相同的无标记基因修饰方法,获得Pcdd-sdt1基因在sacB位点整合的重组菌株。
1.6 发酵实验及发酵产物分析1.6.1 摇瓶发酵:挑取新活化的平板单菌落接入30 mL种子培养基中,200 r/min、37 °C振荡培养10 h。按10%接种量转接30 mL发酵培养基,37 °C、200 r/min振荡培养72 h。
1.6.2 生物量的测定:每隔12 h取发酵液0.5 mL,13000 r/min离心1 min沉淀细胞,洗涤后重悬、稀释,测定菌悬液的OD600值。
1.6.3 尿苷、胞苷和尿嘧啶的测定:取发酵液离心后的上清液,稀释10倍后,用0.22 μm微孔滤膜过滤。采用Agilent 1200 HPLC定量测定尿苷、胞苷及尿嘧啶,色谱条件:色谱柱为phenomenex Gemini 5u C18 110A;流动相为乙腈:水=2:98;流速1.0 mL/min;柱温30 °C;进样量10 μL;紫外检测器波长:270 nm。
2 结果和分析2.1 PRPP合成酶点突变菌株构建及对尿苷合成的影响2.1.1 PRPP合成酶点突变菌株的构建:研究显示,PRS1的Asn114Ser和Leu129Ile点突变,以及B. amyloliquefaciens PRPP合成酶的Asn120Ser和Leu135Ile点突变,都有抗反馈抑制效应。使用DNAMAN软件比对分析,显示B. subtilis PRPP合成酶与B. amyloliquefaciens PRPP合成酶的同源性为95.90%,因此推断B. subtilis的PRPP合成酶相对应的变构位点可能为Asn120和Leu135。因此,设计在B. subtilis的PRPP合成酶中,也引入Asn120Ser 和Leu135Ile点突变。所对应编码序列的改变为A359G、C403A、G405A,通过合成一段393 bp的P′片段,而将上述3个碱基点突变引入prs的编码序列。从B. subtilis TD231-1中,用引物prs-U1/prs-U2,扩增出U片段(1474 bp)。从B. subtilis TD231m中,用引物prs-D1/prs-D2扩增出DCRG片段(3800 bp)。将上述3个片段拼接为UPDCRG片段(5627 bp),并转化B. subtilis TD131。经筛选共获得4个转化子,用引物prs-CX1/prs-CX2对prs基因双向测序,结果如表3所示。重组菌B. subtilis TD232-1的prs基因仅带有预定的3个点突变,而其余3株重组菌,除预定点突变外,都带有1个额外的碱基突变,并导致附加的错义突变。
表3 测序结果及菌株Table 3 The sequencing results and strains
| Sequencing strains | Scheduled mutant base | Additional mutant base | Additional mutant amino acid |
| B. subtilis TD232-1 | + | - | - |
| B. subtilis TD232-2 | + | A446T | Asp149Val |
| B. subtilis TD232-3 | + | A155G | Glu52Gly |
| B. subtilis TD232-4 | + | T935C | Val312Ala |
表选项
2.1.2 PRPP合成酶点突变菌株的发酵:以prs基因野生型菌株TD131,以及野生型prs基因过表达菌株TD231-1分别为对照菌,分别对4株prs基因点突变菌株进行摇瓶发酵和发酵产物分析。结果显示,点突变prs基因的过表达,对细胞生长及发酵液中胞苷的积累没有明显影响(数据未显示),对尿苷和尿嘧啶的积累则产生了显著的影响(图2)。在PRPP合成酶中引入的点突变,均导致尿苷积累量显著提高。其中TD232-3和TD232-4菌株的效果更加突出,其摇瓶发酵72 h尿苷的积累量,分别达到2.47 g/L和2.90 g/L,与prs野生型TD131菌株的1.18 g/L相比,分别提高了109%和146%;与野生型prs过表达菌株TD231-1的1.48 g/L相比,分别提高了67%和96%。结果说明,B. subtilis 的PRPP合成酶的Asn120Ser和Leu135Ile点突变能够促进PRPP的合成,PRPP供给的改善促进了尿苷的合成。PRPP合成酶附加的Glu52Gly或Val312Ala点突变,不能确定它们的酶学效应,但这些附加点突变也有利于尿苷合成。
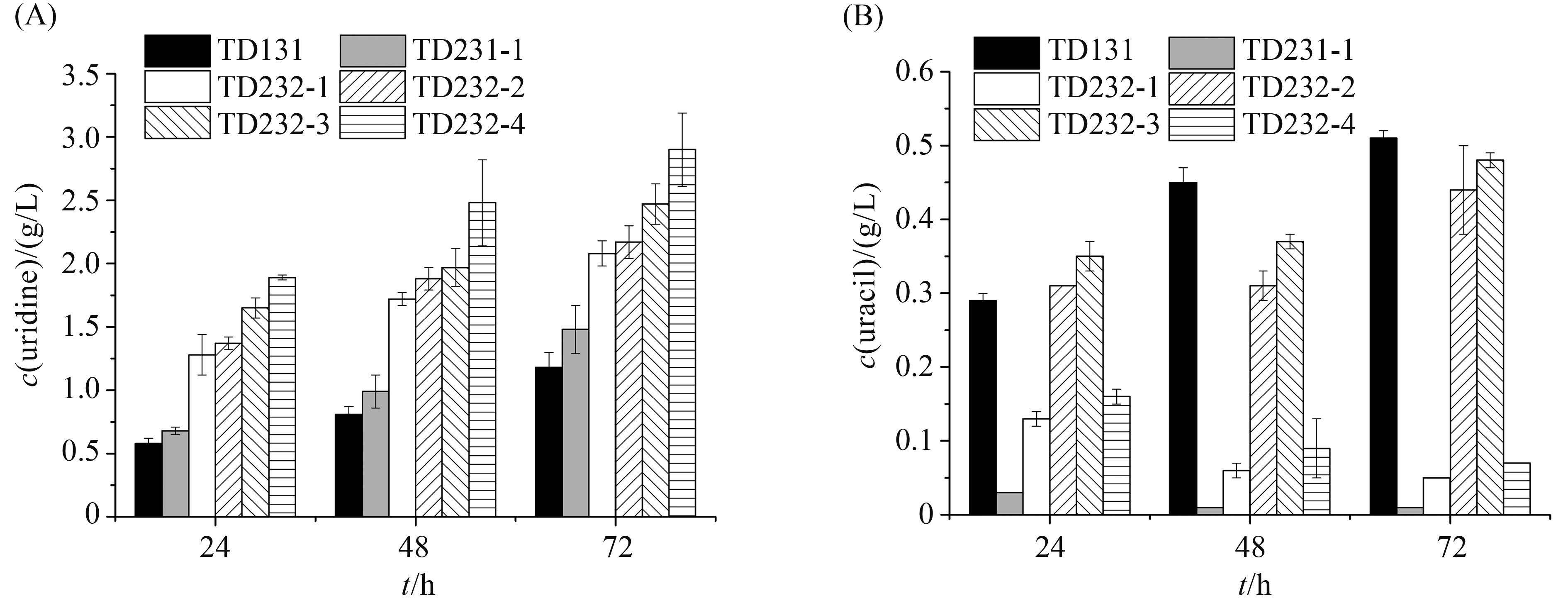 |
| 图2 TD131、TD231-1和TD232-1、2、3、4菌株发酵液中的尿苷(A)及尿嘧啶(B)分析和比较 Figure 2 The analysis and comparison of uridine (A) and uracil (B) of fermentation liquor of strain TD131, TD231-1 and TD232-1, 2, 3, 4. |
| 图选项 |
尿嘧啶是尿苷合成过程的副产物,缺陷嘧啶核苷磷酸化酶(pdp),能够大幅度降低尿嘧啶的生成,同时增加尿苷产量[4, 6]。过表达野生型PRPP合成酶,几乎消除了尿嘧啶的积累。但过表达点突变的PRPP合成酶,对减少尿嘧啶生成的效果并不一致。尿嘧啶的来源,以及尿嘧啶生成与PRPP的关系,还有待研究。
2.2 氨甲酰磷酸合成酶点突变菌株构建及对尿苷合成的影响2.2.1 氨甲酰磷酸合成酶点突变菌株的构建:对E. coli氨甲酰磷酸合成酶的研究显示,其Lys992是与效应物UMP共价结合的氨基酸残基,而其Ser948Phe和Thr977Ala的点突变,也具有抗反馈抑制效应[15, 16, 17]。对B. amyloliquefaciens氨甲酰磷酸合成酶的研究显示,Thr941Phe、Thr970Ala和Lys986Ile点突变,都具有抗反馈抑制效应。使用DNAMAN软件比对分析显示,B. subtilis的氨甲酰磷酸合成酶与B. amyloliquefaciens的同源性为92.81%,据此推断B. subtilis氨甲酰磷酸合成酶相对应的氨基酸残基应该是Thr941、Thr970、Lys986。因此,设计在B. subtilis的氨甲酰磷酸合成酶中引入Thr941Phe、Thr970Ala和Lys986Ile点突变。其编码序列对应的变化为A2821T、C2822T、A2823C、A2908G、A2957T,通过合成一段187 bp的M片段,而将这5个碱基突变引入B. subtilis氨甲酰磷酸合成酶。
从B. subtilis TD232-3中,分别用引物pyrAB-U1/pyrAB-U2q、pyrAB-D1q/pyrAB-D2,扩增出U (718 bp)和D (837 bp)片段。从pC194中,用引物pyrAB-C1/pyrAB-C2扩增出C片段(948 bp)。将这3个片段拼接成UCD片段(2503 bp),转化B. subtilis TD232-3感受态细胞,得到3个氯霉素抗性菌株。用基本培养基和补加50 μg/mL尿嘧啶的基本培养基验证,这3株菌都是尿嘧啶缺陷型,命名为B. subtilis TD234m,作为原位修饰pyrAB基因的受体菌。从TD232-3菌株中,分别用引物pyrAB-U1/pyrAB-U2、pyrAB-D1/pyrAB-D2,扩增U′ (1149 bp)和D′ (974 bp)片段。将U′、M和D′3个片段拼接成U′MD′ (2269 bp)片段,转化B. subtilis TD234m感受态细胞,在基本培养基上筛选得到3个转化子。用引物pyrAB-CX1/ pyrAB-CX2对它们的pyrAB基因进行双向测序,结果如表4。重组菌B. subtilis TD234-9的pyrAB基因仅带有预定的点突变,而其余两株重组菌,除预定点突变外,都带有1个额外的点突变,并导致错义突变。
表4 测序结果及菌株Table 4 The sequencing results and strains
| Sequencing strains | Scheduled mutant base | Additional mutant base | Additional mutant amino acid |
| B. subtilis TD234-8 | + | A3184G | Asn1062Asp |
| B. subtilis TD234-9 | + | - | - |
| B. subtilis TD234-10 | + | A2554G | Met852Val |
表选项
2.2.2 氨甲酰磷酸合成酶点突变菌株的发酵:以出发菌B. subtilis TD232-3为对照菌,分别对3株氨甲酰磷酸合成酶点突变重组菌进行摇瓶发酵实验。发酵结果显示,在氨甲酰磷酸合成酶中引入点突变,显著提高了尿苷积累量(图3-A),对胞苷积累量的提高也有一定影响(图3-B),对细胞生长和尿嘧啶的积累量没有明显影响(数据未显示)。其中没有附加点突变的菌株TD234-9发酵72 h的尿苷积累量为6.97 g/L,与出发菌2.47 g/L相比,提高了182%;胞苷的积累量为0.53 g/L,与出发菌0.35 g/L相比,提高了51%。结果表明,B. subtilis 氨甲酰磷酸合成酶的Thr941Phe、Thr970Ala和Lys986Ile点突变,能够增加UMP合成途径的代谢通量,提高UMP的合成速率,进而显著提高尿苷的合成量,同时对胞苷的合成也有一定的促进作用。氨甲酰磷酸合成酶催化的第一步反应,确实是UMP合成的限速反应。
 |
| 图3 TD232-3和TD234-8、9、10菌株发酵液中的尿苷(A)和胞苷(B)的分析与比较 Figure 3 The analysis and comparison of uridine (A) and cytidine (B) of fermentation liquor of strains TD232-3 and TD234-8, 9, 10. |
| 图选项 |
2.3 异源核苷酸酶的过表达以及对尿苷合成的影响2.3.1 异源核苷酸酶过表达菌株的构建:经过文献搜寻,发现S. cerevisiae的SDT1蛋白(sdt1)是微生物来源的、功能确定的嘧啶核苷酸专一性5′-核苷酸酶。故选择在产尿苷B. subtilis中过表达sdt1基因,以便提高UMP转化为尿苷的反应速率。B. subtilis的cdd基因编码胞苷脱氨酶,其启动子(Pcdd)由3个串联的启动子构成,分别被σE、σB、σA识别[25]。已在实验中观察到Pcdd及前导区具有强表达元件的特性(数据未显示),故选择Pcdd及前导区,用于在染色体sacB基因位点整合表达sdt1基因。以B. subtilis LXZm染色体为模板,用引物sdt-U1/sdt-Cdd2,扩增包括上游同源臂U、Pcdd启动子及前导区在内的UPcdd片段(1310 bp);用引物sdt-D1q/sdt-G2,扩增包括内部同源片段D、选择盒CR及下游同源片段G在内的DCRG片段(3516 bp)。以S. cerevisiae染色体为模板,用引物sdt1q/ sdt2,扩增sdt1的编码序列S(1092 bp)。将这3个片段拼接为UPcdd-SDCRG片段(5918 bp),并转化B. subtilis TD234-9感受态细胞,经过抗性筛选、PCR扩增和引物sdt-CX1/sdt-CX2的双向测序验证,仅得到1个正确的转化子,命名为B. subtilis TD246。
2.3.2 异源核苷酸酶过表达菌株的发酵:以出发菌B. subtilis TD234-9为对照菌,对重组菌TD246 进行摇瓶发酵和产物分析。结果显示(图4),过表达异源的5′-核苷酸酶,显著的提高了尿苷和胞苷在发酵液中的积累量,同时显著降低了尿嘧啶的生成量。摇瓶发酵72 h,TD246菌株的尿苷和胞苷积累量分别为8.16 g/L和1.09 g/L,与出发菌TD234-9的尿苷6.97 g/L、胞苷0.53 g/L相比、分别提高了17%和105%。TD246菌株尿嘧啶积累量仅为0.04 g/L,而出发菌TD234-9则为0.52 g/L。结果表明,来自S. cerevisiae的5′-核苷酸酶(sdt1),被Pcdd启动子及前导区成功的过表达;引入嘧啶核苷酸专一性5′-核苷酸酶,能够显著提高尿苷和胞苷的积累量,同时有效减少尿嘧啶的生成。
 |
| 图4 TD234-9和TD246菌株发酵液中的尿苷(A)、胞苷(B)和尿嘧啶(C)的分析与比较 Figure 4 The analysis and comparison of uridine (A), cytidine (B) and uracil (C) of fermentation liquor of strain TD234-9 and TD246. |
| 图选项 |
3 讨论先前的研究显示,只要pyr操纵子组成型表达,以及嘧啶核苷磷酸化酶(pdp)缺失,B. subtilis就能在发酵液中积累大约400 mg/L左右的尿苷。这一现象说明,细胞内UMP的过量合成是尿苷过量合成的前提;阻断细胞内尿苷主要的转化反应,是尿苷在发酵液中过量积累的必要条件。先前的研究还显示,缺失高丝氨酸脱氢酶(hom),增加L-天冬氨酸的供给;以及过表达prs 基因,提高细胞PRPP的合成能力,可使尿苷在发酵液中的积累量超过1 g/L[6]。这说明L-天冬氨酸和PRPP等UMP合成的主要前体物的供给,是影响UMP过量合成,进而影响尿苷过量合成的另一个重要因素。
在产尿苷的B. subtilis TD131菌株中,进一步过表达带有Asn120Ser和Leu135Ile点突变的PRPP 合成酶,与过表达野生型PRPP合成酶相比,尿苷在发酵液中的积累量提高了40%以上,达到大约2.1 g/L水平。这一结果进一步证实了UMP合成的主要前体物PRPP的供给水平,是限制尿苷过量合成的主要因素之一,同时也为证明B. subtilis PRPP 合成酶的的变构位点与B. amyloliquefaciens 的完全一致,提供了初步的证据。在过表达了突变的PRPP合成酶后,前体物PRPP的供给,可能不再是UMP及尿苷过量合成的限制性因素。在阻断了L-天冬氨酸转化为L-高丝氨酸的代谢后(hom基因缺失),L-天冬氨酸也不再是UMP过量合成的限制性因素。因此,在这种背景下,UMP及尿苷过量合成的主要瓶颈将迁移到UMP从头合成途径的内部。
氨甲酰磷酸合成酶是催化UMP从头合成途径第一步反应的酶,也是一个受UMP反馈抑制的变构酶[14]。由此推断,氨甲酰磷酸合成酶的变构调节机制,可能是限制UMP及尿苷过量合成的另一个关键因素。在氨甲酰磷酸合成酶中引入了Thr941Phe、Thr970Ala和Lys986Ile点突变,结果导致尿苷在发酵液中的积累量大幅度增加(182%),接近7.0 g/L的水平;同时也导致胞苷积累量显著增加(51%)。这一结果表明,B. subtilis氨甲酰磷酸合成酶的Thr941、Thr970和Lys986残基突变,对其活性或反馈抑制的变构调节机制产生了重要的影响,同时也证实了氨甲酰磷酸合成酶催化的反应,是限制UMP及尿苷过量合成的关键因素。但是,氨甲酰磷酸合成酶的突变,所导致的尿苷积累量的提高,并没有达到预想的程度(数倍增加)。推测在UMP从头合成途径中,可能还存在其它限速反应,或者由UMP转化为尿苷的反应,是另一个瓶颈。
在B. subtilis中没有发现催化UMP水解生成尿苷的专一性5′-核苷酸酶,可能由非专一性的磷酸酶催化的这步反应,或许是尿苷合成的限速反应。在B. subtilis中过表达了来自S. cerevisiae的嘧啶核苷酸专一性的5′-核苷酸酶(SDT1),结果导致尿苷的积累量提高了17%,超过8.0 g/L;同时导致胞苷的积累量提高了105%,而尿嘧啶积累量减少了约92%。显然,在B. subtilis中引入SDT1,对于促进UMP和CMP转化为尿苷和胞苷是十分有效的,这步反应也是尿苷过量合成的一个次要显著因素。但是过表达SDT1,对提高尿苷合成的作用有限。说明在这种情况下,胞内UMP水平依然是限制因素;在UMP合成途径中,可能还存在其它的限速反应,这是进一步提高尿苷的合成所需要解决的问题。
过表达SDT1,显著减少了尿嘧啶的生成。尿嘧啶可能来源于UMP的分解,当UMP转化为尿苷或者CMP转化为胞苷的反应,因缺乏专一性5′-核苷酸酶而受到限制时,UMP的胞内水平会有所提高;少量积累的UMP会通过未知的反应转化为尿嘧啶而积累在发酵液中,积累量在0.5 g/L水平左右。当过表达了专一性5′-核苷酸酶SDT1后,加速了UMP到尿苷的转化,降低了UMP的胞内水平,限制了UMP分解生成尿嘧啶的反应,使尿嘧啶在发酵液中的积累量减少了一个数量级,仅为0.05 g/L左右。从这一现象也可以看出,引入5′-核苷酸酶SDT1,对于促进尿苷的合成是十分必要的。
参考文献
| [1] | Wang R. Advances in pyrimidine. Letters in Biotechnology, 2007, 18(3):539-542.(in Chinese) 王锐. 嘧啶核苷的研究进展. 生物技术通讯, 2007, 18(3):539-542. |
| [2] | [2015-04-10]. http://www.genome.jp/kegg-bin/show_pathway? bsu00240. |
| [3] | Tsunemi Y, Asahi S, Doi M. Production of uridine. United States Patent:4880736. 1985-10-18. |
| [4] | Doi M, Asahi S, Tsunemi Y, Akiyama S. Mechanism of uridine production by Bacillus subtilis mutants. Applied Microbiology Biotechnology, 1989, 30(3):234-238. |
| [5] | Cheng YC, Liu KL, Xu QY. The breeding of uridine producing strain. Modern Food Science and Technology, 2007, 23(1):20-22.(in Chinese) 程远超, 刘康乐, 徐庆阳. 尿苷产生菌的选育. 现代食品科技, 2007, 23(1):20-22. |
| [6] | Zhu H, Yang SM, Yuan ZM, Ban R. Metabolic and genetic factors affecting the productivity of pyrimidine nucleoside in Bacillus subtilis. Microbial Cell Factories, 2015, 14(1):54. |
| [7] | Hilden I, Krath BN, Hove-Jensen B. Tricistronic operon expression of the genes gcaD(tms), which encodes Nacetylglucosamine 1-phosphate uridyltransferase, prs, which encodes phosphoribosyl diphosphate synthetase, and ctc in vegetative cells of Bacillus subtilis. Journal of Bacteriology, 1995, 177(24):7180-7284. |
| [8] | Arnvig K, Hove-Jensen B, Switzer RL. Purification and properties of phosphoribosyl-diphosphate synthetase from Bacillus subtilis. European Journal of Biochemistry, 1990, 192(1):195-200. |
| [9] | Eriksen TA, Kadziola A, Bentsen AK, Harlow KW, Larsen S. Structural basis for the function of Bacillus subtilis phosphoribosyl-pyrophosphate synthetase. Nature Structural Biology, 2000, 7(4):303-308. |
| [10] | Zoref E, De Vries A, Sperling O. Mutant feedback-resistant phosphoribosylpyrophosphate synthetase associated with purine overproduction and gout. Phosphoribosylpyrophosphate and purine metabolism in cultured fibroblasts. The Journal of Clinical Investigation, 1975, 56(5):1093-1099. |
| [11] | Becker MA, Smith PR, Taylor W, Mustafi R, Switzer RL. The genetic and functional basis of purine nucleotide feedbackresistant phosphoribosylpyrophosphate synthetase superactivity. The Journal of Clinical Investigation, 1995, 96(5):2133-2141. |
| [12] | Zakataeva NP, Romanenkov DV, Skripnikova VS, Vitushkina MV, Livshits VA, Kivero AD, Novikova AE. Wild-type and feedback-resistant phosphoribosyl pyrophosphate synthetases from Bacillus amyloliquefaciens:purification, characterization, and application to increase purine nucleoside production. Applied Microbiology and Biotechnology, 2012, 93(5):2023-2033. |
| [13] | Bonner ER, D'Elia JN, Billips BK, Switzer RL. Molecular recognition of pyr mRNA by the Bacillus subtilis attenuation regulatory protein PyrR. Nucleic Acids Research, 2001, 29(23):4851-4865. |
| [14] | Nicoloff H, Hubert JC, Bringel F. Carbamoyl-phosphate synthetases(CPS) in lactic acid bacteria and other grampositive bacteria. Lait, 2001, 81(1/2):151-159. |
| [15] | Czerwinski RM, Mareya SM, Raushel FM. Regulatory changes in the control of carbamoyl phosphate synthetase induced by truncation and mutagenesis of the allosteric binding domain. Biochemistry, 1995, 34(42):13920-13927. |
| [16] | Cervera J, Bendala E, Britton HG, Bueso J, Nassif Z, Lusty CJ, Rubio V. Photoaffinity labeling with UMP of lysine 992 of carbamyl phosphate synthetase from Escherichia coli allows identification of the binding site for the pyrimidine inhibitor. Biochemistry, 1996, 35(22):7247-7255. |
| [17] | Delannay S, Charlier D, Tricot C, Villeret V, Piérard A, Stalon V. Serine 948 and threonine 1042 are crucial residues for allosteric regulation of Escherichia coli carbamoylphosphate synthetase and illustrate coupling effects of activation and inhibition pathways. Journal of Molecular Biology, 1999, 286(4):1217-1228. |
| [18] | Fang HT, Liu HY, Chen N, Zhang CL, Xie XX, Xu QY. Sitedirected mutagenesis studies on the uridine monophosphate binding sites of feedback inhibition in carbamoyl phosphate synthetase and effects on cytidine production by Bacillus amyloliquefaciens. Canadian Journal of Microbiology, 2013, 59(6):374-379. |
| [19] | Nakanishi T, Sekimizu K. SDT1/SSM1, a multicopy suppressor of S-II null mutant, encodes a novel pyrimidine 5'-nucleotidase. The Journal of Biological Chemistry, 2002, 277(24):22103-22106. |
| [20] | Shi N, Zhang YJ, Chen HK, Gao YX, Teng MK, Niu LW. Crystal structure of the pyrimidine 5'-nucleotidase from Saccharomyces cerevisiae complexed with uridine 5'-monophosphate provides further insight into ligand binding. Proteins-Structure, Function, and Bioinformatics, 2011, 79(4):1358-1362. |
| [21] | Shevchuk NA, Bryksin AV, Nusinovich YA, Cabello FC, Sutherland M, Ladisch S. Construction of long DNA molecules using long PCR-based fusion of several fragments simultaneously. Nucleic Acids Research, 2004, 32(2):e19. |
| [22] | Spizizen J. Transformation of biochemically deficient strains of Bacillus subtilis by deoxyribonucleate. Proceedings of the National Academy of Sciences of the United States of America, 1958, 44(10):1072-1078. |
| [23] | Anagnostopoulos C, Spizizen J. Requirements for transformation in Bacillus subtilis. Journal of Bacteriology, 1961, 81(5):741-746. |
| [24] | Liu SH, Endo K, Ara K, Ozaki K, Ogasawara N. Introduction of marker-free deletions in Bacillus subtilis using the AraR repressor and the ara promoter. Microbiology, 2008, 154(9):2562-2570. |
| [25] | Song BH, Neuhard J. Chromosomal location, cloning and nucleotide sequence of the Bacillus subtilis cdd gene encoding cytidine/deoxycytidine deaminase. Molecular and General Genetics, 1989, 216(2/3):462-468. |
