 , 孙凯1, 伊佐斯莫娃奥克萨2, 李哲煜1
, 孙凯1, 伊佐斯莫娃奥克萨2, 李哲煜1

1. 哈尔滨工业大学环境学院, 城市水资源与水环境国家重点实验室, 哈尔滨 150090;
2. 圣彼得堡国立大学地球科学研究所, 圣彼得堡, 俄罗斯联邦 199034
收稿日期: 2020-03-29; 修回日期: 2020-09-10; 录用日期: 2020-09-10
基金项目: 国家自然科学基金(No.61674046);城市水资源与水环境国家重点实验室项目(No.2017TS04)
作者简介: 阿里亚·阿不力米提(1993-), 女, E-mail:aliyaabulimit1230@163.com
通讯作者(责任作者): 李哲煜(1973—), 女, 工程师, 博士, 主要研究方向为微流控技术及其在环境微生物方面的应用, 以及纳米表征方法. E-mail:zhylee@hit.edu.cn
摘要:微流控技术是一种基于对微纳流体的操控从而实现特定功能的分析技术,具有微型化、集成化等特点,在生物、化学、医学等领域表现出了很好的发展潜力和应用前景.利用微流控芯片开发细菌培养和抗生素敏感性分析技术,既能对细菌的培养条件实现精确调控,对细菌生长状态进行实时观察,还能显著节省培养时间和使用成本,并缩短抗生素最小抑制浓度的检测周期.有鉴于此,本文针对抗生素敏感分析中微流控技术的研究进展进行评述,以期为相关研究提供一些思路.
关键词:微流控芯片细菌培养抗生素最小抑制浓度(MIC)抗生素敏感性测试(AST)
Advances in antibiotic sensitivity analysis based on microfluidics
ALIYA Abulimiti1
 , SUN Kai1, IZOSIMOVA S. Oksana2, LI Zheyu1
, SUN Kai1, IZOSIMOVA S. Oksana2, LI Zheyu1

1. State Key Laboratory of Urban Water Resource and Environment, School of Environment, Harbin Institute of Technology, Harbin 150090;
2. Institute of Earth Sciences, Saint-Petersburg State University, Saint-Petersburg 199034
Received 29 March 2020; received in revised from 10 September 2020; accepted 10 September 2020
Abstract: Microfluidic technology is a technique that uses microchannels to process micro-fluidics. Microfluidic technology has the characteristic of miniaturization and integration and has great development potential and application prospects in the fields of biology, chemistry and medicine. In terms of bacterial culture and antibiotic sensitivity analysis, microfluidic technology can not only accurately control the culture conditions and real-time observe the growth of bacteria, but also greatly reduce the cost and time of culture and shorten the detection cycle of the minimum inhibitory concentration of antibiotics. This paper introduces several microfluidic chips for bacterial culture, and summarizes the application progress of microfluidic technology in antibiotic sensitivity analysis, hoping to provide some ideas for the design and application of new microfluidic platform.
Keywords: microfluidic chipbacterial cultureantibioticsMICAST
1 引言(Introduction)抗生素对近现代医学贡献巨大, 它有效地减少了细菌感染导致的死亡人数, 挽救了无数人的生命.然而随着抗生素的广泛使用, 抗生素滥用现象也日趋严重, 使得大量细菌产生耐药性, 甚至导致超级细菌的出现(Ayukekbong et al., 2017;Brogan et al., 2016;Lee et al., 2013).仅在美国, 每年因抗药性细菌病原体导致200多万人住院治疗, 2万多人死亡, 医疗费用高达数十亿美元(Syal et al., 2017).缺少针对不同细菌的快速有效的抗生素敏感性测试(AST)方法, 无法指导抗生素的正确使用是导致抗生素滥用的主要原因之一.
细菌抗生素敏感性分析按照原理分为两大类.一类是基于表型的AST方法, 即需要通过观察抗生素对细菌生长代谢的影响获得敏感性信息.这类方法需要在不同种类、不同浓度的抗生素环境下对细菌生长代谢进行有效观察.另一类是在分子层面上的AST方法, 即基因测序.这类方法虽然不需要细菌培养, 但因为只能对特定的基因片段进行检测, 所以极大地限制了所能获取的细菌敏感性信息.传统的基于表型的抗生素敏感性测试主要有扩散法、稀释法和E-实验法(Khan et al., 2019).无论是哪种方法, 都需要完成细菌分离、培养和抗生素敏感性分析实验等步骤, 每一步工作量都很大, 且耗时较长(表 1).而在临床用药中, 时间十分宝贵, 快速准确的用药对挽救病人生命至关重要.因此, 快速进行细菌抗生素敏感分析对指导临床用药、减少抗生素滥用、避免细菌耐药性的产生具有重要意义.
表 1(Table 1)
| 表 1 传统AST方法 Table 1 Traditional AST method | ||||||||||||||||||||
表 1 传统AST方法 Table 1 Traditional AST method
| ||||||||||||||||||||
微流控芯片技术是指在几十至几百μm的微通道中, 调控μL级或更小体积流体的一项技术(Chiu et al., 2017).微流控技术由于其所需样品体积小、反应速率快、兼容性好等优点而受到科研工作者的广泛关注.其中, 基于微流控芯片的细菌培养研究已经在包括细菌的抗生素敏感分析、细菌抗药性、细菌动态变化等方面实现了快速发展(Sibbitts et al., 2017;Chou et al., 2015).特别是在细菌抗生素敏感性分析方面, 微流控芯片技术不仅可以实时监测细菌的形态变化, 而且可以提高分析效率, 节约试剂和时间, 易于集成化、便携化、自动化.
本文通过总结微流控芯片技术在抗生素敏感性分析方面的最新进展, 综述了微流控芯片技术在细菌培养、抗生素浓度梯度的建立和快速AST分析中的应用, 以期为未来微流体平台的设计与应用提供一些思路.
2 基于微流控技术的细菌培养(Bacterial culture based on microfluidic technology)大部分抗生素敏感性分析方法均需要进行细菌预培养, 混合程度、pH值、营养物浓度、温度、空间尺寸等条件均会影响细菌的代谢和生长.传统的试管培养实验通常需要24 h或更长时间才能提供足够高的细胞浓度.与试管培养相比, 利用微流控芯片可以精确调控生长条件, 因此能够缩短培养时间.细菌在微流控芯片中, 只需要6 h甚至更短的时间即可达到所需的浓度.微流控型细菌培养技术主要分为悬浮培养和固定生长两类.其中, 悬浮培养有利于细菌的快速生长, 并适用于多种细菌一起培养;固定化培养能长时间保持细菌活力, 并适用于细菌的原位检测和形态学观察.微流控芯片的结构、尺寸都会对细菌培养产生影响, 随着微流控技术的不断发展, 微流控芯片的结构也越来越多样化, 功能也不尽相同.
2.1 悬浮培养Chen等(2010)探究了不同尺寸的微通道对细菌生长的影响.通过使用不同深度的微通道, 发现具有大微流控比表面积(面积/体积, S/V)的微通道可促进大肠杆菌快速生长, 这是因为在给定浓度的细菌中, 维持细菌生长所需的氧气量与培养基的体积成正比.微流控芯片可以根据需要进行灵活设计, 不仅可以在二维平面内设计微通道, 而且可以利用三维空间实现芯片的高通量、可扩展、多环境细菌培养.Gan等(2011)设计了一种可扩展的多层多通道微流控芯片用于细菌的悬浮培养(图 1A), 芯片分为4层结构, 从上到下依次为气动控制通道、弹性薄膜、L型半环和倒L型半环组成的培养环.该芯片的特点是利用气动控制通道和弹性薄膜组成的蠕动微泵驱动L型半环内的液体促使培养环内液体单向流动, 起到混合搅拌的作用.该芯片具有扩展性好、混合充分、通用性好的特点, 对大肠杆菌、枯草芽孢杆菌等多种细菌的培养效果优于传统摇瓶震荡培养和静态培养.Tang等(2014)设计了一款多通道悬浮培养型微流控芯片用于筛选细菌最佳培养条件(图 1B).该芯片继承了文献(Gan et al., 2011)中的4层芯片结构, 不同的是培养层设计为圣诞树型, 用以产生不同梯度的测试营养素和pH环境.底层为温度层, 通过通入不同温度的水形成不同温度区以筛选不同的温度条件.该芯片具有高通量、细菌生长速度快、工艺小型化和低成本的优势.
图 1(Fig. 1)
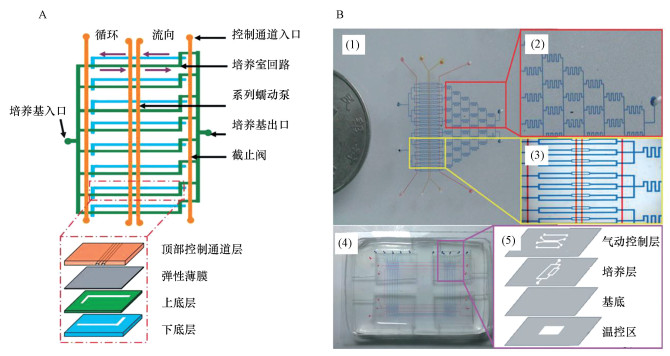 |
| 图 1 用于细菌悬浮培养的可拓展多层多通道微流控芯片(A)和用于筛选最佳细菌培养条件的多通道微流控芯片(B) Fig. 1A scalable multi-channel chip that is capable of bacterial suspension culture(A) and a multi-channel chip used to screen the optimal bacterial culture conditions(B) |
2.2 固定化培养利用固体琼脂糖载体可以在微流控芯片上进行细菌固定化培养.Li等(2014)采用聚二甲基硅氧烷(PDMS)芯片和薄琼脂糖膜的简单组装对细菌进行单层培养(图 2A), 这种简单结构具有单细胞形态学跟踪的优势.但利用琼脂糖进行细菌的固定培养仅限于几代以内, 因为细菌细胞生长和分裂很快, 以至于细胞在几小时内迅速占据培养空间, 细菌堆砌在一起, 难以分辨单个细菌.为解决这个问题, Moffitt等(2012)通过在琼脂糖垫上刻画的亚微米级线性轨道迫使细菌依次排列在轨道中成线性生长, 并允许多余的细胞从两端逃逸, 实现了对细菌的长期培养, 细菌恒温培养可超过30~40代.然而琼脂糖的高孔隙率使其难以改变内部的培养环境, 如果希望长时间测量单细胞水平的分裂细胞, 同时避免培养基交换, 必须通过微型隔室将单个细胞隔离, 并持续提供营养物质让多余的细胞从隔室溢出.Long等(2013)设计了一种微流体装置, 由600个亚微米级的生长通道连接到两个进料通道组成, 形状与梯子相似(图 2B).利用600个亚微米级的窄通道来捕获大肠杆菌并让细菌在窄通道中生长, 主通道通入培养基, 不断地向梯级内的细菌提供新鲜营养物质, 多余的细胞会被推出到主通道中并被流体带走.利用这种装置可以通过介质流从生长通道的两端除去过量的细菌和代谢废物, 使细菌生长超过50代.
图 2(Fig. 2)
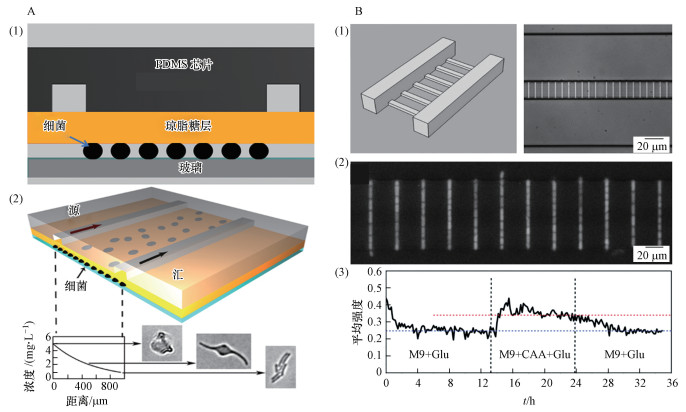 |
| 图 2 用于细菌固定化培养的微流控芯片(A)和横梯式微流控芯片及亚微米通道中生长的大肠杆菌细胞的荧光图像(B) Fig. 2The chip for bacterial immobilization culture(A) and horizontal ladder microfluidic chip and fluorescence image of E.coli cells growing in submicron channels(B) |
3 抗生素浓度梯度的建立(Gradient generators of antibiotic concentration)抗生素浓度梯度的建立可以用于测定抗生素最小生物膜清除浓度(MBEC)(Kim et al., 2010)和最小抑菌浓度(MIC)(Kim et al., 2019)等.但人工配置不同浓度的抗生素耗时长、工作繁复, 而且需要对配置流程熟悉的专业技术人员才能够完成.微流控芯片可以实现抗生素浓度的自动和快速建立, 从而缩短细菌抗生素敏感性测试时间.本节将介绍几种建立抗生素浓度梯度的微流控芯片.
3.1 基于微流控网络结构的浓度梯度生成利用微流控芯片建立抗生素浓度梯度的一种常见方法是利用一组网络结构将抗生素与培养基混合, 生成不同抗生素浓度梯度.这种微流控网络应当具有以下特点(Hattori et al., 2009):①生成具有线性或对数浓度分布的抗生素浓度梯度;②具有紧密的微流控网络结构, 可在芯片上集成;③在很大的流速范围内保持稳定;④能够将生成的浓度分布保留几天到一周.
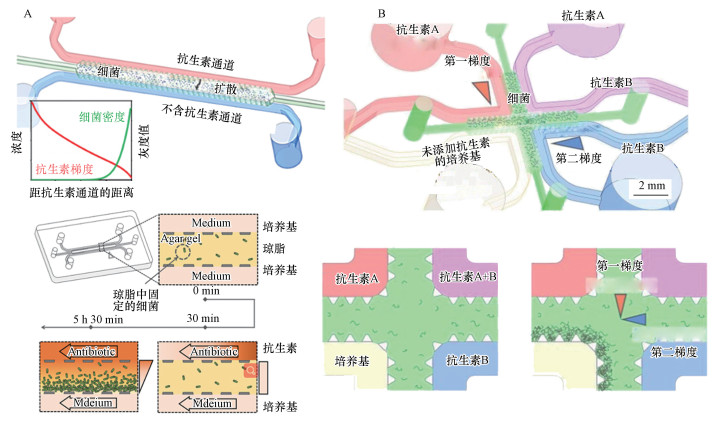
金字塔结构是一种典型的网络结构(Dertinger et al., 2001)(图 3A), 两个入口分别通入抗生素和培养基, 利用高流阻比的结构将入口和出口连通, 可以通过不断叠加依次增加出口个数获得不同抗生素浓度梯度.这种结构形成的浓度分布可以维持数个小时, 因此获得了广泛应用(Jeon et al., 2002).Dicicco等(2014)利用这种装置获得了10组抗生素浓度梯度.通过将多个这种结构集成在一个芯片上, 还可以同时测定多种抗生素的最低抑制浓度(He et al., 2014).但这种结构的占用面积与出口微通道数的平方成正比, 而且产生的浓度分布对流量的变化非常敏感, 流速快则混合不足, 流速慢则相邻浓度梯度间容易发生扩散.针对这些缺点, Hattori等(2009)设计了一种由具有高流阻比的微通道组成的微流控网络(图 3B), 不仅结构紧凑, 而且可将各浓度梯度分离开, 避免扩散.该结构生成的浓度分布稳定性较金字塔结构更好, 至少在3 d内是稳定的.Kim等(2015)利用该网络测定了氨苄青霉素和链霉素对大肠杆菌的最低抑制浓度, 可以在3 h内获得结果, 大大缩短了测试时间, 而且不需要压缩空气源或压力调节器, 只需要基于简单注射器的压缩注射即可满足AST测试需要.
图 3(Fig. 3)
 |
| 图 3 金字塔型浓度梯度生成微流控网络结构(A)和改进的高阻流比通道组成的网络结构(B) Fig. 3Microfluidic network of pyramid type for generating concentration gradient(A) and an improved microfluidic network composed of high resistance to current ratio channels(B) |
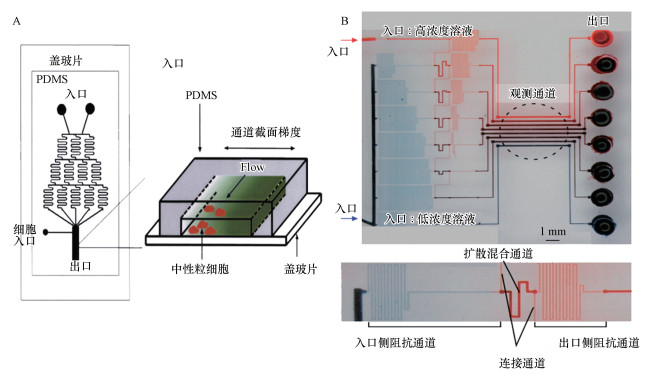
3.2 基于平行通道扩散的浓度梯度生成利用微流控网络结构生成的浓度梯度是动态稳定的, 可用于细菌生物膜抗生素敏感性分析.在静态培养中, 可以利用不同浓度平行通道中的抗生素在琼脂凝胶中扩散形成浓度梯度, 利用泵在通道中不断通入高浓度抗生素维持琼脂凝胶中的抗生素梯度.Chung等(2019)设计了一种微流控系统, 在两个抗生素浓度不同的平行通道之间利用抗生素的扩散建立浓度梯度(图 4A), 只需30 min即可生成线性抗生素浓度梯度, 可在6 h完成AST实验.Kim等(2019)利用微流控平台可在35 min内在细菌捕获琼脂糖凝胶上形成两个正交的浓度梯度(图 4B), 用以研究多种抗生素之间的协同/拮抗作用, 可在7 h内完成各类抗生素分析.Hou等(2014)将时延摄影技术融合在平行扩散微流控系统中, 进一步缩短了AST时间, 整个过程包括数据分析只需2.5~4.0 h, 体现了微流控系统在AST分析上的巨大优势.
图 4(Fig. 4)
 |
| 图 4 基于平行扩散的浓度梯度生成芯片(A.单抗生素平行扩散结构, B.两种抗生素正交扩散结构) Fig. 4Concentration gradient generators based on parallel channel diffusion(A.Parallel channel diffusion for single antibiotic, B.Cross channel diffusion for two kinds of antibiotics) |
3.3 其他浓度梯度生成方法除了上述两类浓度梯度生成微流控系统外, 还有很多生成浓度梯度的方法.比如, 利用旋转产生的离心效应快速实现了高通量的抗生素浓度梯度的生成(Tang et al., 2018), 并可以通过增加圆盘层数实现多种抗生素混合浓度梯度的生成;此外, 可以先在多个腔室中定量预埋不同质量的干燥抗生素, 然后注入细菌悬浮液溶解抗生素形成不同的抗生素浓度(Cira et al., 2012).该装置小巧便携, 虽然无法自动化生成连续的抗生素浓度梯度, 但适用于POC(Point of care)实验(在实验室外或患者护理现场附近进行的实验).
4 基于液滴的微流控系统(Droplet microfluidics for AST)传统的抗生素敏感性分析方法需要将细菌密度提高到可检测的水平(即预培养), 该过程一般需要16~24 h.液滴微流控技术通过无限稀释法, 能够将单个细菌分散到纳升级大小的微液滴中, 每个液滴作为一个单独的生物反应器, 同时含有活性荧光标记染料和不同浓度抗生素.经过细菌培养一代至两代时间, 通过荧光成像检测液滴内是否出现新增细菌, 即可确定MIC值.迄今为止, 检测时间已经可以缩短到1 h内.
4.1 微液滴生成方法基于液滴的微流控系统必须能够产生足够小的微液滴, 液滴通常是由表面诱导的不稳定性产生的, 同时为了保证单分散性一般需加入表面活性剂.对于微液滴的形成方法, 文献(Seemann et al., 2012)进行了详细的总结.Boedicker等(2008)利用多入口的微流控装置(图 5A), 采用混合生成液滴的方法(Adamson et al., 2006;Li et al., 2006)将细菌样本、指示剂和预先形成的抗生素阵列合并通入载液中, 形成纳升大小的液滴来进行细菌抗生素敏感性测试.该方法无需预培养, 在单细胞层面进行抗生素敏感性分析, 大大节约了时间.Postek等(2018)提出了一种产生和分离由亚纳升级液滴组成的乳状液的方法(图 5B).系统首先制备含有不同化学环境的微升级母液滴, 然后利用锥形喷嘴将液母滴乳化, 产生大量分散的亚纳升级微液滴.利用不混溶角鲨烷油将乳化液分离, 形成含有大量微液滴的液槽, 每个液槽含有相同的培养基和不同的抗生素浓度, 可以同时进行多种抗生素敏感性实验.该装置可以通过空间位置对液槽进行标记, 从而避免了多抗生素敏感性实验中需要用不同的染色试剂标记.而且每个液槽中含有成百上千个微液滴, 相当于进行了数千次重复实验, 可以提高试验准确性.
图 5(Fig. 5)
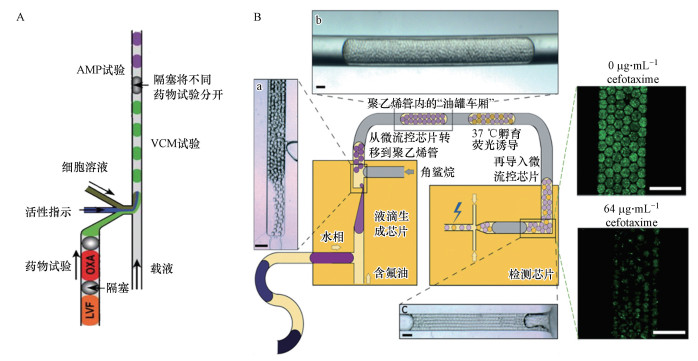 |
| 图 5 多入口混合的纳升级微液滴生成结构(A)和乳化分离生成亚纳升级微液滴及油罐车分段的结构示意图(B) Fig. 5Hybrid generation of nanoscale microdroplets by a multi inlet microfluidic device(A) and microfluidic system for the generation of sub nanoscale microdroplets by emulsification and separation(B) |
4.2 液滴微流控AST系统的集成基于液滴的微流控系统可以进行集成化处理, 将液滴生成、细菌培养、检测整合到一起从而缩短检测时间.Jiang等(2016)介绍了一种基于液滴的微流控数字抗菌药敏感度测试系统(D-AST), 该系统能在2 h内准确地测定最小抑菌浓度(MIC).Kaushik等(2017)将液滴的生成、孵化和检测完全整合到一个单一的、不间断的微流中(图 6A), 提出了一种称为Dropfast的单细胞生物传感平台, 用于细菌生长快速检测和抗菌敏感性评估, 在大肠杆菌庆大霉素敏感性测试中只需培养1 h即可得到细菌抗生素敏感性信息, 该方法代表了基于表型评估细菌耐药性的最快方法.液滴中细菌的检测常用荧光染料标记(Azizi et al., 2018), Keays等(2016)利用光密度测量方法, 无需任何染料辅助即可实现细菌生长的自动连续监测, 能够在1 h内检测到液滴培养的效果.基于液滴的微流控系统还可以通过形成集成阵列的方法进行快速AST测试(图 6B)(Avesar et al., 2017;Jalali et al., 2017;Sabhachandani et al., 2017;Lee et al., 2017).
图 6(Fig. 6)
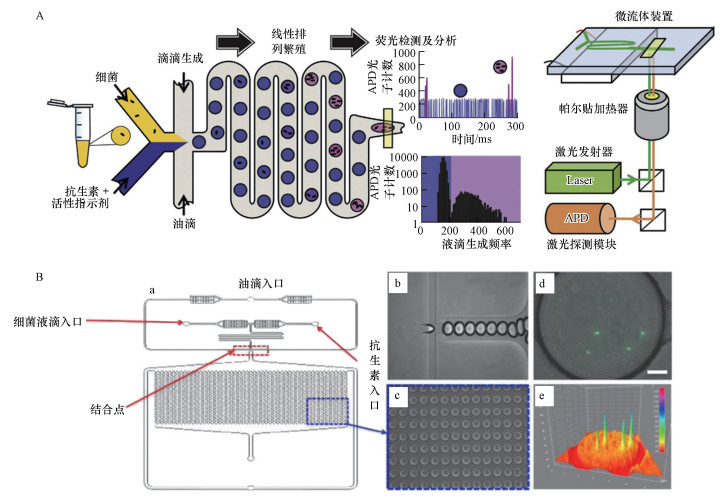 |
| 图 6 集成微流控AST平台(A.Dropfast单细胞生物传感平台, B.基于液滴的微阵列AST系统) Fig. 6Integrated microfluidic AST platform(A.Dropfast single cell biosensor platform, B.Micro array AST system based on droplets) |
虽然液滴微流控技术具有制造成本低、样品体积小、分析时间短、灵敏度高和方便集成化、自动化等优点, 但该方法也有局限性.尽管无需预培养, 但要想捕捉到抗生素对细菌生长、代谢活动的影响, 仍然需要对含有抗生素的液滴中的细菌进行培养(0.5~2.0 h)才能够观察到抗生素是否对细菌生长具有抑制效应, 培养时间过短会降低MIC测量准确性.这限制了细菌敏感性测试时间, 特别是对于生长缓慢的细菌, 这也是基于表型的AST方法无法逾越的壁垒.
4.3 微流控技术与其他先进方法的级联对于基于表型的AST方法, 缩短检测时间的关键在于捕捉抗生素对细菌生长、代谢活动的影响.常用检测方法分为直接检测方法和非直接检测方法(Dai et al., 2016).直接检测方法主要为光学成像, 非直接检测方法包括荧光检测、细菌密度检测、pH检测等.常用方法采用荧光染料标记, 但随着各种新型检测方法的兴起, 细菌检测手段越来越多样化, 已经可以在细胞水平对细胞进行观察, 比如单细胞光学成像跟踪(Baltekin et al., 2017;Li et al., 2019)、电化学传感器(Jo et al., 2018;Kara et al., 2018);此外, 也可以在分子水平对细菌代谢产物变化进行监测, 比如拉曼光谱(SERS)分析(Chang et al., 2019;Rousseau et al., 2018;Liu et al., 2016)、受激拉曼散射(SRS)成像(Hong et al., 2018)、流式细胞技术(Saint et al., 2016)等.这些方法提高了细菌检测的灵敏度和快速性, 通常可以在0.5~4.0 h内完成抗生素敏感性分析.虽然省去了对细菌的处理, 但需要设备支持, 成本较高.
5 结论(Conclusions)本文综述了微流控芯片在细菌抗生素敏感性分析方面的应用, 特别是在细菌培养、抗生素浓度梯度建立、快速抗生素敏感性分析方面, 与传统抗生素敏感性分析方法相比均有优势, 不仅能够缩短细菌预培养、抗生素浓度梯度建立的时间, 简化操作、减小误差, 而且可以通过操纵微流体实现细菌抗生素敏感性快速分析.与不断发展的检测技术相融合, 还可以实时原位观察抗生素作用下的细菌生长和代谢过程.
6 建议(Suggestions)目前基于微流控的细菌抗生素敏感性分析技术仍然在快速发展中.虽然基于微流控的AST技术已经可以实现在1 h内得到细菌针对特定抗生素的敏感性信息, 但这些微流控系统距离商业应用还有一段距离.只有在临床应用中, AST检测的快速性才是有意义的, 因此除了检测的快速性, 还应该关注界面友好性、操作性、对细菌和抗生素种类的普适性及设备的便携性等实用特性, 在现有的快速分析方法的基础上开发满足临床诊断需要的集成化设备.同时, 应该充分发挥基于微流控的AST方法的优势, 利用其可以提供大量的细菌抗生素敏感性信息的特点, 结合临床医生的需要, 开发可以提供多种敏感性信息的自动化设备.相信随着检测技术和微流控AST技术的不断发展, 基于微流控的AST技术有望在临床医学中得到广泛应用.
参考文献
| Adamson D N, Mustafi D, Zhang J X J, et al. 2006. Production of arrays of chemically distinct nanolitre plugs via repeated splitting in microfluidic devices[J]. Lab on a Chip, 6(9): 1178-1186. DOI:10.1039/b604993a |
| Avesar J, Rosenfeld D, Truman-Rosentsvit M, et al. 2017. Rapid phenotypic antimicrobial susceptibility testing using nanoliter arrays[J]. Proceedings of the National Academy of Sciences of the United States of America, 114(29): 5787-5795. DOI:10.1073/pnas.1703736114 |
| Ayukekbong J A, Ntemgwa M, Atabe A N. 2017. The threat of antimicrobial resistance in developing countries:causes and control strategies[J]. Antimicrobial Resistance & Infection Control, 6(1): 1-8. |
| Azizi M, Zaferani M, Dogan B, et al. 2018. Nanoliter-sized microchamber/microarray microfluidic platform for antibiotic susceptibility testing[J]. Analytical Chemistry, 90(24): 14137-14144. DOI:10.1021/acs.analchem.8b03817 |
| Baltekin ?, Boucharin A, Tano E, et al. 2017. Antibiotic susceptibility testing in less than 30 min using direct single-cell imaging[J]. Proceedings of the National Academy of Sciences of the United States of America, 114(34): 9170-9175. DOI:10.1073/pnas.1708558114 |
| Boedicker J Q, Li L, Kline T R, et al. 2008. Detecting bacteria and determining their susceptibility to antibiotics by stochastic confinement in nanoliter droplets using plug-based microfluidics[J]. Lab on a Chip, 8(8): 1265-1272. DOI:10.1039/b804911d |
| Brogan D M, Mossialos E. 2016. A critical analysis of the review on antimicrobial resistance report and the infectious disease financing facility[J]. Globalization and Health, Globalization and Health, 12(1): 1-7. DOI:10.1186/s12992-015-0138-4 |
| Brown D F J, Brown L. 1991. Evaluation of the E test, a novel method of quantifying antimicrobial activity[J]. Journal of Antimicrobial Chemothera, 27(1): 185-190. |
| Chang K, Cheng H, Shiue J, et al. 2019. Antibiotic susceptibility test with surface-enhanced raman scattering in a microfluidic system[J]. Analytical Chemistry, 91(17): 10988-10995. DOI:10.1021/acs.analchem.9b01027 |
| Chen C H, Lu Y, Sin M L Y, et al. 2010. Antimicrobial susceptibility testing using high surface-to-volume ratio microchannels[J]. Analytical Chemistry, 82(3): 1012-1019. DOI:10.1021/ac9022764 |
| Chiu D T, Demello A J, Carlo D D, et al. 2017. Small but perfectly formed? Successes, challenges, and opportunities for microfluidics in the chemical and biological sciences[J]. Chem, 2(2): 201-223. DOI:10.1016/j.chempr.2017.01.009 |
| Chou W L, Lee P Y, Yang C L, et al. 2015. Recent advances in applications of droplet microfluidics[J]. Micromachines, 6(9): 1249-1271. DOI:10.3390/mi6091249 |
| Cira N J, Ho J Y, Dueck M E, et al. 2012. A self-loading microfluidic device for determining the minimum inhibitory concentration of antibiotics[J]. Lab on a Chip, 12(6): 1052-1059. DOI:10.1039/C2LC20887C |
| Dai J, Hamon M, Jambovane S, et al. 2016. Microfluidics for antibiotic susceptibility and toxicity testing[J]. Bioengineering, 3(4): 1-13. |
| Dertinger S K W, Chiu D T, Jeon N L, et al. 2001. Generation of gradients having complex shapes using microfluidic networks[J]. Analytical Chemistry, 73(6): 1240-1246. DOI:10.1021/ac001132d |
| Dicicco M, Neethirajan S. 2014. An in vitro microfluidic gradient generator platform for antimicrobial testing[J]. Biochip Journal, 8(4): 282-288. DOI:10.1007/s13206-014-8406-6 |
| Fluit A C, Visser M R, Schmitz F, et al. 2001. Molecular detection of antimicrobial resistance[J]. Clinical Microbiology Reviews, 14(4): 836-871. DOI:10.1128/CMR.14.4.836-871.2001 |
| Frye J G, Lindsey R L, Rondeau G, et al. 2010. Development of a DNA microarray to detect antimicrobial resistance genes identified in the national center for biotechnology information database[J]. Microbial Drug Resistance, 16(1): 9-19. DOI:10.1089/mdr.2009.0082 |
| Gan M, Su J, Wang J, et al. 2011. A scalable microfluidic chip for bacterial suspension culture[J]. Lab on a Chip, 11(23): 4087-4092. DOI:10.1039/c1lc20670b |
| Hattori K, Sugiura S, Kanamori T. 2009. Generation of arbitrary monotonic concentration profiles by a serial dilution microfluidic network composed of microchannels with a high fluidic-resistance ratio[J]. Lab on a Chip, 9(12): 1763-1772. DOI:10.1039/b816995k |
| He J, Mu X, Guo Z, et al. 2014. A novel microbead-based microfluidic device for rapid bacterial identification and antibiotic susceptibility testing[J]. European Journal of Clinical Microbiology and Infectious Diseases, 33(12): 2223-2230. DOI:10.1007/s10096-014-2182-z |
| Hombach M, Zbinden R, B?ttger E C. 2013. Standardisation of disk diffusion results for antibiotic susceptibility testing using the sirscan automated zone reader[J]. BMC Microbiology, 13(1): 225-233. DOI:10.1186/1471-2180-13-225 |
| Hong W, Karanja C W, Abutaleb N S, et al. 2018. Antibiotic susceptibility determination within one cell cycle at single-bacterium level by stimulated raman metabolic imaging[J]. Analytical Chemistry, 90(6): 3737-3743. DOI:10.1021/acs.analchem.7b03382 |
| Hou Z, An Y, Hjort K, et al. 2014. Time lapse investigation of antibiotic susceptibility using a microfluidic linear gradient 3D culture device[J]. Lab on a Chip, 14(17): 3409-3418. DOI:10.1039/C4LC00451E |
| Jalali F, Ellett F, Irimia D, et al. 2017. Rapid antibiotic sensitivity testing in microwell arrays[J]. Technology, 5(2): 107-114. DOI:10.1142/S2339547817500030 |
| Jeon N L, Baskaran H, Dertinger S K W, et al. 2002. Neutrophil chemotaxis in linear and complex gradients of interleukin-8 formed in a microfabricated device[J]. Nature Biotechnology, 20(7): 826-830. |
| Jiang L, Boitard L, Broyer P, et al. 2016. Digital antimicrobial susceptibility testing using the MilliDrop technology[J]. European Journal of Clinical Microbiology and Infectious Diseases, 35(3): 415-422. DOI:10.1007/s10096-015-2554-z |
| Jo N, Kim B, Lee S, et al. 2018. Aptamer-functionalized capacitance sensors for real-time monitoring of bacterial growth and antibiotic susceptibility[J]. Biosensors and Bioelectronic, 102(7): 164-170. |
| Kara V, Duan C, Gupta K, et al. 2018. Microfluidic detection of movements of Escherichia coli for rapid antibiotic susceptibility testing[J]. Lab on a Chip, 18(5): 743-753. DOI:10.1039/C7LC01019B |
| Kaushik A M, Hsieh K, Chen L, et al. 2017. Accelerating bacterial growth detection and antimicrobial susceptibility assessment in integrated picoliter droplet platform[J]. Biosensors and Bioelectronics, 97(1): 260-266. |
| Keays M C, O'brien M, Hussain A, et al. 2016. Rapid identification of antibiotic resistance using droplet microfluidics[J]. Bioengineered, 7(2): 79-87. DOI:10.1080/21655979.2016.1156824 |
| Khan Z A, Siddiqui M F, Park S. 2019. Current and emerging methods of antibiotic susceptibility testing[J]. Genetic Testing:Care, Consent, and Liability, 9(49): 268-291. |
| Kim K P, Kim Y G, Choi C H, et al. 2010. In situ monitoring of antibiotic susceptibility of bacterial biofilms in a microfluidic device[J]. Lab on a Chip, 10(23): 3296-3299. DOI:10.1039/c0lc00154f |
| Kim S C, Cestellos-Blanco S, Inoue K, et al. 2015. Miniaturized antimicrobial susceptibility test by combining concentration gradient generation and rapid cell culturing[J]. Antibiotics, 4(4): 455-466. DOI:10.3390/antibiotics4040455 |
| Kim S, Li S, Kim J K, et al. 2019. Microfluidic-based observation of local bacterial density under antimicrobial concentration gradient for rapid antibiotic susceptibility testing[J]. Biomicrofluidics, 13(1): 1-11. |
| Kim S, Masum F, Kim J K, et al. 2019. On-chip phenotypic investigation of combinatory antibiotic effects by generating orthogonal concentration gradients[J]. Lab on a Chip, 19(6): 959-973. DOI:10.1039/C8LC01406J |
| Lee C, Cho I H, Jeong B C, et al. 2013. Strategies to minimize antibiotic resistance[J]. International Journal of Environmental Research and Public Health, 10(9): 4274-4305. DOI:10.3390/ijerph10094274 |
| Lee W, Fu C, Chang W, et al. 2017. A microfluidic device for antimicrobial susceptibility testing based on a broth dilution method[J]. Biosensors and Bioelectronic, 87(1): 669-678. |
| Li B, Qiu Y, Glidle A, et al. 2014. Gradient microfluidics enables rapid bacterial growth inhibition testing[J]. Analytical Chemistry, 86(6): 3131-3137. DOI:10.1021/ac5001306 |
| Li H, Torab P, Mach K E, et al. 2019. Adaptable microfluidic system for single-cell pathogen classification and antimicrobial susceptibility testing[J]. Proceedings of the National Academy of Sciences of the United States of America, 116(21): 10270-10279. DOI:10.1073/pnas.1819569116 |
| Li L, Mustafi D, Fu Q, et al. 2006. Nanoliter microfluidic hybrid method for simultaneous screening and optimization validated with crystallization of membrane proteins[J]. Proceedings of the National Academy of Sciences of the United States of America, 103(51): 19243-19248. DOI:10.1073/pnas.0607502103 |
| Liu C Y, Han Y Y, Shih P H, et al. 2016. Rapid bacterial antibiotic susceptibility test based on simple surface-enhanced Raman spectroscopic biomarkers[J]. Scientific Reports, 6(3): 1-15. |
| Long Z, Nugent E, Javer A, et al. 2013. Microfluidic chemostat for measuring single cell dynamics in bacteria[J]. Lab on a Chip, 13(5): 947-954. DOI:10.1039/c2lc41196b |
| Moffitt J R, Lee J B, Cluzel P. 2012. The single-cell chemostat:An agarose-based, microfluidic device for high-throughput, single-cell studies of bacteria and bacterial communities[J]. Lab on a Chip, 12(8): 1487-1494. DOI:10.1039/c2lc00009a |
| Postek W, Gargulinski P, Scheler O, et al. 2018. Microfluidic screening of antibiotic susceptibility at a single-cell level shows the inoculum effect of cefotaxime on E.coli[J]. Lab on a Chip, 18(23): 3668-3677. DOI:10.1039/C8LC00916C |
| Rousseau A N, Espagnon I, Filiputti D, et al. 2018. Culture-free antibiotic-susceptibility determination from single- bacterium Raman spectra[J]. Scientific Reports, S, 8(1): 1-12. DOI:10.1038/s41598-017-17765-5 |
| Sabhachandani P, Sarkar S, Zucchi P C, et al. 2017. Integrated microfluidic platform for rapid antimicrobial susceptibility testing and bacterial growth analysis using bead-based biosensor via fluorescence imaging[J]. Microchimica Acta, 184(12): 4619-4628. DOI:10.1007/s00604-017-2492-9 |
| Saint-Ruf C, Crussard S, Franceschi C, et al. 2016. Antibiotic susceptibility testing of the gram-negative bacteria based on flow cytometry[J]. Frontiers in Microbiology, 7(7): 1-13. |
| Seemann R, Brinkmann M, Pfohl T, et al. 2012. Droplet based microfluidics[J]. Reports on Progress in Physics, 75(1): 1-41. |
| Sibbitts J, Sellens K A, Jia S, et al. 2017. Cellular analysis using microfluidics[J]. Analytical Chemistry, 90(1): 65-85. |
| Syal K, Mo M, Yu H, et al. 2017. Current and emerging techniques for antibiotic susceptibility tests[J]. Theranostics, 7(7): 1795-1805. DOI:10.7150/thno.19217 |
| Tang M, Huang X, Chu Q, et al. 2012. A linear concentration gradient generator based on multi-layered centrifugal microfluidics and its application in antimicrobial susceptibility testing[J]. Lab on a Chip, 18(10): 1452-1460. |
| Tang Y, Gan M, Xie Y, et al. 2014. Fast screening of bacterial suspension culture conditions on chips[J]. Lab on a Chip, 14(6): 1162-1167. DOI:10.1039/C3LC51332G |
| Waites K B, Duffy L B, Bébéar C M, et al. 2012. Standardized methods and quality control limits for agar and broth microdilution susceptibility testing of Mycoplasma pneumoniae, Mycoplasma hominis, and Ureaplasma urealyticum[J]. Journal of Clinical Microbiology, 50(11): 3542-3547. DOI:10.1128/JCM.01439-12 |
