 , 王晓侠1,2, 周立光2, 皮艳霞2,3, 周杰民2, 祝贵兵2
, 王晓侠1,2, 周立光2, 皮艳霞2,3, 周杰民2, 祝贵兵21. 青岛理工大学环境与市政工程学院, 青岛 266033;
2. 中国科学院生态环境研究中心饮用水科学与技术重点实验室, 北京 100085;
3. 哈尔滨商业大学生命科学与环境研究中心, 哈尔滨 150028
收稿日期: 2018-07-25; 修回日期: 2018-09-27; 录用日期: 2018-09-27
基金项目: 国家自然科学基金(No.41671471, 41322012, 21707155);中国科学院的战略重点研究项目(No.XDB15020303);国家重点研发项目(No.2016YFA0602303);广东省"珠江人才计划"本土创新科研团队项目(No.2017BT01Z176);环境模拟与污染控制国家重点联合实验室专项资金(生态环境科学研究中心, 中国科学院)(No.18Z02ESPCR), 中国科学院饮用水科学与技术重点实验室开放研究基金(No.16Z03KLDWST); 中国科学院创新团队国际合作伙伴计划
作者简介: 周利(1963—), 男, 教授(博士), E-mail:qtechzhouli@163.com
摘要: 为研究典型旱地农田土壤硝酸盐异化还原成铵过程(Dissimilatory nitrate reduction to ammonium,DNRA)的群落组成,针对DNRA过程的功能基因nrfA进行高通量测序.根际和非根际、4种典型农作物共16个样品,质控后每个样品得到87000条序列,在相似度≥ 90%下划分到27952个OTUs,选取其中丰度较高的258个代表OTUs进行生态学分析.多样性分析(OTUs水平)结果表明:3/4的作物根际土壤样品中的DNRA群落丰富度、物种多样性和物种均匀度高于相应非根际样品,对比4种作物,粟作物根部土壤DNRA群落多样性最高,玉米作物非根际土壤最低.对代表OTUs进行分类,共定义到6个门(Phylum),19个属(Genus).其中相对丰度最高的3个属为Hyalangium(29.31%)、Chthoniobacter(20.33%)和Nitrospira(13.41%),表明三者在群落组成中占主导地位.结合土壤理化因子分析,DNRA群落相对丰度与NO2--N、TN、含水率、TOM、pH及温度呈显著相关关系.本研究在一定程度上揭示了旱地农田土壤DNRA细菌的群落组成、多样性及与土壤环境因子的关系,为提高氮肥的利用效率和减小环境污染提供理论依据.
关键词:硝酸盐异化还原成铵过程高通量测序功能基因nrfA旱地农田土壤根际和非根际
Community composition of dissimilatory nitrate reduction to ammonium (dnra) bacteria in rhizosphere and non-rhizosphere soil of agricultural lands
ZHOU Li1
 , WANG Xiaoxia1,2, ZHOU Liguang2, PI Yanxia2,3, ZHOU Jiemin2, ZHU Guibing2
, WANG Xiaoxia1,2, ZHOU Liguang2, PI Yanxia2,3, ZHOU Jiemin2, ZHU Guibing2 1. School of Environmental and Municipal Engineering, Qingdao University of Technology, Qingdao 266033;
2. Key Laboratory of Drinking Water Science and Technology, Research Center for Eco-Environment Sciences, Chinese Academy of Sciences, Beijing 100085;
3. Research Center on Life Sciences and Environmental Sciences, Haerbin University of Commerce, Harbin 150028
Received 25 July 2018; received in revised from 27 September 2018; accepted 27 September 2018
Abstract: To explore the community composition of dissimilatory nitrate reduction to ammonium (DNRA) process in typical agricultural dryland soil, high-throughput sequencing targeting on the functional gene nrfA of DNRA bacteria was carried out. A total of 16 samples of rhizosphere and non-rhizosphere soils from 4 typical agricultural lands were collected, and 87000 sequences were obtained for each sample after quality control. Then these sequences were clustered to 27952 OTUs with similarity above 90%, of which 258 representative OTUs with higher abundance were selected for community analysis. Results of diversity analysis (OTUs level) demonstrated that 3/4 of the samples in rhizosphere soil had a higher diversity, richness, and species uniformity of DNRA community than those in the corresponding non-rhizosphere soils. Moreover, it was found that rhizosphere soil of millet crops had the highest diversity of DNRA community while non-rhizosphere soil of maize crops had the lowest one among the investigated crops. Furthermore, the representative OTUs was classified into 6 phyla and 19 genera, and, the three highest richness genera were Hyalangium (29.31%), Chthoniobacter (20.33%) and Nitrospira (13.41%), indicating their dominant role in DNRA community composition. Combined with soil physicochemical factor analysis, the relative abundance of DNRA community was significantly correlated with NO2--N, TN, moisture content, TOM, pH and temperature in the four crops. To some extent, this study revealed the community composition, diversity of DNRA bacteria, and their relationship with soil environmental factors in agricultural dryland soil, providing theoretical basis for improving the utilization efficiency of nitrogen fertilizer and reducing environmental pollution.
Keywords: dissimilatory nitrate reduction to ammonium (DNRA)high-throughput sequencingnrfA functional genearid farmland soilrhizosphere and non-rhizospher
1 引言(Introduction)氮(N)循环驱动主要是通过微生物一系列的氧化还原反应, 来改变氮化合物的存在形态.微生物参与的氮循环过程中硝酸盐涉及的反应过程最多, 包括硝酸盐同化反应、硝酸盐异化还原反应和亚硝酸盐氧化反应(Kraft et al., 2011).土壤中硝酸盐异化还原速度较快, 可分为反硝化过程(Denitrification)和硝酸盐异化还原成铵过程(DNRA).反硝化作用是将NO3-还原成N2而造成氮素损失, 而且还会产生具有温室效应的中间产物N2O (Zhuet al., 2011).DNRA不同于反硝化过程, 它是将NO3-或NO2-还原为NH4+, 产生的NH4+在硝化反应功能微生物作用下重新生成NO2-和NO3-, 然后进行反硝化、DNRA和厌氧氨氧化等过程, 还原为NO2-、NH4+和N2.可以说DNRA在自然系统的氮循环中起着重要作用, 是微生物氮循环的新内容, 也是整个氮循环非常重要的衔接过程, 相较于反硝化反应可以减少温室气体排放, 它的发现对完善全球氮循环具有重要贡献, 在保护生态环境方面也具有非常重要的意义.
随着现代农业和工业的发展, 农业上会大量施用含氮化肥取代传统有机肥.氮肥中的NH4+容易被带有负电荷的土壤胶体吸附, 不易流失.土壤中好氧硝化细菌会将铵态氮转化成硝态氮, 硝酸盐是阴离子, 在土壤系统中移动性好, 当发生降水或农田灌溉时, 硝酸盐容易随水流渗透到地表水和地下水中, 造成氮素的淋失和流失, 导致水体氮污染.DNRA的存在, 有利于氮素在土壤中的蓄持, 减小水体氮污染;铵的生成增强了土壤肥力, 铵可以直接被农作物吸收利用, 有利于作物生长.此外, 由于DNRA细菌和反硝化细菌对底物NO3-存在着竞争, DNRA的存在, 不仅有利于降低土壤中氮素损失, 还可以减少反硝化过程产生的N2O, 减少温室气体的排放.DNRA在氮循环中的作用越来越被广泛关注(贺纪正等, 2013;朱永官等, 2014).
有研究通过对DNRA菌进行纯培养的方式, 研究DNRA基因组成和DNRA群落对环境的响应机制(Rehr and Klemme, 1989);有的通过分子生物学方法研究DNRA菌不同物种之间的密切关系等(Song et al., 2014).以往对DNRA的研究主要集中在水稻田、河口、湖泊和海洋沉积物等厌氧或缺氧环境条件下的土壤, 研究影响DNRA的因素及如何影响(Blackmer and Bremner, 1978; Mmüller et al., 1980; J?rgensen et al., 1984; Maag and Vinther, 1996; Yamulki et al., 1997; Senga et al., 2006).但有关农田旱地土壤生态系统中DNRA细菌群落结构及其相关影响因子尚未被清楚认识和了解, 尤其是对旱地作物根际与非根际土壤中DNRA的研究鲜有报道.
本研究采样点位于广阔的黄河中下游平原, 是我国主要的粮食作物产区之一, 大范围的化肥施用使得农田土壤具有较高的人为氮负荷, 氮循环微生物多样性高.在这样的研究背景下, 为了更好地了解DNRA细菌群落组成, 我们针对北澧河附近冬夏两季旱地农田作物根际和非根际土壤中DNRA的功能基因nrfA进行高通量测序分析, 研究DNRA细菌的多样性、群落结构及其与土壤环境因子的关系.
2 材料与方法(Materials and methods)2.1 样品采集与保存采样地点为河北省北澧河附近旱地农田(27°28′18.49″ N, 114°58′19.45″ E), 选取夏季(8月)和冬季(1月)两个时间点的4种农作物(玉米、棉花、黄豆和粟)的根际及非根际共16个土壤样品为研究对象(表 1), 其中冬季作物为干枯植株.采用抽样法选取植株, 将植株的根部从土壤中整体挖出, 轻轻抖落与根部结合松散的土壤作为非根际土壤样品, 再用无菌的镊子刮取粘连在根系上的薄层土壤(<10 mm)作为根际土壤样品(雍太文等, 2012).将多棵同种作物植株的根际与非根际土壤分别均匀混合在一起作为一个土壤样品(该样品代表了多棵植物的平均水平)(Mroz and Reed, 1991; Smalla et al., 2001; Zhang et al., 2012; Zhu et al., 2015; Su et al., 2018), 用无菌自封袋密封, 于低温(4 ℃)下运回实验室.一部分样品经冷冻干燥后保存在-80 ℃, 其余部分样品于4 ℃下冷藏.
表 1(Table 1)
| 表 1 北澧河旱地4种不同作物根际和非根际的土壤样品信息及理化数据 Table 1 oil sample information and physical and chemical characteristics of rhizosphere and non-rhizosphere from 4 different crops in the dry land of Beilihe | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 1 北澧河旱地4种不同作物根际和非根际的土壤样品信息及理化数据 Table 1 oil sample information and physical and chemical characteristics of rhizosphere and non-rhizosphere from 4 different crops in the dry land of Beilihe
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
2.2 样品理化指标检测对土壤基本理化指标的检测参照《土壤农化分析》(鲍士旦, 2000)中的方法进行.含水率以样品在105 ℃ 4 h的条件下的失水量来计算;总有机质以样品在550 ℃下的烧失量计算(LOI550)(张文河等, 2007);pH值检测方法为:以土壤:水=1 : 5(W/V)的比例, 室温浸提1 h后测量;土壤中NH4+-N、NO3--N和NO2--N含量测定通过用0.01 mol · L-1 CaCl2溶液对土壤样品进行浸提1 h(Houba, 1986), 经0.45 μm微孔滤膜过滤得到上清液, 用连续流动分析仪(SAN plus, Skalar Analytical B.V., Breda, the Netherlands)测定.土壤总氮(TN)、总碳(TC)和总硫(TS)采用元素分析仪(Vario TOC)经高温灼烧的方法进行检测, 首先需将风干的土壤样品过100目筛(0.149 mm), 样品所有理化性质都进行3次重复测定取平均值(表 1).
2.3 样品DNA提取和聚合酶链式反应(PCR)利用FastDNA Spin Kit for Soil DNA (MP Biomedicals, USA)提取试剂盒, 称取约0.33 g冷冻干燥的土壤样品, 参照试剂盒中的提取方法, 提取样品总DNA, 用Nano Drop 2000 UV-Vis Spectrophotometer (Thermo Fisher Scientific, USA)测定DNA浓度, 同时用1%琼脂糖凝胶电泳检测提取DNA的质量, 将DNA样品保存在-20 ℃冰箱中备用.用DNRA细菌功能基因nrfA的特异性引物nrfA-F2aw (5′-CARTGYCAYGTBGARTA-3′)和nrfA-R1 (5′-TWNGGCATRTGRCARTC-3′)对各样品总DNA进行PCR扩增(Welsh et al., 2014).扩增体系为50 μL:10×buffer 5 μL, dNTP (2.5 mmol · L-1) 4 μL, 浓度为10 μmol · L-1的前、后引物各1 μL, BSA 0.5 μL, Taq酶(2.5 U) 0.25 μL, 稀释10倍的DNA模板4 μL, 用ddH2O补足50 μL (Zhu et al., 2015).扩增条件为:95 ℃预变性10 min;95 ℃变性30 s, 52 ℃退火30 s, 72 ℃延伸20 s, 40个循环;72 ℃最后延伸10 min.取5 μL PCR扩增产物用1%凝胶进行电泳检测扩增条带是否单一, 条带大小是否正确.
2.4 高通量测序及生物信息分析用带有Barcode信息的引物(高通量测序时用以区分不同样品)对样品nrfA进行PCR扩增.获得的PCR产物用QIAquick凝胶提取试剂盒(德国)进行回收, 依托Illumina MiSeq PE250平台完成nrfA高通量测序, 由Allwegene公司(奥维森, 北京)构建高通量基因测序文库.经过质量过滤, 得到最终的序列用于OTU分析.
高通量分析遵循以下流程:①从公司获得高质量FA格式文件数据, 通过Excel, Notapad和Bioedit软件筛除低质量序列, 然后利用Qiime和Mothur软件获得非冗余的核酸序列(Schloss et al., 2009).②序列的聚类:用Bioedit软件将非冗余的核酸序列翻译成氨基酸序列, 然后通过Uclust软件将氨基酸序列按照90%的相似度进行聚类(Edgar, 2010; Wang et al., 2018), 得到所有OTUs的序列分配结果.③在OTUs水平进行生态学分析:将每个OTUs代表的氨基酸序列在Genbank数据库进行BLAST(Basic Local Alignment Search Tool)比对并注释;通过编写的Perl脚本计算多样性等(庄林杰等, 2017).
2.5 数据分析根据Bray-Curtis差异度指数进行群落的分类和排序, 分类选择UPGMA算法(Mothur计算), 排序选用主坐标分析(Principle Coordinate Analysis, PCoA).用SPSS 21.0软件(Statistical Product and Service Solutions)进行相关性分析(采用Spearman相关系数), 利用Canoco 4.5进行去趋势对应分析(Detrended Correspondence Analysis, DCA)和去冗余分析(Redundancy analysis, RDA).
3 结果(Results)3.1 DNRA菌高通量测序结果对质控后的每个样品序列进行均一化处理, 取序列数量最小值, 即每个样品抽取87000条序列.使用Qiime、Uchime、Mothur、Framebot等生物信息学分析软件对序列进行严格质量控制并进行多样性分析处理.将每个代表OTU的氨基酸序列在NCBI数据库进行BLAST比对并进行物种注释.结果表明质控优化后的序列均为DNRA细菌的nrfA的氨基酸序列.优化后的高质量序列条数在相似度≥90%下进行聚类(Wang et al., 2018), 得到OTUs数及其覆盖率分布情况如表 2所示.
表 2(Table 2)
| 表 2 高通量测序样品描述 Table 2 Summary of high-throughput sequencing results | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 2 高通量测序样品描述 Table 2 Summary of high-throughput sequencing results
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
根据表中统计结果可以得到, 样品中OTUs数量在2699~7276, 覆盖率均大于96%.采用对优化序列进行随机抽样的方法, 以抽到的序列数与它们所得出的代表OTU的数目构建稀释性曲线(图 1)可以发现, 随着测序条数的增加, 样本稀释曲线渐渐趋于平缓, 说明测序深度合理.
图 1(Fig. 1)
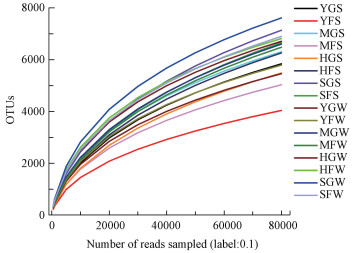 |
| 图 1 DNRA细菌的稀释曲线 Fig. 1The rarefaction curve of nrfA OTUs |
3.2 DNRA菌群α-多样性选取丰富度指数(Chao1 index)、多样性指数(Shannon index)和优势度指数(Simpson index, 指数越高, 群落内物种数量分布越不均, 物种优势度越高, 均匀度越低)为参数, 对DNRA菌群α-多样性进行分析(表 3).由表 3中不同样本的α-多样性指数可以得出:夏季的玉米、棉花、粟和冬季棉花、黄豆及粟作物的根际土壤样品中DNRA菌的Chao1 index和Shannon index高于相应的非根际样品, 对应的根际样品中DNRA菌的Simpson index低于非根际样品.4种作物间进行比较, 发现粟作物根部土壤DNRA群落α-多样性最高, 而玉米作物非根际土壤最低.
表 3(Table 3)
| 表 3 DNRA菌群α-多样性 Table 3 The α-diversity index of DNRA bacteria | |||||||||||||||||||||||||||||||||||||||||||||||||||
表 3 DNRA菌群α-多样性 Table 3 The α-diversity index of DNRA bacteria
| |||||||||||||||||||||||||||||||||||||||||||||||||||
并对DNRA细菌的α-多样性指数进行单个样本T检验(表 4), 检验结果发现不同样品间的α-多样性指数具有显著相关关系(p < 0.05), 同时验证了DNRA细菌的α-多样性结果的可靠性.
表 4(Table 4)
| 表 4 对DNRA细菌的α-多样性指数进行T检验(n=16) Table 4 One-Sample Test for the α-diversity index of DNRA bacteria (n=16) | ||||||||||||||||||||||||||||||||||||
表 4 对DNRA细菌的α-多样性指数进行T检验(n=16) Table 4 One-Sample Test for the α-diversity index of DNRA bacteria (n=16)
| ||||||||||||||||||||||||||||||||||||
3.3 DNRA菌群β-多样性在非限制性排序分析(Unconstrained ordination analysis)中, 基于Bray-Curtis差异度指数, 计算不同样点间的差异, 利用主坐标分析(principal coordinate analysis, PCoA)对旱地DNRA细菌的高通量数据进行β-多样性分析(图 2), 选取解释度最大的两轴作图, 它们对排序结果的解释度分别为33.00%和16.07%.
图 2(Fig. 2)
 |
| 图 2 PCoA排序分析 Fig. 2PCoA of DNRA community in root soil of crops |
图中结果显示:相同季节的同种作物根际和非根际样品间(如:YGS和YFS;MGW和MFW;SGW和SFW等)的DNRA细菌群落组成在两轴上的排序距离均较近;单纯以作物种类为变量的比对条件下(如:YFS、MFS、HFS和SFS), 样品DNRA群落结构在第一轴上的排序较近, 在第二轴上的排序较远;单独对温度进行分析(如:YFS和YFW;HFS和HFW等), DNRA群落结构在两轴上的排序距离均较远.对比不同的变量, 发现根际和非根际对旱地农田土壤中DNRA细菌群落组成影响最小, 作物种类对DNRA细菌群落多样性影响较大, 季节变化(即温度的不同)对DNRA细菌群落影响最大.
3.4 DNRA菌群落结构分析标准化处理后的OTU数据, 选取占序列总数为60%的前258个OTUs作为代表OTUs(Lundberg et al., 2012), 并选取相对丰度较高的前50个OTUs, 绘制热图(Heatmap) (图 3a).热图中的每一个色块代表一个样品对应的OTU, 样品横向排列, OTUs纵向排列, 为了更好地体现差异, 不同OTUs的序列条数通过log(x+1)进行转换.相同作物的土壤样品聚在一起, 与PCoA分析结果吻合.划分到OTU11869的序列条数最多, 通过BLAST比对发现, 该OTU代表的nrfA的序列属于硝化螺菌属(Nitrospira);其次为OTU7569、OTU6479和OTU5667, 均被定义为Chthoniobacter属.Nitrospira属为脱氮除磷菌属, 常被应用于污水处理系统.Chthoniobacter属归于疣微菌门(Verrucomicrobia), 一种革兰氏阴性菌, 在厌氧条件下或以硝酸盐为电子受体进行生命活动, 广泛存在于土壤中, 和线虫存在着共生关系(Sangwan et al., 2004; Kant et al., 2011; Bergmann et al., 2011).
图 3(Fig. 3)
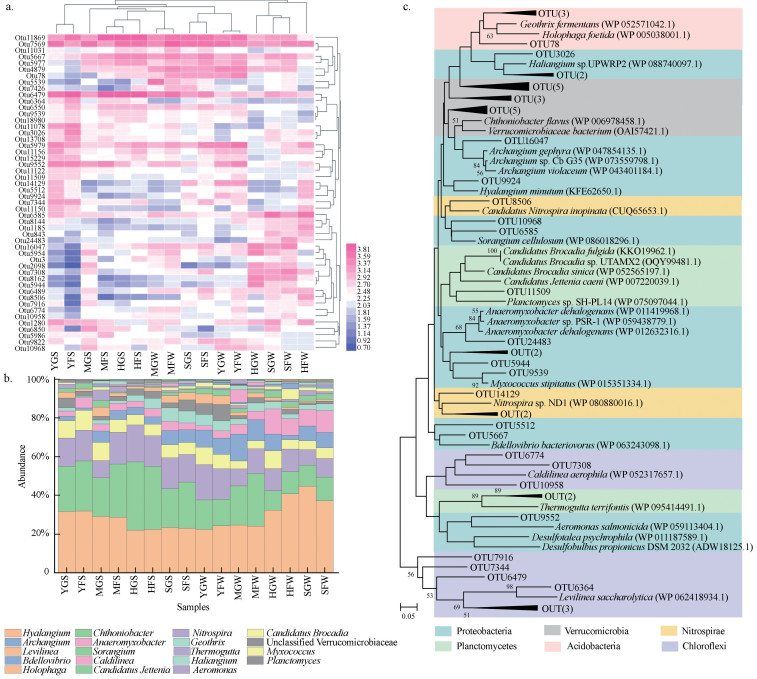 |
| 图 3 不同样点DNRA细菌前50个OTUs的热图(a)及DNRA细菌的相对丰度图(b)和 nrfA的系统发育树图(c) Fig. 3Heatmap of the top 50 OTUs of DNRA bacteria (a), relative abundance of DNRA bacteria (b), phylogenetic tree of nrfA (c) |
将高通量测序获得的序列上传至NCBI, 登录号为SRP158889.对序列进行注释, 共定义到6个门.分别为酸杆菌门(Acidobacteria)、绿弯菌门(Chloroflexi)、硝化螺旋菌门(Nitrospirae)、浮霉菌门(Planctomycetes)、变形菌门(Proteobacteria)和疣微菌门(Verrucomicrobia).其中Proteobacteria、Verrucomicrobia、Nitrospirae分别占总序列条数的47.13%、23.45%和13.41%, 为所有样点中占主导地位的3类微生物.在属(Genus)水平的分类, 共定义到19个属(图 3b).其中, 相对丰度最高的3个属Hyalangium、Chthoniobacter、Nitrospira分别占29.31%、20.33%和13.41%.
选取相对丰度较高的前50个OTUs的氨基酸序列和nrfA的参比氨基酸序列, 用MEGA 6.0软件以邻接法(NJ)构建系统发育树(图 3c)(Zhou et al., 2014).分支节点上的数字表示进行1000次的bootstrap分析所支持的次序, 保留大于50的数值, 图中标尺表示5%序列差异的分支长度, 背景颜色代表门的分类.系统发育树共划分出6个门, 其中Proteobacteria门覆盖最广, 包含占比最多的种属(Hyalangium)和硫酸盐还原菌:脱硫球茎菌属(Desulfobulbus)及脱硫化针菌属(Desulfotalea) (Song et al., 2014)等种属;Planctomycetes门包含了厌氧氨氧化菌(Candidatus Brocadia和Candidatus Jettenia)(Zhu et al., 2018)等.
3.5 DNRA细菌与土壤理化因子相关性分析利用SPSS 21.0对DNRA细菌的相对丰度与土壤环境因子进行相关性分析, 来探索关键环境因子对DNRA群落结构的影响.通过统计学分析结果显示, NH4+ -N、NO2--N、TN、TS、含水率(Moisture Content, MC)、TOM、C/N、pH以及温度(T)对DNRA菌群落均具有显著相关性(p < 0.05, n=16), 且NO2--N、TN、TOM、MC、pH及T对DNRA菌群落间相关性显著(p < 0.01, n=16), NO3--N及TC和DNRA菌群落之间无显著相关性.将具有显著相关性的DNRA种属和环境因子利用Canoco 4.5软件, 对数据进行去趋势对应分析(Detrended Correspondence Analysis, DCA), 得到的最长排序长度为2.023, 小于3, 则选用线性模型的排序方法对数据做RDA分析(图 4).由RDA分析得出, 影响DNRA群落结构的前5的影响因子分别为T、NO2--N、TOM、C/N和MC.相对丰度最高的前5个种属与环境因子之间的相关性:Hyalangium 与MC(r= 0.526, p=0.036)、pH (r= 0.571, p=0.021)呈显著正相关, 与C/N (r= -0.608, p=0.012)呈显著负相关;Chthoniobacter与NH4+-N (r= -0.532, p=0.034)、NO2--N (r= -0.514, p=0.042)、TN (r= -0.568, p=0.022)、MC(r= 0.508, p=0.044)呈显著负相关, 与T(r=0.651, p=0.006)呈显著正相关, Nitrospira与TOM (r=0.609, p=0.012)呈显著正相关;Candidatus Brocadia 与TOM (r= 0.534, p=0.033)呈显著正相关;Archangium与NO2--N (r=0.711, p=0.002)呈显著正相关, 与MC(r= 0.596, p=0.015)呈显著正相关, 和TOM (r= -0.542, p=0.03)呈显著负相关, 与T(r= -0.705, p=0.002)呈显著负相关.
图 4(Fig. 4)
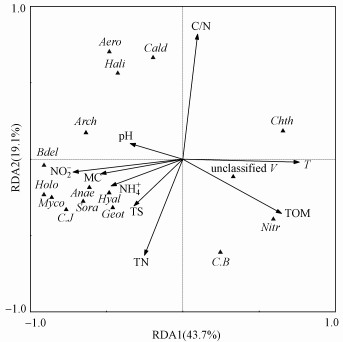 |
| 图 4 DNRA群落与环境因子的RDA分析 Fig. 4RDA analysis of DNRA bactreial community and environmental factors |
4 讨论(Discussion)随着对地球氮循环不断深入的研究, 发现DNRA细菌广泛分布于不同领域, 本论文针对旱地作物根系土壤中DNRA细菌展开研究.有研究表明作物根系的呼吸作用和根系分泌的可被生物利用的有机质的分解形成了根区厌氧环境, 同时凋落物的分解也能够为微生物生长提供无机氮源和有机碳源(Guo et al., 2003; Patra et al., 2006), 推测这些因素都是根际土壤中DNRA菌群落多样性高的环境条件.在自然生态系统条件下, 植物同化作用固定的有机碳会随着植物的死亡或茎叶凋落腐烂储存于土壤有机碳中, 使得土壤环境中有着丰富的碳源.一般认为DNRA过程在氮源缺乏、碳源充足(不稳定碳)的条件下更容易发生, 而反硝化作用则更适合碳源受限条件(傅利剑等, 2005).即与反硝化相比, 在高C/N环境系统中DNRA占据着更重要的地位(Bonin et al., 1996), 本实验测得旱地土壤C/N在14.0~22.2之间, 属于较高程度, 是推动DNRA过程的一个重要因素.在自然环境中DNRA对氮去除的贡献有时会高达90% (Wood et al., 2003).有研究揭示在暴雨之后施撒肥料可以增加有机质矿化, 消耗土壤中的O2, 进而抑制土壤微生物的硝化作用(Ball et al., 2004), 微氧环境中, 细菌的DNRA反应较高溶解氧和厌氧条件下活跃(刘佳, 2007).同时也表明土壤水分的增加可以降低土壤中氧含量, 可以促进土壤细菌进行DNRA反应将土壤中NO3--N(或NO2--N)还原生成NH4+-N以被作物吸收利用.由RDA分析发现土壤含水率与DNRA种群结构具有显著相关性, 且属于主要的影响因子.土壤中一定的含水率对细菌的生长和生命活动是有利的, 可以提高细胞在土壤中的移动速度, 提高细胞中水分含量, 为细胞内物质传递提供良好的介质.作物种类、氮肥填加等人为扰动影响了土壤理化性质, 加之温度等环境因素改变DNRA群落丰度和结构, 从而影响硝酸盐及亚硝酸盐的还原和铵的产生.温度的高低季节性变化将决定该地区微生物的种群结构变化(Ogilivie et al., 1997).韦宗敏等对珠江口沉积物中DNRA细菌进行培养实验发现DNRA菌最适温度为30 ℃ (韦宗敏, 2012).本研究中也发现冬夏两季样品中DNRA群落组成及分布存在差异, 由RDA分析发现温度对DNRA菌群落影响最大, 进一步确认了温度对DNRA细菌的影响.
值得关注的是, 在主要参与亚硝酸盐氧化反应的硝化螺菌属(Nitrospira)常被定义为NOB (Nitrite Oxidizing Bacteria), 同时Nitrospira属中多个物种包含有nrfA和反硝化功能基因, 如菌株Nitrospira ND1的基因组序列中包含了细胞色素c的编码亚硝酸盐还原酶(nrfA) (Ushiki et al., 2017).具有DNRA功能的硝化螺菌属在旱地土壤中大量存在并进行着硝化作用, 林地、草地和农田土壤生态系统中主要的NOB群落是Nitrobacter(硝化杆菌属)和Nitrospira(韩江培, 2015;Wang et al., 2015), 并且Nitrospira种群的多样性高于Nitrobacter(Thomas et al., 2005).Nitrospira的大量存在以及较高丰度, 这是一个普遍现象.在nrfA高通量分析中还发现了Candidatus Brocadia和Candidatus Jettenia属(Anammox细菌) (Zhu et al., 2018).既能进行DNRA反应将土壤中硝酸盐还原为铵, 又能将亚硝酸盐作为底物, 挥发性脂肪酸(VFA)为电子供体进行Anammox反应(Guven et al., 2005;Kartal et al., 2007; Shu et al., 2015).有研究发现, 硝酸盐去除很大程度是由环境中硫化物的浓度决定的, 硫化物对反硝化过程的终端反应产生一定的抑制作用, 进而使产物向铵转变(Tiedje et al., 1983).通过对沉积物的研究发现, 硫化物(如H2S、S2-和S)作为电子供体, 能够为DNRA反应提供电子, 促进DNRA的发生(Sher et al., 2008; Lu et al., 2013).DNRA可以通过无机化能自养硫细菌氧化达到硝酸盐去除的目的, 但有些硫细菌如Desulfovibrio desulfuricans, 不能代谢NO3-, 需要依靠外源NO2-进行代谢(Steenkamp et al., 1981).但由RDA分析发现样品中总硫对DNRA细菌的影响小于其他环境因子, 可能是因为旱地土壤中硫化物的含量明显低于水淹状态下的湿地及河口沉积物中的含量, 导致旱地土壤中的硫细菌较少发挥DNRA作用.
DNRA过程的代谢终产物NH4+, 可被作物更好的吸收利用, 不会产生氮损失, 可以提高氮肥利用率.在自然界, 特别是农田生态系统中, DNRA微生物的存在, 可以更好地维持土壤活力.通过研究DNRA微生物与环境因子之间的相关性, 可以发现对DNRA微生物丰度及组成产生影响的关键因素, 为DNRA微生物在农业中的应用起到指导作用, 探索如何更好发挥DNRA对土壤中的氮保留功效, 减少NO3-的淋流具有重要的研究意义.
5 结论(Conclusions)在黄河中下游平原典型旱地农田土壤中, DNRA细菌广泛分布于不同作物的根际和非根际土壤中.样品中3/4的作物根际土壤样品中的DNRA细菌物种丰富度和多样性高于非根际样品, 且冬夏两季均出现这样的规律;土壤样品中不同的变量对DNRA细菌群落组成影响程度由大到小为:季节变化>作物种类>根际;环境因子NO2--N、TN、TOM、含水率、pH及温度与DNRA菌群落间相关性显著.本研究在一定程度上揭示了旱地农田土壤DNRA细菌的群落组成、多样性及与土壤环境因子的关系, 为提高氮肥的利用效率和减小环境污染提供理论依据.
致谢:感激祝贵兵作者洪堡研究奖学金(No.1152633)的支持, 以及中国科学院青年创新促进会项目.
参考文献
| Ball B C, Mc Taggart I P, Scott A. 2004. Mitigation of greenhouse gasemissions from soil under silage production by use of organicmanures or slow-release fertilizer[J]. Soil Use and Management, 20(3): 287–295.DOI:10.1079/SUM2004257 |
| 鲍士旦. 2000. 土壤农化分析(第3版)[M]. 北京: 中国农业出版社. |
| Bergmann G T, Bates S T, Eilers K G, et al. 2011. The under-recognized dominance of Verrucomicrobia in soil bacterial communities[J]. Soil Biology & Biochemistry, 43(7): 1450–1455. |
| Blackmer A M, Bremner J M. 1978. Inhibitory effect of nitrate on reduction of N2O to N2 by soil microorganisms[J]. Soil Biology and Biochemistry, 10(3): 187–191.DOI:10.1016/0038-0717(78)90095-0 |
| Bonin P. 1996. Anaerobic nitrate reduction to ammonium in two strains isolated from coastal marine sediment:a dissimilatory pathway[J]. FEMS Microbiology Ecology, 19(1): 27–38.DOI:10.1111/fem.1996.19.issue-1 |
| Deng Y, Jiang Y H, Yang Y, et al. 2012. Molecular ecological network analyses[J]. BMC Bioinformatics, 13(1): 113. |
| 都韶婷, 章永松, 林咸永, 等. 2007. 蔬菜积累的硝酸盐及其对人体健康的影响[J]. 中国农业科学, 2007, 40(9): 2007–2014.DOI:10.3321/j.issn:0578-1752.2007.09.021 |
| Edgar R C. 2010. Search and clustering orders of magnitude faster than blast[J]. Bioinformatics, 26(19): 2460.DOI:10.1093/bioinformatics/btq461 |
| 范晓晖, 刘芷宇. 1992. 根际pH环境与磷素利用研究进展[J]. 土壤通报, 1992, 23(5): 238–240.DOI:10.3321/j.issn:0564-3945.1992.05.002 |
| 傅利剑, 郭丹钊, 史春龙, 等. 2005. 碳源及碳氮比对异养反硝化微生物异养反硝化作用的影响[J]. 生态与农村环境学报, 2005, 21(2): 42–45.DOI:10.3969/j.issn.1673-4831.2005.02.009 |
| Guo G X, Deng H, Qiao M, et al. 2013. Effect of long-term wastewaterirrigation on potential denitrification and denitrifying communitiesin soils at the watershed scale[J]. Environmental Science & Technology, 47(7): 3105–3113. |
| Güven D, Dapena A, Kartal B, et al. 2005. Propionate oxidation by and methanol inhibition of anaerobic ammonium-oxidizing bacteria[J]. Applied & Environmental Microbiology, 71(2): 1066–1071. |
| 韩江培. 2015. 设施栽培条件下±壤酸化与盐渍化耦合发生机理研[M]. 杭州: 浙江大学. |
| 贺纪正, 张丽梅. 2013. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98–108. |
| Houba V J G, Novozamsky I, Huybregts A W M, et al. 1986. Comparison of soil extractions by 0.01 M, CaCl2, by EUF and by some conventional extraction procedures[J]. Plant & Soil, 96(3): 433–437. |
| 刘佳.2007.微生物好氧硝酸盐还原产铵研究[D].成都: 四川大学 |
| 蒋自立. 1991. 农作物积累硝酸盐的农业生态因素[J]. 农业环境与发展, 1991, 1: 24–26. |
| J?rgensen K S, Jensen H B, S?rensen J. 1984. Nitrous oxide production from nitrification and denitrification in marine sediment at low oxygen concentrations[J]. Canadian Journal of Microbiology, 30(8): 1073–1078.DOI:10.1139/m84-167 |
| Kant R, Passel M W J V, Palva A, et al. 2011. Genome sequence of Chthoniobacter flavus Ellin428, an aerobic heterotrophic soil bacterium[J]. Journal of Bacteriology, 193(11): 2902–2903.DOI:10.1128/JB.00295-11 |
| Kartal B, Kuypers M M, Lavik G, et al. 2007. Anammox bacteria disguised as denitrifiers:nitrate reduction to dinitrogen gas via nitrite and ammonium[J]. Environmental Microbiology, 9(3): 635–642.DOI:10.1111/emi.2007.9.issue-3 |
| Kraft B, Strous M, Tegetmeyer H E. 2011. Microbial nitrate respiration--genes, enzymes and environmental distribution[J]. Journal of Biotechnology, 155(1): 104–117.DOI:10.1016/j.jbiotec.2010.12.025 |
| 刘文国, 范学科, 马安良. 2002. 植物体对氮吸收和同化过程的研究进展[J]. 杨凌职业技术学院学报, 2002(2): 17–19. |
| Lu W W, Zhang H L, Shi W M. 2013. Dissimilatory nitrate reduction to ammonium in an anaerobic agricultural soil as affected by glucose and free sulfide[J]. European Journal of Soil Biology, 58(58): 98–104. |
| Lundberg D S, Lebeis S L, Paredes S H. 2012. Defining the core Arabidopsis thaliana root microbiome[J]. Nature, 488(7409): 86.DOI:10.1038/nature11237 |
| 马冬云, 郭天财, 宋晓, 等. 2007. 尿素施用量对小麦根际土壤微生物数量及土壤酶活性的影响[J]. 生态学报, 2007, 27(12): 5222–5228.DOI:10.3321/j.issn:1000-0933.2007.12.032 |
| Maag M, Vinther F P. 1996. Nitrous oxide emission by nitrification and denitrification in different soil types and at different soil moisture contents and temperatures[J]. Applied Soil Ecology, 4(1): 5–14.DOI:10.1016/0929-1393(96)00106-0 |
| Mroz G D, Reed D D. 1991. Forest soil sampling efficiency:Matching laboratory analyses and field sampling procedures[J]. Soil Science Society of America Journal, 55(5): 1413–1416.DOI:10.2136/sssaj1991.03615995005500050035x |
| Müller M M, Sundman V, Skujin? J. 1980. Denitrification in low pH spodosols and peats determined with the acetylene inhibition method[J]. Applied and Environmental Microbiology, 40(2): 235–239. |
| Ogilvie B G, Rutter M, Nedwell D B. 1997. Selection by temperature of nitrate-reducing bacteria from estuarine sediments:species composition and competition for nitrate[J]. FEMS Microbiology Ecology, 23(1): 11–22.DOI:10.1111/j.1574-6941.1997.tb00386.x |
| Patra A K, Abbadie L, Clays-Josserand A, et al. 2006. Effects ofmanagement regime and plant species on the enzyme activity andgenetic structure of N-fixing, denitrifying and nitrifying bacterialcommunities in grassland soils[J]. Environmental Microbiology, 8(6): 1005–1016.DOI:10.1111/emi.2006.8.issue-6 |
| Rehr B, Klemme J H. 1989. Formate dependent nitrate and nitrite reduction to ammonia by Citrobacter freundii, and competition with denitrifying bacteria[J]. Antonie Van Leeuwenhoek, 56(4): 311–321.DOI:10.1007/BF00443745 |
| Sangwan P, Chen X, Hugenholtz P, et al. 2004. Chthoniobacter flavus gen. nov. sp nov. the first pure-culture representative of subdivision two, Spartobacteria classis nov. of the phylum Verrucomicrobia[J]. .Applied & Environmental Microbiology, 70(10): 5875–5881. |
| Senga Y, Mochida K, Fukumori R, et al. 2006. N2O accumulation in estuarine and coastal sediments:The influence of H2S on dissimilatory nitrate reduction[J]. Estuarine, Coastal and Shelf Science, 67(1/2): 231–238. |
| Schloss P D. 2009. A high-throughput DNA sequence aligner for microbial ecology studies[J]. Plos One, 4(12): e8230.DOI:10.1371/journal.pone.0008230 |
| Sher Y, Schneider K, Schwermer C U, et al. 2008. Sulfide-induced nitrate reduction in the sludge of an anaerobic digester of a zero-discharge recirculating mariculture system[J]. Water Research, 42(16): 4386–4392.DOI:10.1016/j.watres.2008.07.031 |
| Shu D, He Y, Yue H, et al. 2015. Metagenomic insights into the effects of volatile fatty acids on microbial community structures and functional genes in organotrophic anammox process[J]. Bioresource Technology, 196: 621–633.DOI:10.1016/j.biortech.2015.07.107 |
| Smalla K, Wieland G, Buchner A, et al. 2001. Bulk and rhizosphere soil bacterial communities studied by denaturing gradient gel electrophoresis:plant-dependent enrichment and seasonal shifts revealed[J]. Applied and Environmental Microbiology, 67(10): 4742–4751.DOI:10.1128/AEM.67.10.4742-4751.2001 |
| Song B, Lisa J A, Tobias C R. 2014. Linking DNRA community structure and activity in a shallow lagoonal estuarine system[J]. Frontiers in Microbiology, 5: 460. |
| Steenkamp D J, Peck H D Jr.. 1981. Proton translocation associated with nitrite respiration in Desulfovibrio desulfuricans[J]. The Journal of Biological Chemistry, 256(11): 5450–5458. |
| Su Y, Wang W, Wu D, et al. 2018. Stimulating ammonia oxidizing bacteria (AOB) activity drives the ammonium oxidation rate in a constructed wetland (CW)[J]. Science of the Total Environment, 624: 87–95.DOI:10.1016/j.scitotenv.2017.12.084 |
| 孙羲. 1997. 植物营养原理, 农学, 园艺, 桑茶等专业用[M]. 北京: 中国农业出版社. |
| Tiedje J M, Sexstone A J, Myrold D D, et al. 1983. Denitrification:ecological niches, competition and survival[J]. Antonie van Leeuwenhoek, 48(6): 569–583.DOI:10.1007/BF00399542 |
| Thomas E F, Lisa C H, Christpher D C, et al. 2005. Influence of inorganic nitrogen management regime on the diversity of nitrite-oxidizing bacteria in agricultural grassland soils[J]. Applied & Environmental Microbiology, 71: 8323–8334. |
| Uldahl A.2011.Alternative nitrate reduction pathways in experimentally fertilized new england salt marshes: removal versus recycling of biologically available N[C].21st Biennial Conference of the Coastal and Estuarine Research Federationhttps://link.springer.com/article/10.1007/s12237-014-9858-6 |
| Ushiki N, Fujitani H, Shimada Y, et al. 2017. Genomic Analysis of Two Phylogenetically Distinct Nitrospira Species Reveals Their Genomic Plasticity and Functional Diversity[J]. Frontiers in Microbiology, 8: 1–7. |
| Wang B Z, Zhao J, Guo Z Y, et al. 2015. Differential contributions of ammonia oxidizers and nitrite oxidizers to nitrification in four paddy soils[J]. The ISME Journal, 9: 1062–1075.DOI:10.1038/ismej.2014.194 |
| Wang S, Wang W, Liu L, et al. 2018. Microbial nitrogen cycle hotspots in the plant-bed/ditch system of a constructed wetland with N2O mitigation[J]. Environmental Science & Technology, 52(11): 6226–6236. |
| 韦宗敏.2012.微好氧环境中硝酸盐异化还原成铵的影响研究[D].广州: 华南理工大学http://cdmd.cnki.com.cn/Article/CDMD-10561-1012450921.htm |
| Welsh A, Chee-Sanford J C, Connor L M, et al. 2014. Refined nrfA phylogeny improves PCR-based nrfA gene detection[J]. Applied & Environmental Microbiology, 80(7): 2110–2119. |
| Wood G A, Taylor J C, Godwin R J. 2003. Calibration Methodology for Mapping Within-field Crop Variability using Remote Sensing[J]. Biosystems Engineering, 84(4): 409–423.DOI:10.1016/S1537-5110(02)00281-7 |
| Yamulki S, Harrison R M, Goulding K W T, et al. 1997. N2O, NO and NO2 fluxes from a grassland:effect of soil pH[J]. Soil Biology and Biochemistry, 29(8): 1199–1208.DOI:10.1016/S0038-0717(97)00032-1 |
| 雍太文, 杨文钰, 向达兵, 等. 2012. 不同种植模式对作物根系生长、产量及根际土壤微生物数量的影响[J]. 应用生态学报, 2012, 23(1): 125–132. |
| Zhang L M, Hu H W, Shen J P, et al. 2012. Ammonia-oxidizing archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. Isme Journal, 6(5): 1032–1045.DOI:10.1038/ismej.2011.168 |
| 张文河, 穆桂金. 2007. 烧失法测定有机质和碳酸盐的精度控制[J]. 干旱区地理(汉文版), 2007, 30(3): 137–141. |
| Zhong W H, Cai Z C. 2004. Methods for studying soil microbial diversity[J]. Chinese Journal of Applied Ecology, 58(2): 169–188. |
| Zhou J, Ye D, Feng L, et al. 2011. Phylogenetic Molecular Ecological Network of Soil Microbial Communities in Response to Elevated CO2[J]. Mbio, 2(4): e00122–11. |
| Zhou J M, Song Z Y, Yan D J, et al. 2014. Performance of a haloalkaliphilic bioreactor under different, NO3-/SO42- math Container Loading Mathjax, ratios[J]. Bioresource Technology, 153(1): 216–222. |
| Zhu G, Jetten M S M, Kuschk P, et al. 2010. Potential roles of anaerobic ammonium and methane oxidation in the nitrogen cycle of wetland ecosystems[J]. Applied Microbiology & Biotechnology, 86(4): 1043–1055. |
| Zhu G, Wang S, Ma B, et al. 2018. Anammox granular sludge in low-ammonium sewage treatment:Not bigger size driving better performance[J]. Water Research, 142: 147–158.DOI:10.1016/j.watres.2018.05.048 |
| Zhu G, Xia C, Wang S, et al. 2015. Occurrence, activity and contribution of anammox in some freshwater extreme environments[J]. Environmental Microbiology Reports, 7(6): 961–969.DOI:10.1111/1758-2229.12341 |
| Zhu G, Wang S, Wang Y, et al. 2011. Anaerobic ammonia oxidation in a fertilized paddy soil[J]. Isme Journal, 5(12): 1905–1912.DOI:10.1038/ismej.2011.63 |
| 朱永官, 王晓辉, 杨小茹, 等. 2014. 农田土壤N2O产生的关键微生物过程及减排措施[J]. 环境科学, 2014, 1(2): 792–800. |
| 庄林杰, 夏超, 田晴, 等. 2017. 高通量测序技术研究典型湖泊岸边陆向深层土壤中厌氧氨氧化细菌的群落结构[J]. 环境科学学报, 2017, 37(1): 261–271. |
