复杂疾病相关变异的功能鉴定一直是遗传学研究中的重要问题。肿瘤患者中含有大量的核苷酸变异,包括遗传性变异和体细胞变异,这些变异的致病性判断对肿瘤的风险评估、干预和治疗都具有重要的意义。受到技术限制,目前仍缺乏能够高效评估这些变异功能的高通量筛选平台。近年来,基于CRISPR/Cas9的单碱基编辑器的出现使人们可以在基因组的特定位置上实现C-T [1]和A-G [2]的单点突变,为高通量鉴定点突变的功能提供了可能。2021年2月两篇背靠背发表在Cell杂志上的文章,采用单碱基编辑器CBE (Cytosine Base Editor),对BRCA1/2等DNA损伤应答基因的部分点突变进行了高通量功能筛选[3, 4]。因为这些基因的有害突变对细胞生长有负面影响,通过碱基编辑后不同sgRNA的丰度变化,可以评估其对应的变异对基因功能的影响。然而由于单碱基编辑器工具在基因组不同位点上的编辑效率存在显著差异,并且同一个向导RNA(sgRNA)可能会在基因组上产生不止一种突变,筛选得到的sgRNA对应的基因变异效应可能并不符合预期。
针对这一问题,王晓月团队提出了结合效率校正的单碱基编辑高通量筛选方法。这一方法通过对sgRNA编辑结果的高通量测定,将sgRNA和所产生的变异精确对应,再使用一个效率校正模型去将sgRNA对细胞的效应转化为变异的致病性得分,从而提升变异功能评估的准确性(如图1所示)。作者发现,使用效率校正可将已知致病性突变的检出率提高50%以上。
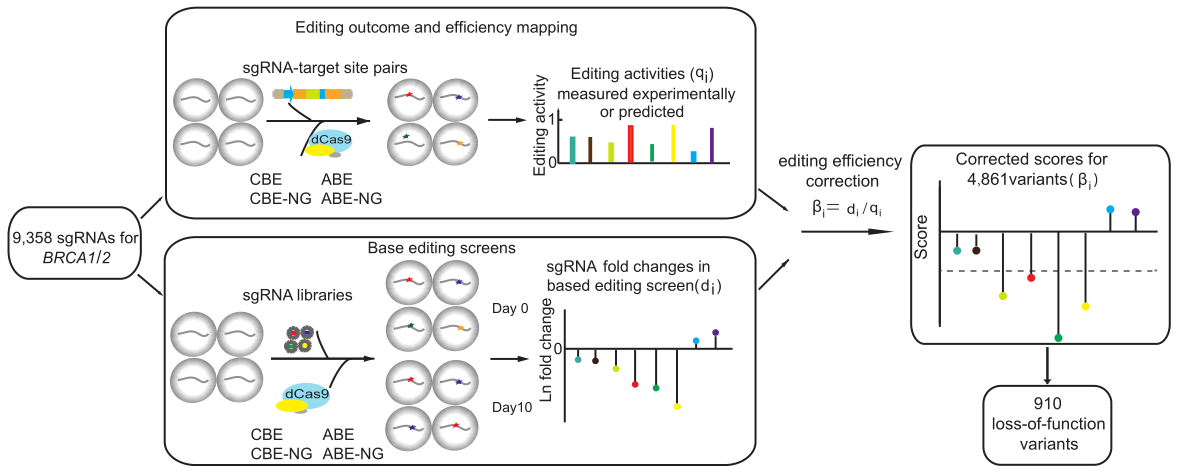
图1结合单碱基编辑效率校正的鉴定有害突变的高通量筛选方法
运用这种方法,王晓月团队使用单碱基编辑工具CBE和ABE(Cytosine Base Editor)对两个肿瘤抑制基因BRCA1和BRCA2的 9000多种变异进行了高通量功能分析,共鉴定出了910种可能影响BRCA1/2功能的变异。这是目前针对BRCA1/2基因全长范围的最大规模的突变功能筛选,筛选得到的有害变异广泛分布于两个基因编码蛋白的不同结构域,为BRCA1/2蛋白的功能解析提供了新的线索。值得注意的是,鉴定出的变异中包括一些在肿瘤家系中存在、而在临床上被判定为意义不明的遗传性变异,说明这些位点的致病性可能需要重新判别。这种基于单碱基编辑的高通量筛选方法可以广泛应用于基因组不同位置的变异功能解析,将为肿瘤等疾病的遗传风险预测和精确诊疗提供重要信息。
本研究工作得到中国医学科学院医学与健康科技创新工程(2017-I2M-4-003,2019-I2M-1-004)等项目的资助。基础医学研究所王晓月教授和梁俊波博士为论文共同通讯作者,黄昌财博士和李光宇硕士为论文的共同第一作者。
论文链接:https://genomebiology.biomedcentral.com/articles/10.1186/s13059-021-02305-2
基础医学研究所
2021年3月16日
