0 引言
【研究意义】畜禽屠宰性能由于与经济效益密切相关,在畜禽育种工作中一直备受重视,但屠宰性状属于数量性状,受多基因控制,易受环境影响,因此采用传统的选育方法进行改良进展缓慢。分子标记由于其结果不受环境影响,不存在等位基因显隐性表型关系,并且可进行早期选择,被认为是缩短育种周期,加快遗传进展的有效手段。但是基因之间往往存在互作效应,因此单基因分子标记往往会出现顾此失彼的现象,而多基因聚合的分子育种技术将有望弥补这方面的缺陷。因此,本研究选择在畜禽研究中被证明与畜禽屠宰性状相关的两个候选基因-肌细胞生成素(MyoG)基因和肌细胞增强因子2a(MEF2a)基因,进行两基因聚合效应对屠宰性状的影响研究,为实现畜禽的多基因聚合育种提供依据。【前人研究进展】由于MyoG基因和MEF2a基因在肌细胞分化和肌肉形成中发挥重要作用,而肌肉的发育与畜禽屠宰性状息息相关,所以这两个基因在畜禽研究中一直被作为屠宰性状的候选基因。MyoG基因是生肌家族因子中最重要的一员,在肌细胞的分化过程中起着重要的作用,能促进肌细胞的增殖和分化[1]。而MEF2基因家族(MEF2a、MEF2b、MEF2c和MEF2d)在骨骼发育、肌肉形成、肝脏纤维化和神经系统发育等多种生理过程中发挥作用,同时它也参与一些疾病的发生,如阿尔茨海默氏症和帕金森氏病等[2]。在骨骼肌发育的过程中,敲除microRNA可导致试验动物在胚胎期致死或骨骼肌发育不全、肌纤维形态异常、肌原细胞凋亡和成肌细胞死亡增加[3-4]。LIU等[5]通过构建小干扰RNA质粒载体获得敲除牛MyoG基因骨骼肌细胞的试验结果显示该基因的表达显著低于其他试验阳性基因。NEVILLE等[6]研究表明,MyoG通过控制成肌细胞的融合和肌纤维的形成而对肌肉的分化起关键作用,是唯一能在所有骨骼肌细胞中表达的基因,是骨骼肌分化所必须的基因。许多骨骼肌和心肌基因的调控区都存在MEF2的结合位点,能够与大多数肌肉特异基因的启动子或增强子直接结合,在所有的肌肉细胞类型中可作为肌源性基因表达的主要调节物[7-9]。LIU[10]等对鸭的MEF2a基因mRNA表达进行探究,发现在平滑肌中的表达量高于心肌和骨骼肌。 JUSZCZUKKUBIAK等[11]对波兰荷斯坦奶牛MEF2a基因启动子区多态性及该基因mRNA表达水平进行研究,结果显示,启动子区域突变的不同基因型个体的背最长肌MEF2a基因mRNA水平显著高于其他基因型个体,因此认为MEF2a的核苷酸序列突变可能作为牛的生长性状指标分子标记。【本研究切入点】MyoG和MEF2a基因在人、小鼠及其他家畜方面已有大量研究,家禽方面近几年的研究也在逐步增多,但水禽方面的研究相对较少,迄今为止还未见到关于鸭的MyoG和MEF2a基因聚合效应分析的报道。虽然植物的多基因聚合育种已取得了初步成功,但动物的聚合育种还未见到成功的报道。本研究在前人研究的基础上进一步探讨MyoG和MEF2a基因之间的关系,探讨聚合基因型与鸭屠宰性状的关联性,为畜禽多基因聚合育种的研究提供基础。【拟解决的关键问题】以贵州省优良地方品种——三穗鸭为研究对象,首次将MyoG和MEF2a基因的聚合基因型与三穗鸭屠宰性状进行关联分析,进行多基因聚合效应的探讨,为家禽屠宰性状分子标记选育提供依据。1 材料与方法
试验于2014年7月采集样品,2014年10月至2015年6月在贵州大学动物科学学院高原山地动物遗传育种与繁殖教育部重点实验室进行相关操作。1.1 试验材料
17周龄三穗鸭240只(公母各半),相同饲养管理水平,翅静脉采血,肝素钠抗凝,置-70℃冰箱保存。屠宰测定按照中华人民共和国农业部制订的《家禽生产性能名词术语和度量统计方法》(2004)进行[12]。1.2 基因组DNA提取与引物设计
采用血液/细胞/组织基因组DNA提取试剂盒(北京天根)提取基因组DNA。根据NCBI上发布的家鸭MyoG基因编码区全序列(GenBank: NW_004676592.1)、家鸭MEF2a基因编码区全序列(GenBank: NW_004676438.1),应用软件Primer Premier 5.0及Oligo 6.0软件设计引物。引物由上海英潍捷基生物技术有限公司合成,引物详细信息见表1。Table 1
表1
表1MyoG、MEF2a基因引物信息
Table 1The primer sequences and their information for PCR amplification of the MyoG and MEF2a genes in Sansui duck
| 引物名称 Primer name | 引物序列(5′→3′) Primer of sequence | 片段大小 Product size (bp) | 退火温度 Annealing temperature (℃) |
|---|---|---|---|
| MyoG-1 | F:GGGAGGGGCACACTTTTATT | 998 | 62.5 |
| R:TCCGTGATTTAATGCCAGCG | |||
| MyoG-1 | F:CACAGCCAGTTCGTTAATGGT | 334 | 65.0 |
| R:AGGTGGCGTGGTTAGAAGTG | |||
| MyoG-2 | R:AGGTGGCGTGGTTAGAAGTG | 284 | 61.0 |
| R: GGCATTGTGCTGCTGTGAT | |||
| MyoG-3 | F: CAGTGGTGTAGTCCATGGCG | 300 | 59.0 |
| R: CAAGCAGGAGCCGTCCTTGT | |||
| MEF2a-1 | F: ACCGAAGAGTGAAATTTCCTGGAT | 268 | 58.1 |
| R: TGTGTTCTGGTAATGCCATCTGT | |||
| MEF2a-2 | F: GTCCTTTATCCTTTTATCTGGAAA | 300 | 61.0 |
| R: GCCTTAAGGAGATAAGAACACAGAT | |||
| MEF2a-3 | F: TCGTGTAGTTTCAGTGGCTCTT | 324 | 62.0 |
| R: GCACCCTCTGTGTTCCTTCT | |||
| MEF2a-4 | F: TGAGCACAGTCCACTATCGG | 379 | 61.3 |
| R: TGCATTCCCTTCTCAAACCA | |||
| MEF2a-5 | F: TGTGTTCCTCATCTCCTTTCC | 318 | 63.9 |
| R: GTTGCTGTTGGCCATTTGTT | |||
| MEF2a-6 | F: ACAAATGGCCAACAGCAACAA | 162 | 64.0 |
| R: TCAACACAGACATGATATTGCACTT | |||
| MEF2a-7 | F: GTTGTTGATTCTCTTGAGACCCG | 375 | 63.7 |
| R: TCTCCCTGTGCAGACCACTT | |||
| MEF2a-8 | F: TCCCTCTTGTTTGCTGAGTACA | 90 | 66.0 |
| R: TCAGCATCTTGCTCTCTCTCA | |||
| MEF2a-9 | F: AGCACGTAATTTTCCATTGTTCTA | 233 | 59.2 |
| R: ACACGCGACAAATCAAGTCA | |||
| MEF2a-10 | F: GAGCATATCCCATGCACTCACA | 255 | 64.8 |
| R: GATGCCGTACCGTAACCTCG | |||
| MEF2a-11 | F: CCACAGACGCATGAATTGTC | 473 | 61.0 |
| R: GAACACAAAACTGAAGAGGCAAC |
新窗口打开
1.3 PCR扩增
PCR扩增采用30 μL反应体系,包括2×Taq PCR Master Mix(北京天根)12 μL,上、下游引物各3.0 μL(浓度均为10 μmol·L-1),模板DNA(50 ng·μL-1)4 μL,ddH2O 8 μL。反应条件如下:94℃预变性4 min,94℃变性30—35 s,退火30—35 s(退火温度如表1所示),72℃延伸30—40 s,35个循环。扩增完成后,72℃再延伸10 min后,4℃保存。1.4 数据处理分析
采用DNAstar进行序列对比拼接,PHASE 2.0软件进行两基因聚合基因型的判定,Excel 2010进行数据整理,SPSS18.0软件中一般线性模型(GLM)进行关联分析,结果以均值±标准差体现。统计模型如下:单基因效应关联分析数学模型:
Y=μ+Gi+S+Gi×S+e(i=1,2)
聚合基因型效应关联分析数学模型:Y=μ+G+e
其中:Y为性状测定值,μ代表群体均值,Gi为MyoG或MEF2a基因型效应,S为样品性别,Gi×S为基因型与性别互作效应,G为MyoG和MEF2a基因的聚合基因型效应,e为随机残差。
2 结果
2.1 PCR 产物检测
扩增目的片段,1.2%琼脂糖电泳检测PCR扩增目的产物片段,结果见图1、2。扩增产物大小与设计引物预期扩增片段长度一致。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1MyoG基因PCR扩增
-->Fig. 1The amplification result of MyoG from PCR
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2MEF2a基因PCR扩增
-->Fig. 2The amplification result of MEF2a from PCR
-->
2.2 PCR扩增产物测序及序列分析
根据目的片段扩增结果,将所有PCR产物送往上海英潍捷基生物技术有限公司进行直接测序。测序结果经序列拼接、对比,并与GenBank上公布的基因参考序列矫正,发现MyoG基因共有6个SNPs(图3、4),分别位于外显子1的g.1131C>T位点,外显子2的g.2186G>A位点和g.2204G>A位点,外显子3的g.2920G>A位点、g.2962C>T位点和g.2977G>C位点,其中,g.2977G>C位点发生的G/C突变使密码子由GAG变为GAC,所编码的氨基酸由谷氨酸变成天冬氨酸。MEF2a基因共有2个SNPs(图5),分别位于外显子11的g.47915G>A位点和g.47918G>A位点,g.47915G>A位点发生的G/A突变使密码子由GAA变为AAA,所编码的氨基酸由谷氨酸变成赖氨酸,g.47918G>A位点的G/A突变引起的密码子由GAT变成AAT,编码的氨基酸由天冬氨酸变成天冬酰胺。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3MyoG基因1131、2920和2977 bp处的碱基变异
-->Fig. 3The sequencing map of the MyoG gene at positions 1131, 2920 and 2977
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4MyoG基因2186、2204 和2962 bp处的碱基变异
-->Fig.4The sequencing map of the MyoG gene at position 2186, 2204 and 2962
-->
2.3 MyoG基因和MEF2a基因遗传学分析
测序结果经序列比对发现,在MyoG基因外显子扩增片段中存在6个SNPs位点,将6个SNPs位点所对应纯合子和杂合子的基因型分别定义为CC、CT、TT;GG、GA;GG、GA;GG、GA、AA;CC、CT和GG、GC、CC型。对几种基因型分析,可知相对应的基因型中TT、GG、GG、GA、CC、GG为优势基因型,等位基因T、G、G、G、C、G则为优势等位基因。在MEF2a基因外显子11中有2个SNPs位点,这两个多态位点属于完全连锁平衡位点,将它们所对应纯合子和杂合子的基因型分别定义为GG、GA型。对基因型进行分析,可知相对应的基因型中GA为优势基因型,等位基因G则为优势等位基因。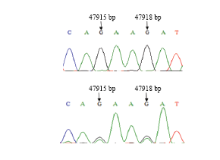 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5MEF2a基因47915和47918 bp处的碱基变异
-->Fig. 5The sequencing map of MEF2a gene at position 47915 and 47918
-->
统计各基因型的个体数,分别计算MyoG基因和MEF2a基因中的SNPs位点的基因型频率和基因频率,计算结果如表2所示。χ2适合性检验结果表明,在MyoG基因中除g.1131C>T外,该基因座其他多态位点均处于Hardy-Weinberg平衡状态(P>0.05)。对MEF2a基因中的2个SNPs进行χ2适合性检验,结果表明,该基因这两个多态位点均未处于Hardy- Weinberg平衡状态(P<0.05),这可能是由于在育种过程中的人为选择或遗传漂变造成的。
Table 2
表2
表2MyoG、MEF2a基因多态位点基因频率及基因型频率
Table 2Gene frequency and genotype frequency of SNPs in MyoG and MEF2a gene
| 基因 Gene | SNPs | 基因型频率 Genotype frequency | 等位基因频率 Gene frequency | χ2(P值) χ2 (P value) | |||
|---|---|---|---|---|---|---|---|
| MyoG | g.1131C>T | CC(68):0.3542 | CT(24):0.1250 | TT(100):0.5208 | C:0.4167 | T:0.5833 | 26.4882(0.0000) |
| g.2186G>A | GG(228):0.9500 | GA(12):0.0500 | AA(0):0.0000 | G:0.9750 | A:0.0250 | 0.0394(0.9805) | |
| g.2204G>A | GG(224):0.9333 | GA(16):0.0667 | AA(0):0.0000 | G:0.9667 | A:0.0333 | 0.0713(0.9650) | |
| g.2920G>A | GG(84):0.3500 | GA(100):0.4167 | AA(56):0.2333 | G:0.5583 | A:0.4417 | 1.4446(0.4856) | |
| g.2962C>T | CC(232):0.9667 | CT(8):0.0333 | TT(0):0.0000 | C:0.9833 | T:0.0167 | 0.0172(0.9914) | |
| g.2977G>C | GG(140):0.5833 | GC(72):0.3000 | CC(28):0.1167 | G:0.7333 | C:0.2667 | 3.2561(0.1963) | |
| MEF2a | g.47915G>A | GG(40):0.1667 | GA(200):0.8333 | AA(0):0.0000 | G:0.5833 | A:0.4163 | 41.2653(0.0000) |
| g.47918G>A | |||||||
新窗口打开
2.4 MyoG基因与MEF2a基因多态性与屠宰性状的相关性分析
MyoG基因多态性与三穗鸭屠宰性状的显著性关系分析如表3所示。可以看出,公鸭的胸肌率在MyoG基因g.1131C>T位点的不同基因型间差异显著,CC型显著高于CT和TT型,TT型显著高于CT型。MyoG基因g.2204G>A的不同基因型对体重和全净膛重有显著影响,GG型均显著高于GA型。其余的性状在各个SNPs的不同基因型间和性别间差异不显著。MEF2a基因的2个突变位点g.47915G>A/g.47918G>A的不同基因型对全净膛率有显著影响(P<0.05),且GA型显著高于GG型,其余的性状在两个SNPs的不同基因型、性别间无差异显著性,分析统计结果如表4所示。Table 3
表3
表3MyoG基因多态位点与屠宰性状的关联分析
Table 3The association results between the genotype of MyoG gene and slaughter traits
| 位点 SNPs | 基因型 Genotype | 性别 Gender | 性状 Traits | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 体重 Weight (g) | 屠体重 Slaughter weight (g) | 半净膛重 Semi-eviscerated weight (g) | 全净膛重 Eviscerated weight (g) | 腿肌重 Leg muscle weight (g) | 胸肌重 Breast muscle weight (g) | 屠宰率 Dressing percentage (%) | 半净膛率 Semi-eviscerated percentage (%) | 全净膛率 Eviscerated percentage (%) | 腿肌率 Leg muscle percentage (%) | 胸肌率 Breast muscle percentage (%) | 瘦肉率 Lean meat percentage (%) | |||
| g.1131C>T | CC(68) | ♀(24) | 1308±76 | 1218±85 | 1108±68 | 986±64 | 118±9 | 122±14 | 93.12±2.25 | 84.76±2.56 | 75.41±2.96 | 12.06±1.35 | 12.35±1.51 | 24.41±2.08 |
| ♂(44) | 1383±136 | 1302±122 | 1133±137 | 1011±119 | 112±20 | 115±20 | 94.20±2.88 | 81.94±5.94 | 73.14±5.20 | 11.16±2.12 | 11.37±1.27b | 22.54±2.92 | ||
| T(68) | 1356±121 | 1272±115 | 1124±116 | 1002±102 | 114±17 | 117±18 | 93.82±2.66 | 82.94±5.10 | 73.94±4.57 | 11.48±1.89 | 11.72±1.40b | 23.20±2.75 | ||
| CT(24) | ♀(20) | 1357±154 | 1268±118 | 1161±112 | 1034±98 | 119±10 | 113±8 | 93.64±3.81 | 85.71±3.57 | 76.40±3.57 | 11.62±1.40 | 10.97±0.89 | 22.60±2.23 | |
| ♂(4) | 1375±113 | 1324±104 | 1216±113 | 1077±93 | 123±19 | 85±21 | 96.29±2.65 | 88.43±1.43 | 78.32±4.03 | 11.42±1.59 | 7.89±1.32a | 19.31±2.41 | ||
| T(24) | 1360±138 | 1277±108 | 1170±102 | 1041±89 | 120±9 | 108±13 | 94.08±3.57 | 86.16±3.38 | 76.72±3.28 | 11.58±1.25 | 10.46±1.49a | 22.05±2.41 | ||
| TT(100) | ♀(56) | 1373±82 | 1263±88 | 1126±93 | 999±87 | 113±23 | 114±26 | 91.99±2.35 | 82.08±5.00 | 72.80±4.59 | 11.29±1.80 | 11.33±1.85 | 22.62±3.07 | |
| ♂(44) | 1377±110 | 1272±111 | 1149±97 | 1026±86 | 123±22 | 109±15 | 92.38±3.44 | 83.45±2.96 | 74.56±3.15 | 11.93±1.79 | 10.64±1.15c | 22.57±2.24 | ||
| T(100) | 1374±93 | 1267±97 | 1136±93 | 1011±86 | 117±23 | 112±22 | 92.16±2.82 | 82.68±4.20 | 73.58±4.04 | 11.57±1.78 | 11.02±1.59c | 22.59±2.68 | ||
| g.2186G>A | GA(12) | ♀(8) | 1295±106 | 1212±115 | 181±83 | 957±91 | 122±9 | 109±23 | 93.50±1.24 | 83.45±0.44 | 73.86±0.94 | 12.70±0.24 | 11.50±3.52 | 24.21±3.76 |
| ♂(4) | 1575±192 | 1471±108 | 1320±142 | 1179±121 | 129±17 | 146±19 | 93.39±3.07 | 83.81±3.69 | 74.85±4.71 | 10.94±1.91 | 12.38±1.03 | 23.32±1.99 | ||
| T(12) | 1388±178 | 1298±171 | 1160±150 | 1031±143 | 124±8 | 121±27 | 93.46±0.80 | 83.57±0.37 | 74.19±0.87 | 12.11±1.03 | 11.79±2.54 | 23.91±2.71 | ||
| GG(228) | ♀(112) | 1363±097 | 1259±89 | 1134±91 | 1008±82 | 115±18 | 114±21 | 92.44±2.55 | 83.28±4.30 | 74.05±4.08 | 11.43±1.59 | 11.25±1.67 | 22.68±2.78 | |
| ♂(116) | 1379±148 | 1293±148 | 1147±144 | 1024±127 | 119±21 | 109±18 | 93.77±3.40 | 83.18±4.57 | 74.25±4.15 | 11.61±1.75 | 10.63±1.28 | 22.25±2.33 | ||
| T(228) | 1371±124 | 1276±123 | 1141±120 | 1016±106 | 117±19 | 111±19 | 93.11±3.06 | 83.23±4.40 | 74.15±4.08 | 11.52±1.66 | 10.93±1.50 | 22.46±2.55 | ||
| g.2204G>A | GA(16) | ♀(8) | 1285±247 | 1200±195 | 1087±161 | 973±145 | 114±1 | 120±24 | 93.66±2.85 | 84.95±3.81 | 76.00±3.35 | 11.86±1.91 | 12.29±0.64 | 24.15±1.27 |
| ♂(8) | 1200±120 | 1131±129 | 976±98 | 861±89 | 104±11 | 91±17 | 94.18±1.29 | 81.28±0.04 | 71.73±0.23 | 12.02±0.01 | 10.52±0.88 | 22.54±0.87 | ||
| T(16) | 1243±166A | 1166±141 | 1031±127 | 917±117a | 109±9 | 106±24 | 93.92±1.83 | 83.12±3.05 | 73.87±3.13 | 11.94±1.10 | 11.40±1.19 | 23.35±1.28 | ||
| GG(224) | ♀(112) | 1364±86 | 1260±83 | 1134±88 | 1007±79 | 116±18 | 113±21 | 92.43±2.50 | 83.17±4.21 | 73.89±4.00 | 11.49±1.58 | 11.19±1.78 | 22.69±2.86 | |
| ♂(112) | 1399±144 | 1311±145 | 1166±140 | 1041±122 | 120±21 | 111±18 | 93.72±3.45 | 83.34±4.63 | 74.45±4.16 | 11.56±1.78 | 10.70±1.33 | 22.26±2.38 | ||
| T(224) | 1381±119B | 1285±120 | 1150±117 | 1024±104b | 118±19 | 112±19 | 93.08±3.06 | 83.26±4.38 | 74.17±4.05 | 11.53±1.67 | 10.95±1.58 | 22.48±2.62 | ||
| g.2920G>A | AA(56) | ♀(16) | 1303±67 | 1229±81 | 1113±63 | 982±61 | 122±5 | 115±20 | 94.30±1.80 | 85.45±2.36 | 75.33±1.81 | 12.46±0.32 | 11.75±2.30 | 24.22±2.37 |
| ♂(40) | 1351±113 | 1261±120 | 1142±115 | 1014±105 | 117±20 | 104±19 | 93.37±4.30 | 84.41±3.12 | 74.94±3.05 | 11.49±1.34 | 10.22±1.29 | 21.71±2.22 | ||
| T(56) | 1337±102 | 1252±108 | 1133±101 | 1004±94 | 118±17 | 107±19 | 93.63±3.71 | 84.71±2.87 | 75.05±2.69 | 11.77±1.21 | 10.65±1.70 | 22.43±2.46 | ||
| GA(100) | ♀(56) | 1365±105 | 1248±86 | 1117±100 | 993±87 | 116±16 | 108±19 | 91.51±2.29 | 81.84±4.67 | 72.83±4.42 | 11.69±1.50 | 10.86±1.78 | 22.55±2.86 | |
| ♂(44) | 1411±175 | 1336±188 | 1168±188 | 1045±164 | 119±21 | 110±17 | 94.59±2.54 | 82.65±5.99 | 73.95±5.41 | 11.50±2.07 | 10.55±1.14 | 22.06±2.39 | ||
| T(100) | 1385±139 | 1287±144 | 1139±144 | 1016±127 | 117±18 | 109±18 | 92.86±2.82 | 82.20±5.19 | 73.32±4.80 | 11.61±1.74 | 10.72±1.51 | 22.34±2.62 | ||
| GG(84) | ♀(48) | 1369±96 | 1274±99 | 1153±88 | 1026±82 | 113±22 | 120±22 | 93.08±2.54 | 84.26±3.57 | 75.01±3.68 | 10.99±1.78 | 11.58±1.56 | 22.58±2.92 | |
| ♂(36) | 1392±161 | 1294±128 | 1148±129 | 1027±111 | 121±22 | 117±21 | 93.16±3.18 | 82.53±3.83 | 73.91±3.51 | 11.81±1.82 | 11.38±1.33 | 23.19±2.27 | ||
| T(84) | 1379±125 | 1283±110 | 1151±105 | 1027±93 | 117±22 | 119±21 | 93.12±2.75 | 83.52±3.69 | 74.54±3.57 | 11.34±1.80 | 11.49±1.43 | 22.84±2.62 | ||
| g.2962C>T | CC(232) | ♀(112) | 1358±96 | 1256±90 | 1129±92 | 1002±83 | 115±18 | 112±20 | 92.52±2.55 | 83.13±4.16 | 73.81±3.88 | 11.47±1.62 | 11.21±1.74 | 22.69±2.84 |
| ♂(120) | 1385±149 | 1299±149 | 1153±145 | 1029±128 | 119±20 | 110±19 | 93.75±3.34 | 83.20±4.50 | 74.27±4.08 | 11.59±1.72 | 10.69±1.30 | 22.28±2.30 | ||
| T(232) | 1372±126 | 1278±125 | 1141±122 | 1016±109 | 117±19 | 111±19 | 93.16±3.03 | 83.17±4.30 | 74.05±3.95 | 11.53±1.66 | 10.94±1.54 | 22.48±2.56 | ||
| CT(8) | ♀(0) | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | |
| ♂(8) | 1363±159 | 1258±122 | 1162±74 | 1047±55 | 127±6 | 127±30 | 92.43±1.86 | 85.55±4.59 | 77.13±4.95 | 12.13±0.09 | 12.02±2.27 | 24.15±2.17 | ||
| T(8) | 1363±159 | 1258±122 | 1162±74 | 1047±55 | 127±6 | 127±30 | 92.43±1.86 | 85.55±4.53 | 77.13±4.95 | 12.13±0.09 | 12.02±2.27 | 24.15±2.17 | ||
| g.2977G>C | CC(28) | ♀(16) | 1293±81 | 1182±75 | 1095±70 | 0977±81 | 116±22 | 111±31 | 91.41±1.68 | 84.76±2.78 | 75.57±4.07 | 11.79±1.52 | 11.20±2.34 | 23.00±3.60 |
| ♂(12) | 1370±213 | 1262±182 | 1121±130 | 1004±109 | 102±26 | 117±17 | 92.21±0.96 | 82.15±3.97 | 73.62±4.34 | 10.14±1.87 | 11.63±0.75 | 21.77±1.52 | ||
| T(28) | 1326±142 | 1216±126 | 1106±91 | 988±86 | 110±23 | 113±24 | 91.75±1.38 | 83.64±3.33 | 74.74±3.96 | 11.09±1.76 | 11.38±1.72 | 22.47±2.77 | ||
| GC(72) | ♀(48) | 1401±67 | 1306±68 | 1157±101 | 1028±92 | 110±22 | 117±20 | 93.22±2.81 | 82.56±5.89 | 73.36±5.54 | 10.69±1.62 | 11.33±1.26 | 22.02±2.58 | |
| ♂(24) | 1399±154 | 1319±142 | 1168±165 | 1042±142 | 121±28 | 119±19 | 94.34±3.18 | 83.26±4.16 | 74.35±3.71 | 11.75±2.76 | 11.45±1.27 | 23.20±3.23 | ||
| T(72) | 1400±99 | 1310±95 | 1161±121 | 1033±107 | 114±24 | 118±19 | 93.59±2.90 | 82.79±5.26 | 73.69±4.91 | 11.04±2.05 | 11.37±1.23 | 22.42±2.78 | ||
| GG(140) | ♀(56) | 1341±111 | 1235±90 | 1119±86 | 993±73 | 120±11 | 111±19 | 92.22±2.35 | 83.50±2.46 | 74.17±1.94 | 12.14±1.29 | 11.23±2.04 | 23.38±2.80 | |
| ♂(84) | 1384±148 | 1298±153 | 1154±147 | 1029±132 | 121±17 | 106±19 | 93.81±3.62 | 83.34±4.82 | 74.34±4.33 | 11.75±1.31 | 10.33±1.24 | 22.09±2.13 | ||
| T(140) | 1366±134 | 1273±134 | 1140±126 | 1015±112 | 120±15 | 108±19 | 93.17±3.23 | 83.40±3.99 | 74.27±3.53 | 11.91±1.29 | 10.69±1.64 | 22.61±2.46 | ||
新窗口打开
Table 4
表4
表4MEF2a基因多态位点与屠宰性状的关联分析
Table 4The association results between the genotype of MEF2a gene and slaughter traits
| 基因型 Genotype | 性别 Genotype | 性状 Traits | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 体重 Weight (g) | 屠体重 Slaughter weight (g) | 半净膛重 Semi-eviscerated weight (g) | 全净膛重 Eviscerated weight (g) | 腿肌重 Leg muscle weight (g) | 胸肌重 Breast muscle weight (g) | 屠宰率 Dressing percentage (%) | 半净膛率 Semi-eviscerated percentage (%) | 全净膛率 Eviscerated percentage (%) | 腿肌率 Leg muscle percentage (%) | 胸肌率 Breast muscle percentage (%) | 瘦肉率 Lean meat percentage (%) | ||
| GG | ♀(20) | 1374±80 | 1260±87 | 1090±97 | 967±87 | 107±29 | 102±20 | 91.67±2.13 | 79.41±6.22 | 70.45±6.11 | 11.12±2.77 | 10.54±1.81 | 21.66±3.46 |
| ♂(20) | 1364±181 | 1266±169 | 1128±171 | 996±155 | 110±11 | 102±23 | 92.78±0.74 | 82.55±1.88 | 72.87±1.99 | 11.12±0.98 | 10.21±1.28 | 21.33±1.79 | |
| T(40) | 1369±132 | 1263±127 | 1109±133 | 982±119 | 109±20 | 102±20 | 92.22±1.61 | 80.98±4.64 | 71.66±4.47b | 11.12±1.96 | 10.37±1.49 | 21.50±2.60 | |
| GA | ♀(100) | 1355±101 | 1255±92 | 1139±89 | 1013±80 | 117±15 | 116±20 | 92.68±2.56 | 84.07±3.25 | 74.75±3.06 | 11.59±1.29 | 11.41±1.73 | 23.01±2.67 |
| ♂(100) | 1390±146 | 1305±147 | 1158±143 | 1036±124 | 121±22 | 112±18 | 93.95±3.63 | 83.33±4.87 | 74.55±4.35 | 11.69±1.84 | 10.78±1.30 | 22.47±2.38 | |
| T(200) | 1372±126 | 1280±124 | 1149±118 | 1024±104 | 119±18 | 114±19 | 93.31±3.17 | 83.70±4.12 | 74.65±3.72a | 11.64±1.57 | 11.10±1.55 | 22.74±2.52 | |
新窗口打开
2.5 聚合基因型与屠宰性状间相关性分析
根据单个SNP位点分析结果,只选取与屠宰性状有显著影响的3个SNPs位点,采用PHASE 2.0软件,进行两基因聚合基因型的判定,选取的性状也是在单基因关联分析时存在显著差异的指标。由于聚合后试验群体个体数较少缘故,故不考虑性别因素。聚合后共得到8种聚合基因型(表5)。从体重指标看,试验群体中CTGGGG型体重除与TTGAGA型个体间差异不显著,与其他基因型个体间均呈现显著差异,且CTGGGA、TTGGGG与CTGGGG基因型间呈差异极显著(P<0.01),TTGAGA与TTGGGG、CTGGGA间存在差异显著(P<0.05);从全净膛重指标看,几种聚合基因型中,除了CTGGGG基因型和TTGAGA基因型间差异不显著,与其他几种基因型间均差异显著;CCGAGA基因型的体重和全净膛重平均值都最高,但个体数量少,有必要扩大样本做进一步验证,其次为CTGGGA基因型,两个指标一致,这是由于两个指标呈正相关引起的。此外,基因型CCGGGA的胸肌率与TTGAGA基因型间表现出显著差异(P<0.05),与CTGGGG、CTGGGA、TTGGGG、TTGGGA间均呈差异极显著关系(P<0.01),CCGGGG聚合基因型的胸肌率平均值最高,其次是CCGGGA基因型;在全净膛率这个指标,TTGAGA基因型的平均值最高,其次为CCGGGA基因型,但各基因型间差异都没达到显著水平。Table 5
表5
表5聚合基因型与屠宰性状关联分析
Table 5Analysis of aggregated genotype and slaughter traits
| 性状 Traits | SNPs/基因型 Genotype | |||||||
|---|---|---|---|---|---|---|---|---|
| g.1131C>T、g.2204G>A、g.47915G>A/g.47918G>A | ||||||||
| CCGGGG (4) | CCGGGA (44) | CCGAGA (4) | CTGGGG (8) | CTGGGA (24) | TTGGGG (28) | TTGGGA (116) | TTGAGA (12) | |
| 体重 Weight (g) | 1280±23 BCbc | 1379±35ABab | 1460±31 Aa | 1168±82Cd | 1442±48Aa | 1439±44Aa | 1369±22ABab | 1225±67Ccd |
| 全净膛重 Eviscerated weight (g) | 960±28 ABbc | 1034±31ABab | 1075±33 Aa | 831±73Cd | 1054±42ABab | 1033±39ABab | 1022±19Aa | 929±60BCc |
| 胸肌率 Breast muscle percentage (%) | 13.65±0.48 Aa | 13.05±0.32Aa | 12.74±0.36 Aa | 9.18±0.76Dd | 10.13±0.44CDc | 10.59±0.41BCbc | 10.44±0.20BCc | 11.49±0.62Bb |
| 全净膛率 Eviscerated rate (%) | 75.00±1.02 Aab | 75.03±1.22Aab | 73.63±0.89Aab | 71.15±2.85A | 72.97±1.65Aab | 71.78±1.52Ab | 74.64±0.75Aab | 75.95±2.33Aa |
新窗口打开
3 讨论
3.1 MyoG基因多态性对肌肉的影响
在不影响肉质风味的前提下,动物能快速生长、肌肉沉积率高一直以来是畜禽育种的目标之一,研究证实,MyoG基因能在所有骨骼肌中表达,其表达程度直接影响畜禽的产肉性能[13]。WEINTRAUB等[14-15]研究表明,敲除MyoG基因的小鼠出生后会发生骨骼肌的发育严重缺陷。缺失MyoG基因的小鼠要比正常小鼠个体小。唐莹等[16]利用PCR-SSCP技术检测京海黄鸡MyoG基因外显子多态性,结果表明,MyoG基因多态与京海黄鸡生长性状相关。ZHANG等[17]再一次证实了MyoG基因多态性对京海黄鸡的生长性状有影响。XUE等[19]报道,肌细胞生成素基因单核苷酸多态性对猪的初生重和背标厚度有显著影响。王健等[20]对太湖鹅MyoG基因研究报道,在外显子1中第108位点处发现碱基C/T突变,推断该基因多态性对太湖鹅早期增重有显著影响。本研究中,共发现三穗鸭MyoG基因6个SNPs,其中g.2977G>C位点发生的G/C突变使密码子由GAG变为GAC;所定义的几种基因型中,MyoG基因g.1131C>T的不同基因型胸肌率有显著影响,CC型显著高于CT和TT型,TT型显著高于CT型,公鸭的胸肌率差异显著。MyoG基因g.2204G>A的不同基因型对体重和全净膛重有显著影响,GG型均显著高于GA型。其余的性状在各个SNPs的不同基因型间和性别间差异不显著。3.2 MEF2a基因多态性
研究表明,多种生理过程与MEF2基因家族相关,如骨骼发育、肌肉形成、肝脏纤维化和神经系统发育等[2]。MEF2a基因属于MEF2基因家族一员,它在其中也扮演着至关重要的角色,NAYA等[21]报道,小鼠敲除MEF2a基因,大多数在第一周就会表现出明显心室扩张,肌纤维碎片等现象而死亡,能存活的个体到了成年因缺乏心脏线粒体也容易发生猝死。LIEB等[22]认为,缺少MEF2基因对冠心病(CAD)不存在影响,然而在2012年,LIU等[23]对不同人群的冠心病(CAD)病例进行分析,结果虽未能证实冠心病的产生与MEF2a基因的多态性有直接关系,但在1 008个研究病例中,有5个病例在MEF2a基因外显子11区域出现了21个碱基缺失,因此推测MEF2a基因的变异可能是造成CAD原因之一。2009年,周艳等[24]报道,MEF2a基因的3个SNPs位点与鸡的部分屠宰性状有显著相关,可推断MEF2a基因可作为影响鸡屠宰性状的候选基因。本研究显示,在三穗鸭MEF2a基因外显子11中发现的2个SNPs位点,分别定义为GG、GA两种基因型,分析表明,GA为优势基因型,等位基因G则为优势等位基因,与屠宰性状进行关联分析表明,MEF2a基因g.47915G>A/g.47918G>A的不同基因型对全净膛率有显著影响,GA型显著高于GG型,其余的性状在两个SNPs的不同基因型、性别间差异不显著。3.3 MyoG、MEF2a基因与三穗鸭屠宰性状的关联
研究表明,MyoG与MEF2a基因具有协同作用。常国斌等[25]研究报道,对鸡肌内脂肪影响最明显的优势单个基因,经两基因或三基因聚合后,并未表现出相应的优势情况;而在扩大样本数进一步研究发现,最佳聚合基因型与相应的单个基因的有利基因型结果一致,且整体效应要高于单个基因型,表现出一定程度的累加效应[26]。其他研究也表明,单个SNP所对应基因型与性状间关联分析结果在不同的品种间通常是不同的,没有多个位点联合分析准确[27-29]。本研究以三穗鸭为研究对象,在单基因关联分析时,3个位点的平均值最高的基因型分别是CC、GG和GA型,但是在聚合效应分析时,并不是CCGGGA基因型的平均值最高,这说明两个基因间存在互作效应,并没有表现出累加效应,这与常国斌等[25-26]的研究结果不一致。在基因间存在交互作用时,如果利用单基因进行分子标记辅助选择往往会顾此失彼,不能收到良好的育种效果,因此本研究的结果表明对影响同一性状的不同基因进行聚合效应分析是十分必要的。本研究结果所显示的平均值最高的聚合基因型个体数量都偏少,只有4个,因此有必要进一步扩大样本进行验证,其次本研究只进行了两个基因的聚合效应分析,证明两个基因间存在互作,然而影响屠宰性状的基因较多,所以有必要进行更多基因的聚合效应分析,同时在其它鸭品种中是否存在与本研究一致的聚合效应也需要做进一步验证,以期得到更真实有效的分子标记,从而为鸭屠宰性状的多基因聚合育种提供依据。再者,通过对不同模式生物的深入研究,对于人类医学进行肌肉相关疾病的研究、治疗也能提供一定的帮助或具有参考意义,与此同时,在进行人类肌肉相关疾病的研究时,也应该考虑从多基因聚合的角度去研究,也许能达到事半功倍的效果。4 结论
本试验首次将鸭的MyoG基因和MEF2a基因进行聚合效应分析,结果单个基因的有利基因型聚合后并不是最佳基因型,说明基因间存在互作效应,但是最佳基因型在本群体中个体数偏少,有必要扩大样本做进一步研究和进行更多个基因聚合效应分析,才能为家禽的多基因聚合育种提供有效的分子标记。(责任编辑 林鉴非)
The authors have declared that no competing interests exist.
