

1. 军事科学院军事医学研究院生命组学研究所 国家蛋白质科学中心 (北京),北京 102206;
2. 北京蛋白质组研究中心,北京 102206;
3. 蛋白质组学国家重点实验室,北京 102206
收稿日期:2020-08-14;接收日期:2020-10-28
基金项目:国家重点研发计划(No. 2016YFB0201702) 资助
摘要:癌症已经被广泛认为是高度异质性的疾病,癌症的早期诊断、分型和预后已成为癌症研究的关注重点。在大数据时代,对海量癌症生物医学数据进行高效的数据挖掘是生物信息学面临的重要挑战。自编码器(Autoencoder) 作为神经网络的一种典型模型,能够通过无监督的方式高效地学习输入数据的特征,进而对生物数据进行整合与挖掘。文中首先介绍了自编码器模型结构并阐述其工作流程,之后结合多种类型的生物医学数据总结自编码器在癌症信息学研究领域的进展,并展望其发展趋势及应用方向。
关键词:自编码器癌症神经网络特征提取
Application of neural network autoencoder algorithm in the cancer informatics research
Xiao Li1,2,3, Jie Ma1,2,3, Fuchu He1,2,3, Yunping Zhu1,2,3


1. National Center for Protein Sciences (Beijing), Beijing Institute of Life Omics, Academy of Military Medical Sciences, Academy of Military Sciences, Beijing 102206, China;
2. Proteome Research Center, Beijing 102206, China;
3. State Key Laboratory of Proteomics, Beijing 102206, China
Received: August 14, 2020; Accepted: October 28, 2020
Supported by: National Key Research and Development Program of China (No. 2016YFB0201702)
Corresponding author: Yunping Zhu. Tel: +86-10-61777058; E-mail: zhuyunping@gmail.com.
Abstract: Cancers have been widely recognized as highly heterogeneous diseases, and early diagnosis and prognosis of cancer types have become the focus of cancer research. In the era of big data, efficient mining of massive biomedical data has become a grand challenge for bioinformatics research. As a typical neural network model, the autoencoder is able to efficiently learn the features of input data by unsupervised training method and further help integrate and mine the biological data. In this article, the primary structure and workflow of the autoencoder model are introduced, followed by summarizing the advances of the autoencoder model in cancer informatics using various types of biomedical data. Finally, the challenges and perspectives of the autoencoder model are discussed.
Keywords: autoencodercancerneural networkfeature extraction
全球癌症的发生率和死亡率正在迅速增长,癌症预计将成为21世纪世界各国人口死亡的主要原因,也是人类延长预期寿命的最大障碍[1]。当前,癌症已经被广泛认为是一类由不同亚型组成的异质性疾病,癌症的早期诊断、分型和预后已成为癌症研究的关注重点[2]。与此同时,高通量技术的发展使得研究者们迎来了“大数据时代”,为癌症研究积累了大量的生物医学数据,包括组学、图像和信号数据[3],这使得从海量数据中挖掘有价值的信息成为一项重要的挑战。
与人类相比,计算机在处理复杂数据时具有得天独厚的优势:基于机器学习方法,计算机可以基于肉眼难以分辨的数据将癌症患者分为不同亚群,这对于人类健康以及癌症预测有着重要意义。研究人员已经开发了许多成熟的算法并将其应用于癌症筛查诊断,如朴素贝叶斯模型(Naive bayesian model,NBM)、支撑向量机(Support vector machine,SVM)、决策树(Decision tree,DT)、人工神经网络(Artificial neural network,ANN) 以及它们的各种衍生算法等。然而,生物数据普遍维度极高而样本数较少[4],在这种高维数据情形下容易出现数据稀疏的情况——这正是所有机器学习方法共同面临的难题,称之为“维度灾难”。为解决高维数据这个问题,降维思想应运而生,目前发展的降维方法包括:主成分分析(Principal components analysis,PCA)、奇异值分解(Singular value decomposition,SVD)、t-分布邻域嵌入算法(t-distribution stochastic neighbour embedding,t-SNE) 以及基于神经网络的自编码器算法(Autoencoder,AE)[5]等。
其中,PCA和SVD都是线性降维方法,无法挖掘出复杂生物数据中可能存在的非线性关系,而且在掌握先验知识时并不能通过参数化等方法对处理过程进行干预。t-SNE虽然是非线性的方法,但由于t分布的尾部很重,它只适用于降到二维或三维,这使得它更多被用于可视化领域。
得益于计算硬件技术的进步,人工神经网络蓬勃发展,各种新的神经网络模型不断被提出,图像识别、自然语言处理等领域的记录不断被刷新。自编码器作为神经网络的一种典型模型,可以将样本数据同时作为神经网络的输入和输出,通过无监督的方式高效地学习输入数据的特征,进而对数据进行整合与挖掘。早在1988年,自编码器的雏形就已经被提出[6],但由于优化难度高而未能得到广泛应用。直到Hinton等[5]首先通过逐层预训练以及微调方法,使得自编码器在高维特征提取任务中取得显著效果,获得了比PCA等模型更有意义的数据投影,它才开始得到广泛的应用。文中首先回顾了自编码器模型并阐述其工作流程,之后结合各组学数据、生物医学图像数据和其他形式的数据,总结自编码器在癌症信息学研究领域的进展,并讨论其主要的应用方向。
1 自编码器模型及应用流程自编码器(AE)包含两个模块:编码器和解码器。如图 1所示,编码器(f) 将原始输入X映射到维度更低的特征空间Z,然后解码器(g) 将Z映射回原始输入空间X。在学习过程中不需要使用样本标签,而是把样本的输入同时作为神经网络的输入和输出,通过最小化重构误差,使得模型从数据中捕获所有显著特征[7]。这种无监督的学习方式极大地提高了模型的通用性。事实上,如果自编码器仅使用线性激活函数,并且损失函数是均方差损失函数,那么它就等价于PCA算法。而作为神经网络,它更可以进行非线性的降维,还能在训练时根据先验知识给样本加权,从而为特征的表达提供更多信息。
 |
| 图 1 自编码器基本结构 Fig. 1 The structure of autoencoder. |
| 图选项 |
自编码器的应用流程如图 2所示,可以概括为以下几个步骤:(1) 数据集的构建,既可以选择序列数据,也可以选择图像数据,还可以整合不同形式的数据;(2) 数据预处理,主要是脏数据清洗和缺失值填补;(3) 根据构造的数据集及对训练结果的要求,选择相应的自编码器模型;(4) 训练模型,通过调节网络参数来达到预期的性能;(5) 提取特征空间Z,进行后续的研究,如分类、聚类、生存分析、通路识别、基因富集分析等。研究人员针对自编码器不同的应用方向、数据特点及优化目标,发展了不同的自编码器模型。
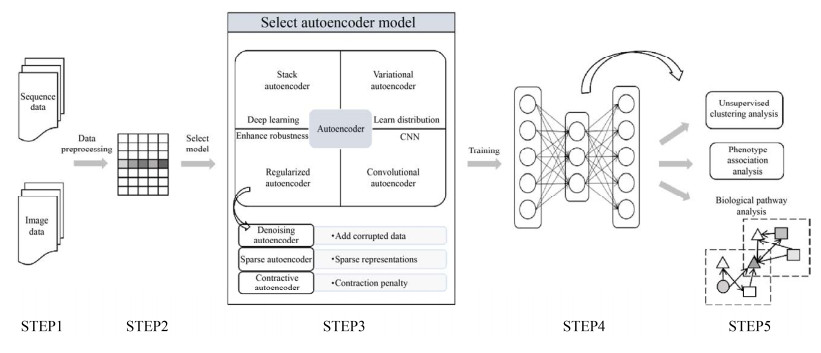 |
| 图 2 自编码器的基本应用流程 Fig. 2 Overview of the workflow of autoencoder. |
| 图选项 |
1.1 栈式自编码器为了避免自编码器层数加深而带来的“梯度消失”、“梯度爆炸”等问题,2007年Bengio等[8]根据“逐层初始化” (Layer-wise pre-training) 的思想,提出了栈式自编码器(Stacked autoencoder,SAE)。其方法是通过逐层非监督的预训练来初始化深度网络的参数,预训练完毕后,最后再对整个网络进行微调。
栈式自编码器具有强大的表达能力及深度神经网络的所有优点。在科研实践中,它最多的应用是与其他自编码器结合,构成栈式降噪自编码器或栈式稀疏自编码器等,在加深网络结构的同时又能获得很好的特征表示。
1.2 正则自编码器衡量一个自编码器性能的很重要的一个标准是模型对输入数据在一定程度下的扰动是否具有鲁棒性,这导致了正则自编码器(Regularized autoencoder) 的出现,并产生了两种不同但都有效的正则化方法[9]:一种办法是在输入中引入随机噪声,基于这种思想,Vincent等[10]提出了降噪自编码器(Denoising autoencoder,DAE);另一种办法是在损失函数中加入不同的约束条件抑制噪声数据带来的扰动,基于这种思想,Ranzato等[11]和Rifai等[12]分别提出了稀疏自编码器(Sparse autoencoder,SAE) 和收缩自编码器(Contractive autoencoder,CAE)。
一个优秀的自编码器模型,应该能从部分“损坏”的原始数据中提取特征,并对数据进行重构。降噪自编码器在进行训练之前,对输入数据X按照某种规则进行加噪“损坏”,使模型必须尝试捕获输入中最显著的特征,以便最佳地消除“损坏”过程的影响。Vincent等[10]提出的损坏方式包括添加服从特定分布的随机噪声或掩码噪声(随机将输入X中特定比例的数据置为0),实验证明,在大多数情况下,掩码噪声已经足够使用。
稀疏自编码器是正则自编码器的一种,它不限制隐藏层的节点数,但对网络进行稀疏性限制,也就是使神经元大部分的时间都是被抑制的(假设激活函数是Sigmoid函数,则神经元输出接近0时认为其被抑制;而若激活函数是Tanh函数,则神经元的输出接近–1时认为它是被抑制的)。之所以要对网络进行稀疏化,是因为隐藏层神经元的维度较高时(可能大于输入层),将无法得到输入的压缩表示。通过加入稀疏性限制,自编码器即使在隐藏层维度较高的情况下仍然可以捕获输入数据中有意义的结构。具体来说,特征的稀疏性可以通过惩罚隐藏神经元的偏置值(使这些偏置参数更负)[11, 13]来实现,也可以通过直接惩罚神经元激活函数的输出(使它们更接近于0) 实现[14]。因此,稀疏自编码器的损失函数增加了对隐藏层的惩罚项,最为广泛应用的就是相对熵(Kullback-Leibler divergence,又称KL散度),其定义为:
为了提高对数据轻微扰动的鲁棒性,收缩自编码器在基础自编码器上添加了正则项,该项是编码器输出的特征向量的平方和,具体形式为编码器f关于输入X的Jacobian矩阵的Frobenius范数,目的是迫使模型学习具有更强收缩作用的映射。对输入扰动的鲁棒性是自编码器降噪的动机之一,降噪自编码器能抵抗小且有限的输入扰动,它针对的是输入,而收缩自编码器使特征提取函数能抵抗极小的输入扰动,针对的是输出。
1.3 变分自编码器变分自编码器(Variational autoencoder,VAE)是“生成式模型”的一种。VAE的核心思想是,假设有一批数据样本X,如果想得到X的分布p(X),但是数据量不够时,模型的估计是不准确的,这时可以将分布形式改一下,引入隐变量Z,可以得到:
| $p(x) = \mathop \sum \limits_Z p\left( {X|Z} \right)P(Z)$ |
VAE通过学习数据潜在的均值和标准差来实现这些特性,模型在重构的损失函数中增加了一个Kullback-Leibler (KL) 散度项,它通过约束隐变量Z以匹配高斯分布来调整权重参数。在VAE中,均值和标准差可以通过允许反向梯度传播的重参数化技巧同时学习[15]。
1.4 卷积自编码器传统的自编码器都是全连接的,这使得每个特征都是全局性的,而这在图像识别中并不适用。研究人员发现,卷积神经网络(Convolutional neural networks,CNN) 之所以在处理图像上有优势,是因为它可以提取隐藏在图像中的局部空间信息,因此很自然地想到如果使用CNN构造编码器和解码器网络,可能会比其他自编码器工作得更好,于是产生了卷积自编码器(Convolutional autoencoder,CAE)[16]。卷积自编码器与传统自编码器的区别在于它的编码器和解码器都是卷积神经网络,即编码器使用的是卷积操作和池化操作,解码器使用反卷积操作和反池化操作。
CNN和CAE之间最主要的区别在于前者将提取的特征进行组合用来分类,因此CNN是一种监督学习方法;而后者通常被用来从输入数据中提取特征重构输入数据。因此CAE在语义分割、图像隐写分析、图像恢复方面都有很好的表现。
神经网络模型的实现及应用对算法和编程能力要求较高,幸运的是,目前许多研究团队已经开发了大量开源的深度学习库可供研究人员使用。如表 1所示,根据研究对象及目的的不同,可以选用不同的深度学习库,如:能够快速处理图像任务的Caffe库,具有高效分布式计算性能的CNTK库,封装程度高、使用简单的Keras库,灵活性和可扩展性好的MXNet库,简洁、支持动态编程的Pytorch库,以及对运行平台和开发语言支持最广泛的TensorFlow库等。
表 1 深度学习框架概况Table 1 Overview of the present deep learning libraries
| Name | Release | Interface language | Organization | Strengths | Source |
| Caffe | 2013 | C++/Python/Matlab | BVLC | Fast, good at image task | https://github.com/BVLC/caffe |
| CNTK | 2014 | C++ | Microsoft | Fast, efficient distributed performance | https://github.com/microsoft/CNTK |
| Keras | 2015 | Python/R | Easy, friendly, highly modular | https://github.com/keras-team/keras | |
| MXNet | 2015 | C++/Python/R/... | DMLC | Flexible, expandable | https://github.com/apache/incubator-mxnet |
| PyTorch | 2017 | Python | Flexible, dynamic programming, friendly | https://github.com/pytorch/pytorch | |
| TensorFlow | 2015 | C++/Python/Go/… | Most extensive support for platforms and languages | https://github.com/tensorflow/tensorflow | |
| BVLC: the berkeley vision and learning center; DMLC: distributed machine learning community. | |||||
表选项
2 自编码器在癌症信息学中的应用传统的机器学习算法对输入特征的依赖性很高,特征提取的优劣会直接影响机器学习算法的预测或分类性能。因此,面对复杂多样的生物数据,选用合适的特征提取算法来帮助研究者得到最优的输入特征至关重要。如表 2所示,这一部分将结合前人的研究介绍各种自编码器如何应用于组学数据、生物医学图像、临床数据等数据特征的提取或整合。
表 2 各种自编码模型应用总结Table 2 Application of various autoencoders
| Model | Input data | Object | References |
| Stack autoencoder | Multi-omics | Clear cell renal cell carcinoma | [28] |
| Pan-cancer | [29-30] | ||
| Single-omic | Breast cancer | [22] | |
| Pan-cancer | [17] | ||
| Images | Breast cancer | [31] | |
| Lung cancer | [32] | ||
| EEG | Epileptic | [33-34] | |
| Denoising autoencoder | Multi-omics | Clear cell renal cell carcinoma | [28] |
| Breast cancer | [35-36] | ||
| Single-omic | Pan-cancer | [17] | |
| Breast cancer | [21-22] | ||
| Images | Lung cancer | [32] | |
| EHRs | – | [37] | |
| Sparse autoencoder | Single-omic | Pan-cancer | [26, 38] |
| Images | Breast cancer | [31] | |
| Basal cell carcinoma | [39] | ||
| Lung cancer | [40] | ||
| Variational autoencoder | Single-omic | Pan-cancer | [18, 20, 26, 41] |
| Multi-omics | Pan-cancer | [27] | |
| Images | – | [42] | |
| Convolutional autoencoder | Single-omic | Pan-cancer | [43] |
| Images | Lung cancer | [40, 44-45] | |
| ECG | Heart | [46] | |
| Contractive autoencoder | ECG | Heart | [47] |
表选项
2.1 自编码器在生命组学数据研究中的应用基因组、转录组和蛋白质组等组学数据都可用来解决生物学中的问题,组学中最常见的输入数据是原始的生物大分子序列(如DNA、RNA、氨基酸序列等)。包括美国国家癌症研究所、美国国家人类基因组研究所和国际癌症基因组联盟在内的大型联盟组织已经对数千个生物样本进行了测序分析,获得了每个样本多种不同的分子特征,包括mRNA、DNA甲基化、拷贝数变异(Copy number variation,CNV)、体细胞突变和蛋白质序列等。这些数据的使用促进了癌症亚型识别、生存预后预测、癌症通路分析、泛癌诊断预测等领域的发展,其中自编码器模型的使用发挥了重要作用。
2.1.1 单组学数据在泛癌分析中,Fakoor等[17]在特征学习步骤中利用稀疏自编码器分析了来自不同类型癌症的基因表达数据,为在非常有限的数据集下进行有效的特征学习提供了参考。Way等[18]使用变分自编码器分析癌症基因组图谱(The cancer genome atlas,TCGA) 转录组数据的研究证明,自编码器在保持生物学洞察力的同时具有学习潜在特征空间的巨大潜力。Chen等[19]基于TCGA基因表达数据,提出了一个基因超集自编码器,通过将一个多层自编码器模型与先验定义的基因集结合,在中间层保留了关键的生物学特征。Palazzo等[7]提出了一个肿瘤突变谱分析流程,通过区分有害和非有害的变异,让自编码器更真实地表示肿瘤细胞特性,这是使用自编码器对大规模泛癌肿瘤数据的突变谱进行低维空间学习的首次尝试。
AE常见的一类应用为针对单一癌症类型进行生存分析或新癌症亚型识别。Wang等[20]使用VAE捕捉到肺癌不同亚型DNA甲基化的表达模式。Tan等[21]基于乳腺癌基因表达数据挖掘代表雌激素受体状态和预后意义的深层特征,这些特征由DAE模型构建,并在下游分析中具有显著的生物学意义。Danaee等[22]使用栈式降噪自编码器提取相关基因的子集并发现潜在的癌症生物标志物,提高了乳腺癌检测分类的性能。
单细胞组学研究(Single cell RNA sequencing,scRNA-seq) 的发展使得大规模获取单细胞转录组特征成为可能[23]。Wang等[24]利用深度自编码器对不同批次的scRNA-seq数据进行批量效应校正;Liang等[25]将AE应用于细胞周期效应的去除,提高了细胞周期描述的准确性和健壮性,这对建立各种细胞图谱和研究肿瘤异质性有帮助。还有研究在血细胞的单细胞RNA测序数据和乳腺癌患者的RNA测序数据上评估和验证了稀疏自编码器和变分自编码器进行基因集分析的可行性[26]。另外,一个名为“Dhaka”[27]的项目使用变分自编码方法,将CNV和scRNA-seq数据降维并转换为更有效的区分肿瘤亚群的特征空间。
2.1.2 多组学数据综合分析利用多组学层次信息,有望为研究生物系统提供更全面的见解。但是,如何对这些异构数据集进行无监督整合是研究者们面临的挑战。我们可以认为每一类组学数据对应一个特征空间,通过整合多组学数据可以连接不同层次的分子特征空间,对阐明各种疾病的分子通路具有重要意义。Liu等[35]使用降噪自编码器从乳腺癌基因表达和拷贝数变异数据中提取深层特征,证实了整合多种数据源有利于患者临床特征和预后相关特征的挖掘。Ma等[4]将领域知识纳入到训练目标中,开发了一种具有网络约束的多视图因式分解自编码器方法,它可以集成多组学数据和领域知识(如分子相互作用网络等),从而准确地预测临床结果。
在其他癌症中,AE也有广泛的应用。Francescatto等[48]整合转录组测序、微阵列和拷贝数变异数据进行神经母细胞瘤预后分析;Chaudhary等[49]基于mRNA表达、微小RNA (microRNA,miRNA) 表达、甲基化和临床信息对肝细胞癌进行亚型异质性的探索;Gu等[28]利用栈式降噪自编码器分析5个肾透明细胞癌(Clear cell renal cell carcinoma,ccRCC) 基因组数据集,成功地发现了两个ccRCC亚型;Zhang等[50]使用AE整合基因表达、拷贝数变异、RNA测序数据,确定出高危神经母细胞瘤两个生存差异显著的子类型;Xu等[29]在栈式自编码器的基础上提出一种深度弹性神经森林框架,利用miRNA、DNA甲基化和基因表达数据成功地对浸润性乳腺癌、多形胶质母细胞瘤和卵巢癌进行了亚型分类。
此外,还有研究结合生物注释进行癌症途径分析或差异表达,如:栈式降噪自编码器多标签学习模型[30]及其可视化工具可用于查看癌症通路中的新基因功能;利用基因表达与DNA甲基化之间相互作用,Kim等[36]发展了乳腺癌综合通路生存预测模型,该模型首先从基因表达和DNA甲基化数据中提取单个通路谱矩阵,在整合图上进行随机游走,然后将降噪自编码器应用于通路图谱,以进一步识别重要的通路特征和基因;Wenric等[41]利用变分自编码器和回归器从特征空间提取基因重要性的排序,以识别与生存相关的基因子集,该研究证明了基于监督学习的基因选择方法在RNA-seq研究中极具潜力。
在基因组层次上,除了编码蛋白的基因之外,还有许多虽然不表达但却参与基因表达调控的“暗物质”,叫作非编码RNA (Non-coding RNA,ncRNAs)。miRNA就是一种小的非编码RNA,通过转录后沉默来调节基因表达,Pyman等[51]使用栈式自编码器和多层感知机(Multi-layer perceptron,MLP) 模型,集成miRNA表达、顺反子注释和序列注释信息构建了一个癌症分类器(Deep cancer classifier,DCC),能够在30多种人体组织中准确预测癌症的存在。还有证据表明,长链非编码RNA (Long noncoding RNA,lncRNA) 在各种生物过程中发挥着重要的作用[52],因此Xuan等[43]构建了一个基于注意力机制和卷积自编码器的卷积神经网络预测模型,从中间层学习lncRNA-疾病的关联形式。
2.2 自编码器在生物医学图像中的应用生物医学成像是另一个活跃的研究领域,基于人工智能(Artificial intelligence,AI) 的计算机辅助诊断(Computer aided diagnosis,CAD) 在智能医疗领域的应用越来越受到重视。该领域研究的通常是患者临床治疗的生物医学图像,如计算机断层扫描(Computed tomography,CT)、磁共振成像、超声波成像等。尽管CNN在图像识别中占据统治地位,但也有研究者使用自编码器开展图像研究,将其用于癌症诊断(如肺癌[53]、乳腺癌[31])、心脏病识别[54-55]或其他精神疾病检测。已经有研究在基准数据集上进行实验,验证了卷积自编码器在特征学习方面的能力和局部结构保存的有效性[56],同样还有研究提出了一种降噪自编码器自组织映射模型[57],在文本、图像的光学识别中证明了其效率、性能和投影能力。Liu等[58]利用深度自编码-分类网络对分类误差和图像重建误差进行联合优化,在小规模数据集的情况下获得了对HEp-2细胞很好的分类效果。
利用组织病理学图像是诊断所有癌症类型的“金标准”[31]。从临床角度来看,直径大于3 mm的结节通常被称为肺结节,而增大的结节更可能会癌变,有研究者分别基于栈式降噪自编码器[32]、卷积自编码器[44-45]开发了针对CT图像或胸部X光检查的诊断模型。
细胞核的组织学特征在疾病的诊断、预后和分析中起着关键作用,Hou等[40]利用稀疏卷积自编码器,成功对细胞核的位置和外观进行了编码,在淋巴细胞区分、淋巴细胞富集区域识别等任务中获得了良好的分类效果。此外,Xu等[31]利用栈式稀疏自编码器,从乳腺癌组织病理学图像中学习到细胞核特征,提高了核检测的精度和效率。
在其他癌症方面,Kharazmi等[39]将稀疏自编码器学习获得的核权值作为过滤器,对病变图像进行卷积,然后将得到的特征图与患者档案信息进行整合,实现对基底细胞癌的早期检测;Vaidhya等[59]利用栈式降噪自编码器从核磁共振图像(Nuclear magnetic resonance imaging,NMRI)中准确分割胶质瘤。此外,还有针对前列腺癌[60-61]、膀胱癌[62]的项目。
不同的自编码器在图像识别领域有自己独特的优势。变分自编码器可以学习健康图像的编码分布,并且能够集成数据分布的先验知识[42];卷积自编码器能够对提取的局部特征进行自编码训练,学习成为鲁棒的特征检测器,通过将这一机制整合到更广泛的疾病分级框架中,可以显著提高准确性;正则自编码器,对输入图像进行“损坏”处理或引入约束条件,可以在一定程度上解决生物医学成像噪声较多的问题。
2.3 自编码器在其他类型数据中的应用除了组学数据、生物医学图像之外,还有其他生物数据被应用于癌症的医学研究。利用可穿戴技术和移动平台,医生能够实时跟踪人们的医疗数据,获取大量生物医学信号,如心电图(Electrocardiogram,ECG)、脑电图(Electroencephalogram,EEG),但是这类数据经常被各种噪声所污染,因此我们有理由相信AE可以在这一领域取得不俗表现。事实上,已经有研究利用卷积自编码器[46]、收缩降噪自编码器[47]处理ECG信号,还有研究利用栈式自编码器处理EEG信号来对癫痫[33-34]进行检测,这都表明自编码器在抵抗噪声以及特征重构方面的强大性能。
另外,栈式降噪自编码器可以被用于处理电子健康档案(Electronic health records,EHRs)[37],为临床决策提供信息,还被用来从急性髓系白血病患者的人口统计学、细胞遗传学、所选基因突变状态和蛋白组数据中挖掘FLT3-ITD突变相关的关键蛋白[63]。
3 自编码器在肿瘤信息学中的应用总结、展望及讨论自编码器能够利用有限的隐层单元提取的特征构建输入数据的近似值,因此可以认为它能够捕获数据本身的特性。通过对自编码器进行不同的优化,包括添加不同的约束或引入随机噪声,可以使自编码器更加适应生物数据中样本小、维度高、噪声多的情况。通过回顾整理前人的研究工作,总结自编码器在癌症信息学研究中有如下应用:(1) 进行特征学习以用于后续的分类、聚类等任务。这是自编码器在这一领域最主要的应用,它既可以处理单类型数据,也可以通过多输入管道处理异构数据,尤其是它可以将多组学数据进行有效整合,为研究癌症生物学机制提供更广阔的视角,这对阐明各种疾病的分子通路具有重要意义。通过分析模型的结构,也可以对最重要的特征进行基因本体注释搜索,促进生物学发现。(2) 进行数据降维和可视化。在某些情况下,我们并不会直接对原始数据进行可视化,而是会先从中捕获某些有意义的特征再进行可视化的分析。自编码器的优势就在于可以调整隐藏层神经元的数目,尽可能多地将信息编码到隐藏节点中,从而使我们可以根据需要将数据直接降到二维、三维或任意维度,其灵活性远远优于t-SNE等方法。(3) 数据去噪或生成新数据。由于实验平台、实验操作等因素的存在,无论是组学数据还是生物医学图像,都不可能获得完全“干净”的实验数据。通过对输入数据添加“噪声”或者对损失函数添加不同的约束,可以使自编码器学会去除这种噪声,从而增加鲁棒性和泛化能力。还可以利用变分自编码器学习数据内在的分布模式,生成有潜在意义的新数据或者从中捕获生物学相关的特征。(4) 图像或信号数据压缩。与JPEG (Joint photographic experts group) 这样的通用无损图片压缩技术相比,图卷积自编码器在压缩图像的同时能够捕获数据中最显著的特征,从而为病理学等相关研究提供关键性的证据。
本文中,我们详细介绍了一种非监督的神经网络学习算法——自编码器。通过对模型的体系结构、输入数据以及本领域的进展进行广泛的回顾,我们讨论总结了自编码器在癌症生物信息学研究中的应用和前景。笔者认为,自编码器凭借其灵活多变的结构以及强大的自我学习能力,能够胜任处理各种复杂生物医学数据的任务。通过整合不同层次的分子特征空间,自编码器可以为研究癌症生物学机制提供更广阔的视角,是进行癌症的生物信息学研究的有力工具。
参考文献
| [1] | Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] | Kourou K, Exarchos TP, Exarchos KP, et al. Machine learning applications in cancer prognosis and prediction. Comput Struct Biotechnol J, 2015, 13: 8-17. DOI:10.1016/j.csbj.2014.11.005 |
| [3] | Min S, Lee B, Yoon S. Deep learning in bioinformatics. Brief Bioinform, 2017, 18(5): 851-869. |
| [4] | Ma TL, Zhang AD. Integrate multi-omics data with biological interaction networks using multi-view factorization auto encoder (MAE). BMC Genomics, 2019, 20(S11): 944. DOI:10.1186/s12864-019-6285-x |
| [5] | Hinton GE, Salakhutdinov RR. Reducing the dimensionality of data with neural networks. Science, 2006, 313(5786): 504-507. DOI:10.1126/science.1127647 |
| [6] | Bourlard H, Kamp Y. Auto-association by multilayer perceptrons and singular value decomposition. Biol Cybern, 1988, 59(4/5): 291-294. |
| [7] | Palazzo M, Beauseroy P, Yankilevich P. A pan-cancer somatic mutation embedding using autoencoders. BMC Bioinformatics, 2019, 20: 655. DOI:10.1186/s12859-019-3298-z |
| [8] | Bengio Y, Lamblin P, Popovici D, et al. Greedy layer-wise training of deep networks//Proceedings of the 19th International Conference on Neural Information Processing Systems. Cambridge, MA, United States: MIT Press, 2007: 153-160. |
| [9] | Bengio Y, Courville A, Vincent P. Representation learning: a review and new perspectives. IEEE Trans Patt Anal Mach Intellig, 2013, 35(8): 1798-1828. DOI:10.1109/TPAMI.2013.50 |
| [10] | Vincent P, Larochelle H, Bengio Y, et al. Extracting and composing robust features with denoising autoencoders//Proceedings of the 25th International Conference on Machine Learning (ICML). New York, NY, United States: ACM, 2008: 1096-1103. |
| [11] | Ranzato MA, Poultney C, Chopra S, et al. Efficient learning of sparse representations with an energy-based model//Proceedings of the 19th International Conference on Neural Information Processing Systems. Cambridge, MA, United States: MIT Press, 2007: 1137-1144. |
| [12] | Rifai S, Vincent P, Muller X, et al. Contractive auto-encoders: explicit invariance during feature extraction//Proceedings of the 28th International Conference on International Conference on Machine Learning (ICML). Madison, WI, United States: Omni Press, 2011: 833-840. |
| [13] | Lee H, Ekanadham C, Ng AY. Sparse deep belief net model for visual area V2//Proceedings of the 20th International Conference on Neural Information Processing Systems. Red Hook, NY, United States: Curran Associates Inc., 2007: 1416-1423. |
| [14] | Ranzato MA, Boureau YL, LeCun Y. Sparse feature learning for deep belief networks//Proceedings of the 20th International Conference on Neural Information Processing Systems. Red Hook, NY, United States: Curran Associates Inc., 2007: 1185-1192. |
| [15] | Kingma D P, Welling M. Auto-encoding variational bayes//2014 2nd International Conference on Learning Representations. Banff, AB, Canada, arXiv preprint arXiv: 1312.6114, 2013. |
| [16] | Masci J, Meier U, Cire?an D, et al. Stacked convolutional auto-encoders for hierarchical feature extraction//Proceedings of the 21th International Conference on Artificial Neural Networks. Berlin, Heidelberg: Springer-Verlag. 2011: 52-59. |
| [17] | Fakoor R, Ladhak F, Nazi A, et al. Using deep learning to enhance cancer diagnosis and classification//Proceedings of the 30th International Conference on Machine Learning(ICML). New York, USA: ACM, 2013, 28. |
| [18] | Way GP, Greene CS. Evaluating deep variational autoencoders trained on pan-cancer gene expression//Proceedings of the 19th International Conference on Neural Information Processing Systems. Long Beach, CA, USA, 2017. |
| [19] | Chen HIH, Chiu YC, Zhang TH, et al. GSAE: an autoencoder with embedded gene-set nodes for genomics functional characterization. BMC Syst Biol, 2018, 12: 142. DOI:10.1186/s12918-018-0642-2 |
| [20] | Wang ZX, Wang YD. Extracting a biologically latent space of lung cancer epigenetics with variational autoencoders. BMC Bioinformatics, 2019, 20: 568. DOI:10.1186/s12859-019-3130-9 |
| [21] | Tan J, Ung M, Cheng C, et al. Unsupervised feature construction and knowledge extraction from genome-wide assays of breast cancer with denoising autoencoders. Pac Symp Biocomput, 2015, 20: 132-143. |
| [22] | Danaee P, Ghaeini R, Hendrix DA. A deep learning approach for cancer detection and relevant gene identification. Pac Symp Biocomput, 2017, 22: 219-229. |
| [23] | Bendall SC, Davis KL, Amir E-AD, et al. Single-cell trajectory detection uncovers progression and regulatory coordination in human B cell development. Cell, 2014, 157(3): 714-725. DOI:10.1016/j.cell.2014.04.005 |
| [24] | Wang TX, Johnson TS, Shao W, et al. BERMUDA: a novel deep transfer learning method for single-cell RNA sequencing batch correction reveals hidden high-resolution cellular subtypes. Genome Biol, 2019, 20: 165. DOI:10.1186/s13059-019-1764-6 |
| [25] | Liang SH, Wang F, Han JC, et al. Latent periodic process inference from single-cell RNA-seq data. Nat Commun, 2020, 11: 1441. DOI:10.1038/s41467-020-15295-9 |
| [26] | Gold MP, LeNail A, Fraenkel E. Shallow sparsely-connected autoencoders for gene set projection. Pac Symp Biocomput, 2019, 24: 374-385. |
| [27] | Rashid S, Shah S, Bar-Joseph Z, et al. Dhaka: variational autoencoder for unmasking tumor heterogeneity from single cell genomic data. Bioinformatics, 2019. DOI:10.1093/bioinformatics/btz095 |
| [28] | Gu TJ, Zhao XW. Integrating multi-platform genomic datasets for kidney renal clear cell carcinoma subtyping using stacked denoising autoencoders. Sci Rep, 2019, 9: 16668. DOI:10.1038/s41598-019-53048-x |
| [29] | Xu J, Wu P, Chen YH, et al. A hierarchical integration deep flexible neural forest framework for cancer subtype classification by integrating multi-omics data. BMC Bioinformatics, 2019, 20: 527. DOI:10.1186/s12859-019-3116-7 |
| [30] | Guan RC, Wang X, Yang MQ, et al. Multi-label deep learning for gene function annotation in cancer pathways. Sci Rep, 2018, 8: 267. DOI:10.1038/s41598-017-17842-9 |
| [31] | Xu J, Xiang L, Liu QS, et al. Stacked sparse autoencoder (SSAE) for nuclei detection on breast cancer histopathology images. IEEE Trans Med Imag, 2016, 35(1): 119-130. DOI:10.1109/TMI.2015.2458702 |
| [32] | Sun W, Zheng B, Qian W. Computer aided lung cancer diagnosis with deep learning algorithms//Medical Imaging 2016: Computer-aided Diagnosis. International Society for Optics and Photonics, 2016, 9785: 97850Z. |
| [33] | Wen TX, Zhang ZN. Deep convolution neural network and autoencoders-based unsupervised feature learning of EEG signals. IEEE Access, 2018, 6: 25399-25410. DOI:10.1109/ACCESS.2018.2833746 |
| [34] | Supratak A, Li L, Guo YK. Feature extraction with stacked autoencoders for epileptic seizure detection//36th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. Chicago, IL, USA: IEEE, 2014: 4184-4187. |
| [35] | Liu Q, Hu PZ. Association analysis of deep genomic features extracted by denoising autoencoders in breast cancer. Cancers (Basel), 2019, 11(4): 494. DOI:10.3390/cancers11040494 |
| [36] | Kim SY, Kim TR, Jeong HH, et al. Integrative pathway-based survival prediction utilizing the interaction between gene expression and DNA methylation in breast cancer. BMC Med Genomics, 2018, 11(S3): 68. DOI:10.1186/s12920-018-0389-z |
| [37] | Miotto R, Li L, Kidd BA, et al. Deep patient: an unsupervised representation to predict the future of patients from the electronic health records. Sci Rep, 2016, 6: 26094. DOI:10.1038/srep26094 |
| [38] | Xiao YW, Wu J, Lin ZL, et al. A semi-supervised deep learning method based on stacked sparse auto-encoder for cancer prediction using RNA-seq data. Comput Methods Progr Biomed, 2018, 166: 99-105. DOI:10.1016/j.cmpb.2018.10.004 |
| [39] | Kharazmi P, Kalia S, Lui H, et al. A feature fusion system for basal cell carcinoma detection through data-driven feature learning and patient profile. Skin Res Technol, 2018, 24(2): 256-264. DOI:10.1111/srt.12422 |
| [40] | Hou L, Nguyen V, Kanevsky AB, et al. Sparse autoencoder for unsupervised nucleus detection and representation in histopathology images. Pattern recognition, 2019, 86: 188-200. DOI:10.1016/j.patcog.2018.09.007 |
| [41] | Wenric S, Shemirani R. Using supervised learning methods for gene selection in RNA-seq case-control studies. Front Genet, 2018, 9: 297. DOI:10.3389/fgene.2018.00297 |
| [42] | Uzunova H, Schultz S, Handels H, et al. Unsupervised pathology detection in medical images using conditional variational autoencoders. Int J Comput Assist Radiol Surg, 2019, 14(3): 451-461. DOI:10.1007/s11548-018-1898-0 |
| [43] | Xuan P, Sheng N, Zhang TG, et al. CNNDLP: a method based on convolutional autoencoder and convolutional neural network with adjacent edge attention for predicting lncRNA-disease associations. Int J Mol Sci, 2019, 20(17): 4260. DOI:10.3390/ijms20174260 |
| [44] | Chen M, Shi XB, Zhang Y, et al. Deep features learning for medical image analysis with convolutional autoencoder neural network. IEEE Trans Big Data, 2017, 1-1. |
| [45] | Wang CM, Elazab A, Jia FC, et al. Automated chest screening based on a hybrid model of transfer learning and convolutional sparse denoising autoencoder. Biomed Eng Online, 2018, 17(1): 63-81. DOI:10.1186/s12938-018-0496-2 |
| [46] | Yildirim O, Tan RS, Acharya UR. An efficient compression of ECG signals using deep convolutional autoencoders. Cognit Syst Res, 2018, 52: 198-211. DOI:10.1016/j.cogsys.2018.07.004 |
| [47] | Xiong P, Wang HR, Liu M, et al. A stacked contractive denoising auto-encoder for ECG signal denoising. Physiol Measur, 2016, 37(12): 2214-2230. DOI:10.1088/0967-3334/37/12/2214 |
| [48] | Francescatto M, Chierici M, Dezfooli SR, et al. Multi-omics integration for neuroblastoma clinical endpoint prediction. Biol Direct, 2018, 13: 5. DOI:10.1186/s13062-018-0207-8 |
| [49] | Chaudhary K, Poirion OB, Lu LQ, et al. Deep learning-based multi-omics integration robustly predicts survival in liver cancer. Clin Cancer Res, 2018, 24(6): 1248-1259. DOI:10.1158/1078-0432.CCR-17-0853 |
| [50] | Zhang L, Lv CK, Jin YQ, et al. Deep learning-based multi-omics data integration reveals two prognostic subtypes in high-risk neuroblastoma. Front Genet, 2018, 9: 477. DOI:10.3389/fgene.2018.00477 |
| [51] | Pyman B, Sedghi A, Azizi S, et al. Exploring microRNA regulation of cancer with context-aware deep cancer classifier. Pac Symp Biocomput, 2019, 24: 160-171. |
| [52] | Guttman M, Rinn JL. Modular regulatory principles of large non-coding RNAs. Nature, 2012, 482(7385): 339-346. |
| [53] | Kumar D, Wong A, Clausi DA. Lung nodule classification using deep features in CT images//12th Conference on Computer and Robot Vision. Halifax, NS, Canada: IEEE, 2015: 133-138. |
| [54] | Roy AG, Conjeti S, Carlier SG, et al. Multiscale distribution preserving autoencoders for plaque detection in intravascular optical coherence tomography//IEEE 13th International Symposium on Biomedical Imaging (ISBI). Prague, Czech Republic: IEEE, 2016: 1359-1362. |
| [55] | Yuan C, Yan Y, Zhou L, et al. Automated atrial fibrillation detection based on deep learning network//IEEE International Conference on Information and Automation (ICIA). Ningbo, China: IEEE, 2016: 1159-1164. |
| [56] | Guo X, Liu X, Zhu E, et al. Deep clustering with convolutional autoencoders// International Conference on Neural Information Processing. Springer, Cham, 2017: 373-382. |
| [57] | Ferles C, Papanikolaou Y, Naidoo KJ. Denoising autoencoder self-organizing map (DASOM). Neural Netw, 2018, 105: 112-131. DOI:10.1016/j.neunet.2018.04.016 |
| [58] | Liu JX, Xu B, Shen LL, et al. HEp-2 cell classification based on a deep autoencoding- classification convolutional neural network//IEEE 14th International Symposium on Biomedical Imaging (ISBI). Melbourne, VIC, Australia: IEEE, 2017: 1019-1023. |
| [59] | Vaidhya K, Thirunavukkarasu S, Alex V, et al. Multi-modal brain tumor segmentation using stacked denoising autoencoders//BrainLes 2015: Brainlesion: Glioma, Multiple Sclerosis, Stroke and Traumatic Brain Injuries. Cham: Springer, 2016: 181-194. |
| [60] | Yan K, Li CY, Wang XY, et al. Comprehensive autoencoder for prostate recognition on MR images//IEEE 13th International Symposium on Biomedical Imaging (ISBI). Prague, Czech Republic: IEEE, 2016: 1190-1194. |
| [61] | Reda I, Shalaby A, El-Ghar MA, et al. A new NMF-autoencoder based CAD system for early diagnosis of prostate cancer//IEEE 13th International Symposium on Biomedical Imaging (ISBI). Prague, Czech Republic: IEEE, 2016: 1237-1240. |
| [62] | Shaker MS, Wael M, Yassine IA, et al. Cardiac MRI view classification using autoencoder//2014 Cairo International Biomedical Engineering Conference (CIBEC). Giza, Egypt: IEEE, 2014: 125-128. |
| [63] | Liang CA, Chen L, Wahed A, et al. Proteomics analysis of FLT3-ITD mutation in acute myeloid leukemia using deep learning neural network. Am J Clin Pathol, 49(1): 119-126. |
