

西南大学 生命科学学院,重庆 400716
收稿日期:2019-03-29;接收日期:2019-07-29
基金项目:国家自然科学基金(No. 31501100)资助
摘要:组蛋白赖氨酸特异性去甲基化酶1A (Histone lysine-specific demethylase 1A,KDM1A)作为组蛋白赖氨酸特异性去甲基化酶(Histone lysine-specific demethylase)家族的一员,在信号传导、染色体重构、胚胎发育、造血和糖脂代谢等生物学过程中起着重要的作用。近年来的研究及临床证据表明,KDM1A的表达与肿瘤的发生发展密不可分,通过与不同的复合物结合并介导不同的下游信号通路,对多种肿瘤的生长增殖起着关键的调节作用,例如前列腺癌、乳腺癌、肺癌和肝癌等。在大多数情况下,KDM1A在肿瘤的发生发展中扮演着促癌基因角色。文中结合近年来有关文献,阐述了KDM1A在多种肿瘤发生及发展中的研究进展,总结了其作用机制,并对以KDM1A为靶点的抑癌治疗的应用前景进行了展望。
关键词:组蛋白赖氨酸特异性去甲基化酶1A肿瘤发生肿瘤发展组蛋白去甲基化酶
Function of histone demethylasehistone lysine-specific demethylase 1A in tumor initiation and progression
Ximei Zhang, Xinli An, Shunqin Zhu, Yan Liu


School of Life Sciences, Southwest University, Chongqing 400716, China
Received: March 29, 2019; Accepted: July 29, 2019
Supported by: National Natural Science Foundation of China (No. 31501100)
Corresponding author: Yan Liu. Tel/Fax: +86-23-68252365; E-mail: liuyan@swu.edu.cn.
Abstract: As a member of the histone lysine-specific demethylase family, KDM1A plays a pivotal role in biological processes including signal transduction, chromatin reprogramming, embryo development, hematopoiesis, glucose and lipid metabolism. Recently, increasing studies and clinical evidences suggest that the expression of KDM1A is related to initiation and development of tumors and plays a key role in regulating of initiation and development of tumors, such as prostate cancer, breast cancer, lung cancer and liver cancer. KDM1A binds to distinct complexes and mediates different downstream signaling pathways. However, KDM1A often plays an oncogenic role in the initiation and development of tumors. Based on the current literatures, we describe the latest research of KDM1A in the initiation and progression of various tumors, and summarize its mechanism of actions, to provide clues for cancer therapy.
Keywords: histone lysine-specific demethylase 1A(KDM1A)tumor initiationtumor developmenthistone demethylase
随着近年来肿瘤在细胞及分子水平研究的不断进展,表观遗传修饰在肿瘤的发生和发展过程中具有不可或缺的作用。表观遗传修饰包括DNA甲基化、组蛋白修饰、染色体重塑、非编码RNA调控[1]。组蛋白赖氨酸修饰对肿瘤的影响是当前的研究热点之一。研究表明H3K27、H3K9、H4K20、H3K79、H2BK5的单甲基化都与基因的激活有关,而H3K27、H3K9以及H3K79的三甲基化则与基因抑制有关,表明组蛋白的甲基化程度能够影响相关基因的表达情况[2],组蛋白的甲基化程度对于维持正常的生命活动是不可或缺的。2004年,Shi和同事第一次发现赖氨酸特异性去甲基化酶(Lysine demethylase 1A,KDM1A也被称为LSD1或AOF2、BHC110),验证了组蛋白的甲基化是可逆的假说[3]。KDM1A除了能够去除组蛋白H3上第4位、第9位赖氨酸上的甲基,还能去除p53上第370位赖氨酸上的甲基,进而抑制p53介导的转录上调及促进凋亡的作用[4]。KDM1A去除DNA甲基转移酶1 (DNA methyltransferase 1,DNMT1)上的甲基后能使其在体内稳定存在[5]。以上的这些研究表明,KDM1A除了靶向组蛋白外,还靶向非组蛋白。根据KDMs的序列同源性和催化机制,KDMs可分为两个家族:1)一类是KDM1s,具有黄素腺嘌呤二核苷酸(Flavin adenine dinucleotide,FAD)依赖的单胺氧化酶,其功能是去除组蛋白H3K4me1/2以及H3K9me1/2上的甲基化,该家族由KDM1A和KDM1B构成。2)另一类去甲基化酶超家族,该家族成员需要二价铁离子(Fe2+)和α-酮戊二酸为辅助因子辅助催化并含Jumonji C (JmjC)结构域[6],根据功能和结构域的不同又可将其细分为7个亚家族(KDM2–8)[7-11]。KDM2–8家族参与多种肿瘤的发生发展过程,其成员的突变、缺失、扩增都可以作为肿瘤的临床诊断指标,如:KDM5C突变导致的失活可能预示着胃癌的存在;KDM4C片段的缺失能够在一定程度预示着前列腺癌的发生等[12]。KDM2–8家族成员均可催化组蛋白的去甲基化修饰,但是具有底物特异性,能通过去甲基化,改变组蛋白的甲基化程度进而影响其转录活性、染色体结构、损伤修复等生理过程[13]。研究进一步表明KDM2–8家族成员在癌症中的作用途径之一是通过调控DNA的合成进展,从而调控细胞周期,影响多种关键基因的转录。因为该家族成员在结构上高度相似,很难针对某一成员特异的酶活性找到匹配的抑制剂,因此目前还没有合适的针对某个成员的药物用于临床[12]。
在KDM1s家族中KDM1A和KDM1B的氨基酸序列和结构高度相似,都含有SWIRM结构域和胺基氧化酶结构域[3],但KDM1B氨基末端包含一个独特的Zf-CW结构域,Zf-CW结构域是一种包含4个半胱氨酸的典型锌指结构,其名称来源于保守的半胱氨酸和色氨酸基团[14]。KDM1s在细胞中靶标组蛋白、非组蛋白的翻译后修饰,从而发挥着不同的功能。在小鼠、线虫、斑马鱼、水稻等中,KDM1s都被证实在生命活动中起着关键的调节作用[15-18],又因为生命活动的失调与肿瘤的发生发展密切相关,KDM1s在肿瘤的病理形成及发展的通路中也发挥着多种功能。
作为KDM1s家族的成员,KDM1A与生物体内不同物质形成不同的复合物从而调控去甲基化酶活性和染色体不同位置上特定底物的结合能力。KDM1A参与多种生物学过程,包括信号传导、染色体重构、转录调控、胚胎发育、有丝分裂、造血功能等。而且KDM1A与多种疾病如Ⅱ型糖尿病[19]、甲型流感[20]、肺结核病[21]、白血病[22]、神经退行性疾病[23]、心血管疾病[24]的发生发展过程有着密切的关系。除此之外,一系列的研究表明,KDM1A与肿瘤的发生[4, 25-26]和发展[27-28]密切相关,其表达量与癌症患者的存活率具有相关性[29]。本文将对KDM1A影响肿瘤发生发展及分子机制进行总结,并在此基础上对今后的研究及应用前景进行了展望。
1 KDM1A简介KDM1A定位于细胞核,是一段由3个区域共852个氨基酸构成的多肽,其催化机制是将氢化物从甲基化的赖氨酸上转移到FAD辅助因子开始,生成不稳定的亚胺中间体,水解后释放甲醛[30]。KDM1A的N端含有一个SWIRM结构域(氨基残基位点172–272),该结构域不仅能与DNA分子结合,而且在蛋白质-蛋白质相互作用以及维持底物结构稳定性方面发挥着重要作用[31]。C端含有胺氧化酶结构域(AOL)并被TOWER结构域(氨基残基位点415–515)分成两个亚结构域[30]。其中一个AOL亚基(氨基残基位点272–415)能与SWIRM结构域相互作用,形成一个FAD结合结构域和催化活性中心[32],催化活性中心与其他胺氧化酶结构域具有相似的结构,但是在底物结合亚结构域上含一个表面带酸性特征的结合“口袋”,该“口袋”通过与组蛋白H3前20个氨基酸的相互作用调控碱性组蛋白尾巴[14],除此之外,AOL-SWIRM结构域内部残基通过改变蛋白质结构和蛋白质与底物之间的相互作用调控KDM1A的催化能力[33]。另一个AOL亚基(氨基残基位点515–852)能结合特异性底物包括蛋白复合体(Protein complexes)、受体(Receptors)、非编码RNA (Non-coding RNA)、非组蛋白底物(Non-histone substrates)、微小-RNA (Micro-RNA),转录因子(Transcription factor,TF)等[34]。近年来的研究指出TOWER结构域能与RE1-沉默转录因子(CoREST,又称RCOR1)共抑制因子相结合,形成杂二聚体复合物,提高了KDM1A的稳定性和催化活性[35]。基因间长非编码RNA (Long intergenic noncoding RNAs,LincRNAs)能够调控染色体状态以及表观遗传。HOTAIR作为一种LincRNAs,因其5′端能与多梳抑制复合物2 (Polycombrepressive complex 2,PRC2)结合,3′端能与KDM1A/CoREST/REST复合物结合,故HOTAIR能够作为支架将PRC2和KDM1A/ CoREST/REST复合物联系在一起,并定位于染色质上,调控H3K4以及H3K27的去甲基化水平,进而对靶基因进行表观修饰[36]。在人类MCF-7乳腺癌细胞的388个c-Myc靶基因中,研究者发现这些基因的启动子可能被肿瘤蛋白HBXIP所占据。HBXIP的表达与c-Myc的靶基因cyclin A、elF4F以及LDHA的表达密切相关。当利用RNAi介导HBXIP的沉默,将会消除这些基因的上调。在结构上,HBXIP能够通过亮氨酸拉链与c-Myc直接作用,并募集HOTAIR和KDM1A,其中HOTAIR作为支架。HBXIP、HOTAIR或者KDM1A的沉默都足以导致体内体外c-Myc促癌进程受到阻断[37]。提示HBXIP/HOTAIR/KDM1A复合物能够作为治疗乳腺癌新的靶点。HOXA11-AS作为一种IncRNA与胃癌发生发展密切相关,研究进一步发现HOXA11-AS能够作为支架将Zeste基因增强子同源物2 (Enhancer of zeste homolog 2,EZH2)和KDM1A募集起来形成EZH2/HOXA11-AS/KDM1A复合物,该复合物能作为胃癌发生发展的关键效应子[38]。在胃癌中,IncRNA FOXD2-AS1与HOXA11-AS具有相似的功能,也能通过将EZH2和KDM1A联系起来并沉默促红细胞生成素产生肝细胞受体(Erythropoie-tin-producing hepatocyte receptor B3,EphB3)的表达进而促进胃癌的发生[39]。以上这些研究表明LincRNA作为KDM1A的支架,将KDM1A与其他多种复合物联系在一起,在肿瘤中发挥着复杂的功能。KDM1A除了通过支架与其他复合物连接发挥作用外,还参与多种转录共抑制复合物的形成如组蛋白去乙酰化酶(Histone deacytylation,HDACs)[40]、核小体重构和去乙酰化复合物(Nucleosome remodeling and deacetylation,NuRDs)[27]、C-端结合蛋白(C-terminal binding proteins,CtBPs)[41]。在与转录活性相关的雌激素受体(Estrogen receptor,ER)[42]、雄激素受体(Androgen receptor,AR)[43]复合物中也发现KDM1A参与其中。KDM1A主要表达在肝脏、胃、脂肪、神经中[44-45]。如图 1所示,KDM1A特殊的结构赋予其独特的功能,参与到多种生物学过程。
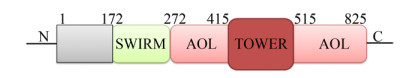 |
| 图 1 KDM1A的结构域体系[14] Fig. 1 Domain architecture of KDM1A[14]. |
| 图选项 |
2 KDM1A与肿瘤恶性肿瘤是环境和遗传因素长期共同作用的结果。癌细胞是一类失去控制并获得持久分裂能力的细胞。基因组的不稳定、自噬、代谢紊乱、端粒的缩短以及活性氧都与癌症的发生发展密切相关。20世纪20年代,奥托·沃泊格(Otto Warburg)发现肿瘤细胞的产能代谢与正常细胞的产能代谢存在较大的差异,即使在有氧条件下,也不会采用绝大数正常细胞所采用的线粒体呼吸作用来提供能量,而是采用糖酵解途径,该现象被称为沃泊格效应(Warburg effect)[46]。沃泊格基于以上的发现,提出这种代谢异常是导致正常细胞转变为癌细胞的根本原因。糖代谢异常被越来越多的研究人员认为是肿瘤发生发展中的重要标志之一。研究表明KDM1A在调控细胞糖代谢中发挥着不可或缺的作用,而糖代谢与癌症的发生发展关系密切[47-48]。
KDM1A作为糖代谢中重要的调节子,通过影响糖代谢进而影响肿瘤的发生发展。KDM1A能通过与低氧诱导因子(Hypoxia inducible factor-1α,HIF1α)协同作用维持胰腺癌细胞的糖酵解过程进而维持胰腺癌细胞不受控制的增殖[47]。在食管癌(Esophageal cancer)细胞中,研究人员通过测定细胞外酸化率(Extracellular acidification rate,ECAR)和耗氧率(Oxygen consumption rate,OCR)来评价细胞内的糖酵解途径和线粒体呼吸途径,该研究证明了KDM1A能通过改变代谢途径促进食管癌细胞的恶性增殖和侵袭[49]。随着研究的不断深入,证明了KDM1A不仅能通过改变糖代谢途径还能通过一些其他途径如利用生物信息学发现KDM1A能够调控多种染色体功能包括染色体在着丝点的重构、异染色质在着丝点的形成、染色体的组装或拆卸以及依赖于甲基化的染色质沉默,提示KDM1A异常表达可以通过调控这些染色质参与多种肿瘤的发生发展过程[28]。大量的研究表明KDM1A在不同类型的肿瘤中发挥着不同的作用(表 1),对于造成这种差异的原因还需要我们进一步探究。
表 1 KDM1A在各种肿瘤中的作用Table 1 The roles of KDM1A in various tumors
| Type of cancer | KDM1A interaction | Functional role of KDM1A | References |
| Prostate cancer | GLUT1 VEGF-A/cyclinA | ↑ Glycolytic process ↓ Proliferation | [50] [51] |
| Non-small cell lung cancer | TIMP3 REST | ↑ Invasion ↑ Oncogenesis | [29] [52] |
| Colorectal cancer | Dickkopf-1 | ↑ Oncogenesis | [53] |
| Breast cancer | HDAC5 RORα2 | ↑ Proliferation ↑ Invasion | [54] [55] |
| Liver cancer | Wnt/β-catenin | ↑ Sorafenib-resistant | [56] |
| Gastric cancer | MicroRNA-329 | ↓ Apoptosis, proliferation, growth | [57] |
| Neuroblastoma | Sestrin 2 | ↑ Apoptosis | [58] |
| Gynecologic malignancies | P62 | ↓ Autophagy | [59] |
表选项
虽然KDM1A在不同肿瘤中发挥着不同的作用,但其含量在大多数肿瘤中都是增加的,推测KDM1A含量的增加与KDM1A获得功能性增加导致表观基因组调控异常的假说是一致的,一定程度上是因为KDM1A调控正常细胞和癌细胞中关键基因的H3K4甲基化程度[60]。
2.1 KDM1A与前列腺癌据了解在2018年新增加的患前列腺癌(Prostate cancer)人数占所有新增加的患癌人数的7.1%,排在肺癌(Lung cancer,11.6%)和乳腺癌(Breast cancer,11.6%)之后。前列腺癌的致死率占患癌人数死亡率的3.8%,是第八大癌症死因[61]。研究显示,KDM1A与前列腺癌的发生发展密切相关。从临床数据来看,KDM1A在去势抵抗性前列腺癌(Castration-resistant prostate cancer,CRPC)患者中具有高表达量[62],而且KDM1A在低分化的前列腺癌细胞中的表达量与前列腺癌的进展及不良预后[63]、Gleason评分、远距离转移都具有良好的正相关性[64]。此外,在前列腺癌组织中,KDM1A表达量的升高伴随着Snail、AR表达水平的升高,钙粘蛋白(E-cadherin)、p53等表达水平下降。除此之外,敲低KDM1A能降低前列腺癌细胞的存活率,并增加前列腺上皮细胞分化标志物的基因表达。利用siRNA抑制KDM1A的表达,不仅能够降低血管内皮生长因子(Vascular endothelial growth factor,VEGF-A)的表达,还能阻断雄激素诱导的前列腺特异性抗原(Prostate specific antigen,PSA)和Tmprss2的表达[51]。
研究显示,作为KDM1A的靶向试剂和表观调节子并依赖于三价铑的复合物1 (Rhodium (Ⅲ) complex 1)能够扰乱人类前列腺癌细胞中KDM1A-H3K4me2之间的相互作用,并增强KDM1A调控的p21、FOXA2 (Forkhead box protein A1)以及骨形态发生蛋白2 (Bone morphogenetic protein,BMP2)等基因启动子的扩增。通过免疫印迹(Western blotting,WB)分析发现,复合物1能够降低葡萄糖转运体(Glucose transporter 1,GLUT1)的表达水平,但是对于KDM1A的表达水平几乎没有影响。在基因水平,当敲除KDM1A后,发现GLUT1的mRNA水平下降了40%。提示复合物1在一定程度上依赖于KDM1A进而调控GLUT1的转录水平[50]。优降宁(Pargyline)作为KDM1A的一种药物抑制剂,能够与FAD形成共价加合物[65],单独使用或者联合用药治疗对细胞生长以及移植瘤模型的生长都具有较大的影响。但是,有研究发现Pargyline在体内[66]和体外[67]对KDM1A的抑制效果较低,提示某些特定的转录或染色体调控对KDM1A的不完全抑制较为敏感。在前列腺癌细胞中,Pargyline能够降低前列腺癌细胞的迁移和侵袭能力,并通过增加体内外钙粘蛋白表达水平,下调氮粘蛋白和波形蛋白的表达来抑制上皮细胞转化(Epithelial-mesenchymal transition,EMT)过程[68]。研究发现雷公藤提取物去甲泽拉木醛(Demethylzeylasteral,T-96)能够逆转胶质瘤(Glioma)细胞的EMT过程,抑制其迁移和侵袭能力[69],表明EMT过程与肿瘤的转移和侵袭过程密切相关。提示抑制KDM1A的表达,能够造成钙粘蛋白、氮粘蛋白、波形蛋白表达改变,并抑制EMT过程进而抑制肿瘤的转移与侵袭,达到抗癌效果。
上述结果明确了KDM1A抑制剂在前列腺癌中的抗癌作用,证明了KDM1A与前列腺癌发生发展密切相关。大多数情况下,KDM1A能够通过雄激素信号通路促进依赖雄激素受体调控雄激素相关蛋白的表达进而诱导前列腺癌的发生,控制前列腺癌的进展。但是因为体内途径的多样性,KDM1A调控前列腺癌的发生发展的具体途径还需要更多的研究去证实。
2.2 KDM1A与乳腺癌乳腺癌(Breast cancer,BC)是女性最易患的癌症同时也是女性死亡率最高的癌症[62]。KDM1A在乳腺癌中的作用主要是与癌细胞的侵袭和转移有关。已有研究显示乳腺癌组织中的KDM1A表达量高于正常的乳腺组织,KDM1A表达量的升高与乳腺癌患者生存降低和不良预后有关[62]。苯环丙胺(Tranylcypromine,TCP)能够非选择性、不可逆地抑制各种单胺氧化酶,广泛用于抑郁症的治疗[70]。已有研究表明较高剂量TCP能够抑制乳腺癌细胞中的KDM1A活性[71]。但又因需要较高浓度的TCP才能抑制肿瘤的生长,所以TCP作为癌症治疗的临床用药具有很大的局限性[72]。因此,一些TCP衍生物如GSK2879522以及ORY-1001等被研发出来,以便更特异性地针对KDM1A,这两种药物目前正处于治疗急性髓性白血病和小细胞肺癌的临床试验第一阶段。以上的研究表明,KDM1A能够作为治疗癌症的新靶点。但KDM1A在乳腺癌中的具体机制还需要进一步研究。研究人员发现过表达组蛋白去乙酰化酶5 (Histone deacetylase 5,HDAC5)能够通过转录后修饰KDM1A蛋白和促进去泛素化酶USP28的稳定性进而维持KDM1A蛋白的稳定性,影响去甲基化的能力。利用shRNA敲除HDAC5后能够抑制细胞的增殖,造成细胞周期阻滞在G0/G1期,诱导凋亡并降低乳腺癌细胞的迁移和菌落形成能力。该研究表明HDAC5-KDM1A轴对于乳腺癌的发生和发展具有促进作用[54]。除此之外,KDM1A介导的维甲酸相关孤儿受体α2 (Retinoic acid-related orphan receptor α2,RORα2)的转录活化对于人类乳腺癌细胞的侵袭有促进作用[55]。作为多种肿瘤转移的抑制因子,乳腺癌转移抑制基因1 (Breast carcinoma metastasis suppressor gene 1,BRMS1)在乳腺癌中通过与KDM1A/CoREST复合物相连接,对乳腺癌细胞进行转录调控进而发挥抑癌作用[73]。另一项研究指出同源异性基因SIX3 (Sine oculis homebox3,SIX3)通过与KDM1A以及转移相关蛋白3 (Metastasis associated protein 3,MAT3)直接作用,特异性募集KDM1A/NURD (MTA3)复合物后抑制WNT1和FOXC2基因,又因为这两种基因能够促进细胞的增殖和侵袭,因此KDM1A在这条途径中发挥着抑制乳腺癌细胞的增殖和侵袭作用[74],说明KDM1A结合不同表观调节物在乳腺癌中发挥的作用是不同的。除此之外,KDM1A的Ser112位点磷酸化对于KDM1A结合到启动子上并发挥去甲基化功能是必需的。体内研究指出,敲除KDM1A损伤了裸鼠体内乳腺癌细胞的转移能力,过表达KDM1A或KDM1A S112D突变体(模拟磷酸化)能够增强癌细胞转移能力,然而KDM1A S112A突变体(模拟非磷酸化)对于癌细胞的转移能力没有影响[75]。说明KDM1A在乳腺癌中的作用与其自身的转录后修饰也有关系。
综合以上结果,KDM1A在乳腺癌中的作用是由不同的表观调节物决定,且KDM1A的作用效应受到明显的转录后修饰,但KDM1A在乳腺癌中的具体机制尚不清楚。
2.3 KDM1A与肺癌在全世界范围内,肺癌(Lung cancer)是人类发病率和致死率最高的癌症[76]。KDM1A在肺癌中,尤其是非小细胞肺癌(Non-small cell lung cancer,NSCLC)中通过促进癌细胞增殖和迁移发挥着致癌效应。在NSCLC细胞系及组织中,KDM1A的mRNA及蛋白质表达水平均普遍高于正常组织,进一步研究发现,KDM1A能够抑制TIMP3基因表达从而促进NSCLC细胞的侵袭[29]。在具有代表性的NSCLC细胞系A549、PC9等中抑制KDM1A的表达,引起细胞周期阻滞在G0/G1期,诱导肺癌细胞生长停滞[72]。对一组小细胞肺癌细胞系(Small cell lung cancer,SCLC)神经内分泌标记基因表达的调查表明,抑制KDM1A后,许多与细胞分化和生长相关的基因的表达发生改变[77]。通过对KDM1A蛋白质定位进行分析,发现这些基因大多数都与KDM1A强烈结合,提示了KDM1A在SCLC细胞系分化中的重要作用[77]。近年来的研究指出Rest作为肿瘤抑制因子[78],在乳腺癌以及SCLC中处于失活状态,通过RNA探针技术发现,敲除KDM1A后,Rest显著上调,提示Rest可能为KDM1A的下游靶基因。后续实验进一步证明了KDM1A能够通过转录抑制Rest从而促进SCLC的发生[52]。
KDM1A的小分子抑制剂如GSK1-LSD1、GSK2879552正在进行肺癌的一期临床试验[79]。研究显示,对同一种KDM1A抑制剂肺癌细胞的敏感性也受到多种因素的影响,具有复杂性。KDM1A抑制剂2-PCPA能够减少NSCLC细胞的增殖、迁移和侵袭,具有EGFR突变的PC9细胞系经2-PCPA治疗后其细胞系的增殖、迁移、侵袭能力比具有Kras突变的A549细胞系有所下降,且具有EGFR突变的PC9细胞系比具有Kras突变的A549的KDM1A的表达量更高。提示在NSCLC细胞中,EGFR突变比Kras突变对于KDM1A抑制剂更敏感[29]。肺癌细胞对于KDM1A抑制剂的敏感性的复杂性不仅于此,对SCLC基因组分析结果揭示了敏感株和耐药株在DNA甲基化模式上存在差异,指出在SCLC细胞株中以及原发性SCLC标本中DNA甲基化模式都能作为KDM1A抑制剂敏感性的生物标记物,进一步的研究指出只有具有敏感性相关的DNA甲基化特征的人源化移植瘤(Patient-derived xenografts,PDX)模型对KDM1A抑制剂具有敏感性[77]。
综合以上研究结果,KDM1A与肺癌的发生发展密切相关,尤其是在调节与细胞生长、分化相关基因方面发挥着重要作用。KDM1A作为治疗肺癌的靶点,其抑制剂作为抗癌药物受到广泛的关注,但不同类型的肺癌细胞对于KDM1A抑制剂的敏感性不同,可能与肺癌细胞中的DNA甲基化程度相关。但关于肺癌细胞对抗癌药物敏感性研究不够深入,需进一步研究。
2.4 KDM1A与肝癌据预测,肝癌(Liver cancer)将成为全球第六大最常见癌症和第四大癌症死亡原因,每年约新增841 000例,死亡782 000人[61]。研究显示,KDM1A与肝癌发生密切相关。临床数据显示,KDM1A过表达更容易形成肿瘤,KDM1A在肝癌组织和肝癌干细胞(Cancer stem-like cells,CSCs)中表达量较高且与肝细胞癌(Hepatocellular carcinoma,HCC)病人的不良预后相关[80]。免疫染色显示,在较高的肿瘤分期(T3–T4)和肿瘤分级(G3)中,KDM1A阳性细胞多于低肿瘤期(T1–T2)和肿瘤分级(G1–G2),表明KDM1A与HCC分化程度密切相关[81]。KDM1A的高乙酰化水平能通过蛋白小体途径抑制KDM1A的表达量及酶活性,进而抑制肝癌CSCs的自我更新和致癌性[80]。在人类肝癌细胞HepG-2中,抑制KDM1A的活性,能够降低葡萄糖的摄取和糖酵解的活性,增加线粒体代谢相关基因启动子上H3K4的甲基化并激活相关基因。提示KDM1A能通过去甲基化H3K4抑制线粒体代谢相关基因的表达[48]。以上研究结果表明KDM1A对肝癌的发生发展具有促进作用。
不仅如此,KDM1A与肝癌的病理发生也密切相关。KDM1A的活性对于经长期索拉菲尼(Sorafenib)治疗的癌细胞出现CSCs特征是必需的。使用KDM1A抑制剂Pargyline和GSK2879552能够显著抑制耐索拉菲尼HCC细胞的干细胞特征,比如说降低干细胞标记物Lgr5、Sox9、Nanog和CD90的mRNA水平,提高分化标记物Alb和Hnf4的mRNA水平,然而对肝脏干细胞/原代细胞标记物CK19的含量没有影响,更重要的是,KDM1A抑制剂能够导致耐索拉菲尼的HCC细胞重新获得对索拉菲尼的敏感性,从而提高索拉菲尼对肝癌的疗效。有研究指出,在PLC肝癌细胞系中的耐索拉菲尼细胞亚系中,KDM1A抑制剂降低β-catenin信号通路的靶基因c-Myc和Cyclin D1的mRNA水平[56]。在miR-302介导的诱导性多能干细胞(Induced pluripotent stem cell,iPSC)重组HCC细胞中,通过抑制KDM1A能够提高HCC对药物的敏感性,同时造成H3K4甲基化程度提高和c-Myc表达抑制[82]。
以上结果表明,KDM1A与肝癌发生发展密切相关。KDM1A作为促癌基因的作用发挥着促进肝癌发生、诱导肝癌CSCs特征并获得耐药性的作用,使得肝癌的治疗变得困难。目前关于KDM1A抑制剂能够提高HCC的药物敏感性的研究取得了一定的进步,但因为肝癌的异质性较强,且KDM1A抑制剂种类繁多,因此进一步明确KDM1A抑制剂对于肝癌的抑制效应并研究其内在的分子机制将是重点方向之一。
2.5 KDM1A与急性髓系白血病急性髓系白血病(Acute myeloid leukemia,AML)作为最常见的血液系统恶性肿瘤,呈高度异质性,其主要表现为髓系白细胞的异常增生以及正常造血细胞的抑制[83]。在血液肿瘤方面,约有60%的AML患者中KDM1A过表达[84]。在造血过程中,KDM1A与混合谱系白血病(Mixed-lineage leukemia,MLL)基因在细胞的分化过程中扮演着重要角色[85]。动物实验显示,在人类MLL-AF9白血病的小鼠模型中,KDM1A能够诱导白血病干细胞(Leukemia stem cell,LSC)潜能,KDM1A在MLL-AF9结合的基因组位点上发挥作用,维持相关致癌程序的表达,从而阻止分化和凋亡[26]。利用sh-RNA抑制KDM1A的表达引起髓系白血病标记基因(CD11b/ITGM和CD86)表达上升,造成细胞增殖抑制以及人类AML细胞克隆能力的下降。同样地,体内抑制KDM1A也会阻断肿瘤的生长,并诱导CD11b和CB86的显著上升[86]。
在临床上,表观调控子的反复突变及异常表达给AML的治疗造成了极大的困扰。靶向治疗主要针对于抑制染色体修饰酶类如KDM1A以及组蛋白甲基化转移酶DOT1L等,其中靶向治疗药物的共同特点是能够诱导髓系细胞分化,解除AML中存在的分化阻滞问题。在用靶向KDM1A和DOT1L并诱导分化的药物处理MLL-AF9驱动的小鼠白血病和MLL重排的PDX的过程中,对染色质动力学进行了比较,有趣的是,虽然KDM1A抑制剂导致了染色质可达性的全面提高,但是用DOT1L抑制剂EPZ4777处理反而导致相反的表型。此外,控制分化的两个转录因子(C/EBPα和PU.1)的下调,使得AML获得对KDM1A抑制剂的耐药性,提示使用KDM1A的药物抑制剂可能是克服AML分化障碍的独特途径[87]。Zeste基因增强子同源物2 (Enhancer of zeste homolog 2,EZH2)是PRC2的催化亚基。EZH2作为组蛋白甲基转移酶(Histone methyltransferase,KMT)催化H3K27的甲基化与作为去甲基化酶的KDM1A具有相反的功能。在AML患者中,EZH2和KDM1A表达量与正常样本相比较均是上调,联合抑制KDM1A和EZH2不仅能下调抗凋亡蛋白Bcl-2 mRNA和蛋白水平,还能上调促凋亡蛋白Bax和色素蛋白C的mRNA和蛋白质水平进而诱导凋亡[88]。
以上结果表明,KDM1A含量与AML的发生具有相关性,尤其是在诱导LSC潜能方面。单独使用KDM1A抑制剂治疗AML,AML易获得耐药性,抗癌效果不明显。联合抑制KDM1A与其他组蛋白甲基化调控子治疗AML将具有巨大的潜能。有研究指出,SP2509作为一种有效的KDM1A抑制剂与广泛的HDAC抑制剂帕比司他(Panobinostat)联合使用比两种药物单独使用更容易提高植入了人类AML细胞的小鼠的存活率,且没有显示出任何毒性[89]。
2.6 KDM1A与口腔癌口腔癌(Oral cancer)是在发展中国家中最常见的癌症,口腔癌组织中的KDM1A表达水平高于正常口腔组织[76]。临床组织芯片显示,KDM1A蛋白在恶性口腔鳞状细胞癌(Oral squamous cell carcinoma,OSCC)中表达量上升,其表达量与肿瘤分级[79],不良预后[90]以及肿瘤的侵袭性和整体生存期的缩短密切相关[91]。在原位口腔癌小鼠模型中,侵袭性HSC-3细胞系中过表达KDM1A能够促进其转移,当敲除KDM1A基因时,肿瘤的转移受到抑制,表明在OSCC的转移过程中KDM1A发挥着重要调控作用。当用特异性的小分子抑制物如GSK-LSD1抑制KDM1A时能够延缓疾病的进程,下调EGF (Epidermal growth factor,EGF)信号通路,减少鳞状细胞癌PDX模型中的促癌基因如MMP13、CTGF、LOXL4以及波形蛋白的表达,增加原先存在的E-钙粘蛋白的表达。此外,基因富集分析指出GSK-LSD1抑制c-Mcy、β-catenin和YAP构成的促癌基因转录网络,同时能够增加p53的表达量并诱导凋亡[56]。利用药物或基因方法抑制KDM1A的表达,能够抑制细胞增殖,削弱转移和侵袭能力,损伤OSCC的化学抗药性以及干细胞稳定性。在动物实验中,腹腔注射KDM1A抑制剂能够抑制OSCC移植瘤的过度生长[92]。褪黑素作为一种内源性分子能够抵抗多种癌症,在过表达KDM1A的口腔癌PDX模型中,褪黑素能够以时间/剂量依赖的方式降低细胞的增殖,同时伴随着褪黑素介导的抑制KDM1A的作用,提示褪黑素在口腔癌中能通过降低KDM1A含量达到抗癌效果[86]。KDM1A作为治疗OSCC的靶点将会受到越来越多的关注。
2.7 KDM1A与胶质瘤胶质瘤作为最常见的颅内恶性肿瘤,其发病率约占全部颅内肿瘤的一半,患者1年生存率约为30%,5年生存率下降到不足5%[92]。有研究者发现,先通过CK1α引起KDM1A的第687位丝氨酸的磷酸化后,GSK3β才能磷酸化KDM1A的第683位丝氨酸位点,KDM1A的Ser683位点磷酸化后,通过结合USP22引起去泛素化,稳定KDM1A。依赖于GSK-3β和USP22的稳定的KDM1A对于组蛋白H3K4的去甲基化是必需的,H3K4的去甲基化抑制BMP2,细胞周期蛋白依赖的激酶抑制剂(Cyclin-dependent kinase inhibitor 1A,CDKN1)以及GATA结合蛋白6 (GATA-binding protein 6,GATA6)的转录,导致肿瘤干细胞的自我更新和胶质瘤母细胞(Glioblastoma,GBM)的发生。在小鼠模型中,颅内注射GSC11 (Glioma stem cells 11,GSC11)后均出现特征明显的胶质母瘤,当利用sh-KDM1A敲除后的GSC11以及sh-USP22敲除的GSC11颅内注射小鼠后发现能够抑制肿瘤的形成。有趣的是,肿瘤发生的抑制现象能被KDM1A的表达所逆转,提示KDM1A和USP22与GCSs的发生密切相关。在人类GBM样本中,也发现KDM1A水平与GSK3β和USP22水平密切相关。在小鼠体内,利用GSK3β的抑制剂硫代谷胱甘肽下调KDM1A引起移植瘤对化学治疗的敏感性和提高生存率[25]。还有研究指出在A172和T98G细胞系中,去泛素化酶USP7能够维持KDM1A稳定性,USP7-KDM1A通过抑制p53信号通路引起G0/G1的阻滞,促进肿瘤的生成和侵袭[93]。以上研究表明,不同的去泛素化酶均能维持KDM1A的稳定性,稳定的KDM1A通过介导不同的下游途径在GBM的发生发展中发挥着重要作用。KDM1A的敲除实验证明,敲除KDM1A能够增加骨髓细胞癌病毒基因同源物(Avian myelocytomatosis viral oncogene homolog,MYC)位点上H3K4的三甲基化,进而提高MYC的表达。MYC表达的提高又反过来调控少突胶质系转录因子2 (Oligodendrocyte lineage,OLIG2)、SOX2以及POU3F2,这些转录因子对于GBM重构成具有干细胞特性的状态是必需的[94]。
已有研究表明联合KDM1A抑制剂TCP和HDAC1抑制剂伏立诺他(Vorinostat)或恩替诺特(Entinostat)处理胶质瘤干细胞(Glioma stem cell,GSCs)后,其存活率降低到30%左右远低于药物单独处理后的存活率,表明GSCs细胞对联合用药较为敏感。为了探究其分子机制,测定了敲除KDM1A后的LN-18细胞系联合/不联合伏立诺他处理的全基因表达改变情况,发现TP53以及家族成员TP73在联合处理下,其表达量显著下调。在原位胶质瘤异种移植瘤模型中,联合伏立诺他(Vorinostat)和TCP两种药物处理该模型,能够引起p53和TAp73表达比单独使用变化更显著,表明了联合用药的高效性。在接下来的研究中应该进一步提高特异性的KDM1A抑制剂透过血脑屏障的能力并在已经进行的实验阶段一期中影响肿瘤的生理学特征进而提高KDM1A抑制剂治疗GBM的特异性[95]。
3 总结与展望KDM1A作为第一个被发现的去甲基化酶,被证实与多种疾病相关,尤其与多种肿瘤的发生发展密切相关。目前的研究证实了KDM1A在不同类型的肿瘤中发挥着不同的作用,但大体上KDM1A是作为促癌基因存在的。特别是KDM1A作为促进糖酵解的重要因子,在维持肿瘤的沃伯格效应中扮演着重要的角色。KDM1A独特和复杂的结构使其能与一系列的蛋白质复合物结合,抑制或激活基因的表达。如图 2所示,KDM1A与不同的启动子、转录因子以及蛋白质复合物相互作用,能够作为重要的调节子调控细胞内的癌症发生过程。
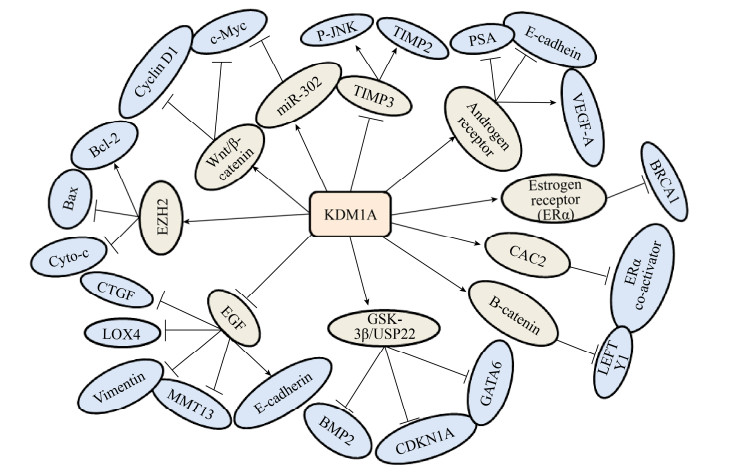 |
| 图 2 KDM1A在肿瘤中的作用机制 Fig. 2 Diagram of the mechanisms of KDM1A in tumor. |
| 图选项 |
目前对于KDM1A在肿瘤中的作用机制研究不够深入。首先,KDM1A能与一系列不同的蛋白质复合物、Non-coding RNAs、Micro-RNAs等结合,另外,KDM1A作用的上下游信号通路调节复杂,需要我们对每种信号通路的调节都作进一步研究。除此之外,KDM1A能够促进糖酵解,进而维持肿瘤细胞的沃泊格效应,使得iPSCs重新编程,从而维持肿瘤细胞的恶性增殖[96]。KDM1A在糖代谢上的作用越来越受到研究人员的关注,但对其机制的了解尚未十分深入。最新研究表明KDM1A能够通过调控关键酶类已糖激酶2 (Hexokinases 2,HK2)的酶活力,促进沃泊格效应。ORY-1001抑制KDM1A,引起HK2酶活力的降低,从而逆转沃泊格效应,抑制癌细胞的增殖[97]。除此之外,KDM1A对肿瘤氨基酸和脂肪酸代谢的影响也将成为重点关注领域。虽然有针对KDM1A抑制剂抑癌效应的研究,但其效应有待进一步研究。总而言之,KDM1A在肿瘤的发生发展中扮演着重要角色,作为肿瘤临床诊断和治疗的重要潜在靶点还需进一步的研究。
参考文献
| [1] | Fu WL, Huang Q. Cancer epigenetics. Clin Biochem Lab Med Foreign Med Sci, 2004, 25(4): 289-290 (in Chinese). 府伟灵, 黄庆. 肿瘤表观遗传学. 国外医学临床生物化学与检验学分册, 2004, 25(4): 289-290. |
| [2] | Barski A, Cuddapah S, Cui KR, et al. High- resolution profiling of histone methylations in the human genome. Cell, 2007, 129(4): 823-837. DOI:10.1016/j.cell.2007.05.009 |
| [3] | Shi YJ, Lan F, Matson C, et al. Histone demethylation mediated by the nuclear amine oxidase homolog LSD1. Cell, 2004, 119(7): 941-953. DOI:10.1016/j.cell.2004.12.012 |
| [4] | Huang J, Sengupta R, Espejo AB, et al. p53 is regulated by the lysine demethylase LSD1. Nature, 2007, 449(7158): 105-108. DOI:10.1038/nature06092 |
| [5] | Wang J, Hevi S, Kurash JK, et al. The lysine demethylase LSD1 (KDM1) is required for maintenance of global DNA methylation. Nat Genet, 2009, 41(1): 125-129. DOI:10.1038/ng.268 |
| [6] | Karytinos A, Forneris F, Profumo A, et al. A novel mammalian flavin-dependent histone demethylase. J Biol Chem, 2009, 284(26): 17775-17782. DOI:10.1074/jbc.M109.003087 |
| [7] | Klose RJ, Yamane K, Bae Y, et al. The transcriptional repressor JHDM3A demethylates trimethyl histone H3 lysine 9 and lysine 36. Nature, 2006, 442(7100): 312-316. DOI:10.1038/nature04853 |
| [8] | Cloos PAC, Christensen J, Agger K, et al. The putative oncogene GASC1 demethylates tri- and dimethylated lysine 9 on histone H3. Nature, 2006, 442(7100): 307-311. DOI:10.1038/nature04837 |
| [9] | Whetstine JR, Nottke A, Lan F, et al. Reversal of histone lysine trimethylation by the JMJD2 family of histone demethylases. Cell, 2006, 125(3): 467-481. |
| [10] | Fodor BD, Kubicek S, Yonezawa M, et al. Jmjd2b antagonizes H3K9 trimethylation at pericentric heterochromatin in mammalian cells. Genes Dev, 2006, 20(12): 1557-1562. DOI:10.1101/gad.388206 |
| [11] | Yamane K, Toumazou C, Tsukada YI, et al. JHDM2A, a JmjC-containing H3K9 demethylase, facilitates transcription activation by androgen receptor. Cell, 2006, 125(3): 483-495. |
| [12] | Wu BH, Wang YP, Han ZG. Research progress of role of JmjC family in tumorigenesis. J Shanghai Jiaotong Univ: Med Sci, 2013, 33(5): 676-680 (in Chinese). 吴炳浩, 王玉平, 韩泽广. JmjC家族在肿瘤发生中作用的研究进展. 上海交通大学学报:医学版, 2013, 33(5): 676-680. |
| [13] | Ma JP, Han J, Zhang JL. Research progress of histone demethylase. Guide China Med, 2017, 15(12): 46-47 (in Chinese). 马佳萍, 韩剑, 张金龙. 组蛋白去甲基化酶的研究进展. 中国医药指南, 2017, 15(12): 46-47. |
| [14] | Stavropoulos P, BlobelG, HoelzA. Crystal structure and mechanism of human lysine-specific demethylase-1. Nat StructMol Biol, 2006, 13(7): 626-632. DOI:10.1038/nsmb1113 |
| [15] | Shao GB, Ding HM, Gong AH, et al. Inheritance of histone H3 methylation in reprogramming of somatic nuclei following nuclear transfer. J Reprod Dev, 2008, 54(3): 233-238. DOI:10.1262/jrd.19173 |
| [16] | Katz DJ, Edwards TM, Reinke V, et al. A C. elegans LSD1 demethylase contributes to germline immortality by reprogramming epigenetic memory. Cell, 2009, 137(2): 308-320. DOI:10.1016/j.cell.2009.02.015 |
| [17] | Zhang J, Tan L, Hou JY, et al. Trans-2- phenylcyclopropylamine induces nerve cells apoptosis in zebrafish mediated by depression of LSD1 activity. Brain Res Bull, 2009, 80(1/2): 79-84. |
| [18] | Wang LJ, Pei ZY, Tian YC, et al. OsLSD1, a rice zinc finger protein, regulates programmed cell death and callus differentiation. Mol Plant-Microbe Int, 2005, 18(5): 375-384. |
| [19] | Hiramoto M, Udagawa H, Ishibashi N, et al. A type 2 diabetes-associated SNP in KCNQ1 (rs163184) modulates the binding activity of the locus for Sp3 and Lsd1/Kdm1a, potentially affecting CDKN1C expression. Int J Mol Med, 2018, 41(2): 717-728. |
| [20] | Shan JY, Zhao BB, Shan Z, et al. Histone demethylase LSD1 restricts influenza A virus infection by erasing IFITM3-K88 monomethylation. PLoS Pathog, 2017, 13(12): e1006773. DOI:10.1371/journal.ppat.1006773 |
| [21] | Chen YC, Chao TY, Leung SY, et al. Histone H3K14 hypoacetylation and H3K27 hypermethylation along with HDAC1 up-regulation and KDM6B down- regulation are associated with active pulmonary tuberculosis disease. Am J Trans Res, 2017, 9(4): 1943-1955. |
| [22] | Togasaki E, Takeda J, Yoshida K, et al. Frequent somatic mutations in epigenetic regulators in newly diagnosed chronic myeloid leukemia. Blood Cancer J, 2017, 7(4): e559. DOI:10.1038/bcj.2017.36 |
| [23] | Maes T, Mascaró C, Ortega A, et al. KDM1 histone lysine demethylases as targets for treatments of oncological and neurodegenerative disease. Epigenomics, 2015, 7(4): 609-626. DOI:10.2217/epi.15.9 |
| [24] | Williams JS, Chamarthi B, Goodarzi MO, et al. Lysine-specific demethylase 1: an epigenetic regulator of salt-sensitive hypertension. Am J Hypertens, 2012, 25(7): 812-817. DOI:10.1038/ajh.2012.43 |
| [25] | Zhou AD, Lin KY, Zhang SC, et al. Nuclear GSK3β promotes tumorigenesis by phosphorylating KDM1A and inducing its deubiquitylation by USP22. Nat Cell Biol, 2016, 18(9): 954-966. DOI:10.1038/ncb3396 |
| [26] | Harris WJ, Huang X, Lynch JT, et al. The histone demethylase KDM1A sustains the oncogenic potential of MLL-AF9 leukemia stem cells. Cancer Cell, 2012, 21(4): 473-487. DOI:10.1016/j.ccr.2012.03.014 |
| [27] | Wang Y, Zhang H, Chen YP, et al. LSD1 is a subunit of the NuRD complex and targets the metastasis programs in breast cancer. Cell, 2009, 138(4): 660-672. DOI:10.1016/j.cell.2009.05.050 |
| [28] | Hayami S, Kelly JD, Cho HS, et al. Overexpression of LSD1 contributes to human carcinogenesis through chromatin regulation in various cancers. Int J Cancer, 2011, 128(3): 574-586. DOI:10.1002/ijc.25349 |
| [29] | Kong LZ, Zhang P, Li W, et al. KDM1A promotes tumor cell invasion by silencing TIMP3 in non-small cell lung cancer cells. Oncotarget, 2016, 7(19): 27959-27974. |
| [30] | Zhou XF, Ma H. Evolutionary history of histone demethylase families: distinct evolutionary patterns suggest functional divergence. BMC Evol Biol, 2008, 8: 294. DOI:10.1186/1471-2148-8-294 |
| [31] | Aravind L, Iyer LM. The SWIRM domain: a conserved module found in chromosomal proteins points to novel chromatin-modifying activities. Genome Biol, 2002, 3(8): research0039.1. |
| [32] | Chen Y, Yang YT, Wang F, et al. Crystal structure of human histone lysine-specific demethylase 1 (LSD1). Proc Natl Acad Sci USA, 2006, 103(38): 13956-13961. DOI:10.1073/pnas.0606381103 |
| [33] | Pilotto S, Speranzini V, Tortorici M, et al. Interplay among nucleosomal DNA, histone tails, and corepressor CoREST underlies LSD1-mediated H3 demethylation. Proc Natl Acad Sci USA, 2015, 112(9): 2752-2757. DOI:10.1073/pnas.1419468112 |
| [34] | Ismail T, Lee HK, Kim C, et al. KDM1A microenvironment, its oncogenic potential, and therapeutic significance. Epigenet Chromatin, 2018, 11(1): 33. |
| [35] | Lee MG, Wynder C, Cooch N, et al. An essential role for CoREST in nucleosomal histone 3 lysine 4 demethylation. Nature, 2005, 437(7057): 432-435. DOI:10.1038/nature04021 |
| [36] | Tsai MC, Manor O, Wan Y, et al. Long noncoding RNA as modular scaffold of histone modification complexes. Science, 2010, 329(5992): 689-693. DOI:10.1126/science.1192002 |
| [37] | Li YH, Wang Z, S hi, et al. HBXIP and LSD1 Scaffolded by lncRNA hotair mediate transcriptional activation by c-Myc. Cancer Res, 2015, 76(2): 293-304. |
| [38] | Sun M, Nie FQ, Wang YF, et al. LncRNA HOXA11-AS promotes proliferation and invasion of gastric cancer by scaffolding the chromatin modification factors PRC2, LSD1 and DNMT1. Cancer Res, 2016, 76(21): 6299-6310. DOI:10.1158/0008-5472.CAN-16-0356 |
| [39] | Xu TP, Wang WY, Ma P, et al. Upregulation of the long noncoding RNA FOXD2-AS1 promotes carcinogenesis by epigenetically silencing EphB3 through EZH2 and LSD1, and predicts poor prognosis in gastric cancer. Oncogene, 2018, 37(36): 5020. DOI:10.1038/s41388-018-0308-y |
| [40] | Hakimi MA, Bochar DA, Chenoweth J, et al. A core-BRAF35 complex containing histone deacetylase mediates repression of neuronal-specific genes. Proc Natl Acad of Sci USA, 2002, 99(11): 7420-7425. DOI:10.1073/pnas.112008599 |
| [41] | Shi YJ, Sawada JI, Sui GC, et al. Coordinated histone modifications mediated by a CtBP co-repressor complex. Nature, 2003, 422(6933): 735-738. DOI:10.1038/nature01550 |
| [42] | Perillo B, Ombra MN, Bertoni A, et al. DNA oxidation as triggered by H3K9me2 demethylation drives estrogen-induced gene expression. Science, 2008, 319(5860): 202-206. DOI:10.1126/science.1147674 |
| [43] | Metzger E, Wissmann M, Yin N, et al. LSD1 demethylates repressive histone marks to promote androgen-receptor-dependent transcription. Nature, 2005, 437(7057): 436-439. DOI:10.1038/nature04020 |
| [44] | Magerl C, Ellinger J, Braunschweig T, et al. H3K4 dimethylation in hepatocellular carcinoma is rare compared with other hepatobiliary and gastrointestinal carcinomas and correlates with expression of the methylase Ash2 and the demethylase LSD1. Human Pathol, 2010, 41(2): 181-189. DOI:10.1016/j.humpath.2009.08.007 |
| [45] | Duteil D, Metzger E, Willmann D, et al. LSD1 promotes oxidative metabolism of white adipose tissue. Nat Commun, 2014, 5: 4093. DOI:10.1038/ncomms5093 |
| [46] | Warburg O. On the origin of cancer cells. Science, 1956, 123(3191): 309-314. DOI:10.1126/science.123.3191.309 |
| [47] | Qin Y, Zhu WW, Xu WY, et al. LSD1 sustains pancreatic cancer growth via maintaining HIF1α- dependent glycolytic process. Cancer Lett, 2014, 347(2): 225-232. DOI:10.1016/j.canlet.2014.02.013 |
| [48] | Sakamoto A, Hino S, Nagaoka K, et al. Lysine demethylase LSD1 coordinates glycolytic and mitochondrial metabolism in hepatocellular carcinoma cells. Cancer Res, 2015, 75(7): 1445-1456. DOI:10.1158/0008-5472.CAN-14-1560 |
| [49] | Kosumi K, Baba Y, Sakamoto A, et al. Lysine- specific demethylase-1 contributes to malignant behavior by regulation of invasive activity and metabolic shift in esophageal cancer. Int JCancer, 2016, 138(2): 428-439. DOI:10.1002/ijc.29714 |
| [50] | Yang C, Wang WH, Liang JX, et al. A rhodium(Ⅲ)-based inhibitor of lysine-specific histone demethylase 1 as an epigenetic modulator in prostate cancer cells. J Med Chem, 2017, 60(6): 2597-2603. DOI:10.1021/acs.jmedchem.7b00133 |
| [51] | Kashyap V, Ahmad S, Nilsson EM, et al. The lysine specific demethylase-1 (LSD1/KDM1A) regulates VEGF-A expression in prostate cancer. Mol Oncol, 2013, 7(3): 555-666. |
| [52] | Jin YJ, Ma D, Gramyk T, et al. Kdm1a promotes SCLC progression by transcriptionally silencing the tumor suppressor Rest. Biochem Biophys Res Commun, 2019, 515(1): 214-221. DOI:10.1016/j.bbrc.2019.05.118 |
| [53] | Huang ZB, Li SZ, Song W, et al. Lysine-specific demethylase 1 (LSD1/KDM1A) contributes to colorectal tumorigenesis via activation of the Wnt/β-Catenin pathway by down-regulating dickkopf-1 (DKK1). PLoS ONE, 2013, 8(7): e70077. DOI:10.1371/journal.pone.0070077 |
| [54] | Cao C, Vasilatos SN, Bhargava R, et al. Functional interaction of histone deacetylase 5 (HDAC5) and lysine-specific demethylase 1 (LSD1) promotes breast cancer progression. Oncogene, 2017, 36(1): 133-145. DOI:10.1038/onc.2016.186 |
| [55] | Kim K, Lee JM, Yu YS, et al. RORα2 requires LSD1 to enhance tumor progression in breast cancer. Sci Rep, 2017, 7(1): 11994. DOI:10.1038/s41598-017-12344-0 |
| [56] | Huang MX, Chen C, Geng J, et al. Targeting KDM1A attenuates Wnt/β-catenin signaling pathway to eliminate sorafenib-resistant stem-like cells in hepatocellular carcinoma. Cancer Lett, 2017, 398: 12-21. DOI:10.1016/j.canlet.2017.03.038 |
| [57] | Cai LS, Chen QX, Fang SY, et al. MicroRNA-329 inhibits cell proliferation and tumor growth while facilitates apoptosis via negative regulation of KDM1A in gastric cancer. J Cell Biochem, 2018, 119(4): 3338-3351. DOI:10.1002/jcb.26497 |
| [58] | Ambrosio S, Saccà CD, Amente S, et al. Lysine- specific demethylase LSD1 regulates autophagy in neuroblastoma through SESN2-dependent pathway. Oncogene, 2017, 36: 6701-6711. DOI:10.1038/onc.2017.267 |
| [59] | Chao A, Lin CY, Chao AN, et al. Lysine-specific demethylase 1 (LSD1) destabilizes p62 and inhibits autophagy in gynecologic malignancies. Oncotarget, 2017, 8(43): 74434-74450. |
| [60] | Hino S, Kohrogi K, Nakao M. Histone demethylase LSD1 controls the phenotypic plasticity of cancer cells. Cancer Sci, 2016, 107(9): 1187-1192. DOI:10.1111/cas.13004 |
| [61] | Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA: Cancer JClin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [62] | Sehrawat A, Gao LN, Wang YL, et al. LSD1 activates a lethal prostate cancer gene network independently of its demethylase function. Proc Natl Acad Sci USA, 2018, 115(18): E4179-E4188. DOI:10.1073/pnas.1719168115 |
| [63] | Kahl P, Gullotti L, Heukamp LC, et al. Androgen receptor coactivators lysine-specific histone demethylase 1 and four and a half LIM domain protein 2 predict risk of prostate cancer recurrence. Cancer Res, 2006, 66(23): 11341-11347. DOI:10.1158/0008-5472.CAN-06-1570 |
| [64] | Wang M, Liu XH, Jiang GJ, et al. Relationship between LSD1 expression and E-cadherin expression in prostate cancer. Int UrolNephrol, 2015, 47(3): 485-490. |
| [65] | Oreland L, Kinemuchi H, Yoo BY. The mechanism of action of the monoamine oxidase inhibitor pargyline. Life Sci, 1973, 13(11): 1533-1541. DOI:10.1016/0024-3205(73)90142-2 |
| [66] | Kauffman EC, Robinson BD, Downes MJ, et al. Role of androgen receptor and associated lysine- demethylase coregulators, LSD1 and JMJD2A, in localized and advanced human bladder cancer. Mol Carcinog, 2011, 50(12): 931-944. DOI:10.1002/mc.20758 |
| [67] | Schmidt DMZ, McCafferty DG. trans-2- phenylcyclopropylamine is a mechanism-based inactivator of the histone demethylase LSD1. Biochemistry, 2007, 46(14): 4408-4416. DOI:10.1021/bi0618621 |
| [68] | Wang M, Liu XH, Guo J, et al. Inhibition of LSD1 by Pargyline inhibited process of EMT and delayed progression of prostate cancer in vivo. Biochem Biophys Res Commun, 2015, 467(2): 310-315. DOI:10.1016/j.bbrc.2015.09.164 |
| [69] | Zhang K, Fu G, Pan GZ, et al. Demethylzeylasteral inhibits glioma growth by regulating the miR-30e-5p/MYBL2 axis. Cell Death Dis, 2018, 9(10): 1035. DOI:10.1038/s41419-018-1086-8 |
| [70] | Lee MG, Wynder C, Schmidt DM, et al. Histone H3 lysine 4 demethylation is a target of nonselective antidepressive medications. Chem Biol, 2006, 13(6): 563-567. DOI:10.1016/j.chembiol.2006.05.004 |
| [71] | Lim S, Janzer A, Becker A, et al. Lysine-specific demethylase 1 (LSD1) is highly expressed in ER-negative breast cancers and a biomarker predicting aggressive biology. Carcinogenesis, 2010, 31(3): 512-520. DOI:10.1093/carcin/bgp324 |
| [72] | Macheleidt IF, Dalvi PS, Lim SY, et al. Preclinical studies reveal that LSD1 inhibition results in tumor growth arrest in lung adenocarcinoma independently of driver mutations. Mol Oncol, 2018, 12(11): 1965-1979. DOI:10.1002/1878-0261.12382 |
| [73] | Qiu RF, Shi H, Wang S, et al. BRMS1 coordinates with LSD1 and suppresses breast cancer cell metastasis. Am J Cancer Res, 2018, 8(10): 2030-2045. |
| [74] | Zheng Y, Zeng Y, Qiu RF, et al. The homeotic protein SIX3 suppresses carcinogenesis and metastasis through recruiting the LSD1/NuRD (MTA3) complex. Theranostics, 2018, 8(4): 972-989. DOI:10.7150/thno.22328 |
| [75] | Feng JX, Xu GY, Liu JW, et al. Phosphorylation of LSD1 at Ser112 is crucial for its function in induction of EMT and metastasis in breast cancer. Breast Cancer Res Treat, 2016, 159(3): 443-456. DOI:10.1007/s10549-016-3959-9 |
| [76] | Nagasawa S, Sedukhina AS, Nakagawa Y, et al. LSD1 overexpression is associated with poor prognosis in basal-like breast cancer, and sensitivity to PARP inhibition. PLoS ONE, 2015, 10(2): e0118002. DOI:10.1371/journal.pone.0118002 |
| [77] | Mohammad HP, Kruger RG. Antitumor activity of LSD1 inhibitors in lung cancer. Mol Cell Oncol, 2016, 3(2): e1117700. DOI:10.1080/23723556.2015.1117700 |
| [78] | Negrini S, Prada I, D'Alessandr R, et al. REST: an oncogene or a tumor suppressor?. Trends Cell Biol, 2013, 23(6): 289-295. DOI:10.1016/j.tcb.2013.01.006 |
| [79] | Alsaqer SF, Tashkandi MM, Kartha VK, et al. Inhibition of LSD1 epigenetically attenuates oral cancer growth and metastasis. Oncotarget, 2017, 8(43): 73372-73386. |
| [80] | Liu CG, Liu LM, Chen XJ, et al. LSD1 stimulates cancer-associated fibroblasts to drive Notch3- dependent self-renewal of liver cancer stem-like cells. Cancer Res, 2018, 78(4): 938-949. DOI:10.1158/0008-5472.CAN-17-1236 |
| [81] | Zhao ZK, Dong P, Gu J, et al. Overexpression of LSD1 in hepatocellular carcinoma: a latent target for the diagnosis and therapy of hepatoma. Tumor Biol, 2013, 34(1): 173-180. |
| [82] | Koga C, Kobayashi S, Nagano H, et al. Reprogramming using microRNA-302 improves drug sensitivity in hepatocellular carcinoma cells. Ann Surg Oncol, 2014, 21(4): 591-600. |
| [83] | Jin J. Targeted therapy of acute myeloid leukemia. J Clin Hematol, 2018, 31(2): 178-180 (in Chinese). 金洁. 急性髓系白血病靶向治疗. 临床血液学杂志, 2018, 31(2): 178-180. |
| [84] | Niebel D, Kirfel J, Janzen V, et al. Lysine-specific demethylase 1 (LSD1) in hematopoietic and lymphoid neoplasms. Blood, 2014, 124(1): 151-152. DOI:10.1182/blood-2014-04-569525 |
| [85] | Vu LP, Luciani L, Nimer SD. Histone-modifying enzymes: their role in the pathogenesis of acute leukemia and their therapeutic potential. Int J Hematol, 2013, 97(2): 198-209. DOI:10.1007/s12185-012-1247-y |
| [86] | Fang JW, Ying HY, Mao T, et al. Upregulation of CD11b and CD86 through LSD1 inhibition promotes myeloid differentiation and suppresses cell proliferation in human monocytic leukemia cells. Oncotarget, 2017, 8(49): 85085-85101. |
| [87] | Cusan M, Cai SF, Mohammad HP, et al. LSD1 inhibition exerts its antileukemic effect by recommissioning PU.1- and C/EBPα-dependent enhancers in AML. Blood, 2018, 131(15): 1730-1742. DOI:10.1182/blood-2017-09-807024 |
| [88] | Wen SJ, Wang JK, Liu PP, et al. Novel combination of histone methylation modulators with therapeutic synergy against acute myeloid leukemia in vitro and in vivo. Cancer Lett, 2018, 413: 35-45. DOI:10.1016/j.canlet.2017.10.015 |
| [89] | Fiskus W, Sharma S, Shah B, et al. Highly effective combination of LSD1 (KDM1A) antagonist and pan-histone deacetylase inhibitor against human AML cells. Leukemia, 2014, 28(11): 2155-2164. DOI:10.1038/leu.2014.119 |
| [90] | Yang CY, Lin CK, Tsao CH, et al. Melatonin exerts anti-oral cancer effect via suppressing LSD1 in patient-derived tumor xenograft models. Oncotarget, 2017, 8(20): 33756-33769. |
| [91] | Wang YL, Zhu YM, Wang Q, et al. The histone demethylase LSD1 is a novel oncogene and therapeutic target in oral cancer. Cancer Lett, 2016, 374(1): 12-21. DOI:10.1016/j.canlet.2016.02.004 |
| [92] | Porter KR, McCarthy BJ, Freels S, et al. Prevalence estimates for primary brain tumors in the United States by age, gender, behavior, and histology. Neuro-Oncol, 2010, 12(6): 520-527. DOI:10.1093/neuonc/nop066 |
| [93] | Yi L, Cui Y, Xu QF, et al. Stabilization of LSD1 by deubiquitinating enzyme USP7 promotes glioblastoma cell tumorigenesis and metastasis through suppression of the p53 signaling pathway. Oncol Rep, 2016, 36(5): 2935-2945. DOI:10.3892/or.2016.5099 |
| [94] | Kozono D, Li J, Nitta M, et al. Dynamic epigenetic regulation of glioblastoma tumorigenicity through LSD1 modulation of MYC expression. Proc Natl Acad Sci USA, 2015, 112(30): E4055-E4064. DOI:10.1073/pnas.1501967112 |
| [95] | Singh MM, Johnson B, Venkatarayan A, et al. Preclinical activity of combined HDAC and KDM1A inhibition in glioblastoma. Neuro-Oncol, 2015, 17(11): 1463-1473. DOI:10.1093/neuonc/nov041 |
| [96] | Sun H, Liang LN, Li Y, et al. Lysine-specific histone demethylase 1 inhibition promotes reprogramming by facilitating the expression of exogenous transcriptional factors and metabolic switch. Sci Rep, 2016, 6: 30903. DOI:10.1038/srep30903 |
| [97] | Shan C, Lgu Z, Li J, et al. ORY-1001 suppresses cell growth and induces apoptosis in lung cancer throuh triggering HK2 mediated Warburg effect. Front Pharmacol, 2018, 9: 1411. DOI:10.3389/fphar.2018.01411 |
