

1. 中国医药工业研究总院 上海医药工业研究院,上海 201203;
2. 复旦大学 药学院,上海 201203;
3. 中国科学院 上海生命科学研究院 植物生理与生态研究所,上海 200032
收稿日期:2020-06-02;接收日期:2020-10-28
基金项目:国家重大新药创制课题(No. 2014ZX09201-001-05), 上海市自然科学基金(No. 15ZR1440300), 上海市产学研合作项目(No. CXY-2013-53)资助
作者简介:胡海峰??博士,博士生导师,研究员,现任中国医药工业研究院院长助理,国药集团健康产业研究院有限公司总经理,上海市领军人才,上海市第十三届政协委员,中国致公党上海市经济与信息化委员会主委等职务。1989年从华东理工大学本科毕业,1998年7月在上海医药工业研究院获得理学博士学位,1999年2月在日本从事日本科技厅特别研究员研究工作,2002年3月从日本回国继续在上海医药工业研究院工作,历任部门副主任、主任、副院长等职务。承担上海市自然科学基金一项、上海市重点攻关项目4项、上海市基础重点项目3项,2011年度被评为上海市优秀技术带头人;“十一五”期间,作为子课题负责人参与3项重大专项项目的研究;“十二五”期间,参与科技部重大仪器专项、微生物药物筛选平台和创新抗耐药抗生素研制及重大品种的综合技术提升等3项重大专项子项目研究。先后担任上海市微生物学会副理事长、《中国医药工业杂志》、《中国抗生素杂志》、《中国天然药物杂志》和《世界临床杂志》编委。承担上海医工院研究生一门专业课教学任务,培养硕士、博士研究生28名,发表论文53篇,申请专利38项,授权专利20项.
摘要:金色链霉菌Streptomyces aureofaciens DM-1是去甲基金霉素的高产菌株。通过Genome Sequencer FLX系统进行测序,得到一条完整的线性基因组序列,长度为6 824 334 bp,GC含量为72.6%。结合软件glimmer 3.02、Genemark和Z-Curve program进行基因预测,最终在其基因组中鉴定出6 431个基因。应用AntiSMASH软件预测其基因组中存在28个次级代谢生物合成基因簇,其中包含了去甲基金霉素生物合成基因簇。其中甲基转移酶CtcK因移码突变提前终止翻译,很可能是去甲基金霉素相对金霉素(CTC)缺失一个甲基的根本原因。研究结果为S. aureofaciens DM-1的功能基因组学和去甲基金霉素高产菌株育种提供了研究基础。
关键词:金色链霉菌DM-1基因组测序去甲基金霉素生物合成基因簇
Genome sequencing of Streptomyces aureofaciens DM-1 and analysis of 6-demethylchlortetracycline biosynthesis gene cluster
Naxin Wu1,2, He Huang3, Taoling Min1, Haifeng Hu1


1. Shanghai Institute of Pharmaceutical Industry, China State Institute of Pharmaceutical Industry, Shanghai 201203, China;
2. School of Pharmacy, Fudan University, Shanghai 201203, China;
3. Institute of Plant Physiology and Ecology, Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences, Shanghai 200032, China
Received: June 2, 2020; Accepted: October 28, 2020
Supported by: State Project For Essential Drug Research and Development (No.2014ZX09201-001-05), Shanghai Municipal Natural Science Foundation (No.15ZR1440300), Shanghai Industry University Research Cooperation Project (No.CXY-2013-53)
Corresponding author: Haifeng Hu. Tel: +86-21-55880207; E-mail: haifenghu88@163.com.
Abstract: Streptomyces aureofaciens DM-1 is a high-yielding 6-demethylchlortetracycline producer. The genome sequencing of DM-1 reveals a linear chromosome containing 6 824 334 bps nucleotides with GC content of 72.6%. In this genome, a total of 6 431 open reading frames were predicted by using glimmer 3.02, Genemark and Z-Curve softwares. Twenty-eight secondary metabolite biosynthetic gene clusters were uncovered by using AntiSMASH gene prediction software, including the complete 6-demethylchlortetracycline biosynthetic gene cluster. A frame-shift mutation in methyltransferase coding region was detected, which may result in the demethylation of chlortetracycline. The complete genome sequence of S. aureofaciens DM-1 provides basic information for functional genomics studies and selection of high-yielding strains for 6-demethylchlortetracycline.
Keywords: Streptomyces aureofaciens DM-1genome sequencing6-demethylchlortetracyclinebiosynthetic gene cluster
去甲基金霉素(6-demethylchlortetracycline)是金色链霉菌Streptomyces aureofaciens合成的四环类抗生素,又称地美环素、去甲氯四环素,在1957年由McCormick首先报道[1]。去甲基金霉素具有抗菌活性强、吸收好、抗菌谱广等优点,现主要用于治疗立克次体病、布氏杆菌病、支原体肺炎、螺旋体病、衣原体病等疾病,对敏感性革兰氏阳性球菌或革兰氏阴性杆菌所引起的轻微感染也有疗效[2]。此外,去甲基金霉素是重要的医药中间体,合成具有抗耐药菌活性的新生代四环类抗生素如米诺环素、替加环素和奥玛环素(PTK0796)[3-6]。去甲基金霉素的菌种选育多利用传统物理和化学因子进行诱变,但因筛选工作量大且无法定向改造而受到很大的限制[7-8]。
随着分子生物学的发展,利用基因工程方法对相关基因和代谢途径进行改造,提高特定代谢产物的产量和合成效率,已成为链霉菌工业菌种选育的一种重要方式[9-11]。通过对微生物基因组的测序,可以从分子水平上了解其遗传变异规律、重要代谢途径和调控机制,为基因工程菌的构建提供理论依据[9]。而在微生物基因组中发现的重要代谢产物生物合成基因簇,对杂合抗生素开发和组合生物学研究有重要意义。
S. aureofaciens DM-1作为去甲基金霉素的产生菌,国际上尚未见相关基因组序列报道。Deng等完成了对金霉素(Chlortetracycline,CTC)工业菌株S. aureofaciens F3的CTC合成基因簇的测序,从分子水平上分析其生物合成途径,并预测了部分合成基因功能[12-13]。本研究采用Genome Sequencer FLX系统[14]测序结合多引物PCR等方法填补基因组缺口(Gaps),获得了S. aureofaciens DM-1的全基因组序列。通过基因组功能注释,有28个次级代谢产物合成基因簇被预测。其中对去甲基金霉素生物合成基因簇进行了详细注释,并与金色链霉菌F3的CTC生物合成簇进行了初步的比较分析。
1 材料与方法1.1 设备与试剂ABI 3730自动测序仪(ABI公司),Roche 454 GS FLX测序平台(Roche公司),PCR System 9700 (ABI公司),低温高速离心机(Eppendorf公司),Kodak紫外凝胶成像仪(Kodak公司),DYY-6C型电泳仪(北京市六一仪器厂)等。
dNTPs、DNA聚合酶和相关缓冲液(TaKaRa公司),AxyPrepTM凝胶回收试剂盒(Axygen公司),1 kb marker (Thermo Scientific),引物(北京赛百盛基因技术公司)。
金色链霉菌S. aureofaciens DM-1 (华北制药集团有限责任公司提供)。
1.2 基因组DNA的提取取少量金色链霉菌DM-1孢子接种于50 mL ISP2液体培养基(酵母膏4.0 g/L,麦芽汁10.0 g/L,葡萄糖4.0 g/L,pH 7.0),摇瓶培养4 d,收集菌体用于提取基因组DNA。基因组DNA提取方法参考文献[9]。
1.3 基因组测序及组装S. aureofaciens DM-1基因组测序工作在国家人类基因组南方研究中心完成。运用鸟枪法将5 μg DNA超声打断到300–500 bp的范围,基于Ampure beads (Agencourt,美国)进行DNA纯化并制备DNA文库,然后利用GS emPCR kit (Roche Applied Science,美国)将DNA固定在磁珠上进行扩增,通过Roche 454 Genome Sequencer FLX进行测序。最后根据测序情况,利用多引物PCR等方法进行缺口填补(Gap closing)。测序完成后,通过454的软件NewblerV2.3将reads进行组装。
1.4 基因功能注释及蛋白同源性分析基因组分析主要包括基因功能注释、RNA及次级代谢产物合成基因簇的分析。基因预测采用软件Glimmer 3.02[15],相关蛋白的功能注释采用Blast软件与软件KEGG (Kyoto encyclopedia of genes and genomes)、NR (Non-redundant protein database)和COG (Cluster of orthologous groups)进行比对。tRNA预测使用tRNAscan-SE-1.23[16],rRNA的预测利用RNAmmer[17]。
1.5 次级代谢产物合成基因簇分析生物合成基因簇的分析采用软件antiSMASH[18]及NRPSpredictor[19]在线软件。
1.6 S. aureofaciens DM-1的去甲基金霉素合成基因簇及与CTC合成基因簇比较分析将S. aureofaciens DM-1的去甲基金霉素生物合成基因簇与已报道的金色链霉菌F3的CTC合成簇(GenBank登录号:HM627755.1)进行比较。
2 结果与分析2.1 基因组DNA的提取最终基因组DNA测得浓度为569 ng/μL。对提取的1 μL S. aureofaciens DM-1基因组进行凝胶电泳鉴定,符合测序要求(图 1)。
 |
| 图 1 S. aureofaciens DM-1基因组DNA电泳图 Fig. 1 Gel electrophoresis of purified genomic DNA of S. aureofaciens DM-1. M: 1 kb marker; DM-1: genomic DNA of S. aureofaciens DM-1. |
| 图选项 |
2.2 基因组测序及组装S. aureofaciens DM-1基因组测序并进行组装,最终得到一条完整的线性基因组序列,长度为6 824 334 bp,然后对所有的低值区域进行补测,最终测序精确度为99.996 7%。
S. aureofaciens DM-1基因组呈线状,长度为6 824 334 bp,GC含量72.6%。基因组鉴定出6 431个蛋白编码基因、69个tRNA基因、27个rRNA基因(GenBank登录号:CP020567)。同时发现DM-1基因组中含有一个线性质粒(GenBank登录号:CP020568),长为252 068 bp (具体见表 1和图 2)。
表 1 S. aureofaciens DM-1的基因组信息Table 1 Genomic information of S. aureofaciens DM-1
| Strain | S. aureofaciens DM-1 |
| Genome (bp) | 6 824 334 |
| Plasmid (bp) | 252 068 |
| GC (%) | 72.6 |
| CDS | 6 431 |
| Average CDS length (bp) | 921 |
| Code density (%) | 83.4 |
| Reads | 443 465 |
| Bases | 229 658 915 |
| Large contigs (≥500 bp) | 134 |
| N50 contig size (bp) | 143 476 |
| Max contig (bp) | 382 311 |
| rRNA | 27 |
| tRNA | 69 |
表选项
 |
| 图 2 S. aureofaciens DM-1基因组圈图 Fig. 2 Cyclic schematic diagram of S. aureofaciens DM-1 genome. |
| 图选项 |
2.3 基因功能注释及蛋白同源性分析基因功能注释:利用KEGG蛋白数据库、NCBI的NR库以及SEED蛋白数据库进行基因功能注释,共有5 582个蛋白具有明确的生物学功能。
COG功能分类:通过搜寻上述蛋白数据库,发现4 124个蛋白具有COG分类。各类基因数量见图 3。
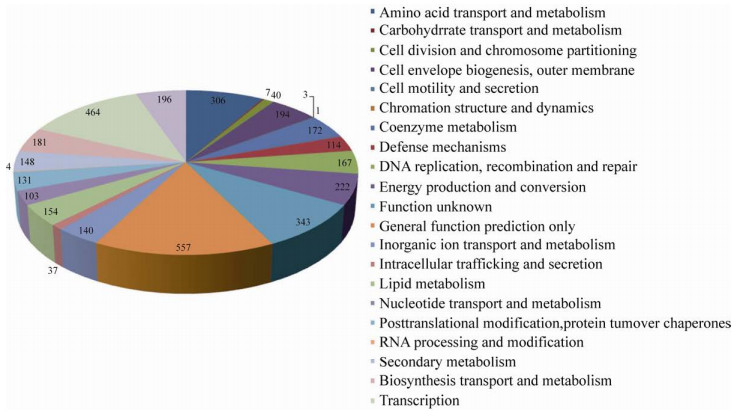 |
| 图 3 S. aureofaciens DM-1基因COG功能分类 Fig. 3 COG functional classification of S. aureofaciens DM-1. |
| 图选项 |
KEGG代谢通路分析:搜寻KEGG蛋白数据库,通过KEGG蛋白数据库构建代谢通路,2 393个蛋白具有KEGG的直系同源(Ortholog)。KEGG代谢通路分类见图 4。
 |
| 图 4 S. aureofaciens DM-1基因KEGG代谢通路分类 Fig. 4 KEGG pathway classification of S. aureofaciens DM-1. |
| 图选项 |
微生物类群匹配分析:通过KEGG蛋白数据库和SEED蛋白数据库匹配度分析,发现所有蛋白匹配的同源蛋白来源于282个物种,其中白丝北里孢菌Kitasatospora setae KM-6054比例最高,达到49.4%。微生物蛋白匹配度分析见图 5。
 |
| 图 5 微生物类群匹配分析 Fig. 5 The matching degree of microbial homologous proteins. |
| 图选项 |
2.4 次级代谢产物合成基因簇分析在S. aureofaciens DM-1基因组中预测到28个次级代谢产物生物合成基因簇,类型包括PKS型、NRPS型、萜类等;可能编码去甲基金霉素在内的18种化合物(表 2)。线性质粒上未发现存在可能的次级代谢产物合成基因簇。
表 2 S. aureofaciens DM-1中预测的次级代谢产物生物合成基因簇Table 2 Predicted biosynthesis clusters in S. aureofaciens DM-1
| No. | Type | Product | Start | Stop | Homologous strains |
| 1 | Terpene | Acarviostatin (11%) | 87 438 | 108 493 | Streptomyces coelicoflavus |
| 2 | Terpene | Hopene (69%) | 118 409 | 145 254 | Streptomyces coelicolor |
| 3 | Siderophore | Scabichelin (20%) | 331 011 | 345 709 | Streptomyces scabiei |
| 4 | Other | - | 504 763 | 548 152 | - |
| 5 | Otherks-Butyrolactone | Neocarzinostatin (4%) | 607 676 | 660 864 | Streptomyces carzinostaticus |
| 6 | Bacteriocin | - | 1 045 041 | 1 056 009 | - |
| 7 | NRPS | Lomaiviticin (6%) | 1 104 350 | 1 167 819 | Salinisporapacifica |
| 8 | Siderophore | - | 1 317 069 | 1 332 792 | - |
| 9 | NRPS | Teleocidin (50%) | 1 504 602 | 1 559 604 | Streptomyces blastmyceticus |
| 10 | Bacteriocin | - | 2 381 686 | 2 393 962 | - |
| 11 | NRPS | Skyllamycin (18%) | 2 448 146 | 2 503 780 | Streptomyces sp. Acta. |
| 12 | Terpene | - | 2 836 249 | 2 858 759 | - |
| 13 | Butyrolactone | - | 3 151 524 | 3 162 639 | - |
| 14 | Thiopeptide | - | 4 081 691 | 4 110 653 | - |
| 15 | T2PKS | Chlortetracycline (74%) | 4 177 690 | 4 220 175 | Streptomyces aureofaciens |
| 16 | Terpene | Sioxanthin (25%) | 4 332 523 | 4 353 629 | Salinisporatropica |
| 17 | T3PKS | Herboxidiene (2%) | 4 585 616 | 4 626 773 | Streptomyces chromofuscus |
| 18 | Bacteriocin-Butyrolactone | Methylenomycin (23%) | 4 696 322 | 4 716 539 | Streptomyces coelicolor |
| 19 | Lantipeptide | - | 5 154 452 | 5 194 693 | - |
| 20 | T3PKS | Herboxidiene (2%) | 5 206 160 | 5 247 305 | Streptomyces clavuligerus |
| 21 | Terpene | Carotenoid (54%) | 5 387 154 | 5 412 340 | Streptomyces avermitilis |
| 22 | NRPS | Teicoplanin (15%) | 5 585 054 | 5 657 779 | Actinoplanesteichomyceticus |
| 23 | T1PKS | FR-008 (76%) | 5 652 207 | 5 803 052 | Streptomyces sp. FR-008 |
| 24 | T1PKS | - | 6 029 872 | 6 076 390 | - |
| 25 | Blactam | Clavams (71%) | 6 101 593 | 6 127 032 | Streptomyces antibioticus |
| 26 | T2PKS | Spore_pigment (83%) | 6 336 477 | 6 378 980 | Streptomyces avermitilis |
| 27 | NRPS | Enduracidin (12%) | 6 446 217 | 6 509 481 | Streptomyces fungicidicus |
| 28 | Terpene | - | 6 623 868 | 6 646 321 | - |
表选项
2.5 S. aureofaciens DM-1的去甲基金霉素合成基因簇及与CTC合成基因簇比较分析参考金色链霉菌S. aureofaciens F3的CTC生物合成基因簇序列(GenBank登录号:HM627755.1),注释包括去甲基金霉素生物合成基因簇核心区在内的62 kb的序列,预测得到42个ORF (表 3)。
表 3 S. aureofaciens DM-1的去甲基金霉素生物合成基因簇基因注释Table 3 Gene annotation in 6-demethylchlortetracycline biosynthetic gene cluster
| Gene | Amino acid (aa) | Strand | Proposed functions |
| Aur_01 | 232 | + | Ctg2 3636 |
| Aur_02 | 281 | - | Methionine minopeptidase |
| Aur_03 | 76 | + | |
| Aur_04 | 442 | - | Major facilitator transporter |
| Aur_05 | 226 | - | |
| Aur_06 | 230 | + | |
| Aur_07 | 60 | + | |
| Aur_08 | 271 | + | |
| Aur_09 | 1 131 | + | Transcriptional regulator, SARP family |
| Aur_10 | 274 | - | ABC-2 type transporter |
| Aur_11 | 341 | - | ABC transporter ATP-binding protein |
| Aur_12 | 289 | - | |
| DM_ctc2 | 539 | + | Drug resistance transporter, EmrB/QacA |
| DM_ctc1 | 209 | + | TetR family transcriptional regulator |
| DM_ctcZ | 347 | + | |
| Aur_16 | 87 | + | |
| DM_ctcY | 440 | - | Sodium/hydrogen exchanger |
| DM_ctcX | 414 | - | Monooxygenase FAD-binding |
| DM_ctcW | 427 | + | Beta-ketoacyl synthase |
| DM_ctcV | 401 | + | Beta-ketoacyl synthase |
| DM_ctcU | 84 | + | Acyl carrier protein |
| DM_ctcT | 609 | + | Asparagine synthase (glutamine-hydrolyzing) |
| DM_ctcS | 165 | - | MarR family transcriptional regulator |
| DM_ctcR | 510 | + | Drug resistance transporter, EmrB/QacA |
| DM_ctcQ | 191 | + | Flavin reductase domain protein FMN-binding |
| DM_ctcP | 555 | - | Halogenase |
| DM_ctcO | 344 | - | Methyltransferase |
| DM_ctcN | 491 | - | Monooxygenase FAD-binding |
| DM_ctcM | 140 | + | |
| DM_ctcL | 363 | + | Aminotransferase |
| DM_ctcK | 318 | - | O-methyltransferase |
| DM_ctcJ | 96 | - | |
| DM_ctcI | 517 | - | AMP-dependent synthetase and ligase |
| DM_ctcH | 138 | - | |
| DM_ctcG | 262 | + | Short-chain dehydrogenase/reductase SDR |
| DM_ctcF | 312 | + | Cyclase/dehydrase |
| DM_ctcE | 541 | + | Monooxygenase FAD-binding |
| DM_ctcD | 256 | + | |
| DM_ctcC | 1 971 | - | GTP binding |
| DM_ctcB | 271 | + | ctcB putative activator |
| DM_ctcA | 357 | - | ctcA putative |
| DM_ctc3 | 401 | - | NAD+ synthase |
表选项
通过将S. aureofaciens DM-1的去甲基金霉素生物合成基因簇与已报道的金色链霉菌F3的CTC合成簇进行比较,发现该合成簇与CTC合成簇相比缺少基因ctc3,该基因编码IS4家族转座酶。根据该基因的功能推测,ctc3可能是与去甲基金霉素合成无关的基因。除此之外,其他基因都具有极高的相似性(> 90%),两者的基因排布也完全一致。鉴于两株菌合成的产物有一个甲基的差别,我们将合成簇内部两个预测甲基化酶进行比对,其氨基酸序列均存在差别。其中DM_ctcK与HM_ctcK相比,其N端由于缺少了一个碱基G,造成移码突变,蛋白翻译提前终止(图 7A),推测去甲基金霉素的合成可能与该甲基转移酶的截短有关。DM_ctcO与其同源蛋白HM_ctcO有5个氨基酸残基的差别,见图 7B。
 |
| 图 6 S. aureofaciens DM-1来源的去甲基金霉素生物合成基因簇结构图 Fig. 6 Genetic architecture of the 6-demethylchlortetracycline biosynthetic gene cluster from S. aureofaciens DM-1. |
| 图选项 |
 |
| 图 7 金霉素与去甲基金霉素合成簇中蛋白同源性比较 Fig. 7 Homology analysis of methyltransferase between chlortetracycline and 6-demethylchlortetracycline biosynthetic gene clusters. (A) Sequence alignment between DM_ctcK and HM_ctcK. (B) Sequence alignment between DM_ctcO and HM_ctcO, * means different amino acids. |
| 图选项 |
3 讨论四环类抗生素是最早使用的广谱抗生素,由于药物滥用等原因造成了严重的耐药性和环境问题[20-21]。人们对第一代四环类抗生素进行结构修饰,得到了具有抗耐药菌活性的第二代和三代四环类抗生素[22]。去甲基金霉素抗菌作用比四环素和土霉素强,比CTC稳定,同时可作为中间体合成第二代和三代四环类抗生素。目前常采用微生物发酵和化学半合成的方法生产去甲基金霉素,其工业生产菌株一般由诱变筛选得到,遗传育种还没有发挥作用。因此,对去甲基金霉素的生产菌株S. aureofaciens DM-1进行全基因组测序,有助于从分子水平上了解其遗传变异规律和次级代谢产物的生物合成途径,为遗传育种提供依据。
本研究通过Roche 454 Genome Sequencer FLX平台,首次获得了去甲基金霉素产生菌S. aureofaciens DM-1的全基因组序列。S. aureofaciens DM-1线状基因组长度为6 824 334 bp,GC含量为72.6%,包含6 431个蛋白编码基因。通过antiSMASH软件,预测得到28个次级代谢产物合成基因簇,其中去甲基金霉素生物合成基因簇为首次报道。
与报道的S. aureofaciens F3的CTC生物合成基因簇对比发现,两合成基因簇有74%的基因相似性。S. aureofaciens DM-1的去甲基金霉素合成簇缺少ctc3基因,该基因编码IS4家族转座酶,推测是与去甲基金霉素合成无关的基因。通过比对两个合成簇的甲基化酶,发现S. aureofaciens DM-1的一个甲基化酶基因DM_ctcK由于N端缺失碱基G,发生移码突变,蛋白翻译提前终止,推测该蛋白的失活是去甲基金霉素合成的主要原因。有文献报道ctcK基因缺失工程菌代谢物中检测到去甲基金霉素,但未检测到金霉素,表明ctcK基因参与金霉素C-6位甲基化[23-24]。而ctcO基因缺失突变株不再合成金霉素,但也没有积累4-氨基脱水四环素或C-4位脱二甲氨基金霉素,而是积累其他代谢产物,因此推测ctcO可能是金霉素生物合成的必需基因,但不是C-4位二甲氨基化酶基因[25]。
通过对去甲基金霉素生物合成基因簇进行注释和分析,有助于其生物合成途径的研究。采用基因工程的方法对合成基因簇上的关键基因进行操作,为适用于工业化生产的工程菌构建提供基础。
参考文献
| [1] | McCormick JRD, Sjolander NO, Hirsch U, et al. A new family of antibiotics: the demethyltetracyclines. J Am Chem Soc, 1957, 79(16): 4561-4563. DOI:10.1021/ja01573a089 |
| [2] | Nelson ML, Levy SB. The history of the tetracyclines. Ann N Y Acad Sci, 2011, 1241(1): 17-32. DOI:10.1111/j.1749-6632.2011.06354.x |
| [3] | Martell MJ, Boothe JH. The 6-deoxytetracyclines. Ⅶ. Alkylated aminotetracyclines possessing unique antibacterial activity. J Med Chem, 1967, 10(1): 44-46. |
| [4] | Nguyen F, Starosta AL, Arenz S, et al. Tetracycline antibiotics and resistance mechanisms. Biol Chem, 2014, 395(5): 559-575. DOI:10.1515/hsz-2013-0292 |
| [5] | Wright PM, Seiple IB, Myers AG. The evolving role of chemical synthesis in antibacterial drug discovery. Angew Chem Int Ed, 2014, 53(34): 8840-8869. DOI:10.1002/anie.201310843 |
| [6] | Garrido-Mesa N, Zarzuelo A, Gálvez J. Minocycline: far beyond an antibiotic. Br J Pharmacol, 2013, 169(2): 337-352. |
| [7] | Daniel MJR, Raymond JE, Oscar SN. 6-demethyltetracyclines and methods for preparing the same: US, 2878289. 1959-03-17. |
| [8] | Growich JA Jr. 7-Chloro-6-demethyl-tetracycline fermentation: US, 3616239. 1971-10-26. |
| [9] | Huang H, Lu XJ, Hu HF. Analysis and determination of complete genome sequence of Actinoplanes sp. N902-109. Chin J Antibiot, 2015, 40(3): 171-177 (in Chinese). 黄鹤, 芦现杰, 胡海峰. Actinoplanes sp. N902-109全基因组序列测定及分析. 中国抗生素杂志, 2015, 40(3): 171-177. DOI:10.3969/j.issn.1001-8689.2015.03.003 |
| [10] | Nakano T, Miyake K, Ikeda M, et al. Mechanism of the incidental production of a melanin-like pigment during 6-demethylchlortetracycline production in Streptomyces aureofaciens. Appl Environ Microbiol, 2000, 66(4): 1400-1404. DOI:10.1128/AEM.66.4.1400-1404.2000 |
| [11] | Wu NX, Huang H, Min TL, et al. TAR cloning and integrated overexpression of 6-demethylchlortetracycline biosynthetic gene cluster in Streptomyces aureofaciens. Acta Biochim Biophys Sin (Shanghai), 2017, 49(12): 1129-1134. DOI:10.1093/abbs/gmx110 |
| [12] | Nakano T, Miyake K, Endo H, et al. Identification and cloning of the gene involved in the final step of chlortetracycline biosynthesis in Streptomyces aureofaciens. Biosci Biotechnol Biochem, 2004, 68(6): 1345-1352. DOI:10.1271/bbb.68.1345 |
| [13] | Zhu T, Cheng XQ, Liu YT, et al. Deciphering and engineering of the final step halogenase for improved chlortetracycline biosynthesis in industrial Streptomyces aureofaciens. Metab Eng, 2013, 19: 69-78. DOI:10.1016/j.ymben.2013.06.003 |
| [14] | Kurtz S, Phillippy A, Delcher AL, et al. Versatile and open software for comparing large genomes. Genome Biol, 2004, 5(2): R12. |
| [15] | Delcher AL, Harmon D, Kasif S, et al. Improved microbial gene identification with GLIMMER. Nucleic Acids Res, 1999, 27(23): 4636-4641. DOI:10.1093/nar/27.23.4636 |
| [16] | Schattner P, Brooks AN, Lowe TM. The tRNAscan-SE, snoscan and snoGPS web servers for the detection of tRNAs and snoRNAs. Nucleic Acids Res, 2005, 33(Suppl_2): W686-W689. |
| [17] | Lagesen K, Hallin PF, R?dland E, et al. RNammer: consistent annotation of rRNA genes in genomic sequences. Nucleic Acids Res, 2007, 35(9): 3100-3108. DOI:10.1093/nar/gkm160 |
| [18] | Blin K, Shaw S, Steinke K, et al. AntiSMASH 5.0: updates to the secondary metabolite genome mining pipeline. Nucleic Acids Res, 2019, 47(W1): W81-W87. DOI:10.1093/nar/gkz310 |
| [19] | R?ttig M, Medema MH, Blin K, et al. NRPSpredictor2 - a web server for predicting NRPS adenylation domain specificity. Nucleic Acids Res, 2011, 39(Suppl_2): W362-W367. DOI:10.1093/nar/gkr323 |
| [20] | Grossman TH. Tetracycline antibiotics and resistance. Cold Spring Harb Perspect Med, 2016, 6(4): a025387. DOI:10.1101/cshperspect.a025387 |
| [21] | Huang FY, An ZY, Moran MJ, et al. Recognition of typical antibiotic residues in environmental media related to groundwater in China (2009-2019). J Hazard Mater, 2020, 399: 122813. DOI:10.1016/j.jhazmat.2020.122813 |
| [22] | Stephens CR, Beereboom JJ, Rennhard HH, et al. 6-deoxytetracyclines. Ⅳ. 1, 2Preparation, C-6 stereochemistry, and reactions. J Am Chem Soc, 1963, 85(17): 2643-2652. DOI:10.1021/ja00900a027 |
| [23] | Lin LZ, Wan YF, Hong WR. Study of ctcK in chlortetracycline biosynthesis gene cluster. J Nat Sci Hunan Normal Univ, 2017, 40(1): 37-43 (in Chinese). 林龙镇, 万云凤, 洪文荣. 金霉素生物合成基因ctcK的研究. 湖南师范大学自然科学学报, 2017, 40(1): 37-43. |
| [24] | Yang WN, Kong LX, Wang Q, et al. Metabolic engineering of a methyltransferase for production of drug precursors demecycline and demeclocycline in Streptomyces aureofaciens. Synth Syst Biotechnol, 2020, 5(3): 121-130. DOI:10.1016/j.synbio.2020.06.001 |
| [25] | Lin LZ. Study on the structural and functional genes of chlortetracycline[D]. Fuzhou: Fuzhou University, 2016 (in Chinese). 林龙镇.金霉素结构功能基因研究[D].福州: 福州大学, 2016. |
