
 , 饶志明
, 饶志明

江南大学 生物工程学院 工业生物技术教育部重点实验室,江苏 无锡 214122
收稿日期:2019-01-19;接收日期:2019-03-13;网络出版时间:2019-06-10
基金项目:中央高校基本科研业务费专项资金(No. JUSRP51708A),江苏高校优势学科建设工程,江苏高校品牌专业建设工程资助项目资助
摘要:从天蓝色链霉菌Streptomyces coelicolor克隆得到海藻糖合酶基因(ScTreS),在大肠杆菌Escherichia coli BL21(DE3)中进行了异源表达,通过Ni-NTA亲和柱对表达产物进行分离纯化得到纯酶,经SDS-PAGE测定其分子量约为62.3 kDa。研究其酶学性质发现该酶最适温度35 ℃;最适pH 7.0,对酸性条件比较敏感。通过同源建模和序列比对分析,对该基因进行定点突变。突变酶K246A比酶活比野生酶提高了1.43倍,突变酶A165T相对提高了1.39倍,海藻糖转化率分别提高了14%和10%。利用突变体重组菌K246A进行全细胞转化优化海藻糖的合成条件并放大进行5 L罐发酵,结果表明:在麦芽糖浓度300 g/L、初始反应温度和pH分别为35 ℃和7.0的条件下,转化率最高达到71.3%,产量为213.93 g/L;当底物浓度增加到700 g/L时,海藻糖产量仍可达到465.98 g/L。
关键词:海藻糖合酶克隆表达定点突变全细胞转化
Heterologous expression of Streptomyces coelicolor trehalose synthase and whole-cell biocatalyst production of trehalose in Escherichia coli
Ao Wu, Xian Zhang, Meijuan Xu, Taowei Yang, Huazhong Li

 , Zhiming Rao
, Zhiming Rao

Key Laboratory of Industrial Biotechnology of Ministry of Education, School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu, China
Received: January 19, 2019; Accepted: March 13, 2019; Published: June 10, 2019
Supported by: Foundamental Research Funds for the Central Universities (No. JUSRP51708A), the Project Funded by the Priority Academic Program Development of Jiangsu Higher Education Institutions, Top-notch Academic Programs Project of Jiangsu Higher Education Institutions
Corresponding author: Huazhong Li. E-mail: hzhli@jiangnan.edu.cn;
Zhiming Rao. Tel: +86-510-85916881; E-mail: raozhm@jiangnan.edu.cn.
Abstract: The trehalose synthase (ScTreS) gene from Streptomyces coelicolor was successfully cloned and heterologously expressed in Escherichia coli BL21(DE3). The protein purified by Ni-NTA affinity column showed an apparent molecular weight (MW) of 62.3 kDa analyzed by SDS-PAGE. The optimum temperature of the enzyme was 35 ℃ and the optimum pH was 7.0; the enzyme was sensitive to acidic conditions. By homologous modeling and sequence alignment, the enzyme was modified by site-directed mutagenesis. The relative activities of the mutant enzymes K246A and A165T were 1.43 and 1.39 times that of the wild type, an increased conversion rate of 14% and 10% respectively. To optimize the synthesis conditions of trehalose, the mutant strain K246A was cultivated in a 5-L fermentor and used for whole-cell transformation. The results showed that with the substrate maltose concentration of 300 g/L at 35 ℃ and pH 7.0, the highest conversion rate reached 71.3%, and the yield of trehalose was 213.93 g/L. However, when maltose concentration was increased to 700 g/L, the yield of trehalose can reach 465.98 g/L with a conversion rate of 66%.
Keywords: trehalose synthaseclone and expressionsite-directed mutagenesiswhole-cell conversion
海藻糖(α-D-吡喃葡萄糖基-(1→1)-α-D-吡喃葡萄糖苷)是一种安全稳定的天然非还原性二糖[1],广泛存在于细菌、酵母和蘑菇等真菌、藻类、昆虫及无脊椎动物中[2]。海藻糖具有保湿性、抗冻抗干燥性,对酸热高度稳定等理化性质和对生物大分子的非特异性保护等生物学功能[3],可作为抗体疫苗和酶的干燥稳定剂,抑制脂肪酸化、防腐的食品添加剂,表面活性剂以及培育抗旱转基因植物的媒介[4]等,在医学、化妆品、食品、农业等行业起着重要作用,具有广阔的应用前景和市场。海藻糖的产量现状仅能基本满足国内市场需求,且大部分被国外垄断,国内的生产工艺、产量和产品纯度等都还具有很大的提升空间。
目前海藻糖的产业化生产主要通过以下3种酶转化法实现:1)磷酸化酶法[5-6];2)双酶法——麦芽寡糖基海藻糖合成酶和麦芽寡糖基海藻糖水解酶[7-8];3)海藻糖合成酶法[9-10]。其中方法一需要消耗价格昂贵的高能磷酸化合物UDP-葡萄糖和6-磷酸葡萄糖,生产成本高,无法大规模生产;方法二是目前工业化运用最多的方法之一[11],效率远高于早期的发酵和酵母提取等方法[12],但同时也存在着低聚糖积累、反应成分复杂、工艺要求较高的弊端;方法三是通过海藻糖合成酶的分子内转糖苷作用将麦芽糖转化为海藻糖,底物专一性强,具有途径简单、生产原料廉价、产物简单等优点,是酶法生产中最理想的工业化生产方法,也是一直以来的研究热点。众多不同来源的海藻糖合酶陆续被发现,性质和三维结构也被解析,不断有****尝试以分子手段如定向进化和基于结构的定点诱变等方法来解决和突破海藻糖合酶稳定性低、转化率不高等限制实际生产应用的瓶颈[13]。其中取得显著效果的有Chou等以已知耐高温海藻糖合成酶序列为参考,将Picrophilus torridus来源的海藻糖合酶(PTTS)中部分氨基酸残基替换突变为脯氨酸得到的突变体N503P. PTTS,海藻糖产量提升了1.3倍,而且在65 ℃条件下的相对活力比野生型提高了39%[14]。还有王青艳等对褐色喜热裂孢菌Thermobifida fusca海藻糖合成酶保守氨基酸残基进行的定点突变,突变酶比活力提高了1.25倍[15]。
文中将来源于天蓝色链霉菌的海藻糖合酶基因(TreS)在大肠杆菌Escherichia coli中进行过表达,得到了重组酶ScTreS,针对其酶学性质进行初步研究,并结合同源建模得到的海藻糖合酶预测结构和多序列比对结果,对TreS保守区和活性中心附近的一些氨基酸进行定点突变,筛选得到了性能提高的突变体。进一步对野生型和突变体的酶学性质进行了对比分析,同时优化了突变体重组菌全细胞转化合成海藻糖的反应条件,为海藻糖合酶的工业化生产应用提供了实验基础。
1 材料与方法1.1 材料1.1.1 菌株与质粒天蓝色链霉菌Streptomyces coelicolor购自广东省微生物菌种保藏中心。大肠杆菌E. coli BL21 (DE3)和质粒pET-28a由本实验室保藏。
1.1.2 酶和试剂EcoRⅠ、Hind Ⅲ限制性内切酶、DNA聚合酶和DpnⅠ等购自TaKaRa公司。高保真PCR酶、同源重组酶克隆试剂盒购自南京诺维赞生物科技有限公司;琼脂糖凝胶DNA回收试剂盒、小量质粒提取试剂盒、细菌DNA基因组提取试剂盒购于上海捷瑞生物工程有限公司;卡那霉素、异丙基-β-D-硫代半乳糖苷(IPTG)购自生工生物工程(上海)股份有限公司;麦芽糖一水合物、葡萄糖、海藻糖二水合物等其他分析纯试剂均购自国药集团化学试剂有限公司。
1.1.3 培养基天蓝色链霉菌培养基:可溶性淀粉20 g/L,KNO3 1 g/L,K2HPO4 0.5 g/L,MgSO4·7H2O 0.5 g/L,NaCl 0.5 g/L,FeSO4 0.01 g/L,pH 7.2–7.4。
LB液体培养基:胰蛋白胨10.0 g/L,酵母粉5.0 g/L,NaCl 10.0 g/L。
TY培养基:酵母粉8.0 g/L,甘油10.0 g/L,胰蛋白胨12.0 g/L,K3PO4 4.02 g/L,NaCl 3 g/L,一水合柠檬酸2.1 g/L,柠檬酸铁铵0.3 g/L,(NH4)2SO4 2.5 g/L,MgSO4·7H2O 0.5 g/L,pH 7.2。
1.2 方法1.2.1 基因提取和引物设计天蓝色链霉菌基因组的提取按照细菌基因组提取试剂盒的方法完成。
通过对NCBI上已有的链霉菌来源的海藻糖合酶基因进行多序列比对,设计并合成同源臂引物ScT-F与ScT-R,序列参见表 1。
表 1 本研究中使用的引物Table 1 Primers used in this study
| Primer name | Primer sequence (5′–3′) | Size (bp) |
| ScT-F | TGGGTCGCGGATCCGAATTCATGATCGTCAACGAGCCCGT | 40 |
| ScT-R | TCGAGTGCGGCCGCAAGCTTTCAGGCGGCGTCCTTGCGCA | 40 |
| K246A-F | CTCAAGCGGGTCCGCGCAGAGATCGACGCCCACTA | 35 |
| A165T-F | TTCGTCGACACCGAGACGTCCAACTGGACCTTCGA | 35 |
| F178Y-F | GTCCGCAAGCAGTACTACTTCCACCGCTTCTTCTC | 35 |
| F179W-F | CGCAAGCAGTACTTCTGGCACCGCTTCTTCTCCCA | 35 |
| Q125A-F | AACCACACCAGCGACGCACACCCGTGGTTCCAGGA | 35 |
| I361A-F | CGCGCCAACATCGGTGCACGCAGGCGCCTCGCCCC | 35 |
| T169S-F | GAGGCCTCCAACTGGAGCTTCGACCCGGTCCGCAA | 35 |
| E340D-F | GAAATGGTCACCGACGACGAACGCGACTACATG | 33 |
| L76A-F | CCGCCCTTCTTCAAGGCACCGCTGAGGGACGGCGG | 35 |
| D188A-F | TTCTCCCACCAGCCGGCACTCAACTACGAGAACCC | 35 |
| E331A-F | CTGCGCAACCACGACGCACTGACCCTCGAAATGGT | 35 |
| pET28a-2254-R | GCCTTACTGGTTAGCAGAATG | 21 |
| The italic sequences are the homologous arm and the underlined sequences are the mutated sites. | ||
表选项
1.2.2 基因的克隆和重组表达载体构建以提取的基因组为模板,使用引物ScT-F和ScT-R扩增,将纯化得到的PCR产物与用EcoRⅠ和Hind Ⅲ酶切回收的pET28a载体以一定比例混合用同源重组试剂盒进行连接。最后将连接产物转化入E. coli BL21 (DE3)感受态细胞中,在LB固体平板培养,菌落PCR筛选鉴定后挑取阳性转化子至LB液体培养基中培养,并提取得到重组质粒pET28a-ScTreS送至上海瑞迪生物科技有限公司测序,验证是否成功构建重组菌株。
1.2.3 突变位点的选择和突变体的构建根据海藻糖合酶氨基酸序列,采用http://swissmodel.expasy.org网站服务器进行海藻糖合酶三维结构的构建,并与不同来源的海藻糖合酶氨基酸序列进行比对分析。本着以侧链最短且不带电荷的丙氨酸或其他相应关键氨基酸残基替换的原则,分别选择L76、Q125、A165、T169、F178、F179、D188、K246、E331、E340、I361位点进行突变。突变引物参见表 1。
定点突变选择以大引物PCR定点突变的方法[16-18]进行。以野生型的重组表达质粒pET28a-ScTreS为模板使用高保真酶进行PCR反应。在上游引物中引入突变点,下游引物采用质粒pET28a上下游的一段序列作为通用引物(pET28a-2254-R),第一步PCR反应扩增的产物作为含有突变序列的大片段引物,在第二步PCR反应中再次变性延伸得到含完整突变基因的全长质粒。反应条件:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火1 min,72 ℃延伸3 min,5个循环;95 ℃变性30 s;68 ℃延伸6 min,20个循环;68 ℃充分延伸12 min。反应结束用DpnⅠ消化PCR产物中的模板之后将其转化E. coli BL21 (DE3)。挑取菌落PCR鉴定结果阳性的转化子送上海瑞迪生物科技有限公司进行测序。
1.2.4 TreS酶的表达和纯化由-40 ℃冰箱取出菌种,于固体LB平板划线活化,挑取单菌落接入含有卡那霉素(50 μg/mL)抗性的10 mL LB培养液中。37 ℃过夜培养后按照1% (V/V)的接种量转接入含相同浓度抗生素的50 mL LB培养基中继续培养2–3 h,待OD600达到0.6–0.8时加入终浓度为0.5 mmol/L的IPTG,16 ℃过夜培养。离心收集菌体,用50 mmol/L磷酸钠缓冲液(pH 7.0)洗涤菌体并悬浮。超声破碎(破碎1 s,间隔3 s,共破碎8–10 min),12 000 r/min低温离心20 min,取上清得重组菌粗酶液。
将粗酶液用0.2 μm滤膜过滤处理,通过Ni-NTA亲和层析,利用不同浓度的咪唑进行梯度洗脱获得纯化蛋白。并采用Bradford法测定蛋白浓度[19-20]。
1.2.5 酶活测定反应体系包含100 g/L麦芽糖,50 mmol/L的pH 7.0磷酸钠缓冲液,30 μg纯酶,35 ℃水浴保持1 h,沸水浴10 min终止反应。使用HPLC法检测酶活。高效液相色谱检测条件为:RID检测器,NH2柱(Agilent 5 μm, 4.6 mm×250 mm),流动相为80%乙腈,流速1 mL/min,柱温40 ℃。
酶活定义为:每1 min生成1 μmol海藻糖的酶量为1个酶活力单位。
1.2.6 酶学性质酶的最适反应温度和最适反应pH:分别在25 ℃、30 ℃、35 ℃、40 ℃、45 ℃温度下测定海藻糖合酶的酶活,分析获得酶的最适反应温度。分别在不同pH 5.5、6.0、6.5、7.0、7.5、8.0的反应体系下测定海藻糖合酶的酶活,研究酶的最适反应pH。
酶的热稳定性:取适量纯酶液在35 ℃、40 ℃、45 ℃保温一定时间后取样检测其残余酶活力,考察酶的热稳定性。
1.2.7 突变体的发酵罐培养及全细胞转化生产海藻糖将突变体BL21/pET28a-K246A按1.2.4方法活化并转接入50 mL LB大瓶,培养5–6 h后接种至装有2.5 L TY培养基的5 L发酵罐中低转速培养,待菌体浓度OD600达到18–20时,添加0.2 mmol/L的IPTG,28 ℃高转速诱导培养12 h左右,发酵液用6 000 r/min离心30 min,去除上清,收集菌体,进行全细胞转化优化反应条件。
2 结果与分析2.1 基因克隆及重组载体构建提取天蓝色链霉菌基因组作为模板PCR扩增目的基因TreS。按1.2.2方法构建重组菌,并送样基因测序得到TreS序列,核酸大小1 701 bp (图 1A),GenBank的Protein ID为AZQ37515.1。
 |
| 图 1 Streptomyces coelicolor TreS基因的克隆(A)和表达及纯化分析(B) Fig. 1 The result of amplification and expression of target TreS gene of Streptomyces coelicolor. (A) The amplification result. (B) SDS-PAGE of the purified TreS from E. coli BL21(DE3). M: marker; 1: crude enzyme; 2–3: purified TreS |
| 图选项 |
2.2 突变位点的选择同源建模及利用Discovery Studio以麦芽糖为配体,进行分子对接结果见图 2。海藻糖合成酶具有典型的(β/α) 8桶装催化结构域,底物结合在蛋白表面的凹槽内,与分子内转糖苷作用活性中心靠近酶表面的报道相符[21-22]。根据已有的结构研究和分析结果,推测保守序列Ⅰ区的His121和Ⅳ区的His329介导底物结合,保守序列Ⅱ区和Ⅲ区形成活性中心,其中的Asp218和Glu260分别作为亲核基团和质子供体发挥作用[23-25]。实验设计选择L76、Q125、A165、T169、F178、F179、D188、K246、E331、E340、I361等残基进行定点突变,按1.2.3方法构建突变重组菌,将验证含重组质粒的阳性克隆子送往公司测序,除去部分未突变的原始菌株外,其他转化子突变位置和氨基酸均符合预期突变。
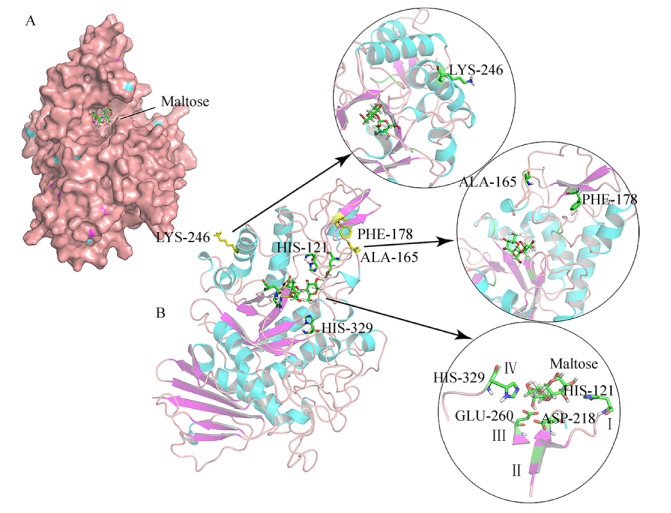 |
| 图 2 ScTreS结合底物麦芽糖的表面结构图(A)和底物结合位点、保守结构域以及部分突变氨基酸三维结构图(B) Fig. 2 The binding model of ScTreS 3D molecular with maltose substrate. (A) A surface structure of ScTreS with maltose. (B) The location of substrate binding site, conserved domain (Ⅰ, Ⅱ, Ⅲ, Ⅳ), and mutant amino acids (A165, F178, K246) |
| 图选项 |
2.3 野生重组酶与突变酶的表达纯化按1.2.4的方法将取得的重组菌和突变体粗酶液用Ni-NTA琼脂糖凝胶柱纯化。取少量粗酶液和纯化产物进行SDS-PAGE分析,如图 1B和图 3所示,含有重组质粒pET28a-TreS的大肠杆菌在分子量62 kDa处有明显蛋白表达带,所得纯化酶液单一,可见TreS在E. coli BL21 (DE3)中能够正常表达。野生酶与突变酶酶活结果如表 2所示,突变酶K246A与野生酶比酶活提高1.43倍;A165T和F178Y提高约1.39倍和1.18倍。已报道如谷氨酸棒杆菌Corynebacterium glutamicum、天蓝色链霉菌Streptomyces coelicolor (Gene ID: 8250619)、阿维链霉菌Streptomyces avermitilis、灰色链霉菌Streptomyces griseus、弯曲热单孢菌Thermomonospora curvate来源的TreS酶活分别为55、56、57、60和65 U/mg[26]。可见野生型ScTreS酶活略偏低,而A165T和K246A的酶活则基本相持平或更高。另外以100 g/L的海藻糖为底物,在相同条件下反应测得ScTreS逆向酶活为20.9 U/mg。
 |
| 图 3 野生型ScTreS和突变株纯化的SDS-PAGE分析 Fig. 3 SDS-PAGE of the purified ScTreS and mutants from E. coli BL21(DE3). M: marker |
| 图选项 |
表 2 重组野生型和突变体ScTreS的相对酶活Table 2 Relative activities of wild type ScTreS and mutants
| Enzyme/ mutant | Specific activity (U/mg) | Relative activity (%) |
| Wild type | 47.9±0.6 | 100.0±1.2 |
| K246A | 68.5±0.8 | 143.0±1.7 |
| A165T | 66.5±0.3 | 139.0±0.7 |
| F178Y | 56.5±1.2 | 118.0±2.5 |
| F179W | 48.8±0.4 | 102.0±0.8 |
| Q125A | 43.2±0.5 | 90.3±1.1 |
| I361A | 7.3±0.5 | 15.2±0.9 |
| T169S | 41.1±0.9 | 85.9±1.8 |
| E340D | 32.3±0.5 | 67.4±1.1 |
| L76A | 0 | 0 |
| D188A | 0 | 0 |
| E331A | 0 | 0 |
表选项
2.4 野生酶与突变酶的酶学性质比较2.4.1 野生酶与突变酶的最适温度比较酶在不同温度下测得的酶活结果见图 4A,突变酶与野生酶ScTreS的最适反应温度并没有发生明显变化,都为35 ℃左右。当温度大于35 ℃时突变体Q125A酶活即迅速下降,而野生酶和其他突变体在40 ℃以下仍能保持较高酶活,温度大于40 ℃时,酶活开始逐渐下降。
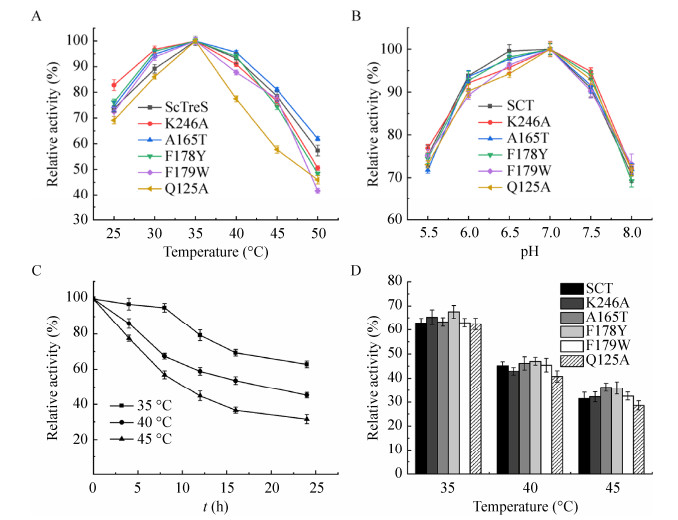 |
| 图 4 野生酶和突变酶的最适温度(A)、最适pH (B)和温度稳定性(C-D) Fig. 4 Optimal temperature (A), optimal pH (B) and thermal stability (C, D) of wild and mutant enzyme |
| 图选项 |
2.4.2 野生酶与突变酶的最适pH比较实验结果表明(图 4B),重组酶和突变酶的最适反应pH都为7.0,野生酶和突变体K246A、A165T、F179W在pH 6.0–7.5之间始终保持大于90%的较高酶活力。酸性条件下,突变体K246A、A165T的相对酶活与野生酶对比相对较低,而在碱性条件下则相反。
2.4.3 野生酶和突变酶热稳定性的比较将野生酶于35 ℃、40 ℃和45 ℃静置并取样检测酶的剩余活力,结果见图 4C。在不同温度保持12 h后,35 ℃下酶液仍约有80%酶活,40 ℃和45 ℃条件下分别剩余约60%和45%的酶活;而在24 h后,酶液在35 ℃、40 ℃、45 ℃条件下的酶活分别下降到约62%、45%、31%;可见野生酶活力在45 ℃以下时比较稳定,在45 ℃时则变得较差。
将突变酶分别置于35 ℃、40 ℃、45 ℃保持24 h后取样测其残余酶活并与野生酶进行对比,结果如图 4D,突变体的热稳定性与野生酶的范围相近,仅发生了细微变化,没有明显的提高或降低。
2.4.4 野生酶与突变酶合成海藻糖的能力比较取适量野生型ScTreS和突变株K246A、A165T、F178Y、F179W、Q125A纯酶,在50 mmol/L磷酸钠缓冲液(pH 7.0)、100 g/L麦芽糖底物浓度体系下,35 ℃、150 r/min条件反应,按时取样进行HPLC检测。结果如图 5所示,反应8 h时,突变株K246A海藻糖转化率达到最高,为71.1%,比野生型提高约14%;A165T最高转化率66.8%,相对提高约10%;F178Y相对提高约6%;F178W基本与野生酶持平;而Q125A转化率比野生型降低约5%。
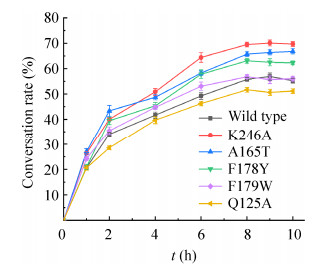 |
| 图 5 野生型ScTreS和突变株K246A、A165T、F178Y、F179W、Q125A合成海藻糖能力的比较 Fig. 5 Comparison of the ability of wild-type ScTreS and mutant enzyme K246A, A165T, F178Y, F179W and Q125A to synthesize trehalose |
| 图选项 |
2.5 海藻糖合酶突变体全细胞转化合成海藻糖的条件优化2.5.1 温度对全细胞转化合成海藻糖的影响温度通过影响酶促反应的效率而影响海藻糖的合成。控制初始反应pH 7.0,底物浓度200 g/L,菌体量OD600=20,其他条件相同的情况下,分别在25 ℃、30 ℃、35 ℃、40 ℃、45 ℃下进行全细胞转化合成海藻糖,结果如图 6A所示。当反应温度为35 ℃时,转化率达到最高,达到64.5%,同时伴随生成12.7%的葡萄糖;在45 ℃时转化率较低,仅为50.7%,并生成12.5%的葡萄糖。
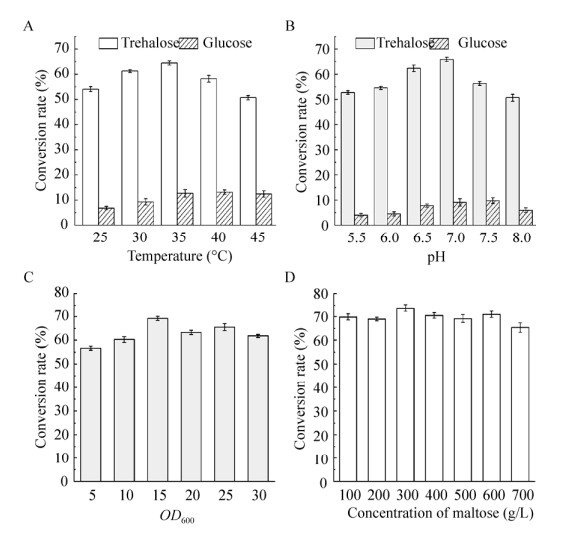 |
| 图 6 温度(A)、pH (B)、菌体生长密度(C)和底物浓度(D)对突变重组菌BL21/pET28a-K246A全细胞转化合成海藻糖的影响 Fig. 6 Influence of temperature (A), pH (B), cell growth density (C) and substrate concentration (D) on the transformation of trehalose in mutant recombinant BL21/pET28a-K246A whole cells |
| 图选项 |
2.5.2 初始pH对全细胞转化合成海藻糖的影响维持反应温度35 ℃,底物浓度200 g/L,菌体量OD600为20,其他条件相同的情况下,在初始体系pH分别为5.5、6.0、6.5、7.0、7.5、8.0条件下进行全细胞转化合成海藻糖。pH对海藻糖制备的影响结果见图 6B,pH小于7.0时,全细胞的转化率随pH的升高而增加,在pH 6.5和7.0时转化率较高,分别为62.4%和65.9%,副产物葡萄糖的转化率分别为7.82%和9.1%;而当pH升高到8.0时,转化率则下降到50.7%。
2.5.3 菌体量对全细胞转化合成海藻糖的影响实际应用中为了满足生产效率和降低生产成本,将菌体浓度控制在适宜的水平也是至关重要的。控制其他条件相同,在反应温度35 ℃,初始pH 7.0,底物浓度200 g/L,菌体量OD600分别为5、10、15、20、25、30条件下进行全细胞转化合成海藻糖。结果如图 6C,转化体系细胞OD600为15时,海藻糖产量最高,转化率为69.2%。
2.5.4 底物浓度对全细胞转化合成海藻糖的影响控制反应温度35 ℃,初始pH 7.0,菌体量OD600为15,在麦芽糖浓度分别为100、200、300、400、500、600、700 g/L条件下进行生物转化。全细胞转化率结果如图 6D所示,并没有表现出明显的底物抑制效果,底物浓度300 g/L转化率达到最高,约73.7%。当底物浓度增加到700 g/L时,转化率也能维持在65.3%左右。
2.5.5 5 L罐全细胞转化用1 L pH 7.0、50 mmol/L的磷酸钠缓冲液配置的麦芽糖溶液悬浮适量BL21/pET28a-K246A菌体,使其OD600为15,35 ℃、200 r/min条件下于罐上转化,液相检测转化液中海藻糖、麦芽糖及葡萄糖的浓度。结果如图 7所示,300 g/L底物浓度下,共生成213.93 g/L海藻糖,21.35 g/L葡萄糖,转化率71.3%。700 g/L底物浓度下,共生成465.98 g/L海藻糖,42.73 g/L葡萄糖,转化率66.5%。
 |
| 图 7 重组菌BL21/pET28a-K246A分别在底物麦芽糖浓度300 g/L (A)和700 g/L (B)条件下的5 L罐转化 Fig. 7 The whole-cell bioconversion with recombiant BL21/pET28a-K246A under the condition of 300 g/L (A) or 700 g/L (B) maltose in 5 L fermenter |
| 图选项 |
3 讨论文中从天蓝色链霉菌中克隆出一种新型海藻糖合酶并在大肠杆菌BL21 (DE3)中进行了异源表达。测得其相应的酶学性质参数,最适反应温度35 ℃,最适pH 7.0,海藻糖转化率最高为65%。同源建模模拟得到ScTreS的三维结构并进行底物对接。根据序列比对的结果和对三维模型的分析,选取活性中心附近的部分氨基酸残基进行了定点突变。推测突变体L76A、D188A、E331A、Q125A、T169S、I361A和E340D影响破坏了底物结合位点和催化中心,导致酶完全失活或降低;A165和F178分别位于底物结合的凹槽的正上方, 突变后表面结构发生改变,使底物更容易进入到酶的活性中心,所以比酶活得到了提高,海藻糖转化率也提高了10%和6%;而K246在保守序列Ⅱ区、Ⅲ区对应结构后的第4个α螺旋的转角,突变为丙氨酸后,酶的疏水性增加,底物的催化结构域也发生细微变化,对酶活力产生了影响,使得海藻糖转化率提高了14%。对此还需要进一步的饱和突变以及晶体结构的研究分析来进行佐证。
酶法催化比全细胞催化对反应条件更加敏感,容易失活,而全细胞催化更稳定,可重复利用率高,文中的全细胞重复利用3次后海藻糖产量仍有54.7%,另外全细胞催化不仅在反应过程中会消耗一部分葡萄糖副产物,有利于后期海藻糖的分离纯化;而且省去了酶法催化前期所需破碎细胞的过程,工艺流程更简便优化,经济效益更高。所以为了契合实际生产,本研究对利用突变株BL21/pET28a-K246A全细胞转化合成海藻糖的反应条件也进行了探索优化。结果表明在温度35 ℃、pH 7.0、底物浓度300 g/L的最适条件下,海藻糖转化率为73.7%;改变底物浓度,转化率也基本大于63%,在高浓度底物700 g/L时的转化率分别为66%,符合目前已报道和实际应用的最高水平[27],与经过透性化等工艺处理的细胞海藻糖转化率水平相比,转化率相近或更高,在效率和生产工艺上都更有优势。综上所述,文中所构建的天蓝色链霉菌海藻糖合成酶和突变株及其相关研究结果为海藻糖合成酶生产海藻糖的工业化应用进一步奠定了理论和实验基础。
参考文献
| [1] | Crowe JH, Crowe LM, Chapman D. Preservation of membranes in anhydrobiotic organisms: the role of trehalose.Science, 1984, 223(4637): 701–703.DOI: 10.1126/science.223.4637.701 |
| [2] | Paul MJ, Primavesi LF, Jhurreea D, et al. Trehalose metabolism and signaling.Annu Rev Plant Biol, 2008, 59: 417–441.DOI: 10.1146/annurev.arplant.59.032607.092945 |
| [3] | Roth R, Sussman M. Trehalose synthesis in the cellular slime mold Dictyostelium discoideum.Biochim et Biophys Acta, 1966, 122(2): 225–231.DOI: 10.1016/0926-6593(66)90064-6 |
| [4] | Huang RB. Trehalose: A Novel Sugar in the New Century.Beijing: Chemical Industry Press, 2010(in Chinese). 黄日波. 海藻糖: 21世纪的新型糖类.北京: 化学工业出版社, 2010. |
| [5] | Kern C, Wolf C, Bender F, et al. Trehalose-6-phosphate synthase from the cat flea Ctenocephalides felis and Drosophila melanogaster: gene identification, cloning, heterologous functional expression and identification of inhibitors by high throughput screening.Insect Mol Biol, 2012, 21(4): 456–471.DOI: 10.1111/imb.2012.21.issue-4 |
| [6] | Zhao SM, Fu FL, Gou L, et al. Cloning and truncation modification of trehalose-6-phosphate synthase gene from Selaginella pulvinata.Gene, 2013, 512(2): 414–421.DOI: 10.1016/j.gene.2012.09.052 |
| [7] | Carpinelli J, Kr?mer R, Agosin E. Metabolic engineering of Corynebacterium glutamicum for trehalose overproduction: role of the TreYZ trehalose biosynthetic pathway.Appl Environ Microbiol, 2006, 72(3): 1949–1955.DOI: 10.1128/AEM.72.3.1949-1955.2006 |
| [8] | Maruta K, Hattori K, Nakada T, et al. Cloning and sequencing of trehalose biosynthesis genes from Arthrobacter sp. Q36.Biochim Biophys Acta (BBA) - Gen Subj, 1996, 1289(1): 10–13.DOI: 10.1016/0304-4165(95)00139-5 |
| [9] | Liang JY, Huang RB, Huang Y, et al. Cloning, expression, properties, and functional amino acid residues of new trehalose synthase from Thermomonospora curvata DSM 43183.J Mol Catal B: Enzym, 2013, 90: 26–32.DOI: 10.1016/j.molcatb.2013.01.014 |
| [10] | Zhang R, Pan YT, He SM, et al. Mechanistic analysis of trehalose synthase from Mycobacterium smegmatis.J Biol Chem, 2011, 286(41): 35601–35609.DOI: 10.1074/jbc.M111.280362 |
| [11] | Song XG, Tang SS, Jiang L, et al. Integrated biocatalytic process for trehalose production and separation from maltose.Ind Eng Chem Res, 2016, 55(40): 10566–10575.DOI: 10.1021/acs.iecr.6b02276 |
| [12] | Schiraldi C, di Lernia I, de Rosa M. Trehalose production: exploiting novel approaches.Trends Biotechnol, 2002, 20(10): 420–425.DOI: 10.1016/S0167-7799(02)02041-3 |
| [13] | Cai X, Seitl I, Mu WM, et al. Biotechnical production of trehalose through the trehalose synthase pathway: current status and future prospects.Appl Microbiol Biotechnol, 2018, 102(12): 2965–2976. |
| [14] | Chou HH, Chang SW, Lee GC, et al. Site-directed mutagenesis improves the thermostability of a recombinant Picrophilus torridus trehalose synthase and efficiency for the production of trehalose from sweet potato starch.Food Chem, 2010, 119(3): 1017–1022.DOI: 10.1016/j.foodchem.2009.08.010 |
| [15] | Wang QY, Chen FZ, Huang FB, et al. Study on site-directed mutagenesis and kinetics of trehalose synthase from Thermobifida fusca.J Guangxi Agric Biol Sci, 2007, 26(2): 115–119.(in Chinese). 王青艳, 陈发忠, 黄福宝, 等. Thermobifida fusca海藻糖合成酶的定点突变及其动力学性质研究.广西农业生物科学, 2007, 26(2): 115-119. |
| [16] | Ke SH, Madison EL. Rapid and efficient site-directed mutagenesis by single-tube 'megaprimer' PCR method.Nucleic Acids Res, 1997, 25(16): 3371–3372.DOI: 10.1093/nar/25.16.3371 |
| [17] | Ling MM, Robinson BH. Approaches to DNA mutagenesis: an overview.Anal Biochem, 1997, 254(2): 157–178.DOI: 10.1006/abio.1997.2428 |
| [18] | Sambrook J. Rapid and efficient site-directed mutagenesis by the single-tube megaprimer PCR method.Nat Methods, 2004, 1: 181–182.DOI: 10.1038/nmeth1104-181 |
| [19] | Kruger NJ. The Bradford method for protein quantitation//Walker JM, Ed. Basic Protein and Peptide Protocols. Totowa: Humana Press, 1994, 32: 9-15. |
| [20] | Walker JM. The Protein Protocols Handbook.3rd ed. Clifton.New Jersey: Humana Press, 2009. |
| [21] | Caner S, Nguyen N, Aguda A, et al. The structure of the Mycobacterium smegmatis trehalose synthase reveals an unusual active site configuration and acarbose-binding mode.Glycobiology, 2013, 23(9): 1075–1083.DOI: 10.1093/glycob/cwt044 |
| [22] | Wang YL, Chow SY, Lin YT, et al. Structures of trehalose synthase from Deinococcus radiodurans reveal that a closed conformation is involved in catalysis of the intramolecular isomerization.Acta Crystallogr Sect D: Biol Crystallogr, 2014, 70: 3144–3154.DOI: 10.1107/S1399004714022500 |
| [23] | Mirza O, Skov LK, Remaud-Simeon M, et al. Crystal structures of amylosucrase from Neisseria polysaccharea in complex with D-glucose and the active site mutant Glu328Gln in complex with the natural substrate sucrose.Biochemistry, 2001, 40(30): 9032–9039.DOI: 10.1021/bi010706l |
| [24] | Ravaud S, Robert X, Watzlawick H, et al. Trehalulose synthase native and carbohydrate complexed structures provide insights into sucrose isomerization.J Biol Chem, 2007, 282(38): 28126–28136.DOI: 10.1074/jbc.M704515200 |
| [25] | Zhang DH, Li N, Lok SM, et al. Isomaltulose synthase (PalI) of Klebsiella sp. LX3. Crystal structure and implication of mechanism.J Biol Chem, 2003, 278(37): 35428–35434.DOI: 10.1074/jbc.M302616200 |
| [26] | Liang JY. Site-directed mutagenesis of active center of trehalose synthase and preparation of trehalulose[D]. Nanning: Guangxi University, 2013 (in Chinese). 梁甲元.海藻糖合成酶活性中心的定点突变及其制备海藻酮糖的研究[D].南宁: 广西大学, 2013.http://cdmd.cnki.com.cn/Article/CDMD-10593-1014379229.htm |
| [27] | Liu HL, Yang SJ, Liu Q, et al. A process for production of trehalose by recombinant trehalose synthase and its purification.Enzyme Microb Technol, 2018, 113: 83–90.DOI: 10.1016/j.enzmictec.2017.11.008 |
