
 , 卢元1
, 卢元1

1 清华大学 化学工程系,北京 100084;
2 沈阳师范大学 生命科学学院,辽宁 沈阳 110034
收稿日期:2018-05-12;接收日期:2018-06-28; 网络出版时间:2018-07-14 基金项目:国家自然科学基金(No. 21706144)资助
作者简介:卢元 清华大学化学工程系助理教授、博士生导师。研究的焦点在于发展和应用无细胞合成等世界最前沿的技术,突破天然生命体系的限制,高效合成和精准改造核酸、蛋白质等生物大分子,以解决生物制造、人类与动物健康等领域最具挑战性的科学与工程难题。目前已在Proc Natl Acad Sci USA等杂志上发表 30多篇高影响学术杂志论文,拥有5项国际授权专利、12项国内授权专利。研究成果得到了相关领域国内外同行专家的广泛关注和高度评价,并产生了日益增大的国际影响力。更多信息请浏览课题组网站:http://LuLab.org
摘要:无细胞非天然蛋白质合成作为蛋白质研究的新兴手段,已成功用于表征蛋白质分子间、蛋白质与核酸分子间相互作用等基础科学研究及医药蛋白、蛋白质材料等工业生产领域。无细胞非天然蛋白质合成系统不需维持细胞的生长,无细胞膜阻碍,可依据研究目的添加基因元件或化学物质从而增强工程设计和过程调控的自由性;也可赋予蛋白质新的特性、结构及功能,如可实现蛋白翻译后修饰、反应手柄引入、生物物理探针及多聚蛋白质合成等。文中系统地综述了目前应用于无细胞蛋白质合成系统中的非天然氨基酸嵌入方法,包括全局抑制及基于正交翻译体系的终止密码子抑制、移码抑制、有义密码子再分配和非天然碱基等方法的研究进展,及非天然氨基酸在蛋白质修饰、生物物理探针、酶工程、蛋白质材料以及医药蛋白质生产等领域的应用进展,并分析了该体系的发展前景及广泛工业化应用的机遇与挑战。
关键词:无细胞合成生物学 蛋白质工程 非天然氨基酸 非天然蛋白质
Recent advances in cell-free unnatural protein synthesis
Wei Gao1,2, Ning Bu2

 , Yuan Lu1
, Yuan Lu1

1 Department of Chemical Engineering, Tsinghua University, Beijing 100084, China;
2 College of Life Sciences, Shenyang Normal University, Shenyang 110034, Liaoning, China
Received: May 12, 2018; Accepted: June 28, 2018; Published: July 14, 2018
Supported by: National Natural Science Foundation of China (No. 21706144)
Corresponding author:Ning Bu. E-mail: buning@sohu.com
Yuan Lu. Tel/Fax: +86-10-62780127; E-mail: yuanlu@tsinghua.edu.cn
Abstract: Cell-free unnatural protein synthesis (CFUPS) as an emerging approach for protein engineering research, has been successfully applied in basic scientific studies (e.g., protein-protein interaction, protein-nucleic acid interaction) and industrial production (e.g., pharmaceutical proteins, protein materials). CFUPS can improve the engineering freedom and the process control by allowing free addition of genetic elements and chemicals as research purposes. It also can give protein novel characteristics, structures and functions, including post-translational modification of proteins, incorporation of reaction handles, synthesis of biophysical probes and polymeric protein. This article systematically reviews the unnatural amino acid incorporation methods (global suppression, stop codon suppression, frameshift suppression and unnatural base-pairs) and the application advances of unnatural amino acids in protein modifications, biophysical probes, enzyme engineering, biomaterials and biopharmaceutical protein production. The opportunities and challenges of the CFUPS system development and the wide application of industrialization are also illustrated with details.
Keywords: cell-free synthetic biology protein engineering unnatural amino acids (UNAAs) unnatural proteins
蛋白质作为重要的生物大分子,是所有生物体维持结构、功能的必需物质。自然状态下,20种天然氨基酸(Natural amino acids, NAAs)可产生结构功能多样的天然蛋白质,但这些天然蛋白质已不能满足蛋白质工程及蛋白药物生产等领域的研究需要,而带有新型侧链基团的非天然氨基酸(Unnatural amino acids, UNAAs)[1]的嵌入可赋予蛋白质新的化学性质、结构及功能[2],打开了新蛋白质工程的大门,为生物研究、生物治疗学及合成生物学提供了新的途径,已成为这些领域中关键的新兴应用[3]。在基础科学研究领域,将其用于蛋白质结构及动力学研究,利用原核生物无细胞蛋白表达系统实现了真核生物蛋白翻译后修饰[4];一些可交联的UNAAs已被嵌入蛋白质来表征蛋白质之间配体-受体[2]及蛋白质与核酸之间的相互作用[5];增强蛋白质的光学及热学稳定性,用于构建纳米开关[1]等。在工业化生产研究领域,非天然蛋白质可作为新型生物高分子材料、蛋白药物及共价大分子抑制剂,为材料科学和医药蛋白的开发与生产创造了新的契机,如蛋白质聚合物[6]、硒蛋白[7]、磷酸化蛋白质[8]及抗体-药物结合物[9]的合成等。因此,合成嵌入UNAAs的非天然蛋白质是蛋白质工程发展中具有革命性的一步。
探究历史,非天然蛋白质的合成[10]具有里程碑意义的进展要追溯到1989年,Schultz团队首先通过琥珀抑制的方法将合成的UNAAs嵌入到β-内酰胺酶中,并发现嵌入多种不同UNAAs,可促进对蛋白质结构及功能的研究[11]。自此,蛋白质中嵌入UNAAs的方法得到了快速发展。目前较成熟的方法主要包括两大类,分别是基于天然翻译体系的全局抑制方法及基于正交翻译体系(Orthogonal translation systems, OTSs)的琥珀抑制、移码抑制、有义密码子再分配及非天然碱基对的嵌入方法[12]。
最初,非天然蛋白质主要依托细胞体系,但需要维持细胞自身良好的生长状态,以合成目标蛋白。由于细胞内代谢通路复杂,而目标蛋白合成途径非细胞内主反应,导致产物产量较低,纯化步骤繁琐;且某些UNAAs不易穿过细胞膜屏障进入细胞,并会对细胞产生不同程度的毒性,从而降低了细胞对UNAAs的利用率,限制UNAAs嵌入效率;同时,由于细胞膜的阻碍,反应的可调控性能较差。这些导致体内UNAAs的嵌入效率较低,特别是在多位点UNAAs嵌入时尤为明显。但随着无细胞蛋白合成系统的出现及日渐成熟[13-15],无细胞非天然蛋白质合成系统(Cell-free unnatural protein synthesis, CFUPS) (图 1)应运而生。CFUPS以外源DNA或mRNA为模板,向反应体系中添加底物、能量物质、多种辅因子和非天然组分,包括OTS中的正交氨酰tRNA合成酶/正交tRNA (o-aaRS/o-tRNA)对及UNAAs等,在细胞提取物提供的多种酶的作用下合成非天然蛋白质。其中细胞提取物来源已从原核细胞扩大至真核细胞及哺乳动物,包括大肠杆菌[16]、酵母细胞[17]、小麦胚芽细胞[18]、昆虫细胞[19]和兔网织红细胞[20]。CFUPS与细胞非天然蛋白质合成相比具有诸多的优点(表 1)[12-13]:该系统生产周期短,整个生产过程仅需1-2 d;无细胞膜阻碍,规避了UNAAs选择性问题且不会对细胞产生毒性作用,利于重新分配遗传密码[2]和精细调控反应进程,进一步扩大蛋白质功能[12]。虽然CFUPS已得到极大发展,但同一条肽链上不同UNAAs的多位点嵌入,仍是该领域的瓶颈,在CFUPS和细胞体系均只能实现2-3个位点的嵌入[24-25];而同一条肽链上同种UNAAs的多位点嵌入已取得很大进展,均能实现10个或10个以上UNAAs[6, 22-23]的嵌入,但CFUPS中非天然蛋白质表达水平远远高于细胞体系。如Yanagisawa团队利用CFUPS实现了单一位点包含BocKme1 (Nε-(叔丁氧羰基)-Nε-甲基-1-赖氨酸)的人组蛋白H3的高于细胞体系约10倍的表达[21];另外Yanagisawa团队[21]也对基于E. coli的细胞及无细胞体系中人组蛋白H3变体的表达量进行比较,结果发现当嵌入2个BocKme1时,CFUPS的H3变体表达量高于细胞体系约70倍。因此,CFUPS已逐渐成为强大的非天然蛋白质合成平台,目前已成功从实验室投入到实际生产应用中,用于抗体-药物结合物[9]、潜在的疫苗和显像剂、装饰性病毒样颗粒的生产[26]及酶的开发改造[27]等工业加工过程,但目前还未达到无细胞天然蛋白质合成的100 L的生产规模[28],因此要实现CFUPS的工厂化应用,仍需更多科研工作者的不懈努力。
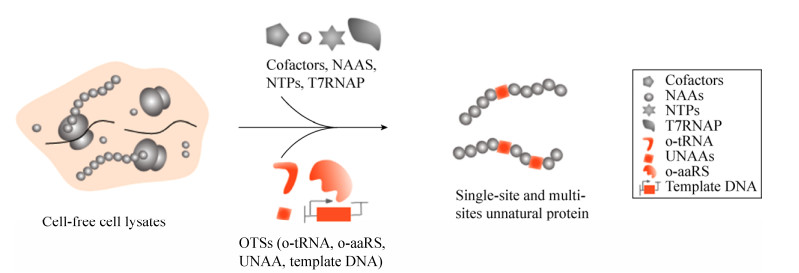 |
| 图 1 无细胞非天然蛋白质合成系统 Figure 1 Cell-free unnatural protein synthesis system. |
| 图选项 |
表 1 无细胞及细胞非天然蛋白质合成系统比较Table 1 Comparison of cell-free and cellular protein synthesis system
| Characteristics | Cell-free system | Cellular system |
| Design-build-test cycle | 1-2 days | 1-2 weeks |
| Cell membrane permeability of UNAAs | No hindrance | Difficult for UNAAs with charges or bulky groups |
| Toxicity of unnatural components at high concentrations | Low | High |
| Regulation of transcription and translation | Easy | Hard |
| Different UNAAs in the same peptide chain | 3[25, 43] | 2[24, 29] |
| Yield (mg/L)a | 430 | 44.5 |
| Downstream process | Easier | Redundancy |
| a: refers to the expression of human histone H3 with single BocK36me1 in E. coli-based and cell-free unnatural protein synthesis system[21]. | ||
表选项
本文首先介绍了CFUPS中UNAAs的不同嵌入方法,主要包括基于天然翻译体系的全局抑制方法和基于OTSs的终止密码子抑制、移码抑制、有义密码子再分配和非天然碱基对方法,以及它们应用的优势及挑战,接着对CFUPS的主要应用进行回顾,最后分析了当前CFUPS嵌入方法面临的挑战及可能的应对策略。
1 无细胞体系UNAAs嵌入的方法CFUPS作为合成生物学新兴的研究手段,已发展为强大的UNAAs嵌入平台,用来改进天然蛋白质的结构及功能特性[2]。目前,该体系采用的UNAAs嵌入方法主要包括基于天然翻译体系的全局抑制,以及基于OTSs的终止密码子抑制、移码抑制、有义密码子再分配和非天然碱基对的方法,本节会对这些方法的发展应用进行详细论述。
1.1 全局抑制全局抑制(Global suppression)是最早发展的UNAAs嵌入方法(图 2 A-a, b),其利用天然蛋白质翻译体系合成嵌入UNAAs的非天然蛋白质。全局抑制以一种(或多种)特定氨基酸的营养缺陷型菌株为宿主菌,向反应体系中补充菌株所缺陷氨基酸的非天然类似物,在天然翻译机制的背景下,这种非天然类似物即可取代该天然氨基酸的位点,在全局范围内实现UNAAs嵌入,从而改变蛋白质的理化性质及功能(图 2A)[2]。早在60多年前,Cohen及其同事成功在大肠杆菌细胞内用蛋氨酸类似物SeMet全局替代Met[30]。但分子量大或带电荷的UNAAs很难穿过细胞膜屏障,这大大限制了体系对UNAAs的利用率,且某些UNAAs本身或一定量的累积会对细胞产生毒性作用,甚至使细胞致死[31]。因此研究者们发展了无细胞膜阻碍的CFUPS,在使用CFUPS嵌入UNAAs时,首先要制备营养缺陷型细胞的提取物[31],再添加反应所需的NAAs (除去被替换的NAA)及UNAAs。若未除尽体系中所有将被替换的NAA,即会引起该种NAA与其非标准类似物之间的竞争,使UNAAs与NAAs按照两者的浓度比率嵌入蛋白质[12]。Singh-Blom等使用色氨酸枯竭型CFUPS,表达了具有生物活性的含有色氨酸类似物5-氟色氨酸或6-氟色氨酸修饰的链霉抗生物素蛋白,且嵌入效率高达100%[31],为UNAAs的广泛嵌入提供了新的可能。全局抑制利用天然翻译机制,不需进化翻译组分和改变目标蛋白遗传信息,即可实现多个位点一种(或多种) UNAAs的高效嵌入,且不会生成截短的蛋白质;另外,基于无细胞体系的开放性和自由性,无UNAAs的毒性及运输问题,可提高体系对UNAAs的利用效率。但较难实现位点特异性UNAAs嵌入。
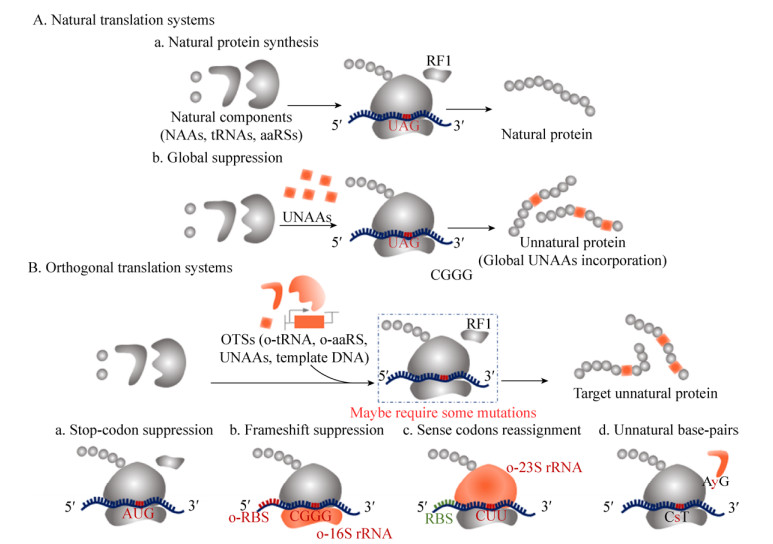 |
| 图 2 无细胞体系中UNAAs嵌入方法 Figure 2 Methods of UNAAs incorporation in cell-free system. |
| 图选项 |
1.2 终止密码子抑制在蛋白质工程领域,有时需要对一个(或几个)位点进行特定的修饰,以监测及表征蛋白质的某些结构及功能特性,但全局抑制无法实现UNAAs的位点特异性嵌入,因此研究者们发展了一套位点特异性嵌入UNAAs的方法。Schultz及其同事首先在大肠杆菌提取物中,将UNAA嵌入蛋白质中被琥珀终止密码子取代的有义密码子位点[11],称为终止密码子抑制(Stop codon suppression)方法(图 2 B-a)。在天然翻译体系中,64个三联密码子中的3个终止密码子赭石(UAA)、乳白(UGA)和琥珀(UAG)通常不编码任何氨基酸,而是与释放因子识别,终止翻译过程。但在某些生物体天然翻译机制中,终止密码子可编码特定的氨基酸[32],如詹氏甲烷球菌Methanococcus jannaschii的终止密码子(UAG)可编码酪氨酸[33],研究者们由此发展了终止密码子抑制的方法,即用3个终止密码子编码UNAAs,从而合成非天然蛋白质[12]。应用该方法进行UNAAs嵌入常基于詹氏甲烷球菌的OTS,包括某种UNAA及与其正交的o-tRNA/o-aaRS对,有时还包括正交延伸因子(o-EF-Tu)及正交核糖体(o-Ribosome)。为实现及提高UNAAs嵌入的精准度,关键的一点是进化OTS,使其可不再识别NAAs,只特异性识别正交的UNAA[35]。OTSs的正交性越强,NAAs在tRNACUA上的交叉装载的频率越低,提高UNAAs嵌入效率[12]。但在原核生物中,释放因子1 (RF1)会特异性识别琥珀密码子(UAG),与o-tRNA产生竞争,从而产生截短的蛋白质,使嵌入效率降低。考虑到这些因素,Lajoie构建了一株将基因组中所有编码UAG终止密码子(321个TAG)突变成TAA,且敲除prfA基因的重组大肠杆菌,在该菌株中琥珀密码子与OTS完全正交,并不被本体的翻译机制识别,且不会产生截短的蛋白质,从而进一步提高了UNAAs嵌入效率[36]。Hong等在去除RF1重编码的大肠杆菌CFUPS中成功实现单位点和多位点对-炔丙氧基-L-苯丙氨酸(p-propargyloxy-L-phenylalanine, pPaF)的嵌入,与含有RF1的CFUPS相比,单一位点嵌入pPaF的超折叠绿色荧光蛋白(Superfolder green fluorescent protein, sfGFP)产量高出2.5倍[22]。此外,热敏RF1突变体的热失活以及利用抗体抑制RF1已成功应用于CFUPS系统[37]。但宿主菌RF1的敲除,通常会影响细胞提取物活性,降低CFUPS蛋白产量,限制其广泛应用。而最近,Jewett利用菌株改造工程,以C321.ΔA[36]为出发菌,构建了5种核酸酶(rna、rnb、mazF、endA和rne)、两种蛋白酶(ompT和lon)以及8种影响氨基酸、能量和氧化还原稳定性的基因(gdhA、gshA、sdaA、sdaB、speA、tnaA、glpK和gor)功能单一或组合失活的重组菌株,建立了RF1敲除的高产CFUPS平台,其中,C321.ΔA.759粗提物中单位点4-乙酰基-L-苯基丙氨酸(4-Acetyl-L-phenylalanine, pAcF)嵌入的sfGFP表达量比通用的BL21 Star (DE3)粗提取物提高92%,高于MCJ.559 (C13.ΔA endA csdA- (C13: 13 TAG再编码为TAA))粗提物145%[38],实现了高嵌入效率及高非天然蛋白质产量的双重目标。终止密码子抑制在一定程度上扩大了生物体遗传编码,与重编码菌株细胞提取物手段相结合,可有效提高UNAAs嵌入效率。但终止密码子数量有限,导致可嵌入的UNAAs数量有限,此外,OTSs的正交性也是限制UNAAs嵌入效率的主要因素。因此扩大UNAAs的遗传编码和进化OTSs是提高嵌入效率的必然选择。
1.3 移码抑制为实现单(或多)位点特异性UNAAs嵌入,研究者们发展了移码抑制(Frameshift suppression) (图 2 B-b)的嵌入方法,扩大了UNAAs的密码子库,提高了嵌入效率,也解决了单一蛋白中多个特异位点UNAAs嵌入问题。移码抑制是基于含有一个扩大的反密码子环的移码抑制tRNA (Frameshift suppressor tRNA)导致密码子被超过3个核碱基的反密码子解码的现象[12]。通过该方法,四碱基[39]甚至五碱基密码子[40]已被成功地用于CFUPS中非天然蛋白质合成。Hohsaka及其同事在CFUPS中用CGGG四联密码子成功将丹酰化非天然氨基酸(Dansylated non-natural amino acids)嵌入链霉亲和素,这也是第一次将相对较大的丹酰化氨基酸嵌入蛋白质,且比琥珀抑制具有更高的嵌入效率[41]。此外,该方法适用的细胞粗提物来源已从大肠杆菌扩展到昆虫[19]和兔网织红细胞[42]。通过使用不同的四联密码子和它们的同源预氨酰化tRNAs的方法,已将2个[39]或3个[43]不同的UNAAs成功嵌入目标蛋白。目前移码抑制的密码子也扩展至五联密码子,Hohsaka等在大肠杆菌CFUPS中使用五联密码子CGGUA,并添加预氨酰化的tRNAUACCG,成功在链霉菌抗生物素蛋白mRNA的Tyr54位点嵌入对-硝基苯丙氨酸(p-nitrophenylalanine, nitroPhe),产生了全长的链霉菌抗生物素蛋白,且CGGN1N2 (N表示任意核糖核苷酸)的另外15个密码子也被其互补的预化学酰化的移码抑制tRNA成功解码[40]。尽管移码抑制已很大程度扩展了UNAAs的遗传编码,但天然核糖体识别四联或五联反密码子tRNA的能力有限,解码效率不高,同时可能会导致蛋白质组的错读与蛋白质的错误合成,产生截短的蛋白质,因此需要对天然的核糖体进行改造,使它朝着更有效解码四联体密码子的方向进化[44]。
1.4 有义密码子再分配鉴于遗传密码的简并性和tRNA反密码子对密码子识别的摆动性,30–40个有义密码子足以编码生物体的遗传信息,因此,超过20个有义密码子是潜在的可重新分配的密码子[45],这种为有义密码子重新分配其他NAAS (或UNAAs)的方法即为有义密码子再分配(图 2 B-c),目前使用该方法已成功将150多种UNAAs嵌入蛋白质中[44]。如在酵母线粒体中将CUN密码子(N代表A、U、G、C)从编码亮氨酸转变为编码苏氨酸[45],这种重编码也需要突变新的正交tRNA/aaRS对。而CFUPS在遗传密码再分配领域具有一定优势,纯化组分[46]和天然tRNAs缺失的细胞粗提物的无细胞翻译系统[47],均可选择性地提供纯化的tRNAs,从而基本上创建一个可自定义遗传密码的平台,其中选中的密码子保留“空白”以重新分配UNAAs[48]。Iwane等[49]构建了含有32个tRNASNN (S = G或C)的体外转录物的CFUPS,利用预先装载UNAAs的tRNASNNs代替可装载NAAs的冗余tRNASNNs,实现了3个冗余密码子的再分配,并成功表达了由20个NAAs和3个UNAAs组成的线性肽的32聚体。或者,Terasaka等突变50S核糖体亚基的肽基转移酶中心位点,消除其利用某些天然tRNAs进行翻译的能力[50]。将这种核糖体与含补偿位点突变的tRNA一起使用,可进行遗传密码重编码,为遗传密码重写和非天然蛋白质合成提供潜在新策略。
1.5 非天然碱基对上述的UNAAs嵌入方法均依赖于天然碱基组成的密码子-反密码子相互作用,使用无义抑制tRNA[51]或四联抑制tRNA[52]嵌入UNAAs,但是天然碱基数量有限,使这些方法在多种不同的UNAAs嵌入蛋白质的效率受到限制,且翻译过程中抑制tRNA与释放因子或天然tRNA之间的竞争也会降低UNAAs嵌入效率[53]。在这些方面,使用非天然碱基对(Unnatural base-pairs,UBPs) (图 2 B-d)扩大UNAAs的遗传编码更有优势[18],UBPs是人工设计合成的可行使或模拟天然核酸功能、而又具有相对独立性的人造碱基对,在翻译过程中,可将UNAAs分配给含有非天然碱基的额外密码子[18]在蛋白特定位点嵌入UNAAs。Bain研究组成功实现了非天然碱基对isoG-isoC的DNA的体外复制和转录[54],并借助人工合成含有反密码子CU(isoG)的tRNA,在体外翻译系统中成功将3-碘化酪氨酸嵌入到一段小肽中[55]。Hirao开发了2-氨基-6-(2-噻吩基)嘌呤(用s表示)和吡啶-2-酮(用y表示)的UBPs (s-y),该UBPs可通过聚合酶链式反应(Polymerase chain reaction, PCR)嵌入DNA或RNA的特异位点,可在T7转录的大肠杆菌CFUPS中将3-氯酪氨酸(3-chlorotyrosine)嵌入到人Ras蛋白的32位点[18]。Romesberg团队发展出了一对较为优异的非天然碱基对dNaM-dTPT3[54],其复制的保真性已基本接近天然碱基[56]。但非天然碱基有时结构不稳定,会在异构体间相互转换,且某些在复制和转录过程中仍有较低保真性[57],导致正确的模板数量较少,从而影响UNAAs嵌入效率,因此,提高UBPs本身的化学稳定性及其与模板复制、转录及翻译过程中各组分的正交性尤为重要[58]。
2 正交翻译体系优化迄今,通过OTSs嵌入到目标蛋白中的UNAAs已达150余种,在蛋白质的标记、稳定性改变、定位监测、酶活性提高及细胞信号传导等方面有着广泛的应用[44]。OTSs包括某种UNAA及与其正交的o-tRNA/o-aaRS对、延伸因子及核糖体。为提高嵌入效率,必须确保这些组分与宿主翻译机器的正交性(指o-tRNA不携带任何NAAs;o-aaRS不能氨酰化任何内源性tRNAs;o-aaRS不能接受任何NAAs作为底物),o-tRNA/o-aaRS对通常来源于亲缘关系遥远的生物[59],通过定向进化以改善其与UNAAs的兼容性,实现特异位点嵌入。而进化的o-aaRS酰化活性通常低于其天然祖先,非天然蛋白质的合成可基于OTSs,使用终止密码子抑制、移码抑制、有义密码子再分配和非天然碱基对的方法在特异位点高效嵌入UNAAs。因为CFUPS与OTSs的兼容性要明显优于细胞系统,所以正交翻译方法通常应用在无细胞非天然蛋白质合成体系中。但与其天然祖先相比,工程化的o-aaRSs通常表现出较低的酰化活性,尤其是对非同源抑制tRNA[45, 60-61],而CFUPS的开放环境允许定量添加纯化的o-aaRS、o-tRNA和UNAAs,可通过调整正交翻译组分浓度来提高UNAAs嵌入效率;在真核CFUPS中添加大肠杆菌来源的OTSs时,增加Mg2+浓度有助于UNAAs嵌入[62]。此外,细胞生长过程中o-aaRS和o-tRNA的过表达及UNAAs的补充常导致细胞毒性,而CFUPS不需依赖完整的活细胞,也就规避了这一问题,可优先高效合成嵌入UNAAs的非天然蛋白质,甚至难溶性蛋白质[5]。
虽然目前OTSs在CFUPS中已得到了很好的应用,但由于正交翻译组分在不同嵌入方法间的适应性不同,为提高UNAAs嵌入效率,需更有效进化包括o-aaRSs、o-tRNAs、核糖体及EF-Tu在内的OTSs。首先需开发更有效的o-aaRS/o-tRNA进化筛选及表达优化平台,提高o-aaRS与o-tRNA的正交性和表达水平,以提高非天然蛋白质产量[45]。另外,终止密码子抑制中,需提高抑制tRNA与终止密码子的识别效率,抑制或去除RF1与终止密码子的识别,提高抑制tRNA的通读效率[23]。移码抑制中,可进化核糖体的16S rRNA及模板链上的核糖体结合位点,以提高对四联或五联密码子的解码效率,避免出现蛋白质组错读的现象[63]。为进一步提高有义密码子再分配UNAAs的嵌入效率,可寻找并利用具有开放密码子(基因组中未使用的密码子)的天然物种或创建合成生物体,还可开发有义密码子重新分配筛选方法来测试o-tRNA/o-aaRS对的正交性,以开发出正交性强或完全正交的o-tRNA/o-aaRS对[45]。使用UBPs时,由于非天然碱基对本身结构不稳定,因此,提高非天然碱基对稳定性及其与模板复制、转录及翻译过程中各组分的正交性是提高UNAAs嵌入效率的主要途径。这些优化将是非天然蛋白质合成领域的重大进步,也将为基于正交翻译体系CFUPS创造更广阔的应用前景。
3 无细胞合成非天然蛋白质的应用3.1 蛋白质修饰蛋白质位点特异性修饰(图 3)是蛋白质标记和选择性添加结构元素的基本技术,已经成为探测蛋白质结构和功能以及产生具有增强的或新的特性蛋白质的有力工具。蛋白质修饰的理想状态是获得均一的同质混合物,但通常的偶联反应涉及用亲电试剂修饰赖氨酸、半胱氨酸或丝氨酸的亲核侧链[64],通常会产生不同性质蛋白质缀合物的异质混合物;另外,也可通过酶催化的生物反应或化学反应用化学标签[65]修饰特定多肽序列,或用半合成方法表达蛋白质连接(Expressed protein ligation, EPL)[66],将正交化学反应官能团引入蛋白质中,以精确控制结合位点和化学计量比,但后续处理过程较复杂。而CFUPS平台的应用可基本解决这些问题。将含有酮基、叠氮基、炔基和磷酸基等正交化学反应性侧链的UNAAs嵌入蛋白质的特异位点,可合成结构明确的蛋白质缀合物[4],除实现对结合位点和反应化学计量比的精确控制外,也可优化蛋白质缀合物的物理和生物学性质[59],是一种极具吸引力的蛋白质修饰方式。如Ohno基于大肠杆菌CFUPS,将酵母琥珀抑制tRNATyr/突变的酪氨酰-tRNA合成酶对作为3-叠氮基酪氨酸(3-azidotyrosine)的载体,以大鼠钙调蛋白(CaM)为模型蛋白,分别获得了在72、78、80或100氨基酸位点处携带叠氮基酪氨酸的几个非天然CaM分子,用三芳基膦和生物素的缀合化合物对这些蛋白进行翻译后修饰,产生了位点选择性生物素化的CaM分子[67]。
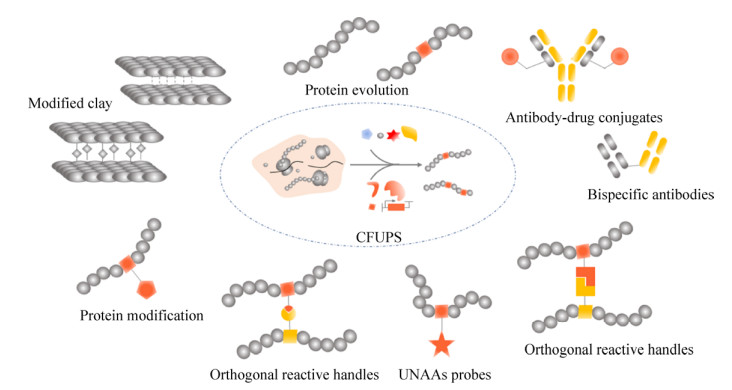 |
| 图 3 CFUPS中UNAAs应用 Figure 3 Applications of UNAAs in CFUPS. |
| 图选项 |
另外,应用UNAAs对蛋白质进行翻译后修饰(Post-translational modifications, PTMs),也是UNAAs在蛋白质修饰方面的一个重要应用。天然存在的PTMs会影响蛋白质结构稳定及功能,有时也会对相应修饰蛋白的定位及相互作用产生影响,此外,不同PTMs信号之间会相互干扰,导致每种PTMs不能执行其特定的功能[68],而应用CFUPS嵌入UNAAs可作为这种问题的有效解决手段[12]。这种方法可将同质天然PTMs模拟物嵌入蛋白质中,对药物蛋白质的生产来讲,可规避细胞体系常伴随着的批次间的变化,如通过CFUPS将特定的PTMs (例如单糖基化和二糖基化的UNAAs)共翻译成蛋白质[69];或引入具有反应性手柄的UNAAs,对已合成的蛋白质进行翻译后修饰,如磷酸酪氨酸类似物[70-71]等。
3.2 蛋白质中的UNAAs探针UNAAs作为高灵敏度的生物物理探针(图 3),可结合到重组蛋白质的特定位点,用于目标蛋白质的定量检测及结构和功能的精确分析[31, 72]。由于在细胞提取物制备过程中损害了细胞本身的氨基酸代谢途径,并且可以使用特异性抑制剂,使同位素标记的干扰最小化[73];也可经过透析,去除部分内源的氨基酸,以减少对同位素标记的UNAAs的稀释。因此,相比细胞体系,CFUPS可以在使用最少量昂贵氨基酸条件下进行稳定的同位素标记,产生的含有同位素标记UNAAs的蛋白质,可用于FT-IR、NMR表征及MS定量检测[12]。除13C/15N标记的氨基酸外,19F[74]取代的UNAAs也可用于无背景NMR研究[31, 75];此外,重原子取代的氨基酸的使用也大大促进了X射线晶体结构测定[76]。Abe以脱硫弧菌黄素单核苷酸(FMN)结合蛋白作为模型蛋白,应用基于小麦胚芽的CFUPS,将3-叠氮基-L-酪氨酸(3-azido-L-tyrosine)嵌入FMN结合位点的Tyr35残基位点,并通过荧光素-三芳基膦衍生物对该UNAA进行特定的化学修饰,检测辅因子结合蛋白的结构变化[77]。
另一类重要的生物物理探针即荧光UNAAs。荧光探针的高灵敏度性使其成为蛋白质工程中普遍使用的研究方法[12]。荧光UNAAs可用于快速测定蛋白质产量,无放射性标记,因此不需要液体闪烁计数及在磷光屏上曝光[78]。如将荧光性UNAA (BODIPYFL)嵌入烟碱型乙酰胆碱受体,可以对蛋白进行单分子荧光检测[79]。在特异位点嵌入荧光探针也可用于探测蛋白质定位、构象和蛋白质之间相互作用的信息[80],将4-氰基苯丙氨酸和4-乙炔基苯丙氨酸等UNAAs嵌入到蛋白质中,利用这些氨基酸的固有荧光研究蛋白质分子内过程,如构象变化和蛋白质折叠[81]。Kirill使用体外预先装载BODIPY-tRNACCUCys将BODIPY荧光团引入钙调蛋白(CaM),同时,通过琥珀抑制方法在相同的多肽上嵌入对叠氮基苯丙氨酸(p-azido-l-phenylalinine,pAzF)及其与四甲基罗丹明二苯并环辛烯(TAMRA-DIBO)的翻译后缀合,通过一个翻译后衍生步骤成功引入两个荧光标记,并通过单分子FRET分析监测CaM在暴露于Ca2+或螯合剂时构象的显著变化[82]。引入多种荧光UNAAs还可实现对蛋白质构象和完整性的研究[12]。Anderson等使用琥珀抑制和四碱基解码的混合方法,可通过FRET监测蛋白质完整性[83];Nataka及其同事使用荧光UNAAs相关光谱筛选MMP-9抑制剂[84];Sisido小组通过将一对供体淬灭剂UNAAs嵌入链霉亲和素中,得到了局部蛋白质结构的信息[85];此外,嵌入UNAAs也可监测生物素在链霉亲和素上的结合结果[72, 86]。
3.3 蛋白质进化传统的酶或蛋白质工程依赖于用其余的19种NAAs中的一种取代某一位点的某种氨基酸来改变酶或蛋白质的功能特性[87],嵌入UNAAs的工程技术开始产生具有更高稳定性和改变催化性质的酶[87]。近年,残基特异性和位点特异性嵌入方法正在成为产生具有改变或改善功能酶的优选方法。嵌入UNAAs能够提高半胱氨酸蛋白酶底物结合灵敏度[88]。细菌磷酸三酯酶以很快的速率催化农药氧磷的水解,并被认为接近其进化极限。Ugwumba等[89]为了测试是否可以通过嵌入UNAAs来改善其自然进化的更新率,及探测外周活性位点残基在催化循环过程非化学步骤中的作用(底物结合和产物释放),在CFUPS中,用非天然的L-(7-羟基香豆素-4-酰)乙基甘氨酸(Hco)和L-(7-甲基香豆素-4-酰)乙基甘氨酸(Mco)以及天然的亮氨酸、苯丙氨酸和色氨酸取代309位天然存在的酪氨酸。动力学分析表明Hco的7-羟基,特别是当其处于去质子化状态时,有助于促进底物转化的限速产物释放步骤,提高其天然活性8-11倍。这些结果表明,UNAAs为已有的酶工程和进化提供了新的有价值功能空间。
3.4 蛋白质药物过去十年中,基于非天然蛋白质药物的研究在生物医药领域受到高度关注,且已被应用于临床规模药物蛋白及疫苗的生产[90]。例如利用UNAAs形成共价的蛋白质-蛋白质和蛋白质-小分子连接,形成均匀的抗体-药物结合物,能够增强对靶向细胞的有效性[91]。此外,Patel及其同事创造了含有炔丙基甘氨酸的萤光素酶变体以及携带pAzF的scFv与IM9蛋白的融合构建体。然后研究人员将IM9-scFv融合蛋白与萤光素酶偶联,并将所得的偶联物用于体内检测B细胞淋巴瘤细胞[92]。Swartz及其同事开发了一种用于生产装饰性病毒样颗粒的新型流水线,可用作潜在的疫苗和显像剂[26]。此外,抗体药物结合物(Antibody-drug conjugates, ADCs)目前已经成为靶向高效递送细胞毒性药物治疗肿瘤的有效方法,已有超过40种ADCs处于临床开发阶段[93],且有两个高效靶向递送细胞毒性药物治疗肿瘤的ADSs已通过监管部门的审批[94]。Zimmerman等在CFUPS中成功表达含有pAzF的曲妥珠单抗变体,随后将其与带有DBCO的MMAF药物部分偶联,合成位点特异性ADC,产量达到约250 μg/mL,且经细胞毒性测试为有效[9]。Ugwumba等用非天然氨基酸Hco标记西尼罗河病毒非结构蛋白酶,该酶对蛋白抑制剂及小配体分子非常敏感,可用于探测蛋白质鉴定新的抑制剂,使其成为发现新药的有力工具[95]。这些进展表明了CFUPS在生产新型治疗药物(图 3)方面是实用且有效的,目前已成为新药研发过程中快速而有效的工具之一。
3.5 蛋白质材料UNAAs嵌入产生新型序列明确的聚合物以用于生物材料合成的多功能应用。Albayrak和Swartz报道了在CFUPS中含有2个或3个位点特异性嵌入UNAAs的蛋白质的直接聚合,其允许铜催化的叠氮化物-炔烃环加成形成线性或分支的蛋白质聚合物[6]。Maza等合成9种UNAAs,通过与荧光团或衍生树脂反应分析不同的生物复合物的最佳连接臂长度,优化了制备生物复合物的参数[96]。Katti等将3种具有较长侧链的UNAAs ((±)-2-氨基庚二酸、5-氨基戊酸和DL-2-氨基辛酸)作为改性剂修饰钠蒙脱石(Na-MMT)粘土,X射线衍射结果表明,在用3种UNAAs修饰后,Na-MMT粘土的层间距离增加;细胞培养实验显示用3种氨基酸修饰的Na-MMT粘土仍保持了原有的生物相容性[97]。
4 结论与展望无细胞非天然蛋白质合成系统作为灵活嵌入非天然氨基酸的强大技术平台,已成为细胞内非天然蛋白质合成的重要补充手段之一。无细胞非天然蛋白质合成解决了UNAAs的跨膜及胞内运输障碍、细胞生长毒性问题,目前已成功合成蛋白药物、病毒样颗粒、膜蛋白和蛋白质材料等多领域的非天然功能蛋白质,在指导细胞内非天然蛋白质合成体系及研究蛋白质间相互作用、开发蛋白质药物、新型蛋白材料方面发挥着至关重要的作用。将来无细胞非天然蛋白质合成系统需要朝着4个方面发展,分别是:1)开发更有效的OTSs,提高非天然氨基酸的嵌入效率;2)实现在目标蛋白的多个位点嵌入多种UNAAs,扩大非天然蛋白质的种类;3)解决UNAAs多样性带来的生物不相容性,使非天然蛋白质的产量得到大幅度的上升;4)进一步降低非天然蛋白质合成的成本,使其能够带来可观的经济效益,促进工业化应用。最大限度去开发无细胞非天然蛋白质合成平台的潜力,使其能够更好地应用于科学基础研究及工业生产研究。
参考文献
| [1] | Wang L. Genetically encoding new bioreactivity.New Biotechnol, 2017, 38: 16–25.DOI: 10.1016/j.nbt.2016.10.003 |
| [2] | Soye BJD, Patel JR, Isaacs FJ, et al. Repurposing the translation apparatus for synthetic biology.Curr Opin Chem Biol, 2015, 28: 83–90.DOI: 10.1016/j.cbpa.2015.06.008 |
| [3] | Hoesl MG, Budisa N. Recent advances in genetic code engineering in Escherichia coli.Curr Opin Biotechnol, 2012, 23(5): 751–757.DOI: 10.1016/j.copbio.2011.12.027 |
| [4] | Liu CC, Schultz PG. Adding new chemistries to the genetic code.Annu Rev Biochem, 2010, 79(1): 413–444.DOI: 10.1146/annurev.biochem.052308.105824 |
| [5] | Hong SH, Kwon YC, Jewett M. Non-standard amino acid incorporation into proteins using Escherichia coli cell-free protein synthesis.Front Chem, 2014, 2: 34. |
| [6] | Albayrak C, Swartz JR. Cell-free co-production of an orthogonal transfer RNA activates efficient site-specific non-natural amino acid incorporation.Nucl Acids Res, 2013, 41(11): 5949–5963.DOI: 10.1093/nar/gkt226 |
| [7] | Br?cker MJ, Ho J ML, Church GM, et al. Recoding the genetic code with selenocysteine.Angew Chem, 2014, 53(1): 319–323.DOI: 10.1002/anie.v53.1 |
| [8] | Park HS, Hohn MJ, Umehara T, et al. Expanding the genetic code of Escherichia coli with phosphoserine.Science, 2011, 333(6046): 1151–1154.DOI: 10.1126/science.1207203 |
| [9] | Zimmerman ES, Heibeck TH, Gill A, et al. Production of site-specific antibody-drug conjugates using optimized non-natural amino acids in a cell-free expression system.Bioconjugate Chem, 2014, 25(2): 351–361.DOI: 10.1021/bc400490z |
| [10] | Zhang P, Lin J, Lu Y. Advances in cell-free protein synthesis.Chin J Bioprocess Engin, 2018, 16(1): 59–66.(in Chinese). 张鹏, 林骏, 卢元. 无细胞蛋白质合成的研究进展.生物加工过程, 2018, 16(1): 59-66.DOI:10.3969/j.issn.1672-3678.2018.01.007 |
| [11] | Noren CJ, Anthony-Cahill SJ, Griffith MC, et al. A general method for site-specific incorporation of unnatural amino acids into proteins.Science, 1989, 244(4901): 182–188.DOI: 10.1126/science.2649980 |
| [12] | Quast RB, Mrusek D, Hoffmeister C, et al. Cotranslational incorporation of non-standard amino acids using cell-free protein synthesis.FEBS Lett, 2015, 589(15): 1703–1712.DOI: 10.1016/j.febslet.2015.04.041 |
| [13] | Lu Y. Cell-free synthetic biology: engineering in an open world.Synthet Syst Biotechnol, 2017, 2(1): 23–27.DOI: 10.1016/j.synbio.2017.02.003 |
| [14] | Welsh JP, Lu Y, He XS, et al. Cell-free production of trimeric influenza hemagglutinin head domain proteins as vaccine antigens.Biotechnol Bioeng, 2012, 109(12): 2962–2969.DOI: 10.1002/bit.v109.12 |
| [15] | Lu Y, Chan W, Ko BY, et al. Assessing sequence plasticity of a virus-like nanoparticle by evolution toward a versatile scaffold for vaccines and drug delivery.Proc Natl Acad Sci USA, 2015, 112(40): 12360–12365.DOI: 10.1073/pnas.1510533112 |
| [16] | Goerke AR, Swartz JR. High-level cell-free synthesis yields of proteins containing site-specific non-natural amino acids.Biotechnol Bioeng, 2009, 102(2): 400–416.DOI: 10.1002/bit.v102:2 |
| [17] | Hodgman CE, Jewett MC. Characterizing IGR IRES-mediated translation initiation for use in yeast cell-free protein synthesis.New Biotechnol, 2014, 31(5): 499–505.DOI: 10.1016/j.nbt.2014.07.001 |
| [18] | Hirao I, Ohtsuki T, Fujiwara T, et al. An unnatural base pair for incorporating amino acid analogs into proteins.Nat Biotechnol, 2002, 20(2): 177–182.DOI: 10.1038/nbt0202-177 |
| [19] | Taki M, Tokuda Y, Ohtsuki T, et al. Design of carrier tRNAs and selection of four-base codons for efficient incorporation of various nonnatural amino acids into proteins in Spodoptera frugiperda 21 (Sf21) insect cell-free translation system.J Biosci Bioeng, 2006, 102(6): 511–517.DOI: 10.1263/jbb.102.511 |
| [20] | Li SW, Millward S, Roberts R. In vitro selection of mrna display libraries containing an unnatural amino acid.J Am Chem Soc, 2002, 124(34): 9972–9973.DOI: 10.1021/ja026789q |
| [21] | Yanagisawa T, Takahashi M, Mukai T, et al. Multiple site-specific installations of Nε-monomethyl-L-lysine into histone proteins by cell-based and cell-free protein synthesis.Chembiochem, 2014, 15(12): 1830–1838.DOI: 10.1002/cbic.201402291 |
| [22] | Hong SH, Ntai I, Haimovich AD, et al. Cell-free protein synthesis from a release factor 1 deficient Escherichia coli activates efficient and multiple site-specific nonstandard amino acid incorporation.ACS Synth Biol, 2014, 3(6): 398–409.DOI: 10.1021/sb400140t |
| [23] | Johnson DBF, Xu JF, Shen ZX, et al. RF1 knockout allows ribosomal incorporation of unnatural amino acids at multiple sites.Nat Chem Biol, 2011, 7(11): 779–786.DOI: 10.1038/nchembio.657 |
| [24] | Chen SX, Fahmi NE, Wang L, et al. Detection of dihydrofolate reductase conformational change by FRET using two fluorescent amino acids.J Am Chem Soc, 2013, 135(35): 12924–12927.DOI: 10.1021/ja403007r |
| [25] | Kajihara D, Abe R, Iijima I, et al. FRET analysis of protein conformational change through position-specific incorporation of fluorescent amino acids.Nat Methods, 2006, 3(11): 923–929.DOI: 10.1038/nmeth945 |
| [26] | Lu Y, Welsh JP, Chan W, et al. Escherichia coli-based cell free production of flagellin and ordered flagellin display on virus-like particles.Biotechnol Bioeng, 2013, 110(8): 2073–2085.DOI: 10.1002/bit.v110.8 |
| [27] | Ravikumar Y, Nadarajan SP, Hyeon YT, et al. Incorporating unnatural amino acids to engineer biocatalysts for industrial bioprocess applications.Biotechnol J, 2015, 10(12): 1862–1876.DOI: 10.1002/biot.v10.12 |
| [28] | Zawada JF, Yin G, Steiner AR, et al. Microscale to manufacturing scale-up of cell-free cytokine production—a new approach for shortening protein production development timelines.Biotechnol Bioeng, 2011, 108(7): 1570–1578.DOI: 10.1002/bit.23103 |
| [29] | Chatterjee A, Sun SB, Furman JL, et al. A versatile platform for single- and multiple-unnatural amino acid mutagenesis in Escherichia coli.Biochemistry, 2013, 52(10): 1828–1837.DOI: 10.1021/bi4000244 |
| [30] | Munier R, Cohen GN. Incorporation of structural analogues of amino acids in bacterial proteins.Biochim Biophys Acta, 1956, 21(3): 592–593.DOI: 10.1016/0006-3002(56)90207-4 |
| [31] | Singh-Blom A, Hughes RA, Ellington AD. An amino acid depleted cell-free protein synthesis system for the incorporation of non-canonical amino acid analogs into proteins.J Biotechnol, 2014, 178: 12–22.DOI: 10.1016/j.jbiotec.2014.02.009 |
| [32] | Ambrogelly A, Palioura S, S?ll D. Natural expansion of the genetic code.Nat Chem Biol, 2006, 3(1): 29–35. |
| [33] | Wang L, Brock A, Herberich B, et al. Expanding the genetic code of Escherichia coli.Science, 2001, 292(5516): 498–500.DOI: 10.1126/science.1060077 |
| [34] | Xie JM, Schultz PG. An expanding genetic code.Methods, 2005, 36(3): 227–238.DOI: 10.1016/j.ymeth.2005.04.010 |
| [35] | Normanly J, Kleina LG, Masson JM, et al. Construction of Escherichia coli amber suppressor tRNA genes: Ⅲ. Determination of tRNA specificity.J Mol Biol, 1990, 213(4): 719–726.DOI: 10.1016/S0022-2836(05)80258-X |
| [36] | Lajoie MJ, Rovner AJ, Goodman DB, et al. Genomically recoded organisms expand biological functions.Science, 2013, 342(6156): 357–360.DOI: 10.1126/science.1241459 |
| [37] | Agafonov DE, Huang YW, Grote M, et al. Efficient suppression of the amber codon in E. coli in vitro translation system.FEBS Lett, 2005, 579(10): 2156–2160.DOI: 10.1016/j.febslet.2005.03.004 |
| [38] | Martin RW, Des Soye BJ, Kwon YC, et al. Cell-free protein synthesis from genomically recoded bacteria enables multisite incorporation of noncanonical amino acids.Nat Commun, 2018, 9: 1203.DOI: 10.1038/s41467-018-03469-5 |
| [39] | Hohsaka T, Ashizuka Y, Taira H, et al. Incorporation of nonnatural amino acids into proteins by using various four-base codons in an Escherichia coli in vitro translation system.Biochemistry, 2001, 40(37): 11060–11064.DOI: 10.1021/bi0108204 |
| [40] | Hohsaka T, Ashizuka Y, Murakami H, et al. Five-base codons for incorporation of nonnatural amino acids into proteins.Nucl Acids Res, 2001, 29(17): 3646–3651.DOI: 10.1093/nar/29.17.3646 |
| [41] | Hohsaka T, Muranaka N, Komiyama C, et al. Position-specific incorporation of dansylated non-natural amino acids into streptavidin by using a four-base codon.FEBS Lett, 2004, 560(1/3): 173–177. |
| [42] | Taira H, Fukushima M, Hohsaka T, et al. Four-base codon-mediated incorporation of nonnatural amino acids into proteins in a eukaryotic cell-free translation system.J Biosci Bioeng, 2005, 99(5): 473–476.DOI: 10.1263/jbb.99.473 |
| [43] | Ohtsuki T, Manabe T, Sisido M. Multiple incorporation of non-natural amino acids into a single protein using tRNAs with non-standard structures.FEBS Lett, 2005, 579(30): 6769–6774.DOI: 10.1016/j.febslet.2005.11.010 |
| [44] | Dumas A, Lercher L, Spicer CD, et al. Designing logical codon reassignment-expanding the chemistry in biology.Chem Sci, 2015, 6(1): 50–69.DOI: 10.1039/C4SC01534G |
| [45] | O'donoghue P, Ling JQ, Wang YS, et al. Upgrading protein synthesis for synthetic biology.Nat Chem Biol, 2013, 9(10): 594–598.DOI: 10.1038/nchembio.1339 |
| [46] | Shimizu Y, Inoue A, Tomari Y, et al. Cell-free translation reconstituted with purified components.Nat Biotechnol, 2001, 19(8): 751–755.DOI: 10.1038/90802 |
| [47] | Ahn JH, Hwang MY, Oh IS, et al. Preparation method for Escherichia coli S30 extracts completely dependent upon tRNA addition to catalyze cell-free protein synthesis.Biotechnol Bioproc Eng, 2006, 11(5): 420–424.DOI: 10.1007/BF02932309 |
| [48] | Forster AC, Tan ZP, Nalam MNL, et al. Programming peptidomimetic syntheses by translating genetic codes designed de novo.Proc Natl Acad Sci USA, 2003, 100(11): 6353–6357.DOI: 10.1073/pnas.1132122100 |
| [49] | Iwane Y, Hitomi A, Murakami H, et al. Expanding the amino acid repertoire of ribosomal polypeptide synthesis via the artificial division of codon boxes.Nat Chem, 2016, 8(4): 317–325.DOI: 10.1038/nchem.2446 |
| [50] | Terasaka N, Hayashi G, Katoh T, et al. An orthogonal ribosome-tRNA pair via engineering of the peptidyl transferase center.Nat Chem Biol, 2014, 10(7): 555–557.DOI: 10.1038/nchembio.1549 |
| [51] | Cload ST, Liu DR, Froland WA, et al. Development of improved tRNAs for in vitro biosynthesis of proteins containing unnatural amino acids.Chem Biol, 1996, 3(12): 1033–1038.DOI: 10.1016/S1074-5521(96)90169-6 |
| [52] | Moore B, Persson BC, Nelson CC, et al. Quadruplet codons: implications for code expansion and the specification of translation step size.J Mol Biol, 2000, 298(2): 195–209.DOI: 10.1006/jmbi.2000.3658 |
| [53] | Short GF, Golovine SY, Hecht SM. Effects of release factor 1 on in vitro protein translation and the elaboration of proteins containing unnatural amino acids.Biochemistry, 1999, 38(27): 8808–8819.DOI: 10.1021/bi990281r |
| [54] | Li LJ, Degardin M, Lavergne T, et al. Natural-like replication of an unnatural base pair for the expansion of the genetic alphabet and biotechnology applications.J Am Chem Soc, 2014, 136(3): 826–829.DOI: 10.1021/ja408814g |
| [55] | Bain JD, Switzer C, Chamberlin R, et al. Ribosome-mediated incorporation of a non-standard amino acid into a peptide through expansion of the genetic code.Nature, 1992, 356(6369): 537–539.DOI: 10.1038/356537a0 |
| [56] | Seo YJ, Hwang GT, Ordoukhanian P, et al. Optimization of an unnatural base pair toward natural-like replication.J Am Chem Soc, 2009, 131(9): 3246–3252.DOI: 10.1021/ja807853m |
| [57] | Chen F, Dong MX, Ge M, et al. Artificial base and synthetic life.Bull Chin Acad Sci, 2016, 31(4): 457–466.(in Chinese). 陈非, 董梦醒, 葛猛, 等. 人造碱基与人工合成生命.中国科学院院刊, 2016, 31(4): 457-466. |
| [58] | Hirao I, Kimoto M. Unnatural base pair systems toward the expansion of the genetic alphabet in the central dogma.Proc Japan Acad, Ser B, 2012, 88(7): 345–367.DOI: 10.2183/pjab.88.345 |
| [59] | Kim CH, Axup JY, Schultz PG. Protein conjugation with genetically encoded unnatural amino acids.Curr Opin Chem Biol, 2013, 17(3): 412–419.DOI: 10.1016/j.cbpa.2013.04.017 |
| [60] | Albayrak C, Swartz JR. Using E. coli-based cell-free protein synthesis to evaluate the kinetic performance of an orthogonal tRNA and aminoacyl-tRNA synthetase pair.Biochem Biophys Res Commun, 2013, 431(2): 291–295.DOI: 10.1016/j.bbrc.2012.12.108 |
| [61] | Nehring S, Budisa N, Wiltschi B. Performance analysis of orthogonal pairs designed for an expanded eukaryotic genetic code.PLoS ONE, 2012, 7(4): e31992.DOI: 10.1371/journal.pone.0031992 |
| [62] | Quast RB, Claussnitzer I, Merk H, et al. Synthesis and site-directed fluorescence labeling of azido proteins using eukaryotic cell-free orthogonal translation systems.Analyt Biochem, 2014, 451: 4–9.DOI: 10.1016/j.ab.2014.01.013 |
| [63] | Wang KH, Neumann H, Peak-Chew SY, et al. Evolved orthogonal ribosomes enhance the efficiency of synthetic genetic code expansion.Nat Biotechnol, 2007, 25(7): 770–777.DOI: 10.1038/nbt1314 |
| [64] | Baslé E, Joubert N, Pucheault M. Protein chemical modification on endogenous amino acids.Chem Biol, 2010, 17(3): 213–227.DOI: 10.1016/j.chembiol.2010.02.008 |
| [65] | O'hare HM, Johnsson K, Gautier A. Chemical probes shed light on protein function.Curr Opin Struct Biol, 2007, 17(4): 488–494.DOI: 10.1016/j.sbi.2007.07.005 |
| [66] | Muir TW. Semisynthesis of proteins by expressed protein ligation.Ann Rev Biochem, 2003, 72(1): 249–289.DOI: 10.1146/annurev.biochem.72.121801.161900 |
| [67] | Ohno S, Matsui M, Yokogawa T, et al. Site-selective post-translational modification of proteins using an unnatural amino acid, 3-azidotyrosine.J Biochem, 2007, 141(3): 335–343. |
| [68] | Venne AS, Kollipara L, Zahedi RP. The next level of complexity: crosstalk of posttranslational modifications.Proteomics, 2014, 14(4/5): 513–524. |
| [69] | Fahmi NE, Dedkova L, Wang BX, et al. Site-specific incorporation of glycosylated serine and tyrosine derivatives into proteins.J Am Chem Soc, 2007, 129(12): 3586–3597.DOI: 10.1021/ja067466n |
| [70] | Serwa R, Wilkening I, Del Signore G, et al. Chemoselective staudinger-phosphite reaction of azides for the phosphorylation of proteins.Angew Chem Int Ed Engl, 2009, 48(44): 8234–8239.DOI: 10.1002/anie.200902118 |
| [71] | Serwa R, Majkut P, Horstmann B, et al. Site-specific PEGylation of proteins by a Staudinger-phosphite reaction.Chem Sci, 2010, 1(5): 596–602.DOI: 10.1039/c0sc00324g |
| [72] | Taki M, Hohsaka T, Murakami H, et al. A non-natural amino acid for efficient incorporation into proteins as a sensitive fluorescent probe.FEBS Lett, 2001, 507(1): 35–38.DOI: 10.1016/S0014-5793(01)02935-0 |
| [73] | Su XC, Loh CT, Qi R, et al. Suppression of isotope scrambling in cell-free protein synthesis by broadband inhibition of PLP enymes for selective 15N-labelling and production of perdeuterated proteins in H2O.J Biomol NMR, 2011, 50(1): 35–42.DOI: 10.1007/s10858-011-9477-5 |
| [74] | Neerathilingam M, Greene LH, Colebrooke SA, et al. Quantitation of protein expression in a cell-free system: efficient detection of yields and 19F NMR to identify folded protein.J Biomol NMR, 2005, 31(1): 11–19.DOI: 10.1007/s10858-004-5357-6 |
| [75] | Loscha KV, Herlt AJ, Qi RH, et al. Multiple-site labeling of proteins with unnatural amino acids.Angew Chem Int Ed Engl, 2012, 51(9): 2243–2246.DOI: 10.1002/anie.201108275 |
| [76] | Kiga D, Sakamoto K, Kodama K, et al. An engineered Escherichia coli tyrosyl-tRNA synthetase for site-specific incorporation of an unnatural amino acid into proteins in eukaryotic translation and its application in a wheat germ cell-free system.Proc Natl Acad Sci USA, 2002, 99(15): 9715–9720.DOI: 10.1073/pnas.142220099 |
| [77] | Abe M, Ohno S, Yokogawa T, et al. Detection of structural changes in a cofactor binding protein by using a wheat germ cell-free protein synthesis system coupled with unnatural amino acid probing.Prot: Struct, Funct, Bioinf, 2007, 67(3): 643–652.DOI: 10.1002/prot.21341 |
| [78] | Ozawa K, Loscha KV, Kuppan KV, et al. High-yield cell-free protein synthesis for site-specific incorporation of unnatural amino acids at two sites.Biochem Biophys Res Commun, 2012, 418(4): 652–656.DOI: 10.1016/j.bbrc.2012.01.069 |
| [79] | Zhang WH, Otting G, Jackson CJ. Protein engineering with unnatural amino acids.Curr Opin Struct Biol, 2013, 23(4): 581–587.DOI: 10.1016/j.sbi.2013.06.009 |
| [80] | Abe R, Jeong HJ, Arakawa D, et al. Ultra Q-bodies: quench-based antibody probes that utilize dye-dye interactions with enhanced antigen-dependent fluorescence.Sci Rep, 2014, 4: 4640. |
| [81] | Miyake-Stoner SJ, Miller AM, Hammill JT, et al. Probing protein folding using site-specifically encoded unnatural amino acids as FRET donors with tryptophan.Biochemistry, 2009, 48(25): 5953–5962.DOI: 10.1021/bi900426d |
| [82] | Cui ZL, Mureev S, Polinkovsky ME, et al. Combining sense and nonsense codon reassignment for site-selective protein modification with unnatural amino acids.ACS Synth Biol, 2017, 6(3): 535–544.DOI: 10.1021/acssynbio.6b00245 |
| [83] | Anderson RD, Zhou J, Hecht SM. Fluorescence resonance energy transfer between unnatural amino acids in a structurally modified dihydrofolate reductase.J Am Chem Soc, 2002, 124(33): 9674–9675.DOI: 10.1021/ja0205939 |
| [84] | Nakata H, Ohtsuki T, Sisido M. A protease inhibitor discovery method using fluorescence correlation spectroscopy with position-specific labeled protein substrates.Analyt Biochem, 2009, 390(2): 121–125.DOI: 10.1016/j.ab.2009.03.049 |
| [85] | Taki M, Hohsaka T, Murakami H, et al. Position-specific incorporation of a fluorophore-quencher pair into a single streptavidin through orthogonal four-base codon/anticodon Pairs.J Am Chem Soc, 2002, 124(49): 14586–14590.DOI: 10.1021/ja017714+ |
| [86] | Murakami H, Hohsaka T, Ashizuka Y, et al. Site-directed incorporation of fluorescent nonnatural amino acids into streptavidin for highly sensitive detection of biotin.Biomacromolecules, 2000, 1(1): 118–125.DOI: 10.1021/bm990012g |
| [87] | Ravikumar Y, Nadarajan SP, Yoo TH, et al. Unnatural amino acid mutagenesis-based enzyme engineering.Trends Biotechnol, 2015, 33(8): 462–470.DOI: 10.1016/j.tibtech.2015.05.002 |
| [88] | Poreba M, Kasperkiewicz P, Snipas SJ, et al. Unnatural amino acids increase sensitivity and provide for the design of highly selective caspase substrates.Cell Death Different, 2014, 21(9): 1482.DOI: 10.1038/cdd.2014.64 |
| [89] | Ugwumba IN, Ozawa K, Xu ZQ, et al. Improving a natural enzyme activity through incorporation of unnatural amino acids.J Am Chem Soc, 2011, 133(2): 326–333.DOI: 10.1021/ja106416g |
| [90] | Murray CJ, Baliga R. Cell-free translation of peptides and proteins: from high throughput screening to clinical production.Curr Opin Chem Biol, 2013, 17(3): 420–426.DOI: 10.1016/j.cbpa.2013.02.014 |
| [91] | Axup JY, Bajjuri KM, Ritland M, et al. Synthesis of site-specific antibody-drug conjugates using unnatural amino acids.Proc Natl Acad Sci USA, 2012, 109(40): 16101–16106.DOI: 10.1073/pnas.1211023109 |
| [92] | Patel KG, Ng PP, Kuo CC, et al. Cell-free production of Gaussia princeps luciferase-antibody fragment bioconjugates for ex vivo detection of tumor cells.Biochem Biophys Res Commun, 2009, 390(3): 971–976.DOI: 10.1016/j.bbrc.2009.10.087 |
| [93] | Donaghy H. Effects of antibody, drug and linker on the preclinical and clinical toxicities of antibody-drug conjugates.mAbs, 2016, 8(4): 659–671.DOI: 10.1080/19420862.2016.1156829 |
| [94] | Bakhtiar R. Antibody drug conjugates.Biotechnol Lett, 2016, 38(10): 1655–1664.DOI: 10.1007/s10529-016-2160-x |
| [95] | Ugwumba IN, Ozawa K, de la Cruz L, et al. Using a genetically encoded fluorescent amino acid as a site-specific probe to detect binding of low-molecular-weight compounds.Assay Drug Dev Technol, 2011, 9(1): 50–57.DOI: 10.1089/adt.2010.0306 |
| [96] | Maza JC, McKenna JR, Raliski BK, et al. Synthesis and incorporation of unnatural amino acids to probe and optimize protein bioconjugations.Bioconjugate Chem, 2015, 26(9): 1884–1889.DOI: 10.1021/acs.bioconjchem.5b00424 |
| [97] | Katti KS, Ambre AH, Peterka N, et al. Use of unnatural amino acids for design of novel organomodified clays as components of nanocomposite biomaterials.Philosoph Trans R Soc A: Math, Phys Eng Sci, 2010, 368(1917): 1963–1980.DOI: 10.1098/rsta.2010.0008 |
