

复旦大学 生命科学学院 遗传学研究所,上海 200433
收稿日期:2017-09-08;接收日期:2017-12-11
摘要:基于微滴式数字聚合酶链式反应(Droplet digital polymerase chain reaction, ddPCR)设计一种检测肠癌游离循环DNA (Circulating cell free DNA, cfDNA)中KRAS (V-Ki-ras2 Kirsten ratsarcoma viral oncogene homolog)基因突变的新方法并评估其灵敏度和准确性。根据肠癌病人KRAS基因的突变类型设计并合成,采用ddPCR扩增并评估其灵敏度和准确性;根据AMRS-PCR引物设计原理设计KRAS基因的实时定量PCR扩增引物并评估其准确性,进而比较ddPCR和qPCR二者之间的优缺点;最后针对52例肠癌病人的cfDNA采用ddPCR进行检测,研究ddPCR在cfDNA KRAS基因突变检测的应用。成功使用ddPCR和qPCR两种方法对KRAS野生型及7种突变型建立检测方法,使用质粒标准品及实际样品验证该两种方法可行并对其假阳性率、线性范围及检测下限等性能进行了评价,最后成功对52例临床患者和20例正常人的血浆cfDNA样本进行检测,临床灵敏度为97.64%,临床特异性为81.43%。ddPCR的检测性能优于qPCR,LOD达到个位数DNA拷贝,最低可确认突变浓度达到0.01%-0.04%。样本提取效率在方法学建立中也十分重要,直接影响到灵敏度和Cut Off值的判定。临床患者检测结果显示其KRAS突变率接近报道水平。
关键词:微滴式数字聚合酶链式反应 肠癌游离循环DNA KRAS 液态活检
Detection of KRAS mutation in colorectal cancer patients' cfDNA with droplet digital PCR
Yuwen Luo, Yao Li


Institute of Genetics, School of Life Sciences, Fudan University, Shanghai 200433, China
Received: September 8, 2017; Accepted: December 11, 2017
Corresponding author:Yao Li. Tel: +86-21-51630559; E-mail: yaoli@fudan.edu.cn
Abstract: This study aims to develop a new method for the detection of KRAS mutations related to colorectal cancer in cfDNA, and to evaluate the sensitivity and accuracy of the detection. We designed a method of cfDNA based KRAS detection by droplets digital PCR (ddPCR). The theoretical performance of the method is evaluated by reference standard and compared to the ARMS PCR method. Two methods, ddPCR and qPCR, were successfully established to detect KRAS wild type and 7 mutants. Both methods were validated using plasmid standards and actual samples. The results were evaluated by false positive rate, linearity, and limit of detection. Finally, 52 plasma cfDNA samples from patients and 20 samples from healthy people were tested, the clinical sensitivity is 97.64%, clinical specificity is 81.43%. ddPCR method shows higher performance than qPCR. The LOD of ddPCR method reached single digits of cfDNA copies, it can detect as low as 0.01% to 0.04% mutation abundance.
Key words: droplet digital PCR colorectal cancer cell free DNA KRAS liquid biopsy
游离循环DNA (Circulating cell free DNA,cfDNA)是一种细胞外游离状态的DNA,长度一般在180 bp左右,肿瘤病人体内的cfDNA部分来源于肿瘤细胞,被称为循环肿瘤DNA (Circulating cell free tumor DNA,ctDNA)[1]。理论上,通过检测肿瘤病人cfDNA中肿瘤相关基因突变应与肿瘤组织中含有的基因突变一致,进而可实现肿瘤基因的微创检测,即液体活检,本方法可克服传统检测方法对组织样本的依赖性等难题[2]。目前,基因突变常用的检测方法主要有:直接测序法、实时荧光定量法、高分辨率熔解曲线、质谱及高效液相色谱法等[3]。然而,鉴于外周血中cfDNA的丰度较低,其有大量野生型基因背景,故采用传统方法进行检测的难度较大。如实时荧光定量PCR仅能在野生型背景中检出30 pg左右的突变;测序法虽然准确度更高,但最低只能测得1%左右,而且成本较高。
微滴式数字PCR (Droplet digital pCR,ddPCR)是一种同时具备高灵敏度和高准确性的PCR检测方法,对于突变检测的理论检测下限可有效达到0.001%[4]。其利用油包水原理将PCR反应体系分散为数万个微滴,每个微滴都是一个单模板PCR反应室。模板DNA在微滴中扩增后释放荧光信号。通过对每个微滴荧光信号的逐一收集来统计PCR扩增阴性和阳性微滴的数量,并通过泊松分布换算成核酸的初始拷贝浓度,进而对模板初始浓度进行精确到单拷贝的定量[5]。微滴内的模板拷贝数为个位数而微滴本身的体积极小,所以相对而言,反应体系中低丰度突变基因的相对丰度很高,因此每个微滴独立检测时的灵敏度会获得大幅提升[6]。微滴式数字PCR可以在拥有更高精确度和结果稳定性的同时拥有以前分子检测方法所无法达到的检测下限(Limits of detection,LOD)[7]。同时,多篇文献证明ddPCR对于肝素等多种PCR抑制剂都有良好的耐受特性[8-9],可以避免复杂体液中可能存在的PCR酶抑制剂对实验的影响,从而提高检测的稳定性和重复性。
目前国内对于数字PCR的检测性能研究较少,所以本文旨在摸索一种基于ddPCR的cfDNA KRAS基因突变检测方法,并与现有常规检测方法(如荧光定量PCR)比较线性范围、LOD、重复性等,从而为数字PCR的液态活检应用提供更多参考与支持。同时,国内外同类研究大多都还在使用实验经验值与技术间横向对比的方法判定灵敏度,而没有使用量化的统计学算法,所以本研究基于泊松分布的数学模型进行灵敏度分析,对目前数字PCR实用化开发具有很大的补充完善价值。其最终目的是为充分挖掘数字PCR的应用潜力,客观评价其检测性能,并为其临床化液态活检提供实践依据。
1 材料与方法1.1 标准品处理选择KRAS基因12和13号外显子上7个突变型作为备选基因,包括G12C、G12V、G12D、G12R、G12S、G12A、G13D,并结合野生型基因的质粒标准品进行合成(宝生物工程(大连)有限公司,中国)。实验检测前采用限制性内切酶XbaⅠ(宝生物工程(大连)有限公司,中国)进行线性化处理。
1.2 血浆cfDNA的提取52例结病患血浆标本来自于复旦大学附属肿瘤医院确诊结直肠癌患者,20例平均年龄45岁健康人对照血浆标本均来自于郑州大学附属医院。按照临床常规方法采用无抗凝剂真空采血管采集静脉血,使用Cell-Free DNA? BCT (218962,Streck Inc. USA)将5 mL全血以1 600×g速度离心20 min吸取上清得到血浆,再以1 6000×g速度离心10 min去除血浆中的残留细胞。在采集分离当天使用QIAamp? Circulating Nucleic Acid Kit (Qiagen)试剂盒进行cfDNA提取,核酸特异性结合到QIAamp Mini离心柱上,污染物流走。通过三步洗涤步骤完全去除二价阳离子和蛋白等PCR抑制物,结合在离心柱上的纯核酸用试剂盒中的洗脱缓冲液洗脱,洗脱体积为50 μL。在说明书操作流程基础上,将蛋白酶K处理条件延长为60 ℃孵育1 h,洗脱体积为30 μL,于-20 ℃保存备用。
1.3 微滴式数字PCR引物设计及扩增基于Beacon Designer 8.10 (Primer Biosoft)软件(http://www.softpedia.com/get/Science-CAD/Beacon-Designer.shtml)设计引物探针,经Blast比对并预实验验证筛选特异性。引物探针所针对的扩增子为7种常见KRAS突变基因型与野生型KRAS,在检测过程中每一种突变型引物探针都与对应的野生型进行双重检测,由于KRAS野生型的cfDNA来源于人类正常组织,所以可以作为KRAS突变检测的内参使用。所有突变型和野生型使用通用上下游引物(5′-GACTGAATATAAACT TGTGGTA-3′; 5′-GTCCACAAAATGATTCTGA-3′),分别针对每种突变型设计FAM标记的Taqman探针,并对于KRAS野生型设计HEX标记的Taqman探针,具体引物序列如表 1所示,所有引物由宝生物工程(大连)有限公司化学合成。
表 1 微滴式数字PCR探针序列Table 1 Sequence of digital PCR probes
| Type | Sequence |
| G12C | FAM-5′-ACTCTTGCCTACGCCACAAG-3′-BHQ1 |
| G12S | FAM-5′-ACTCTTGCCTACGCCACTAG-3′-BHQ1 |
| G12R | FAM-5′-ACTCTTGCCTACGCCACGAG-3′-BHQ1 |
| G12V | FAM-5′-ACTCTTGCCTACGCCAACAG-3′-BHQ1 |
| G12D | FAM-5′-ACTCTTGCCTACGCCAT CAG-3′-BHQ1 |
| G12A | FAM-5′-ACTCTTGCCTAC GCCAGCAG-3′-BHQ1 |
| G13D | FAM-5′-ACTCTT GCCTACGTCACCAG-3′-BHQ1 |
| W T | HEX-5′-ACTCTTGCCTACGCCACCAG-3′-BHQ1 |
表选项
将每个配制好的PCR反应体系加入微滴发生卡(Bio-Rad)中间一排的8个“Sample”孔内,为保证8个孔的压力均一,在PCR反应体系不足8个时,剩余孔用20 μL 1×buffer control (Bio-Rad)补足,加样时注意不能产生气泡。在微滴发生卡(Bio-Rad)最底层一排8个“Oil”孔中各加入70 μL微滴生成油(Bio-Rad),8孔全部加满。微滴发生卡(Bio-Rad)通过QX200? Droplet Generator(Bio-Rad)仪器制备为20 000个反应微滴,制备完成后微滴位置将处于微滴发生卡(Bio-Rad)顶层一排的8个“Sample”孔内,最后将微滴转移入96孔板并于普通PCR仪上进行扩增。在PCR过程中,由于微滴本身热传导不具备流体一样的对流特点,所以为保证微滴间热传导更充分,升降温速度设定在2.5 ℃/s。PCR反应结束后将96孔板放入QX200? Droplet Reader (Bio-Rad),并在软件QuantaSoft (Bio-Rad)上设定检测模式为RED,同时检测FAM和HEX的荧光信号。仪器会自动分析每个样品的每个微滴中荧光信号,然后,由QuantaSoft完成对数据的泊松分布换算,获得靶序列在PCR反应体系中的拷贝数浓度(单位:copies/μL)及突变浓度。
1.4 聚合酶链式扩增阻碍突变系统引物设计及检测参考聚合酶链式扩增阻碍突变系统ARMS-PCR原理设计实时定量PCR引物探针,下游使用通用引物,上游引物根据不同基因型设计,并设计PNA探针,PNA探针可起到阻遏作用来竞争下游提高反应的特异性,具体引物序列如表 2所示;PNA探针(野生型)序列为5′-TGGAGCTGGT GGCGTAGGC-P04-3′,HEX标记的Taq-man探针序列为5′-HEX-TCTGAATTAGCTGTATCGTCAAG GCACTCT-BHQ1-3′。
表 2 qPCR引物序列Table 2 Sequence of qPCR primers
| Type | Sequence (5′-3′) |
| G12C | AACTTGT GGTAGTTGGAGCGT |
| G12S | ATAAACTTG TGGTAGTTGGAGCTA |
| G12R | ATAAACTT GTGGTAGTTGGAGCCC |
| G12V | AAACTT GTGGTAGTTGGAGCGGT |
| G12D | CTTGTG GTAGTTGGAGCTTA |
| G12A | AACTTGTGG TAGTTGGAGCTGC |
| G13D | GTGGTAGTTG GAGCTGGTAA |
| Universal | ACCTCTATTGTTGGATCATATTCGT C |
表选项
1.5 方法学论证1.5.1 cfDNA得率验证由于cfDNA的提取得率对于检测灵敏度至关重要,所以在样本提取后会首先检测OD260 (OD260 > 1)以及OD260/OD280 (1.6 < OD260/OD280 < 1.9)对其cfDNA得率及纯度进行评估,并结合后续样本实验检测结果,评价cfDNA的提取得率。
1.5.2 理论检测性能比较通过稀释标准品进行检测,比较ddPCR与qPCR的LOD、定量下限(Limit of quantification,LOQ)、线性范围、变异系数(CV%)、特异性、单个样品检测成本等参数。
1.5.3 实际样本检测使用验证后的ddPCR方法对临床病理学实验及影像学确诊的52例结直肠癌病人样本和20例健康人样本进行检测。再根据实际样本检测结果,比较ddPCR所得结果和现有研究中的中国人群KRAS突变分布比例的一致性,并根据以上的统计学分析结果划定两种检测方法的Cut Off值。
1.5.4 检测下限计算在任何一种检测方式的硬件可接受动态范围内,检测背景(Limit of blank,LOB)、LOD、LOQ的范围在依次排开,具体关系如图 1所示。其中,浓度低于LOB的样本无法测得阳性,浓度高于LOB但低于LOD的样本无法稳定检出但检出有可能为阳性,需要重复检测;浓度高于LOD低于LOQ的样本可稳定检出但无法精确定量,高于LOD即可出具可靠的临床报告;浓度在LOQ范围内的样本可以达到精确定量检测的目的;高于LOQ低于总动态范围的样本可以判定阳性但定量误差较大;大于动态范围浓度的样本将表现出过饱和阳性的检测结果,无法定量。LOD作为区分方法学能否可靠检出结果的分水岭,是检测方法最重要的一项参数,直接关系到临床报告的可靠性。而LOD的值等于该检测方法假阳性水平的正向95%统计学分位数,所以我们可以通过估计阴性临床样本的假阳性水平来计算LOD,并以LOD来定义灵敏度。
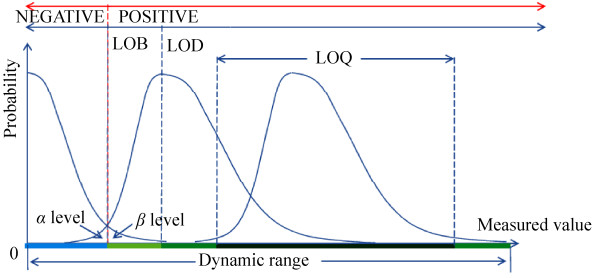 |
| 图 1 检测方法动态范围关系图 Figure 1 Dynamic range schematic of diagnostic method. |
| 图选项 |
1.6 统计学分析所有实验数据均采用SPSS v21.0 (http://spss. en.softonic.com/)统计学分析软件进行统计分析,qPCR计量资料以正态分布均数±标准差表示,ddPCR计量资料以泊松分布的数学期望显示。由于正态分布与泊松分布间、Ct值结果与拷贝数结果间不能以常规检验方法检验差异显著性,故以某一估计总体的数学期望值/均值是否处于另一估计总体的95%置信区间外来衡量差异是否显著。
2 结果2.1 ddPCR理论性能检测经过反复优化成功建立了基于ddPCR 7个位点的KRAS检测方法(反应体系及反应条件)。为验证反应特异性,以非特异模板进行不同突变型引物探针间的交叉反应实验,每个反应重复3次。不同突变型之间、突变型与野生型之间的交叉反应为零。标准品实验中的特异性为100%。
如图 2A和2B所示,ddPCR检测中平均每孔微滴数超过10 000个,由于微滴中平均包裹核酸拷贝数符合泊松分布,而10 000个以上的微反应单位已经达到进行数字PCR泊松分布分析的最小抽样量[10]。如图 2所示,G12C与G12D最低测得浓度为0.01%,其余突变型测得最低浓度为0.04%。所有基因型0.1%及以上浓度的CV都小于25%,使用标准品检测过程中突变含量0%的样品都没有出现假阳性,线性方程的R2都大于0.98,图 3为各检测位点的线性曲线。
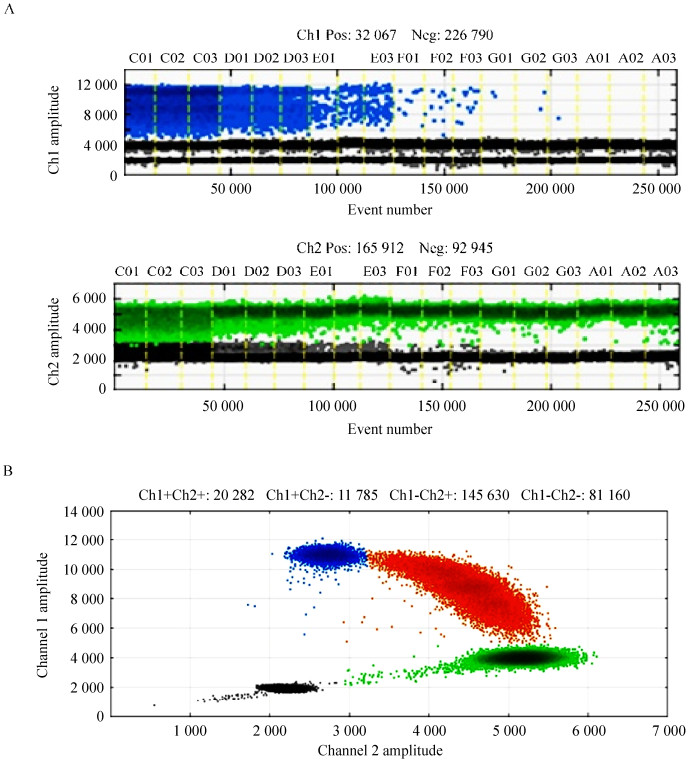 |
| 图 2 ddPCR扩增微滴图(以G12D标准品梯度稀释检测结果为例). (A) ddPCR扩增微滴一维图,Ch1为FAM通道(突变型),Ch2为HEX通道(野生型).黑灰色微滴为无扩增阴性微滴,蓝色和绿色微滴分别为FAM和HEX通道中PCR阳性扩增微滴,突变比例从左至右依次降低. (B) ddPCR扩增微滴二维图,纵轴为FAM荧光增量,横轴为HEX荧光增量,微滴在坐标系内聚类为4簇:左下为无模板微滴,左上为仅有FAM信号的微滴,右下为仅有HEX信号的微滴,右上为同时发出FAM与HEX信号的双阳性微滴. Figure 2 ddPCR amplification chart (G12D as an example). (A) ddPCR 1D amplification charts, Ch1 is FAM Channel (mutant type) and Ch2 is HEX channel (wild type). In these charts, black points signify PCR negative droplets, blue and green points respectively signify PCR positive droplets in FAM channel and HEX channel, the mutation abundances descend from left to right. (B) ddPCR 2D amplification chart, Y axis shows amplitude in FAM channel and X axis shows amplitude in HEX channel, the droplets are separated in 4 clusters: lower left points signify no template droplets, upper left points signify the FAM signal positive droplets, lower right points signify HEX signal positive droplets, upper right points signify double positive droplets in both FAM and HEX channel. |
| 图选项 |
 |
| 图 3 KRAS基因不同突变型标准品梯度稀释线性方程图. (A) KRAS基因G12C突变型标准品梯度稀释线性方程图. (B) KRAS基因G12D突变型标准品梯度稀释线性方程图. (C) KRAS基因G12A突变型标准品梯度稀释线性方程图. (D) KRAS基因G12V突变型标准品梯度稀释线性方程图. (E) KRAS基因G12R突变型标准品梯度稀释线性方程图. (F) KRAS基因G12S突变型标准品梯度稀释线性方程图. (G) KRAS基因G13D突变型标准品梯度稀释线性方程图. G12D及G12C的浓度梯度为100%-0.01%,10倍稀释;其余突变型的稀释梯度为1%、0.2%、0.1%、0.04% Figure 3 ddPCR linear curves of KRAS mutant abundances. (A) shows linear curve of KRAS G12C type. (B) shows linear curve of KRAS G12D type. (C) shows linear curve of KRAS G12A type. (D) shows linear curve of KRAS G12V type. (E) shows linear curve of KRAS G12R type. (F) shows linear curve of KRAS G12S type. (G) shows linear curve of KRAS G13D type. The samples of G12D and G12C are tenfold diluted from 100% to 0.01%, the dilution gradients of other mutant types are 1%, 0.2%, 0.1% and 0.04%. |
| 图选项 |
2.2 健康人样本的ddPCR检测及理论LOD值使用20个健康人样本作为阴性对照进行ddPCR检测,在检测中大部分结果都显示阴性(图 4),各突变类型的总反应体系中假阳性结果都小于2 copies/μL,假阳性孔中的阳性微滴数量为1-2 droplets。
 |
| 图 4 阴性对照检测的微滴一维图 Figure 4 1D droplets chart of negative control tests. |
| 图选项 |
本研究中ddPCR的理论LOD值依据两种准则建立:一种是以泊松分布在采样量极大时无限接近正态分布的原理,假设阴性对照的假阳性结果符合正态分布,将其LOD判定在假阳性结果均数的正态分布95%置信区间以外(LOD判定法1)。另一种是依据于ddPCR中阳性微滴符合泊松分布,通过公式FPR=false droplets number/wells来计算不同方法的假阳性率(False positive rate,FPR),并以FPR的泊松分布正向95%置信区间分位数为真阳性阈值(微滴数)[11],同时以真阳性阈值作为LOD的负方向95%置信区间分位数来推测LOD的数学期望值(LOD判定法2),具体判定结果见表 3,后续实验采用对应拷贝数更高、结果更保守的LOD判定法2。
表 3 两种不同方法判定的ddPCR KRAS突变检测法的LODTable 3 LODs of ddPCR KRAS detection evaluated by 2 different algorithms
| LOD | G12C | G12S | G12R | G12V | G12D | G12A | G13D |
| LOD algorithm 1 (copies/well) | 1.39 | 1.63 | 2.26 | 2.21 | 1.80 | 1.72 | 1.80 |
| LOD algorithm 2 (positive droplets/well) | 3.00 | 3.00 | 3.00 | 3.00 | 3.00 | 3.00 | 3.00 |
| Note: LOD algorithm 1 is according to normal distribution, LOD algorithm 2 is according to Poisson distribution, There are significant differences between LODs and negative controls in all mutant type, P < 0.05. | |||||||
表选项
由于QX200 ddPCR仪的微滴上限为20 000个,而微滴体积为0.89 nL,每个反应体系20 μL,所以当阳性微滴数已知时可以通过泊松分布校正公式copies/μL=20·(-ln(1-PD/20 000))/0.89计算反应体系中的总拷贝数,式中PD代表反应中的阳性微滴数。当LOD=3 positive droplets/well时,对应的拷贝数为3.49拷贝,鉴于QX200 ddPCR的理论检测上限为100 000拷贝,定量比较精确的结果为低于其总微滴数的20 000拷贝左右,所以当反应体系中野生型对照达到20 000拷贝时(上样量足够),3.49拷贝对应的突变含量为0.017%。由于统计学分析最低可检测突变含量低于线性梯度稀释实验中的最低值0.01%及0.04%,且该浓度下测定值CV < 25%,所以有理由确认数字PCR在KRAS检测中的突变含量LOD为0.01%-0.04%左右。
2.3 实时定量PCR理论性能检测如图 5所示,非特异模板无扩增曲线或Ct > 35,空白对照无扩增。最低测得0.1%突变含量的样本,Ct值变异系数在12%以内,如果按每个Ct浓度差异2倍来估算的话,定量的变异系数约为50%左右。由于qPCR需以标准品测定值为参照,建立标准曲线以对样品定量(定量结果都以已知量标定),所以定量误差无法在标准品检测中体现。而对于样品检测而言,精度除受Ct值误差影响以外,完全依赖于建立方法所用标准品的质量及其稀释准确度。
 |
| 图 5 qPCR浓度梯度Ct值曲线 Figure 5 Ct/concentration gradient curve of qPCR. |
| 图选项 |
2.4 健康人样本的实时定量PCR检测及理论LOD值使用16个健康人样本作为阴性对照进行实时定量PCR检测,多数无扩增,Ct值记为50,少部分有Ct值高于40个循环的扩增曲线,以Ct值置信区间作为判定实时定量PCR LOD的方法。如表 4所示,在上样量不变的情况下,由于LOD值小于0.1%浓度样本的Ct值,因此,G12S并无法可信地判定0.1%突变为真阳性。
表 4 qPCR法在各突变检测位点的理论LOD (Ct值)Table 4 The oretical LODs of qPCR methods in all mutant type (Ct value)
| Mutanttype | G12S | G12R | G12C | G12D | G12A | G12V | G13D |
| LOD | 27.23 | 35.98 | 30.59 | 32.82 | 37.23 | 35.00 | 34.33 |
| Note: There are significant differences between LODs and negative controls in all mutant type, P < 0.05. | |||||||
表选项
2.5 误差与重复性图 6为综合分析比较ddPCR与qPCR在LOQ内所有测得值CV的结果,ddPCR在所有突变含量下的CV水平都要低于qPCR,证明其重复性更好且精确性更高。
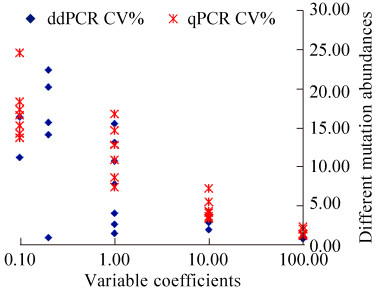 |
| 图 6 ddPCR与qPCR变异系数对照图 Figure 6 Comparison of variable coefficients between ddPCR and qPCR. X axis shows. |
| 图选项 |
2.6 ddPCR实际样本检测结果首先使用ddPCR方法对所有样本的7种KRAS突变位点做了一次检测,然后对于测得结果低于理论LOD值的样本提高1倍上样量,提高核酸绝对量并重复试验,重复后测得为阴性突变结果和低于理论LOD值的样本记为纯野生型。所有测得拷贝数浓度都除以PCR体系稀释比得到原始浓度,并将突变型除以总cfDNA拷贝数得到突变含量(表 5)。低于LOD及难以确认的样本,增加1倍上样量重复验证,并参考肿瘤手术病理学检测结果及帕尼单抗耐药情况进行验证。
表 5 基于ddPCR测得临床样本KRAS野生型、突变型拷贝数换算出的突变含量Table 5 Clinical sample mutation abundances calculated from ddPCR detection results of KRAS wild and mutant type copy numbers
| Sample | G12R | G12C | G12A | G12S | G12D | G12V | G13D |
| 1 | 4.48% | 10.13% | 0.00% | 14.89% | 0.00% | 1.52% | 10.52% |
| 2 | 8.76% | 0.00% | 0.00% | 9.79% | 2.12% | 0.00% | 1.86% |
| 3 | 3.51% | 0.00% | 2.39% | 4.99% | 0.00% | 0.00% | 5.54% |
| 4 | 20.73% | 4.76% | 4.41% | 15.00% | 2.78% | 3.88% | 12.18% |
| 5 | 37.50% | 19.60% | 17.65% | 38.14% | 47.10% | 26.83% | 45.51% |
| 6 | 3.35% | 4.35% | 0.00% | 12.89% | 0.00% | 1.50% | 16.29% |
| 7 | 1.33% | 0.10% | 0.25% | 0.80% | 0.26% | 0.30% | 0.00% |
| 8 | 0.00% | 0.00% | 1.96% | 0.00% | 8.09% | 4.44% | 0.00% |
| 9 | 14.15% | 0.00% | 0.00% | 22.48% | 0.00% | 0.00% | 7.69% |
| 10 | 12.28% | 8.23% | 5.24% | 8.11% | 0.00% | 3.06% | 0.00% |
| 11 | 65.12% | 0.00% | 54.55% | 75.12% | 55.10% | 71.05% | 0.00% |
| 12 | 44.83% | 0.00% | 38.14% | 10.42% | 43.50% | 4.59% | 0.00% |
| 13 | 45.65% | 0.00% | 42.86% | 55.45% | 20.62% | 1.93% | 13.73% |
| 14 | 0.00% | 0.00% | 8.97% | 0.00% | 17.53% | 3.61% | 0.00% |
| 15 | 0.00% | 50.00% | 0.00% | 44.13% | 4.11% | 0.00% | 0.00% |
| 16 | 1.11% | 0.37% | 4.63% | 1.39% | 0.28% | 0.40% | 0.00% |
| 17 | 11.71% | 0.00% | 5.19% | 18.75% | 7.53% | 3.19% | 0.00% |
| 18 | 23.00% | 0.00% | 21.11% | 2.95% | 5.98% | 0.00% | 0.00% |
| 19 | 14.89% | 0.00% | 5.14% | 6.86% | 0.00% | 3.41% | 0.00% |
| 20 | 0.00% | 0.06% | 0.00% | 0.13% | 0.06% | 0.03% | 0.00% |
| 21 | 1.64% | 1.25% | 0.00% | 4.97% | 0.00% | 1.35% | 0.00% |
| 22 | 3.10% | 2.27% | 1.26% | 17.43% | 1.23% | 8.11% | 0.00% |
| 23 | 13.73% | 0.00% | 15.79% | 0.00% | 0.00% | 1.26% | 0.00% |
| 24 | 50.00% | 0.00% | 3.23% | 56.00% | 2.74% | 23.08% | 0.00% |
| 25 | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% |
| 26 | 0.00% | 28.57% | 0.00% | 0.00% | 0.00% | 0.00% | 13.73% |
| 27 | 0.00% | 6.04% | 0.00% | 0.00% | 5.41% | 0.00% | 0.00% |
| 28 | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 1.57% | 0.00% |
| 29 | 0.00% | 0.00% | 0.00% | 2.32% | 0.00% | 0.00% | 0.00% |
| 30 | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 1.19% | 0.00% |
| 31 | 0.00% | 1.45% | 0.00% | 3.38% | 62.79% | 0.00% | 0.00% |
| 32 | 0.00% | 0.00% | 0.00% | 0.54% | 0.00% | 1.71% | 0.00% |
| 33 | 0.48% | 0.00% | 0.00% | 9.88% | 0.00% | 6.54% | 0.00% |
| 34 | 0.00% | 0.00% | 0.00% | 0.00% | 56.90% | 0.00% | 0.00% |
| 35 | 0.00% | 0.00% | 10.83% | 0.00% | 0.00% | 0.00% | 0.00% |
| 36 | 0.00% | 0.00% | 0.00% | 0.00% | 45.00% | 0.00% | 0.00% |
| 37 | 0.00% | 0.00% | 0.00% | 35.38% | 0.81% | 0.00% | 0.00% |
| 38 | 0.00% | 33.33% | 25.00% | 1.99% | 48.28% | 1.01% | 13.95% |
| 39 | 0.00% | 10.45% | 4.19% | 0.00% | 5.51% | 6.35% | 0.00% |
| 40 | 0.00% | 0.98% | 1.43% | 1.13% | 0.46% | 0.69% | 0.00% |
| 41 | 0.00% | 6.18% | 3.34% | 0.00% | 1.15% | 0.00% | 1.39% |
| 42 | 0.00% | 0.00% | 4.19% | 0.00% | 5.23% | 8.31% | 0.00% |
| 43 | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 3.60% | 0.00% |
| 44 | 0.00% | 1.57% | 0.00% | 0.00% | 1.10% | 0.00% | 0.00% |
| 45 | 0.00% | 0.00% | 0.00% | 1.16% | 11.50% | 0.00% | 0.00% |
| 46 | 0.00% | 1.58% | 0.00% | 0.00% | 0.52% | 1.08% | 0.89% |
| 47 | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 3.33% |
| 48 | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% |
| 49 | 0.00% | 6.25% | 12.00% | 0.00% | 0.00% | 11.29% | 3.41% |
| 50 | 0.00% | 0.40% | 0.00% | 0.00% | 2.24% | 0.32% | 0.00% |
| 51 | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% | 0.00% |
| 52 | 0.00% | 2.57% | 0.49% | 0.00% | 0.00% | 0.59% | 0.00% |
| Note: the bold numbers show the positive repeated verification results of samples under LOD (false negative in single test). The numbers with underline show the negative repeated verification results of samples under LOD (false positive in single test). | |||||||
表选项
在所有52例临床样本364个反应的检测中,如以2.2中的LOD进行判定,需重复验证的结果总共47个反应,验证后,其中假阳性44反应,假阴性3反应。如果以灵敏度=真阳性例数/(真阳性例数+假阴性例数)计算临床灵敏度,以特异性=真阴性例数/(真阴性例数+假阳性例数),则临床灵敏度为97.64%,临床特异性为81.43%。
2.7 cfDNA提取得率本次研究cfDNA提取效率通过野生型突变型拷贝数总量计算(图 7),平均提取效率为5.19 ng/mL。测得结果相比OD260比色结果更低,但数据更稳定,我们认为这主要是由于样品内杂质干扰了比色定量的结果,故采用ddPCR检测数据。
 |
| 图 7 本研究中52个结直肠癌病人血液cfDNA提取效率 Figure 7 Blood cfDNA extraction efficiency of 52 colorectal cancer patients' sample in this study. |
| 图选项 |
依据泊松分布原理,为保证实验结果落在其95%置信区间内,样本总量中则至少拥有3个拷贝以上的目的基因。对20 000个微滴中的DNA分子来说,如果要测得0.1%的突变,则模板量必须达到3 000个拷贝野生型DNA中含有3个拷贝的突变型DNA,本研究中将此称为液态活检的样本极限LOD。以本次提取效率估计,采血量与LOD相关表格见表 6。
表 6 以本次研究结果建立的采血量与样本极限LOD关系表Table 6 Relationship between total blood capacity and sample LOD according to the result of this study
| Sample LOD | Total copies | Mass | Extraction efficiency (ng/mL) | Total blood requirement (mL) |
| ?1/1 000 | ?3 000 | ?10 ng | 5.19 | ~2 |
| ?1/10 000 | ?30 000 | ?100 ng | ~20 | |
| ?1/100 000 | ?300 000 | ?1 μg | ~200 |
表选项
3 讨论KRAS基因属于RAS基因家族,是EGFR信号通路中的重要基因之一,位于染色体12p12.1上,编码p21蛋白[12]。其突变型不依赖刺激信号的激活,即不受上游EGFR基因状态的影响,始终处于激活状态,其功能状态不可控,导致肿瘤的持续增殖。在大约38%的转移性结直肠癌中都发生了KRAS突变,其中 > 96%的突变发生在12-13号密码子上[13]。由于KRAS突变后不再依赖于其他信号的调控,这会导致抗EGFR靶向药物或者EGFR抑制剂在癌症治疗中失效[14],并对西妥昔单抗等药物的治疗效果产生重大影响。已有研究表明,中国结直肠癌患者中,KRAS的突变频率为30%-40%左右,和世界人群一样,中国结直肠癌患者的KRAS突变中主要包括12号及13号外显子上的7种突变[15]。通过检测KRAS突变可对癌症进行及时的侦测及预后,并能基于个体化用药原则对治疗方案进行指导。现在很多的肿瘤基因检测都是使用肿瘤组织的石蜡包埋块作为DNA模板来源,虽可准确地检出肿瘤组织中癌症相关突变,但要获取肿瘤组织进行检测面临两个难题。首先一些肿瘤组织并不便于使用传统的穿刺或手术方法获得;其次,获取肿瘤组织本身对患者的身体都会带来创伤。除此之外,虽然获得了肿瘤组织,但肿瘤组织的包埋过程、保存状态及肿瘤细胞个体的异质性都会影响检测结果的准确性[16]。
液态活检最早于1974年由Sorrells RB应用关节腔的滑液分析来诊断滑膜疾病[17]。结合当下的核酸、细胞检测技术的发展,液态活检这个概念近期被重新提出,其专注方向开始偏向于血液中的游离循环核酸(DNA和RNA)和循环肿瘤细胞。液态活检也由一种概念和技术迅速发展成为肿瘤生物学研究领域的热点,并逐步由实验室研究步入临床应用阶段[18]。液态活检中的液态指的是体液样本,其中包括了血液、胸水、腹水、脑脊液、尿液及其他体液等,其检测对象主要有两大类:一类是核酸、另一类是细胞,对于体液中的cfDNA检测正属于其中的一类。液态活检在临床疗效监测、预后判断及临床分期、肿瘤个体化治疗靶标等方向提供了一条微创检测的途径。相较于传统的组织活检或固相活检,体液从临床获得样本途径非常简便,可以做到随时取样、随时监测,也不会造成医源性播散[19]。同时有研究表明,通过对于游离核酸液态活检方法得到的突变含量相比于荧光原位杂交(FISH)等基于肿瘤组织的检测方法来说,和肿瘤的死亡率等指标拥有更一致的联系[20]。已有文献表明在使用TKI药物的非小细胞肺癌的伴随诊断中,使用ddPCR进行液态活检,其可靠性高于传统方法[21-22]。
在本研究理论检测性能验证中,ddPCR理论拷贝数LOD为3.49 copies/well,理论突变浓度LOD达到0.01%-0.04%。线性范围内R2良好,定量误差在25%以内。
将qPCR的Ct值转化为突变浓度百分比,以其正态分布与ddPCR的LOD及LOQ的泊松分布进行统计学检验。则ddPCR在LOD推算的灵敏度上比qPCR高出一个数量级,定量变异系数低于qPCR,线性范围也比qPCR更广(P < 0.05),从而可以得到更精准更灵敏的理论检测性能。同时,ddPCR相比ARMS PCR等方法可方便地进行双重PCR,以野生型基因拷贝数作为内参来提高突变浓度的定量准确度。ddPCR对于拷贝数的绝对定量原理,使其检测性能不再受制于扩增曲线、Ct值以及标准品的限制。
在本次实验的临床样本检测结果中,可确认的真阳性样本最低浓度为0.06%,在LOD以上的检测范围中重复性较好。从技术角度来看,ddPCR技术可以满足现有癌症液态活检的需求,且检测性能优于qPCR。在实际使用中,ddPCR测得病人样本中的KRAS平均突变率为35.43%,接近现有报道中的中国结直肠癌人群中的突变率(38%左右),结果无显著差异(P > 95)。而具体突变位点的的比例则差异很大(P < 0.05),笔者认为这主要是因为52例样本的取样量有限,无法完全体现总体分布,同时也受方法学之间差异的影响。在对临床样本检测结果重复验证后得到的临床灵敏度为97.64%,临床特异性为81.43%。
本次研究的平均cfDNA提取效率均一性相差很大。这一方面是在样本保存和运输过程中导致的,另一方面也可能由于样本间个体差异导致的。提取效率的不均一性直接导致了样本间灵敏度的差异,所以对于液态活检领域,样本提取的方法学研究和标准化同样十分重要。对于突变含量的检测灵敏度不仅取决于核酸绝对量的检测灵敏度,还受到提取所得cfDNA总量的影响。所以在标定ddPCR对于cfDNA检测的Cut Off值时,首先,应当将ddPCR对于目的基因拷贝数检测的LOD作为判?ut Off值的基础;同时,Cut Off值和实际灵敏度是随着反应体系内的cfDNA总量变化的。
参考文献
| [1] | Li SW, Han L, Ma P, et al. New hope of tumor diagnosis-circulating free DNA detection.Chin J Lab Pathol, 2015, 7(2): 119–121, 89.(in Chinese). 黎四维, 韩露, 马培, 等. 肿瘤诊断的新希望-循环游离DNA检测.实用检验医师杂志, 2015, 7(2): 119-121, 89. |
| [2] | Snyder MW, Kircher M, Hill AJ, et al. Cell-free DNA comprises an in vivo nucleosome footprint that informs its tissues-of-origin.Cell, 2016, 164(1/2): 57–68. |
| [3] | Abbosh C, Birkbak NJ, Wilson GA, et al. Phylogenetic ctDNA analysis depicts early-stage lung cancer evolution.Nature, 2017, 545(7655): 446–451.DOI: 10.1038/nature22364 |
| [4] | Hindson BJ, Ness KD, Masquelier DA, et al. High-throughput droplet digital PCR system for absolute quantitation of DNA Copy number.Anal Chem, 2011, 83(22): 8604–8610. |
| [5] | Low H, Chan SJ, Soo GH, et al. Clarity? digital PCR system: a novel platform for absolute quantification of nucleic acids.Anal Bioanal Chem, 2017, 409(7): 1869–1875.DOI: 10.1007/s00216-016-0131-7 |
| [6] | Jones M, Williams J, G rtner K, et al. Low copy target detection by droplet digital PCR through application of a novel open access bioinformatic pipeline, 'definetherain'.J Virol Methods, 2014, 202: 46–53.DOI: 10.1016/j.jviromet.2014.02.020 |
| [7] | Whale AS, Huggett JF, Cowen S, et al. Comparison of microfluidic digital PCR and conventional quantitative PCR for measuring copy number variation.Nucleic Acids Res, 2012, 40(11): e82.DOI: 10.1093/nar/gks203 |
| [8] | Dingle TC, Sedlak RH, Cook L, et al. Tolerance of droplet-digital PCR vs real-time quantitative PCR to inhibitory substances.Clin Chem, 2013, 59(11): 1670–1672.DOI: 10.1373/clinchem.2013.211045 |
| [9] | Ra?ki N, Dreo T, Gutierrez-Aguirre I, et al. Reverse transcriptase droplet digital PCR shows high resilience to PCR inhibitors from plant, soil and water samples.Plant Methods, 2014, 10(1): 42.DOI: 10.1186/s13007-014-0042-6 |
| [10] | Tsui NB, Kadir RA, Chan KC, et al. Noninvasive prenatal diagnosis of hemophilia by microfluidics digital PCR analysis of maternal plasma DNA.Blood, 2015, 117(13): 3684–3691. |
| [11] | Milbury CA, Zhong Q, Lin J, et al. Determining lower limits of detection of digital PCR assays for cancer-related gene mutations.Biomol Detect Quantific, 2014, 1(1): 8–22.DOI: 10.1016/j.bdq.2014.08.001 |
| [12] | Cicenas J, Tamosaitis L, Kvederaviciute K, et al. KRAS, NRAS and BRAF mutations in colorectal cancer and melanoma.Med Oncol, 2017, 34(2): 26.DOI: 10.1007/s12032-016-0879-9 |
| [13] | Knijn N, Mekenkamp LJ, Klomp M, et al. KRAS mutation analysis: a comparison between primary tumours and matched liver metastases in 305 colorectal cancer patients.Br J Cancer, 2011, 104(6): 1020–1026.DOI: 10.1038/bjc.2011.26 |
| [14] | Sameen S, Barbuti R, Milazzo P, et al. Mathematical modeling of drug resistance due to KRAS mutation in colorectal cancer.J Theor Biol, 2016, 389: 263–273. |
| [15] | Xu ZN, Yang YF. Influences of storage status and time on the mutations of EGFR gene.China J Tradit Chin Med Pharm, 2017, 32(3): 1029–1032.(in Chinese). 许仲宁, 杨宇飞. 晚期大肠癌中医证素在KRAS、BRAF、NRAS基因突变类型中的分布差异性分析.中华中医药杂志, 2017, 32(3): 1029-1032. |
| [16] | Qian K, Zhang Y. Influences of storage status and time on the mutations of EGFR gene.Chin Clin Oncol, 2014, 19(2): 132–135.(in Chinese). 钱坤, 张毅. 组织样本保存状态及时间对EGFR基因突变检测结果的影响.临床肿瘤学杂志, 2014, 19(2): 132-135. |
| [17] | Sorrells RB. Synovioanalysis ("liquid biopsy").J Ark Med Soc, 1974, 71(1): 59–62. |
| [18] | Zhu MH. Liquid Biopsy: opportunities and challenges of the development of pathology.Chin J Pathol, 2015, 44(9): 617–618.(in Chinese). 朱明华. 液态活检:病理学发展的机遇和挑战.中华病理学杂志, 2015, 44(9): 617-618. |
| [19] | Wang C, Li YM, Fang XD. Research progress on liquid biopsy in oncology and its clinical applications.Hereditas (Beijing), 2017, 39(3): 220–231.(in Chinese). 王晨, 李艳明, 方向东. 肿瘤液态活检的研究进展及其临床应用.遗传, 2017, 39(3): 220-231. |
| [20] | Kinugasa H, Nouso K, Tanaka T, et al. Droplet digital PCR measurement of HER2 in patients with gastric cancer.Br J Cancer, 2015, 112(10): 1652–1655.DOI: 10.1038/bjc.2015.129 |
| [21] | Zhang Y. Detection of EGFR in plasma cfDNA with ddPCR and analysis of the correlation with EGFR-TKI curative effect[D]. Beijing: Beijing Union Medical College, Chinese Academy of Medical Sciences Peking Union Medical College, Chinese Academy of Medical Sciences, Tsinghua University School of Medicine, 2016 (in Chinese). 张雨. ddPCR法检测血浆游离DNA EGFR基因突变及与EGFR-TKI疗效的相关性分析[D]. 北京: 北京协和医学院中国医学科学院, 北京协和医学院, 中国医学科学院, 清华大学医学部, 2016. |
| [22] | Watanabe M, Kawaguchi T, Isa SI, et al. Multiplex ultrasensitive genotyping of patients with non-small cell lung cancer for epidermal growth factor receptor (EGFR) mutations by means of picodroplet digital PCR.EBioMedicine, 2017, 21: 86–93.DOI: 10.1016/j.ebiom.2017.06.003 |
