

齐鲁工业大学 生物工程学院,山东 济南 250353
收稿日期:2016-06-22;接收日期:2016-12-6 基金项目:山东省自主创新及成果转化专项(No. 201422CX02602) 资助
摘要: Pxa1p为酿酒酵母Saccharomyces cerevisiae过氧化物酶体上的膜蛋白,与Pxa2p组成二聚体,参与转运长链脂肪酸进入过氧化物酶体过程。热带假丝酵母能够发酵烷烃和脂肪酸生产长链二元酸,而过氧化物酶体中发生的β-氧化会消耗产生的长链二元酸造成产率降低。本研究以热带假丝酵母Candida tropicalis1798为宿主菌,通过基于PCR片段的同源单交换法,快速构建ctpxa1基因敲除菌株C. tropicalis1798-pxa1。利用半定量RT-PCR技术,检测ctpxa1基因在C. tropicalis1798、C. tropicalis1798-pxa1的表达量,灰度值比值为2.03,表明ctpxa1在C. tropicalis1798-pxa1中的表达被弱化。经144 h发酵,C. tropicalis1798-pxa1比C. tropicalis1798的十二碳二元酸产量明显提升,其产出浓度为10.3 g/L,比野生型菌株C. tropicalis 1798提高了94.3%。
关键词: 热带假丝酵母 长链二元酸 ctpxa1p基因 过氧化物酶体
Effect of ctpxa1 gene deletion in Candida tropicalis on long chain dicarboxylic acid accumulation
Cheng Cheng, Junqing Wang, Tengfei Wang, Xiaohui Yang, Ruiming Wang


College of Biological Engineering, Qilu University of Technology, Jinan 250353, Shandong, China
Received: June 22, 2016; Accepted: December 6, 2016
Supported by: Shandong Province Independent Innovation and Achievement Transformation Project (No. 201422CX02602)
Corresponding authors:Ruiming Wang. Tel: +86-531-89631076; E-mail: ruiming3k@163.com
Abstract: Candida tropicalis uses alkanes and fatty acids to produce long chain dicarboxylic acids. However, the yield can be affected by β-oxidation in peroxisomes. Pxa1p was a membrane protein of Saccharomyces cerevisiaeperoxisomes. Pxa1p and Pxa2p form a dimer that is involved in transporting of long chain fatty acids into peroxisomes, but the similar transporting system of Candida tropicalis has not yet been reported. In this study, a ctpxa1 gene deletion strain named C. tropicalis1798-pxa1 was constructed by homologous single exchange method using PCR fragment. The expression of ctpxa1 gene in C. tropicalis1798, C. tropicalis1798-pxa1 was detected by semi-quantitative RT-PCR, and the ratio of gray value was 2.03, implying that the expression of ctpxa1 in C. tropicalis1798-pxa1 was weakened. After 144 h fermentation, the dodecanedioic acid production of C. tropicalis1798-pxa1 was increased 94.3% than the former strain, the maximum yield was 10.3 g/L.
Key words: Candida tropicalis long chain dicarboxylic acid ctpxa1pgene peroxisomes
长链二元酸是一类重要的化工原料,可用于合成人工麝香类香料、尼龙、工程塑料、热熔胶、树脂、高级润滑油、耐寒增塑剂及液晶等物质。热带假丝酵母是重要的工业菌种,能够利用烷烃和脂肪酸作为唯一碳源和能源合成长链二元酸[1-3],当以烷烃或脂肪酸为碳源时,在细胞内可形成大量的过氧化物酶体,同时诱导生成脂肪酸β-氧化酶系[4-5],二元酸可以进入过氧化物酶体经β-氧化降解为细胞生命活动提供能量,但该过程通常会导致二元酸的过量消耗,因此对于提高烷烃的转化率和二元酸的产量是极为不利的[6-8]。许家齐等研究结果表明,在酿酒酵母Saccharomyces cerevisiae过氧化物酶体上存在一条能够特异性转运长链脂肪酸的转运体系,其中Pxa1p为过氧化物酶体的膜蛋白,与Pxa2p组成二聚体,参与转运长链脂肪酸进入过氧化物酶体中,是长链脂肪酸β-氧化重要环节之一[9-11],而Dulermo等则发现解脂耶氏酵母Yarrowia lipolytica中存在类似于S.cerevisiae的脂肪酸转运系统YlPxa1p-YlPxa2p,但该系统功能与S.cerevisiae的Pxa1p-Pxa2p转运体系具有较大的差异性[12-15],而关于其他酵母菌种特别是假丝酵母菌株的相关转运体系尚未见报道。
本研究通过BLAST方法在NCBI数据库中发现C.tropicalisMYA-3404中存在一种与S.cerevisiae中Pxa1p蛋白氨基酸序列一致性为41%的蛋白CTRT_01043(NCBI No.EER36303.1),并命名为CtPxa1p。为进一步验证CtPxa1p蛋白的功能,通过重叠PCR构建ctpxa1p基因敲除的片段pxa1p1-kanr,酶切后经电转化至C.tropicalis 1798中,利用同源单交换的原理[16],快速构建ctpxa1p基因单敲除菌株C.tropicalis1798-pxa1。
1 材料与方法1.1 材料1.1.1 菌种、质粒、引物热带假丝酵母购自中国工业微生物菌种保藏中心(CICC),编号为1798;pPIC 9K为本实验室保藏。
1.1.2 试剂限制性内切酶购自TaKaRa公司。Pfu DNA聚合酶、T4 DNA连接酶、DNA marker及相关分子生物学试剂盒均为生工生物工程(上海) 股份有限公司产品。酵母浸粉和蛋白胨为北京奥博星生产。其他试剂均为国产分析纯。
1.1.3 培养基种子培养基:葡萄糖20 g/L,蛋白胨20 g/L,酵母浸粉10 g/L。
发酵培养基:葡萄糖62 g/L,酵母浸粉2 g/L,(NH4)2SO41 g/L,VB1 0.2 g/L,NaCl 2 g/L,KH2PO4 8 g/L,Na2HPO4·12H2O 10.08 g/L,尿素3 g/L,Mg2SO4·7H2O 6.15 g/L。
1.2 方法1.2.1 同源臂pxa1p1和抗性基因kanr获取用S.cerevisiae中Pxa1p的氨基酸序列(GenBank Accession No.AJW19428.1) 进行Blast搜索,获得热带假丝酵母中同源序列,并以此设计引物pxa1p F1和pxa1p R1。使用上海生工酵母基因组DNA抽提试剂盒提取C.tropicalis 1798菌体的DNA,并以此为模板,使用引物pxa1p F1和pxa1p R1进行PCR扩增,PCR扩增条件为:95 ℃预变性5 min;94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min,4 ℃保存。扩增获得长度为553 bp用于pxa1p基因敲除的同源臂pxa1p1;使用高纯度质粒小量快速提取试剂盒提取质粒pPIC 9K,以质粒pPIC 9K为模板,使用引物kan F1和kan R1进行PCR扩增,PCR扩增条件为:95 ℃预变性5 min;94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸3.5 min,30个循环;72 ℃延伸10 min,4 ℃保存。扩增获得长度为1 561 bp G418抗性基因片段kanr;使用SanPrep柱式DNA胶回收试剂盒(上海生工生物工程股份有限公司) 胶回收制得的pxa1p1片段与kanr片段,保存于-20 ℃备用。
1.2.2 敲除片段的构建以胶回收制得的pxa1p1片段与kanr片段为模板,pxa1p F1和kan R1为引物进行重叠延伸PCR扩增,PCR扩增条件为:1)95 ℃预变性5 min;94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸3.5 min,5个循环;72 ℃延伸2 min;2)95 ℃预变性5 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸4.5 min,30个循环;72 ℃延伸10 min,4 ℃保存。扩增得到长度为2 148 bp的同源重组片段pxa1p1-kanr,过程如图 1所示,使用SanPrep柱式DNA胶回收试剂盒进行胶回收,保存于-20 ℃备用。
 |
| 图 1 C. tropicalis1798-pxa1工程菌的构建流程 Figure 1 Flowchart of construction of C. tropicalis1798-pxa1. |
| 图选项 |
1.2.3 同源重组片段pxa1p1-kanr酶切及浓缩同源重组片段pxa1p1-kanr经EcoR I酶切后,加入酶切产物1/10体积的3 mol/L NaAc和2.5倍体积的无水乙醇,-20 ℃冷藏20 min,12 000 r/min离心5 min,倒掉上清液;加入300 μL 70%乙醇重悬清洗一次;12 000 r/min离心5 min,倒掉上清液;37 ℃风干30 min,除去乙醇,加16 μL去离子水重悬DNA。使用核酸超微量分光光度计(BioFuture MD2000) 测定回收DNA浓度,并最终获得浓度在500-1 000 ng/μL之间的DNA溶液。
1.2.4 热带假丝酵母感受态制备及电击转化挑取新鲜固体YPD培养基表面的C.tropicalis 1798单菌落,接种20 mL液体YPD培养基30 ℃培养16-18 h,接种过夜培养菌体5-50 mL新鲜液体YPD培养基30 ℃培养10 h,使菌体浓度达到1×108个/mL,3 000 r/min离心10 min收集菌体,倒掉上清,菌体用20 mL无菌水悬浮,洗涤2次;离心收集菌体,用1 mL 1 mol/L山梨醇重新悬浮菌体,洗涤1次,转移至5 mL离心管中;离心收集菌体,移液器吸净上清,加入0.2 mL 1 mol/L山梨醇混匀菌体,使菌体呈浓浆状,取0.2-0.4 mL的菌悬液分装在1.5 mL的离心管,每管中加入10 μL (1 μg) DNA,混匀,冰浴10 min;将转化菌悬液全部转入已预冷的转化池中,1 500 V、5 ms下使用Eppendorf Mulultiporator电转化仪进行转化;用等体积的1 mol/L山梨醇将转化菌悬液从转化池中洗出,取200 μL涂布于添加了一定浓度G418的YPD平板上,30 ℃培养箱培养2-3 d,筛选抗G418的菌株。
1.2.5 阳性重组菌株的鉴定挑取上述单菌落于50 mL液体YPD培养基中,30 ℃培养12-14 h,使用Ezup柱式酵母基因组DNA抽提试剂盒(上海生工生物工程股份有限公司) 提取基因组,以提取的基因组为模板,pxa1p F1、kan R1为引物进行PCR扩增,扩增产物利用1%琼脂糖凝胶电泳进行验证,最终获得阳性重组菌株。
1.2.6 C.tropicalis 1798-pxa1菌株ctpxa1基因表达量利用UNIQ柱式Trizol总RNA抽提试剂盒提取总RNA,并按照TUREscript 1st Strand cDNA Synthcsis Kit的要求将总RNA反转录合成cDNA,作为ctpxa1基因PCR扩增的模板,以pxa1p F2、pxa1p R2为引物进行PCR扩增,扩增产物利用1%琼脂糖凝胶电泳进行验证,并通过Quantity One软件对灰度值进行分析,计算C.tropicalis1798与C.tropicalis1798-pxa1菌株中ctpxa1基因的灰度值比值。
1.2.7 种子培养取100 μL甘油管保藏的热带假丝酵母接种于50 mL液体YPD培养基,30 ℃、220 r/min培养12 h,然后在装有固体YPD培养基的平板上划线,30 ℃恒温培养箱培养12-16 h,挑取单菌落,转接于装液量为50 mL种子培养基的250 mL三角瓶,30 ℃、220 r/min摇床培养12 h[17-19]。
1.2.8 发酵培养取种子培养液转接于装液量为50 mL发酵培养基的500 mL挡板瓶中,接种量为10%,30 ℃、220 r/min摇床培养,每2 h取样,发酵液稀释一定倍数,蒸馏水为对照,测定OD600,待生长稳定后调节发酵液pH为7.5,加入5%十二烷烃,进入产酸期,在产酸期,每12 h调节pH到7.5。
1.2.9 十二碳二元酸的提取与测定将发酵液离心,回收上层未利用的十二烷,用滤纸过滤上清液。取3 mL过滤液置于碘量瓶中,加适量玻璃珠,在沸水浴中加热,用3 mol/L盐酸调pH至3.0,加入100 mL乙醚,摇瓶至十二碳二元酸完全溶解于乙醚中,静置30 min后取50 mL乙醚提取液于100 mL烧杯内,在通风橱中吹去乙醚,得白色固体十二碳二元酸。将提取到的二元酸加入25 mL 95%中性乙醇溶解,加入2滴酚酞,用标准氢氧化钠溶液滴定,记录消耗的氢氧化钠体积,计算十二碳二元酸的产量。
2 结果与分析2.1 氨基酸序列比对目前关于酵母中pxa1p基因功能的研究主要集中在S.cerevisiae和Y.lipolytica中。通过NCBI分别检索S.cerevisiae中Pxa1p (870 aa) 和Y.lipolytica中YlPxa1p (NCBI No.AJW19428.1,930 aa) 蛋白的氨基酸序列,并利用DNAMAN进行序列比对分析,结果如图 2所示。分析发现,C.tropicalis中的CtPxa1p长度为732 aa,短于S.cerevisiae和Y.lipolytica中对应的蛋白序列,同时虽然C.tropicalis中的CtPxa1p序列与S.cerevisiae和Y.lipolytica中对应的蛋白序列都属于ABC跨膜转运蛋白家族,并定位于过氧化物酶体膜上,但与两者相比氨基酸序列一致性较低,分别仅为41%和38%,因此C.tropicalis中的CtPxa1p蛋白的具体功能还需要进一步研究。
 |
| 图 2 氨基酸序列相似性分析 Figure 2 Putative amino acid sequence alignment. |
| 图选项 |
2.2 C.tropicalis 1798-pxa1工程菌的构建以C.tropicalis 1798基因组为模板,以pxa1p F1、pxa1p R1为引物,PCR克隆同源臂pxa1p1,获得大小为550 bp左右的条带,结果如图 3A所示,与理论值553 bp接近。以pPIC 9K基因组为模板,以kan F1、kan R1为引物,克隆kanr基因,获得大小为1 600 bp左右的条带(图 3B),与理论值1 561 bp接近。重叠PCR将同源臂pxa1p1与kanr基因连接起来,得到pxa1p1-kanr片段,获得大小为2 100 bp左右的条带(图 3C),与理论值2 148 bp接近,该结果表明通过重叠PCR方法成功获得了pxa1p1和kanr的融合片段。
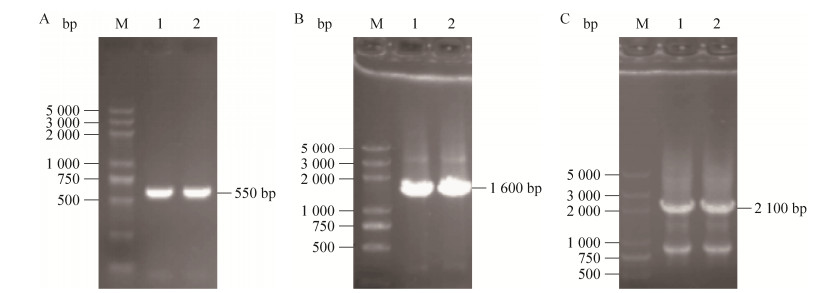 |
| 图 3 PCR扩增目的基因 Figure 3 PCR amplification of the target gene. (A) PCR product of homology arms pxa1p1 gene. M: marker DL5000; 1-2: 550 bp DNA fragment of pxa1p1 gene from C. tropicalis 1798. (B) PCR product of kanr gene. M: marker DL5000; 1-2: 1 600 bp DNA fragment of kanr gene from plasmid pPIC 9K. (C) Product of overlap PCR. M: marker DL5000; 1-2: 2 100 bp DNA fragment by overlap PCR. |
| 图选项 |
2.3 pxa1p1-kanr电转化及鉴定用EcoR Ⅰ酶切1 mL pxa1p1-kanr片段,并用乙醇浓缩法浓缩获得的酶切片段,使用核酸超微量分光光度计(BioFuture MD2000) 测定回收DNA浓度,结果显示DNA浓度为564.32 ng/μL。将10 μL回收片段与100 μL C.tropicalis1798感受态细胞混合并电转化,并使用引物pxa1p F3和kan R1对含G418(终浓度800 μg/mL) 平板上生长菌落的基因组进行PCR扩增,扩增产物利用1%琼脂糖凝胶电泳进行验证,获得大小为2 200 bp左右的条带(图 4),与理论值2 169 bp接近,表明pxa1p1-kanr与pxa1发生同源重组,完成ctpxa1基因的敲除。
 |
| 图 4 工程菌C. tropicalis1798-pxa1的PCR验证 Figure 4 PCR verification of C. tropicalis1798-pxa1. M: marker DL5000; 1-2: 3 700 bp PCR product of C. tropicalis1798-pxa1. |
| 图选项 |
2.4 C.tropicalis 1798、C.tropicalis 1798-pxa1菌株中ctpxa1基因表达量提取C.tropicalis1798、C.tropicalis1798-pxa1总RNA进行半定量RT-PCR,扩增产物利用1%琼脂糖凝胶电泳进行验证,结果如图 5所示,通过Quantity One软件对灰度值进行分析,ctpxa1基因在C.tropicalis1798、C.tropicalis1798-pxa1灰度值比值为2.03,证明ctpxa1基因在C.tropicalis1798-pxa1的表达量下降。
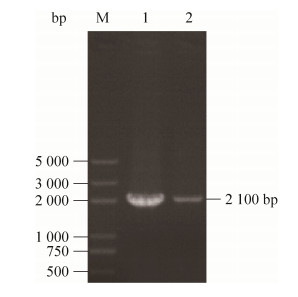 |
| 图 5 PCR扩增目的基因 Figure 5 PCR amplification of the target gene. M: marker DL5000; 1: 2 100 bp PCR product of C. tropicalis1798; 2: 2 100 bp PCR product of C. tropicalis1798-pxa1. |
| 图选项 |
2.5 C.tropicalis 1798、C.tropicalis 1798-pxa1生长曲线取种子培养液转接于装液量为50 mL发酵培养基的500 mL挡板瓶中,接种量为10%,30 ℃、220 r/min,每2 h取样一次,发酵液稀释一定倍数,蒸馏水为对照,测定OD600,结果如图 6所示。从生长曲线可以看出C.tropicalis1798与C.tropicalis1798-pxa1生长情况相近,表明以葡萄糖为碳源培养时,ctpxa1基因敲除对C.tropicalis 1798的生长没有显著影响。
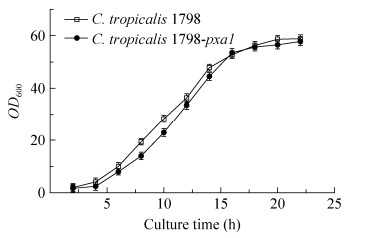 |
| 图 6 C. tropicalis 1798和C. tropicalis 1798-pxa1生长曲线 Figure 6 The growth curves of C. tropicalis 1798 and C. tropicalis 1798-pxa1. |
| 图选项 |
2.6 以十二烷为底物C.tropicalis 1798-pxa1发酵验证发酵培养12 h后加入5 mL的十二烷烃,每12 h调节pH到7.5并取样,用标准氢氧化钠溶液滴定,测得十二碳二元酸的浓度,结果如图 7所示。结果显示C.tropicalis1798-pxa1比C.tropicalis1798产十二碳二元酸的量明显提升,其产出浓度为10.3 g/L,比C.tropicalis1798产十二碳二元酸提高了94.3%,同时产酸阶段的时间也由野生型的120 h延长至144 h,表明ctpxa1基因敲除提高了热带假丝酵母产十二碳二元酸的能力,同时也延长了产酸期。
 |
| 图 7 C. tropicalis 1798和C. tropicalis 1798-pxa1发酵十二烷烃曲线 Figure 7 Fermentation curves of dodecane by C. tropicalis 1798 and C. tropicalis 1798-pxa1. |
| 图选项 |
3 讨论在二元酸发酵过程中,烷烃氧化为一元脂肪酸后,有两条代谢途径:ω-氧化和β-氧化。ω-氧化将一元酸的ω碳末端进一步氧化,形成目的产物二元酸,而β-氧化则将中间产物一元酸以及目的产物降解,从而进入TCA循环,为细胞生长提供能量。目前针对热带假丝酵母的基因工程改造多集中在提高ω-氧化的过程上,以降低β-氧化为目的的菌株改造报道较少,Picataggio等[20]阻断β-氧化中脂酰辅酶A氧化酶提高了长链二元酸的转化率。高弘等[21-22]通过敲除发现肉毒碱乙酰转移酶基因(CAT) 阻断乙酰辅酶A由过氧化物酶体到线粒体过程的转运,减少烷烃通过β-氧化途径的消耗,能够提高烷烃的转化率,但CAT基因的缺失会影响菌体代谢短链和中链脂肪酸的过程,进而影响酵母的生长和产酸的过程。长链脂肪酸转运蛋白Pxa1p定位在S.cerevisiae中的过氧化物酶体膜上,特异性介导长链脂肪酸进入过氧化物酶体的过程,但该蛋白在假丝酵母中与长链二元酸的消耗的关系尚未明晰。本研究首选通过基因比对分析找到热带假丝酵母潜在的长链脂肪酸转运蛋白,并通过基因敲除手段对该蛋白的功能进行分析,探索阻碍部分长链脂肪酸进入过氧化物酶体,减少长链脂肪酸消耗的新途径。构建菌株C.tropicalis 1798-pxa1较原始菌株C.tropicalis 1798十二碳二元酸产量提高了94.3%,产出浓度为10.3 g/L。由此推测ctpxa1基因的敲除,阻断了部分长链脂肪酸进入过氧化物酶体中,减弱了β-氧化反应与长链二元酸的消耗,提高了长链二元酸的产量。
酵母菌中目的基因敲除常用的方法是双交换同源重组[23-24],即采用以质粒为载体的同源双交换法,获取目的基因上下游两段基因序列作为同源臂,构建穿梭载体或自杀载体并转化到目的菌株中,转化后需先后发生两次单交换,实现目的基因的敲除[25]。本研究与双交换同源重组之间的区别在于目的基因的敲除利用的是一次单交换,并且通过重叠PCR方法直接构建热带假丝酵母敲除元件pxa1p-kanr,省去构建基因敲除载体及测序的过程。该方法具有简单、稳定、易行和节约时间等优点,同时由于所构建的片段较短,因此转化效率相对较高。该方法对其他微生物的基因敲除也有一定的借鉴意义。
参考文献
| [1] | Hattori K, Suzuki T. Microbial production of d-arabitol by n-alkane-grown Candida tropicalis.Agric Biol Chem, 2014, 38(10): 1875–1881. |
| [2] | Ferdinandusse S, Denis S, van Roermund CWT, et al. Identification of the peroxisomal β-oxidation enzymes involved in the degradation of long-chain dicarboxylic acids.J Lipid Res, 2004, 45(6): 1104–1111.DOI: 10.1194/jlr.M300512-JLR200 |
| [3] | Wang JX, Ding HY, He LF, et al. Influence of growth factor and emulsifier on dodecanedioic acid fermentation by Candida tropicalis.China Brew, 2011(5): 87–89.(in Chinese). 王佳新, 丁海燕, 何连芳, 等. 生长因子和乳化剂对热带假丝酵母产十二碳二元酸发酵的影响.中国酿造, 2011(5): 87-89. |
| [4] | Lasorsa FM, Scarcia P, Erdmann R, et al. The yeast peroxisomal adenine nucleotide transporter: characterization of two transport modes and involvement in ΔpH formation across peroxisomal membranes.Biochem J, 2004, 381(3): 581–585.DOI: 10.1042/BJ20040856 |
| [5] | Van Roermund CWT, Ijlst L, Majczak W, et al. Peroxisomal fatty acid uptake mechanism in Saccharomyces cerevisiae.J Biol Chem, 2012, 287(24): 20144–20153.DOI: 10.1074/jbc.M111.332833 |
| [6] | Wang JJ, Zhang B, Chen SL. Oleaginous yeast Yarrowia lipolytica mutants with a disrupted fatty acyl-CoA synthetase gene accumulate saturated fatty acid.Process Biochem, 2011, 46(7): 1436–1441.DOI: 10.1016/j.procbio.2011.03.011 |
| [7] | Beopoulos A, Mrozova Z, Thevenieau F, et al. Control of lipid accumulation in the yeast Yarrowia lipolytica.Appl Environ Microbiol, 2008, 74(24): 7779–7789.DOI: 10.1128/AEM.01412-08 |
| [8] | Court DL, Sawitzke JA, Thomason LC. Genetic engineering using homologous recombination.Annu Rev Genet, 2002, 36(1): 361–388.DOI: 10.1146/annurev.genet.36.061102.093104 |
| [9] | Choi BK, Bobrowicz P, Davidson RC. Use of combinatorial genetic libraries to humanize N-linked glycosylation in the yeast Pichia pastoris.Proc Natl Acad Sci USA, 2003, 100(9): 5022–5027.DOI: 10.1073/pnas.0931263100 |
| [10] | Liu SC, Li C, Fang XC, et al. Optimal pH control strategy for high-level production of long-chain α, ω-dicarboxylic acid by Candida tropicalis.Enzyme Microb Technol, 2004, 34(1): 73–77.DOI: 10.1016/j.enzmictec.2003.09.001 |
| [11] | Beopoulos A, Nicaud JM, Gaillardin C. An overview of lipid metabolism in yeasts and its impact on biotechnological processes.Appl Microbiol Biotechnol, 2011, 90(4): 1193–1206.DOI: 10.1007/s00253-011-3212-8 |
| [12] | Liu SC, Li SL, Yang D, et al. Fermentation of 1, 13-tridecane dicarboxylic acid in an industrial pilot plant.Petrochem Technol, 2002, 31(7): 558–561.(in Chinese). 刘树臣, 李淑兰, 杨东, 等. 十三碳二元酸发酵工业试验.石油化工, 2002, 31(7): 558-561. |
| [13] | Green KD, Turner MK, Woodley JM. Candida cloacae oxidation of long-chain fatty acids to dioic acids.Enzyme Microb Technol, 2000, 27(3/5): 205–211. |
| [14] | Ma YC, Yu HM, Pan WY, et al. Identification of nitrile hydratase-producing Rhodococcus ruber TH and characterization of an amiE-negative mutant.Bioresour Technol, 2010, 101(1): 285–291.DOI: 10.1016/j.biortech.2009.07.057 |
| [15] | De Crescenzo G, Grothe S, Zwaagstra J, et al. Real-time monitoring of the interactions of transforming growth factor-β (TGF-β) isoforms with latency-associated protein and the ectodomains of the TGF-β type Ⅱ and Ⅲ receptors reveals different kinetic models and stoichiometries of binding.J Biol Chem, 2001, 276(32): 29632–29643.DOI: 10.1074/jbc.M009765200 |
| [16] | Teder T, Boeglin WE, Brash AR. Lipoxygenase-catalyzed transformation of epoxy fatty acids to hydroxy-endoperoxides: a potential P450 and lipoxygenase interaction.J Lipid Res, 2014, 55(12): 2587–2596.DOI: 10.1194/jlr.M054072 |
| [17] | Uccelletti D, Farina F, Mancini P, et al. KlPMR1 inactivation and calcium addition enhance secretion of non-hyperglycosylated heterologous proteins in Kluyveromyces lactis.J Biotechnol, 2004, 109(1/2): 93–101. |
| [18] | van Roermund CWT, Drissen R, van den Berg M, et al. Identification of a peroxisomal ATP carrier required for medium-chain fatty acid β-oxidation and normal peroxisome proliferation in Saccharomycescerevisiae.Mol Cell Biol, 2001, 21(13): 4321–4329.DOI: 10.1128/MCB.21.13.4321-4329.2001 |
| [19] | Cho YH, Lee HJ, Lee JE, et al. Fast determination of multiple-reaction intermediates for long-chain dicarboxylic acid biotransformation by gas chromatography-flame ionization detector.J Microbiol Biotechnol, 2015, 25(5): 704–708.DOI: 10.4014/jmb.1502.02026 |
| [20] | Picataggio S, Rohrer T, Deanda K, et al. Metabolic engineering of Candida tropicalis for the production of long-chain dicarboxylic acids.Nat Biotechnol, 1992, 10(8): 894–898.DOI: 10.1038/nbt0892-894 |
| [21] | Gao H, Zhang J, Hua YT, et al. Effects of carnitine acetyltransferase gene knockout on long chain dicarboxylic acid production and metabolism of Candida tropicalis.Acta Microbiol Sin, 2005, 45(1): 102–105.(in Chinese). 高弘, 张剑, 华玉涛, 等. 肉毒碱乙酰转移酶基因敲除对长链二元酸生产代谢过程的影响.微生物学报, 2005, 45(1): 102-105. |
| [22] | Gao H, Huang YM, Liu M, et al. Effects of CAT gene knockout on DCA13 metabolic network of Candida tropicalis.J Chem Ind Eng (China), 2006, 57(9): 2157–2161.(in Chinese). 高弘, 黄英明, 刘铭, 等. CAT基因敲除对热带假丝酵母产DCA13代谢网络的影响.化工学报, 2006, 57(9): 2157-2161. |
| [23] | Xu Y, Tu Z. Application and progress of filamentous fungi gene targeting.J Food Sci Biotechnol, 2007, 26(1): 120–126.(in Chinese). 许杨, 涂追. 丝状真菌基因敲除技术研究进展.食品与生物技术学报, 2007, 26(1): 120-126. |
| [24] | Xiang Z, Chen XZ, Zhang LH, et al. Development of a genetic transformation system for Candida tropicalisbased on a reusable selection marker of URA3gene.Hereditas (Beijing), 2014, 36(10): 1053–1061.(in Chinese). 项峥, 陈献忠, 张利华, 等. 利用可重复使用的URA3标记基因建立热带假丝酵母基因敲除系统.遗传, 2014, 36(10): 1053-1061. |
| [25] | Xue KJ, Liu B, Chang SH, et al. A high efficient method to knockout target gene by two-step homologous recombination in Pichia pastoris.Lett Biotechnol, 2010, 21(5): 650–654.(in Chinese). 薛奎晶, 刘波, 唱韶红, 等. 一种两步基因同源重组敲除酵母靶标蛋白基因的方法.生物技术通讯, 2010, 21(5): 650-654. |
