

1. 武汉大学药学院 组合生物合成与新药发现教育部重点实验室,湖北 武汉 430072
2. 军事医学科学院放射与辐射医学研究所 国家蛋白质科学中心 (北京) 北京蛋白质组研究中心 蛋白质组学国家重点实验室,北京 102206
摘要:蛋白质泛素化是以泛素单体和泛素链作为信号分子,共价修饰细胞内其他蛋白质的一种翻译后修饰形式。不同蛋白质底物、同一底物的不同氨基酸修饰位点以及同一位点上泛素链连接方式的不同均可导致细胞效应的差异。蛋白质泛素化在真核细胞内广泛存在,除了介导蛋白质的26S蛋白酶体降解途径之外,还广泛参与了基因转录、蛋白质翻译、信号传导、细胞周期控制以及生长发育等几乎所有的生命活动过程。泛素链的形成及其修饰过程的任何失调均可导致生物体内环境的紊乱,从而产生严重的疾病。文中结合实验室研究,综述了泛素的发现历史、基因特点、晶体结构,特别是泛素链的组装过程、结构、功能以及与人类相关疾病关系的新进展,可为这些疾病的治疗靶点和药物靶标的研究提供思路。
关键词: 泛素泛素链晶体结构赖氨酸残基疾病
Progress in ubiquitin, ubiquitin chain and protein ubiquitination
Qiuyan Lan1, 2, Yuan Gao2, Yanchang Li2, Xuechuan Hong1, Ping Xu1, 2


1. Key Laboratory of Combinatorial Biosynthesis and Drug Discovery (Wuhan University), Ministry of Education, Wuhan University School of Pharmaceutical Sciences, Wuhan 430072, Hubei, China
2. State Key Laboratory of Proteomics, Beijing Proteome Research Center, Beijing Institute of Radiation Medicine, National Center for Protein Sciences Beijing, Beijing 102206, China
Received: March 29, 2015; Accepted: June 4, 2015
Supported by: National Basic Research Program of China (973 Program) (Nos. 2011CB910600, 2013CB911200), National HighTechnology Research and Development Program (863 Program) (Nos. SS2012AA020502, 2011AA02A114), National Natural ScienceFoundation of China (Nos. 31070673, 31170780), National Mega Projects for Key Infectious Diseases (No. 2013zx10003002), KeyProjects in the National Science & Technology Pillar Program (No. 2012BAF14B00).
Corresponding authors: Ping Xu. Tel: +86-10-83147777-1314; Fax: +86-10-80705155; E-mail: xupingghy@gmail.com
Abstract: Protein ubiquitination is one of the most important and widely exist protein post-translational modifications in eukaryotic cells, which takes the ubiquitin and ubiquitin chains as signal molecules to covalently modify other protein substrates. It plays an important roles in the control of almost all of the life processes, including gene transcription and translation, signal transduction and cell-cycle progression, besides classical 26S protesome degradation pathway. Varied modification sites in the same substrates as well as different types of ubiquitin linkages in the same modification sites contain different structural information, which conduct different signal or even determine the fate of the protein substrates in the cell. Any abnormalities in ubiquitin chain formation or its modification process may cause severe problem in maintaining the balance of intracellular environment and finally result in serious health problem of human being. In this review, we discussed the discovery, genetic characteristics and the crystal structure of the ubiquitin. We also emphasized the recent progresses of the assembly processes, structure and their biological function of ubiquitin chains. The relationship between the disregulation and related human diseases has also been discussed. These progress will shed light on the complexity of proteome, which may also provide tools in the new drug research and development processes.
Keywords: ubiquitinubiquitin chainscrystal structurelysineiseases
蛋白质是细胞的主要组成成分,也是细胞功能的直接执行者。蛋白质功能的发挥依赖于细胞内多样的蛋白质翻译后修饰,其中泛素化修饰在真核细胞内普遍存在。已知人体内参与蛋白质泛素化的泛素激活酶E1、泛素耦联酶E2、泛素连接酶E3以及脱泛素化酶DUB等的编码基因数目超过1 000种,远多于细胞内负责重要生物学功能的蛋白质磷酸化相关激酶和磷酸酶的基因数量[1, 2, 3],预示着这种翻译后修饰的重要性和调控的复杂性。在泛素化蛋白质的结构上,由于底物蛋白上存在多个赖氨酸残基,这些赖氨酸位点中的一个或多个可同时被泛素修饰,形成了蛋白质翻译后修饰的宏观不均一性。即使在同一修饰位点也可形成不同赖氨酸参与的同质泛素链或者混合泛素链的修饰形式;而进一步深入研究还发现泛素分子的N端 (甲硫氨酸的游离氨基) 也可发生泛素化修饰形成线性泛素化链,从而产生复杂多样的蛋白质泛素化修饰中的微观不均一性[4, 5]。这些都从不同侧面反映了真核细胞中蛋白质泛素化修饰的普遍性、结构的多样性、调控的复杂性及功能的重要性[2]。
由于蛋白质的泛素化修饰影响甚至决定着被修饰蛋白质的命运,因此泛素化及其信号途径的任何突变或失调均可引发包括癌症、神经退行性疾病在内的多种严重而且难治愈的人类疾病。因此深入研究泛素及其修饰系统,鉴定泛素化底物蛋白,了解其调控机制对于对相关疾病分子机制的理解和治疗具有重要意义。本文就泛素和泛素链的结构、功能及泛素化修饰的多样性和复杂性等方面进行综述,并结合本实验室现有工作,介绍国际上相关方面研究进展和发展趋势。
1 泛素的发现 泛素最初在分离促胸腺生成素时在胸腺中被发现,并被认为是普遍存在于真核生物中的免疫生成性多肽UBIP。不同于促胸腺生成素仅促进T细胞分化的作用,泛素能同时促进T细胞和B细胞的分化[6]。随后Goldknopf和Busch发现泛素C末端可共价连接到组蛋白H2A赖氨酸残基的ε-氨基上[7],揭示了泛素可修饰其他蛋白质的特性。1978年,Hershko、Ciechanover以及他们的同事发现兔网织红细胞的无细胞裂解物具有依赖于ATP的降解某些底物蛋白质的功能[8]。随后Hough及其合作者从这种兔网织红细胞裂解物中成功地纯化到了能够降解多聚泛素-蛋白偶联物的26S蛋白酶体[9],完善了泛素-蛋白酶体系统学说。而泛素-蛋白酶体在系列生理过程中的重要作用及其分子机制是由Finely及其合作者阐明的[10],即泛素能够修饰底物蛋白,并以此为信号将底物引导到蛋白酶体进行特异性降解,从而维持机体内环境的稳态[11]。
2 高度保守的泛素基因及其编码的蛋白质序列 泛素这种普遍存在于真核生物体内的小蛋白在编码基因的结构和序列上都是非常保守的。已知在细胞中编码泛素蛋白的基因有多个,且在不同的物种中这些基因或基因簇在染色体上的位置各异。在不同的生理状态下,这些由不同基因座编码的泛素基因可特异地开启,并按照要求表达特定量的泛素蛋白,以满足不同生理条件的需要。
表1展示了泛素基因在不同物种中的分布情况。其中拟南芥中的泛素编码基因最多,高达14种,它们在染色体上的分布也最为分散。这些基因按照序列特点可分为多聚泛素编码基因、类泛素基因以及泛素延伸基因3类。如ubq3、ubq4、ubq10、ubq11和ubq14等多聚泛素基因都包含228 bp的泛素串联重复区域,但在同义替代、泛素编码区域的数量、非泛素部分C末端氨基酸以及染色体定位等方面存在一定的差异,而类泛素基因ubq7、ubq8、ubq9和ubq12也包含串联重复的泛素编码区域,但是至少每个重复基因编码一个发生了氨基酸替换的蛋白[12]。
表1 不同物种中泛素基因座位分布Table 1 Ubiquitin genes and their loci in varied species
| Species | Gene | Chromosome |
| Saaccharomyces cerevisiae | ubi1 | Ⅸ |
| ubi2 | Ⅺ | |
| ubi3, ubi4 | Ⅻ | |
| Arabidopsis thaliana | ubq12, ubq13 | Ⅰ |
| ubq2, ubq6, ubq7 | Ⅱ | |
| ubq1, ubq5, ubq8 | Ⅲ | |
| ubq10, ubq11, ubq14 | Ⅳ | |
| ubq3, ubq4, ubq9 | Ⅴ | |
| Drosophila | ubiquitin-5E | ⅹ |
| ubiquitin-63E | 3L | |
| Nematode | Polyubiquitin | Unknown |
| ubiquitin-60S ribosomal protein L40 | Unknown | |
| ubq-1, ubq-2 | Ⅲ | |
| Musculus | ubc | Ⅴ |
| uba52a | Ⅷ | |
| rps27a, ubb | Ⅺ | |
| mCG1048340 | XIV | |
| Homo sapiens | ubiquitin C | Ⅻ |
| Ubb | XVII | |
| ubiquitin A-52 residue ribosomal protein fusion product 1, ubiquitin-60S ribosomal protein L40 precursor | XIX |
表选项
除拟南芥外,大多数生物的泛素编码基因通常表达成多聚泛素前体、N端泛素基团分别与核糖体多肽L40或S27共融合表达的UbL40和UbS27融合蛋白 (图1) 等3种不同的前体结构。在酵母中,这几种泛素前体分别为由ubi1、ubi2和ubi3产生的融合蛋白以及由ubi4产生的多聚泛素链。有趣的是这4个基因都含有不同的内含子序列,其中ubi1、ubi2、ubi3和ubi4在5′端和3′端均有内含子的分布,但这些内含子序列相似度不高,表明它们可能具有不同的调节功能。ubi1 位于酵母基因组Ⅸ号染色体上,ubi2位于Ⅺ号染色体上。这两个基因的表达产物均含一个泛素多肽和一个完全相同的含52个残基的前体肽。ubi3位于Ⅻ号染色体上,编码一个泛素多肽和一个包含了76个氨基酸残基的前体肽,且这一前体肽与Ubi1和Ubi2前体肽的氨基酸序列相似性较低[13]。ubi4也位于酵母基因组的Ⅻ号染色体上,编码一个由5个泛素基团首尾相连而成的多聚泛素。该多聚泛素是高温、饥饿或氨基酸类似物刺激等应激条件下泛素的主要供应者[14]。这4种基因都能在指数生长的细胞中表达,但在稳定期中仅ubi3和ubi4可表达。
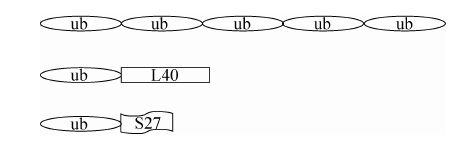 |
| 图1 泛素分子不同前体结构的示意图[16] Fig.1 Schematic representation of different ubiquitin precursor’s structures[16]. |
| 图选项 |
这4个基因编码的泛素的氨基酸序列完全相同,但它们各有不同的尾部序列。ubi1、ubi2 和ubi3的尾部片段含有预测的DNA结合区域以及类TFⅢA核酸结合区域,因此这些尾部区域可能与特定DNA结合。另有研究者认为泛素单体本身作为一种修饰泛素分子的蛋白,而未处理的泛素前体则可能是蛋白水解作用的一种调节信号[13]。这些泛素融合蛋白基因表达的蛋白可被特异性的内肽酶切开,释放出序列一致的泛素分子,进入细胞内游离的泛素库,供机体使用。而与核糖体蛋白融合表达的泛素单体不仅提供了细胞必需的泛素分子,还作为伴侣蛋白加速核糖体的组装,参与蛋白质合成机器的调控[15]。
尽管细胞内编码泛素的基因众多,但这一由76个氨基酸组成的蛋白质经过长时间的生物进化仍能在氨基酸序列上高度保守,这与该蛋白质在细胞内的重要功能相符。研究显示这些泛素分子在同种生物中的氨基酸序列完全一致,即使在不同的物种如芽殖酵母、植物和人体中也仅有4个氨基酸存在差异 (图2)。
 |
| 图2 不同物种中泛素氨基酸序列比对结果 Fig.2 Ubiquitin amino acids sequence alignment result of different species. |
| 图选项 |
不仅泛素本身的氨基酸序列保守,多聚泛素前体和UbL40的氨基酸序列在大多数物种中也是完全一致的,说明编码这两种蛋白的基因座之间存在频繁的重组事件。通过对编码这些泛素基因的密码子分析,研究者发现UbS27基因座的重组并不像多聚泛素或UbL40基因座那样均匀。对比预测的UbS27的二级结构与人类泛素分子的NMR结构显示泛素蛋白的整体结构似乎比序列更加保守,推测泛素结构限制了对UbS27序列退化的容忍程度。进一步研究推测多聚泛素基因座很可能作为一种重组的供体或者底物以维持非等位泛素基因的稳定,同时也为新的类泛素结构的产生提供序列源泉。比较泛素和类泛素蛋白质的序列,泛素蛋白中76个氨基酸有60个可发生替换形成类泛素蛋白,但新形成的类泛素蛋白分子的功能却没有发生改变,因此泛素折叠的结构需要而不是泛素蛋白的功能需要促成了泛素家族进化上的保守性[16]。
3 泛素蛋白质的结构 泛素以一种特殊的由5个β折叠包围着一个α螺旋的形式存在。这种SSHSSS的结构在类泛素化蛋白中也存在[16]。泛素分子表面存在几个对于其发挥生物学功能不可或缺的重要的疏水表面,其中由Ile44、Leu8、Val70以及His68组成的疏水口袋介导泛素单体与蛋白酶体和大多数泛素结合结构域 (UBDs) 结合,且这些保守序列对于细胞分裂以及蛋白酶体降解功能的发挥是必需的[17, 18]。以Ile36为中心,加上Leu71和Leu73形成的疏水表面介导了与泛素链的相互作用以及与HECT类泛素连接酶、去泛素化酶[19]和泛素结合结构域[20]的识别。由Gln2、Phe4和Thr12组成的表面可能参与转运以及与泛素特异性蛋白酶结构域和UBAN结构域的相互作用[21]。而由Thr12、Thr14、Glu34、Lys6和Lys11组成的TEK-box对于有丝分裂相关蛋白的降解是必需的[22]。
4 蛋白质的泛素化修饰和泛素链的特异性 泛素蛋白本身可作为信号分子,经由泛素激活酶E1、泛素偶联酶E2和泛素连接酶E3的级联反应共价修饰到底物蛋白质的赖氨酸残基上,由此实现蛋白质的泛素化。单个泛素分子修饰底物蛋白上的单个赖氨酸残基,可形成单泛素化修饰。多个泛素分子修饰底物蛋白上的多个赖氨酸残基,可形成多泛素化修饰。由于泛素分子本身携带有7个赖氨酸残基,已经共价修饰到底物蛋白上的泛素分子的赖氨酸残基还可继续发生泛素化修饰,形成泛素链。与此对应,泛素分子N端的甲硫氨酸也有一个自由的氨基,该氨基可串联泛素分子,形成线性泛素链。因此细胞中可存在单泛素化、多泛素化以及8类不同形式的同质泛素链修饰。如果一个泛素链上结合泛素本身不同的赖氨酸残基,形成混合或分支泛素链,则进一步增加了泛素链的复杂性 (图3)。定量蛋白质组学研究表明上述泛素链在细胞中均广泛存在[23],但其浓度存在一定的差异。在对数生长期酵母细胞中,七类由赖氨酸连接的同质泛素链K6∶K11∶K27∶K29∶K33∶K48∶K63的百分比约为11∶28∶9∶3∶4∶29∶16[24]。
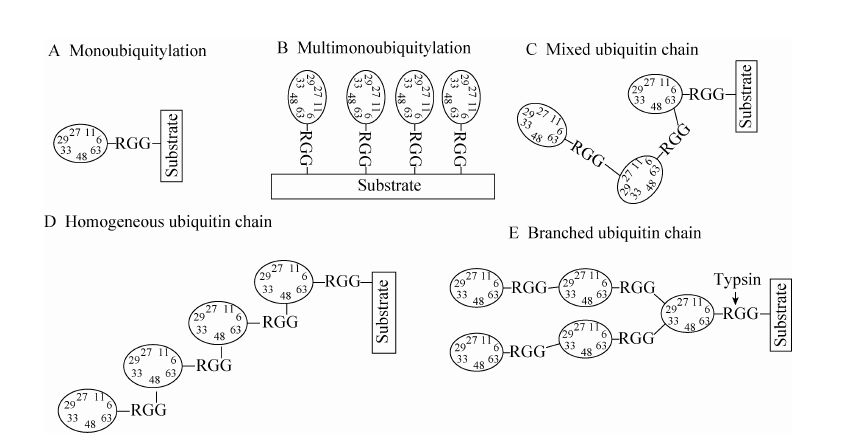 |
| 图3 不同泛素链修饰的示意图 Fig.3 Schematic representation of the different Ub modifications. |
| 图选项 |
5 泛素链的空间结构 不同的赖氨酸连接形成的泛素链空间结构各异。同质K48泛素链彼此紧紧包裹,产生一种紧凑的球状构象[25]。这种链通过与受体蛋白作用,将蛋白导向蛋白酶体并进行特异性降解。研究表明至少需要4个通过K48连接的泛素组成的泛素链才能将底物蛋白导向蛋白酶体[26]。泛素单体表面的疏水性残基Leu8、Ile44和 Val70对于蛋白酶体识别K48连接的泛素链十分重要[27]。在近中性pH (pH 6.7) 条件下,该泛素链的整体结构非常类似一种关闭的形式,其Ile44疏水口袋彼此相对[26, 27, 28];而K6和K11连接的泛素二聚体和泛素四聚体则呈现紧凑的空间结构。由于缺少分子间相互作用,Leu8、Ile44和Val70形成的疏水性口袋暴露在外,这有利于识别NEMO等伴侣蛋白[4, 20]。K11连接的泛素链也存在一定的结构灵活性,即在K11连接的二聚泛素链上有不对称的表面基团,并覆盖着其α螺旋,或者泛素单体通过Ile36口袋对称地相互作用[29, 30]。
在泛素结构中,M1和K63位的残基在空间上接近,因此M1和K63连接的泛素二聚体有相似但不完全相同的空间构象。它们的泛素间因没有任何联系从而都采取等效开放构象[31]。大部分含有泛素结合结构域的结合伴侣通过这种泛素链识别泛素基团之间的距离和灵活 性[32]。K27、K29、K33泛素链由于丰度低,富集困难,针对其晶体结构的研究还较少。
多样的泛素间连接形式形成了多样的泛素链结构。这些多样的结构可与相关蛋白的结构域发生特异而多样的相互作用,从而将底物蛋白导向蛋白酶体、溶酶体或其他信号通路,形成复杂的信号网络,最终调控细胞的重要生命活动。
6 不同泛素链的组装方式及其生物学功能 6.1 K48泛素链 以Sic1 (Cyclin依赖的激酶抑制剂) 为例,其泛素化可分为两步。首先是在cullin-RING泛素连接酶SCFCdc4与其泛素耦联酶Cdc34作用下,添加第一个泛素到Sic1上。随后快速延伸K48连接的泛素链。Cdc34酸性环偏好蛋白质底物泛素化中的K48连接的泛素链,攻击SCF结合的Cdc34-泛素硫酯从而持续合成K48连接的泛素链[33]。
和泛素链合成的严格受控相似,泛素链的降解也具有一定的特异性。在酵母中脱泛素化酶Ubp15与人的USP7同源,可快速移除泛素单体或二聚体以及K63连接的长泛素链,但移除K48连接的泛素链非常慢。这种对长泛素链的耐受性使K48链修饰的底物处于游离状态,直到某种泛素连接酶促使其发生降解[34]。相反,Ubp12很容易从末端开始裂解所有的泛素链。
K48泛素链的主要作用是负反馈调节蛋白质稳定性。已知K48泛素链的缺乏对细胞是致死的[24]。该泛素链通过与受体蛋白作用,将蛋白导向蛋白酶体降解[35, 36]。K48连接的泛素链也参与DNA损伤应答。当DNA损伤时,RNF8和RNF168泛素连接酶转移到DNA损伤位点,介导蛋白JMJD2A、JMJD2B和L3MBTL1的泛素化,一方面引导JMJD2A和JMJD2B到蛋白酶体降解,从而解除由于JMJD2A和JMJD2B结合在甲基化的组蛋白H4K20上而造成的53BP1 (肿瘤抑制因子p53结合蛋白1) 无法结合,促进CHK2的高效磷酸化,并激活p53。另一方面L3MBTL1的泛素化会促进VCP/p97和 RNF8/168依赖的染色体提取效应[37]。
6.2 K63泛素链 大多数情况下,合成K63连接的多聚泛素链依赖于一个额外的蛋白——UEV。该蛋白与泛素耦联酶Ubc13形成异二聚体[38]。E1催化硫醇转移反应将供体泛素加载到Ubc13的半胱氨酸活性位点上,此后第二个泛素单体加到UEV和E2上,第63位的赖氨酸侧链靠近供体泛素,并与Ubc13形成硫酯键。由此,K63能够攻击硫酯键使供体泛素从Ubc13的活性位点上转移到受体泛素的第63位赖氨酸侧链上,形成异肽键。最后供体泛素转移到受体位点成为受体泛素,继续后续的泛素化。经过多个循环可形成K63连接的泛素链[39]。另外含JAMM/MPN+ 结构的脱泛素化酶对K63泛素链的共有特异性也在实验中得到了证明[40]。
研究还发现哺乳动物细胞中存在一种可溶性因子ESCRT0以及它的组件STAM和Hrs,可选择性地结合K63连接的泛素链,阻止其与蛋白酶体结合,并将底物蛋白特异地定位到溶酶体上[41]。因此,K63泛素链在哺乳动物细胞中主要参与调节DNA损伤修复、线粒体遗传[42]、核糖体和NF-κB的功能[23,43-45]等非蛋白水解信号途径,并与遗传性帕金森病相关[46]。但在芽殖酵母中,HECT类泛素连接酶Rsp5能以K63连接的泛素链修饰Mga2-p120,将其转位到蛋白酶体中完成降解过程。同时该研究也发现了蛋白酶体对长的K63泛素链的偏好性[47]。
以亨廷顿舞蹈症为例,在年轻的亨廷顿蛋白基因敲入鼠中,K48泛素链特异性泛素连接酶Ube3a (也称E6AP) 含量很高,它以K48泛素链修饰突变的亨廷顿蛋白,并将其引导到蛋白酶体,进行特异性降解。随着年龄的增长,Ube3a的含量逐渐减少,蛋白酶体能够有效降解的突变亨廷顿蛋白逐渐减少,导致突变亨廷顿蛋白被K63泛素链修饰,进而发生蛋白质聚集,出现选择性神经退行性疾病,并最终形成亨廷顿舞蹈症[48]。
6.3 K11泛素链 K11连接的泛素链在体内和体外均作为蛋白酶体的高效靶向信号[49, 50]。不同于其他链,K11泛素链的高效合成依赖于泛素表面的TEK-box。有意思的是APC/C底物中同样发现了同源的TEK-box结构,它可促进成核反应。拓扑结构研究发现APC/C是一种Ring finger类泛素连接酶[51],猜测APC/C识别底物和泛素表面相同的TEK-box基序,以此促进第一个泛素到底物赖氨酸残基的转移。之后APC/C与其特异性的泛素耦联酶UbcH10借助泛素表面的TEK-box高效特异地组装K11链,从而实现严格的细胞周期调控[22]。
除泛素连接酶外,近年也有研究鉴定到了K11泛素链的其他特异性底物,如泛素耦联酶Ubc6。该酶主要合成K11泛素链,且在内质网相关降解途径中发挥作用[24]。另外OTU家族的去泛素化酶Cezanne特异性地对K11泛素链修饰的底物进行去泛素化[29],再次说明了DUB对泛素链的特异性。
6.4 K6泛素链 K6连接的泛素链可由Rad6 (酵母直接同源UbcH2) 催化形成。有趣的是在合适的E3存在时,Rad6也可以主要催化K48连接的泛素链合成。类似的情况在p53泛素化时也会发生。当存在E6AP时,UbcH5c主要催化p53发生K48泛素链修饰;但是当BRCA1/BARD1存在时,UbcH5c主要催化生成K6泛素链[52]。此外,红褐蛋白环指蛋白1 (MGRN1) 能以K6泛素链修饰α-tubulin[53]。除泛素化修饰的特异性外,最新研究表明去泛素化酶USP8能特异性消化Parkin蛋白上K6泛素链,从而招募Parkin到去极化的线粒体中,最后经线粒体自噬得以消除[54]。
K6连接的泛素链的生理功能至今不甚明了。已有的研究表明K6泛素链能够修饰一种肿瘤抑制因子BRCA1。BRCA1本身可与BARD1组成异二聚体,具有E3泛素连接酶的活性,催化泛素链的形成,并参与包括DNA修复、转录调节以及细胞周期检验点控制等[55]多种细胞过程。BRCA1的突变可能造成遗传性胸癌或卵巢癌[56]等多种人类严重疾病。虽然BRCA1泛素化后续的信号传递机制尚不清楚,但是这足以说明K6连接的泛素链对机体正常生命活动的重要性。
6.5 K27、29、33泛素链 由于K27/29/33连接的泛素链在生物体内的含量偏低,富集困难,因此针对这几种泛素链的研究还较少。但是仅有的一些研究已经表明这几种泛素链在生命活动中也扮演着重要的角色。
泛素连接酶TRAF6在组装K63连接的泛素链中发挥作用,但也促进K6、K27和K29泛素链在帕金森疾病相关蛋白DJ-1和α-synucluin上的组装,从而促进它们在细胞质中的积累。在宿主与病原相互作用时,两种不同的K27泛素链修饰NF-κB调节亚基IFFγ (也叫NEMO)。在病毒感染时,泛素连接酶TRIM23添加K27泛素链到IFFγ上,激活IFFγ并产生抗病毒干扰素β。相反,当志贺氏杆菌感染时,细菌泛素连接酶IpaH9.8添加另一种K27泛素链到IFFγ上,促进其降解,从而抑制NF-κB信号和宿主防御应答[57, 58]。转录调节蛋白Jun的半衰期也严格地受到K27泛素链的调控。在多个类型的细胞中很多泛素连接酶能特异性地催化Jun发生K27泛素链修饰,进而经由TSG101和HRS的识别将其转位到溶酶体,完成蛋白质自身的降解,以应答不同的刺激[59]。此外,最新的研究还发现K27连接的泛素链也在抑制自身免疫性疾病中发挥作用[60]。
Deltex (DTX) 和AIP4是果蝇deltex与其抑制基因的人类直系同源基因,参与抑制Notch信号途径。这两种基因分别编码RING-H2和HECT类泛素连接酶。研究发现这两种分子相互影响,并且部分共定位于内吞膜泡中。AIP4主要组装K29连接的泛素链,并将自身与DTX泛素化,引导DTX靶向溶酶体降解,表明K29连接形式与溶酶体降解和内吞作用相关[61]。另外K29连接的泛素链也参与蛋白酶体靶向信号,与招募底物蛋白解折叠伴侣蛋白 (如VCP/Cdc48p) 的过程相关[62]。
最近研究发现一种新的泛素连接酶CUL3-KLHL20可催化CRN7的K33泛素链修饰。修饰后的CRN7定位于高尔基体网反面,从而稳定F肌动蛋白,并促进post-Golgi转 运[63, 64]。在T细胞中也发现了K33的泛素链。这些K33泛素链是由泛素连接酶Cbl-b和Itch共同催化TCR-ζ的泛素化修饰形成的,可抑制TCR-ζ信号的磷酸化及与酪氨酸激酶Zap-70的联系,最终约束TCR信号[65]。
6.6 线性泛素链 泛素分子N端的甲硫氨酸残基 (M1) 也可与另一个泛素分子C末端的羧基共价结合,形成线性泛素链[31]。一个600 kDa的E3复合物LUBAC负责形成线性泛素链。该复合体包括泛素连接酶HOIL-1、HOIL-1相互作用蛋白HOIP和SHARPIN等三部分。由于HOIL-1含有与泛素相互作用的结构域UBL,而HOIP上有与蛋白酶体结合的结构域UBA,推测LUBAC具有将靶蛋白导向蛋白酶体降解的作用[66]。LUBAC功能多样,不仅参与炎症抑制和免疫应答调 节[67],还在炎症和固有免疫中参与NOD2信号传递[68]、NF-κB信号激活[69]、TNF介导的基因诱导[70]、负反馈调节RIG-1和TRIM25介导的1型干扰素感应[71]以及巨噬细胞Toll样受体2 (TLR2) 应答[72]等多种信号传递过程 。
现今研究最清楚的是其在TNF受体1 (TNFR1) 中的作用。当有TNF信号刺激时,TNF与TNFR1交联形成TNF-RSC,交联的TNFR1招募TNFR1相关的死亡结构域 (TRADD) 和受体相互作用蛋白PIP1、TNFR相关因子 (TRAF2) 被招募到TRADD,cIAP (细胞凋亡抑制蛋白) 1和2转移到TRAF2,LUBAC转移到Ciap (有E3连接酶活性),并形成泛素化。LUBAC随后以线性泛素链修饰RIP1和NF-κB必需的调节因子NEMO。cIAPs和LUBAC使用不同的泛素链赋予TNF-RSC不同的基因活化功能,使NF-κB激酶 (IKK) 和TAK/TAB (TNF-β活化激酶1和TAK1结合蛋白) 复合物在TNF-RSC中实现正确定位和差异调节[73]。
而最新的研究表明在骨髓衍生巨噬细胞中一种新的HOIL-1L作为NLRP3/ASC炎性小体活化的必需调节子。当NLRP3受到刺激时,NLRP3炎性小体适配器蛋白凋亡相关斑点样蛋白ASC发生线性泛素链修饰,从而在细胞质中由分散的状态转变为明显的区域分布。此外LUBAC能够活化caspase 1从而以一种独立于NF-κB的形式促进IL-1β的形成。这说明LUBAC也可以作为固有免疫的一种调节因子[74, 75]。
6.7 混合和分支泛素链 相对于同质泛素链,混合和分支泛素链的组装和拓扑学结构更加复杂,对其进行研究也更加困难。但已有的结果显示混合和分支泛素链同样具有重要的生物学功能。
目前对于混合和分支泛素链的组装机制研究比较清楚的是APC/C组装的分支泛素链。首先Ube2C以含K11、K48、K63的短泛素链修饰APC/C的底物,之后Ube2S添加大约6个K11连接的泛素分子到这些短泛素链上,从而在底物上形成分支泛素链。这些分支泛素链能快速地将底物从APC/C复合物中移出,导向蛋白酶体,发生降解。这一过程能介导包括Cdc20在内的许多APC/C底物的降解,从而促使纺锤体检验点的去组装,使细胞退出有丝分裂[76, 77]。另外,Kaposi’s肉瘤相关疱疹病毒K5基因产物也能以K11和K63连接的混合泛素链修饰组织相容性复合物MHCⅠ,从而促进MHCⅠ的内吞作用[78]。而K63和M1连接的混合泛素链能修饰白细胞介素1相关激酶IRAK1、类Toll受体1/2激动剂1/2、髓样分化初反应基因88 MyD88以及IRAK4等,从而活化IκB激酶 (IKK) 复合物[79]。
6.8 单泛素化修饰 单泛素化并不像通常的多聚泛素链一样将底物蛋白导向蛋白酶体进行降解,而是受体内化、膜泡分选[23]、转录激活、DNA修复[80]、Ras活化[81]和基因沉默的信号[82]。最先鉴定到的泛素化组蛋白H2A即是单泛素化蛋白,但这种修饰事件的功能至今未证明。在酵母中H2B是唯一已知的存在泛素化修饰的组蛋白。在组蛋白乙酰化或泛素化缺陷的突变中,一种SWI/SNF染色体重构因子定位到GAL1上游激活序列,但是在二者皆突变时不会出现。这说明H3的乙酰化与H2B的泛素化在转录激活中存在相同的功能[83]。另外H3 第4位赖氨酸的甲基化与染色体沉默相关。通过突变抑制Rad6介导的H2B的第123位赖氨酸的泛素化,可使Set1介导的组蛋白H3第4位的赖氨酸残基无法发生甲基化,从而造成染色体端粒沉默[84]。此外,H2B的C末端螺旋参与了核小体间的相互作用。因此H2B的C末端第123位赖氨酸残基的泛素化可能破坏这些核小体间的相互作用,改变局部和整体的染色体折叠形式,从而影响基因的表达[85]。
7 泛素突变对机体生存能力的影响 Finley通过同源重组的方式以特定标记基因HIS、TRP1、LEU分别替换UBI1、UBI2和UBI4等泛素编码基因,并突变UBI3基因中编码泛素的部分,形成酵母染色体上泛素基因完全缺失的菌株。在此基础上转化进一个携带CUP1启动子驱动的泛素基因的质粒,由此成功构建了与野生型酵母细胞泛素表达水平相当的酵母菌株。该工程菌株的构建大大简化了酵母细胞中泛素基因的进一步改造[14, 15, 86]。
研究发现K6、K11、K27、K29、K33等非常规赖氨酸残基连接的泛素链在体内均广泛存在,并且都可将底物蛋白质靶向蛋白酶体降 解[41]。但这些多样的连接形式又有不同的冗余功能,从而造成了泛素修饰影响的广泛性和功能多样性。突变研究表明K48R的突变无法维持酵母的生存,且只保留第48位赖氨酸也不足以维持酵母的生存。而保留K29、K33和K48三个赖氨酸的泛素突变体尽管可维持酵母的生长,但会导致严重的生长阻滞。各种泛素突变株在YPD培养基中的生长速率为:WT= R11=R11R63>R27>R11R27R63>R6R11R27R63,这表明突变对细胞增殖有一种累积的负效应[24]。
泛素分子内部含有由13个脂肪族氨基酸形成的疏水核心和两个极性氨基酸 (Thr7和Gln41) 组成的溶剂难接近区。为了检测这些内部的氨基酸如何影响蛋白功能,有研究者以丙氨酸扫描诱变策略测定丙氨酸替换突变对于酵母生长的影响。他们发现除I30A和L43A突变外的其余13种氨基酸突变对酵母生长没有毒性,而I30A突变会造成泛素化蛋白降解的缺陷,L43A突变会造成细胞中游离泛素链的缺失,二者都造成了蛋白水解作用的缺陷,从而导致细胞无法生长[87, 88]。
8 泛素-蛋白酶体系统失调和人类疾病 蛋白质泛素化修饰在细胞周期、细胞分裂、细胞分化、细胞发育、对细胞内外刺激的应答、神经元网络形态的形成、细胞表面受体调节、离子通道和分泌途径、DNA修复、转录调节和基因沉默、长时记忆、生物周期节律、免疫调节、炎症应答以及细胞器的形成等细胞生命活动的方方面面均起着重要的作用[35, 89]。这些过程的失调可引发如帕金森病、阿尔茨海默症、亨廷顿症、普里昂症以及肌萎缩性脊髓侧索硬化症等多种严重的人类疾病[90]。
Uba6 (E1)-Use1 (E2) 介导底物蛋白泛素化的级联反应,通过调节这些蛋白的特异性降解实现对鼠胚胎发育的有效调控。如在鼠胚胎发育时期缺失Uba6会造成海马和杏仁体神经元结构的改变,树突刺密度的减少以及多种行为的紊乱。在Uba6缺陷鼠中,虽然Ube3a的底物Arc的量减少了,但与树突刺功能相关的Ube3a和Shank3在杏仁体中的水平得到了提升。因此Uba6和Use1在鼠的胚胎纤维母细胞中促进Ube3a发生K48泛素链修饰,从而促进其转运到蛋白酶体降解。该现象在人细胞模型的研究也得到了证实,显示了这一分子对于胚胎发育的重要性[91]。
另外,去泛素化酶CYLD与家族性圆柱瘤密切相关。CYLD是一种肿瘤抑制因子,可对非K48泛素链发挥去泛素化作用。正常情况下,当受体活化时,会引发TRAF2、TRAF6和NEMO (IKKγ) 亚基发生K63泛素链修饰,从而活化IKK复合物,磷酸化IκB。随后IκB被K48泛素链修饰,导向蛋白酶体发生降解,实现NFκB信号的下调。突变的CYLD可引发NFκB信号的上调,使得机体耐受凋亡等刺激的能力增强,从而形成肿瘤[92]。
9 展望 近年来,泛素这一信号分子的重要性得到了越来越多的关注和证明。但随着研究的深入,更多的泛素及其修饰的生物学问题逐渐显现,有待多学科的科学家去探索。在泛素化蛋白研究的技术方面,高覆盖定量蛋白质组学技术的飞速发展为泛素-蛋白酶体系统的深入研究提供了契机。抗体、表位标签、串联亲和纯化、定点突变等策略使得富集低丰度的泛素化蛋白质底物成为可能。在数据分析方面,生物信息学人才的加盟为蛋白质泛素化的系统生物学研究提供了便利。他们系统收集、汇总、处理、存储和呈现了泛素/泛素化相关的信息和数据,成功构建了包括UbiProt (泛素化修饰底物蛋白数据库:http://ubiprot.org.ru/)、hUbiquitome (实验验证的人类泛素化相关蛋白质数据库:http://202.38.126.151/hmdd/hubi/)、E3Miner (使用文本挖掘方法建立的泛素化E3数据库:http://e3miner.biopathway.org/e3miner.html)、E3Net (升级的E3Miner:http://pnet.kaist.ac.kr/ e3net/)等系列数据库。这些数据库的构建不仅集中了特定的泛素相关信息,而且为构建细胞内泛素相关信号通路网络及泛素化与其他翻译后修饰的复杂关联奠定了基础,也为更好地预测泛素化位点和泛素化修饰的基序提供了线 索[93]。如何解读这些海量数据并且用生物学的方法验证这些数据隐含的生物学信息、如何获得各种不同连接形式的泛素链是一直以来困扰科学界的难题。最近Tatjana Schneider等利用密码子扩张和以点击化学为基础的聚合作用合成了人工泛素链,为我们分析复杂的泛素信号提供了契机[94]。未来人们将继续关注泛素系统的时空活性及其在多种生理过程中所扮演的重要角色,并就泛素链及泛素相关酶类的特异性进行探究。
此外,人们对以泛素-蛋白酶体系统关键蛋白作为药靶的可行性也给予了越来越多的关注。如何将基础研究所得结果快速转变为实际应用,以期找到治疗某些疾病的方法是我们需要面对的重大问题。未来人们希望鉴定所有的泛素修饰酶,揭示泛素化引导的分子信号及其生物学功能,建立泛素化与其他翻译后修饰的串联相互作用网络,从而发现与某些疾病相关的关键蛋白的泛素化靶点,开发单靶点或多靶点药物,为人类战胜现今无法治疗的疾病作出贡献[57]。
参考文献
| [1] | Schwartz DC, Hochstrasser M. A superfamily of protein tags: ubiquitin, SUMO and related modifiers. Trends Biochem Sci, 2003, 28(6): 321-328. |
| [2] | Wong BR, Parlati F, Qu KB, et al. Drug discovery in the ubiquitin regulatory pathway. Drug Discov Today, 2003, 8(16): 746-754. |
| [3] | Sun LJ, Chen ZJ. The novel functions of ubiquitination in signaling. Curr Opin Cell Biol, 2004, 16(2): 119-126. |
| [4] | Komander D, Rape M. The ubiquitin code. Annu Rev Biochem, 2012, 81: 203-229. |
| [5] | Kulathu Y, Komander D. Atypical ubiquitylation-the unexplored world of polyubiquitin beyond Lys48 and Lys63 linkages. Nat Rev Mol Cell Biol, 2012, 13(8): 508-523. |
| [6] | Goldstein G, Scheid M, Hammerling U, et al. Isolation of a polypeptide that has lymphocyte-differentiating properties and is probably represented universally in living cells. Proc Natl Acad Sci USA, 1975, 72(1): 11-15. |
| [7] | Goldknopf IL, Busch H. Isopeptide linkage between nonhistone and histone 2A polypeptides of chromosomal conjugate-protein A24. Proc Natl Acad Sci USA, 1977, 74(3): 864-868. |
| [8] | Hershko A, Ciechanover A, Rose IA. Resolution of the ATP-dependent proteolytic system from reticulocytes: a component that interacts with ATP. Proc Natl Acad Sci USA, 1979, 76(7): 3107-3110. |
| [9] | Hough R, Pratt G, Rechsteiner M. Purification of two high molecular weight proteases from rabbit reticulocyte lysate. J Biol Chem, 1987, 262(17): 8303-8313. |
| [10] | Finley D, Varshavsky A. The ubiquitin system: functions and mechanisms. Trends Biochem Sci, 1985, 10(9): 343-347. |
| [11] | Jentsch S. The ubiquitin-conjugation system. Annu Rev Genet, 1992, 26: 179-207. |
| [12] | Callis J, Carpenter T, Sun CW, et al. Structure and evolution of genes encoding polyubiquitin and ubiquitin-like proteins in Arabidopsis thaliana ecotype Columbia. Genetics, 1995, 139(2): 921-939. |
| [13] | Ozkaynak E, Finley D, Solomon MJ, et al. The yeast ubiquitin genes: a family of natural gene fusions. EMBO J, 1987, 6(5): 1429-1439. |
| [14] | Finley D, Özkaynak E, Varshavsky A. The yeast polyubiquitin gene is essential for resistance to high temperatures, starvation, and other stresses. Cell, 1987, 48(6): 1035-1046. |
| [15] | Finley D, Bartel B, Varshavsky A. The tails of ubiquitin precursors are ribosomal proteins whose fusion to ubiquitin facilitates ribosome biogenesis. Nature, 1989, 338(6214): 394-401. |
| [16] | Catic A, Ploegh HL. Ubiquitin-conserved protein or selfish gene? Trends Biochem Sci, 2005, 30(11): 600-604. |
| [17] | Shih SC, Sloper-Mould KE, Hicke L. Monoubiquitin carries a novel internalization signal that is appended to activated receptors. EMBO J, 2000, 19(2): 187-198. |
| [18] | Dikic I, Wakatsuki S, Walters KJ. Ubiquitin-binding domains—from structures to functions. Nat Rev Mol Cell Biol, 2009, 10(10): 659-671. |
| [19] | Hu M, Li PW, Li MY, et al. Crystal structure of a UBP-family deubiquitinating enzyme in isolation and in complex with ubiquitin aldehyde. Cell, 2002, 111(7): 1041-1054. |
| [20] | Rahighi S, Ikeda F, Kawasaki M, et al. Specific recognition of linear ubiquitin chains by NEMO is important for NF-kappaB activation. Cell, 2009, 136(6): 1098-1109. |
| [21] | Sloper-Mould KE, Jemc JC, Pickart CM, et al. Distinct functional surface regions on ubiquitin. J Biol Chem, 2001, 276(32): 30483-30489. |
| [22] | Jin LY, Williamson A, Banerjee S, et al. Mechanism of ubiquitin-chain formation by the human anaphase-promoting complex. Cell, 2008, 133(4): 653-665. |
| [23] | Galan JM, Haguenauer-Tsapis R. Ubiquitin lys63 is involved in ubiquitination of a yeast plasma membrane protein. EMBO J, 1997, 16(19): 5847-5854. |
| [24] | Xu P, Duong DM, Seyfried NT, et al. Quantitative proteomics reveals the function of unconventional ubiquitin chains in proteasomal degradation. Cell, 2009, 137(1): 133-145. |
| [25] | Cook WJ, Jeffrey LC, Kasperek E, et al. Structure of tetraubiquitin shows how multiubiquitin chains can be formed. J Mol Biol, 1994, 236(2): 601-609. |
| [26] | Satoh T, Sakata E, Yamamoto S, et al. Crystal structure of cyclic lys48-linked tetraubiquitin. Biochem Biophys Res Commun, 2010, 400(3): 329-333. |
| [27] | Varadan R, Walker O, Pickart C, et al. Structural properties of polyubiquitin chains in solution. J Mol Biol, 2002, 324(4): 637-647. |
| [28] | Ryabov Y, Fushman D. Interdomain mobility in di-ubiquitin revealed by NMR. Proteins, 2006, 63(4): 787-796. |
| [29] | Bremm A, Freund SMV, Komander D. Lys11-linked ubiquitin chains adopt compact conformations and are preferentially hydrolyzed by the deubiquitinase Cezanne. Nat Struct Mol Biol, 2010, 17(8): 939-947. |
| [30] | Matsumoto ML, Wickliffe KE, Dong KC, et al. K11-linked polyubiquitination in cell cycle control revealed by a K11 linkage-specific antibody. Mol Cell, 2010, 39(3): 477-484. |
| [31] | Komander D, Reyes-Turcu F, Licchesi JDF, et al. Molecular discrimination of structurally equivalent lys 63-linked and linear polyubiquitin chains. EMBO Rep, 2009, 10(5): 466-473. |
| [32] | Sims JJ, Cohen RE. Linkage-specific avidity defines the lysine 63-linked polyubiquitin-binding preference of rap80. Mol Cell, 2009, 33(6): 775-783. |
| [33] | Petroski MD, Deshaies RJ. Mechanism of lysine 48-linked ubiquitin-chain synthesis by the cullin-ring ubiquitin-ligase complex scf-cdc34. Cell, 2005, 123(6): 1107-1120. |
| [34] | Schaefer JB, Morgan DO. Protein-linked ubiquitin chain structure restricts activity of deubiquitinating enzymes. J Biol Chem, 2011, 286(52): 45186-45196. |
| [35] | Glickman MH, Ciechanover A. The ubiquitin-proteasome proteolytic pathway: destruction for the sake of construction. Physiol Rev, 2002, 82(2): 373-428. |
| [36] | Finley D, Sadis S, Monia BP, et al. Inhibition of proteolysis and cell cycle progression in a multiubiquitination-deficient yeast mutant. Mol Cell Biol, 1994, 14(8): 5501-5509. |
| [37] | Mallette FA, Richard S. K48-linked ubiquitination and protein degradation regulate 53BP1 recruitment at DNA damage sites. Cell Res, 2012, 22(8): 1221-1223. |
| [38] | Hofmann RM, Pickart CM. Noncanonical MMS2-encoded ubiquitin-conjugating enzyme functions in assembly of novel polyubiquitin chains for DNA repair. Cell, 1999, 96(5): 645-653. |
| [39] | Eddins MJ, Carlile CM, Gomez KM, et al. Mms2-Ubc13 covalently bound to ubiquitin reveals the structural basis of linkage-specific polyubiquitin chain formation. Nat Struct Mol Biol, 2006, 13(10): 915-920. |
| [40] | Cooper EM, Cutcliffe C, Kristiansen TZ, et al. K63-specific deubiquitination by two JAMM/MPN+ complexes: BRISC-associated Brcc36 and proteasomal Poh1. EMBO J, 2009, 28(6): 621-631. |
| [41] | Nathan JA, Kim HT, Ting L, et al. Why do cellular proteins linked to K63-polyubiquitin chains not associate with proteasomes? EMBO J, 2013, 32(4): 552-565. |
| [42] | Fisk HA, Yaffe MP. A role for ubiquitination in mitochondrial inheritance in saccharomyces cerevisiae. J Cell Biol, 1999, 145(6): 1199-1208. |
| [43] | Deng L, Wang C, Spencer E, et al. Activation of the IκB kinase complex by TRAF6 requires a dimeric ubiquitin-conjugating enzyme complex and a unique polyubiquitin chain. Cell, 2000, 103(2): 351-361. |
| [44] | Spence J, Gali RR, Dittmar G, et al. Cell cycle-regulated modification of the ribosome by a variant multiubiquitin chain. Cell, 2000, 102(1): 67-76. |
| [45] | Springael JY, Galan JM, Haguenauer-Tsapis R, et al. NH4+-induced down-regulation of the Saccharomyces cerevisiae Gap1p permease involves its ubiquitination with lysine-63-linked chains. J Cell Sci, 1999, 112(9): 1375-1383. |
| [46] | Lim KL, Chew KCM, Tan JMM, et al. Parkin mediates nonclassical, proteasomal-independent ubiquitination of synphilin-1: implications for lewy body formation. J Neurosci, 2005, 25(8): 2002-2009. |
| [47] | Saeki Y, Kudo T, Sone T, et al. Lysine 63‐linked polyubiquitin chain may serve as a targeting signal for the 26S proteasome. EMBO J, 2009, 28(4): 359-371. |
| [48] | Bhat KP, Yan S, Wang CE, et al. Differential ubiquitination and degradation of huntingtin fragments modulated by ubiquitin-protein ligase E3A. Proc Natl Acad Sci USA, 2014, 111(15): 5706-5711. |
| [49] | Chaney J, Das C. Understanding K11-polyubiquitin recognition at the 26S proteasome. FASEB J, 2013, 27: 785.5. |
| [50] | Zhang Z, Lü XD, Yin WC, et al. Ter94 ATPase complex targets k11-linked ubiquitinated ci to proteasomes for partial degradation. Dev Cell, 2013, 25(6): 636-644. |
| [51] | Rape M, Reddy SK, Kirschner MW. The processivity of multiubiquitination by the APC determines the order of substrate degradation. Cell, 2006, 124(1): 89-103. |
| [52] | Wu-Baer F, Lagrazon K, Yuan W, et al. The BRCA1/BARD1 heterodimer assembles polyubiquitin chains through an unconventional linkage involving lysine residue K6 of ubiquitin. J Biol Chem, 2003, 278(37): 34743-34746. |
| [53] | Srivastava D, Chakrabarti O. Mahogunin-mediated α-tubulin ubiquitination via noncanonical K6 linkage regulates microtubule stability and mitotic spindle orientation. Cell Death Dis, 2014, 5: e1064. |
| [54] | Durcan TM, Tang MY, Pérusse JR, et al. USP8 regulates mitophagy by removing K6-linked ubiquitin conjugates from parkin. EMBO J, 2014, 33(21): 2473-2491. |
| [55] | Scully R, Livingston DM. In search of the tumour-suppressor functions of BRCA1 and BRCA2. Nature, 2000, 408(6811): 429-432. |
| [56] | Welcsh PL, Owens KN, King MC. Insights into the functions of BRCA1 and BRCA2. Trends Genet, 2000, 16(2): 69-74. |
| [57] | Malynn BA, Ma A. Ubiquitin makes its mark on immune regulation. Immunity, 2010, 33(6): 843-852. |
| [58] | Arimoto KI, Funami K, Saeki Y, et al. Polyubiquitin conjugation to NEMO by triparite motif protein 23 (TRIM23) is critical in antiviral defense. Proc Natl Acad Sci USA, 2010, 107(36): 15856-15861. |
| [59] | Ikeda H, Kerppola TK. Lysosomal localization of ubiquitinated jun requires multiple determinants in a lysine-27-linked polyubiquitin conjugate. Mol Biol Cell, 2008, 19(11): 4588-4601. |
| [60] | Liu J, Han CF, Xie B, et al. Rhbdd3 controls autoimmunity by suppressing the production of IL-6 by dendritic cells via K27-linked ubiquitination of the regulator NEMO. Nat Immunol, 2014, 15(7): 612-622. |
| [61] | Chastagner P, Israël A, Brou C. Itch/AIP4 mediates deltex degradation through the formation of K29-linked polyubiquitin chains. EMBO Rep, 2006, 7(11): 1147-1153. |
| [62] | Pickart CM. Ubiquitin in chains. Trends Biochem Sci, 2000, 25(11): 544-548. |
| [63] | Yuan WC, Lee YR, Lin SY, et al. K33-linked polyubiquitination of coronin 7 by Cul3-KLHL20 ubiquitin E3 ligase regulates protein trafficking. Mol Cell, 2014, 54(4): 586-600. |
| [64] | Baumann K. Post-translational modifications: lys33-linked ubiquitin in post-golgi transport. Nat Rev Mol Cell Biol, 2014, 15(6): 365. |
| [65] | Huang HN, Jeon MS, Liao LJ, et al. K33-linked polyubiquitination of T cell receptor-zeta regulates proteolysis-independent T cell signaling. Immunity, 2010, 33(1): 60-70. |
| [66] | Kirisako T, Kamei K, Murata S, et al. A ubiquitin ligase complex assembles linear polyubiquitin chains. EMBO J, 2006, 25(20): 4877-4887. |
| [67] | Gerlach B, Cordier SM, Schmukle AC, et al. Linear ubiquitination prevents inflammation and regulates immune signalling. Nature, 2011, 471(7340): 591-596. |
| [68] | Damgaard RB, Nachbur U, Yabal M, et al. The ubiquitin ligase XIAP recruits LUBAC for NOD2 signaling in inflammation and innate immunity. Mol Cell, 2012, 46(6): 746-758. |
| [69] | Tokunaga F, Nakagawa T, Nakahara M, et al. SHARPIN is a component of the NF-κB-activating linear ubiquitin chain assembly complex. Nature, 2011, 471(7340): 633-636. |
| [70] | Tokunaga F, Sakata SI, Saeki Y, et al. Involvement of linear polyubiquitylation of NEMO in NF-κB activation. Nat Cell Biol, 2009, 11(2): 123-132. |
| [71] | Inn KS, Gack MU, Tokunaga F, et al. Linear ubiquitin assembly complex negatively regulates RIG-I- and TRIM25-mediated type I interferon induction. Mol Cell, 2011, 41(3): 354-365. |
| [72] | Zak DE, Schmitz F, Gold ES, et al. Systems analysis identifies an essential role for SHANK-associated RH domain-interacting protein (SHARPIN) in macrophage toll-like receptor 2 (TLR2) responses. Proc Natl Acad Sci USA, 2011, 108(28): 11536-11541. |
| [73] | Rieser E, Cordier SM, Walczak H. Linear ubiquitination: a newly discovered regulator of cell signalling. Trends Biochem Sci, 2013, 38(2): 94-102. |
| [74] | Rodgers MA, Bowman JW, Fujita H, et al. The linear ubiquitin assembly complex (LUBAC) is essential for NLRP3 inflammasome activation. J Exp Med, 2014, 211(7): 1333-1347. |
| [75] | Minton K. Inflammasomes: ubiquitin lines up for inflammasome activity. Nat Rev Immunol, 2014, 14(9): 580-581. |
| [76] | Meyer HJ, Rape M. Enhanced protein degradation by branched ubiquitin chains. Cell, 2014, 157(4): 910-921. |
| [77] | Meyer HJ, Rape M. Processive ubiquitin chain formation by the anaphase-promoting complex. Semin Cell Dev Biol, 2011, 22(6): 544-550. |
| [78] | Boname JM, Thomas M, Stagg HR, et al. Efficient internalization of MHC I requires lysine-11 and lysine-63 mixed linkage polyubiquitin chains. Traffic, 2010, 11(2): 210-220. |
| [79] | Emmerich CH, Ordureau A, Strickson S, et al. Activation of the canonical IKK complex by K63/M1-linked hybrid ubiquitin chains. Proc Natl Acad Sci USA, 2013, 110(38): 15247-15252. |
| [80] | Rajendra E, Oestergaard VH, Langevin F, et al. The Genetic and biochemical basis of FANCD2 monoubiquitination. Mol Cell, 2014, 54(5): 858-869. |
| [81] | Baker R, Lewis SM, Sasaki AT, et al. Site-specific monoubiquitination activates ras by impeding GTPase-activating protein function. Nat Struct Mol Biol, 2012, 20(1): 46-52. |
| [82] | Johnson ES. Ubiquitin branches out. Nat Cell Biol, 2002, 4(12): E295-E298. |
| [83] | Robzyk K, Recht J, Osley MA. Rad6-dependent ubiquitination of histone H2B in yeast. Science, 2000, 287(5452): 501-504. |
| [84] | Sun ZW, Allis CD. Ubiquitination of histone H2B regulates H3 methylation and gene silencing in yeast. Nature, 2002, 418(6893): 104-108. |
| [85] | White CL, Suto RK, Luger K. Structure of the yeast nucleosome core particle reveals fundamental changes in internucleosome interactions. EMBO J, 2001, 20(18): 5207-5218. |
| [86] | Ecker DJ, Khan MI, Marsh J, et al. Chemical synthesis and expression of a cassette adapted ubiquitin gene. J Biol Chem, 1987, 262(8): 3524-3527. |
| [87] | Lee SY, Pullen L, Virgil DJ, et al. Alanine scan of core positions in ubiquitin reveals links between dynamics, stability, and function. J Mol Biol, 2014, 426(7): 1377-1389. |
| [88] | Roscoe BP, Thayer KM, Zeldovich KB, et al. Analyses of the effects of all ubiquitin point mutants on yeast growth rate. J Mol Biol, 2013, 425(8): 1363-1377. |
| [89] | Ciechanover A. The ubiquitin-proteasome pathway: on protein death and cell life. EMBO J, 1998, 17(24): 7151-7160. |
| [90] | Ciechanover A, Brundin P. The ubiquitin proteasome system in neurodegenerative diseases: sometimes the chicken, sometimes the egg. Neuron, 2003, 40(2): 427-446. |
| [91] | Lee Peter CW, Dodart JC, Aron L, et al. Altered Social behavior and neuronal development in mice lacking the Uba6-Use1ubiquitin transfer system. Mol Cell, 2013, 50(2): 172-184. |
| [92] | Singhal S, Taylor MC, Baker RT. Deubiquitylating enzymes and disease. BMC Biochem, 2008, 9(Suppl 1): S3. |
| [93] | Lu L, Li D, He FC. Bioinformatics advances in protein ubiquitination. Hereditas, 2013, 35(1): 17-26 (in Chinese). 卢亮, 李栋, 贺福初. 蛋白质泛素化修饰的生物信息学研究进展. 遗传, 2013, 35(1): 17-26. |
| [94] | Schneider T, Schneider D, Rosner D, et al. Dissecting ubiquitin signaling with linkage-defined and protease resistant ubiquitin chains. Angew Chem Int Ed Engl, 2014: 53(47): 12925-12929. |
