
 , 张延平2
, 张延平2 

1. 江南大学药学院,江苏 无锡 214122
2. 中国科学院微生物研究所,北京 100101
3. 中国科技大学生命科学院,安徽 合肥 230022
4. 中国科学院天津工业生物技术研究所,天津 300308
摘要:1,2,4-丁三醇 (BT) 是一种在工业中有多种用途的重要的非天然化合物。文中通过将外源基因xdh和mdlC导入大肠杆菌BW25113表达,并敲除了xylA、xylB、yagE、yjhH、yiaE和ycdW等木糖和中间产物代谢旁路基因,构建了能够将D-木糖转化为BT的重组菌株。为优化BT合成途径,针对BT合成途径中的限速步骤——3-脱氧-D-甘油-戊酮糖酸的脱羧反应,进行了新酶的筛选和评价,获得了可显著提高反应效率的新的 2-酮酸脱羧酶——KivD,并构建了表达该酶的重组菌株BW-025。在此基础上,通过初步条件优化,将BT产量提高至2.38 g/L;进一步调节途径中各个酶的表达量,探究了它们对BW-025合成BT的影响,最终获得了BT产量较BW-025提高了48.62%的重组菌株BW-074。
关键词: D-木糖1,2,4-丁三醇大肠杆菌2-酮酸脱羧酶途径优化
Optimization of 1,2,4-butanetriol synthetic pathway in Escherichia coli
Lei Sun1, Fan Yang2, 3, Taicheng Zhu2, Xinghua Li2, Hongbing Sun4, Yin Li2, Zhenghong Xu1

 , Yanping Zhang2
, Yanping Zhang2

1. Laboratory of Pharmaceutical Engineering, School of Pharmaceutical Science, Jiangnan University, Wuxi 214122, Jiangsu, China
2. CAS Key Laboratory of Microbial Physiological and Metabolic Engineering, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
3. School of Life Sciences, University of Science and Technology of China, Hefei 230022, Anhui, China
4. National Engineering Laboratory for Industrial Enzymes, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308,China
Received: March 25, 2015; Accepted: May 4, 2015
Supported by: The Knowledge Innovation Project of the Chinese Academy of Sciences (No. KSCX2-EW-G-5), Beijing MunicipalNatural Science Foundation (No. 31170039).
Corresponding authors: Zhenghong Xu. Tel/Fax: +86-510-85918206; E-mail: zhenghxu@jiangnan.edu.cnYanping Zhang. Tel/Fax: +86-10-64807351; E-mail: zhangyp@im.ac.cn
Abstract: 1,2,4-Butanetriol (BT) is an important non-natural chemical with a variety of industrial applications. A recombinant Escherichia coli biosynthesizing BT from D-xylose was constructed by heterologously expressing xdh and mdlC, and knocking out competing pathway genes including xylA, xylB, yjhE, yagH and ycdW. To optimize BT synthesis pathway, the third catalytic step that catalyzes the decarboxylation reaction of 3-deoxy-D-glycero-pentulosonic acid was identified as a potential bottleneck. Consequently, 2-keto acid decarboxylases from three different microorganisms were screened, and the kivD gene from Lactococcus lactis was found to increase BT titer by 191%. The improved strain BW-025 reached a final BT titer of 2.38 g/L under optimized transformation conditions. Attempts on synthetic pathway optimization were also made by fine-tuning the expression levels of each enzyme involved in the whole pathway based on BW-025. As a result, an xdh overexpressed recombinant strain, BW-074 was finally generated, with 48.62% higher BT production than that of BW-025.
Keywords: D-xylose1,2,4-butanetriolEscherichia coli2-keto acid decarboxylasepathway optimization
1,2,4-丁三醇 (1,2,4-butanetriol,简称BT) 是一种重要的非天然多元醇,在军工、医药等领域具有广泛的应用价值。BT最有潜力的应用是作为1,2,4-丁三醇三硝酸酯 (BTTN) 的合成前体,后者可代替硝化甘油作为推进剂和含能增塑剂,并具有更优的理化性质[1]。医药方面,BT可被用作阳离子脂质体和多种药物的合成前体[2, 3, 4]。此外,BT还可用于合成聚氨酯泡沫、卷烟添加剂和彩色显影剂等[5, 6, 7]。
目前BT的商业生产多采用NaBH4催化的苹果酸二酯还原或Ru-C催化的苹果酸还原等化学合成法[8, 9, 10]。这些合成工艺都存在反应条件苛刻、环境污染严重、生产危险性大、副产物多等弊端。生物合成法具有原料成本低廉、反应条件温和、环境友好、安全高效等优点。2003年,Niu等首先提出了BT的生物合成法[10]。该方法以D-木糖或L-阿拉伯糖为底物,经过四步酶催化 (图1) 实现。整个工艺使用了莓实假单胞菌Pseudomonas fragi和大肠杆菌Escherichia coli两种微生物作为催化剂。之后,研究者将BT转化途径的四步酶都在E. coli中进行表达,用E. coli即可完成整个BT的生物转化,大大简化了转化工艺[11]。但从目前已有报道来看,用重组E. coli生产BT的催化效率较低,导致BT产量无法满足工业生产的需求[3, 12, 13]。如果要进一步提高BT的转化效率,首先必须对BT合成途径进行系统地优化,找出催化限速步骤,精确调节催化各个步骤酶的表达量,以实现最大的转化通量。
本文在构建BT合成工程菌的基础上,针对关键限速步骤进行了新酶的筛选,并分别调节了途径中各个酶的表达量,显著提高了重组菌株利用木糖合成BT的效率。
1 . 材料与方法1.1 菌株及质粒 实验所用菌株及质粒见表1。
1.2 酶与试剂 PrimeSTAR® HS DNA聚合酶和T4 DNA连接酶为TaKaRa (大连) 公司产品。限制性内切酶购自Fermentas公司。2×Taq PCR StarMix with Loading Dye购自北京康润诚业生物科技有限公司。各试剂盒购自Omega Bio-tek公司。1,2,4-丁三醇标准品购自Sigma公司。酵母提取物、胰蛋白胨为Oxoid公司产品。其余试剂如D-木糖、异丙基硫代半乳糖苷 (IPTG)、硫酸卡那霉素 (Kanamycin)、盐酸四环素 (Tetracycline hydrochloride)、NaCl和CaCO3等均为国产。引物由英潍捷基 (上海) 公司合成。
表1 本文所用的菌株及质粒Table 1 Plasmids and strains used in this work
| Plasmids or strains | Main characteristics | Reference or source |
| Plasmids | ||
| pET30a | Skeleton plasmid for cloning, Kanr | Lab collection |
| pACYC184 | Skeleton plasmid for cloning, Cmr, Tcr | Lab collection |
| pKmTsSacB | Skeleton plasmid for cloning, Kanr | Lab collection |
| pTAK117 | Used for cloning T1T2 terminator | Lab collection |
| pET30a-xdh-T1T2-adhP-miniPtac | pET30a based vector, containing xdh-T1T2-adhP-miniPtac cassette, Kanr | This study |
| pET30a-xdh-T1T2-adhP-Ptac | pET30a based vector, containing xdh-T1T2-adhP-Ptac cassette, Kanr | This study |
| pKmTsSacB-xdh-adhP | pKmTsSacB based vector, containing xylA up-xdh-T1T2-adhP-miniPtac-xylB down cassette, Kanr | This study |
| pET30a-miniPtac-mdlC | pET30a based vector with miniPtac, carrying one copy of codon optimized mdlC gene, Kanr | This study |
| pET30a-miniPtac-kivD | pET30a based vector with miniPtac, carrying one copy of codon optimized kivD gene, Kanr | This study |
| pET30a-miniPtac-aepY | pET30a based vector with miniPtac, carrying one copy of codon optimized aepY gene, Kanr | This study |
| pET30a-miniPtac-ipdC | pET30a based vector with miniPtac, carrying one copy of codon optimized ipdC gene, Kanr | This study |
| pET30a-Ptac-kivD | pET30a based vector with Ptac, carrying one copy of codon optimized kivD gene, Kanr | This study |
| pET30a-miniPtac-2kivD | pET30a based vector with miniPtac, carrying two copies of codon optimized kivD gene, Kanr | This study |
| pACYC184-Ptac-xdh-T1T2 | pACYC184 based vector, containing Ptac-xdh-T1T2 cassette, Cmr, Tcr | This study |
| pACYC184-miniPtac-xdh-T1T2 | pACYC184 based vector, containing miniPtac-xdh-T1T2 cassette, Cmr, Tcr | This study |
| pACYC184-Ptac-adhP-T1T2 | pACYC184 based vector, containing Ptac-adhP-T1T2 cassette, Cmr, Tcr | This study |
| pACYC184-miniPtac-adhP-T1T2 | pACYC184 based vector, containing miniPtac-adhP-T1T2 cassette, Cmr, Tcr | This study |
| pACYC184-Ptac-yjhG | pACYC184 based vector with Ptac, carrying one copy of codon optimized yjhG gene, Cmr, Tcr | This study |
| pACYC184-miniPtac-yjhG | pACYC184 based vector with miniPtac, carrying one copy of codon optimized yjhG gene, Cmr, Tcr | This study |
| Strains | ||
| BW25113 | Wild type, used as the host strain | Lab stock |
| DH5α | Commercial transformation host for cloning | TaKaRa |
| BW-004 | BW25113/△yjhH::FRT & △yagE::FRT & △ycdW::FRT & △yiaE::FRT | This study |
| BW-010 | BW-004/△xylAB::xdh-T1T2-adhP-miniPtac | This study |
| BW-011 | BW-010/pET30a-miniPtac-mdlC | This study |
| BW-025 | BW-010/pET30a-miniPtac-kivD | This study |
| BW-055 | BW-010/pET30a-Ptac-kivD | This study |
| BW-056 | BW-010/pET30a-Ptac-kivD & pACYC184-miniPtac-yjhG | This study |
| BW-058 | BW-010/pET30a-Ptac-kivD & pACYC184-Ptac-yjhG | This study |
| BW-059 | BW-010/pET30a-miniPtac-2kivD | This study |
| BW-063 | BW-010/pET30a-miniPtac-aepY | This study |
| BW-064 | BW-010/pET30a-miniPtac-ipdC | This study |
| BW-066 | BW-010/pET30a-Ptac-kivD & pACYC184-miniPtac-adhP-T1T2 | This study |
| BW-067 | BW-010/pET30a-Ptac-kivD & pACYC184-Ptac-adhP-T1T2 | This study |
| BW-073 | BW-010/pET30a-miniPtac-kivD & pACYC184-miniPtac-xdh-T1T2 | This study |
| BW-074 | BW-010/pET30a-miniPtac-kivD & pACYC184-Ptac-xdh-T1T2 | This study |
表选项
1.3 遗传操作 DNA的克隆、酶切、连接、感受态细胞制备和转化、质粒提取等基本基因操作技术参照《分子克隆实验指南》[14]及试剂盒供应商提供的操作手册进行。文中所用引物见表2。
表2 本文所用的引物Table 2 Primers used in this work
| Primers | Sequences (5′-3′)/restriction sitesa | Plasmid constructed or function | |
| T1T2-F-BamHⅠ | TTAggatccAAAAGGCCATCCGTCAGGATG | pET30a-xdh-T1T2-adhP- miniPtac & pET30a-xdh-T1 T2-adhP-Ptac | |
| T1T2-R-NotⅠ | ATAgcggccgcTTGGCTGTTTTGGCGGATG | ||
| adhP-F-NotⅠ | GGCGCGGCCGCTTAGTGACGGAAATCAATCACCATG | ||
| adhP-R-miniPtac-XhoⅠ | GGCCTCGAGTTGACAATTAATCATCGGCTCGTATAATGTGTGGTCACACAGGAGATATCATATGAAGGCTGCAGTTGTTACGAAG | ||
| adhP-R-Ptac-XhoⅠ | GGCCTCGAGTTGACAATTAATCATCGGCTCGTATAATGTGTGGAATTGTGAGCGGATAACAATTTCACACAGGAGATATCATATGAAGGCTGCAGTTGTTACGAAG | ||
| xylA up-F-SalⅠ | ATCCAGCTGATTTTGTTATTATTGGCGGTTAGCG | pKmTsSacB-xylAup-xdh- T1T2-adhP-miniPtac-xylB down | |
| xylA up-R | GAGACGGGTAGATAGCAGAAGACATATTGAACTCCATAATCAGGTAATGCC | ||
| xylB down-F | CGAGCCGATGATTAATTGTCAACAGCTCACGTTATCCCCTGC | ||
| xylB down-R-XhoⅠ | TACGAGCTCACGTGAGTTATTCGCTGTATTCTTCC | ||
| Expression cassette 1-F | GGCATTACCTGATTATGGAGTTCAATatgTCTTCTGCTATCTACCCGTCTC | ||
| Expression cassette 1-R | GGTCAGGCAGGGGATAACGTGAGCTGTTGACAATTAATCATCGGCTCG | ||
| xdh-F | CAGGAGATATCATatgTCTTCTGCTATCTACCCGTCTC | pACYC184-Ptac-xdh-T1T2 & pACYC184-miniPtac-xdh- T1T2 | |
| xdh-R-AhdⅠ | TTAGACTCTCAGTCTTGGCTGTTTTGGCGGATG | ||
| Ptac-F-AvaⅠ | GACctcgggccatcatcatcatcattcttctgg | ||
| miniPtac-R-xdh | GATAGCAGAAGACATATGATATCTCCTGTGTGACCACAC | ||
| Ptac-R-xdh | GATAGCAGAAGACATATGATATCTCCTGTGTGAAATTGTTATCC | ||
| 续表2 | |||
| yjhG-F | CACACAGGAGATATCATATGTCTGTTCGCAATATTTTTGCTGAC | pACYC184-miniPtac-yjhG & pACYC184-Ptac-yjhG | |
| yjhG-R-DrdⅠ | CCCGACCCGGTCGTCTCAGTTTTTATTCATAAAATCGCGC | ||
| Ptac-F-AhdⅠ | GGCgactgagagtcccatcatcatcatcattcttctgg | ||
| miniPtac-R-yjhG | CGAACAGACATATGATATCTCCTGTGTGACCACAC | ||
| Ptac-R-yjhG | CGAACAGACATATGATATCTCCTGTGTGAAATTGTTATCC | ||
| kivD-F | GGATAACAATTTCACACAGGAGATATCATATGTACACCGTTGG | pET30a-Ptac-kivD | |
| kivD-R-NcoⅠ | CCATGGTTAAGATTTGTTCTGTTCAGCGAAC | ||
| Ptac-F-SgrAⅠ | TATcgccggtgCCATCATCATCATCATTCTTCTGG | ||
| Ptac-R-kivD | GTGAGCGGATAACAATTTCACACAGGAGATATC | ||
| KivD-F-NcoⅠ | ATTCCATGGAGGAGATATCATATGTACACCGTTGG | pET30a-miniPtac-2kivD | |
| KivD-R-BamHⅠ | TAAGGATCCTTAAGATTTGTTCTGTTCAGCGAAC | ||
| Ptac-adhP-AvaⅠ | ATACTCGGGGAAGCAGCTCCAGCCTACACA | pACYC184-miniPtac-adhP- T1T2 & pACYC184-Ptac- adhP-T1T2 | |
| adhP-R-AhdⅠ | GATGACTCTCAGTCTACCGCCTTTGAGTGAGCTGATAC |
表选项
1.3.1 重组质粒的构建 基因xdh由Genewiz公司进行密码子优化并合成,然后被插入质粒pET30a的NdeⅠ和Bgl Ⅱ位点之间,从而获得pET30a-xdh。基因adhP克隆自E. coli BW25113基因组,引物为adhP-F-NotⅠ和adhP-R-miniPtac-XhoⅠ。通过PCR 将引物中的组成型启动子序列miniPtac (或诱导型启动子序列Ptac) 与adhP序列融合。T1T2终止子扩增自pTAK117载体,引物为T1T2-F-BamHⅠ和T1T2-R-NotⅠ。将两段基因片段分别插入pET30a-xdh,获得了重组质粒pET30a-xdh-T1T2-adhP-miniPtac。以该质粒为模板扩增出xdh-T1T2-adhP-miniPtac片段 (引物为expression cassette 1-F和expression cassette 1-R),并通过PCR与xylA的上游区域和xylB的下游区域融合,获得xylA up-xdh-T1T2-adhP- miniPtac-xylB down片段,然后将其插入载体pKmTsSacB的XhoⅠ和SalⅠ位点,构建了pKmTsSacB-xdh-adhP。
重组质粒pET30a-miniPtac-mdlC的基因合成、测序验证和质粒的亚克隆构建均委托Genewiz公司完成。其中miniPtac为启动子,并加入AAGGAG的RBS序列,mdlC为根据大肠杆菌密码子的偏好性,在不改变MdlC氨基酸序列的前提下,对MdlC的编码序列进行密码子优化得到的核苷酸序列,将质粒pET30a的SgrAⅠ和NcoⅠ的之间的序列替换为商业合成的miniPtac-mdlC的SgrAⅠ和NcoⅠ的位点之间的序列,保持pET30a的其他序列不变,将序列正确的重组质粒命名为pET30a-miniPtac-mdlC。pET30a-miniPtac-kivD、pET30a-miniPtac-aepY和pET30a-miniPtac-ipdC等带有2-酮酸脱羧酶的编码基因的重组质粒构建方法相同。pET30a-miniPtac-2kivD是通过将另一拷贝的kivD基因插入pET30a-miniPtac-kivD而获得的。
载体pACYC184用于基因xdh、yjhG和adhP的表达。将扩增自pET30a-xdh-T1T2-adhP- miniPtac的xdh-T1T2片段与miniPtac或Ptac融合后插入pACYC184,便分别获得了重组质粒pACYC184-miniPtac-xdh-T1T2和pACYC184- Ptac-xdh-T1T2。pACYC184-miniPtac-adhP-T1T2和pACYC184-Ptac-adhP-T1T2的构建方法相同。将扩增自E. coli BW25113基因组的yjhG基因与miniPtac或Ptac融合后插入pACYC184的AhdⅠ和DrdⅠ位点,则获得了重组质粒pACYC184- miniPtac-yjhG和pACYC184-Ptac-yjhG。
1.3.2 基因敲除及验证 采用λ Red法[15, 16, 17]敲除了已知的导致D-木糖转用的4种旁路基因yagE、yjhH、yiaE和ycdW,获得菌株BW-004。通过两轮重组的方法将基因xdh和adhP一起整合至宿主细胞的基因组中,置换木糖异构酶和木酮糖激酶的编码基因xylA和xylB,以构建重组菌株BW-010。具体过程为:第一轮,通过将重组质粒pKmTsSacB-xdh-adhP转入BW-004,并在卡那平板上筛选出整合至xylAB位点的单交换型;在第二轮中,在含蔗糖的琼脂平板上筛选出双交换的阳性转化株。最后通过菌落PCR进行相应的验证。
1.3.3 重组菌株的构建 将构建好的重组质粒以电击转化方法导入底盘细胞BW-010,在质粒相应的抗性平板上进行阳性菌株的筛选,并通过菌落PCR加以验证。
1.4 培养基和转化条件 种子培养基为液体LB培养基 (含10 g/L胰蛋白胨、5 g/L酵母提取物和10 g/L NaCl),培养条件为37 ℃、200 r/min。取1 mL过夜活化的种子培养物至100 mL LB液体培养基 (装于500 mL三角摇瓶),37 ℃、200 r/min条件下预培养10 h,带有诱导型表达基因 (含Ptac启动子) 的菌株还需指数生长中期 (OD600=0.4) 加入200 mmol/L IPTG以诱导蛋白质表达。
之后对种子液OD600进行测定和计算,取一定量的菌液离心后再以20 mL补加了20 g/L D-木糖作为唯一碳源的M9培养基[13] (含17.08 g/L Na2HPO4·12H2O、3.0 g/L KH2PO4、0.5 g/L NaCl、1.0 g/L NH4Cl、1 mmol/L MgSO4·7H2O、0.1 mmol/L CaCl2、10 mmol/L NaHCO3和1 μL/mL微量金属离子溶液) 重悬,然后在30 ℃/33.5 ℃/37 ℃、 200 r/min条件下转化72 h。微量金属离子溶液包含:3.7 g/L (NH4)6 (Mo7O24)·4H2O、2.9 g/L ZnSO4·7H2O、15.8 g/L MnCl2·4H2O、24.7 g/L H3BO3和2.5 g/L CuSO4·5H2O。另外可向培养基中加入100 mmol/L MOPS和2.5 g/L CaCO3来控制pH的下降。每种设置3个平行。
在上述培养和转化过程中,视菌株所带质粒情况添加相应的抗生素。卡那霉素和盐酸四环素的工作浓度分别为50 μg/mL和10 μg/mL。
1.5 胞外代谢物的检测 胞外底物、产物和副产物如木糖、木糖酸、乙酸、乳酸、BT等物质的浓度测定采用高效液相色谱法。取1 mL菌液室温下10 000×g离心 2 min,取上清用孔径为0.22 μm的无菌滤膜过滤,采用高效液相色谱法检测,具体设备与条件为:柱型Bio-Rad Aminex® HPX-87H Ion Exclusion Column (300 mm × 7.8 mm),流动相为5 mmol/L H2SO4,流速0.5 mL/min,柱温15 ℃,进样量10 μL,检测器为Agilent Technologies 1260 RID (Refractive index detector)。
通过检测标准品的相应数值计算样品中各物质的含量。
2 . 结果与分析2.1 大肠杆菌中丁三醇合成途径的构建 在构建BT的合成途径之前,我们首先对宿主细胞E. coli BW25113进行了必要的代谢工程改造。如图1所示,通过λ Red法敲除了已知D-木糖竞争旁路基因yagE、yjhH、yiaE和ycdW,获得菌株BW-004。在此基础上,将新月柄杆菌 Caulobacter crescentus的D-木糖脱氢酶的编码基因xdh和大肠杆菌E. coli中的一个醇脱氢酶的编码基因adhP一起整合至宿主细胞的基因组中,置换木糖异构酶和木酮糖激酶的编码基因xylA和xylB,成功构建了重组菌株BW-010。在本研究中,BW-010被用作构建利用D-木糖合成BT的工程菌株的底盘细胞。
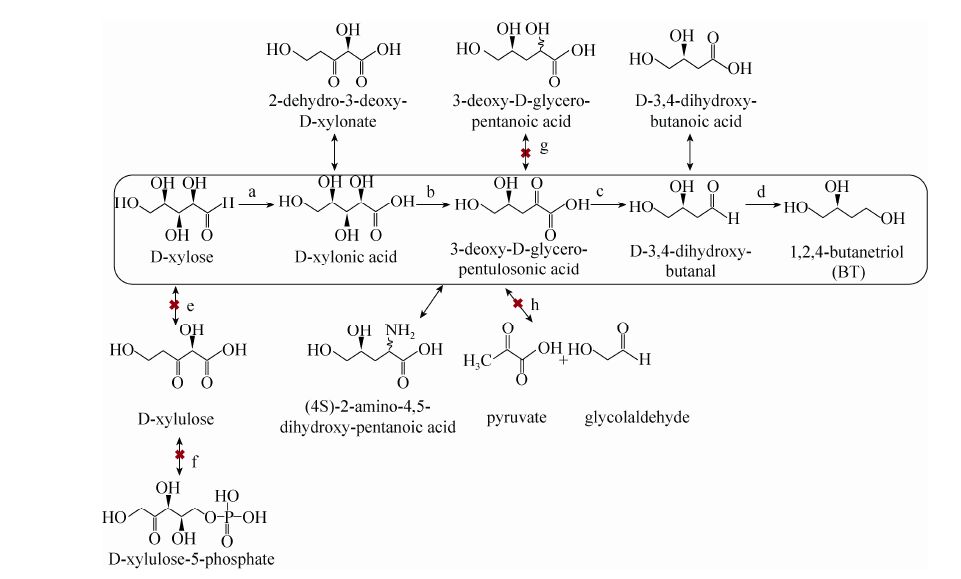 |
| 图1 E. coli重组细胞中D-木糖合成1,2,4-丁三醇的代谢途径 Fig.1 The BT synthetic pathway from D-xylose in the recombinant E. coli. The BT synthetic pathway (in the box) and by-passes (outside the box). Enzymes (coding genes,source) involved: a: D-xylose dehydrogenase (xdh,C. crescentus), b: D-xylonic acid dehydratase (yjhG and yagF,E. coli), c: benzoylformate decarboxylase (mdlC,P. putida), d: alcohol dehydrogenase (adhP,E. coli), e: xylose isomerase (xylA,E. coli), f: xylulokinase (xylB,E. coli), g: 2-Keto acid dehydrogenase (yiaE and ycdW,E. coli), h: 2-Keto-3-deoxy-d-xylonate aldolase (yagE and yjhH,E. coli). The × denotes gene disruption. |
| 图选项 |
之后将BT合成途径中的另一个异源基因,来自恶臭假单胞菌Pseudomonas putida的苯甲酰甲酸脱羧酶编码基因mdlC,与组成型启动子miniPtac融合,并插入多拷贝质粒pET30a。通过电击转化将构建的质粒pET30a-miniPtac-mdlC导入BW-010,成功得到了BT合成途径组装完全的E. coli重组菌株BW-011。该菌株中,BT合成途径第1步和第3步的催化蛋白Xdh和MdlC为异源蛋白,前者的基因被插入到E. coli染色体上,后者通过表达载体引入细胞;催化第2步和第4步的蛋白YjhG和AdhP为E. coli内源蛋白,前者利用E. coli本身携带的基因进行表达,后者的基因被合成后插入到染色体上增加一个拷贝。该菌株在20 g/L木糖中转化 72 h,可成功地检测到BT的积累,产量为0.3 g/L (图2)。
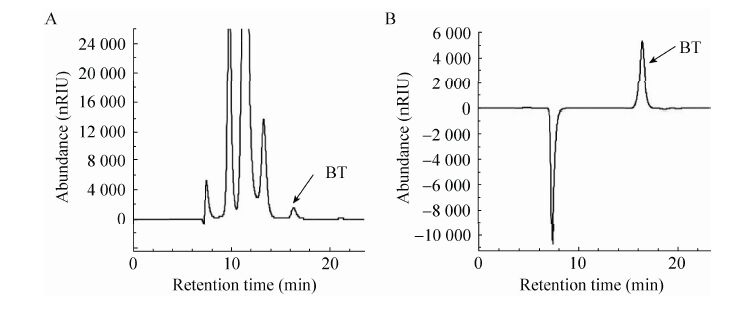 |
| 图2 高效液相检测色谱图 Fig.2 The HPLC analysis of BT conversion broth. (A) Supernatant from BW-011 broth. (B) Pure BT sample. |
| 图选项 |
2.2 2-酮酸脱羧酶的筛选 为了优化BT的合成途径,需要首先识别出整个途径的限速步骤。由于自然界中尚未发现天然催化3-脱氧-D-甘油-戊酮糖酸脱羧反应的酶,只能选择其他的2-酮酸脱羧酶替代,因此第3步催化反应很可能是整个反应的限速步骤。在上述构建BT途径中,我们参考之前的报 道[10, 11],选择了来自恶臭假单胞菌P. putida的苯甲酰甲酸脱羧酶。为进一步优化途径,我们通过在KEGG等数据库中检索类似的基团反应,以期筛选出比MdlC更有效催化3-脱氧-D-甘油-戊酮糖酸脱羧反应的2-酮酸脱羧酶。
基于“倾向于选择能催化与BT合成途径中第3步反应相似基团反应,但与MdlC的亲缘关系较远的2-酮酸脱羧酶”的思路,我们选择了3种可能可用的2-酮酸脱羧酶 (表3),其中IpdC和KivD同属于吲哚丙酮酸脱羧酶,AepY为磷酸烯醇丙酮酸脱羧酶。对上述3种2-酮酸脱羧酶进行克隆,以miniPtac为启动子,连接到pET30a载体中。将构建成功的重组表达载体pET30a-miniPtac-kivD、pET30a-miniPtac-aepY和pET30a-miniPtac-ipdC分别转化BW-010感受态细胞,获得重组菌株BW-025、BW-063和BW-064。
表3 筛选获得的2-酮酸脱羧酶 Table 3 2-Keto acid decarboxylase screened in this work
| 2-Keto acid decarboxylase (Enzyme, coding gene) | Source |
| α-Ketoisovalerate decarboxylase (KivD, kivD) | Lactococcus lactis |
| Phosphonopyruvate decarboxylase (AepY, aepY) | Bacteroides fragilis |
| Indole-3-pyruvate decarboxylase (IpdC, ipdC) | Enterobacter cloacae |
表选项
将重组菌株BW-025、BW-063、BW-064与BW-011一起进行摇瓶转化实验,比较其BT的合成能力 (图3)。与BW-011相比,BW-025的1,2,4-丁三醇产量提高了191%,产量最高,BW-064提高了6.2%,而BW-063几乎不产1,2,4-丁三醇,表明L. lactis的α-酮异戊酸脱羧酶 (KivD) 比MdlC对3-脱氧-D-甘油-戊酮糖酸的脱羧反应有更高的催化效率,能够提高整条途径的通量,从而提高重组菌株的1,2,4-丁三醇产量;E. cloacae的吲哚丙酮酸脱羧酶 (IpdC) 催化3-脱氧-D-甘油-戊酮糖酸脱羧反应的能力与MdlC相近;而B. fragilis的磷酸丙酮酸脱羧酶 (AepY) 不能催化该反应。
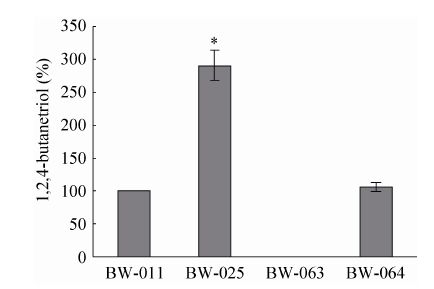 |
| 图3 表达不同2-酮酸脱羧酶的重组菌株的1,2,4-丁三醇产量 Fig.3 Effects of different 2-keto acid decarboxylases on BT production. |
| 图选项 |
2.3 1,2,4-丁三醇转化条件的初步优化 主要考察了转化温度、初始细胞密度和控制转化pH对BW-025重组菌株合成BT的影响。如图4所示,BW-025在33.5 ℃下72 h合成的BT产量最高,分别比30 ℃和37 ℃高出209.64%和16.57%。BT产量随着初始菌量的增加而增加,在OD600为30时达到最大,初始OD600提升到40时,BT产量反而有所下降。整个转化过程中,由于木糖酸和其他酸性中间产物的积
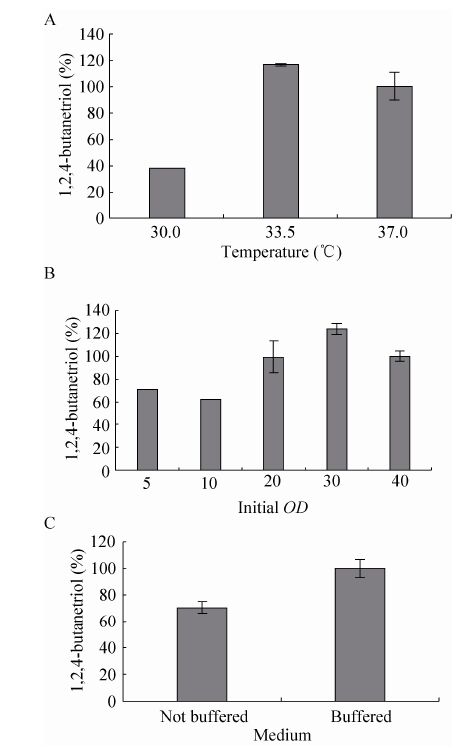 |
| 图4 不同转化条件对BW-025合成1,2,4-丁三醇的影响 Fig.4 Comparison of BT production with different conversion conditions. BT production of BW-025 with different temperatures(A),initial cell densities (OD600) (B) and pH control (C) were evaluated. |
| 图选项 |
累,pH值不断下降 (结果未显示)。通过向培养基中添加100 mmol/L MOPS与0.05 g轻质CaCO3,可有效减缓转化体系pH值的下降。BW-025的BT产量也在72 h内提高了41.57%。综合以上各最优条件,BW-025在72 h最高可产BT 2.38 g/L。
2.4 丁三醇合成途径中各步酶表达量的调节优化 在筛选到KivD后,菌株的BT产量得到显著提高,证明第3步酶确实是整个BT合成的限速步骤。为了考察继续提高KivD的表达量是否进一步增强1,2,4-丁三醇的合成,我们首先考察了提高KivD表达量对BT合成的影响。分别通过更换强启动子和增加拷贝数的方法,将重组质粒pET30a-Ptac-kivD和pET30a-miniPtac- 2kivD分别转化至BW-010中,构建了重组菌株BW-055和BW-059。两者与BW-025的摇瓶发酵72 h后的1,2,4-丁三醇产量如图5所示,与BW-025相比,BW-055的丁三醇产量降低了32.1%,BW-059降低了6.9%。可推测KivD不是现有1,2,4-丁三醇合成途径的限速酶。
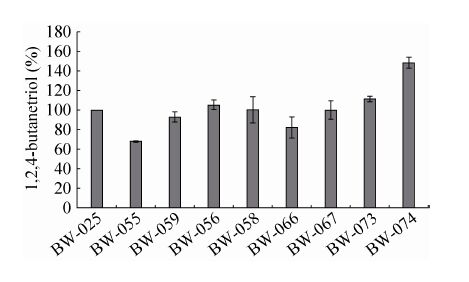 |
| 图5 在BW-025基础上分别过表达编码途径中四种酶的基因对1,2,4-丁三醇产量的影响 Fig.5 Effects of expression levels of kivD,yihG,adhP and xdh on BT production of BW-025,respectively. |
| 图选项 |
为了确定其他催化步骤是否限制了BT的合成,将重组质粒pACYC184-miniPtac-yjhG和pACYC184-Ptac-yjhG分别与pET30a-miniPtac- kivD共同转入BW-010感受态细胞,得到了催化第二步反应的酶YjhG过表达的重组菌株BW-056和BW-058;将重组质粒pACYC184- miniPtac-adhP-T1T2和pACYC184- Ptac-adhP- T1T2分别与pET30a-miniPtac-kivD共同转入BW-010感受态细胞,获得催化第四步反应的酶AdhP过表达的重组菌株BW-066和BW-067;通过将重组质粒pACYC184-miniPtac- xdh-T1T2和pACYC184-Ptac-xdh-T1T2分别与pET30a- miniPtac-kivD共同转入BW-010感受态细胞,则构建了催化第一步反应的酶Xdh过表达的重组菌株BW-073和BW-074。
将上述6种重组菌株与BW-025一起进行摇瓶转化实验,比较其BT的合成能力 (图5)。与BW-025相比,过表达了YjhG的重组菌株BW-056和BW-058的BT产量略有提高,但均不明显;BW-025过表达AdhP后,BT产量有所下降。而过表达Xdh的重组菌株BW-073和BW-074其BT产量较BW-025均有提高,尤其是编码基因xdh诱导型表达的BW-074的BT产量,提高了48.62%。
3 . 讨论 丁三醇是一种具有重要应用前景的多元醇,但作为一种非天然化合物,其生物转化途径仍面临着途径效率低的问题,因此对于BT合成途径的优化至关重要。为了实现此目的,首先应当识别整个催化过程的限制性步骤。Cao等利用重组E. coli转化木糖生产木糖酸可达到理论得率的近88%[18],Valdehuesa等报道了用重组E. coli转化木糖生产BT时,有82.28%的木糖转化为木糖酸,但只有10.25%的木糖最终转化为BT[3, 19],这表明后三步酶催化可能是整个途径的限速步骤。
由于第三步脱羧反应是非天然催化反应,同时脱羧反应又是不可逆反应,可推测该步反应极有可能是整个反应的限制性步骤[20],因此选择从脱羧反应入手优化BT合成途径。之前所有的报道,都沿用Niu等采用的来自恶臭假单胞菌的苯甲酰甲酸脱羧酶MdlC[3, 10, 11, 12, 21]。为了寻找与3-脱氧-D-甘油-戊酮糖酸更加匹配的底物,我们尝试了其他微生物来源的不同类型的脱羧酶:吲哚丙酮酸脱羧酶 (IpdC和KivD),磷酸烯醇丙酮酸脱羧酶 (AepY)。体内实验表明来自乳酸乳球菌L. lactis的α-酮异戊酸脱羧酶KivD的催化活力比MdlC提高了191%。因此,通过对于新的2-酮酸脱羧酶的筛选很大程度上解决了第三步酶对整个催化途径的限制。
在途径通量的优化过程中,一个限速反应的解决通常会导致其他反应会成为新的限制步骤。在筛选到KivD的工作基础上,我们通过对其他三步酶的表达量进行调节,进一步考察了其他3个反应是否会成为新的限速步骤。研究表明,YjhG的过表达对于BT产量的提升不明显,表明此时YjhG的表达量并不构成对整个途径的限制。醇脱氢酶AdhP的过量表达对于BT产量有一定的副作用,由于E. coli体内的AdhP具有多种功能,具有较宽的底物范围[22, 23, 24, 25],因此推测AdhP的过表达会对宿主的代谢造成较大的干扰。Xdh的过表达使得整个途径的催化效率又提高了48.62%,这表明在KivD的限制性因素得到解决后,Xdh的表达可能成为整个反应新的限速步骤。因此,本研究为BT的合成途径优化提供了很好的参考,后续工作可以在Xdh和KivD过表达菌株的基础上,对合成途径进行新一轮的优化,以进一步提高BT的产量。
除了对BT途径的优化外,本文还对BT的转化条件进行了初步的探索。转化初始菌量的提高可显著增加BT的转化效率,这是可以理解的。但当OD600达到40时,BT的产量却有所下降。我们推测,过高的菌量可能会造成溶氧不足,引起中间代谢产物的积累。我们在转化发酵液中确实检测到乙酸、乳酸和乙二醇等的积累 (结果未显示),这些代谢副产物可能会对重组菌的生长和代谢造成不利影响,从而最终影响到BT的转化效率。
综上所述,本文首先成功构建了以20 g/L D-木糖为底物合成0.3 g/L BT的E. coli工程菌株BW-011。之后对BT合成途径中的瓶颈进行了分析,确定了3-脱氧-D-甘油-戊酮糖酸的脱羧反应这一限速步骤,选出了几种可能催化该反应的2-酮酸脱羧酶并在底盘细胞BW-010中表达,找出了催化效率更高的新酶KivD,获得了BT产量更高的重组菌株BW-025。探究了温度、初始细胞密度和pH等条件对BW-025合成BT的影响,转化条件优化 (转化温度33.5 ℃、初始OD600在20-40、向培养基中添加缓冲剂) 后BW-025在72 h内最高可合成BT 2.38 g/L。与此同时,进一步对BW-025的BT合成途径中各个酶的表达量进行调节,评价了其对BT合成的影响,其中诱导型过表达xdh的重组菌株BW-074的BT得率较BW-025提高了48.62%,表明通过此方法对BT合成途径进行优化的思路是可行的。
参考文献
| [1] | Gouranlou F, Kohsary I. Synthesis and characterization of 1,2,4-butanetrioltrinitrate. Asian J Chem, 2010, 22(6): 4221-4228. |
| [2] | Ren T, Liu DX. Synthesis of cationic lipids from 1,2,4-butanetriol. Tetrahedron Lett, 1999, 40(2): 209-212. |
| [3] | Valdehuesa KNG, Liu HW, Ramos KRM, et al. Direct bioconversion of d-xylose to 1,2,4-butanetriol in an engineered Escherichia coli. Process Biochem, 2014, 49(1): 25-32. |
| [4] | Cai ZY, Wang ML. One-step synthesis of (S)-1,2,4-butanetriol via (S)-3-hydroxy-γ- butyrolactone. J Hefei Univ Technol, 2010, 33(6): 915-917 (in Chinese). 蔡征宇, 王明亮. (S)-3-羟基-γ-丁内酯一步法合成(S)-1,2,4-丁三醇的研究. 合肥工业大学学报(自然科学版), 2010, 33(6): 915-917. |
| [5] | Lau MK. Synthesis and downstream purification of 1,2,4-butanetriol. UMI Microform 3348145. Ann Arbor, MI, USA: Pro Quest LLC, 2006. |
| [6] | Pisacane FJ. 1,2,4-Butanetriol: analysis and Synthesis. Silver Spring: Naval Surface Weapons Center, 1982. |
| [7] | Luo AL, Qiao JJ, Song XC. Synthesis of 1,2,4- hydroxyl-butane. Northwest Pharmaceut J, 2007, 22(3): 144-145 (in Chinese). 罗阿利, 乔建军, 宋新潮. 1,2,4-丁三醇的合成工艺研究. 西北药学杂志, 2007, 22(3): 144-145. |
| [8] | Tandon VK, Van Leusen AM, Wynberg H. Synthesis of enantiomerically pure (S)-(+)-3-hydroxytetrahydrofuran, and its (R)-enantiomer, from malic or tartaric acid. J Org Chem, 1983, 48(16): 2767-2769. |
| [9] | Hanessian S, Ugolini A, Dubé D, et al. Facile access to (S)-1,2,4-butanetriol and its derivatives. Can J Chem, 1984, 62(11): 2146-2147. |
| [10] | Niu W, Molefe MN, Frost JW. Microbial synthesis of the energetic material precursor 1,2,4-butanetriol. J Am Chem Soc, 2003, 125(43): 12998-12999. |
| [11] | Frost JW, Niu W. Microbial synthesis of d-1,2,4-butanetriol: US, WO 2008/091288 A2. 2008-07-31. |
| [12] | Sun WL, Lu XY, Zong H, et al. Biosynthesis of D-1,2,4-butanetriol by an engineered Escherichia coli. Microbiol China, 2014, 41(10): 1948-1954 (in Chinese). 孙文龙, 陆信曜, 宗红, 等. 代谢工程改造大肠杆菌合成D-1,2,4-丁三醇. 微生物学通报, 2014, 41(10): 1948-1954. |
| [13] | Li XH, Cai Z, Li Y, et al. Design and construction of a non-natural malate to 1,2,4-butanetriol pathway creates possibility to produce 1,2,4-butanetriol from glucose. Sci Rep, 2014, 4: 5541. |
| [14] | Sambrook J, Russell DW. Molecular Cloning: A Laboratory Manual. 3rd ed. New York: Cold Spring Harbor Laboratory Press, 2001. |
| [15] | Datsenko KA, Wanner BL. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci USA, 2000, 97(12): 6640-6645. |
| [16] | Han C, Zhang WC, You S. Advances in the red-mediated recombination. China Biotechnol, 2003, 23(12): 17-21 (in Chinese). 韩聪, 张惟材, 游松. Red同源重组技术研究进展. 中国生物工程杂志, 2003, 23(12): 17-21. |
| [17] | Yu HM, Ma YC. Gene knockout strategies for metabolic pathway regulation in industrial microbes. Chin J Biotechnol, 2010, 26(9): 1199-1208 (in Chinese). 于慧敏, 马玉超. 工业微生物代谢途径调控的基因敲除策略. 生物工程学报, 2010, 26(9): 1199-1208. |
| [18] | Cao YJ, Xian M, Zou HB, et al. Metabolic engineering of Escherichia coli for the production of xylonate. PLoS ONE, 2013, 8(7): e67305. |
| [19] | Liu HW, Valdehuesa KNG, Nisola GM, et al. High yield production of d-xylonic acid from d-xylose using engineered Escherichia coli. Bioresour Technol, 2012, 115: 244-248. |
| [20] | Yim H, Haselbeck R, Niu W, et al. Metabolic engineering of Escherichia coli for direct production of 1,4-butanediol. Nat Chem Biol, 2011, 7(7): 445-452. |
| [21] | Abdel-Ghany SE, Day I, Heuberger AL, et al. Metabolic engineering of arabidopsis for butanetriol production using bacterial genes. Metab Eng, 2013, 20: 109-120. |
| [22] | Reid MF, Fewson CA. Molecular characterization of microbial alcohol dehydrogenases. Crit Rev Microbiol, 1994, 20(1): 13-56. |
| [23] | Shafqat J, Höög JO, Hjelmqvist L, et al. An ethanol-inducible MDR ethanol dehydrogenase/ acetaldehyde reductase in Escherichia coli. Eur J Biochem, 1999, 263(2): 305-311. |
| [24] | Machielsen R, Uria AR, Kengen SWM, et al. Production and characterization of a thermostable alcohol dehydrogenase that belongs to the aldo-keto reductase superfamily. Appl Environ Microbiol, 2006, 72(1): 233-238. |
| [25] | Thomas LM, Harper AR, Miner WA, et al. Structure of Escherichia coli AdhP (ethanol-inducible dehydrogenase) with bound NAD. cta Crystallogr Sect F: Struct Biol Cryst Commun, 2013, 69(Pt 7): 730–732.. |
