邓文芳1,3, 吴佳鹏1

 , 洪义国1
, 洪义国1
 , 刘晓晗1,3, 胡耀豪2
, 刘晓晗1,3, 胡耀豪2 1. 广州大学大湾区环境研究院, 珠三角水质安全与保护教育部重点实验室, 广东 广州 510006;
2. 中国科学院南海海洋研究所, 热带海洋环境国家重点实验室, 广东 广州 510301;
3. 广州大学环境科学与工程学院, 广东 广州 510006
收稿日期:2020-11-23;修回日期:2021-02-08;网络出版日期:2021-03-11
基金项目:国家自然科学基金(42006122,9185111);广东省自然科学基金粤穗联合基金(2020A1515110597,2019B1515120066);广州市教育局高校青年人才项目(202032795)
作者简介:吴佳鹏,博士,广州大学大湾区环境研究院讲师。研究方向为近海微生物氮循环过程。主持承担国家自然科学基金青年基金项目、广东省基金面上项目和联合基金青年项目、广州市科技计划项目和广州市教育局青年人才项目,参与973项目、国家自然科学基金重大研究计划项目和广东省重点项目等多项科研项目。发表SCI索引学术论文30余篇,论文总被引200余次。担任中国毒理学会环境与生态毒理学专业委员会会员.
*通信作者:吴佳鹏, Tel/Fax: +86-20-39337914;E-mail: wujiapeng@gzhu.edu.cn;
洪义国, E-mail: yghong@gzhu.edu.cn.
摘要:真菌反硝化过程的发现打破了反硝化过程只发生在原核生物中的传统认识,是对全球微生物氮循环过程的重要补充。真菌参与的反硝化过程由于缺乏N2O还原酶,其终产物为具有强辐射效应的温室气体N2O。真菌在环境中分布广泛,生物量巨大,故真菌反硝化作用对全球N2O释放通量的贡献是不容忽视的。近年来许多研究表明,真菌反硝化过程是自然环境中N2O产生的重要途径。本文对反硝化真菌的发现、多样性及分布、产生N2O的机制和活性测定方法等几个方面进行综述,并对未来的研究提出展望。
关键词:真菌反硝化群落组成反硝化机制活性测定N2O贡献
The diversity distribution and N2O production driven by fungal denitrification in different natural ecosystems
Deng Wenfang1,3, Wu Jiapeng1

 , Hong Yiguo1
, Hong Yiguo1
 , Liu Xiaohan1,3, Hu Yaohao2
, Liu Xiaohan1,3, Hu Yaohao2 1. Institute of Environmental Research at Greater Bay Area; Key Laboratory for Water Quality and Conservation of the Pearl River Delta, Ministry of Education, Guangzhou University, Guangzhou 510006, Guangdong Province, China;
2. State Key Laboratory of Tropical Oceanography(LTO), South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, Guangdong Province, China;
3. Shcool of Environmental Science and Engineering, Guangzhou University, Guangzhou 510006, Guangdong Province, China
Received: 23 November 2020; Revised: 8 February 2021; Published online: 11 March 2021
*Corresponding author: Jiapeng Wu, Tel/Fax: +86-20-39337914;E-mail: wujiapeng@gzhu.edu.cn;
Yiguo Hong, E-mail: yghong@gzhu.edu.cn.
Foundation item: Supported by National Natural Science Foundation of China (42006122, 9185111), by the Natural Science Foundation of Guangdong Province (2020A1515110597, 2019B1515120066) and by the Young Talent Project of Guangzhou Education Bureau (202032795)
Abstract: Nitrous oxide (N2O) is a potent greenhouse gas which generates approximately 300-fold powerful greenhouse effect than carbon dioxide (CO2) and is one of the main compounds that causing ozone-depletion in stratospheric. Nitrification and denitrification of microorganism is the most important path for N2O production. It has long been considered that denitrification only occurred in prokaryote until in 1970's two Japanese scientists found that fungi like Fusarium oxysporum also had the ability to denitrifying. Unlike bacterial denitrification (bDNF), N2O is the end-product of fungal denitrification process (fDNF) due to the lack of nitrous oxide reductase (nosZ). It means that fDNF usually produce more N2O than bDNF. Recent studies have proved that fDNF contributed over 50% of the total N2O emission in soil and 70% in high permeable sediments thus it shouldn't be neglect. In this review, we elucidated the species composition, denitrification mechanism, measurements and N2O-contribution of fDNF base on the current studies. At last, we discussed some problems remain to be solved on the research of fDNF and suggest prospects for future studies.
Keywords: fungal denitrificationfungal communitydenitrification mechanismactivity determinationN2O contribution
氮循环是生态系统物质循环的关键步骤,在为生命提供氮源和维持地球稳态方面起到了重要作用[1]。微生物参与的氮循环主要有以下六个过程:同化作用、氨化作用、硝化作用、反硝化作用、厌氧氨氧化作用和固氮作用[2]。其中微生物的反硝化作用是指微生物将NO3–和NO2–向气态的NO、N2O和N2逐步还原的过程[3]。长期以来,反硝化作用被认为是原核生物独有的特征,而真菌反硝化过程的发现则更新了传统观念[4]。传统上认为,影响全球N2O通量的主要生物来源是微生物的硝化和反硝化作用。近期的研究表明,真菌反硝化比细菌反硝化对土壤排放的N2O的贡献更大[5]。同时,有研究发现作为大气N2O主要来源的陆海界面沉积物,其中存在的真菌反硝化过程(fDNF)对N2O的释放也发挥着至关重要的作用[6]。因此,有必要进一步认识自然环境中存在的真菌反硝化过程,以加深对环境中N2O的微生物来源的了解。本文综述了国内外有关反硝化真菌的研究进展,包括反硝化真菌的在环境中的多样性及分布,反硝化机制、反硝化活性测定方法及其在自然环境氮循环中扮演的角色等方面的介绍。
1 真菌反硝化过程的发现 真菌反硝化是一个新兴的概念,最早于1972年由Bollag和Tung观察到在低氧浓度条件下尖孢镰刀菌(Fusarium oxysporum)和茄病镰刀菌(Fusarium solani)能还原亚硝酸盐并释放出N2O,但他们不能解释这一现象涉及的生理反应[7]。直到1991年,Shoun和Tanimoto通过培养F. oxysporum的纯菌株并测定其细胞萃取液中相关反硝化还原酶的活性,首次证实该菌株在厌氧及存在NO3–/NO2–的条件下是通过反硝化作用产生N2O,且由其内存在独特的细胞色素P450(P-450dNIR)参与还原反应[8]。真菌反硝化概念的提出打破了以往认为反硝化作用仅存在于原核生物的观念,而F. oxysporum也成为一种研究反硝化真菌生理生态和系统发育的模式生物。在1992年,Shoun等研究发现,许多来自Fusarium、Gibberella、Nectria、Cylindrocarpon和Trichoderma属的真菌具有反硝化能力,能够在厌氧条件下将NO3–或NO2–还原成NO或N2O[9]。Tanimoto等在早期利用抑制剂研究真菌的反硝化作用机制时发现,与仅添加亚硝酸盐的处理组相比,当添加含氮抑制剂时,反硝化产物中能检测出更高的氮氧化物的含量。随后他们利用了叠氮化物(azide)、水杨基异羟肟酸(salicylhydroxamic acid,SHAM)和氯化铵(ammonium chloride)等与亚硝酸盐混合培养F. oxysporum MT-811,结果发现添加亚硝酸盐和SHAM的处理组最终生成的N2O的量远远超过了与亚硝酸盐添加量的化学计量比,而与两种添加物的总量近乎相等。添加了叠氮化物和亚硝酸盐的处理组除了N2O还观察到了N2,生成的N2总量与叠氮化物的量相等,说明N2来源于叠氮化物。
但若培养基不添加亚硝酸盐,则没有N2O或N2的产生。因此,Tanimoto等认为,含氮化合物本身无法诱导反硝化系统,但能够与亚硝酸盐通过真菌反硝化系统进行共反硝化(Co-denitrification)反应从而产生N2O或N2[10-11]。
随着研究的深入,真菌反硝化过程的分子机制也随之被阐明[12–14]。真菌具有与细菌相似的反硝化系统,细菌反硝化是将NO3–/NO2–逐步还原为N2(NO3–→NO2–→NO→N2O→N2)的过程,参与的还原酶分别为硝酸盐还原酶(nitrate reductase,Nar)、亚硝酸盐还原酶(nitrite reductase,Nir)、一氧化氮还原酶(nitric oxide reductase,Nor)以及氧化亚氮还原酶(nitrous oxide reductase,Nos)[15]。真菌反硝化系统缺乏编码N2O还原酶的基因(nosZ)而不具备将N2O还原成N2的能力,故真菌反硝化的路径比细菌的短,是将NO3–或NO2–逐步还原成N2O的过程[15]。驱动真菌反硝化作用的酶位于真菌的线粒体膜上,与细菌相似的是真菌反硝化作用不需要严格厌氧,在好氧、微好氧以及微厌氧环境中均可以发生,因此人们预测在好氧与厌氧并存的环境中,真菌反硝化作用在驱动氮循环方面发挥重要作用[5, 15]。
2 反硝化真菌的种类及其环境分布 据Mothapo等研究报道,从1972年至2014年间有关反硝化真菌的研究报告中共发现60个属119种反硝化真菌[15]。这些源自富集、纯培养或者土壤样品的反硝化真菌有90%属于子囊菌门(Ascomycota),其余分别属于担子菌门(Basidiomycota,7%)和接合菌门(Zygomycota,3%)。而在子囊菌门中,有46%的反硝化真菌属于粪壳菌纲(Sordariomycetes),24%属于散囊菌纲(Eurotiomycetes),另有9%和8%分别属于酵母纲(Saccharomycetes)和座囊菌纲(Dothideomycetes)。代表性的子囊菌真菌包括链格孢菌(Alternaria alternata),灰质芽孢杆菌(B.cinerea)以及以下属内的种:曲霉属(Aspergillus)、镰刀菌属(Fusarium)、青霉属(Penicillium)和木霉属(Trichoderma)[15]。
基于NCBI数据库,搜集反硝化真菌P450nor基因序列构建进化树如图 1所示[16]。在子囊菌门内的Sordariomycetes纲中分布有最丰富的反硝化真菌属,包括Fusarium、Chaetomium、Trichoderma、Metarhizium等属,其次是Eurotiomycetes纲的Aspergillus、Trichophyton、Histoplasma等属,Leotiomycetes纲有Pseudogymnoascus、Glare、Marssonina和Sclerotinia属,而Dothideomycetes纲有Bonordeniella、Macrophomina及Bipolaris属。在P450nor基因进化树上不同纲的反硝化真菌之间相互聚集成簇(如Leotiomycetes纲Pseudogymnoascus属与Eurotiomycetes纲的Trichophyton属),推测在进化过程中,不同纲的反硝化真菌的P450nor基因发生了水平转移,导致反硝化真菌18S rRNA与P450nor基因进化不同步,从而出现进化树相同纲的反硝化真菌分布在不同簇[17]。
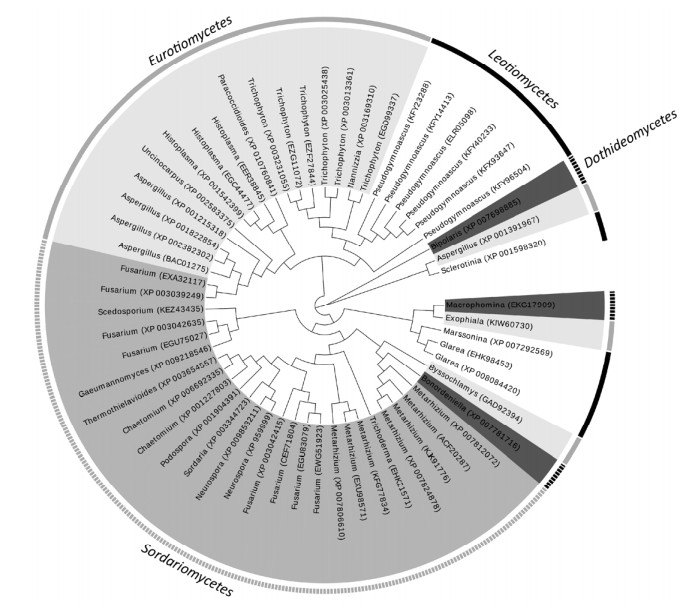 |
| 图 1 基于P450nor基因的反硝化真菌系统发育树 Figure 1 Phylogenetic tree of fungal denitrifiers based on P450nor gene. |
| 图选项 |
表 1汇总了近年来不同生境中反硝化真菌的种属组成以及鉴定方法。通常从土壤环境中识别和表征反硝化真菌的方法包括3个步骤,首先是将土壤样品中的真菌进行分离和纯培养,随后在液体培养基(如察氏培养基、甘油-蛋白胨培养基、马铃薯-葡萄糖培养基等)测试这些真菌纯菌株产生N2O的活性,最后将这些具有反硝化活性的真菌基于形态学特征或者真菌ITS、18S rRNA基因或者真菌nirK功能基因等序列分析后进行属或种水平上的鉴定[15]。
表 1. 不同生境中反硝化真菌在属水平上的群落组成 Table 1. Community composition of fungal denitrifiers at genus level in different habitats
| Environment | Identification methods | Genus | References | ||||
| Arable soils | Fungal nirK gene | Aspergillus | Trichoderma | Chaetomium | Byssochlamys | Actinomucor | [1] |
| Fusarium | Trichocladium | Talaromyces | |||||
| Cattle impacted soils | Pure culture Morphology | Aspergillus | Trichoderma | Verticillium | Monographella | Eurotium | [18] |
| Fusarium | Gibberella | Acremonium | Pseudallescheria | Humicola | |||
| Penicillium | Scopulariopsis | Clonostachys | |||||
| Conventional farming | Pure culture Fungal ITS gene | Aspergillus | Penicillium | Gibberella | Neosartorya | Bionectria | [19] |
| Fusarium | Trichoderma | Hypocrea | Neocosmospora | ||||
| Integrated crop and livestock system | Aspergillus | Penicillium | Gibberella | Mortierella | Hypocrea | ||
| Fusarium | Trichoderma | Ophiocordyceps | |||||
| Organic farming | Aspergillus | Penicillium | Metarhizium | Mortierella | Gibberella | ||
| Fusarium | |||||||
| Plantation forestry | Aspergillus | Penicillium | Bionectria | Trichoderma | Mucor | ||
| Fusarium | |||||||
| Abandoned field | Fusarium | Trichoderma | Bionectria | Cladosporium | Phoma | ||
| Penicillium | Gibberella | ||||||
| Groundwater and sediment | Pure culture Fungal SSU rRNA and ITS gene | Aspergillus | Penicillium | Neolinocarpon | Phialocephala | Coniochaeta | [20] |
| Fusarium | Teberdinia | Rhodosporidium | Paecilomyces | Apiospora | |||
表选项
据Xu等研究,在不同的农田土壤(红粘土、冲积土和黑土)中nirK型反硝化真菌的丰度为9.92×108–3.15×109 copies/g[1]。基于引物nirKfF/ nirKfR的PCR及高通量测序表明,不同农田土壤中含有的nirK型反硝化真菌具有高度的相似性,主要来自Hypocreales、Sordariales、Eurotiales和Mucorales目,以及8个属:Fusarium、Trichocladium、Chaetomium、Talaromyces、Trichoderma、Aspergillus、Byssochlamys和Actinomucor,其中Hypocreales纲和Fusarium属的反硝化真菌丰度最高[1]。他们认为长期的耕作活动导致Fusarium属数量的积累,使得不同类型农田土壤具有相似的nirK型反硝化真菌群落。而真菌nirK基因丰度与土壤N2O的排放呈正相关关系,也表明反硝化真菌在农田土壤产生N2O的过程中起重要作用[1]。Jirout等用琼脂平板法从Bohemia南部的牧场土壤样品中分离真菌并测定其反硝化活性,其中有63.8%的真菌菌株具有反硝化活性,而这些菌株隶属于13个属(如表 1所示),主要来自Hypocreaceae和Trichocomaceae[18]。Mothapo等分离了5种农业土壤(传统农田、综合农牧田、有机农田、人工林和废弃农田)中的真菌并测定其反硝化活性,基于ITS基因引物ITS-1/ITS-2鉴定的结果表明,大多数产N2O的真菌菌株属于Sordariomycetes、Eurotiomycetes和Dothideomycetes纲,而五种不同农业土壤中均有分布 Fusarium、Penicillium、Aspergillus和Trichoderma属的反硝化真菌[19]。除了农田环境,近年来Jasrotia等[20]从铀和硝酸盐污染的地下环境中分离了反硝化真菌。研究发现来自Teberdinia、Aspergillus、Coniochaeta、Neolinocaron、Fusarium、Penicillium、Apiospora、Phialocephala和Paecilomyces属的反硝化真菌能够将NO3–还原成N2O。在此之前的研究仅发现Fusarium、Cylindrocarpon和Trichosporon属的真菌能够还原NO3–为N2O[20]。
上述研究表明,Fusarium、Penicillium、Aspergillus和Trichoderma属反硝化真菌广泛分布在陆地环境中。目前,已有众多针对土壤反硝化真菌多样性的研究,但对其他环境(如海洋)中的反硝化真菌群落仍需要进一步探索。近期有研究表明,在雅浦海沟深海沉积物中分离了两株具有反硝化能力的枝孢菌,这表明在深海环境中同样存在反硝化真菌,且其群落组成以及对N2O的排放贡献仍是未知,有待进一步探索[21]。
3 真菌反硝化过程的机制 迄今,研究者对F. oxysporum和C. tonkinense的反硝化系统在细胞内的定位、参与反硝化过程的底物和酶的作用机制已有较为全面的研究,它们的反硝化系统如图 2所示[22]。
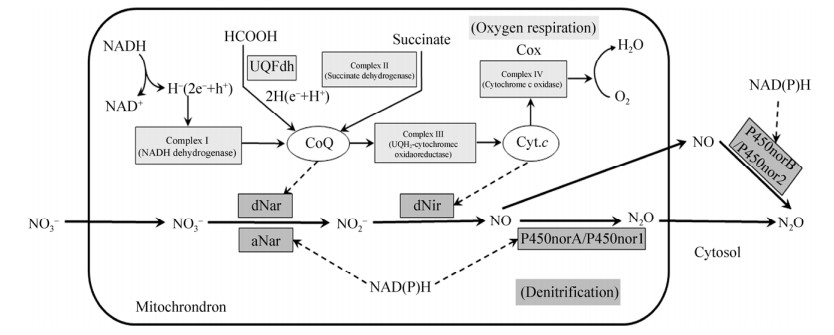 |
| 图 2 F. oxysporum和C. tonkinense与线粒体呼吸链相关的反硝化系统[22] Figure 2 Mitochondria-denitrifying system of F. oxysporum and C. tonkinense coupling to respiratory chain.The key enzymes indenitrifying system of F. oxysporum contains dNar, dNir (NirK) and P450norA/P450norB, while the key enzymes indenitrifying system of C. tonkinense contains aNar, dNir (NirK) and P450nor1/P450nor2[22]. |
| 图选项 |
1996年,Kobayashi等通过细胞分离和免疫荧光法技术观察到F. oxysporum的Nar和C. tonkinense的Nir均分布在线粒体上。他们通过分离Nar和Nir,发现呼吸底物如苹果酸、丙酮酸、琥珀酸和甲酸等能为Nar和Nir提供电子还原NO3–或NO2–,而呼吸链抑制剂如鱼藤酮、抗霉素A和噻吩甲酰三氟丙酮等能够显著抑制真菌Nar或Nir的活性[14]。这些证据表明真菌反硝化过程发生在线粒体上,真菌反硝化过程与呼吸链合成ATP相偶联[14]。Kobayashi等还认为,F. oxysporum的Nar系统利用甲酸提供电子的特性类似于大肠杆菌(E. coil)的硝酸盐还原系统,其中可能存在与呼吸链相关的甲酸脱氢酶(formate dehydrogenase,Fdh)系统支持硝酸盐的厌氧还原[14]。而后Uchimura等研究证实,在F. oxysporum的线粒体中检测到了Fdh的活性[23]。研究还发现,F. oxysporum的细胞内至少存在两种类型的Nar,一种是位于线粒体膜上的异化型硝酸盐还原酶(dissimilatory nitrate reductase,dNar),一种是存在于可溶组分中的同化型硝酸盐还原酶(assimilatory nitrate reductase,aNar),其中aNar仅从NADH或NADPH中接收电子,而dNar从甲酸等呼吸底物中接收电子[23]。进一步的分析表明,Fdh是通过UQ/UQH2池从甲酸转移电子到dNar,Fdh-Nar的耦合参与了真菌NO3–呼吸[23]。对部分纯化后的dNar进行光谱学分析显示,该酶含有钼蛋白、细胞色素b以及铁硫蛋白。无论是Fdh-Nar的耦合还是酶的组成,dNar参与的NO3–呼吸与大肠杆菌(Escherichia coli)的非反硝化型NO3–呼吸系统十分相似,而Fdh-Nar的耦合从未在反硝化细菌中发现[23]。据Takaya等研究报道,F. oxysporum在缺氧或反硝化条件下能同时进行O2呼吸和NO3–呼吸,两种呼吸同享线粒体的呼吸链,是一种杂合的呼吸过程[24]。在这一过程中,dNar至少可以从三条途径接收电子(图 2):泛醌-甲酸脱氢酶(UQFdh)、线粒体呼吸链酶复合物Ⅰ (NADH脱氢酶)或线粒体呼吸链酶复合物Ⅱ (琥珀酸脱氢酶)。酶活性测定实验表明,Fdh-Nar的耦合反应在三条电子传输路径中具有最高的活性,因此是dNar接收电子的主要途径[24]。
除了在F. oxysporum细胞内发现的特殊Fdh-Nar耦合NO3–呼吸系统,Watsuji等认为,与F. oxysporum具有亲缘关系的C. tonkinense在特定条件下可能进行硝酸盐的反硝化[25]。经过一系列的酶活性检测实验,Watsuji等在C. tonkinense的细胞胞浆内检测出一种依赖NADH的同化型NO3–还原酶(assimilartory nitrate reductase,aNar)的活性,而未检测出dNar[25]。在探究碳氮源对C. tonkinense反硝化过程的影响实验中,Watsuji等发现在特定条件下(初始含氧随后厌氧),当NO3–是唯一的氮源时,aNar和aNir仅被诱导用于同化过程[25]。借助同位素标记法(15NaNO3和14NaNO3),他们发现在另外添加NH4+和可发酵糖(葡萄糖或者半乳糖)后,NO3–出现消耗,且同位素标记结果表明生成的N2O来源于NO3–[25]。此外,添加Nar抑制剂(钨酸盐)后也观察到NO3–的消耗和N2O的产生过程受到明显抑制。因此,Watsuji等认为NH4+和可发酵糖(葡萄糖或者半乳糖)作为电子供体的存在诱导了C. tonkinense发生硝酸盐反硝化[25]。这是首次发现aNar利用同化的系统达到异化(反硝化)的目的,本被认为仅能进行亚硝酸盐反硝化的C. tonkinense在特定条件下存在aNar进行硝酸盐反硝化。另有研究称,在厌氧条件下aNar可将NO3–还原成NH4+,此过程可利用底物产能使细胞生长,称为真菌的氨发酵[26]。
F. oxysporum和C. tonkinense具有不同类型的Nar,但均含有NirK (含铜型dNir)和P450nor,由于缺乏N2O还原酶,所以终产物是具有强烈温室效应的N2O[22]。1995年,Kobayashi等在F. oxysporum中首次发现和分离了异化型亚硝酸盐还原酶(dissimilatory nitrite reductase,dNir),该酶由含有两个铜原子的亚基组成,分子量为41.8 kDa,能从菌体自身合成的类天青蛋白和细胞色素c549中接收电子,将NO2–还原为NO,同时耦合线粒体合成ATP,免疫染色结果显示dNir位于线粒体的内外膜之间[13–14]。1999年,Kubota等通过破碎C. tonkinense的细胞在可溶组分和膜组分中均检测出Nir的活性。经过纯化、SDS-PAGE检测及光谱学分析显示,两种Nir在性质上相同,是一种亚基大小为67 kDa的同源二聚体[27]。酶活性抑制实验显示,C. tonkinense的Nir可以被二乙基二硫代氨基甲酸强烈抑制,结合其吸收光谱的结果意味着这是同F. oxysporum类似的含铜型dNir (CuNir)[27]。在C. tonkinense的线粒体中,呼吸底物如苹果酸、丙酮酸和琥珀酸可以为dNir作用过程提供电子[14]。以丙酮酸和苹果酸为碳源的dNir的活性被鱼藤酮和抗霉素抑制而不被噻吩甲酰三氟丙酮(TTFA)抑制,而以琥珀酸为碳源的dNir活性被抗霉素和TTFA抑制而不被鱼藤酮抑制[14]。因此,Kobayashi等认为C. tonkinense的dNir与O2呼吸过程共享线粒体电子传递系统,与细菌的Nir同是经过呼吸链酶复合物Ⅲ(UQH2-细胞色素c氧化还原酶)来得到电子[14]。
以F. oxysporum和C. tonkinense为代表的真菌在反硝化过程中后一步的酶是P450nor,这是一种具有脂氧合酶活性和P450性质的血红素蛋白,能将NO还原成N2O,在线粒体和细胞质中均有分布[28]。P450nor属于细胞色素P450超基因家族,编号是CYP55[22]。细胞色素P450具有加单氧酶的作用,能参与重要的生理反应如机体解毒、固醇类激素的合成等,在真核和原核生物中均有存在[29]。尽管P450的分子大小多种多样且广泛分布于生物体中,但都起源于同一个基因[30]。比起真核生物的P450s,CYP55的起源与细菌P450s具有更近的亲缘关系,有研究推测该基因可能是从放线菌中水平转移到真菌内而产生的[22]。
在F. oxysporum中基因CYP55A1编码两种P450nor同工酶P450norA和P450norB,由于两者编码的起始密码子不同,使得P450norA作用于线粒体内,而P450norB作用于细胞质中,以NADH为电子供体。在C. tonkinense中也含有两种P450nor同工酶:位于线粒体的P450nor1(由CYP55A2基因编码)和位于细胞质的P450nor2(由CYP55A3基因编码)。其中P450nor1只能利用NADH提供电子,而P450nor2更倾向于从NADPH中接受电子[22, 31]。P450nor参与反硝化过程的整体反应式为:2NO+NADH+H+→N2O+H2O+NAD+[24]。真菌的P450nor从NADH(或者NADPH)中接受电子的特性与传统的P450从其他蛋白质组分(如P450还原酶和铁氧还蛋白)中接受电子形成了鲜明的对比,同时P450nor参与的还原过程不需要耦合呼吸链,这也是真菌反硝化系统的独特性之一[24, 30–31]。
从真菌反硝化作用的发现至今,科学家们对真菌反硝化系统的机制也进行了深入的研究。其中,真菌F. oxysporum和C. tonkinense的反硝化系统已被详细的阐明,在它们的反硝化系统中dNar或aNar同dNir和P450nor参与了反硝化,N2O是最终产物。研究发现,只有少数的真菌比如F. oxysporum和Gibberella fujikuroi (Swada) Wollenw.能够利用Fdh-Nar耦合的系统将NO3–还原为NO2–,大多数的真菌以NO2–为反硝化的起点[15]。而Kim等表明真菌nirK与细菌nirK拥有共同的祖先,许多含有nirK同源物的真菌也含有P450nor的同源物,表明这种同时含有nirK和P450nor类型的反硝化系统在真菌中普遍存在[32]。尽管真菌Nar和Nir与细菌具有许多相似性,但真菌P450nor却与细菌的Nor具有较大的差别,这与反硝化真菌线粒体起源于细菌的假说有一定的矛盾[33]。另外,以真菌F. oxysporum和C. tonkinense为代表的反硝化机制能否代表大部分真菌的反硝化作用机制?除了这两种真菌反硝化机制是否还存在其他不同的真菌反硝化作用机制仍是未知,这些问题值得我们进一步探究。
4 真菌反硝化活性的测定 传统上认为,农业和自然土壤中的微生物硝化和反硝化作用是全球范围内N2O的主要来源,其中由真菌反硝化作用排放的N2O约占总N2O排放量的34%–42%[34]。基于现有的研究,常用于测定真菌反硝化对N2O的贡献的方法为基质诱导呼吸抑制法(SIRIN),而同位素法则常用来溯源土壤N2O的产生途径,该部分将围绕这两种主要方法进行介绍。
4.1 基质诱导呼吸抑制法(SIRIN)[35] SIRIN法最初于1973和1975年由Anderson和Domsch用来测定土壤真菌和细菌的生物量及相对活性,而后科学家们改良此法用于测定土壤真菌和细菌对N2O产生的相对贡献[35-36]。该方法是以葡萄糖或蛋白胨作为细菌和真菌的碳源诱导基质,利用生物抑制剂,通常是放线菌酮(cycloheximide,C15H23NO4)和链霉素(streptomycin,C21H39N7O12)来分别选择性抑制真菌和细菌合成蛋白质的活性。其中放线菌酮能够抑制真核生物60S核糖体亚单位肽基转移酶的活性,从而抑制真菌的反硝化活性。而链霉素可以抑制原核生物的蛋白质的初始合成,引起信使RNA的误读,以此抑制细菌的活性。除抑制剂外,还可配合使用一定浓度的乙炔来抑制样品中微生物的硝化作用对N2O排放量造成的影响[35, 37]。SIRIN法通过分组添加相应的抑制剂(放线菌酮、链霉素或两种结合),利用气相色谱仪测定各组在不同时间段内的N2O浓度,再结合相应公式即可计算出真菌对N2O产生的贡献及速率[38]。
该方法在样品(土壤、沉积物或水体)测定之前,需要先进行预实验来确定抑制剂的最佳浓度,避免高浓度的抑制剂对细菌或真菌造成非靶向抑制。确定最佳抑制剂浓度需要设置四组实验处理:(1)空白对照组(A),不添加抑制剂;(2)放线菌酮组(B),系列浓度的放线菌酮;(3)链霉素组(C),系列浓度的链霉素;(4)双抑制剂组合组(D),不同浓度组合的两种抑制剂。设置3组以上的平行实验,在一系列时间梯度(如4、12、20和32 h)内利用气相色谱仪测定瓶中N2O的浓度并与时间进行回归来计算N2O的产生速率[38-39]。利用抑制剂加性比(IAR)的值(式1)来判断两种抑制剂是否具有非靶向效应,当IAR的值接近1时,相应浓度抑制剂效果最佳[36]。
 | 公式(1) |
确定抑制剂最佳浓度后,设置与预实验相似的四组处理,采用微宇宙培养实验测定样品中真菌产生N2O的速率(式2)及贡献(式3)[38]。
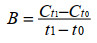 | 公式(2) |
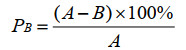 | 公式(3) |
同理,代入数据可求出样品中细菌和非生物部分产生N2O的速率及相对排放贡献。
表 2统计了近年来使用SIRIN法研究时选用的抑制剂浓度以及各自对N2O排放的抑制率,其中放线菌酮的浓度最低为1 mg/g,最高为15 mg/g,链霉素的浓度最低为2 mg/g,最高为8 mg/g。使用抑制剂法判别土壤的微生物作用时,抑制效果的差异可能是土壤理化性质、生物群落组成不同或者抑制剂法本身的缺陷造成的。因为不排除在土壤样品中部分微生物对抑制剂具有耐受性的情况。因此,选择适合的抑制剂浓度是有效使用该方法的关键。最佳的抑制剂浓度应对样品中微生物的呼吸速率没有显著影响,且不应抑制非目标微生物的活性,否则测定结果将出现较大的误差。Yanai等利用SIRIN法研究了印度尼西亚加里曼丹中部可耕地泥炭土中的真菌反硝化作用[40]。结果发现,与未添加抑制剂的土壤相比,链霉素的添加减少了31%的泥炭土N2O排放率,而放线菌酮的添加则减少了81%的N2O排放率,这说明真菌是泥炭土N2O排放的主要贡献者[40]。章伟等发现米槠天然林土壤中真菌的活性显著高于细菌,但两种微生物对土壤N2O的贡献量相当[41]。Chen等利用同浓度组合的抑制剂研究了五种农业土壤中真菌、细菌对N2O排放的贡献,其中除了种植园林业土壤中的真菌对N2O的贡献大于细菌外,其余四种土壤中真菌和细菌对N2O的贡献相同[38]。Wei等指出,施用肥料后农田土壤N2O的排放主要是由真菌的反硝化作用驱动[42]。
表 2. 不同环境土壤中使用的抑制剂浓度及对微生物产生N2O的抑制效果 Table 2. The concentrations of inhibitors used in different types of soils and the inhibitory effects on N2O emission by microbe
| Soils | Inhibitor concentration gradient/(mg/g) | Inhibitor concentration/(mg/g) | Inhibitory rate on N2O emission/% | References | |||||
| Cycloheximide | Streptomycin | Cycloheximide | Streptomycin | Cycloheximide | Streptomycin | ||||
| Red clay soil | - | 8 | 5 | 66 | 27 | [1] | |||
| Alluvial soil | 5 | 5 | 19 | 55 | |||||
| Black soil | 8 | 5 | 53 | 22 | |||||
| Conventional farming soil | (0, 0.5, 1, 2, 3, 5) | 1 | 2 | 40 | 38 | [38] | |||
| Integrated crop and Livestock soil | 44 | 45 | |||||||
| Organic farming soil | 40 | 54 | |||||||
| Plantation forestry soil | 50 | 30 | |||||||
| Abandoned field soil | 51 | 41 | |||||||
| Peat soil | - | 15 | 3 | 81 | 31 | [40] | |||
| Forest soil | - | 15 | 3 | 51 | 44 | [41] | |||
| Cropland soil | (0, 2, 5, 10) | (0, 2, 5, 8, 12) | 5 | 8 | 84 | 20 | [42] | ||
| Tea soil | (0, 2, 6, 10) | (0, 2, 6, 12) | 10 | 6 | 70 | 23 | [50] | ||
表选项
直至今天SIRIN法仍是研究土壤真菌和细菌对N2O的相对贡献的主要方法,具备良好的研究基础。该法操作简单,可以在短时间内快速测定土壤中微生物的活性比,是实验室较为常用的一种测定真菌反硝化活性的方法。但是该方法是从总体上粗略估计样品真菌和细菌对N2O的相对贡献大小,而且底物的选择和抑制剂的浓度均会影响某些微生物的活性(耐受或不耐受),使结果出现偏差等。为此,可以结合使用其他测定方法(如同位素法),彼此之间的相互验证得出更为可靠有效的数据[41]。
4.2 同位素方法 由于土壤中具有复杂的微生物群落,要判断土壤N2O的来源在以往是一项困难,而同位素及相关仪器的发展则带来了有效解决方法。N2O的稳定同位素特征值如δ15N、δ18O和SP等是区别土壤N2O产生和消耗过程(硝化、细菌反硝化、真菌反硝化和硝化反硝化)的有效指标[43]。由于同位素的分馏效应的影响,在反硝化过程中导致生成的N2O与其前体分子相比出现δ15Nbulk值(产物N2O和前体分子NO2–/NO3–的δ15N之间的差值)的降低及δ18O值的升高,同时与未受还原作用影响的N2O相比,N2O还原过程会导致残余N2O的δ15Nbulk和δ18O值的升高。此外,δ18O还会受到反硝化中间产物与水之间发生氧交换的影响。因此,在解读N2O的氮氧同位素数据时必须考虑反硝化过程中同位素分馏和氧交换对N2O的同位素组成造成的影响[44]。
在N2O分子中,根据15N所处的位置可分为中心位15Nα(14N-15N-O)和末端位15Nβ(15N-14N-O),它们的相对丰度分别用δ15Nα和δ15Nβ表示,两者的差值称为位嗜值(site preference,SP=δ15Nα-δ15Nβ)。SP值在分析N2O产生途径(如硝化和反硝化)的相对贡献比例中不会受到N2O前体分子的同位素影响,基于前人的纯培养研究提供的各种N2O生成途径的SP值的差异性数据,可以用作区分N2O来源的重要依据[45]。Stuka等表明F. oxysporum和C. tonkinense的SP值约为(37.0±2.3)‰,明显区别于反硝化细菌纯培养中测定的SP值(约为0‰),而真菌反硝化产生N2O的SP值与在硝化细菌纯培养中测定的平均SP值(约为33‰)之间没有明显的差异[46]。他还表示真菌反硝化过程和细菌硝化过程的δ15N和δ18O是不同的,因此为了能更准确判断N2O的来源,多种同位素特征值的互相结合综合判断是解决该问题的有效手段。
近期Wankel等利用15N、18O同位素富集系数、SP值和δ17O特征值揭示了德国北海Sylt岛上的K?nigshafen沿海潮间带沉积物中的真菌反硝化作用及铁的氧化还原过程是驱动海岸沉积物产生N2O的重要途径[6]。他们发现在结合Δδ15N和SP值来区别N2O来源途径时会出现数据重叠(如真菌反硝化和细菌、古菌氨氧化),难以判断重叠部分数据所代表的N2O产生来源[6]。所以Wankel等还借助三氧同位素技术[47]来进一步准确区分N2O的产生来源。通过比较稳态时的Δ17ON2O和Δ17ONO2,他们发现在高NO3–条件培养下,有70%–80%的N2O是从NO2–中通过还原而不是氨氧化途径转化而来的。同时结合升高的SPN2O值和微弱变化的Δδ15N,再通过同位素质量平衡法计算出4个主要生物过程如细菌反硝化、真菌反硝化、氨氧化及硝化反硝化各自作用过程产生N2O的相对贡献值从而推断真菌反硝化和化学反硝化作用可能是海岸沉积物N2O排放的主要途径[6]。
同位素法不影响微生物活性,能够有效区分N2O的来源,但同位素标记物的不均匀扩散或受同位素前体转化过程的干扰等都会影响测定结果。在自然环境中是同时存在多种生物/非生物途径产生N2O,因此,仅靠纯培养得出的SP值来判断复杂环境中N2O的来源是不准确的,所以多种同位素特征值的结合判断能够更加准确区分环境中N2O的微生物来源[45]。
除上述两种方法可以测定真菌反硝化活性外,还可以对环境样品中的真菌进行分离和纯培养再测定其反硝化活性[48]。不同的研究可以根据需求选择测定方法,同位素方法具有数据更准确可靠、对微生物不具有侵扰性等优点,但同时这种方法对实验设备的要求更高,实验操作和计算过程较为复杂。而抑制剂法可通用于各种实验室,操作简单,经济实惠,但抑制剂浓度和添加量却对微生物活性有影响,需做好预实验和实验控制,以避免其对非目标微生物的抑制作用,数据的可信度较同位素方法差。
5 不同环境中真菌反硝化的速率及贡献 自Shoun等[8]发现真菌反硝化作用以来,越来越多的研究表明反硝化真菌的种类丰富且广泛分布于不同的环境(如农田、茶园、湿地、森林、牧场、半干旱草原)中对N2O的排放有重要贡献。在不同抑制剂作用下,N2O总量减少的百分比比值作为真菌与细菌产生N2O相对贡献比(F: B),可用来判断环境中哪类微生物对N2O的产生起主导作用[38]。表 3总结了近年来在不同环境研究中的F: B比值以及真菌的反硝化速率。
表 3. 不同环境真菌与细菌的N2O相对贡献比及真菌反硝化速率 Table 3. Relative contribution ratio of fungi to bacteria and fungal denitrification rate in different environments
| Environment | Fungi/% | Bacteria/% | F: B | Fungal denitrification rate | Units | References |
| Cropland | 84±1 | 20±3 | 4.2 | 509 | μg N/(kg·h) | [42] |
| Conventional farming land | 39.9±5.4 | 38.1±5.5 | 1.2±0.5 | 3.2 | nmol N2O/(g·h) | [38] |
| Integrated crop and livestock land | 44.4±4.9 | 45.4±8.6 | 1.2±0.4 | 3.7 | ||
| Organic farming land | 40.0±8.4 | 54.2±6.7 | 0.8±0.2 | 7.0 | ||
| Plantation forestry | 50.7±4.7 | 30.5±6.9 | 2.4±0.6 | 3.3 | ||
| Abandoned field | 51.3±6.8 | 41.5±4.1 | 1.4±0.2 | 3.0 | ||
| Peat land | 81 | 31 | 2.6 | 17.1–35.9 | ng N/(g·h) | [40] |
| Forest | 50.8±11.3 | 43.7±11.8 | 1.15 | 45.0 | μg N/(kg·h) | [41] |
| Tea soil | 70 | 23 | 3.0 | 6.7 | mg N2O/(kg·h) | [50] |
| Wetland | 34.3–35.1 | 1.46–1.59 | 20.0–25.0 | 0.12–0.27 | mg N/(kg·d) | [51] |
表选项
Wei等认为施肥后土壤表面含有丰富的有机质以及有氧/缺氧条件更适合真菌生长,所以对比未施肥的土壤,施肥后的农田土壤表面发生的真菌反硝化作用是主要的N2O来源[42]。Chen等探究了真菌在不同农田土壤(常规农业田、综合作物和牲畜地、有机农业田、人工林、废弃农田)中对N2O的排放起到的作用,发现在偏酸性(pH=5.3)的人工林土壤中真菌主导N2O的产生,pH可能是影响真菌和细菌活性和生物量的关键环境因子[38]。Herold等证实真菌反硝化作用受土壤pH的影响较小,真菌比细菌能够在更广泛的土壤pH中生长,而细菌群落的反硝化潜势随pH的降低而减小[49]。Huang等的研究也表明,在高酸性(pH=3.8)的茶土中,真菌和细菌反硝化的贡献分别为70%和23%,真菌反硝化主导了茶园土壤中N2O的产生,其反硝化速率为6.7 mg N2O/(kg·h)[50]。Seo等利用SIRIN法探究了Louisiana沼泽森林湿地的沉积物在不同氧化还原电位(–200 mV,–100 mV,0 mV,+100 mV,+250 mV,+400 mV)下真菌和细菌的反硝化情况[51]。结果发现,在强还原条件(Eh < 100 mV)下,细菌反硝化占主导地位。当氧化还原电位在+250 mV(适度还原条件)和+400 mV (微氧化条件)时,即处于有氧/厌氧交界条件下,真菌与细菌反硝化的贡献值分别为34.3%–35.1%和1.46%–1.59%,真菌反硝化起到主导作用,其反硝化速率为0.12–0.27 mg N/(kg·d)。除此之外,还有真菌反硝化作用在可耕种的泥炭土以及森林中对N2O的排放均起到重要的作用,真菌对土壤N2O的排放贡献也受到科学家们越来越多的关注[40-41]。
6 展望 自然环境中的真菌反硝化过程对N2O的产生有重要作用,陆地土壤环境的真菌反硝化作用研究已有众多报道,而其他环境如海洋、地下河口等的真菌反硝化作用还鲜有报道。近期有研究表明,在印度果阿海岸(Goa)的沉积物中能分离出反硝化真菌,还有研究发现东太平洋深海沉积物中也存在反硝化真菌[52-53]。因此,研究不同环境中真菌反硝化作用介导N2O释放过程是势在必行的,未来的研究可以注重以下几个方面:(1)有必要探索更多环境中真菌反硝化作用对N2O的排放贡献;(2)真菌反硝化活性测定方法需要优化,更有效的功能基因引物需要开发:随着分子生物学的发展,引物的开发为分析土壤反硝化真菌的群落组成、丰度等提供了有效的手段,但目前的反硝化真菌功能基因引物还存在一定的缺陷,有必要开发更有效的真菌反硝化引物;(3)真菌反硝化过程机制的研究需进一步拓展,目前真菌反硝化的过程机制研究主要是基于两种真菌F. oxysporum和C. tonkinense的研究,其他反硝化真菌的作用机理还有待进一步探索研究。增进对反硝化真菌群的认识,有利于人们进一步掌握全球氮循环的情况,从而为将来控制和改善氮排放污染问题提供理论基础。
References
| [1] | Xu HF, Sheng R, Xing XY, Zhang WZ, Hou HJ, Liu Y, Qin HL, Chen CL, Wei WX. Characterization of fungal nirK-containing communities and N2O emission from fungal denitrification in arable soils. Frontiers in Microbiology, 2019, 10: 117. DOI:10.3389/fmicb.2019.00117 |
| [2] | Kuypers MMM, Marchant HK, Kartal B. The microbial nitrogen-cycling network. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [3] | Yoon S, Song BK, Phillips RL. Ecological and physiological implications of nitrogen oxide reduction pathways on greenhouse gas emissions in agroecosystems. FEMS Microbiology Ecology, 2019, 95(6): fiz066. DOI:10.1093/femsec/fiz066 |
| [4] | Tsuruta S, Takaya N, Zhang L, Shoun H, Kimura K, Hamamoto M, Nakase T. Denitrification by yeasts and occurrence of cytochrome P450nor in Trichosporon cutaneum. FEMS Microbiology Letters, 1998, 168(1): 105-110. DOI:10.1111/j.1574-6968.1998.tb13262.x |
| [5] | Phillips R, Grelet G, McMillan A, Song BK, Weir B, Palmada T, Tobias C. Fungal denitrification: Bipolaris sorokiniana exclusively denitrifies inorganic nitrogen in the presence and absence of oxygen. FEMS Microbiology Letters, 2016, 363(4): fnw007. DOI:10.1093/femsle/fnw007 |
| [6] | Wankel SD, Ziebis W, Buchwald C, Charoenpong C, de Beer D, Dentinger J, Xu ZJ, Zengler K. Evidence for fungal and chemodenitrification based N2O flux from nitrogen impacted coastal sediments. Nature Communications, 2017, 8: 15595. DOI:10.1038/ncomms15595 |
| [7] | Bollag JM, Tung G. Nitrous oxide release by soil fungi. Soil Biology & Biochemistry, 1972, 4: 271-276. |
| [8] | Shoun H, Tanimoto T. Denitrification by the fungus Fusarium oxysporum and involvement of cytochrome P450 in the respiratory nitrite reduction. The Journal of Biological Chemistry, 1991, 266(17): 11078-11082. DOI:10.1016/S0021-9258(18)99130-1 |
| [9] | Shoun H, Kim DH, Uchiyama H, Sugiyama J. Denitrification by fungi. FEMS Microbiology Letters, 1992, 94(3): 277-282. DOI:10.1111/j.1574-6968.1992.tb05331.x |
| [10] | Tanimoto T, Hatano K, Kim D, Uchiyama H, Shoun H. Co-denitrification by the denitrifying system of the fungus Fusarium oxysproum. FEMS Microbiology Letters, 1992, 93(2): 177-180. DOI:10.1111/j.1574-6968.1992.tb05086.x |
| [11] | Su F, Takaya N, Shoun H. Nitrous oxide-forming codenitrification catalyzed by cytochrome P450nor. Bioscience, Biotechnology, and Biochemistry, 2004, 68(2): 473-475. DOI:10.1271/bbb.68.473 |
| [12] | Nakahara K, Tanimoto T, Hatano K, Usuda K, Shoun H. Cytochrome P-45055A1(P-450dNIR) Acts as nitric oxide reductase employing NADH as the direct electron donor. The Journal of Biological Chemistry, 1993, 268(11): 8350-8355. DOI:10.1016/S0021-9258(18)53102-1 |
| [13] | Kobayashi M, Shoun H. The Copper-containing dissimilatory nitrite reductase involved in the denitrifying system of the fungus Fusarium oxysporum. Journal of Biological Chemistry, 1995, 270(8): 4146-4151. DOI:10.1074/jbc.270.8.4146 |
| [14] | Kobayashi M, Matsuo Y, Takimoto A, Suzuki S, Maruo F, Shoun H. Denitrification, a novel type of respiratory metabolism in fungal mitochondrion. The Journal of Biological Chemistry, 1996, 271(27): 16263-16267. DOI:10.1074/jbc.271.27.16263 |
| [15] | Mothapo N, Chen HH, Cubeta MA, Grossman JM, Fuller F, Shi W. Phylogenetic, taxonomic and functional diversity of fungal denitrifiers and associated N2O production efficacy. Soil Biology&Biochemistry, 2015, 83: 160-175. |
| [16] | Higgins SA, Welsh A, Orellana LH, Konstantinidis KT, Chee-Sanford JC, Sanford RA, Schadt CW, Loffler FE. Detection and diversity of fungal nitric oxide reductase genes (p450nor) in agricultural soils. Applied and Environmental Microbiology, 2016, 82(10): 2919-2928. DOI:10.1128/AEM.00243-16 |
| [17] | Kizawa H, Tomura D, Oda M, Fukamizu A, Hoshino T, Gotoh O, Yasui T, Shoun H. Nucleotide sequence of the unique nitrate/nitrite-inducible cytochrome P-450 cDNA from Fusarium oxysporum. The Journal of Biological Chemistry, 1991, 266(16): 10632-10637. DOI:10.1016/S0021-9258(18)99270-7 |
| [18] | Jirout J, ?imek M, Elhottová D. Fungal contribution to nitrous oxide emissions from cattle impacted soils. Chemosphere, 2013, 90(2): 565-572. DOI:10.1016/j.chemosphere.2012.08.031 |
| [19] | Mothapo N, Chen HH, Cubeta M, Shi W. Nitrous oxide producing activity of diverse fungi from distinct agroecosystems. Soil Biology & Biochemistry, 2013, 66: 94-101. |
| [20] | Jasrotia P, Green SJ, Canion A, Overholt WA, Prakash O, Wafula D, Hubbard D, Watson DB, Schadt CW, Brooks SC, Kostka JE. Watershed-scale fungal community characterization along a pH gradient in a subsurface environment cocontaminated with uranium and nitrate. Applied and Environmental Microbiology, 2014, 80(6): 1810-1820. DOI:10.1128/AEM.03423-13 |
| [21] | He GY, Xu W, Guo SS, Liu WH, Luo ZH. Diversity and denitrification capability of cultivable fungi from deep-sea sediments of Yap Trench. Journal of Applied Oceanography, 2018, 37(2): 229-240. (in Chinese) 何高阳, 徐炜, 郭双双, 刘文华, 骆祝华. 雅浦海沟深海沉积物可培养真菌多样性及其反硝化能力研究. 应用海洋学学报, 2018, 37(2): 229-240. DOI:10.3969/J.ISSN.2095-4972.2018.02.010 |
| [22] | Shoun H, Fushinobu S, Jiang L, Kim SW, Wakagi T. Fungal denitrification and nitric oxide reductase cytochrome P450nor. Philosophical Transactions of the Royal Society B Biological Sciences, 2012, 367(1593): 1186-1194. DOI:10.1098/rstb.2011.0335 |
| [23] | Uchimura H, Enjoji H, Seki T, Taguchi A, Takaya N, Shoun H. Nitrate reductase-formate dehydrogenase couple involved in the fungal denitrification by Fusarium oxysporum. Journal of Biochemistry, 2002, 131(4): 579-586. DOI:10.1093/oxfordjournals.jbchem.a003137 |
| [24] | Takaya N, Kuwazaki S, Adachi Y, Suzuki S, Kikuchi T, Nakamura H, Shiro Y, Shoun H. Hybrid respiration in the denitrifying mitochondria of Fusarium oxysporum. Journal of Biochemistry, 2003, 133(4): 461-465. DOI:10.1093/jb/mvg060 |
| [25] | Watsuji TO, Takaya N, Nakamura A, Shoun H. Denitrification of nitrate by the fungus Cylindrocarpon tonkinense. Bioscience, Biotechnology, and Biochemistry, 2003, 67(5): 1115-1120. DOI:10.1271/bbb.67.1115 |
| [26] | Zhou ZM, Takaya N, Nakamura A, Yamaguchi M, Takeo K, Shoun H. Ammonia fermentation, a novel anoxic metabolism of nitrate by fungi. The Journal of Biological Chemistry, 2002, 277(3): 1892-1896. DOI:10.1074/jbc.M109096200 |
| [27] | Kubota Y, Takaya N, Shoun H. Membrane-associated, dissimilatory nitrite reductase of the denitrifying fungus Cylindrocarpon tonkinense. Archives of Microbiology, 1999, 171(3): 210-213. DOI:10.1007/s002030050701 |
| [28] | Shoun H, Sudo Y, Seto Y, Beppu T. Purification and properties of a cytochrome P-450 of a fungus, Fusarium oxysporum. Journal of Biochemistry, 1983, 94(4): 1219-1229. DOI:10.1093/oxfordjournals.jbchem.a134467 |
| [29] | Zhou JG, Jiang Y, Liu DL. Cytochrome P450nor of fungi and denitrification. Chemistry of Life, 2002, 22(4): 308-311. (in Chinese) 周建刚, 江月, 刘德立. 真菌细胞色素P450nor与反硝化作用. 生命的化学, 2002, 22(4): 308-311. DOI:10.3969/j.issn.1000-1336.2002.04.003 |
| [30] | Zhang L, Takaya N, Kitazume T, Kondo T, Shoun H. Purification and cDNA cloning of nitric oxide reductase cytochrome P450nor (CYP55A4) from Trichosporon cutaneum. European Journal of Biochemistry, 2001, 268(11): 3198-3204. DOI:10.1046/j.1432-1327.2001.02206.x |
| [31] | Usuda K, ToritsukaN, MatsuoY, Kim DH, Shoun H. Denitrifification by the fungus Cylindrocarpon tonkinense: anaerobic cell growth and two isozyme forms of cytochrome P-450nor. Applied and Environmental Microbiology, 1995, 61(3): 883-889. DOI:10.1128/aem.61.3.883-889.1995 |
| [32] | Kim SW, Fushinobu S, Zhou S, Wakagi T, Shoun H. Eukaryotic nirK genes encoding copper-containing nitrite reductase: originating from the protomitochondrion?. Applied and Environmental Microbiology, 2009, 75(9): 2652-2658. DOI:10.1128/AEM.02536-08 |
| [33] | Huang C, He QM, Wu HD, Peng XY. Study progress on the mechanism of dissimilatory nitrate reduction in fungi. Microbiology, 2009, 36(7): 1052-1057. (in Chinese) 黄灿, 何清明, 邬红东, 彭绪亚. 真菌异化硝酸盐还原机理的研究进展. 微生物学通报, 2009, 36(7): 1052-1057. |
| [34] | Ibraim E, Wolf B, Harris E, Gasche R, Wei J, Yu LF, Kiese R, Eggleston S, Butterbach-Bahl K, Zeeman M, Tuzson B, Emmenegger L, Six J, Henne S, Mohn J. Attribution of N2O sources in a grassland soil with laser spectroscopy based isotopocule analysis. Biogeosciences, 2019, 16(16): 3247-3266. DOI:10.5194/bg-16-3247-2019 |
| [35] | Anderson JP, Domsch KH. Measurement of bacterial and fungal contributions to respiration of selected agricultural and forest soils. Canadian Journal of Microbiology, 1975, 21(3): 314-322. DOI:10.1139/m75-045 |
| [36] | Anderson JPE, Domsch KH. Quantification of bacterial and fungal contributions to soil respiration. Archiv Für Mikrobiologie, 1973, 93(2): 113-127. DOI:10.1007/BF00424942 |
| [37] | Huang Y, Long XE. Contribution of fungi to soil nitrous oxide emission and their research methods: a review. Chinese Journal of Applied Ecology, 2014, 25(4): 1213-1220. (in Chinese) 黄莹, 龙锡恩. 真菌对土壤N2O释放的贡献及其研究方法. 应用生态学报, 2014, 25(4): 1213-1220. |
| [38] | Chen HH, Mothapo NV, Shi W. The significant contribution of fungi to soil N2O production across diverse ecosystems. Applied Soil Ecology, 2014, 73: 70-77. DOI:10.1016/j.apsoil.2013.08.011 |
| [39] | Ma ST, Shan J, Yan XY. N2O emissions dominated by fungi in an intensively managed vegetable field converted from wheat-rice rotation. Applied Soil Ecology, 2017, 116: 23-29. DOI:10.1016/j.apsoil.2017.03.021 |
| [40] | Yanai Y, Toyota K, Morishita T, Takakai F, Hatano R, Limin SH, Darung U, Dohong S. Fungal N2O production in an arable peat soil in Central Kalimantan, Indonesia. Soil Science and Plant Nutrition, 2007, 53(6): 806-811. DOI:10.1111/j.1747-0765.2007.00201.x |
| [41] | Zhang W, Gao R, Chen SD, Ma HL, Gao Y, Zhu XM, Yin YF, Yang YS, Guan HL. Fungal contribution to N2O production in soil for a natural Castanopsis carlesii forest in Wuyi nature reserve, southeastern China. Journal of Subtropical Resources and Environment, 2013, 8(2): 28-34. (in Chinese) 章伟, 高人, 陈仕东, 马红亮, 高艳, 朱祥妹, 尹云锋, 杨玉盛, 官惠玲. 米槠天然林土壤真菌对N2O产生的贡献. 亚热带资源与环境学报, 2013, 8(2): 28-34. DOI:10.3969/j.issn.1673-7105.2013.02.004 |
| [42] | Wei W, Isobe K, Shiratori Y, Nishizawa T, Ohte N, Otsuka S, Senoo K. N2O emission from cropland field soil through fungal denitrification after surface applications of organic fertilizer. Soil Biology & Biochemistry, 2014, 69: 157-167. |
| [43] | Yamamoto A, Akiyama H, Nakajima Y, Hoshino YT. Estimate of bacterial and fungal N2O production processes after crop residue input and fertilizer application to an agricultural field by 15N isotopomer analysis. Soil Biology&Biochemistry, 2017, 108: 9-16. |
| [44] | Rohe L, Anderson TH, Braker G, Flessa H, Giesemann A, Lewicka-Szczebak D, Wrage-Monning N, Well R. Dual isotope and isotopomer signatures of nitrous oxide from fungal denitrification-a pure culture study. Rapid Communications in Mass Spectrometry, 2014, 28(17): 1893-1903. DOI:10.1002/rcm.6975 |
| [45] | Lin W, Fang FL, Zhang W, Ding JJ, Li YZ, Xu CY, Li QZ. A review on development of stable isotope technique in the studies of N2O formation mechanism. Chinese Journal of Applied Ecology, 2017, 28(7): 2344-2352. (in Chinese) 林伟, 房福力, 张薇, 丁军军, 李玉中, 徐春英, 李巧珍. 稳定同位素技术在土壤N2O溯源研究中的应用. 应用生态学报, 2017, 28(7): 2344-2352. |
| [46] | Sutka RL, Adams GC, Ostrom NE, Ostrom PH. Isotopologue fractionation during N2O production by fungal denitrification. Rapid Communications in Mass Spectrometry, 2008, 22(24): 3989-3996. DOI:10.1002/rcm.3820 |
| [47] | Fang YT, Koba K, Makabe A, Takahashi C, Zhu WX, Hayashi T, Hokari AA, Urakawa R, Bai E, Houlton BZ, Xi D, Zhang SS, Matsushita K, Tu Y, Liu DW, Zhu FF, Wang ZY, Zhou GY, Chen DX, Makita T, Toda H, Liu XY, Chen QS, Zhang DQ, Li YD, Yoh M. Microbial denitrification dominates nitrate losses from forest ecosystems. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(5): 1470-1474. DOI:10.1073/pnas.1416776112 |
| [48] | Lavrent'ev RB, Zaitsev SA, Sudnitsyn II, Kurakov AV. Nitrous oxide production by fungi in soils under different moisture levels. Moscow University Soil Science Bulletin, 2008, 63(4): 178-183. DOI:10.3103/S0147687408040054 |
| [49] | Herold BM, Elizabeth MB, Daniell JT. Fungal and bacterial denitrification are differently affected by long-term pH amendment and cultivation of arable soil. Soil Biology & Biochemistry, 2012, 54: 25-35. |
| [50] | Huang Y, Xiao X, Long XE. Fungal denitrification contributes significantly to N2O production in a highly acidic tea soil. Journal of Soils and Sediments, 2017, 17(6): 1599-1606. DOI:10.1007/s11368-017-1655-y |
| [51] | Seo DC, Delaune RD. Fungal and bacterial mediated denitrification in wetlands: Influence of sediment redox condition. Water Research, 2010, 44(8): 2441-2450. DOI:10.1016/j.watres.2010.01.006 |
| [52] | Cathrine SJ, Rahukumar C. Anaerobic denitrification in fungi from the coastal marine sediments off Goa, India. Mycological Research, 2009, 113: 100-109. DOI:10.1016/j.mycres.2008.08.009 |
| [53] | Wei X, Guo SS, Gong LF, He GY, Pang KL, Luo ZH. Cultivable fungal diversity in deep-sea sediment of the East Pacific Ocean. Geomicrobiology Journal, 2018, 35(9): 790-797. DOI:10.1080/01490451.2018.1473531 |
