刘浩东, 闫荣曼, 张传伦


南方科技大学海洋科学与工程系, 海洋地球古菌组学重点实验室, 广东 深圳 518055
收稿日期:2020-02-29;修回日期:2020-05-19;网络出版日期:2020-07-02
基金项目:国家自然科学基金(41906136,41530105,91851210);广东省自然科学基金(2018B030311016);科技部国家重点研发计划全球变化及应对专项(2018YFA0605800);南方科技大学深圳海洋地球古菌组学重点实验室项目(ZDSYS201802081843490)
作者简介:张传伦, 南方科技大学海洋与工程系讲席教授(2017-)。1994年获美国德州农工大学博士学位, 1994-1998年在美国橡树岭国家实验室从事博士后研究工作; 1998年起历任美国密苏里大学助理教授、美国佐治亚大学海洋系副教授、终身教授; 2008-2016年兼任同济大学海洋学院讲座教授。长期致力于生物地球化学方向的研究和人才培养, 创建了海洋地球古菌组学深圳市重点实验室。擅长整合脂类标记物、稳定同位素和分子DNA等交叉手段来研究微生物在环境及能源、地质历史演化及在全球碳氮循环中的作用; 研究课题主要包括河口及近海生态、深部海洋及洋中脊热液等极端环境地质(构造)微生物, 天然气水合物生物地球化学, 海洋和陆相古菌的生物地球化学过程及气候和环境变化指标的开发应用.
*通信作者:张传伦, Tel:+86-755-88018785;Fax:+86-755-88018785;E-mail:zhangcl@sustech.edu.cn.
摘要:海洋浮游古菌MGII是海洋表层水体中最丰富的古菌类群。自1992年被发现以来,如今依然没有被成功分离纯化。前人基于16S rRNA基因的研究认为MGII可以被分为MGIIa、MGIIb和MGIIc三个亚类。近年来,对大量的宏基因组测序数据的分析表明,MGII在分类学上属于广古菌门热源体纲下的一个目,包含MGIIa和MGIIb两个科。以前通过16S rRNA基因高通量测序结果得出的少量MGIIc,在宏基因组测序的数据中并没有找到,因此最近两年的研究认为MGII主要由MGIIa和MGIIb组成。本文综述了海洋浮游古菌MGII的丰度和多样性分布特征、潜在的生态功能、生态关系以及培养等方面的研究进展,比较了MGIIa和MGIIb的异同点,并对当前的研究热点和趋势进行了讨论和展望。
关键词:海洋浮游古菌MGII16S rRNA基因宏基因组测序培养
Advances in marine group Ⅱ archaea research
Haodong Liu, Rongman Yan, Chuanlun Zhang


Key Laboratory of Marine Archaea Geo-Omics, Department of Ocean Science and Engineering, Southern University of Science and Technology, Shenzhen 518055, Guangdong Province, China
Received: 29 February 2020; Revised: 19 May 2020; Published online: 2 July 2020
*Corresponding author: Chuanlun Zhang, Tel: +86-755-88018785; Fax: +86-755-88018785; E-mail: zhangcl@sustech.edu.cn.
Foundation item: Supported by the National Natural Science Foundation of China (41906136, 41530105, 91851210), by the Key Project of Natural Science Foundation of Guangdong Province (2018B030311016), by the Ministry of Science and Technology Award (2018YFA0605800) and by the Shenzhen Key Laboratory of Marine Archaea Geo-Omics, Southern University of Science and Technology (ZDSYS201802081843490)
Abstract: Marine Group Ⅱ (MGII) is the most abundant archaeal group in surface ocean waters. At present, no pure culture of the group has been isolated successfully since they were discovered in 1992. Analysis of the 16S rRNA genes has revealed that MGII mainly consist of two groups, MGIIa and MGIIb, which perform photoheterotrophy and potentially play an important role in marine carbon cycle. Phylogenetic classification based on the 16S rRNA gene assigned MGIIa and MGIIb as two families within an order-level lineage under Thermoplasmata. The phylogenetic position of a third group, MGIIc, is ambiguous due to the lack of their 16S rRNA gene sequences and absence in the metagenome data. This mini-review aims to provide the latest information on the distribution, abundance and diversity, metabolic capabilities and potential ecological functions, as well as efforts being made in enrichment and isotope labeling studies of MGII. Insights on future research directions are also provided.
Keywords: marine planktonic archaeamarine group Ⅱ (MGII)16S rRNA genemetagenomic sequencingcultivation
古菌是地球生物圈的重要组成部分,但是关于古菌的研究历史与真核生物和细菌相比还非常短暂。1977年,Carl Woese和George Fox发现产甲烷菌的核糖体小亚基RNA (SSU rRNA)基因序列和细菌具有极大的差别[1],进而提出以产甲烷菌为代表的“古细菌”的概念,作为生命的第三域与细菌和真核生物并列。从20世纪70年代末到90年代初,“古细菌”的研究开启了以传统的分离培养技术和分子生物学技术并重的时期。除甲烷菌外,嗜盐菌和嗜热菌等生存在极端环境中的“古细菌”也得到了较大的研究进展。到1990年,Woese等正式将“古细菌”命名为古菌[2],主要包括泉古菌门和广古菌门两大类。当时人们对古菌的认识一直和极端环境联系起来,认为古菌只存在于高盐、高温或缺氧等极端环境中,直到1992年,随着海洋浮游古菌的发现,这一观念发生了根本性改变。
1992年,Fuhrman等[3]首次报道了在太平洋100 m和500 m的水体样品中存在大量不同于极端环境中的古菌类群,认为它们是新的古菌类群。同年,DeLong[4]也发现近海水体中存在大量的新古菌类群,它们与生存于极端环境中的古菌显著不同。基于对16S rRNA基因的系统发育分析,DeLong正式提出MGI和MGII,前者成为泉古菌的一个分支,后者成为广古菌的一个分支(图 1)。在1997年和2001年,MGIII和MGIV也相继被发现[5-6],但是它们的丰度远远低于MGI和MGII。
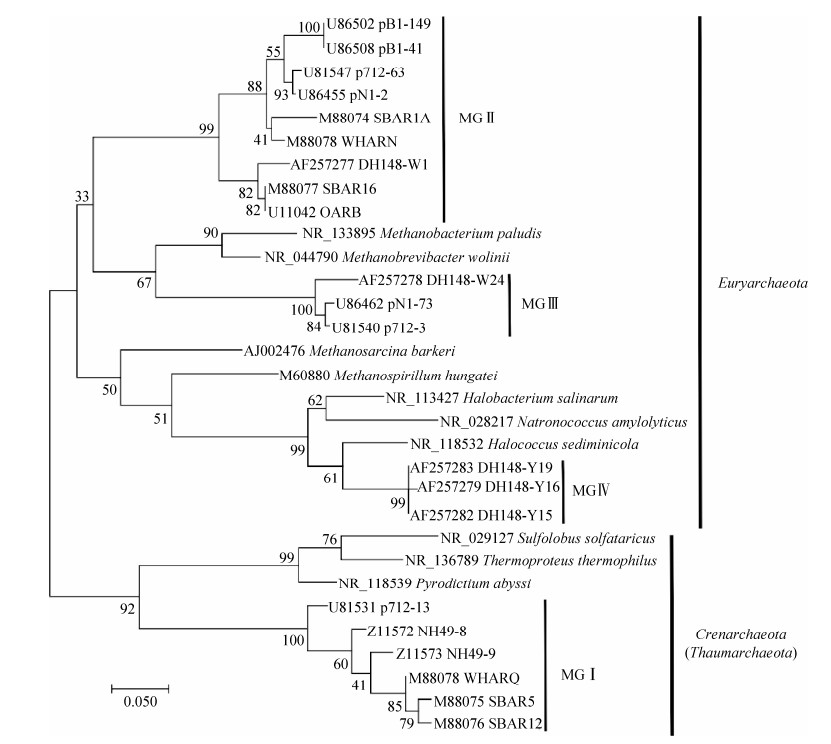 |
| 图 1 用最大似然法构建的早期研究中的古菌系统进化树(16S rRNA基因序列源自参考文献[4-6]) Figure 1 using maximum-likelihood method. (The 16S rRNA gene sequences were collected from references[4-6]) |
| 图选项 |
海洋浮游古菌占海洋水体中原核生物的三分之一以上[7],在全球碳氮循环和生态环境的动态平衡中起着重要的驱动和调节作用。它们主要由MGI、MGII、MGIII和MGIV四个类群组成。MGI逐渐被认知具有氨氧化功能[8-9]并在2005年被成功分离[10],最终被命名为奇古菌门[11]。MGII-MGIV都属于广古菌门,至今没有纯菌株分离出来[12-13]。
纯培养菌株的获得对于海洋浮游古菌的研究是一个重要的突破口,因此MGI的研究目前比其他海洋浮游古菌要深入很多[14]。比如,它们的细胞形状和大小、生理生化特性、细胞膜脂的组成等方面的研究已经具有确定的结论[10, 14-15]。MGII在海洋中同样大量存在,并在表层海洋中占海洋古菌的绝大多数,它们对碳、氮等元素循环具有重要的潜在影响[12]。但是由于缺乏纯培养菌株,关于它们的研究就相对难以深入。然而,分子生物学技术的飞速发展,特别是宏基因组技术的广泛应用,极大促进了我们对MGII的认知[16-19]。本文从以下几个方面总结了前人关于MGII的研究成果,并对当前MGII的研究热点和研究趋势进行了讨论。
1 MGII丰度和多样性的分布特征 目前大量的研究结果表明MGII几乎只分布在海洋水体环境中,在海洋表层沉积物中偶尔会检测到极少的MGII,而陆地环境中不存在MGII。因此,研究探讨MGII的分布特征主要是研究MGII在全球海洋水体中的分布特征。MGII在全球海洋中的分布具有普遍性,它们在各种海洋水体环境中都有发现,但是主要集中在海洋表层水体[12-13]。
Karner等通过DAPI染色计数法和polyFISH计数法对开阔大洋水体样品的古菌和细菌进行定量,并结合大陆架海域的研究结果,估算出全球海洋水体中约有1.3×1028个古菌细胞和3.1×1028个细菌细胞,其中大约有1.0×1028个MGI细胞[7]。当前尚无文献报道全球海洋中MGII的生物量,但是近年来的研究表明MGII广泛分布于各大洋和边缘海的表层海洋水体中[13],并且它们在温暖季节的近岸海域经常会出现短暂的季节性暴发现象,最高时可以占所有原核生物总量的40%以上[20],因此MGII也是海洋水体中含量丰富的一类古菌。
MGII丰度的整体分布规律是温暖海水比寒冷海水更丰富,近海比远海更丰富,表层海水比深层海水更丰富。在近海、河口等区域MGII呈现出非常明显的季节性变化,特别是亚热带、温带以及部分寒带的近海水体中MGII常在春夏季节大爆发。一项基于FISH定量的研究表明在地中海近岸海域中古菌的相对丰度最高时可达到微生物总量的60%以上[21]。但在寡营养的热带和亚热带开阔大洋区域MGII的季节性变化不明显,在南大洋的大部分海域MGII以及北冰洋开阔海域MGII的季节性变化也不明显。
热带、亚热带海洋的近岸海域MGII的丰度一般都比较高,在表层水或河流入海口等有机质比较丰富的海域中,它们的16S rRNA基因丰度普遍能达到108 copies/L的水平,次表层一般降到107,到深层水进一步降低[7, 18]。在开阔大洋,表层水MGII的16S rRNA基因丰度一般都在106– 107 copies/L,深层水一般降到105–106 copies/L[22]。在海水混合剧烈的海域,比如南海东北部,其深层水中也会出现丰度较高的MGII,其16S rRNA基因丰度能够达到107 copies/L,接近表层水中MGII的丰度[23]。温带海洋中MGII的分布与热带和亚热带海洋类似,但是季节性变化更明显,所以变化范围更大。在近岸海域的春夏季节MGII暴发时其16S rRNA基因丰度可达108 copies/L,最高时可接近109[24-25],比热带和亚热带的近岸海域MGII的丰度还高。而在秋冬季节,其16S rRNA基因丰度迅速降低到106–107 copies/L[26-27]。温带海洋开阔海域MGII的丰度比较低,与热带、亚热带海洋的开阔海域类似。此外,浮游态和附着态的MGII在近岸与远洋也有差异,一般来说,近岸海域浮游态的MGII会更高一些,而在远洋附着态MGII的比例增加[28]。南大洋MGII的16S rRNA基因丰度一般都在106 copies/L,而且表层水到深层水变化不大,在所有原核生物中的比例一般都不超过5%[29-30]。北冰洋表层海水中MGII的16S rRNA基因丰度一般为106–107 copies/L,个别近岸海域最高可达108,显著高于南大洋[31],但在深层水和开阔海域较低,一般都低于106– 107 copies/L,在有些站位甚至低于检测限[32-34],与南大洋类似。
MGII是海洋水体中多样性最高的古菌类群,在大部分海域其多样性都高于海洋奇古菌[20, 35-38]。MGII与细菌以及真核藻类之间的水平基因转移比海洋奇古菌与细菌和真核藻类之间的水平基因转移更加频繁,大约29.7%的MGII和23.9%的海洋奇古菌的基因是从细菌那里通过水平基因转移获得的[39-40]。频繁的水平基因转移很可能是MGII具有丰富的多样性的重要因素之一。同属于广古菌门的MGIII和MGIV的丰度和多样性都远低于MGII[6, 41]。基于16S rRNA基因的研究认为MGII主要分为MGIIa、MGIIb、MGIIc三个亚类,其中MGIIa和MGIIb的丰度和多样性均远远高于MGIIc[42-43]。
近年来基于大量关于海洋微生物宏基因组学方面的研究,发现目前MGII的MAGs (metagenome-assembled genomes)都属于MGIIa和MGIIb,而没有发现MGIIc的MAGs,因此认为MGII主要由MGIIa和MGIIb组成[17, 19, 44]。2018年Rinke等[17]对270个MGII MAGs进行了系统进化研究,认为MGII属于广古菌门热源体纲下的一个目,并将其命名为Candidatus Poseidoniales,包含MGIIa和MGIIb两个科,分别命名为Candidatus Poseidonaceae和Candidatus Thalassarchaeaceae。他们进一步将MGIIa划分为11个属,MGIIb划分为10个属。不久后,Tully在提供了一些新的MGII MAGs的基础上也对MGII进行了系统进化分析[19],他也认为MGII由MGIIa和MGIIb组成,共包含17个进化枝,其中9个属于MGIIa,8个属于MGIIb。在最新的关于MGII的宏基因组学研究中,又有更多的MGII MAGs被组装出来[44],它们在分类学上被归入了Rinke等提出的不同的属。未来需要更详尽的研究来证明MGII是否还有更多的类群尚未被发现,从而不断丰富和统一MGII在进化和分类学上的研究。
近两年已发表的论文中尚未发现属于MGIIc的MAGs,这很可能是由于它们在海洋水体中的分布范围主要集中在深层海水(图 2),而且丰度比较低等原因造成的。经过大量的文献搜集和查阅工作,我们发现2015年发表的一篇文献报道了在深层海水中存在少量MGIIc的MAGs,只是这些MAGs的完整度不高[16],而且都没有拼接出16S rRNA基因, 因此没有被引起重视。但实际分析结果表明MGII是由MGIIa、MGIIb和MGIIc组成(图 2)。由于当前所发表的文献中MGIIc的MAGs太少且质量较低,它们的进化地位、分类命名和生态功能等方面的信息有待于将来更多的对深层海水宏基因组学和宏转录组学等方面的研究工作来逐渐拓展和完善。
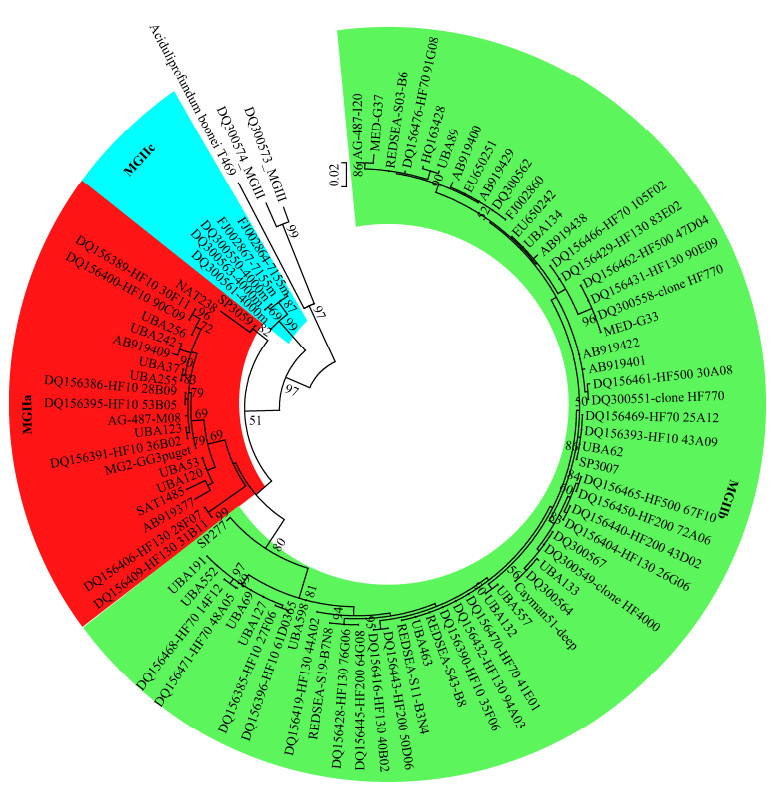 |
| 图 2 用长度大于1200 bp的16S rRNA基因序列通过最大似然法构建的MGII系统进化树 Figure 2 Phylogenetic tree of MGII based on 16S rRNA genes (> 1200 bp) using maximum-likelihood method. The bold items in different branches indicate 16S rRNA genes that were extracted from metagenome-assembled genomes of MGII. The outgroup consists of Aciduliprofundum boonei and MGIII |
| 图选项 |
我们从公共数据库NCBI中选择了长度大于1200 bp的MGII 16S rRNA基因序列,并结合从宏基因组数据拼接的MGII MAGs中提取出的16S rRNA基因序列,进行了MGII进化树的构建(图 2)。可以看出MGIIb的多样性是最高的,MGIIa的多样性次之,MGIIc的多样性最低。这和它们的分布范围是相一致的,MGIIb在表层到深层海水都有发现,MGIIa主要分布在表层海水,MGIIc则主要分布在个别海域的深层海水[23, 42]。本文作者课题组对南海东北部和马里亚纳海沟水体中古菌多样性的研究表明,不论是在边缘海还是在远洋海域,MGIIa主要分布在表层海水,而MGIIb从表层海水到深层海水都存在,只是在表层水中相对较少[12, 23]。在南海东北部从表层到3200 m的底层水体中都未发现MGIIc,而在马里亚纳海沟8000 m以深的水体中发现了少量的MGIIc。本课题组对珠江口水体中的MGII进行了丰度、多样性和宏基因组学方面的分析[18],发现富营养的珠江口区域MGII主要由MGIIa组成,并且它们主要是附着在颗粒物上,这与寡营养的南海东北部和马里亚纳海沟水体中MGIIb占主导有显著差异。对一个完整度达到93%的MGIIa的MAG进行注释后发现,珠江口MGIIa含有大量有机质降解相关酶的编码基因,比如其基因组中糖苷水解酶的编码基因比例较高,有助于它们降解利用结构复杂的糖类物质[18]。
由于含有丰富的有机质降解酶的相关编码基因,MGIIa往往在有机质丰富的温暖海水中占据主导地位,比如春夏季节的近岸海域中MGIIa的含量一般都比较高,有时伴随着浮游植物的大量增加而出现暴发式生长的现象,甚至MGIIa的单个OUT (分类操作单元)的丰度可以达到原核生物量的30%[45]。而MGIIb在有机质贫乏的寒冷的海水中占主导,比如在冬天的近岸海域[46-50]。在热带亚热带远洋的表层水中,MGIIa的丰度虽然较低,但是依然高于MGIIb。然而叶绿素最大层和深层水中MGIIb则比较多[23, 51]。由此可见MGIIa更适应温暖的富营养的海水,而MGIIb相对来说更适应寒冷的寡营养的海水。在河口区由于有机质来源比较丰富,MGIIa的丰度一般都高于MGIIb,但后者的多样性更高[52-53]。
前人研究结果表明温度是决定MGII在全球分布情况的极其重要的调控因素,因此对MGII在不同纬度海域的分布进行了归纳总结[12]。此外,离岸距离的远近决定了海水营养的贫富状态,对MGII的分布影响也比较大[28]。在河口区域由于淡水和海水的混合形成的盐度梯度也对MGII的分布具有显著的影响[18]。不同水深的MGII的限制性影响因素也不同,比如表层和次表层水体中MGII主要受温度和光合有效辐射的影响,而深层水的MGII与盐度以及表层水的初级生产力相关[20]。除以上主要因素外,其他环境因素也在一定程度上对MGII的分布产生影响,比如营养盐浓度(氮、磷、硅的无机化合物)、季节变化、海水的混合等[19, 24, 47, 54-58]。综上所述,MGII的分布特征受多种环境因素的综合影响。
2 MGII的生态功能 由于尚无纯培养菌株,目前关于MGII的生态功能方面的研究主要借助于宏基因组学、宏转录组学和宏蛋白组学的方法。在已有的纯培养的古菌中,与MGII亲缘关系最近的是一株从深海热液喷口分离的同属于广古菌门的Aciduliprofundum boonei,这是一株嗜热嗜酸能够还原硫酸盐和铁的异养古菌[59],MGII的宏基因组方面的注释就是以Aciduliprofundum boonei的基因组和新陈代谢的特性为基准进行的。
2012年,Iverson等率先拼接组装出第一个完整的MGII MAG。该MAG的基因组大小为2.06 Mb,来自于表层海水,基于16S rRNA基因的系统发育分析表明该MAG属于MGIIa[60]。2015年,Martin-Cuadrado等又拼接出了MGIIb的较为完整的MAGs[50]。基于MGIIa和MGIIb的宏基因组数据的分析,发现自养途径相关的编码基因在MGII中都不存在,而蛋白质、脂类和糖类等有机物大分子降解酶的编码基因在MGII中普遍存在,说明MGII属于异养代谢类型[12]。真光层海水中的MGII大多含有能够利用光能的视紫红质的编码基因[17, 39],具有利用光能生成ATP的潜力但是不具备光合作用的潜力,这部分MGII属于光能异养代谢型。深层海水中的MGII都不含有视紫红质基因[16-17],没有利用光能的能力,但与浅层水中含有视紫红质编码基因的MGII相比,深层海水中的MGII含有更多与氨基酸、糖类和脂类的转运和代谢相关的基因[40, 61]。
近年来的研究结果表明,MGIIa和MGIIb在代谢途径上存在显著的相同点和不同点(图 3)。比如宏基因组学的分析表明,二者都含有较多大分子肽酶、脂类降解酶、糖基转移酶和糖苷水解酶等大分子代谢相关酶的编码基因,也含有不少简单糖类、寡肽、二肽、氨基酸等小分子有机质跨膜转运蛋白的编码基因,它们都具有三羧酸循环和磷酸戊糖途径所需要的酶的编码基因[17, 19, 44, 62]。富集培养的结果表明浮游植物能够刺激MGII的生长[28]。稳定同位素标记的培养结果表明MGII能够吸收浮游植物产生的蛋白质[63]。
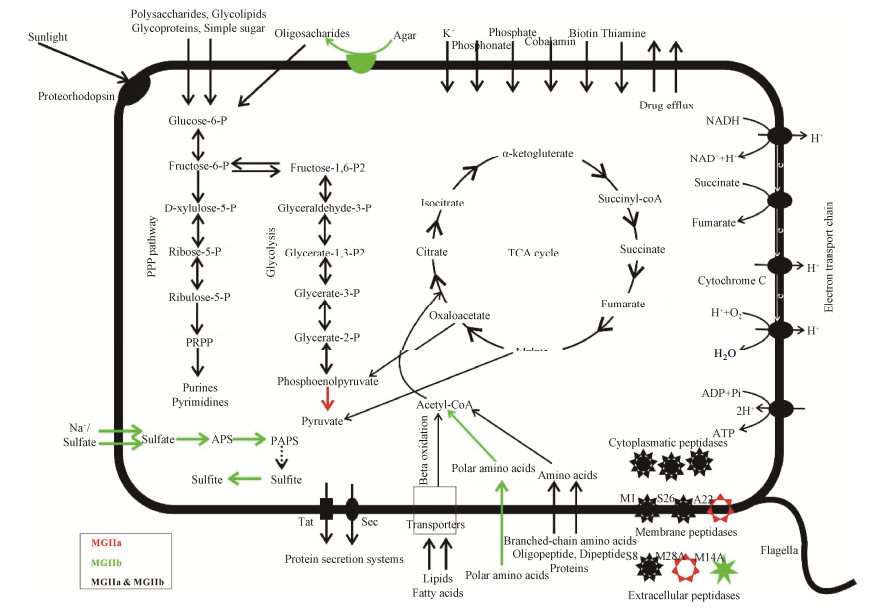 |
| 图 3 MGIIa和MGIIb的代谢潜能 Figure 3 The potential metabolism of MGIIa and MGIIb. Components in red and blue indicate the function/ encoding genes that are only or mainly present in MGIIa and MGIIb, respectively. Components in black indicate the function/encoding genes that are present in both MGIIa and MGIIb. The dotted line indicates the related encoding gene that is not detected. This figure was produced according to references [12, 16, 44] |
| 图选项 |
MGIIa和MGIIb在代谢上的不同点主要体现在相关代谢途径关键酶是否存在以及代谢底物的偏好性上。比如在糖酵解途径的最后一步,丙酮酸激酶催化磷酸烯醇式丙酮酸和ADP生成丙酮酸和ATP,所有的MGIIa都含有该酶的编码基因,而绝大多数MGIIb则没有[44],这表明MGIIa在单糖的代谢能力上比MGIIb有优势。春夏季节的近海海水中MGII以MGIIa为主,它们含有丰富的糖类转移和代谢相关的酶,近海中浮游态MGII的相对丰度高于远洋,而远洋海水中附着态的MGII的相对丰度更高一些[64],可能是由于近海MGIIa对溶解有机碳的利用能力比远洋MGIIb更强,这与MGIIa具备完整的糖酵解途径而MGIIb大多缺少完整的糖酵解途径是一致的。
在氨基酸的代谢能力上,MGIIb比MGIIa有优势。比如MGIIb除了能和MGIIa一样具有支链氨基酸的转运和代谢相关酶的编码基因外,MGIIb还具有MGIIa中不存在的极性氨基酸的转运和代谢相关酶的编码基因[44]。此外,MGIIb具有吸收和还原硫酸盐相关酶的编码基因,这有利于它们在还原态的硫化合物浓度很低的冬季海水中进行生存和繁殖,而MGIIa中则没有这些酶的编码基因[44]。硫酸盐还原能力的存在很可能是MGIIb主导冬季海水中MGII群落结构的一个重要原因。
除了在对简单有机质的利用上MGIIa偏好利用单糖MGIIb偏好氨基酸之外,在复杂有机质的利用上二者也有一些区别,这主要体现在MGIIb比MGIIa在利用难降解复杂有机质方面的潜力更大,比如MGIIb含有降解琼脂、几丁质等复杂有机质相关酶的编码基因,而MGIIa中则很少见或不存在。这可能也是为什么MGIIa主要分布在表层水,而MGIIb在深层水中远多于MGIIa的原因之一[16, 50]。无论是表层水中的MGIIa,还是深层水中的MGIIb,它们大多具有鞭毛相关的编码基因[19],这表明它们具有主动游动寻找食物的潜能。而在叶绿素最大层的MGIIb则不含有鞭毛结构,但它们具有较多的降解各种有机化合物相关酶的编码基因,包括降解琼脂相关酶的编码基因[50]。
宏基因组数据表明,附着在颗粒物上的MGII大多都富含一种依赖TonB转运体的编码基因,这些转运体能够将外界的大分子有机质转移到细胞内[28]。这些附着态的MGII基本上也都含有鞭毛和表面附着蛋白的编码基因[60],这表明它们具备运动和附着在颗粒物上的潜能。宏转录组数据表明MGII确实具有降解大分子有机质的活性,尤其在深层水中,对大分子有机质的降解活性很高[16]。深海MGII的转录组中包含丰富的α-甘露糖苷酶(GH38)、支链淀粉酶(GH57)、4-α-葡聚糖转移酶(GH77)、几丁质酶(GH18/CBM5和GH20/CBM5)等糖类代谢酶的相关转录本,并且具有20多种降解蛋白质的胞外肽酶的编码基因及其对应的转录本[16]。这说明MGII在深层海水中的新陈代谢是很活跃的,对大分子有机质有较强的降解能力。在地中海西北部的中深层海洋中,MGII所吸收利用的浮游植物产生的胞外聚合物占所有被吸收利用的胞外聚合物的50%以上[65],这表明MGII在中深层海洋中对大分子有机质的利用具有重要的作用。除了在降解大分子有机质方面具有重要的贡献,宏蛋白组学的研究也表明MGII在深海中能够转移和吸收肽类和氨基酸,表明它们对小分子有机质的降解方面也具有重要作用[66]。标记培养试验证明中层海洋和深层海洋中的MGII能够吸收利用亮氨酸和碳酸氢盐[67],虽然目前尚未知道MGII利用无机碳酸氢盐的方式。
很多证据表明MGII是需氧微生物,它们大多都能够通过氧化磷酸化获取能量,进行有氧呼吸[12, 17]。但是也有部分证据表明一些MGII在氧气含量较低的环境中也可以生存,也能够进行厌氧呼吸,属于兼性厌氧微生物。比如它们含有琥珀酸脱氢酶、延胡索酸还原酶、二甲基亚砜或二硫化物还原酶[68-69],属于MGIIb的2个属O3和Q1含有硝酸盐还原酶,这些证据表明在氧气浓度极低或者无氧的条件下,一些MGII可以利用琥珀酸、延胡索酸、二甲基亚砜、二硫化物、硝酸盐等物质作为电子受体进行厌氧呼吸。
3 MGII与其他生物的互作关系 MGII分布广泛,在近海的丰度非常高,与其他类群的海洋生物之间存在密切的互作关系。作为数量巨大的降解者,MGII是海洋生态系统中的重要组成部分,它们对维护海洋生态系统的平衡,促进全球海洋碳氮硫等元素循环方面具有重要意义。
从宏基因组学和宏转录组学的研究结果已经得知MGII具备附着在颗粒物和降解大分子有机质的潜力,这也与它们常常在海洋藻类暴发的同时或随后也出现暴发性生长的现象相一致[45, 47-48]。这些现象和研究结果表明了MGII与浮游植物之间存在非常密切的关系。基于分子生态学和相关性分析研究表明,MGII与一些海洋浮游藻类之间存在普遍的关联性[70]。比如在三月下旬的圣佩德罗边缘海出现了球形棕囊藻的暴发,同时引起了MGII的大量增殖[45],二者的丰度随时间的变化存在显著的相关性。在圣巴巴拉海峡对浮游古菌的连续观测表明MGII的间歇性暴发跟叶绿素a相关联[46]。加利福尼亚湾中的MGII与叶绿素a的丰度之间也存在显著的相关性[28]。从珠江口到南海的海域中真核藻类的暴发常常会引起MGII的暴发[18]。春季时MGII在北大西洋东部的北海近岸水域中的暴发有时甚至能达到所有细胞总数的30%以上,占古菌细胞总数的90%以上,这也跟浮游植物的大量增殖有密切关系[24]。
MGII不仅与真核藻类之间存在广泛而密切的生态关系,和原核藻类之间的关系可能也比较紧密。比如在南海的吕宋海峡附近的海域,MGII的分布和原绿球藻具有相似的特征[23, 71]。MGII与自养的MGI在深层水中也存在较为显著的相关性[20]。基于16S rRNA基因相对丰度的研究表明MGII与异养细菌之间也存在潜在的生态关系,这些细菌类群主要包括SAR11、SAR406、SAR324、SAR86、SAR202等类群以及拟杆菌门和绿弯菌门的部分类群,MGII与这些异养细菌的丰度存在显著相关性。而且丰度较高的MGII OTU与这些细菌中的大多数OTU都以正相关为主,但与SAR86和SAR406的部分OTU也表现出了负相关性[20, 58, 72-73]。
MGII与光能自养的真核藻类和原核藻类之间存在密切的生态关系很可能是由于需要利用它们产生的有机质。此外,根据现阶段的研究成果推测,MGII可能无法独自完成生长繁殖和新陈代谢活动,而必须依赖其他的海洋生物,因为MGIIa和MGIIb的基因组中不都含有钴胺素(维生素B12)和生物素(维生素B7)这两种重要的生长因子合成酶的编码基因,而这两种物质是它们进行生命活动所必需的[13, 60]。但与MGII具有显著相关性的原绿球藻和MGI都具有合成这两种物质的能力[74-77]。MGII与异养细菌的相关性很可能是由于MGII和这些异养细菌都需要从藻类获取有机质,在这一点上,MGII与异养细菌是竞争的关系。而当有机质比较丰富的时候,MGII对一些能够产生钴胺素和生物素的异养细菌则可能存在一定的偏利共生关系。因为MGII与一些异养细菌有时表现为正相关的关系,有时表现为负相关的关系[20]。
虽然目前尚无MGII的纯培养菌株,但是借助于宏基因组技术,MGII的相关病毒的研究也已经开展。比如在海洋中广泛分布的双链DNA病毒Magroviruses很可能就是以MGII为主要的宿主[78],这种有尾噬菌体具有来自于古菌的几乎完整的复制元件,其基因组大小介于65–100 kb。完整的MGII病毒MAGs的注释信息显示它们含有衣壳蛋白、门户蛋白、末端酶和DNA聚合酶的编码基因。通过使用不同蛋白的序列进行系统进化分析,可以将Magroviruses分成3类,分别是Group A、B和C[78]。对多种蛋白进行进化分析的结果表明Group A和B是在进化上相对稳定的类群,它们与嗜盐古菌的病毒Haloviruses具有一定的亲缘关系。而Group C很可能是进化速度相对比较快一个类群,因为基于不同的蛋白进化分析结果显示它们往往处于不同的进化位置。Group A和B中都存在类似DnaQ的核酸外切酶的编码基因,而Group C中则不存在。该酶与病毒基因组在复制过程中的勘校功能密切相关,有助于降低病毒的突变率,也许这就是为什么Group A和B比Group C在进化上更稳定的一个重要因素。值得一提的是,基于序列相似度分析的结果表明该酶的编码基因很可能是从细菌基因组获得的,这意味着Magroviruses的基因大部分都来自于MGII,但也存在从细菌基因组中获取部分基因的可能性[78]。除了上述3个类群外,还有1个与MGII病毒有关的类群被暂时定为Group D,但是目前尚未组装出属于该类群的完整基因组[78]。在北太平洋的东部热带海域MGII丰度比较高的表层海水中发现一些新的古菌病毒,虽然无法确定它们的宿主,但是由于该海域中的古菌类群主要是MGII,所以这些新发现的噬菌体的宿主很有可能是MGII[79]。此外,在全球其他很多海域也发现了数十个MGII病毒的不完整的基因组序列,其中有些病毒基因组和Magroviruses的基因组是相同的[80]。以上这些研究结果主要是关于表层海水中MGII相关病毒的宏基因组学研究,深层海水中的MGII相关病毒是否具有相似的特征尚不清楚。与MGII在全球海洋表层水中的广泛分布相似,Magroviruses在全球的表层海水中也具有广泛的分布,它们的丰度仅次于SAR11的噬菌体和蓝藻噬菌体[78]。在大于0.2 μm孔径滤膜上的Magroviruses丰度可以忽略不计,它们绝大多数都是从0.2 μm孔径滤膜过滤后的海水中收集的[78]。这说明Magroviruses能够裂解MGII细胞,MGII在暴发性增长后其丰度迅速降低很可能就是Magroviruses对MGII的裂解造成的。需要注意的是,上述研究都是基于对基因组的分析而得出的结论。要直接证明Magroviruses与MGII的病毒宿主关系还有待于MGII纯菌株的成功分离。
4 MGII的培养 传统的培养方法在MGII研究中的应用非常少。这主要是因为在海水中广泛分布的MGII的培养极其困难,与MGI通过氧化铵盐获取能量的自养生活方式不同,MGII通过降解有机质进行异养生活方式,而海洋水体中有大量的细菌也进行类似的生活方式,比如属于变形菌的SAR11和SAR86[13]。它们都是很难分离培养的海洋异养微生物,目前也只获得了SAR11的纯菌株[81],而MGII和SAR86的分离培养依然是当今海洋微生物学的难点。
至今,已报道的关于MGII的培养试验主要是同位素标记培养和共培养。用3H标记的亮氨酸作为底物培养边缘海表层海水中的微生物实验表明,原位海水中平均35% (6%–61%)的MGII具有吸收利用亮氨酸的活性,尤其在3月份,具有亮氨酸代谢活性的MGII的比例达到最高,比具有亮氨酸代谢活性的细菌的比例还高[25]。分别用3H标记的亮氨酸和用14C标记的碳酸氢盐作为底物培养中深层海水的微生物实验表明,在中深层海水中MGII能够利用无机碳和有机碳[67]。虽然它们的生长速率低于细菌,具有吸收利用亮氨酸活性的MGII的比例也低于细菌,但是具有吸收利用碳酸氢盐活性的MGII的比例高于细菌。具有吸收亮氨酸活性的MGII高于MGI,在200–2000 m的水层中具有吸收利用碳酸氢盐的MGII的比例也高于MGI,但是在3000 m水深则低于MGI,对亮氨酸和碳酸氢盐具有吸收利用活性的MGII所占比例随着深度的增加而降低[67]。
Orsi等[63]用13C标记的碳酸氢钠和15N标记的硝酸钠作为底物培养一种名为Micromonas pusilla的绿藻,然后从该藻提取标记的蛋白质对原位海水中的微生物进行培养,再通过DNA-SIP (DNA稳定同位素探针)的方法研究海水中的微生物对该种藻类蛋白的利用。结果表明,浮游态微生物中吸收利用该藻类蛋白质的能力较强的主要是玫瑰杆菌、SAR86和MGII。虽然测序结果中没有检测到附着态MGII能够吸收利用这些藻类蛋白质,但是qPCR的结果表明附着态MGII也具有吸收利用藻类蛋白质的能力,只是吸收利用蛋白质的能力没有浮游态MGII强[63]。除了用同位素标记后的藻类蛋白质培养海水中的微生物来检测它们对蛋白质的吸收利用情况,Orsi等还做了藻类与海洋微生物共培养的实验。他们将Micromonas pusilla加入海水样品中对微生物进行培养,72 h后发现浮游态MGII增加了5倍,它们每天的净增长率达到0.44–0.67,只稍微低于海洋中浮游细菌的净增长率[28],却比海水中自然状态下所有广古菌的净增长率高一个数量级[67]。浮游态MGII在藻类存在的情况下快速生长可能跟它们含有大量的ABC转运器有关,因为ABC转运器能够帮助浮游态MGII迅速获取浮游藻类产生的易降解的溶解有机质,从而使得它们的数量远远超过附着态MGII。另外,附着在0.8–3.0 μm悬浮颗粒有机质(POM)上的MGII在加入浮游动物Salpa sp.后也有显著增加,可能是由于它们能够利用浮游动物排泄的POM。但是附着在直径大于3 μm颗粒物上的MGII却没有显著增加,说明附着态MGII的代谢底物和浮游态MGII可能有所不同[28]。
以上研究报道了MGII的同位素标记培养实验和富集培养实验,证明了MGII可以吸收利用碳酸氢盐、氨基酸和藻类蛋白质。近年来,本文作者课题组进行了大量的MGII富集培养实验,尝试了不同的有机质作为底物对原位海水中的MGII进行培养,比如氨基酸、葡萄糖、丙酮酸钠、绿藻藻粉等。但是上述有机底物大多没有对MGII的生长起到非常明显的刺激作用。近期我们尝试用球形棕囊藻和MGII共培养的方法对原位海水中的MGII进行富集培养,发现经过短暂(1–2 d)的培养后MGII的丰度增加得非常快,不同试验组中MGII的丰度可增加6–10倍。但随后MGII的丰度迅速降低。据此我们认为球形棕囊藻对MGII生长的刺激作用是一个快速而短暂的过程。后续工作还要摸索更多条件以找到球形棕囊藻促进MGII生长的主要因素。
综合本课题组的研究进展和国际同行所发表的文献来看,至今为止MGII的富集培养实验尚未取得重大突破,依然还没有可传代培养的MGII富集培养物的相关报道,分离纯化更是当今研究MGII的热点和难点。虽然借助于宏基因组学技术已经取得了大量的MGII基因相关的信息,但是其生理生化和新陈代谢方面的直接证据依然有待于MGII纯培养菌株的获得才能够验证。关于MGII的膜脂中是否含有GDGT (甘油二烷基甘油四醚)以及它们是否对重建古海洋海水温度的指标TEX86具有影响一直没有定论[22, 82],主要是因为目前尚未获得MGII的纯培养菌株,无法给出最直接的证据。
5 讨论和展望 从MGII的发现到现在近30年的研究历程中,关于MGII的丰度、多样性、生态功能、生态关系等方面的研究取得了长足的进展。然而这些研究大多数是国际上报道的,与国际同行相比,国内对MGII的研究还非常少,比如近30年来与MGII研究相关的中文报道仅有少数几篇[83-87],而且这些报道主要是基于16S rRNA基因研究中国近海海域中微生物群落组成和多样性,在宏基因组方面关于MGII的研究还有欠缺,在富集培养方面目前还处于探索阶段。鉴于MGII在全球海洋的分布极其广泛而且数量庞大,对全球碳循环和气候变化具有重要的潜在影响,因此国内在这方面的研究很有必要深入下去。结合国内外关于MGII的研究进展,我们对MGII的认知依然还存在很大的不足,主要体现在以下几个方面。
(1) MGII在全球海洋的生物量是多少?我们根据前人的研究结果[7]推导出全球海洋中大约含有3×1027个MGII细胞,但是该研究结果没有考虑到近岸海域MGII在春夏季节的大暴发,因此我们认为这个结果低估了MGII的生物量。准确的MGII生物量估算需要考虑边缘海和大洋的差异,还要考虑不同季节的差异,有待于将来在更细致和全面的研究工作的基础上来解决这一问题。
(2) MGII的生活方式是什么?根据宏基因组学、宏转录组学和标记培养的实验结果认为MGII是异养或光能异养微生物[12-13, 63],但是同位素标记培养实验的结果发现很高比例的MGII具有吸收碳酸氢盐的活性[67],因此MGII中的这些类群是不是混合营养型还不得而知。
(3) MGII的起源和演化进程是怎样的?由于缺少MGII的纯培养菌株和化石证据,所以还无法确定MGII的起源和演化问题。目前和MGII亲缘关系最近的已经成功分离培养的古菌是Aciduliprofundum boonei,它是从深海热液喷口中分离出来的一株嗜热嗜酸的异养古菌[88]。与MGII同属于广古菌门的MGIII和MGIV也大多分布在深海中。根据长度大于1200 bp的16S rRNA基因构建的系统进化树,我们发现MGIIc位于树的根部,而且它们都来自于深海。因此MGIIc是否是MGII最早的类群以及它们是否起源于海底热液喷口呢?这需要更多的证据来加以论证。
(4) MGII在生物地球化学循环过程中的重要作用是如何体现的?MGII的膜脂对海洋古温度指标TEX86的影响一直存在争议,包括最新在不同海域的研究结果[89-90]。另外,异养古菌是微型生物碳泵(MCP)的组成部分,但是它们究竟具有多大贡献目前尚未得知。MGII能降解有机质已经得到了确切的证据,它们基因组中有很多有机质降解相关酶的编码基因[12-13, 16]。但是它们吸收碳酸氢盐后是通过哪些酶或者哪些途径利用的都尚不清楚。
(5) MGII难以分离培养的原因是什么?现在很多关于MGII的生理生化以及有机地化方面的关键科学问题都需要MGII的纯培养菌株来揭示答案。可是MGII难培养的决定因素仍不得而知,根据宏基因组提供的信息是否能够找到MGII培养实验的突破点,这些都是当前和将来一段时间内亟待解决的重要而又非常具有挑战性的问题。
References
| [1] | Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain:the primary kingdoms. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(11): 5088-5090. DOI:10.1073/pnas.74.11.5088 |
| [2] | Woese CR, Kandler O, Wheelis ML. Towards a natural system of organisms:proposal for the domains Archaea, Bacteria, and Eucarya. Proceedings of the National Academy of Sciences of the United States of America, 1990, 87(12): 4576-4579. DOI:10.1073/pnas.87.12.4576 |
| [3] | Fuhrman JA, McCallum K, Davis AA. Novel major archaebacterial group from marine plankton. Nature, 1992, 356(6365): 148-149. DOI:10.1038/356148a0 |
| [4] | DeLong EF. Archaea in coastal marine environments. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89(12): 5685-5689. |
| [5] | Fuhrman JA, Davis AA. Widespread Archaea and novel Bacteria from the deep sea as shown by 16S rRNA gene sequences. Marine Ecology Progress Series, 1997, 150: 275-285. DOI:10.3354/meps150275 |
| [6] | López-García P, Moreira D, López-López A, Rodríguez-Valera F. A novel haloarchaeal-related lineage is widely distributed in deep oceanic regions. Environmental Microbiology, 2001, 3(1): 72-78. DOI:10.1046/j.1462-2920.2001.00162.x |
| [7] | Karner MB, DeLong EF, Karl DM. Archaeal dominance in the mesopelagic zone of the Pacific Ocean. Nature, 2001, 409(6819): 507-510. DOI:10.1038/35054051 |
| [8] | Venter JC, Remington K, Heidelberg JF, Halpern AL, Rusch D, Eisen JA, Wu DY, Paulsen I, Nelson KE, Nelson W, Fouts DE, Levy S, Knap AH, Lomas MW, Nealson K, White O, Peterson J, Hoffman J, Parsons R, Baden-Tillson H, Pfannkoch C, Rogers YH, Smith HO. Environmental genome shotgun sequencing of the Sargasso Sea. Science, 2004, 304(5667): 66-74. DOI:10.1126/science.1093857 |
| [9] | Schleper C, Jurgens G, Jonuscheit M. Genomic studies of uncultivated archaea. Nature Reviews Microbiology, 2005, 3(6): 479-488. DOI:10.1038/nrmicro1159 |
| [10] | K?nneke M, Bernhard AE, de la Torre JR, Walker CB, Waterbury JB, Stahl DA. Isolation of an autotrophic ammonia-oxidizing marine archaeon. Nature, 2005, 437(7058): 543-546. DOI:10.1038/nature03911 |
| [11] | Brochier-Armanet C, Boussau B, Gribaldo S, Forterre P. Mesophilic Crenarchaeota:proposal for a third archaeal phylum, the Thaumarchaeota. Nature Reviews Microbiology, 2008, 6(3): 245-252. DOI:10.1038/nrmicro1852 |
| [12] | Zhang CL, Xie W, Martin-Cuadrado AB, Rodriguez-Valera F. Marine Group Ⅱ Archaea, potentially important players in the global ocean carbon cycle. Frontiers in Microbiology, 2015, 6: 1108. |
| [13] | Santoro AE, Richter RA, Dupont CL. Planktonic marine archaea. Annual Review of Marine Science, 2019, 11: 131-158. DOI:10.1146/annurev-marine-121916-063141 |
| [14] | Stahl DA, de la Torre JR. Physiology and diversity of ammonia-oxidizing Archaea. Annual Review of Microbiology, 2012, 66: 83-101. DOI:10.1146/annurev-micro-092611-150128 |
| [15] | Elling FJ, K?nneke M, Lipp JS, Becker KW, Gagen EJ, Hinrichs KU. Effects of growth phase on the membrane lipid composition of the thaumarchaeon Nitrosopumilus maritimus and their implications for archaeal lipid distributions in the marine environment. Geochimica et Cosmochimica Acta, 2014, 141: 579-597. DOI:10.1016/j.gca.2014.07.005 |
| [16] | Li M, Baker BJ, Anantharaman K, Jain S, Breier JA, Dick GJ. Genomic and transcriptomic evidence for scavenging of diverse organic compounds by widespread deep-sea archaea. Nature Communications, 2015, 6: 8933. DOI:10.1038/ncomms9933 |
| [17] | Rinke C, Rubino F, Messer LF, Youssef N, Parks DH, Chuvochina M, Brown M, Jeffries T, Tyson GW, Seymour JR, Hugenholtz P. A phylogenomic and ecological analysis of the globally abundant Marine Group Ⅱ archaea (Ca. Poseidoniales ord. nov.). The ISME Journal, 2019, 13(3): 663-675. DOI:10.1038/s41396-018-0282-y |
| [18] | Xie W, Luo HW, Murugapiran SK, Dodsworth JA, Chen SZ, Sun Y, Hedlund BP, Wang P, Fang HY, Deng MH, Zhang CL. Localized high abundance of Marine Group Ⅱ archaea in the subtropical Pearl River Estuary:implications for their niche adaptation. Environmental Microbiology, 2018, 20(2): 734-754. DOI:10.1111/1462-2920.14004 |
| [19] | Tully BJ. Metabolic diversity within the globally abundant Marine Group Ⅱ Euryarchaea offers insight into ecological patterns. Nature Communications, 2019, 10(1): 271. DOI:10.1038/s41467-018-07840-4 |
| [20] | Parada AE, Fuhrman JA. Marine archaeal dynamics and interactions with the microbial community over 5 years from surface to seafloor. The ISME Journal, 2017, 11(11): 2510-2525. DOI:10.1038/ismej.2017.104 |
| [21] | Fuhrman JA, Ouverney CC. Marine microbial diversity studied via 16S rRNA sequences:cloning results from coastal waters and counting of native archaea with fluorescent single cell probes. Aquatic Ecology, 1998, 32(1): 3-15. DOI:10.1023/A:1009974817127 |
| [22] | Lincoln SA, Wai B, Eppley JM, Church MJ, Summons RE, DeLong EF. Planktonic Euryarchaeota are a significant source of archaeal tetraether lipids in the ocean. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(27): 9858-9863. |
| [23] | Liu HD, Zhang CL, Yang CY, Chen SZ, Cao ZW, Zhang ZW, Tian JW. Marine Group Ⅱ dominates planktonic Archaea in water column of the Northeastern South China Sea. Frontiers in Microbiology, 2017, 8: 1098. DOI:10.3389/fmicb.2017.01098 |
| [24] | Pernthaler A, Preston CM, Pernthaler J, DeLong EF, Amann R. Comparison of fluorescently labeled oligonucleotide and polynucleotide probes for the detection of pelagic marine bacteria and Archaea. Applied and Environmental Microbiology, 2002, 68(2): 661-667. |
| [25] | Alderkamp AC, Sintes E, Herndl GJ. Abundance and activity of major groups of prokaryotic plankton in the coastal North Sea during spring and summer. Aquatic Microbial Ecology, 2006, 45(3): 237-246. |
| [26] | DeLong EF, Taylor LT, Marsh TL, Preston CM. Visualization and enumeration of marine planktonic Archaea and bacteria by using polyribonucleotide probes and fluorescent in situ hybridization. Applied and Environmental Microbiology, 1999, 65(12): 5554-5563. DOI:10.1128/AEM.65.12.5554-5563.1999 |
| [27] | Mincer TJ, Church MJ, Taylor LT, Preston C, Karl DM, DeLong EF. Quantitative distribution of presumptive archaeal and bacterial nitrifiers in Monterey Bay and the North Pacific Subtropical Gyre. Environmental Microbiology, 2007, 9(5): 1162-1175. DOI:10.1111/j.1462-2920.2007.01239.x |
| [28] | Orsi WD, Smith JM, Wilcox HM, Swalwell JE, Carini P, Worden AZ, Santoro AE. Ecophysiology of uncultivated marine euryarchaea is linked to particulate organic matter. The ISME Journal, 2015, 9(8): 1747-1763. |
| [29] | Simon M, Gl?ckner FO, Amann R. Different community structure and temperature optima of heterotrophic picoplankton in various regions of the Southern Ocean. Aquatic Microbial Ecology, 1999, 18(3): 275-284. |
| [30] | Church MJ, DeLong EF, Ducklow HW, Karner MB, Preston CM, Karl DM. Abundance and distribution of planktonic Archaea and Bacteria in the waters west of the Antarctic Peninsula. Limnology and Oceanography, 2003, 48(5): 1893-1902. DOI:10.4319/lo.2003.48.5.1893 |
| [31] | Amano-Sato C, Akiyama S, Uchida M, Shimada K, Utsumi M. Archaeal distribution and abundance in water masses of the Arctic Ocean, Pacific sector. Aquatic Microbial Ecology, 2013, 69(2): 101-112. DOI:10.3354/ame01624 |
| [32] | Kirchman DL, Elifantz H, Dittel AI, Malmstrom RR, Cottrell MT. Standing stocks and activity of Archaea and Bacteria in the western Arctic Ocean. Limnology and Oceanography, 2007, 52(2): 495-507. |
| [33] | Alonso-Sáez L, Sánchez O, Gasol JM, Balagué V, Pedrós-Alio C. Winter-to-summer changes in the composition and single-cell activity of near-surface Arctic prokaryotes. Environmental Microbiology, 2008, 10(9): 2444-2454. DOI:10.1111/j.1462-2920.2008.01674.x |
| [34] | Galand PE, Lovejoy C, Pouliot J, Vincent WF. Heterogeneous archaeal communities in the particle-rich environment of an arctic shelf ecosystem. Journal of Marine Systems, 2008, 74(3/4): 774-782. |
| [35] | Massana R, DeLong EF, Pedrós-Alió C. A few cosmopolitan phylotypes dominate planktonic archaeal assemblages in widely different oceanic provinces. Applied and Environmental Microbiology, 2000, 66(5): 1777-1787. DOI:10.1128/AEM.66.5.1777-1787.2000 |
| [36] | Bano N, Ruffin S, Ransom B, Hollibaugh JT. Phylogenetic composition of Arctic Ocean archaeal assemblages and comparison with Antarctic assemblages. Applied and Environmental Microbiology, 2004, 70(2): 781-789. DOI:10.1128/AEM.70.2.781-789.2004 |
| [37] | Xia XM, Guo W, Liu HB. Dynamics of the bacterial and archaeal communities in the Northern South China Sea revealed by 454 pyrosequencing of the 16S rRNA gene. Deep Sea Research Part Ⅱ:Topical Studies in Oceanography, 2015, 117: 97-107. DOI:10.1016/j.dsr2.2015.05.016 |
| [38] | Pereira O, Hochart C, Auguet JC, Debroas D, Galand PE. Genomic ecology of Marine Group Ⅱ, the most common marine planktonic Archaea across the surface ocean. MicrobiologyOpen, 2019, 8(9): e00852. |
| [39] | Frigaard NU, Martinez A, Mincer TJ, DeLong EF. Proteorhodopsin lateral gene transfer between marine planktonic Bacteria and Archaea. Nature, 2006, 439(7078): 847-850. DOI:10.1038/nature04435 |
| [40] | Deschamps P, Zivanovic Y, Moreira D, Rodriguez-Valera F, López-García P. Pangenome evidence for extensive interdomain horizontal transfer affecting lineage core and shell genes in uncultured planktonic thaumarchaeota and euryarchaeota. Genome Biology and Evolution, 2014, 6(7): 1549-1563. DOI:10.1093/gbe/evu127 |
| [41] | López-García P, López-López A, Moreira D, Rodríguez-Valera F. Diversity of free-living prokaryotes from a deep-sea site at the Antarctic Polar Front. FEMS Microbiology Ecology, 2001, 36(2/3): 193-202. |
| [42] | Galand PE, Casamayor EO, Kirchman DL, Potvin M, Lovejoy C. Unique archaeal assemblages in the Arctic Ocean unveiled by massively parallel tag sequencing. The ISME Journal, 2009, 3(7): 860-869. DOI:10.1038/ismej.2009.23 |
| [43] | Xia XM, Guo W, Liu HB. Basin scale variation on the composition and diversity of Archaea in the Pacific Ocean. Frontiers in Microbiology, 2017, 8: 2057. DOI:10.3389/fmicb.2017.02057 |
| [44] | Orellana LH, Ben Francis T, Krüger K, Teeling H, Müller MC, Fuchs BM, Konstantinidis KT, Amann RI. Niche differentiation among annually recurrent coastal Marine Group Ⅱ Euryarchaeota. The ISME Journal, 2019, 13(12): 3024-3036. DOI:10.1038/s41396-019-0491-z |
| [45] | Needham DM, Fuhrman JA. Pronounced daily succession of phytoplankton, archaea and bacteria following a spring bloom. Nature Microbiology, 2016, 1: 16005. DOI:10.1038/nmicrobiol.2016.5 |
| [46] | Murray AE, Wu KY, Moyer CL, Karl DM, DeLong EF. Evidence for circumpolar distribution of planktonic Archaea in the Southern Ocean. Aquatic Microbial Ecology, 1999, 18(3): 263-273. |
| [47] | Galand PE, Gutiérrez-Provecho C, Massana R, Gasol JM, Casamayor EO. Inter-annual recurrence of archaeal assemblages in the coastal NW Mediterranean Sea (Blanes Bay Microbial Observatory). Limnology and Oceanography, 2010, 55(5): 2117-2125. DOI:10.4319/lo.2010.55.5.2117 |
| [48] | Hugoni M, Taib N, Debroas D, Domaizon I, Jouan Dufournel I, Bronner G, Salter I, Agogué H, Mary I, Galand PE. Structure of the rare archaeal biosphere and seasonal dynamics of active ecotypes in surface coastal waters. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(15): 6004-6009. DOI:10.1073/pnas.1216863110 |
| [49] | Galand PE, Lovejoy C, Vincent WF. Remarkably diverse and contrasting archaeal communities in a large arctic river and the coastal Arctic Ocean. Aquatic Microbial Ecology, 2006, 44(2): 115-126. |
| [50] | Martin-Cuadrado AB, Garcia-Heredia I, Moltó AG, López-úbeda R, Kimes N, López-García P, Moreira D, Rodriguez-Valera F. A new class of marine Euryarchaeota group Ⅱ from the mediterranean deep chlorophyll maximum. The ISME Journal, 2015, 9(7): 1619-1634. DOI:10.1038/ismej.2014.249 |
| [51] | Tian JW, Fan L, Liu HD, Liu JW, Li Y, Qin QL, Gong Z, Chen HT, Sun ZB, Zou L, Wang XC, Xu HZ, Bartlett D, Wang M, Zhang YZ, Zhang XH, Zhang CL. A nearly uniform distributional pattern of heterotrophic bacteria in the Mariana Trench interior. Deep Sea Research Part Ⅰ:Oceanographic Research Papers, 2018, 142: 116-126. DOI:10.1016/j.dsr.2018.10.002 |
| [52] | Crump BC, Baross JA. Archaeaplankton in the Columbia River, its estuary and the adjacent coastal ocean, USA. FEMS Microbiology Ecology, 2000, 31(3): 231-239. DOI:10.1111/j.1574-6941.2000.tb00688.x |
| [53] | Liu JW, Yu SL, Zhao MX, He BY, Zhang XH. Shifts in archaeaplankton community structure along ecological gradients of Pearl Estuary. FEMS Microbiology Ecology, 2014, 90(2): 424-435. |
| [54] | Massana R, Taylor LT, Murray AE, Wu KY, Jeffrey WH, DeLong EF. Vertical distribution and temporal variation of marine planktonic archaea in the Gerlache Strait, Antarctica, during early spring. Limnology and Oceanography, 1998, 43(4): 607-617. DOI:10.4319/lo.1998.43.4.0607 |
| [55] | Wuchter C, Abbas B, Coolen MJL, Herfort L, Van Bleijswijk J, Timmers P, Strous M, Teira E, Herndl GJ, Middelburg JJ, Schouten S, Damsté JSS. Archaeal nitrification in the ocean. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(33): 12317-12322. DOI:10.1073/pnas.0600756103 |
| [56] | Levipan HA, Qui?ones RA, Urrutia H. A time series of prokaryote secondary production in the oxygen minimum zone of the Humboldt current system, off central Chile. Progress in Oceanography, 2007, 75(3): 531-549. DOI:10.1016/j.pocean.2007.08.029 |
| [57] | Beman JM, Sachdeva R, Fuhrman JA. Population ecology of nitrifying Archaea and Bacteria in the southern California Bight. Environmental Microbiology, 2010, 12(5): 1282-1292. |
| [58] | Beman JM, Steele JA, Fuhrman JA. Co-occurrence patterns for abundant marine archaeal and bacterial lineages in the deep chlorophyll maximum of coastal California. The ISME Journal, 2011, 5(7): 1077-1085. DOI:10.1038/ismej.2010.204 |
| [59] | Reysenbach AL, Flores GE. Electron microscopy encounters with unusual thermophiles helps direct genomic analysis of Aciduliprofundum boonei. Geobiology, 2008, 6(3): 331-336. DOI:10.1111/j.1472-4669.2008.00152.x |
| [60] | Iverson V, Morris RM, Frazar CD, Berthiaume CT, Morales RL, Armbrust EV. Untangling genomes from metagenomes:revealing an uncultured class of marine Euryarchaeota. Science, 2012, 335(6068): 587-590. DOI:10.1126/science.1212665 |
| [61] | Baker BJ, Sheik CS, Taylor CA, Jain S, Bhasi A, Cavalcoli JD, Dick GJ. Community transcriptomic assembly reveals microbes that contribute to deep-sea carbon and nitrogen cycling. The ISME Journal, 2013, 7(10): 1962-1973. DOI:10.1038/ismej.2013.85 |
| [62] | Li X, Qin L. Metagenomics-based drug discovery and marine microbial diversity. Trends in Biotechnology, 2005, 23(11): 539-543. DOI:10.1016/j.tibtech.2005.08.006 |
| [63] | Orsi WD, Smith JM, Liu ST, Liu ZF, Sakamoto CM, Wilken S, Poirier C, Richards TA, Keeling PJ, Worden AZ, Santoro AE. Diverse, uncultivated bacteria and archaea underlying the cycling of dissolved protein in the ocean. The ISME Journal, 2016, 10(9): 2158-2173. DOI:10.1038/ismej.2016.20 |
| [64] | Aylward FO, Eppley JM, Smith JM, Chavez FP, Scholin CA, Delong EF. Microbial community transcriptional networks are conserved in three domains at ocean basin scales. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(17): 5443-5448. DOI:10.1073/pnas.1502883112 |
| [65] | Boutrif M, Garel M, Cottrell MT, Tamburini C. Assimilation of marine extracellular polymeric substances by deep-sea prokaryotes in the NW Mediterranean Sea. Environmental Microbiology Reports, 2011, 3(6): 705-709. DOI:10.1111/j.1758-2229.2011.00285.x |
| [66] | Bergauer K, Fernandez-Guerra A, Garcia JAL, Sprenger RR, Stepanauskas R, Pachiadaki MG, Jensen ON, Herndl GJ. Organic matter processing by microbial communities throughout the Atlantic water column as revealed by metaproteomics. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(3): E400-E408. DOI:10.1073/pnas.1708779115 |
| [67] | Herndl GJ, Reinthaler T, Teira E, Van Aken H, Veth C, Pernthaler A, Pernthaler J. Contribution of Archaea to total prokaryotic production in the deep Atlantic Ocean. Applied and Environmental Microbiology, 2005, 71(5): 2303-2309. DOI:10.1128/AEM.71.5.2303-2309.2005 |
| [68] | Martin-Cuadrado AB, Rodriguez-Valera F, Moreira D, Alba JC, Ivars-Martínez E, Henn MR, Talla E, López-García P. Hindsight in the relative abundance, metabolic potential and genome dynamics of uncultivated marine archaea from comparative metagenomic analyses of bathypelagic plankton of different oceanic regions. The ISME Journal, 2008, 2(8): 865-886. DOI:10.1038/ismej.2008.40 |
| [69] | Moreira D, Rodríguez-Valera F, López-García P. Analysis of a genome fragment of a deep-sea uncultivated Group Ⅱ euryarchaeote containing 16S rDNA, a spectinomycin-like operon and several energy metabolism genes. Environmental Microbiology, 2004, 6(9): 959-969. DOI:10.1111/j.1462-2920.2004.00644.x |
| [70] | Lima-Mendez G, Faust K, Henry N, Decelle J, Colin S, Carcillo F, Chaffron S, Ignacio-Espinosa JC, Roux S, Vincent F, Bittner L, Darzi Y, Wang J, Audic S, Berline L, Bontempi G, Cabello AM, Coppola L, Cornejo-Castillo FM, d'Ovidio F, De Meester L, Ferrera I, Garet-Delmas MJ, Guidi L, Lara E, Pesant S, Royo-Llonch M, Salazar G, Sánchez P, Sebastian M, Souffreau C, Dimier C, Picheral M, Searson S, Kandels-Lewis S, Tara Oceans coordinators, Gorsky G, Not F, Ogata H, Speich S, Stemmann L, Weissenbach J, Wincker P, Acinas SG, Sunagawa S, Bork P, Sullivan MB, Karsenti E, Bowler C, De Vargas C, Raes J. Ocean plankton. Determinants of community structure in the global plankton interactome. Science, 2015, 348(6237): 1262073. DOI:10.1126/science.1262073 |
| [71] | Jiao N, Luo T, Zhang R, Yan W, Lin Y, Johnson ZI, Tian J, Yuan D, Yang Q, Zheng Q, Sun J, Hu D, Wang P. Presence of Prochlorococcus in the aphotic waters of the western Pacific Ocean. Biogeosciences, 2014, 11(8): 2391-2400. |
| [72] | Gilbert JA, Dupont CL. Microbial metagenomics:beyond the genome. Annual Review of Marine Science, 2011, 3: 347-371. DOI:10.1146/annurev-marine-120709-142811 |
| [73] | Steele JA, Countway PD, Xia L, Vigil PD, Beman JM, Kim DY, Chow CET, Sachdeva R, Jones AC, Schwalbach MS, Rose JM, Hewson I, Patel A, Sun FZ, Caron DA, Fuhrman JA. Marine bacterial, archaeal and protistan association networks reveal ecological linkages. The ISME Journal, 2011, 5(9): 1414-1425. DOI:10.1038/ismej.2011.24 |
| [74] | Sa?udo-Wilhelmy SA, Gómez-Consarnau L, Suffridge C, Webb EA. The role of B vitamins in marine biogeochemistry. Annual Review of Marine Science, 2014, 6: 339-367. DOI:10.1146/annurev-marine-120710-100912 |
| [75] | Doxey AC, Kurtz DA, Lynch MDJ, Sauder LA, Neufeld JD. Aquatic metagenomes implicate Thaumarchaeota in global cobalamin production. The ISME Journal, 2015, 9(2): 461-471. DOI:10.1038/ismej.2014.142 |
| [76] | Heal KR, Qin W, Ribalet F, Bertagnolli AD, Coyote-Maestas W, Hmelo LR, Moffett JW, Devol AH, Armbrust EV, Stahl DA, Ingalls AE. Two distinct pools of B12 analogs reveal community interdependencies in the ocean. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(2): 364-369. DOI:10.1073/pnas.1608462114 |
| [77] | Ahlgren NA, Chen YY, Needham DM, Parada AE, Sachdeva R, Trinh V, Chen T, Fuhrman JA. Genome and epigenome of a novel marine Thaumarchaeota strain suggest viral infection, phosphorothioation DNA modification and multiple restriction systems. Environmental Microbiology, 2017, 19(6): 2434-2452. DOI:10.1111/1462-2920.13768 |
| [78] | Philosof A, Yutin N, Flores-Uribe J, Haron I, Koonin EV, Béjà O. Novel abundant oceanic viruses of uncultured marine group Ⅱ Euryarchaeota. Current Biology, 2017, 27(9): 1362-1368. DOI:10.1016/j.cub.2017.03.052 |
| [79] | Vik DR, Roux S, Brum JR, Bolduc B, Emerson JB, Padilla CC, Stewart FJ, Sullivan MB. Putative archaeal viruses from the mesopelagic ocean. PeerJ, 2017, 5: e3428. DOI:10.7717/peerj.3428 |
| [80] | Nishimura Y, Watai H, Honda T, Mihara T, Omae K, Roux S, Blanc-Mathieu R, Yamamoto K, Hingamp P, Sako Y, Sullivan MB, Goto S, Ogata H, Yoshida T. Environmental viral genomes shed new light on virus-host interactions in the ocean. mSphere, 2017, 2(2): e00359-16. |
| [81] | Rappé MS, Connon SA, Vergin KL, Giovannoni SJ. Cultivation of the ubiquitous SAR11 marine bacterioplankton clade. Nature, 2002, 418(6898): 630-633. DOI:10.1038/nature00917 |
| [82] | Besseling MA, Hopmans EC, Bale NJ, Schouten S, Damsté JSS, Villanueva L. The absence of intact polar lipid-derived GDGTs in marine waters dominated by Marine Group Ⅱ:implications for lipid biosynthesis in Archaea. Scientific Reports, 2020, 10(1): 294. DOI:10.1038/s41598-019-57035-0 |
| [83] | Pan XJ, Jiao NZ. Advances in research of marine archaea. Marine Sciences, 2001, 25(2): 20-23. (in Chinese) 潘晓驹, 焦念志. 海洋古菌的研究进展. 海洋科学, 2001, 25(2): 20-23. |
| [84] | Ren LC, Li MY, Bao SX. The investigation of the marine archaea diversity. Life Science Research, 2006, 10(2): 67-70. (in Chinese) 任立成, 李美英, 鲍时翔. 海洋古菌多样性研究进展. 生命科学研究, 2006, 10(2): 67-70. |
| [85] | Zhao MX, Li DW, Xing L. Using archaea biomarker index TEX86 as a paleo-sea surface temperature proxy. Marine Geology & Quaternary Geology, 2009, 29(3): 75-84. (in Chinese) 赵美训, 李大伟, 邢磊. 古菌生物标志物古海水温度指标TEX86研究进展. 海洋地质与第四纪地质, 2009, 29(3): 75-84. |
| [86] | Lai DX, Xie W, Fan L, Tao JC, Hu AY, Wang P, Zhu YQ, Zhang CL. The spatial patterning of Marine Group Ⅱ archaea in Chinese coastal and esturine waters. Bulletin of National Natural Science Foundation of China, 2018, 32(5): 459-470. (in Chinese) 赖登训, 谢伟, 范陆, 陶建昌, 胡安谊, 王鹏, 朱元清, 张传伦. MGII古菌在中国近海和河口的空间分布特征. 中国科学基金, 2018, 32(5): 459-470. |
| [87] | Zhu SQ, Liu JW, Zheng YF, Liu J, Meng Z, Liu XY, Wang YW, Zhang XH. An overview of archaeal community structure in the Chinese coastal areas. Marine Sciences, 2019, 43(5): 123-134. (in Chinese) 朱尚清, 刘吉文, 郑艳芬, 刘骥, 孟哲, 刘晓月, 王雅文, 张晓华. 中国近海区域古菌群落结构研究概述. 海洋科学, 2019, 43(5): 123-134. |
| [88] | Reysenbach AL, Liu YT, Banta AB, Beveridge TJ, Kirshtein JD, Schouten S, Tivey MK, Von Damm KL, Voytek MA. A ubiquitous thermoacidophilic archaeon from deep-sea hydrothermal vents. Nature, 2006, 442(7101): 444-447. DOI:10.1038/nature04921 |
| [89] | Besseling MA, Hopmans EC, Bale NJ, Schouten S, Sinninghe Damsté JS, Villanueva L. The absence of intact polar lipid-derived GDGTs in marine waters dominated by Marine Group Ⅱ:implications for lipid biosynthesis in Archaea. Scientific Reports, 2020, 10(1): 294. DOI:10.1038/s41598-019-57035-0 |
| [90] | Ma CL, Coffinet S, Lipp JS, Hinrichs KU, Zhang CL. Marine Group Ⅱ Euryarchaeota contribute to the archaeal lipid pool in Northwestern Pacific Ocean surface waters. Frontiers in Microbiology, 2020, 6: 1034. |
