张文洪1, 杨雁霞1, 杨正凤1, 黄遵锡1,2,3, 李俊俊1,2,3, 唐湘华1,2,3, 杨云娟1,2,3, 吴倩1,2,3, 慕跃林1,2,3, 韩楠玉1,2,3, 许波1,2,3


1. 云南师范大学 生命科学学院, 云南 昆明 650500;
2. 生物能源持续开发利用教育部工程研究中心, 云南 昆明 650500;
3. 云南省生物质能与环境生物技术重点实验室, 云南 昆明 650500
收稿日期:2018-10-19;修回日期:2019-01-24;网络出版日期:2019-03-07
基金项目:国家自然科学基金(31860299,31560305);国家重点研发计划2017年度项目(2017YFB0308400);国家重点项目(2017YFB0308401)
*通信作者:许波, Tel:+86-871-5920830;Fax:+86-871-5920952;E-mail:xubo128028@163.com.
摘要:[目的] 对滇金丝猴粪便微生物来源的β-半乳糖苷酶进行异源表达和纯化,并研究其酶学性质。[方法] 从滇金丝猴粪便微生物的宏基因组中克隆出一个β-半乳糖苷酶基因galRBM20_1,对该基因进行异源表达和酶学性质分析。构建含有T7强启动子的pEASY-E2-galRBM20_1质粒,转化至大肠杆菌BL21(DE3),经IPTG诱导表达后进行酶学性质研究。[结果] 滇金丝猴粪便来源的β-半乳糖苷酶(galRBM20_1)最适pH为5.0,在pH 4-7之间能保留70%及其以上的活性。最适温度为45℃,在37℃和45℃下耐受1 h,酶活不变。特别的是,该酶具有良好的NaCl稳定性,经1-5 mol/L的NaCl作用1 h后,相对酶活均能超过初始酶活:当NaCl的作用浓度为4 mol/L时,β-半乳糖苷酶相对酶活最高(146%);当NaCl的作用浓度为5 mol/L时,β-半乳糖苷酶的相对酶活仍达到135%。[结论] 本研究从滇金丝猴粪便微生物的宏基因库中克隆得到β-半乳糖苷酶基因galRBM20_1,并成功在大肠杆菌BL21(DE3)表达,首次从动物胃肠道宏基因组中获得具有耐盐和转糖基产Galactooligosaccharides(GOS)性能的β-半乳糖苷酶。该酶具有良好的耐盐性,和较广的pH作用范围,使其在食品、生物技术领域和环保方面的发展具有良好的应用价值。
关键词:滇金丝猴粪便微生物宏基因组β-半乳糖苷酶耐盐性转糖基
Expression and characterization of β-galactosidase from fecal microbes of Rhinopithecus bieti
Wenhong Zhang1, Yanxia Yang1, Zhengfeng Yang1, Zunxi Huang1,2,3, Junjun Li1,2,3, Xianghua Tang1,2,3, Yunjuan Yang1,2,3, Qian Wu1,2,3, Yuelin Mu1,2,3, Nanyu Han1,2,3, Bo Xu1,2,3


1. School of Life Sciences, Yunnan Normal University, Kunming 650500, Yunnan Province, China;
2. Engineering Research Center of Sustainable Development and Utilization of Biomass Energy, Ministry of Education, Kunming 650500, Yunnan Province, China;
3. Key Laboratory of Yunnan for Biomass Energy and Biotechnology of Environment, Kunming 650500, Yunnan Province, China
Received: 19 October 2018; Revised: 24 January 2019; Published online: 7 March 2019
*Corresponding author: Xu Bo, Tel:+86-871-5920830;Fax:+86-871-5920952;E-mail:xubo128028@163.com.
Foundation item: Supported by the National Natural Science Foundation of China (31860299, 31560305); The National Key Research and Development Program of China (2017YFB0308400); National Key Project (2017YFB0308401)
Abstract: [Objective] The study was to express and characterize β-galactosidase from the fecal microbes of Rhinopithecus bieti. [Methods] A β-galactosidase gene galRBM20_1 was cloned from the fecal microbial metagenome of Rhinopithecus bieti. The pEASY-E2-galRBM20_1 plasmid was constructed and transformed into Escherichia coli BL21 (DE3). The expression was induced by Isopropyl β-D-1-thiogalactopyranoside and the enzymatic properties were studied. [Results] The optimum pH of β-galactosidase galRBM20_1 was 5.0, and the enzyme retained more than 70% activity between pH 4 and 7. The optimum temperature is 45℃ and the enzyme was stable at 37℃ and 45℃, retaining nearly 100% activity after incubation for 1 h. In addition, the enzyme had strong NaCl stability. After 1 h of 1 to 5 mol/L NaCl treatment, the relative enzyme activity maintained 100%; when the concentration of NaCl was 4 mol/L, the activity of β-galactosidase galRBM20_1 was the highest (146%); it still had activity after treatment with 2.5 mol/L NaCl at 45℃ for 24 h. [Conclusion] β-galactosidase galRBM20_1 has good salt tolerance and wide pH range.
Keywords: Rhinopithecus bietifecal microbial metagenomeβ-galactosidasesalt-toleranttransglycosylation
β-半乳糖苷酶(EC 3.2.1.23)又名β-D-半乳糖苷半乳糖水解酶和乳糖酶,能够催化共轭糖非还原末端半乳糖残基的水解,将乳糖水解为葡萄糖和半乳糖[1-2];同时该酶具有转糖基活性,可将乳糖催化形成低聚半乳糖。因此,β-半乳糖苷酶被广泛运用于食品工业中乳糖的水解,提高乳制品的消化率、甜度、溶解度和风味等领域中[3-4]。
乳糖是哺乳动物乳汁及乳制品中存在的主要碳水化合物,正常情况下必须被β-半乳糖苷酶水解为单糖、半乳糖或葡萄糖才能被机体吸收利用。统计表明,世界上70%人都存在乳糖酶缺乏症[5]。由于体内缺乏β-半乳糖苷酶,未被消化的乳糖在结肠内被肠道细菌分解成短链脂肪酸和氢气、甲烷、二氧化碳等气体,从而引起腹胀、腹痛、腹鸣等症状[6]。β-半乳糖苷酶添加到乳制品中,是解决全球乳糖不耐受症患者的乳糖消耗问题、克服全球乳糖不耐受症问题影响的有效方法[7]。此外,利用β-半乳糖苷酶的转糖基活性,可将乳糖上的半乳糖残基转移到糖受体上产生低聚半乳糖(Galactooligosaccharides,GOS),低聚半乳糖是一种重要的益生元,能够促进益生肠道细菌增殖,改善肠道健康[8]。
β-半乳糖苷酶来源广泛,微生物来源的β-半乳糖苷酶主要来自大肠杆菌、乳酸杆菌、酵母菌、芽孢杆菌和霉菌等[9]。由于微生物来源的β-半乳糖苷酶具有催化活性高、产量高、易于表达等特点,因此具有广泛的用途。根据氨基酸序列的相似性,β-半乳糖苷酶可归入5种糖苷水解酶(Glycoside hydrolases,GH)家族,包括1、2、35、42和59家族[10]。一般来说,真菌来源的β-半乳糖苷酶主要属于GH35家族,细菌来源的β-半乳糖苷酶主要属于GH2和GH42家族,而GH2家族的β-半乳糖苷酶在乳糖水解和GOS合成方面更有效[11]。
依据报道,已获得的β-半乳糖苷酶主要来源于纯培养的微生物菌株。由于自然界中仅有约0.1%-1%的微生物能通过传统纯培养分离方法获得[12],大大限制了β-半乳糖苷酶的开发利用。宏基因组学技术避开了微生物分离培养的问题,使得从环境中获得大量未培养微生物基因资源成为现实,极大地扩展了微生物酶及基因资源的利用空间,是发现新型生物催化剂的有效方法[13]。目前,利用宏基因组学技术,研究者已从土壤、海洋、热泉、小麦秸秆降解物等环境样品中获得β-半乳糖苷酶,并对其进行了克隆表达及鉴定[14-17]。然而,动物胃肠道微生物宏基因组来源的β-半乳糖苷酶很少见报道。
胃肠道微生物是动物长期进化的结果,蕴含着丰富的微生物酶类基因资源。前期研究中结合培养及宏基因组Shotgun测序,我们从滇金丝猴粪便微生物中获得多种酶类基因[18-19],并对木聚糖酶等进行了异源表达和鉴定[20-21]。本研究从滇金丝猴粪便微生物宏基因组中克隆得到β-半乳糖苷酶基因galRBM20_1,经大肠杆菌异源表达后,对其酶学性质进行研究。此外,由于β-半乳糖苷酶被广泛应用于食品工业行业、生物技术和医药等领域,耐盐与否与其应用前景息息相关,故本文也谈到对该β-半乳糖苷酶基因galRBM20_1表达的蛋白进行耐盐性研究。
1 材料和方法 1.1 材料、酶和载体 滇金丝猴粪便微生物宏基因组Fosmid文库混合质粒,实验室保藏;LA-Taq酶、dNTP Mix、2×GC buffer Ⅰ、限制性内切酶、载体pMD19-T,购自大连宝生物工程有限公司;表达载体pEASY-E2购自北京全式金生物技术有限公司;一步快速克隆试剂盒(ClonExpressⅡOne Step Cloning Kit),购自诺唯赞生物科技有限公司。
1.2 试剂和仪器 质粒提取试剂盒、胶回收纯化试剂盒为美国Omega公司产品,购自天根生化科技有限公司;Ni-NTA Agarose,购自QIAGEN公司;Trans1-T1、BL-21(DE3),购自北京全式金生物技术有限公司;氨苄青霉素钠,购自宝生物工程有限公司;2-硝基苯基β-D-吡喃半乳糖苷(oNPG)、4-硝基苯基β-D-吡喃半乳糖苷(pNPG),购自Sigma-Aldrich公司。Qubit荧光定量仪、Qubit DNA Quantification Kit,赛默飞世尔科技有限公司;细胞超声破碎仪,宁波新芝生物科技股份有限公司;纯水仪,锐思捷科学仪器有限公司;离心机,Eppendorf公司;凝胶成像仪、琼脂糖凝胶电泳仪、SDS-PAGE蛋白电泳仪和PCR仪等,为Bio-Rad公司;紫外分光光度计,德国耶拿分析仪器股份公司。
1.3 基因克隆和序列分析
1.3.1 基因克隆: 设计引物galRBM20_1DNAF (5′-ATGGCCTGCCGGCGTAGGGGCGTT-3′)和galRBM20_1DNAR (5′-CCACTGGTTGTCCAGG TTTTTCGTA-3′),以滇金丝猴粪便微生物宏基因组Fosmid文库混合质粒为模板,进行Touch-down PCR扩增β-半乳糖苷酶基因galRBM20_1。PCR反应体系:2×GC bufferⅠ25.0 μL,2.5 mmol/L dNTP Mix 4.0 μL,LA-Taq DNA Polymerase 1 U,10 μmol/L galRBM20_1 DNAF 1.0 μL,10 μmol/L galRBM20_1 DNAR 1.0 μL,宏基因组文库的Fosmid混合质粒0.5 μL,dd H2O补足50.0 μL。PCR反应程序为:94 ℃ 5 min;94 ℃ 30 s;63 ℃ 30 s (每个循环降0.5 ℃);72 ℃ 1 min,20个循环;94 ℃ 30 s,53 ℃ 30 s,72 ℃ 1 min,10个循环;72 ℃ 10 min。随后将其克隆到载体pMD19-T/galRBM20_1,转化到感受态细胞Trans1-T1/galRBM20_1,经阳性克隆验证(菌液PCR)后,保种并测序。
1.3.2 序列分析: 使用BLAST服务器(http://blast.ncbi.nlm.nih.gov/Blast.cgi)完成核苷酸和氨基酸序列比对。使用BioXM 2.6预测蛋白质分子量大小。信号肽预测使用SignalP 4.1服务器(http://www.cbs.dtu.dk/services/SignalP/)。使用MEGA6.0进行多序列比对分析和制作系统进化树,ESPript 3.0 (Http://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgi)进行比对结果美化。
1.4 大肠杆菌重组表达系统的构建 根据β-半乳糖苷酶基因galRBM20_1的全长序列以及一步快速克隆试剂盒使用说明设计表达引物:galRBM20_1NdeF (5′-TAAGAAGGAGAT ATACATATGGAATTGGCTGCCGGCGTAGGGGCG-3′)与galRBM20_1XhoR (5′-GTGGTGGTGGT GGTGCTCGAGACAAATTGCAAAGGAATAT-3′),其中galRBM20_1NdeF和galRBM20_1XhoR分别含有限制性内切酶NdeⅠ(CATATG)和XhoⅠ (CTCGAG)的识别位点。取含有载体pMD19-T/ galRBM20_1的大肠杆菌菌株Trans1-T1/ galRBM20_1,以0.1%的量接种于LB(含100 mg/L Amp)的液体培养基中,37 ℃、180 r/min培养12-16 h后,提取质粒pMD19-T/galRBM20_1。将质粒pMD19-T/galRBM20_1作为模板,使用galRBM20_1NdeF和galRBM20_1XhoR两种引物PCR,得到含有内切酶NdeⅠ和XhoⅠ识别位点的基因片段galRBM20_1/NdeⅠ/XhoⅠ。使用限制性内切酶NdeⅠ与XhoⅠ切割表达载体pEASY-E2使其线性化。采用一步快速克隆试剂盒将基因片段galRBM20_1/Nd-eⅠ/XhoⅠ克隆到线性化的表达载体pEASY-E2上,连接得到原核表达质粒pEASY-E2/galRBM20_1,转化大肠杆菌BL-21 (DE3)获得重组大肠杆菌菌株BL-21 (DE3)/ galRBM20_1的阳性克隆子,命名为BL-21 (DE3)/ galRBM20_1,送上海生工测序。
1.5 蛋白的表达与纯化 取大肠杆菌菌株BL-21(DE3)/galRBM20_1以0.1%的量接种于LB(含100 mg/L Amp)的液体培养基中,37 ℃、180 r/min摇床培养约3-4 h (OD600到0.6-1.0)后,加入IPTG(终浓度为0.7 mmol/L)在20 ℃和160 r/min摇床下培养10 h以诱导重组蛋白产生。8000 r/min离心6 min,收集菌体,用Tris-HCl (PH 7.0)缓冲液重悬菌体,之后再冰水浴中超声破碎细胞(300 W,超声开5 s,超声关7 s)。超声破碎的细胞液体经冷冻离心机4 ℃、12000 r/min离心10 min,重复2-3次(无杂细胞碎片)收集上清。将上清通过亲和层析柱Ni-NTA Agarose进行蛋白纯化,纯化后的蛋白即为含有6×His-tag的β-半乳糖苷酶galRBM20_1。以空载体pEASY-E2转化BL-21(DE3)作为阴性对照检测β-半乳糖苷酶galRBM20_1的活性和通过SDS- PAGE检测纯化后酶的纯度。
1.6 酶活性的测定 将2-硝基苯基β-D-吡喃半乳糖苷(oNPG)溶于0.1 mol/L的柠檬酸-磷酸氢二钠缓冲液(pH 5.0),配制成浓度为2 mmol/L的底物溶液。取450 μL的底物于45 ℃水浴预热5 min,加入50 μL纯化并适当稀释的酶液,反应10 min。之后加入1 mL 1 mol/L Na2CO3终止反应,测定波长在405 nm处的吸光值。酶活单位定义:一个酶活单位(U)即在pH 5.0、45 ℃条件下每分钟水解底物oNPG生成1 μmol oNP所需的酶量。
1.7 重组蛋白galRBM20_1的酶学性质研究
1.7.1 重组β-半乳糖苷酶galRBM20_1的最适温度和最适pH的测定: 在pH 7.0的缓冲液中,于不同温度(0、10、20、30、40、45、50、55、60、65、70、80 ℃)条件下测定β-半乳糖苷酶galRBM20_1的酶活力,每个温度设1个对照和3个重复,以最高活力计为100%绘制最适温度曲线。
分别配制pH 2.2-10.0的不同缓冲液(pH 2.2-8.0:0.1 mol/L柠檬酸-磷酸氢二钠缓冲液;pH 8.0-10.0:甘氨酸-氢氧化钠缓冲液),将底物oNPG溶于上述配制的不同pH缓冲液中,并将β-半乳糖苷酶用相应的pH缓冲液稀释。在45 ℃的条件下测定各pH下的酶活力,以最高活力计为100%,每个pH条件下设1个对照和3个重复绘制最适pH曲线。
1.7.2 重组β-半乳糖苷酶galRBM20_1的温度稳定性和pH稳定性的测定: 在温度为37 ℃、45 ℃和50 ℃下,对β-半乳糖苷酶galRBM20_1分别预处理10、20、30、40、50、60 min后,在45 ℃、pH 5.0条件下测定β-半乳糖苷酶经不同温度和时间处理后的剩余酶活。以未经处理的酶液活力计为100%,每个温度下设1个对照和3个重复,以时间为横坐标,相对酶活(剩余酶活)为纵坐标绘制温度稳定性曲线。
将纯化后的原酶液用不同pH (pH 3-11)的缓冲液稀释后,置于37 ℃恒温水浴中放置1 h后置于冰上,在45 ℃、pH 5.0 (0.1 mol/L pH 5.0柠檬酸-磷酸氢二钠)条件下测定β-半乳糖苷酶的剩余酶活力。以未经处理的酶液活力计为100%,每个pH下设1个对照和3个重复,绘制pH稳定性曲线。
1.7.3 重组β-半乳糖苷酶galRBM20_1的底物特异性和动力学参数测定: 在45 ℃、pH 5.0的条件下,以不同底物包括oNPG、pNPG、乳糖和其他硝基苯衍生物等测定底物特异性。动力学方程则使用不同浓度的oNPG (0.1-2.0 mmol/L)、pNPG (0.50-1.75 mmol/L)和乳糖(20-80 mmol/L)在一级反应时间下进行测定,根据Lineweaver- Burk法计算出Km、Vmax和kcat等值。
1.7.4 金属离子和化学试剂对重组β-半乳糖苷酶galRBM20_1的影响测定: 将各种金属离子和化学试剂(Na+、Li+、Ca2+、K+、Fe2+、Zn2+、Mg2+、Na+、Mn2+、Fe3+、Pb2+、Ni2+、Co2+、Ag+、Cu2+、Hg2+、Al3+、EDTA、SDS、CTAB、β-Mercaptoethanol、Triton X-100、Tween-80)加入到酶促反应体系中,使其终浓度为1 mmol/L(其中Triton X-100、Tween-80终浓度为0.5%(体积比)),在45 ℃、pH 5.0条件下测定β-半乳糖苷酶活力。以不加金属离子和有机试剂的酶活力为100%,每种体系下设1个对照和3个重复。
1.8 NaCl对重组β-半乳糖苷酶galRBM20_1的影响和稳定性的测定 用pH 5.0 (0.1 mol/L pH 5.0柠檬酸-磷酸氢二钠)的缓冲液配制不同浓度的NaCl溶液(0.5- 5 mol/L)。在45 ℃下,向不同浓度的NaCl溶液加入底物和酶液进行酶促反应,以反应体系中不含NaCl的反应酶活力为100%,每种不同浓度的NaCl下设1个对照和3个重复测定NaCl对β-半乳糖苷酶galRBM20_1的影响。将适当稀释的酶液加入不同浓度的NaCl溶液中,使NaCl终浓度为0.5-5.0 mol/L,45 ℃恒温水浴中放置1 h和24 h后,于45 ℃、pH 5.0 (0.1 mol/L pH 5.0柠檬酸-磷酸氢二钠)条件下测定β-半乳糖苷酶的剩余酶活力。以未经NaCl处理的酶活力为100%,测定NaCl稳定性。
1.9 重组β-半乳糖苷酶galRBM20_1的水解和转糖基性能测定 将纯化后的β-半乳糖苷酶分别加入到1 mL 5% (W/V)的乳糖溶液中,在45 ℃、pH 5.0条件下反应,间隔不同时间取样(30 min、2 h、8 h、12 h),煮沸5 min使酶变性失活,用正丁醇︰乙醇︰水(5:3:2, V:V:V)作为展开剂,进行薄层层析(TLC)分析。
将纯化后的β-半乳糖苷酶加入到25% (W/W)的乳糖溶液中,在45 ℃、pH 5.0条件下反应24 h,煮沸5 min使酶变性失活,以灭活后的酶液反应组做阴性对照进行TLC分析。
2 结果和分析 2.1 基因galRBMn20_1的克隆和序列分析 通过PCR扩增得到β-半乳糖苷酶基因galRBM20_1,其核苷酸序列已在GenBank中保存(登录号MH177893)。该基因全长1893 bp,(G+C)%含量54.9%,起始密码子ATG, ,终止密码子TAA,编码630个氨基酸,分子量为71.58 kDa,等电点为9.3,无信号肽序列。与GenBank中的序列比对发现,galRBM20_1的氨基酸序列与已报道的嗜粘蛋白-艾克曼菌(Akkermansia Muciniphila)来源的GH35家族β-半乳糖苷酶具有88%的最大相似性(GenBank ID: WP_094136891)。与GenBank中GH35家族β-半乳糖苷酶的氨基酸多序列比对发现,保守性氨基酸残基为57-YVFWN-61、95-RPGPY-99、160-ENEYG-164、280-HGGT-283、535-VWVNG-539、543- GRYW-546、561-LKKGEN-566等(图 1)。将galRBM20_1与不同家族来源的β-半乳糖苷酶的氨基酸序列进行系统进化分析表明(图 2),该进化树分为两个不同的大分支。galRBM20_1与GH35家族细菌来源的β-半乳糖苷酶具有高度亲缘性,且共同与高等真核生物和真菌来源的β-半乳糖苷酶处于同一分支,与GH42和GH2家族的β-半乳糖苷酶亲缘性较远,处于不同的两个大分支。表明galRBM20_1是GH35家族β-半乳糖苷酶的新成员。
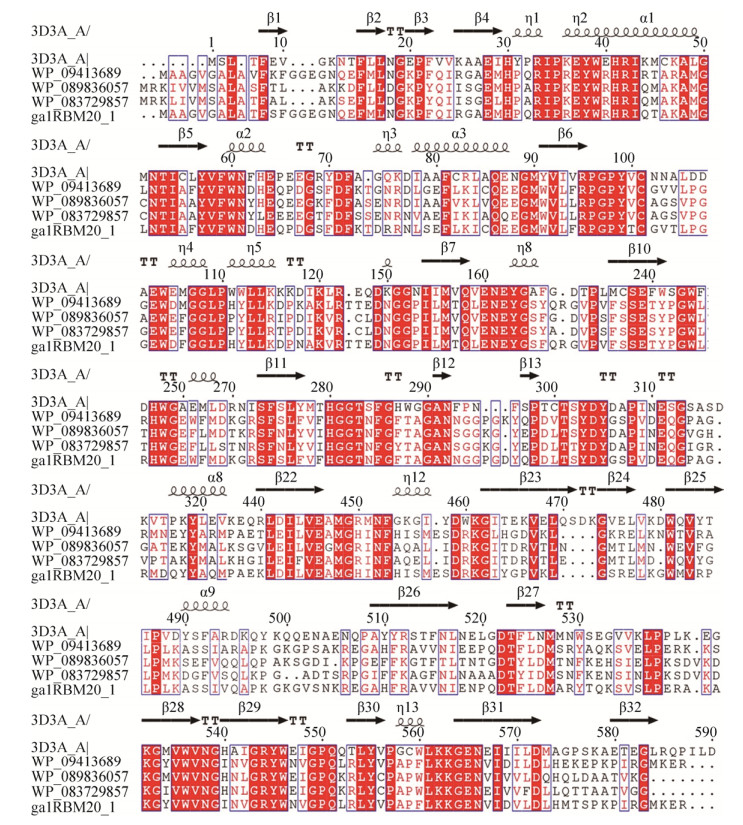 |
| 图 1 galRBM20_1的多序列比对分析 Figure 1 Multiple sequence alignment analysis of galRBM20_1. β-galactosidase of 3D3A Chian A (GenBank: AE015928; PBD: 3D3A) from Bacteroides thetaiotaomicron was used as the secondary structural template. WP_09413689, WP_089836057 and WP_083729875 are from Akkermansia Muciniphila, Chitinophaga filiformis and Bacteroidetes bacterium, respectively. |
| 图选项 |
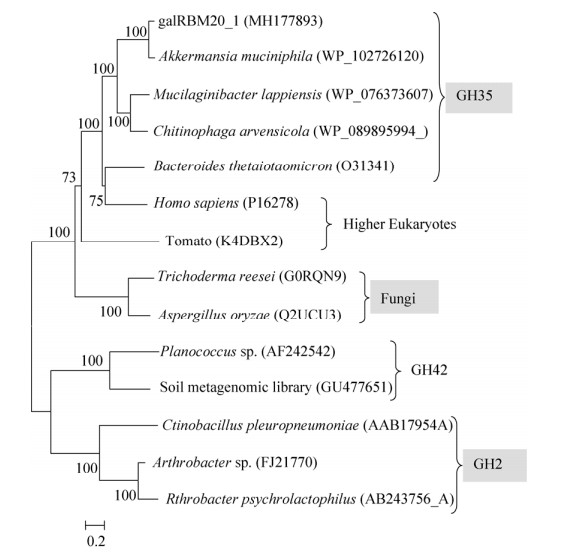 |
| 图 2 galRBM20_1与不同家族来源β-半乳糖苷酶氨基酸序列的系统进化分析 Figure 2 Phylogenetic analysis of the amino acid sequence of galRBM20_1 and β-galactosidase from different family sources. The phylogenetic tree analysis was constructed using the Neighbor-Joining (NJ) method using MEGA 6.0. The number near the branch line indicates the percentage of reliability of the branch in the test (Sequences O31341, P16278, K4DBX2, G0RQN9, Q2UCU3 from UniProt, the rest from GenBank). |
| 图选项 |
2.2 galRBM20_1基因在大肠杆菌的诱导表达及纯化 重组质粒pEASY-E2/galRBM20_1成功转化到大肠杆菌BL-21(DE3)细胞中,重组菌株经IPTG诱导后表达重组蛋白(galRBM20_1)。经离心收集菌体后再经超声波破碎、离心收集粗酶液(上清),该粗酶液经Ni-NTA Agarose树脂纯化后得到的纯酶液用SDS-PAGE检测如图 3,蛋白分子量约71.58 kDa,大小与预测蛋白分子量相符,纯化后的酶液无杂蛋白,达到电泳要求。
 |
| 图 3 β-半乳糖苷酶galRBM20_1的SDS-PAGE分析 Figure 3 SDS-PAGE analysis of β-galactosidase galRBM20_1. Lane M: protein molecular weight marker; lane 1: cell lysate of E. coli harboring empty pEasy-E2 following IPTG induction; lane 2: cell lysate from induced transformant harboring pEASY-E2/galRBM20_1; lane 3: recombinant galRBM20_1 purified protein. |
| 图选项 |
2.3 重组β-半乳糖苷酶galRBM20_1的酶学性质
2.3.1 最适温度和温度稳定性: 根据结果显示(图 4),重组β-半乳糖苷酶galRBM20_1在45 ℃条件下相对酶活力最高(图 4-A),比商业β-半乳糖苷酶(最适温度40 ℃)的最适温度略高。在37-50 ℃之间,该酶的相对酶活均在60 %以上。经37 ℃、45 ℃和50 ℃分别处理0-1 h后发现,在37 ℃和45°下仍能保留100%的活性,经50 ℃处理10 min,剩余酶活为50%,处理20 min,剩余酶活为20% (图 4-B)。
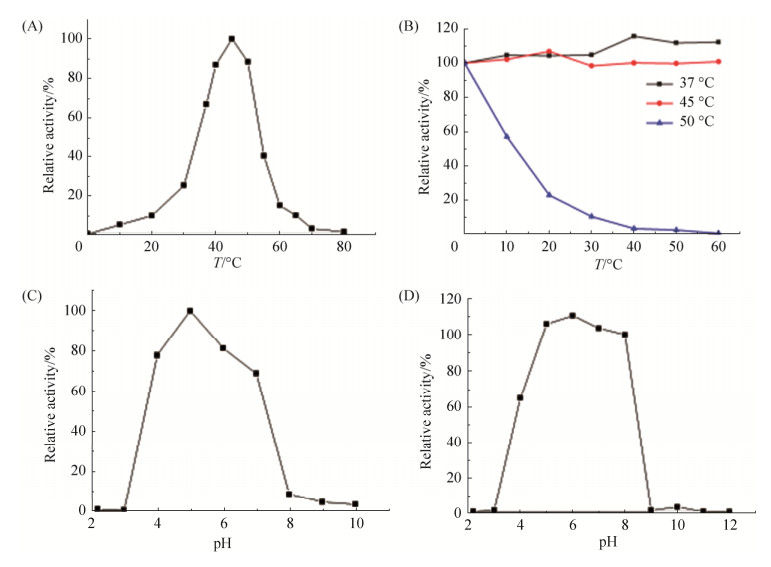 |
| 图 4 温度和pH对β-半乳糖苷酶galRBM20_1的影响 Figure 4 Effect of temperature and pH on the activity of β-galactosidase galRBM20_1. A: Optimum temperature of β-galactosidase galRBM20_1; B: Temperature stability of β-galactosidase galRBM20_1; C: Optimum pH of β-galactosidase galRBM20_1; D: pH stability of β-galactosidase galRBM20_1. |
| 图选项 |
2.3.2 最适pH和pH稳定性: galRBM20_1在pH 5.0时,相对酶活力最高,且pH 4-7仍能保持70%及其以上的活性(图 4-C)。表明在天然牛乳的pH值(pH 6.5)范围内,该酶具有良好的反应潜能。pH 4-8处理1 h后,酶活均在60%以上,其中pH 5-8,酶活均在100%左右(图 4-D)。
2.3.3 底物特异性和动力学参数: β-半乳糖苷酶galRBM20_1对底物oNPG具有较高的比活性,其次是pNPG和乳糖,对其他硝基苯衍生物没有检测到活性(数据未显示)。其水解oNPG、pNPG和乳糖的动力学参数如下(表 1)。
表 1. β-半乳糖苷酶galRBM20_1水解底物oNPG、pNPG和乳糖的动力学参数 Table 1. Kinetic parameters of β-galactosidase galRBM20_1 on substrates oNPG, pNPG and lactose
| Substrate | Vmax/(mmol/min·g) | Km/(mmol/L) | Kcat/(s-1) | Kcat/Km/(1/mmol/L·s) |
| oNPG | 3.41±0.02 | 0.60±0.05 | 224.34±2.45 | 373.01±3.56 |
| pNPG Lactose | 2.03±0.06 4.56±2.2 | 0.86±0.04 36.48±3.0 | 133.55±2.03 300.00±1.89 | 155.24±2.31 8.23±0.79 |
表选项
2.3.4 金属离子和化学试剂的影响: 1 mmol/L的Hg2+、Ag2+、SDS完全抑制β-半乳糖苷酶galRBM20_1的活性,Ag2+对该酶的活性具有很强的抑制作用,Triton X-100对该酶具有很强激活作用(表 2)。与大多数报道一致,大部分二价金属离子对该β-半乳糖苷酶都具有激活作用,Mg2+是生理离子,与β-半乳糖苷酶的效率增加有关(Kcat上升),因此增加了对底物的亲和力(Km下降)[22]。
表 2. 金属离子和化学试剂对重组酶活性的影响 Table 2. Effects of metal ions and chemical agents on enzyme activity
| Ions or chemicals | Relative activity/% |
| Control | 100±0.01 |
| Na+ | 116.31±0.04 |
| K+ | 110.53±0 |
| Mg2+ | 106.74±0.04 |
| Ni2+ | 119.71±0.02 |
| Pb2+ | 104.39±0.02 |
| Co2+ | 109.53±0.05 |
| Cu2+ | 106.4±0.04 |
| Mn2+ | 108.79±0.02 |
| Ca2+ | 77.21±0.04 |
| Fe2+ | 96.61±0.04 |
| β-mercaptoethanol | 109.57±0.03 |
| Zn2+ | 114.27±0.02 |
| Fe3+ | 112.49±0.05 |
| CTAB | 115.05±0.03 |
| EDTA | 106.57±0.02 |
| Tween 80 (0.5% V/V) | 90.65±0.08 |
| Triton X-100 (0.5% V/V) | 159.86 |
| Ag2+ | 35.96±0.04 |
| SDS | 0 |
| Hg2+ | 0 |
表选项
2.4 NaCl浓度的影响和稳定性 当反应体系中NaCl的浓度为1 mol/L时,相对酶活达153%;且NaCl的浓度在0-2 mol/L时,相对酶活均在100%以上;5 mol/L的NaCl的反应体系中,β-半乳糖苷酶galRBM20_1剩余20%酶活(图 5-A)。
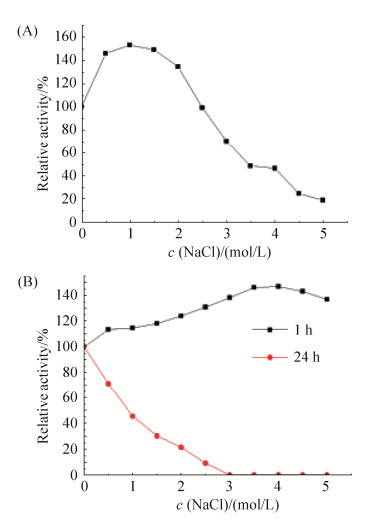 |
| 图 5 NaCl对β-半乳糖苷酶galRBM20_1的影响和稳定性 Figure 5 Effect and stability of NaCl on β-galactosidase galRBM20_1; A: Effect of NaCl on β-galactosidase galRBM20_1; B: NaCl stability of β-galactosidase galRBM20_1. |
| 图选项 |
β-半乳糖苷酶galRBM20_1经浓度为1-5 mol/L的NaCl在45 ℃处理1 h后,其相对酶活均能保持在100%以上,当NaCl的作用浓度为4 mol/L时,相对酶活达到146%的最高值,当NaCl的作用浓度为5 mol/L时,仍有135%的相对酶活(图 5-B)。经浓度为1-5 mol/L的NaCl在45 ℃处理24 h后发现,当NaCl的作用浓度为1 mol/L时,仍具有45%的活性,这些结果表明该酶具有良好的NaCl稳定性。
2.5 水解和转糖基性能测定 反应的混合物经过薄层层析分析显示(图 6-A),当β-半乳糖苷酶galRBM20_1与5% (W/V)的乳糖反应30 min后,部分乳糖已被水解。当反应时长达12 h时,乳糖几乎完全被水解,说明该酶能有效水解乳糖。β-半乳糖苷酶常用于乳制品行业中无乳糖牛奶的生产,故其在无糖牛奶的生产中具有巨大的潜力。
 |
| 图 6 β-半乳糖苷酶galRBM20_1的水解和转糖基活性测定 Figure 6 Hydrolysis and transglycosylation activity of β-galactosidase galRBM20_1. A: TLC analysis of β-galactosidase galRBM20_1 for degradation of lactose. Lane M is a standard mixture of lactose, galactose, and glucose; 1, 2, 3 and 4 are a mixture of reaction for 30 min, 2 h, 8 h and 12 h respectively; B: TLC analysis of β-galactosidase galRBM20_1 transglycosylation activity. Lane M: mixture of lactose, galactose, glucose; lane 1: negative control, 25% (W/V) lactose without beta-galactosidase galRBM20_1; lane 2: reaction mixture of β-galactosidase galRBM20_1 and 25% (W/V) lactose for 24 h. |
| 图选项 |
β-半乳糖苷酶galRBM20_1与25% (W/V)的乳糖反应24 h后,经薄层层析(TLC)分析见部分低聚半乳糖(GOS)产生(图 6-B)。泳道M为乳糖、半乳糖和葡萄糖的标准混合物,泳道1为阴性对照,即高温灭活后的β-半乳糖苷酶galRBM20_1与乳糖的混合反应体系,泳道2为实验组,即β-半乳糖苷酶galRBM20_1与乳糖的混合反应体系。图中可以看出,泳道2的反应体系较泳道1除了含有乳糖以外还有半乳糖、葡萄糖和低聚半乳糖(GOS),表明该酶除了能水解乳糖生成半乳糖和葡萄糖以外,还具有转糖基产GOS的活性,即在乳糖的半乳糖一侧又结合上了1-4个分子的半乳糖。
3 讨论 本研究将滇金丝猴粪便微生物宏基因组来源的β-半乳糖苷酶基因galRBM20_1在大肠杆菌中成功克隆并表达,对纯化后的重组β-半乳糖苷酶galRBM20_1酶学性质研究发现,该酶能较好地将乳糖水解为半乳糖和葡萄糖,其最适作用温度为45 ℃,与来自类芽孢杆菌(Paenibacillus barengoltzii)[23]和盐单胞菌属(Halomonas sp. S62)[24]的β-半乳糖苷酶类似,在50 ℃下耐受10 min具有50%的剩余酶活。其最适pH为5.0,介于真菌中来源β-半乳糖苷酶的最适pH (2.5-5.4)[8],与乳酸曲霉(Aspergillus lacticoffeatus)[25]来源的β-半乳糖苷酶最适pH一致,相比于在大肠杆菌内表达的其他细菌来源的β-半乳糖苷酶(最适pH 7.0-7.5)偏酸。与其他环境微生物宏基因组来源的β-半乳糖苷酶比较发现(表 3),该酶在pH 4-7之间仍能保持70%及以上的活性,说明在天然牛乳的酸碱水平(pH 6.5)范围内能很好发挥其水解作用,适用于水解牛乳和乳清中的乳糖。
表 3. 重组酶galRBM20_1与一些宏基因库和其他微生物来源的β-半乳糖苷酶酶学性质比较 Table 3. Comparison of enzymatic properties of recombinant enzyme galRBM20_1 with some macrogen libraries and other microbial-derived β-galactosidase
| Microbial source | GH family | Opt temp./℃ | Opt pH | Specific of pH | Production GOS | Salt-tolerant (NaCl) | Reference |
| Rhinopithecus bieti fecal microbial metagenome | 35 | 45 | 5.0 | 4–7 has more than 70% activities | GOS | 130% active at 2 mol/L and 40% active at 4 mol/L | This study |
| soil metagenomic library | _ | 50 | 7.0 | 5.0–7.5 has more than 65% activities | GOS | _ | [26] |
| Akkermansia muciniphila | 3–4 has more than 60% activities | ||||||
| 35 | 42 | 3.5 | 6–8 has more than 80% activities | _ | _ | [27] | |
| Soil metagenomic library | 42 | 38 | 7.0 | 5.5–9.5 has more than 80% activities | _ | _ | [28] |
| Hot springs metagenomic library | _ | 55 | 7.5 | 5.0–7.5 has more than 60% activities | GOS | _ | [16] |
| _ | |||||||
| Marine metagenomic library | 1 | 40 | 6.5 | _ | _ | _ | [15] |
| Psychrophilic Planococcus | |||||||
| Wheat straw | 42 | 42 | 6.5 | _ | 0% active at 4 mol/L | [29] | |
| degradant microbial | |||||||
| metagenomic | . | 90% active at 2 mol/L | |||||
| library | 2 | 50 | 7.5 | _ | [17] |
表选项
galRBM20_1与人类胃肠道细菌(Akkermansia Muciniphila)来源的GH35家族β-半乳糖苷酶Am0874的氨基酸序列经ClustalW比对后发现具有74%的相似性(UniProt: O31341),两者最适温度比较接近,galRBM20_1在37 ℃和45 ℃下耐受1 h后酶活未改变,而Am0874经37 ℃和42 ℃耐受1 h后酶活显著降低[27]。虽然两者的来源都是细菌,但最适pH均在真菌来源的β-半乳糖苷酶最适pH范围内,可能是由于两者的原始菌株长期生存于动物胃肠道,为适应复杂的代谢活动而使其基因的功能趋向于更高等的生物(真菌)。在天然牛乳的降解以及工业生产低聚半乳糖(GOS)方面,galRBM20_1更有优良的应用潜力。金属离子及化学试剂的影响有明显的差异,Mg2+对galRBM20_1的活性具有促进作用,而对Am0874具有强力的抑制作用。
此外,已报道的耐盐β-半乳糖苷酶仅来自于小麦秸秆降解物微生物宏基因组和南极嗜冷平板球菌(Planococcus)。对β-半乳糖苷酶galRBM20_1进行耐盐性研究发现,反应环境中NaCl的浓度为2 mol/L时,该酶具有130%的相对酶活,比来自小麦秸秆降解物微生物宏基因组中的β-半乳糖苷酶高出40%。当NaCl的浓度为4 mol/L时,该酶仍然具有40%以上的酶活,比来自于南极嗜冷平板球菌(Planococcus)的耐盐β-半乳糖苷酶高出2倍以上。经5 mol/L的NaCl处理1 h后的β-半乳糖苷酶galRBM20_1的活性较原始酶活(100%)提高到135% (图 5-B),经1 mol/L的NaCl处理24 h后,仍然剩余45%的酶活。相比于其他微生物及宏基因组来源的β-半乳糖苷酶,本实验研究的重组β-半乳糖苷酶galRBM20_1具有良好的耐盐性,在食品工业行业、生物技术领域报告基因[29]等方面具有良好的应用前景。
据已报道的β-半乳糖苷酶,宏基因组来源的糖基转移酶较少[30],尤其是动物胃肠道宏基因组,且具有转糖基活性的β-半乳糖苷酶大多来自第2家族。而本文研究的β-半乳糖苷酶galRBM20_1的基因序列经BLASTn比对后,与收录于NCBI中第35家族的β-半乳糖苷酶基因序列相似性最高,仍然具有转糖基产GOS的活性。
研究发现,乳杆菌来源的β-半乳糖苷酶虽具有水解乳糖和生产低聚半乳糖的优良性质,但在原始菌株内表达量偏低、纯化困难、不易获得大量纯酶,不利于大规模的生产和使用。宏基因组提供了所研究环境样品中所有微生物包括迄今为止尚未被培养的微生物的集体遗传物质,并且是揭示新基因和生物途径的重要途径[31-32]。由分离宏基因组DNA、构建文库和功能驱动筛选产生的文库构成的功能宏基因组学是分离全新和全长酶的唯一策略[32-33]。因此,将β-半乳糖苷酶基因转入基因工程菌中进行异源表达可获得能够用于大规模生产的目的蛋白。本实验利用了pEASY-E2载体和大肠杆菌BL-21(DE3)的原核表达系统,将滇金丝猴粪便微生物宏基因组文库来源的β-半乳糖苷酶基因galRBM20_1成功异源表达。表达出的β-半乳糖苷酶galRBM20_1具有良好水解乳糖和转糖基产GOS的能力,且基因galRBM20_1来源于动物胃肠道,在水解牛奶和生产低乳糖乳制品方面具有良好的安全性和有效性,在乳制品的生产和加工中将具有可观的应用潜力。
References
| [1] | Vasileva N, Ivanov Y, Damyanova S, Kostova I, Godjevargova T. Hydrolysis of whey lactose by immobilized β-galactosidase in a bioreactor with a spirally wound membrane. International Journal of Biological Macromolecules, 2016, 82: 339-346. DOI:10.1016/j.ijbiomac.2015.11.025 |
| [2] | Pereira-Rodriguez á, Fernández-Leiro R, González-Siso MI, Cerdán ME, Becerra M, Sanz-Aparicio J. Structural basis of specificity in tetrameric Kluyveromyces lactis β-galactosidase. Journal of Structural Biology, 2012, 177(2): 392-401. DOI:10.1016/j.jsb.2011.11.031 |
| [3] | Grosová Z, Rosenberg M, Rebro? M. Perspectives and applications of immobilised β-galactosidase in food industry-a review. Czech Journal of Food Sciences, 2008, 26(1): 1-14. DOI:10.17221/1134-CJFS |
| [4] | Sharma SK, Li SH, Micic M, Orbulescu J, Weissbart D, Nakahara H, Shibata O, Leblanc RM. β-galactosidase Langmuir monolayer at air/X-gal subphase interface. The Journal of Physical Chemistry B, 2016, 120(48): 12279-12286. DOI:10.1021/acs.jpcb.6b09020 |
| [5] | Chanalia P, Gandhi D, Attri P, Dhanda S. Purification and characterization of β-galactosidase from probiotic Pediococcus acidilactici and its use in milk lactose hydrolysis and galactooligosaccharide synthesis. Bioorganic Chemistry, 2018, 77: 176-189. DOI:10.1016/j.bioorg.2018.01.006 |
| [6] | Felicilda-Reynaldo RFD, Kenneally M. Digestive enzyme replacement therapy:pancreatic enzymes and lactase. Medsurg Nursing, 2016, 25(3): 182-185. |
| [7] | Harju M, Kallioinen H, Tossavainen O. Lactose hydrolysis and other conversions in dairy products:technological aspects. International Dairy Journal, 2012, 22(2): 104-109. DOI:10.1016/j.idairyj.2011.09.011 |
| [8] | Saqib S, Akram A, Halim SA, Tassaduq R. Sources of β-galactosidase and its applications in food industry. 3 Biotech, 2017, 7(1): 79. |
| [9] | Juajun O, Nguyen TH, Maischberger T, Iqbal S, Haltrich D, Yamabhai M. Cloning, purification, and characterization of β-galactosidase from Bacillus licheniformis DSM 13. Applied Microbiology and Biotechnology, 2011, 89: 645-654. DOI:10.1007/s00253-010-2862-2 |
| [10] | Cheng JJ, Romantsov T, Engel K, Doxey AC, Rose DR, Neufeld JD, Charles TC. Functional metagenomics reveals novel β-galactosidases not predictable from gene sequences. PLoS One, 2017, 12(3): e0172545. DOI:10.1371/journal.pone.0172545 |
| [11] | Gans J, Wolinsky M, Dunbar J. Computational improvements reveal great bacterial diversity and high metal toxicity in soil. Science, 2005, 309(5739): 1387-1390. DOI:10.1126/science.1112665 |
| [12] | Wei DZ, Chen SX, Wang XL, Yuan QS, Yu JT. Properties of β-galactosidase from Bacillus Stearothermophilus. Microbiology China, 2001, 28(1): 18-22. (in Chinese) 魏东芝, 陈少欣, 王筱兰, 袁勤生, 俞俊棠. 嗜热脂肪芽孢杆菌β-半乳糖苷酶的性质. 微生物学通报, 2001, 28(1): 18-22. DOI:10.3969/j.issn.0253-2654.2001.01.006 |
| [13] | Lorenz P, Eck J. Screening for novel industrial biocatalysts. Engineering in Life Sciences, 2004, 4(6): 501-504. DOI:10.1002/elsc.200402159 |
| [14] | Matsuzawa T, Yaoi K. Screening, identification, and characterization of a novel saccharide-stimulated β-glycosidase from a soil metagenomic library. Applied Microbiology and Biotechnology, 2017, 101(2): 633-646. DOI:10.1007/s00253-016-7803-2 |
| [15] | Wierzbicka-Wo? A, Bartasun P, Cie?liński H, Kur J. Cloning and characterization of a novel cold-active glycoside hydrolase family 1 enzyme with β-glucosidase, β-fucosidase and β-galactosidase activities. BMC Biotechnology, 2013, 13: 22. DOI:10.1186/1472-6750-13-22 |
| [16] | Deng J, Zhang Q, Wei YL, Lin LB. A β-galactosidase derived from hot springs metagenome and its characteristics. Food and Fermentation Industries, 2014, 40(5): 60-64. (in Chinese) 邓建, 张琦, 魏云林, 林连兵. 来源于热泉宏基因组的β-半乳糖苷酶及其特征. 食品与发酵工业, 2014, 40(5): 60-64. |
| [17] | Maruthamuthu M, Van Elsas JD. Molecular cloning, expression, and characterization of four novel thermo-alkaliphilic enzymes retrieved from a metagenomic library. Biotechnology for Biofuels, 2017, 10: 142. DOI:10.1186/s13068-017-0808-y |
| [18] | Xu B, Xu WJ, Li JJ, Dai LM, Xiong CY, Tang XH, Yang YJ, Mu YL, Zhou JP, Ding JM, Wu Q, Huang ZX. Metagenomic analysis of the Rhinopithecus bieti fecal microbiome reveals a broad diversity of bacterial and glycoside hydrolase profiles related to lignocellulose degradation. BMC Genomics, 2015, 16: 174. DOI:10.1186/s12864-015-1378-7 |
| [19] | Dai LM, Deng M, Huang ZX, Wang YJ, Li JJ, Zhou JP, Mu YL, Xu B. Gene diversity of the glycosyl hydrolase family 10 xylanase in the fecal microorganism of Rhinopithecus bieti. Microbiology China, 2016, 43(1): 44-50. (in Chinese) 戴利铭, 邓梦, 黄遵锡, 王余娇, 李俊俊, 周峻沛, 穆跃林, 许波. 滇金丝猴粪便微生物GH10家族木聚糖酶基因多样性. 微生物学通报, 2016, 43(1): 44-50. |
| [20] | Xu B, Dai LM, Li JJ, Deng M, Miao HB, Zhou JP, Mu YL, Wu Q, Tang XH, Yang YJ, Ding JM, Han NY, Huang ZX. Molecular and biochemical characterization of a novel xylanase from Massilia sp. RBM26 isolated from the feces of Rhinopithecus bieti. Journal of Microbiology and Biotechnology, 2016, 26(1): 9-19. |
| [21] | 戴利铭.滇金丝猴粪便微生物木聚糖降解酶研究.云南师范大学生命科学学院硕士学位论文, 2016. https://www.ixueshu.com/document/73b30e8ae4beb49905d245eaa5878b66.html |
| [22] | Sutendra G, Wong S, Fraser ME, Huber RE. β-galactosidase (Escherichia coli) has a second catalytically important Mg2+ site. Biochemical and Biophysical Research Communications, 2007, 352(2): 566-570. DOI:10.1016/j.bbrc.2006.11.061 |
| [23] | Liu Y, Chen Z, Jiang ZQ, Yan QJ, Yang SQ. Biochemical characterization of a novel β-galactosidase from Paenibacillus barengoltzii suitable for lactose hydrolysis and galactooligosaccharides synthesis. International Journal of Biological Macromolecules, 2017, 104: 1055-1063. DOI:10.1016/j.ijbiomac.2017.06.073 |
| [24] | Wang GX, Gao Y, Hu B, Lu XL, Liu XY, Jiao BH. A novel cold-adapted β-galactosidase isolated from Halomonas sp. S62:gene cloning, purification and enzymatic characterization. World Journal of Microbiology and Biotechnology, 2013, 29(8): 1473-1480. DOI:10.1007/s11274-013-1311-7 |
| [25] | Cardoso BB, Silvério SC, Abrunhosa L, Teixeira JA, Rodrigues LR. β-galactosidase from Aspergillus lacticoffeatus:a promising biocatalyst for the synthesis of novel prebiotics. International Journal of Food Microbiology, 2017, 257: 67-74. DOI:10.1016/j.ijfoodmicro.2017.06.013 |
| [26] | Wang SD, Guo GS, Li L, Cao LC, Tong L, Ren GH, Liu YH. Identification and characterization of an unusual glycosyltransferase-like enzyme with β-galactosidase activity from a soil metagenomic library. Enzyme and Microbial Technology, 2014, 57: 26-35. DOI:10.1016/j.enzmictec.2014.01.007 |
| [27] | Guo BS, Zheng F, Crouch L, Cai ZP, Wang M, Bolam DN, Liu L, Voglmeir J. Cloning, purification and biochemical characterisation of a GH35 beta-1, 3/beta-1, 6-galactosidase from the mucin-degrading gut bacterium Akkermansia muciniphila. Glycoconjugate Journal, 2018, 35(3): 255-263. DOI:10.1007/s10719-018-9824-9 |
| [28] | Wang K, Li G, Yu SQ, Zhang CT, Liu YH. A novel metagenome-derived β-galactosidase:gene cloning, overexpression, purification and characterization. Applied Microbiology and Biotechnology, 2010, 88(1): 155-165. DOI:10.1007/s00253-010-2744-7 |
| [29] | Sheridan PP, Brenchley JE. Characterization of a salt-tolerant family 42 β-galactosidase from a psychrophilic antarctic Planococcus isolate. Applied and Environmental Microbiology, 2000, 66(6): 2438-2444. DOI:10.1128/AEM.66.6.2438-2444.2000 |
| [30] | Rabausch U, Juergensen J, Ilmberger N, B?hnke S, Fischer S, Schubach B, Schulte M, Streit WR. Functional screening of metagenome and genome libraries for detection of novel flavonoid-modifying enzymes. Applied and Environmental Microbiology, 2013, 79(15): 4551-4563. DOI:10.1128/AEM.01077-13 |
| [31] | Simon C, Daniel R. Achievements and new knowledge unraveled by metagenomic approaches. Applied Microbiology and Biotechnology, 2009, 85(2): 265-276. DOI:10.1007/s00253-009-2233-z |
| [32] | Iqbal HA, Feng ZY, Brady SF. Biocatalysts and small molecule products from metagenomic studies. Current Opinion in Chemical Biology, 2012, 16(1/2): 109-116. |
| [33] | Schmeisser C, Steele H, Streit WR. Metagenomics, biotechnology with non-culturable microbes. Applied Microbiology and Biotechnology, 2007, 75(5): 955-962. DOI:10.1007/s00253-007-0945-5 |
