周怡璇1,2#, 周心童1#, 杨健1, 胡新玲1, 米凯霞1,2


1. 中国科学院微生物研究所, 中国科学院病原微生物与免疫学重点实验室, 北京 100101;
2. 中国科学院大学存济医学院, 北京 101408
收稿日期:2019-01-30;修回日期:2019-03-25;网络出版日期:2019-07-11
基金项目:国家重点研发计划(2018YFC1603900,2017YFA0505901);国家自然科学基金(31600114,31700128)
*通信作者:米凯霞, Tel:+86-10-64806082, Fax:+86-10-64807468, E-mail:mik@im.ac.cn.
#并列第一作者
摘要:[目的] 丝氨酸/苏氨酸蛋白激酶K(Serine/Threonine protein kinases K)是分枝杆菌类似真核样的蛋白激酶,预测在分枝杆菌的生长和新陈代谢等生理过程中起着重要的作用,解析PknK的生物功能及作用机制,将为结核病的防治提供一定的理论基础。[方法] 通过基因敲除等遗传方法获得结核分枝杆菌疫苗株BCG的pknK敲除菌株△pknK、回补菌株pMV361-pknK/△pknK和过表达菌株pMV261-pknK/BCG;对获得的菌株进行生长曲线测定和抗药性分析;通过pulldown-MS方法及生物信息学方法鉴定了PknK相互作用蛋白。[结果] 监测各种分枝杆菌△pknK、pMV361-pknK/△pknK和pMV261-pknK/BCG生长,确定PknK负调控BCG生长;抗药性分析显示PknK降低BCG的耐药性;pulldown-MS方法显示PknK与丝氨酸/苏氨酸蛋白激酶PknA和双组分系统中的反应调节因子MtrA、TrcR、MoxR等蛋白相互作用。[结论] 研究发现PknK调控分枝杆菌的生长和耐药性,我们的研究为深入研究PknK在结核分枝杆菌中的功能奠定了基础。
关键词:丝氨酸/苏氨酸蛋白激酶PknK生长耐药性
Characterization of Serine/Threonine protein kinases K (PknK) in Mycobacterium
Yixuan Zhou1,2#, Xintong Zhou1#, Jian Yang1, Xinling Hu1, Kaixia Mi1,2


1. CAS Key Laboratory of Pathogenic Microbiology and Immunology, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
2. Savaid Medical School, University of Chinese Academy of Sciences, Beijing 101408, China
Received: 30 January 2019; Revised: 25 March 2019; Published online: 11 July 2019
*Corresponding author: Kaixia Mi, Tel:+86-10-64806082, Fax:+86-10-64807468, E-mail:mik@im.ac.cn.
Foundation item: Supported by the Ministry of Science and Technology of China (2018YFC1603900, 2017YFA0505901) and by the National Natural Science Foundation of China (NSFC) (31600114, 31700128)
#These authors contributed equally to this work
Abstract: [Objective] Serine/Threonine Protein Kinases K (PknK) is a eukaryotic-like Ser/Thr protein kinase in Mycobacterium, with important roles in cell growth and signaling transduction. However, its underlying mechanism of action is not completed. [Methods] The pknK knockout strain △pknK was obtained by phage specialized transduction meanwhile the complement strain pMV361-pknK/ΔpknK and the overexpressing strain with pMV261-pknK/BCG was construed for further analysis. The growth curve and resistance of the obtained strains were determined. PknK-interacting proteins were identified by pulldown-MS. [Results] PknK affected Mycobacterium growth, and △pknK had growth advantage over BCG and pMV261-pknK/BCG. Knockout pknK led to increased multiple antibiotic susceptibility. We identified the binding proteins for PnK in BCG using in pulldown combined with Mass Spectrometry. [Conclusion] PknK negatively regulates BCG growth and increases antibiotic susceptibility in mycobacteria. The PknK binding proteins include a Ser/Thr protein kinase PknA, and two component system regulators such as MtrA, MoxR1 and TrcR, which is expected to be a resource for understanding the PknK-mediated signaling pathways in Mycobacterium, thus facilitating new therapeutic strategies for antibiotic-resistant infections.
Keywords: STPKsPknKgrowthantibiotic resistance
结核病(Tuberculosis,TB)是一种由结核分枝杆菌(Mycobacterium tuberculosis,M. tb)引起的慢性传染病,可感染全身多个器官,但是以肺部感染最为常见。根据世界卫生组织(World Health Organization,WHO)全球结核病疫病情况的调查,截止至2017年,全球约有558000例结核病患者,其中82%的患者感染的是耐多药结核病(Multidrug-resistant tuberculosis,MDR-TB)[1]。报告表明,结核病在今天仍然是严重威胁全球公共卫生安全的重大隐患,防痨工作依然任重而道远。
1882年,德国细菌学家科赫首次证实了结核病的病原菌是结核分枝杆菌。1921年,针对结核病的疫苗——卡介苗(Mycobacterium bovis Bacillus Calmette-Guerin,BCG)开始在全球广泛使用,对幼儿的播散性结核病和肺结核病具有良好的保护作用[2]。自1944年起,链霉素、异烟肼、吡嗪酰胺、乙胺丁醇以及利福平的先后发现,开启了抗生素药物的“黄金时代”[3]。然而,随着越来越多药物的广泛应用,结核分枝杆菌对这些药物的耐受也在不断增加,并随之出现了能够耐受多种药物的耐多药结核病以及一些具有广泛耐药能力的广泛耐药结核病(Extensively drug-resistant tuberculosis,XDR-TB)[4]。传统的药物在耐多药结核的治疗中疗效不佳,药物研发受限于结核相关基础研究的匮乏。因此加深结核基础研究,将为结核病治疗提供新的思路及理论基础。
在原核生物中磷酸化的信号级联反应广泛存在。丝氨酸/苏氨酸蛋白激酶(Serine/Threonine Protein Kinases,STPKs)是原核生物体一类重要的激酶[5]。STPKs通过磷酸化细胞内的底物蛋白,调节大多数信号转导通路,对细胞的增殖和分化起着重要的控制作用[6-7]。在结核分枝杆菌中,STPKs共有11种,根据序列相似程度分为5个类型,分别是Clade Ⅰ (PknA,PknB,PknL)、Clade Ⅱ (PknD,PknE,PknH)、Clade Ⅲ (PknF,PknI,PknJ)、Clade Ⅳ (PknG)和Clade Ⅴ (PknK)[8]。PknK是结核分枝杆菌中最大的STPK,其生物功能还有待发掘。目前对于PknK的研究主要集中在蛋白组数据的分析,组学的分析结果显示PknK不是分泌蛋白[9],而是存在于结核分枝杆菌的细胞壁、细胞质及细胞膜上[10]。其存在的位置预示PknK在细菌内部的信号转导中起着重要的作用[11-12]。一项研究显示PknK在调控分枝杆菌的生长中起着重要的作用[13]。但是,目前还缺乏系统性理解PknK在分枝杆菌生长等功能中发挥的作用。
分枝杆菌疫苗株BCG中PknK与结核分枝杆菌H37Rv中的PknK序列相同,因此本研究以分枝杆菌疫苗株BCG为研究材料,利用噬菌体介导的同源重组技术敲除pknK基因,敲除菌株△pknK的生长优于野生型BCG,敲除菌株△pknK对检测的抗生素利福平、异烟肼、环丙沙星、红霉素的耐受程度明显下降。由于PknK的功能都是通过与其相互作用的蛋白发挥作用,利用pulldown-MS方法,我们获得了PknK相互作用候选蛋白,包括丝氨酸/苏氨酸蛋白激酶PknA、双组分系统中的反应调节因子、膜相关蛋白等,PknK通过与这些蛋白的相互作用,从而调控细菌生长和耐药性。本研究为深入探讨PknK在结核分枝杆菌中的功能奠定了基础。
1 材料和方法 1.1 材料
1.1.1 菌株、细胞株、质粒和培养基: 牛型结核分枝杆菌和大肠杆菌(Escherichia coli) DH5α感受态细胞、BL21(DE3)感受态细胞,人单核巨噬细胞系THP-1细胞株,质粒p0004s、phAE159、pMV261、pMV361、pET23b、pGEX-5x。BCG培养所用培养基为7H9 (液体,含10% ADS与0.05% Tween 80)和7H11 (固体,含10% ADS)。LB培养基用于大肠杆菌培养。
1.1.2 主要仪器和试剂: PCR仪(北京东胜创新生物科技有限公司),EPS-300电泳仪、HE-120多功能水平电泳槽、VE 180微型垂直电泳槽、Tanon-5200全自动化学发光处理系统(上海天能科技有限公司),凝胶图像分析系统(DNR Bio-imaging System公司),ECM399电转仪(BTX公司),752紫外可见分光光度计(上海菁华科技有限公司),EASY-nLCIITM-nano-HPLC系统(PROXEON),LTQ-Orbitrap质谱仪(Thermo),HCl、NaCl、NaOH、葡萄糖、Na2HPO4·12H2O、KH2PO4、KCl、EDTA、DTT、IAM、TFA (国药集团化学试剂有限公司),冰醋酸、异丙醇、无水乙醇、甲醇、甘油(北京化工厂),胰蛋白胨、酵母提取物(OXOID),琼脂粉、Tween-80、Tween-20、DMSO、利福平(Sigma),卡那霉素、潮霉素、异烟肼、环丙沙星(BIO BASIC INC),红霉素、PMSF、HEPES、IPTG、SDS(AMRESCO),琼脂糖(Biowest),7H9、7H11(Becton Dickinson),GoodView DNA染料(上海赛百盛基因),T4 DNA连接酶、Q5 DNA聚合酶(NEB),Blue Plus Protein Marker、Trans2K Plus DNA Marker (全式金生物技术有限公司),2×Power Taq PCR MasterMix (北京百泰克生物技术有限公司),Tris、甘氨酸(NOVON),限制性内切酶:Van91 Ⅰ、Pac Ⅰ、BamH Ⅰ、Hind Ⅲ、EcoR Ⅰ、Hpa Ⅰ、Not I、Hpa Ⅰ (Thermo),GST、Ni-NAT Agarose (Qiagen),抗体His-Tag Mouse mAb、HRP标羊抗小鼠IgG二抗、鼠抗GST标签单克隆抗体(普利莱生物技术有限公司)。
1.2 敲除菌株的构建
1.2.1 敲除载体的构建和噬菌体的扩增: 提取BCG的基因组,并以该基因组为模板,通过PCR扩增pknK的左右臂,左臂引物是pknK-LL和pknK-LR;右臂引物是pknK-RL和pknK-RR (表 1)。使用限制性内切酶Van91 Ⅰ对扩增产物pknK-L (左臂)和pknK-R (右臂)进行酶切,回收酶切产物,定向克隆至中间载体p0004S上,并转化DH5α感受态细胞,LB培养基(潮霉素100 mg/L)培养,挑选单菌落提质粒测序,进而获得测序正确的载体p0004-pknK。使用Pac Ⅰ分别对p0004-pknK和phAE159进行酶切,回收酶切产物并连接,将体外包装获得的完整质粒转化至HB101感受态中,LB培养基(潮霉素100 mg/L)培养,最终获得正确的多元件穿梭载体phAE159-0004-pknK-L/R。将phAE159-0004-pknK-L/R电击转化耻垢分枝杆菌标准菌株(mc2 155),7H11平板上(潮霉素75 mg/L) 30 ℃恒温培养获得噬菌斑,扩增噬菌体,提高滴度至1010/mL。
表 1. 实验用引物汇总表 Table 1. Oligonucleotide primers used in this study
| Primers | Sequences (5′→3′) | Purposes |
| pknK-LL | TTTTTTTTCACAAAGTGTGACACACACTCCATCGCTC | Clone the left arm of pknK |
| pknK-LR | TTTTTTTTCACTTCGTGATCCAGCCTCAAG CAGTTCC | |
| pknK-RL | TTTTTTTTCACAGAGTGTCTTGCAGCCACAAGACGAT | Clone the right arm of pknK |
| pknK-RR | TTTTTTTTCACCTTGTGGGCCTGAAGCTTGTCCAGAT | |
| pknK-InL | CTGCTTTGCGCTTCGATACC | To confirm pknK knocked out |
| pknK-InR | TCTGCACGATTGCCAGAAGT | |
| pknK-LLL | CGGCAGTATCGAGCCTATGG | To confirm pknK knocked out |
| pknK-RRR | CCTGTGGAAACTGGTGCGTA | |
| I-R(F) | GACACACCAACAGCATGGT | To confirm pknK knocked out |
| I-L(R) | TCGACGACCCTAGAGTCC | |
| pknK-F | CCCAAGCTTTGACCGACGTTGATCCGCACGCG | To clone pknK fragment |
| pknK-R | GCTCAATTGCAAGATCTGCACGACGATACG | |
| pET23b-pknK-F | CCCAAGCTTACCGACGTTGATCCGCACGCG | To clone pknK fragment |
| pET23b-pknK-R | ATTTGCGGCCGCGGCCAGTCCCGCATCGACCAG | |
| pknA-F | CCGGAATTCAGCCCCCGAGTTGGCGTGAC | To clone pknA fragment |
| pknA-R | CGCGGATCCTCATTGCGCTATCTCGTATCGGG |
表选项
1.2.2 基因敲除: 接种BCG至7H9培养基中,37 ℃摇床培养至对数期(OD600为0.6-0.8),用MP Buffer清洗菌体2次,再用7H9培养基重悬,并与之前得到的噬菌体混匀,37 ℃恒温箱中培养24 h,全部均匀涂布在7H11平板上(潮霉素75 mg/L),7H9培养基(潮霉素75 mg/L) 37 ℃恒温培养2-3周。挑选单克隆鉴定。
1.2.3 敲除菌株的验证: 分别设计3对鉴定引物(表 1),即目的基因内部引物、目的基因左右臂远端引物和载体内部引物,并通过PCR验证目的基因是否被敲除(图 1)。若目的基因被正确敲除,则内部引物pknK-InL和pknK-InR不能成功扩增,同时远端引物和载体内部引物能进一步确认载体片段是否插入正确部位。
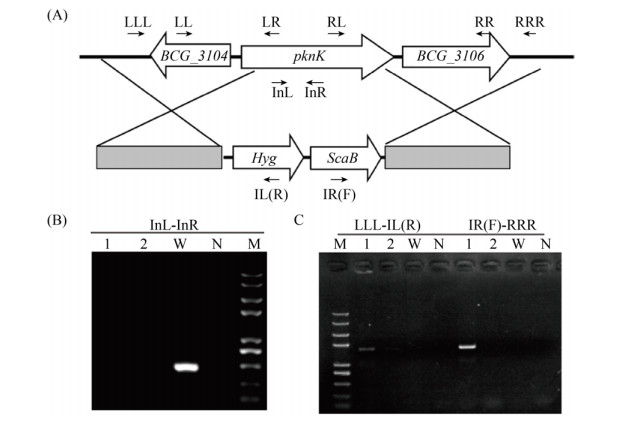 |
| 图 1 敲除菌株的构建模型及鉴定图谱 Figure 1 Generation of the △pknK strain. A: Genomic organization of the pknK gene locus. Genes are shown as large arrows in their orientation. The small arrows show the primer pairs for PCR. B: The location of hygromycin cassette is indicated (bottom panel). No PCR product was obtained using primers InL and InR to amplify the coding sequences of pknK. C: The PCR products of the upstream and downstream regions of pknK were amplified using the primer pairs LLL/IL(R) and IR(F)/RRR, respectively. |
| 图选项 |
1.2.4 回补菌株的构建: 以BCG基因组为模板,PCR实验扩增目的基因pknK,引物(表 1)为pknK-F和pknK-R。使用限制性内切酶Hpa Ⅰ和Hind Ⅲ对扩增产物和载体pMV361进行酶切,并连接酶切产物,转化DH5α感受态细胞,LB培养基(卡那霉素50 mg/L)培养。挑选单菌落进行基因测序,将测序正确的pMV361-pknK电击转入△pknK感受态细胞中,得到回补菌株pMV361-pknK/△pknK。
1.3 药物胁迫实验 培养野生型菌株BCG、敲除菌株△pknK至OD600为0.8-1.0,将上述菌液转接于新鲜培养基中,调节OD600至0.1,加入抗结核一、二线药物利福平(Rifampicin)、异烟肼(Isoniazid)、环丙沙星(Ciprofloxacin)、红霉素(Erythromycin),进行药物处理,同时设立无药物处理对照组。37 ℃培养72 h,取药物处理组和对照组菌液测OD600。将药物处理组结果与对照组结果进行对比,二者比值越大,则菌株对该药物的耐药性越大,反之则耐药性越小。
1.4 过表达菌株的构建 以BCG基因组为模板,PCR实验扩增目的基因pknK,引物(表 1)为pknK-F和pknK-R。使用限制性内切酶Hpa Ⅰ和Hind Ⅲ对扩增产物和载体pMV261进行酶切,并连接酶切产物,转化DH5α感受态细胞,LB培养基(卡那霉素50 mg/L)培养。挑选单菌落测序,将测序正确pMV261-pknK与pMV261分别电击转入BCG感受态细胞中。挑选单菌落,并进行PCR验证,得到过表达菌株pMV261-pknK/BCG及对照菌株pMV261/BCG。
1.5 生长曲线测定 接种野生型BCG、敲除菌株△pknK、回补菌株pMV361-pknK/△pknK、过表达菌株pMV261- pknK/BCG及对照菌株pMV261/BCG至7H9培养基中,每个菌株至少3组平行,37 ℃摇床培养至对数期(OD600为0.6-0.8)。分别调OD600达到0.1,37 ℃摇床培养,每隔12 h取样测1次OD600值,记录实验数据,绘制生长曲线。
1.6 pulldown-MS实验
1.6.1 构建PknK表达菌株: 以BCG基因组为模板,利用PCR扩增目的基因pknK,引物(表 1)为pET23b-pknK-F和pET23b-pknK-R。使用限制性内切酶Not I和Hind Ⅲ对扩增产物和载体pET23b进行酶切,并连接酶切产物,转化DH5α感受态细胞,LB培养基(氨苄青霉素100 mg/L)培养。挑选单菌落测序,将测序正确pET23b-pknK电击转入BL21(DE3)感受态细胞中,得到PknK的表达菌株。
1.6.2 体外pulldown: 将PknK表达菌株进行转接,并进行异丙基硫代半乳糖苷(IPTG,0.5 mmol/L)诱导表达。低温收菌、冰上超声、低温离心,收取上清与His-beads混合孵育,然后用磷酸盐缓冲液冲洗,得到PknK和His-beads的混合物。同时收集生长至对数期的BCG菌体,裂解菌体获得总蛋白样品,与PknK-His-beads的混合物进行孵育、清洗、洗脱,将洗脱液进行SDS-PAGE,进行银染。与对照蛋白对比,选取特异性条带切胶,并进行质谱(Mass spectrometry,MS)检测。
1.6.3 质谱检测: 将切下的胶体用洗脱剂去除染料颜色,然后用二硫苏糖醇(DL-Dithiothreitol,DTT,25 mmol/L)还原蛋白质,并用碘乙酰胺(55 mmol/L)烷基化蛋白质。之后加入胰酶,置于37 ℃温箱中,在碳酸氢铵(50 mmol/L)中过夜消化。用1%的三氟乙酸在50%的乙腈水溶液中提取多肽2次,用真空离心蒸发浓缩器浓缩,0.1%甲酸的缓冲液中复溶。以0.25 μL/min的流速流经EASY-nLCIITM-nano-HPLC系统,该系统直接与LTQ-Orbitrap质谱仪相连。首先使用Xcalibur 2.0.7软件对数据进行采集,然后使用Proteome Discover软件对原始数据文件进行处理,生成峰值列表文件。实验独立重复3次,用于MS检索的蛋白数据库从UniProt下载。
1.7 体外蛋白互作验证
1.7.1 构建PknA表达菌株: 以BCG基因组为模板,使用PCR扩增目的基因pknA,引物(表 1)为pknA-F和pknA-R。使用限制性内切酶EcoR Ⅰ和Hpa Ⅰ对扩增产物和载体pGEX-5x进行酶切,连接酶切产物并转化DH5α感受态细胞,LB培养基(氨苄青霉素100 mg/L)培养。挑取单菌落测序,将测序正确的pGEX-5x-pknA热激转化BL21(DE3)感受态细胞,得到PknA表达菌株。
1.7.2 体外pulldown实验: IPTG (0.5 mmol/L)低温诱导PknK表达菌株,利用His-beads纯化得到PknK蛋白;IPTG (0.5 mmol/L)低温诱导PknA表达菌株,利用GST-beads纯化得到PknA蛋白。将二者按摩尔浓度1:1混合,在4 ℃共孵育1 h,然后将混合液通过GST-beads。收取洗脱液进行SDS-PAGE检测,并利用抗His特异性抗体进行Western blotting检测。
2 结果和分析 2.1 分枝杆菌pknK敲除菌株的构建 首先,我们利用分枝杆菌噬菌体特异转导法(Mycobacteriophages specialized transduction)[13]构建候选基因的敲除菌株(图 1-A)。随机挑选2个克隆(分别标注1、2)培养并提取基因组DNA,通过特异性PCR扩增进行验证:利用引物InL/InR检测pknK基因内部片段,在野生型BCG菌株中可以检测到PCR产物,在pknK敲除菌株中没有检测到PCR产物(图 1-B)。利用载体上的引物和pknK左右臂远端引物进行扩增,样品1获得了PCR产物,说明潮霉素基因置换了pknK,基因敲除成功(图 1-C),我们获得了敲除菌株△pknK。
2.2 PknK负调控BCG生长 获得敲除菌株△pknK后,我们首先检测了PknK对细菌的生长是否有影响。△pknK和BCG接种于7H9培养基中培养,每12 h取样测定OD600值。在前期48 h,△pknK生长比BCG稍微有生长优势;在后期,与野生型BCG比,敲除菌株△pknK有明显的生长优势;回补菌株pMV361-pknK/△pknK可以部分回补PknK介导的快速生长,其生长速度介于两者之间(图 2-A)。同时,过度表达pknK菌株pMV261-pknK/BCG的生长速度低于其对照菌株pMV261/BCG (图 2-B)。生长曲线检测结果说明PknK负调控BCG生长。
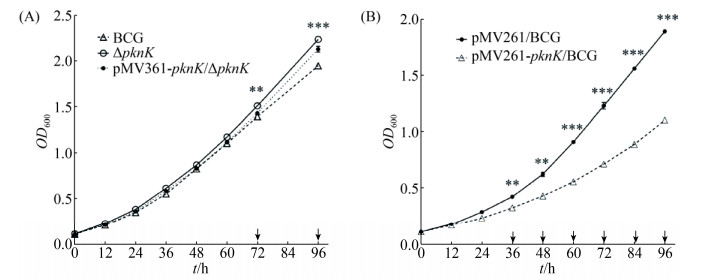 |
| 图 2 PknK负调控BCG的生长 Figure 2 PknK negatively regulate BCG growth. A: The mycobacterial strains, BCG, △pknK and pMV361-pknK/△pknK, were incubated in 7H9 medium and the growth rates were determined. B: The mycobacterial strains, pMV261-pknK/BCG and pMV261/BCG were growth in 7H9 medium with kanamycin. Representative growth curves are shown. Data are shown as the mean±SD of three replicates. A significant difference in the OD600 was observed between the strain pair △pknK and BCG or pMV261-pknK/BCG and pMV261/BCG as pointed by arrow (two-sided student's t-test, **: P < 0.01, ***: P < 0.001). Data represent the results of three independent experiments. |
| 图选项 |
2.3 PknK降低BCG耐药性 细菌的生长状态与药物的作用密切相关。由于PknK负调控BCG生长,PknK可能会影响BCG的药物耐受性,因此我们检测了野生型菌株BCG和敲除菌株△pknK对抗结核药物的耐药性。检测的药物包括一线药物利福平和异烟肼,治疗多耐结核药物红霉素和环丙沙星。因为PknK本身影响细菌的生长,为了分析PknK对BCG药物敏感性的影响,我们采用药物处理OD600值与非药物处理组OD600值的比例作为评估指标。调节菌液的OD600到0.1时,把培养物分为2份,一份添加药物,继续培养72 h,检测OD600值(处理组,treatment group);同时,另一份培养物,在没有药物处理的条件下培养,72 h后检测OD600值(对照组,control group),处理组的OD600值与对照组的OD600值比值显示药物处理对菌株生长的影响。图 3结果显示,敲除菌株△pknK对利福平、异烟肼、环丙沙星、红霉素的耐受程度明显下降。该结果显示,PknK减低BCG耐药性。
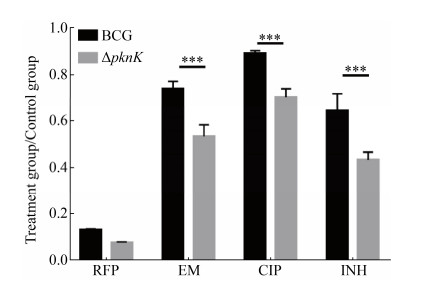 |
| 图 3 PknK降低BCG的耐药性 Figure 3 PknK decreases antibiotic susceptibility in mycobacteria. The ratio of the OD600 of the treatment group to the OD600 of the control group indicates the antibiotic susceptibility in mycobacteria. The results are presented as the mean±SD of three replicates. A significant difference in ratio was observed between △pknK and BCG (two-sided Student's t-test, ***: P < 0.001). Data represent the results of three independent experiments. RFP: rifampicin; EM: erythromycin; CIP: ciprofloxacin; INH: isoniazid. |
| 图选项 |
2.4 系统分析PknK相互作用的蛋白 为了进一步了解PknK的生物功能,我们利用pulldown-MS的方法筛选与PknK蛋白相互作用的候选蛋白(图 4-A)。为了获得直接与PknK相互作用的蛋白,我们在大肠杆菌中表达了PknK蛋白,与Ni-NTA Beads孵育,用磷酸盐缓冲液冲洗获得Ni-NTA beads-PknK后,与BCG裂解物孵育,清洗后SDS-PAGE分离复合物。因为银染的敏感度比较高,可能会显示出部分非特异性条带,因此用于PknK功能不相关的蛋白进行对照以排除非特异性蛋白条带。MSMEG_2415是一种蚯蚓血红蛋白,MSMEG_1641是一种五肽重复序列蛋白,都不是PknK的底物[5, 11],因此作为负对照。表达、纯化MSMEG_2415、MSMEG_1641和PknK蛋白。银染的SDS-PAGE中,红色箭头分别标记靶蛋白:MSMEG_2415、PknK和MSMEG_1641;黑色箭头所示为PknK经过体外pulldown后的特异条带,切割特异条带胶体进行质谱分析(图 4-B)。经过3次独立实验,我们筛选了部分score值大于10、Unique Peptides大于2的蛋白数据,按score大小进行排序(表 2),得到了多个在体外可能与PknK结合的蛋白质。获得的PknK相互作用的候选蛋白与细菌生长、转录调控等功能相关,包括丝氨酸/苏氨酸蛋白激酶PknA、双组分系统中的反应调节因子、膜相关蛋白等(表 2)。
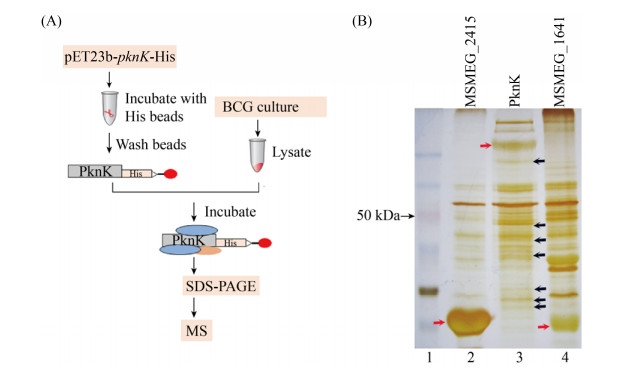 |
| 图 4 Pulldown-MS方法鉴定PknK互作蛋白 Figure 4 Identification of PknK interacting proteins using the pulldown-MS assay. A: The workflow for the PknK pulldown-MS assay. B: Capture and enrichment of specific binding proteins by bait proteins. Lane 1, molecular weight marker proteins. MSMEG_2415 (lane 2), PknK (lane 3), MSMEG_1641 (lane 4) are corresponding bait proteins, respectively. The corresponding pulldown proteins were separated by SDS-PAGE gel and visualized by silver staining. The red arrows indicate the bait proteins. The black arrows indicate the PknK interacting proteins pulldown by PknK. |
| 图选项 |
表 2. PknK pulldown-MS方法鉴定的PknK相互作用候选蛋白 Table 2. PknK interacting proteins identified by PknK pulldown-MS
| Accession | Gene name | Description | Score | Coverage | Unique peptides | Function |
| A0A0H3M1M2 | BCG_0505c | Probable transcriptional regulatory protein | 902.78 | 67.09 | 29 | Transcription |
| A1KKE3 | pafA | Proteasome accessory factor A | 492.99 | 61.07 | 25 | Proteasome system |
| A0A0H3M2C1 | coaX | Type Ⅲ pantothenate kinase | 283.76 | 48.53 | 10 | Kinase |
| A0A0H3MBY6 | BCG_3824c | Putative two component transcriptional regulatory protein | 98.21 | 47.86 | 6 | Transcription |
| A0A0H3MAR7 | mtrA | Two component sensory transduction transcriptional regulatory protein mtrA | 95.04 | 50.88 | 8 | Transcription |
| A0A0H3M2D6 | BCG_0074c | Probable transcriptional regulatory protein | 78.43 | 43.03 | 8 | Transcription |
| A0A0H3M3T1 | trcR | Two-component system regulator trcR | 42.43 | 28.40 | 5 | Transcription |
| A0A0H3M8I4 | pfkA | ATP-dependent 6-phosphofructokinase | 39.25 | 24.49 | 6 | Kinase |
| A0A0Y1BRF1 | BCG_1541 | Transcriptional regulator MoxR1 | 30.04 | 29.30 | 6 | Transcription |
| A0A0H3M2C1 | pknA | Transmembrane serine/threonine-protein kinase A | 17.67 | 22.05 | 7 | Kinase |
| A0A0H3M0V2 | BCG_0215 | Probable conserved MCE associated membrane protein | 14.06 | 15.57 | 2 | Membrane |
表选项
在质谱分析筛选出的靶标蛋白中,我们挑选了PknA与PknK进行了体外pulldown实验验证。首先,我们构建了PknA的表达质粒pGEX-5x-pknA,并转化得到表达菌株。纯化的GST-PknA与PknK-His混合孵育,用GST-tag进行pulldown,收集洗脱液并进行Western blotting检测。结果显示,GST-PknA与PknK-His共孵育时,最终的洗脱液能够被His抗体检测到,即带有GST-tag的PknA能够与PknK结合,并使一同被洗脱下来的携带有His-tag的PknK被His抗体检测到;而GST与PknK-His共孵育时,最终的洗脱液不能被His抗体检测到,即单独表达的GST无法与PknK结合(图 5-A)。同时,洗脱液中的GST-PknA及GST被GST抗体检测到,而PknK-His不能被检测到,说明实验系统是有效的(图 5-B)。该实验证明,在体外条件下,PknK可以与PknA蛋白进行相互作用,所以PknK可能是通过与PknA等蛋白相互作用从而影响细菌的生长(图 6)。
 |
| 图 5 PknA与PknK相互作用的体外验证 Figure 5 PknK interacts with PknA. Examination of the interaction between PknK (A) and PknA (B) using affinity tagged pulldown assays. The purified proteins were obtained from BL21(DE3) cells expressing GST-PknA or PknK-His. The corresponding purified proteins were diluted 100-fold and 2 μL of the diluted sample were loaded as the input. The interactions were examined by Western blotting using an antibody against GST (for PknA) or His (for PknK). Data represent the results of three independent experiments. |
| 图选项 |
 |
| 图 6 PknK参与通路的模式图 Figure 6 Model of the effect of PknK on the growth and antibiotic susceptibility in mycobacteria. |
| 图选项 |
3 讨论 丝氨酸/苏氨酸蛋白激酶K(Serine/Threonine protein kinases K,PknK)是分枝杆菌类似真核样的蛋白激酶,在分枝杆菌的生长和新陈代谢等生理过程中起着重要的作用[5, 14-18]。由于STPKs在分枝杆菌的生长及在细菌感染的宿主中存活起着重要的作用,它们是潜在的药物靶点[6, 19]。PknK是分枝杆菌丝氨酸/苏氨酸蛋白激酶家族中重要的成员之一,目前关于其功能的研究尚不全面。本研究显示,PknK对BCG的生长具有负调控作用;同时,敲除pknK基因会导致多重抗生素耐药性,说明PknK在分枝杆菌对多种抗生素耐药中起着重要作用。利用pulldown-MS方法,获得与PknK相互作用的候选蛋白,这些候选蛋白与细菌生长、转录调控等功能相关。我们的工作为深入研究PknK在分枝杆菌的分子调控机制奠定了基础。
我们的研究与之前的报道一致[20],PknK调控分枝杆菌的生长。研究显示敲除pknK基因的BCG菌株(△pknK)在生长速度方面明显快于野生型BCG,回补菌株可以部分回补减低生长速度(图 2-A)。过表达菌株pMV261-pknK/BCG的生长速度要明显慢于对照组菌株(图 2-B)。分枝杆菌的抗生素敏感性与细菌生长状态相关[21-22],敲除菌株△pknK对利福平、红霉素、环丙沙星及异烟肼的耐受性明显下降(图 3)。我们的结果显示,敲除pknK基因的BCG菌株(△pknK)生长速度比野生型BCG生长速度快,导致多重抗生素易感性增加。进一步的研究需要解决介导PknK功能在抗生素耐药中的具体分子机制。
磷酸化在细菌的信号转导中起着重要的作用。在分枝杆菌中,存在双组分系统和真核样的蛋白激酶通过磷酸化进行细菌信号的传递。为了更加系统地了解PknK在分枝杆菌生长和耐药中发挥的作用,我们利用pulldown-MS方法,筛选获得与PknK相互作用的候选蛋白(表 2)。生物信息学分析表明,双组分系统中的反应调节因子及PknA与PknK相互作用。PknA属于丝氨酸/苏氨酸蛋白激酶家族[17],其通过磷酸化参与了多种信号通路,包括Ⅱ型脂肪酸合成系统[23]、细胞形态塑造[15]、细胞伸长[16]及细胞分裂[18]等多种通路,影响分枝杆菌的生长速度。最近的研究显示,磷酸化调节MtrA与DNA的结合,调控MtrA操纵子的基因转录,调控分枝杆菌的耐药性[24-25]。我们的结果提示双组分系统中的反应调节因子MtrA、TrcR、MoxR及PknA可被PknK通过相互作用和磷酸化进行反馈调节,从而调控分枝杆菌的生长和耐药性(图 6)。我们的结果也揭示PknK是一个潜在的药物靶点。今后,对PknK如何对相互作用蛋白进行磷酸化、信号传导的深入研究,将会加深分枝杆菌的生长和耐药的分子调控机制的理解,为结核病的治疗提供新的思路及理论基础。
References
| [1] | WHO. Global tuberculosis report 2018. Geneva: World Health Organization, 2018. |
| [2] | Mangtani P, Abubakar I, Ariti C, Beynon R, Pimpin L, Fine PEM, Rodrigues LC, Smith PG, Lipman M, Whiting PF, Sterne JA. Protection by BCG vaccine against tuberculosis: a systematic review of randomized controlled trials. Clinical Infectious Diseases, 2014, 58(4): 470-480. DOI:10.1093/cid/cit790 |
| [3] | Miku?ová K, Ekins S. Learning from the past for TB drug discovery in the future. Drug Discovery Today, 2017, 22(3): 534-545. DOI:10.1016/j.drudis.2016.09.025 |
| [4] | Seung KJ, Keshavjee S, Rich ML. Multidrug-resistant Tuberculosis and extensively drug-resistant Tuberculosis. Cold Spring Harbor Perspectives in Medicine, 2015, 5(9): a017863. DOI:10.1101/cshperspect.a017863 |
| [5] | Wu FL, Liu Y, Jiang HW, Luan YZ, Zhang HN, He X, Xu ZW, Hou JL, Ji LY, Xie Z, Czajkowsky DM, Yan W, Deng JY, Bi LJ, Zhang XE, Tao SC. The Ser/Thr protein kinase protein-protein interaction map of M. tuberculosis. Molecular & Cellular Proteomics, 2017, 16(8): 1491-1506. |
| [6] | Caballero J, Morales-Bayuelo A, Navarro-Retamal C. Mycobacterium tuberculosis serine/threonine protein kinases: structural information for the design of their specific ATP-competitive inhibitors. Journal of Computer-Aided Molecular Design, 2018, 32(12): 1315-1336. DOI:10.1007/s10822-018-0173-3 |
| [7] | Prisic S, Husson RN. Mycobacterium tuberculosis serine/threonine protein kinases. Microbiology Spectrum, 2014, 2(5). DOI:10.1128/microbiolspec.MGM2-0006-2013 |
| [8] | Narayan A, Sachdeva P, Sharma K, Saini AK, Tyagi AK, Singh Y. Serine threonine protein kinases of mycobacterial genus: phylogeny to function. Physiological Genomics, 2007, 29(1): 66-75. DOI:10.1152/physiolgenomics.00221.2006 |
| [9] | De Souza GA, Leversen NA, M?len H, Wiker HG. Bacterial proteins with cleaved or uncleaved signal peptides of the general secretory pathway. Journal of Proteomics, 2011, 75(2): 502-510. DOI:10.1016/j.jprot.2011.08.016 |
| [10] | Mawuenyega KG, Forst CV, Dobos KM, Belisle JT, Chen J, Bradbury EM, Bradbury ARM, Chen X. Mycobacterium tuberculosis functional network analysis by global subcellular protein profiling. Molecular Biology of the Cell, 2005, 16(1): 396-404. DOI:10.1091/mbc.e04-04-0329 |
| [11] | Kumar P, Kumar D, Parikh A, Rananaware D, Gupta M, Singh Y, Nandicoori VK. The Mycobacterium tuberculosis protein kinase K modulates activation of transcription from the promoter of mycobacterial monooxygenase operon through phosphorylation of the transcriptional regulator VirS. Journal of Biological Chemistry, 2009, 284(17): 11090-11099. DOI:10.1074/jbc.M808705200 |
| [12] | Malhotra V, Okon BP, Clark-Curtiss JE. Mycobacterium tuberculosis protein kinase K enables growth adaptation through translation control. Journal of Bacteriology, 2012, 194(16): 4184-4196. DOI:10.1128/JB.00585-12 |
| [13] | Bardarov S, Bardarov Jr S, Pavelka Jr MS, Sambandamurthy V, Larsen M, Tufariello J, Chan J, Hatfull G, Jacobs Jr WR. Specialized transduction: an efficient method for generating marked and unmarked targeted gene disruptions in Mycobacterium tuberculosis, M. bovis BCG and M. smegmatis. Microbiology, 2002, 148(10): 3007-3017. DOI:10.1099/00221287-148-10-3007 |
| [14] | Chopra P, Singh B, Singh R, Vohra R, Koul A, Meena LS, Koduri H, Ghildiyal M, Deol P, Das TK, Tyagi AK, Singh Y. Phosphoprotein phosphatase of Mycobacterium tuberculosis dephosphorylates serine-threonine kinases PknA and PknB. Biochemical and Biophysical Research Communications, 2003, 311(1): 112-120. DOI:10.1016/j.bbrc.2003.09.173 |
| [15] | Kang CM, Abbott DW, Park ST, Dascher CC, Cantley LC, Husson RN. The Mycobacterium tuberculosis serine/threonine kinases PknA and PknB: substrate identification and regulation of cell shape. Genes & Development, 2005, 19(14): 1692-1704. |
| [16] | Nagarajan SN, Upadhyay S, Chawla Y, Khan S, Naz S, Subramanian J, Gandotra S, Nandicoori VK. Protein kinase A (PknA) of Mycobacterium tuberculosis is independently activated and is critical for growth in vitro and survival of the pathogen in the host. Journal of Biological Chemistry, 2015, 290(15): 9626-9645. DOI:10.1074/jbc.M114.611822 |
| [17] | Sajid A, Arora G, Gupta M, Upadhyay S, Nandicoori VK, Singh Y. Phosphorylation of Mycobacterium tuberculosis Ser/Thr phosphatase by PknA and PknB. PLoS One, 2011, 6(3): e17871. DOI:10.1371/journal.pone.0017871 |
| [18] | Thakur M, Chakraborti PK. GTPase activity of mycobacterial FtsZ is impaired due to its transphosphorylation by the eukaryotic-type Ser/Thr kinase, PknA. Journal of Biological Chemistry, 2006, 281(52): 40107-40113. DOI:10.1074/jbc.M607216200 |
| [19] | Khan MZ, Kaur P, Nandicoori VK. Targeting the messengers: serine/threonine protein kinases as potential targets for antimycobacterial drug development. IUBMB Life, 2018, 70(9): 889-904. DOI:10.1002/iub.1871 |
| [20] | Malhotra V, Arteaga-Cortés LT, Clay G, Clark-Curtiss JE. Mycobacterium tuberculosis protein kinase K confers survival advantage during early infection in mice and regulates growth in culture and during persistent infection: implications for immune modulation. Microbiology, 2010, 156(9): 2829-2841. DOI:10.1099/mic.0.040675-0 |
| [21] | Wayne LG, Hayes LG. An in vitro model for sequential study of shiftdown of Mycobacterium tuberculosis through two stages of nonreplicating persistence. Infection and Immunity, 1996, 64(6): 2062-2069. |
| [22] | Wallis RS, Patil S, Cheon SH, Edmonds K, Phillips M, Perkins MD, Joloba M, Namale A, Johnson JL, Teixeira L, Dietze R, Siddiqi S, Mugerwa RD, Eisenach K, Ellner JJ. Drug tolerance in Mycobacterium tuberculosis. Antimicrobial Agents and Chemotherapy, 1999, 43(11): 2600-2606. DOI:10.1128/AAC.43.11.2600 |
| [23] | Molle V, Brown AK, Besra GS, Cozzone AJ, Kremer L. The condensing activities of the Mycobacterium tuberculosis type Ⅱ fatty acid synthase are differentially regulated by phosphorylation. Journal of Biological Chemistry, 2006, 281(40): 30094-30103. DOI:10.1074/jbc.M601691200 |
| [24] | Carette X, Platig J, Young DC, Helmel M, Young AT, Wang Z, Potluri LP, Moody CS, Zeng JM, Prisic S, Paulson JN, Muntel J, Madduri AVR, Velarde J, Mayfield JA, Locher C, Wang TS, Quackenbush J, Rhee KY, Moody DB, Steen H, Husson RN. Multisystem analysis of Mycobacterium tuberculosis reveals kinase-dependent remodeling of the pathogen-environment interface. mBio, 2018, 9(2): e02333-17. |
| [25] | Li XJ, Li JJ, Hu XL, Huang LG, Xiao J, Chan J, Mi KX. Differential roles of the hemerythrin-like proteins of Mycobacterium smegmatis in hydrogen peroxide and erythromycin susceptibility. Scientific Reports, 2015, 5: 16130. DOI:10.1038/srep16130 |
