周恩民, 李文均


中山大学生命科学学院, 有害生物控制与资源利用国家重点实验室, 广东 广州 510275
收稿日期:2017-10-30;修回日期:2018-01-06;网络出版日期:2018-01-24
基金项目:国家自然科学基金(31470139,31600103);博士后科学基金(2016M602569);广东省高等学校珠江****岗位计划(2014)
作者简介:周恩民, 中山大学, 生命科学学院, 有害生物控制与资源利用国家重点实验室, 博士后。2015年毕业于云南大学获得博士学位。主要围绕着热泉环境高温微生物资源和生态开展研究工作, 通过改进分离培养策略, 同时结合分子生态学的技术手段, 对中国滇藏和美国大盆地地热系统中微生物的多样性及特定生态学过程的功能微生物进行了研究获得了大量的高温微生物资源, 并对部分类群在热泉环境中的生态功能进行了探索。已在相关学术杂志上发表第一或并列第一作者SCI论文8篇, 参与发表 40余篇; 主持国家自然科学基金青年基金1项, 相关科研成果参与并获得2017年度云南省自然科学二等奖(排名第五)
*通信作者:李文均, Tel/Fax:+86-20-84111727;E-mail:liwenjun3@mail.sysu.edu.cn
摘要:自然界中绝大部分的微生物仍是未培养的,称之为未培养微生物或微生物“暗物质”。对其进行研究不仅有助于认识微生物多样性及其代谢特征,加深对环境中微生物参与的生态学过程的理解,还有利于重构生命之树,揭示微生物的进化历程,具有重要的科学意义。同时未培养微生物是发现新基因资源和新活性物质的巨大宝库。随着现代分子生物学研究方法和培养技术的成熟和完善,从环境中直接破译未培养微生物的遗传信息,并实现培养逐渐成为可能。本文主要介绍了基于宏基因组技术和单细胞基因组技术或两者结合运用,研究环境中未培养微生物的主要方法和挑战,总结分析了目前已经解析的未培养微生物的主要类群,并对未来研究的机遇进行了展望。
关键词: 未培养微生物 微生物“暗物质” 宏基因组 单细胞基因组
Uncultivated microorganisms study: methods, opportunities and challenges
Enmin Zhou, Wenjun Li


State Key Laboratory of Pest Control and Resource Utilization, School of Life Sciences, Sun Yat-Sen University, Guangzhou 510275, Guangdong Province, China
Received 30 October 2017; Revised 6 January 2018; Published online 24 January 2018
*Corresponding author: Wenjun Li, Tel/Fax: +86-20-84111727; E-mail: liwenjun3@mail.sysu.edu.cn
Supported by the National Natural Science Foundation of China (31470139, 31600103), by the China Postdoctoral Science Foundation (2016M602569) and by the Guangdong Province Higher Vocational Colleges and Schools Pearl River Scholar Funded Scheme (2014)
Abstract: The majority of microbial species in the environment remains uncultivated, called uncultivated microorganisms or microbial "dark matter". Unraveling the mysteries of these microbial "dark matter" is especially important to understand the diversity of microbes and their metabolic characteristics. These data can provide insights into the microbes involved in ecological processes, and insights into the early diversification of microbial lineages and the evolution of Bacteria and Archaea. DNA genome sequences of microbial "dark matter" could be recovered from the environment samples by population binning of metagenomics and single-cell genomics, independently or combined synergistically. The metabolic potential could be predicted based on bioinformatics analysis. In this mini-review, we briefly introduce the methods and challenges in this area, summarize the main groups of microbial "dark matter" that has been explored, and indicate the future research opportunities.
Key words: uncultivated microorganisms microbial "dark matter" metagenomics single-cell genomics
微生物是地球上数量最为众多、形式最为多样的生命形式,据估计其细胞总量高达4×1030-6×1030,是地球生物圈的重要组成部分;同时微生物细胞生长繁殖快,具有丰富的生理代谢功能,是生态过程的重要参与者,被认为是地球上元素生物地球化学循环与能量流动的重要引擎[1]。长久以来我们对微生物世界的认识主要来源于基于平板分离法得到的纯培养菌株,然而早在1898年,澳大利亚微生物学家Heinrich Winterberg就已经观察到通过细胞计数和平板计数2种方法得到的样品中微生物总量存在着巨大差别[2]。将近90年后,Staley和Konopka将这一现象总结为The Great Plate Count Anomaly[3],虽然一个多世纪过去了,这个基本微生物问题仍然没有得到很好的解决。之后随着基于生物Marker基因分子系统发育框架的构建[4],以及不依赖于培养的分子技术在微生物领域的广泛应用,特别是利用组学技术如基于系统发育基因(如16S rRNA基因)、宏基因组、单细胞基因组来研究环境中的微生物,使我们能够更加全面和深刻地理解微生物多样性及其与人类健康、环境保护和气候变化等之间的紧密联系。通过这种方法,研究人员从环境中发现了大量的未培养微生物(uncultivated microorganisms),很多都是生命之树上的新分支(candidate divisions)。基于“主要未培养微生物(uncultivated majority)”的范围和规模,研究人员借用天体物理学中“暗物质(dark matter)”的概念,直接使用“暗物质(dark matter)”、“生物暗物质(biological dark matter)”或“微生物暗物质(microbial dark matter)”等术语来代表它们,以此来引起人们对未培养微生物的重视[5-6]。
通过深入了解环境中微生物多样性,并建立微生物类群与代谢特征之间的联系,可以加深我们对生态系统中微生物参与的生态学过程及其行使的生态学功能的理解。然而针对微生物“暗物质”,仅仅是通过分子手段(分子Marker或部分基因组信息)知道它们存在于环境中的,却难以在实验室实现培养,并且缺少基因组信息,因此对其代谢潜能、生态功能的认识十分有限。随着高通量测序技术的迅猛发展,基于宏基因组和单细胞基因组技术,借助生物信息学分析手段,从环境中直接获得并解读未培养微生物的基因组信息逐渐成为可能,包括破译一些潜在新门级别的未培养微生物基因组信息,让我们有机会揭开环境中大量存在的未培养微生物的神秘面纱,进而认识环境中微生物的多样性,理解其生理生态潜能和进化历程,给微生物学研究领域带来了巨大变革。本文简要回顾当前基于宏基因组技术和单细胞基因组技术或两者结合运用,研究未培养微生物的主要方法和挑战,总结分析目前已经解析的未培养微生物的主要类群,并对未来研究进行展望。
1 未培养微生物研究的主要方法和挑战 毋庸置疑,从复杂环境样品中直接获得未培养微生物的基因组信息并进行解读存在着巨大挑战和风险,但是随着测序技术的不断革新和发展,特别是宏基因组和单细胞基因组技术的应用,以及相关生物信息学分析工具的完善,基于严格的质控和相关技术的补充应用,让我们能够获得宏基因组组装的基因组(metagenome-assembled genomes,MAGs)和单细胞基因组(single amplified genomes,SAGs)。通过这些方法可以从环境中得到部分未培养微生物的基因组信息,并探索其代谢潜能,有利于发现全新的生理代谢功能和过程,进而完善对生态系统过程及功能的了解;同时还有助于拓展对微生物多样性的认识,有利于重构生命之树,解析生命起源及其进化历程。
宏基因组技术是通过直接将环境样品中所有微生物的DNA提取出来,构建宏基因组文库,然后一起测序,利用基因组学方法研究环境样品所包含的全部微生物的遗传组成及其群落功能。通过宏基因组技术挖掘未培养微生物主要包括以下几个步骤:(1)样品总DNA的提取。需要结合样品自身的特点利用手工提取方法[7],或宏基因组提取试剂盒,以获取足量的总DNA用于后续建库测序。但值得注意的是不同的提取策略其提取效果可能会存在很大的偏差[8],因此针对同一批次、同一类型的样品尽量采用标准化的提取策略。(2)建库测序。可以借助不同的平台来进行宏基因组测序,但不同的测序平台在读长、通量以及错误率上存在差异,将直接影响后续的拼接效果。(3)序列拼接。基于一些特异性的算法[9-10],借助拼接软件(如SPAdes[11],MetaVelvet[12]等)将测序所得的reads组装成各个Contigs/Scaffolds。(4)基因组分装(Genome binning)和分析。依据序列核苷酸的组成特征(如GC含量,密码子使用情况,四核苷酸多态性,序列相似性等)、测序深度及覆盖度等,结合样品自身的特性,利用binning软件(如GroopM[13],MaxBin[14],MetaBAT[15],Canopy[16],ESOM[17],CONCOCT[18],MetaWatt[19]和DASTool[20]等),从而获得宏基因组组装基因组(MAGs)。并利用CheckM检验各MAGs的完整度及污染度[21],并手工调整以获得高质量的基因组信息。筛选每个MAGs中保守的标记基因并基于全基因组进行系统发育分析以判断其系统发育信息。针对微生物“暗物质”类群,进行基因预测以获得其基因信息,并完成基因组的功能注释,结合注释信息,预测其代谢潜能。通过该方法,研究人员已经获得了部分未培养微生物的首个完整或接近完整的基因组信息[6, 22]。宏基因组学技术的一大优势是样品制备过程相对简单和可变,可以应用到任何能够获得足量总DNA的样品,总DNA的提取方法也可以根据样品特性来进行调整和完善,这是其他方法如单细胞基因组技术所不能比拟的。理论上通过改进总DNA提取过程,结合一定的测序深度,能够全面反映样品中的所有遗传信息,从而获得所有微生物的全基因组信息。但该技术存在的主要挑战是,测序获得的基因信息是来自于环境中丰度各异的不同物种的集合体,同时还存在种群内部菌株水平的异质性,因此想从中得到单一物种的独立基因组信息并不容易。特别是针对具有复杂微生物群落结构的环境样品(如土壤),或者是群落中的那些低丰度类群以及缺少完整的参考基因的未培养微生物(如一些潜在新门级别的类群)的基因组时。但是随着测序通量、读长的增加,以及基因组组装和binning算法的完善,该技术也可以从部分复杂环境样品中获得MAGs[23]。
单细胞基因组技术是通过从样品中分选出单一细胞,然后利用全基因组扩增和测序,借助生物信息学分析软件组装获得单细胞基因组(SAGs),从最基本的生物学单位(单个细胞)来研究样品中的微生物[24]。通过单细胞基因组技术挖掘未培养微生物主要包括以下几个步骤:(1)单细胞分选。目前可以通过不同的方式来分选样品中的细胞,包括梯度稀释、细胞捕获、显微操作、流式分选以及微流控芯片技术等。其中梯度稀释和显微操作分选过程相对随机,分选目标难以实现特异性,并且通量低;基于流式细胞仪的分选过程可以参照细胞的大小、形态、荧光信号等表型特征,高效分选出不同类型的微生物细胞,但是大型高效的分选型流式细胞仪往往价格昂贵,并且需要专业技术人员进行操作和维护。基于微流控芯片的单细胞分选技术,在具有分选效率高的基础上还可以结合荧光和光谱类检测方法,对特定表型细胞实现特异性分选,是近年来较为主流和热门的单细胞分选手段。目前,已有成熟的商业化微流控芯片[25],也有很多实验室开发了多样化的微流控芯片设备,如:中国科学院青岛生物能源与过程研究所开发的“FOCOT (Facile One-Cell-One-Tube)”的微流控平台[26],中国科学院微生物研究所近期开发的“MSP (Microfluidic Streak Plate)”[27]。(2)单细胞基因组扩增。分选获得单细胞之后进行细胞裂解,以释放基因组DNA,然后通过多重置换扩增(MDA,multiple displacement amplification)进行全基因组扩增[28],使单细胞基因组DNA从飞克级别扩增到纳克至微克级别,以用于下游实验。(3)未培养微生物的筛选和测序。后续一般先通过PCR扩增和测序对分选获得的单细胞进行初步鉴定,选择感兴趣的未培养微生物类群进行全基因组测序,并参照组装得到的单细胞基因组中单拷贝保守基因(SCMs,single-copy conserved markers)的数目来确定所获得的单细胞基因组的完整度。单细胞基因组技术的挑战主要在于从复杂环境样品中筛选单细胞以及单细胞所含的DNA量太低而必须要借助于全基因组扩增。在环境样品中紧密粘附在固体颗粒表面或者呈聚集生长状态的微生物细胞,往往要经过相应的前期处理来富集和分散细胞,以便于细胞分选;另外在分选过程中还要保证细胞的完整性。由于单细胞极微量的基因组DNA需要大量扩增,对污染问题特别敏感,因此对样品制备要求严格,需要考虑所使用的试剂、仪器等[24]。后续的序列质控(核苷酸使用频率,SCMs的拷贝数,与可能的污染源库进行比对),同时借助一些软件(如DeconSeq[29],Anvi’o[30],ProDeGe[31])可以在一定程度上剔除单细胞基因组中的污染序列[6, 32]。此外在单细胞基因组多轮扩增中往往会引入嵌合体,扩增过程本身也存在一定程度的偏好性。针对这个问题,目前可以借助一些特殊的算法(如SPAdes[11]和IDBA-UD[33]),同时合并同一物种的多个单细胞基因组(ANI>95%)来提高单细胞基因组的质量[6, 34]。
宏基因组技术和单细胞基因组技术各有优劣,相互补充,2种技术组合应用,是挖掘未培养微生物的有力手段[35]。宏基因组技术不需要分选单细胞和MDA,而单细胞基因组技术可以直接揭示系统发育位置和潜在功能联系[36],同时为从宏基因组序列中binning出单个基因组提供依据。此外通过宏基因组技术得到的reads和contigs能够指导多个相近单细胞基因组的组装,有效提高单细胞基因组质量[37]。2种技术补充应用,可以从环境样品中得到未培养微生物近乎完整的基因组信息[6, 32]。
2 未培养微生物研究的主要进展 2.1 利用宏基因组技术挖掘未培养微生物 Tyson等[22]利用宏基因组技术,并借助第一代测序平台从酸性矿山废水(AMD)的粉色生物膜中获得了2个未培养细菌的基因组(i.e., Leptospirillum group Ⅱ和Ferroplasma group Ⅱ),并且对它们的营养需求和生物地球化学循环功能进行了解析。细菌类群WWE1的第一个基因组Candidatus ‘Cloacamonas acidaminovorans’是通过相似的技术手段从厌氧消化池中获得,基因组分析揭示该类群与其他未知微生物营互养生长[38]。可以利用富集手段来降低样品中微生物的多样性,以利于宏基因组的组装和分析。古菌类群Korarchaeota的基因组Candidatus‘Korarchaeum cryptofilum’,便是基于该类群对高浓度的SDS有一定的耐受能力,通过富集并结合宏基因组测序得到的[39]。此外还有Candidatus‘Accumulibacter phosphatis’ strain UW-1 [40],Candidatus‘Nitrospira defluvii’[41]和Candidatus‘Liberibacter asiaticus’[42]等。
二代测序技术的出现大大降低了测序成本,同时加速了利用宏基因组技术挖掘未培养微生物的进程。前期主要是通过对Fosmid文库中的大基因组片段进行测序,然后拼接获得MAGs,如古菌新类群Candidatus ‘Caldiarchaeum subterraneum’[43]和细菌新类群Candidatus‘Acetothermum autotrophicum’[44]的基因组。van de Vossenberg等[45]使用一代、二代测序技术相结合的策略,从富集培养物中获得了海洋来源的厌氧氨氧化细菌Candidatus‘Scalindua profunda’的基因组,通过解析发现该菌株可以利用小分子有机酸及寡肽,能够以硝酸盐、亚硝酸盐和金属氧化物作为电子受体,这些特性与淡水环境中的厌氧氨氧化细菌存在很大差别。细菌新类群Candidatus‘Fodinabacter communificans’[46]、Candidatus‘Accumulibacter sp.’ strain UW-2[47]以及具有光合异养潜能的放线菌新类群Candidatus ‘Actinomarinidae’的基因组[48]都是通过这种方法得到。针对多样性较低的样品,只通过二代测序也成功地获得了部分未培养微生物的基因组信息。Lynch等[49]从高海拔Atacama沙漠火山碎石样品中获得Pseudonocardia sp.的MAGs,能够编码完整的氧化大气中不同小分子气体物质(如H2,CO等)并固定二氧化碳的分子通路。Mondav等[50]从永久冻土解冻区获得Candidatus‘Methanoflorens stordalenmirensis’的MAG,具有氧化氢并产甲烷的基因,该物种是一类基于甲烷的应对气候变暖正反馈调节的重要类群。Hua等[51]采用“分而治之(divide and conquer)”的组装策略,同时参考宏转录组数据,从AMD系统中获得11个MAGs,其中10个属于稀有类群,同时揭示了这些微生物适应低pH、高重金属环境的机制。目前随着具有更长读长的三代测序技术的不断完善,将更有利于从环境样品中直接重构基因组。Zhalnina等[52]基于PacBio测序平台从农业土壤富集物中组装得到第一个土壤环境来源的氨氧化古菌Ⅰ.1b类群Candidatus‘Nitrososphaera evergladensis’的MAG。
借助深度测序可以从样本中获得大量的MAGs,从而更好地理解样品中的微生物及其过程,同时完善我们对微生物系统发育和进化的认识。Anantharaman等[23]利用宏基因组技术从美国科罗拉多州的一个地下蓄水层沉积物和地下水样本中,获得了2500多个MAGs,涵盖了近80%的已知细菌门类,包含47个新门级的细菌类群,该研究揭示了地下微生物的多样性及不同微生物之间的互作方式,及其参与的碳、氮、氢等重要元素的生物地球化学循环过程。加州大学伯克利分校的Jillian F. Banfield[53]研究团队基于公共数据库中的基因组和1011个从不同环境中获得的新的MAGs,重构生命之树,揭示了细菌类群巨大的多样性,其中多样性最高的分支被称为“候选门辐射群(candidate phyla radiation,CPR)”,目前还没有纯培养物。Parks等[54]从1500多个公开的宏基因组中组装获得了7903个MAGs,大大拓展了细菌和古菌的多样性,其中包含17个新门级别的细菌和3个新门级别的古菌,还包括245个CPR类群的未培养微生物。此外宏基因组技术在解析环境中病毒多样性方面也显示出一定的优势。Paez-Espino等[55]分析了全球10类生境的3024个宏基因组样本,最终得到了125842个部分及完整的病毒基因组,将已知的病毒基因数量提高了16.6倍,这些病毒基因可编码279万多个蛋白,且其中75%和已分离培养的病毒无序列相似性,还构建了首个全球病毒的分布图。
2.2 基于单细胞基因组技术挖掘未培养微生物 单细胞基因组技术是挖掘未培养微生物的有力手段,特别是针对微生物群落中的稀有类群。第一个低盐环境下的氨氧化古菌Candidatus‘Nitrosoarchaeum limnia’ SFB1的基因组就是通过这种方法得到的[56]。Marcy等[5]利用该技术成功解析了人类口腔中丰度只有0.7%-1.9%的细菌类群TM7的基因组,并对该类群菌株进行了分离培养[57]。McLean等[58]解析了全球分布的未培养细菌类群TM6,Youssef等[59]解析了陆地和海洋生态系统中广泛存在的未培养细菌类群OP11,Fullerton等[60]解析了地球深部生物圈中绿弯菌门的未培养类群的全基因组。Swan等[61]获得了海洋主要微生物类群Deltaproteobacteria cluster SAR324、Gammaproteobacteria clusters ARCTIC96BD-19和Agg47,以及部分Oceanospirillales成员的全基因组,并解析其化能无机自养代谢途径,这些类群在海洋碳循环中扮演着重要角色。Rinke等[6]利用流式细胞仪分选结合单细胞基因组测序,从海洋、淡水、热液口等9种不同环境样品中获得了201个SAGs,涵盖29个未培养微生物类群,基于这些数据进一步解析了不同类群之间的系统进化关系,并提出2个超门,同时发现了很多新奇的代谢途径。另外单细胞基因组技术还能够更好地解析不同微生物之间的互作关系,Hongoh等[62]利用单细胞基因组技术从白蚁Reticulitermes speratus的肠道共生原生生物Trichonympha agili中获得未培养细菌菌株Rs-D17的SAG,并对三者之间的关系进行了阐述。Engel等[63]揭示了蜜蜂肠道内生菌Gilliamella apicola和Snodgrassella alvi种内不同菌株之间蛋白编码基因的差异。
2.3 混合策略挖掘未培养微生物 宏基因技术和单细胞基因组技术各有优劣,相较于单一技术,采用混合策略将提升我们对未培养微生物的挖掘和理解。单细胞基因组可以参照宏基因组技术获得的序列来提高基因组的完整度[56],同时单细胞基因组也可以用于指导并校正宏基因组数据的组装拼接[64]。Nobu等[65]通过融合两种技术获得了15个门的35个基因组草图,其中包括3个潜在新门Atribacteria、Hydrogenedentes和Marinimicrobia的第一个基因组。另外还可以借助单细胞基因组技术对宏基因组技术获得的特定类群的功能进行专门研究。Mason等[66]利用单细胞基因组技术证实墨西哥湾漏油事故中的优势类群Oceanospirillales (宏基因组研究结果)具有石油烃降解功能。Dodsworth等[32]借助这种方法解析了细菌新类群OP9的全基因组,并对其代谢特征进行了预测。斯坦福大学的Stephen Quake研究团队最近提出了MINI-宏基因组技术[67],该技术基于微流控芯片从环境样品中获得含有5-10个细胞的子样品,从而降低样品中微生物复杂度,然后利用宏基因组技术对不同的子样品进行研究,这种方法一方面保留了单细胞的高分辨率,另外利用不同子样品中微生物细胞的共存模式还能提高单个基因组的binning。他们利用该技术从美国黄石公园的热泉样品中获得了29个新的微生物基因组。
3 总结和展望 测序技术的革新和发展,特别是宏基因组和单细胞基因组技术的成功应用,同时借助相应生物信息学工具,让我们有机会更加全面地了解自然环境中微生物的多样性,并且能够从复杂环境中得到大量未培养微生物的基因组信息。这些信息促进了我们对生命之树各个分支及其进化历程的认识,同时有助于我们对生物多样性和生态系统功能,特别是新的代谢特征的理解。大规模测序工作如人类微生物组计划(Human Microbiome Project)[68]、地球微生物组计划(Earth Microbiome Project)[69]、细菌古菌基因组百科全书(Genomic Encyclopedia of Bacteria and Archaea)[70]等的成功实施,加速了我们对微生物多样性和功能的认识,在未培养微生物研究方面也取得了一些实质性的进展,表 1、表 2分别列示了目前已经解析的新门级别古菌和细菌类群“暗物质”,但现在仍然是我们深入挖掘未培养微生物的绝佳时机。目前为止,生命之树仍有很多分支没有基因组信息[71]。随着国际上主要微生物组计划的实施,例如美国已经实施的国家微生物组计划(National Microbiome Initiative),我国即将实施的中国微生物组计划(China Microbiome Initiative)[72],中、美、德等国科学家呼吁组织的国际微生物组计划(International Microbiome Initiative)[73],以及专门针对微生物“暗物质”美国能源部联合基因组研究中心(DOE-JGI)启动的细菌古菌基因组百科全书-微生物“暗物质”计划(Genomic Encyclopedia of Bacteria and Archaea-Microbial Dark Matter,GEBA-MDM)等,可以预见,公共数据库中基因组数据集将不断增加,越来越多的微生物“暗物质”将被发现并解析。最近基因组标准协会(Genomic Standards Consortium,GSC)基于基因组的拼装质量、完整度和污染程度制定了有关SAG和MAG的质量标准[74],该标准的建立将促进大规模的比较研究,有利于研究人员从组学大数据中获得未培养微生物的基因组信息,为后续的发展奠定标准和基础。
表 1. 近年来通过宏基因组和单细胞基因组技术获得的古菌潜在新门 Table 1. Archaeal candidate phyla with one or more members having partial or complete MAGs or SAGs
| Candidate phylum | Genomic datasets | First description in reference |
| Aenigmarchaeota (DSEG) | SAG | Homestake Mine[6] |
| Aigarchaeota (pSL4; HWCG-I) | MAG, SAG | Geothermal water stream from a subsurface mine in Japan[6, 43] |
| Bathyarchaeota (MCG) | MAG | Marine sediment[75] |
| Diapherotrites (pMC2A384) | SAG | Homestake Mine[6] |
| Geoarchaeota | MAG | Acidic iron mats in Yellowstone National Park[76] |
| Heimdallarchaeota | MAG | Marine sediments (Loki’s Castle and Aarhus Bay)[77] |
| Korarchaeota | MAG | Obsidian Pool, Yellowstone National Park[78] |
| Lokiarchaeota | MAG | Arctic Mid-Ocean Ridge[79] |
| Nanoarchaeota | MAG | Submarine hot vent[80] |
| Nanohaloarchaeota | MAG, SAG | Ponds of Bras del Port salterns, Spain[81] |
| Odinarchaeota | MAG | Hot spring metagenomes (Yellowstone National Park and Radiata Pool)[77] |
| Pacearchaeota | MAG | Aquifer adjacent to the Colorado River (USA)[82] |
| Parvarchaeota (ARMAN) | MAG, SAG | A drift of the Richmond Mine, Northern California[6, 83] |
| Thorarchaeota | MAG | Sulfate-methane transition zone in the White Oak River estuary sediments[84] |
| UAP1-3 | MAG | Assembled from public metagenomes[54] |
| Verstraetearchaeota | MAG | Cellulose-degrading anaerobic digesters[85] |
| Woesearchaeota | MAG | Aquifer adjacent to the Colorado River (USA)[82] |
表选项
表 2. 近年来通过宏基因组和单细胞基因组技术获得的细菌潜在新门 Table 2. Bacterial candidate phyla with one or more members having partial or complete MAGs or SAGs
| Candidate phylum | Genomic datasets | First description in reference |
| Acetothermia (OP1/KB1 group) | MAG, SAG | Obsidian Pool, Yellowstone National Park[6, 78] |
| Aerophobetes (CD12) | SAG | Sakinaw Lake[6] |
| Aminicenantes (OP8) | SAG | Obsidian Pool, Yellowstone National Park[6, 78] |
| Atribacteria (OP9/JS1) | MAG, SAG | Obsidian Pool, Yellowstone National Park[6, 32, 78] |
| BD1-5 | MAG | Groundwater samples[86] |
| Berkelbacteria (ACD58) | MAG | Aquifer adjacent to the Colorado River (USA)[87] |
| BRC1 | SAG | Etoliko Lagoon and Sakinaw Lake[6] |
| Calescamantes (EM19) | SAG | Great Boiling Spring and Gongxiaoshe hot spring[6] |
| Cloacimonetes (WWE1) | MAG, SAG | Municipal Anaerobic Sludge Digester[6, 38] |
| CPR (RIF1-46 and SM2F11) | MAG | Aquifer sediments and groundwater, USA[23] |
| EM3 (former OP2) | SAG | Obsidian Pool, Yellowstone National Park[6, 78] |
| Fervidibacteria (OctSpA1-106) | SAG | Octopus Spring sediment[6] |
| Gracilibacteria (GN02) | MAG, SAG | Guerrero Negro hypersaline microbial mat[6, 86] |
| Hydrogenogenetes (BRC1/NKB19) | MAG, SAG | Bulk soil and rice roots[6] |
| Kryptonia | MAG | High-temperature pH-neutral geothermal springs[88] |
| KSB3 | MAG | Anaerobic wastewater treatment bioreactor[89] |
| Latescibacteria (WS3) | SAG | Wurtsmith Air Force Base, Michigan[6, 90] |
| Marinimicrobia (SAR406) | MAG, SAG | Subsurface of Atlantic and Pacific oceans[6] |
| Melainabacteria | MAG | Human gut and groundwater[91] |
| Microgenomates (OP11) | MAG, SAG | Obsidian Pool, Yellowstone National Park[78, 86] |
| NC10 | MAG | Aquatic microbial formations in flooded caves[92] |
| Omnitrophica (OP3) | MAG, SAG | Obsidian Pool, Yellowstone National Park[78] |
| Parcobacteria (OD1) | MAG, SAG | Obsidian Pool, Yellowstone National Park[78, 86] |
| PER | MAG, SAG | Groundwater samples[86] |
| Poribacteria | SAG | Marine sponge-associated[93] |
| Saccharibacteria (TM7) | MAG, SAG | Peat bog (TM means Torf, Mittlere schicht)[5, 94] |
| SBR1093 | MAG | Activated sludge from wastewater treatment system[95] |
| SR1 | MAG, SAG | Hydrocarbon-contaminated aquifer (SR, “Sulfur River”)[96] |
| Tectomicrobia | MAG, SAG | Marine sponge[97] |
| TM6 | SAG | Peat bog (TM means Torf, Mittlere schicht)[58, 94] |
| UBP1-17 | MAG | Assembled from public metagenomes[54] |
| WS1 | SAG | Wurtsmith Air Force Base, Michigan[6, 90] |
| WWE3 | MAG | Anaerobic sludge digester[87] |
表选项
3.1 未培养微生物研究的后基因组时代 基于未培养微生物MAGs和SAGs信息获得的代谢潜能,仅仅只是预测,如果没有其他数据支撑,只能作为一个线索,而非有力证据。因此在后基因组时代,未培养微生物的研究将以基因组功能预测为基础,重点开展原位功能活性测试。为此,部分研究人员选择了宏转录组和宏蛋白质组,在转录和翻译层面上来检测目的基因的表达[83]。这种方法在微生物多样性较低的环境如热泉、酸性尾矿、生物反应器等生境具有一定的优势,但该方法不能直接对微生物本身及其功能基因代谢产物进行检测。同位素标记实验对解决这一问题具有优势。在原始生境中使用放射性或稳定性同位素进行标记,然后利用荧光原位杂交(FISH,fluorescence in situ hybridization)结合显微放射自显影技术(microautoradiography,MAR-FISH)[98]或纳米级二次离子质谱技术(nano-scale secondary ion mass spectrometry,FISH-Nano-SIMS)[99]、拉曼光谱技术(Raman-FISH)[100]可以揭示特定微生物类群的同化代谢特征。此外如果用系统发生芯片(PhyloChip)来代替荧光原位杂交同时结合Nano-SIMS,可以同时对多个类群的功能进行研究(Chip-SIP)[101]。类似的高通量方法还有利用物质的拉曼效应,直接对微生物细胞内各种化合物进行定性、定量分析,并结合细胞分选,然后进行单细胞基因组测序。另外通过对未培养微生物中特定功能基因进行异源表达并检测其活性[75],同时基于合成生物学,在模式生物中对整个操纵子进行研究,也是非常有效的手段[102]。
3.2 未培养微生物的分类命名及可培养化 目前微生物的分类命名系统主要是基于纯培养菌株的信息构建的,大部分通过分子手段发现的未培养微生物并没有按照Linnean的双名法进行命名,而只是用简单的字母和数字来标识,这种标识既不能表明表型特征或生态学功能,也不能反映分类学信息,常常出现混乱的情况,给研究和交流工作带来了极大的限制。虽然也提出了暂定种(Candidatus)[103]这个概念,但因其操作性不强,使用范围非常有限;同时由于没有对暂定种的命名进行专门审阅,大约有30%的命名是不恰当的[104]。因此能够兼顾未培养微生物和所有已生效发表的纯培养菌株的分类体系,以及针对未培养微生物的命名法亟待建立。随着技术的进步,目前越来越多的基因组信息可用于区分不同的物种,因此美国****Whitman[105]向原核生物系统学国际委员会(ICSP,International Committee on Systematics of Prokaryotes)提出使用DNA序列作为新的分类标准材料的提案。Konstantinidis等[106]基于未培养微生物的基因组信息和预测的表型特征提出了一套与现有命名系统并行的命名方式,但形成一套能同时兼顾纯培养和未培养菌株的分类命名系统,并得到大家的广泛认可和接受,尚需时日。
获得微生物的纯培养菌株是进行深入科学研究和开发应用的基础,微生物细胞能否被培养在一定程度上取决于是否找到了适宜的培养方法。未培养微生物往往具有特殊的生长需求,包括温度、pH、含氧量、营养源、生长因子、信号物质等。另外这些微生物在实验室条件下作为一种适应策略可能会形成活的但不能培养(viable but non-culturable)或休眠状态[107]。通过对未培养微生物基因组的详细分析,进而获得一些指导信息,将有助于实现未培养微生物的可培养化。另外培养手段的创新也很重要,如基于允许微生物与环境及其他物种交流代谢物思想设计的ichip (isolation chip)技术,大大提高了可培养微生物的多样性[108]。基于未培养微生物的全基因组信息,深入研究其代谢特性,并结合其原位生长环境特征,改进完善分离培养策略,实现免培养技术和纯培养手段的有效结合,能够大大增强未培养微生物类群的可培养性,从而提高我们对未培养微生物的认识和理解。
3.3 功能导向性的未培养微生物挖掘 未培养微生物蕴藏着大量的未知功能基因和代谢潜能,在生物能源、生物技术和环境领域具有重要的应用潜力。Lewis[109]指出未培养微生物是探寻新抗生素的重要来源,可以解决目前病原微生物的抗药性和耐药性问题。Lackner等[110]利用宏基因组技术构建海绵共生细菌Candidatus‘Entotheonella factor’和Candidatus‘Entotheonella gemina’的基因组,并证明它们是海绵中生物活性物质的主要产生者。Ling等[111]利用iChip装置,通过原位培养,从土壤中获得一种隶属于β-变形杆菌“Eleftheria terrae sp.”类群的未培养微生物,该菌株能够产生一种称之为“Teixobactin”的酯肽,该物质可以阻碍革兰氏阳性细菌细胞壁的合成,具有广谱的抗菌活性,同时很难产生抗药性。环境中未培养微生物的不断发现和解析,将大大促进功能导向性的研究工作,使未培养微生物来源的新基因和新活性物质的挖掘出现新的机遇。
此外值得注意的是,微生物“暗物质”除了包括那些已知的未培养微生物类群之外,还包括那些目前未探明(undiscovered)的生命,主要是指基于现有的技术手段还未探测到的生命类群[112]。譬如由于现有技术的限制(比如与通用16S rRNA基因引物的错配、现有测序技术和深度的限制等),或者目前还未获得它们相应生境的样品,以至于还检测不到它们。在未探明的生命中很有可能会发现生命三域之树以外的分支。全球范围内的极端环境,如热泉、盐湖、喀斯特洞穴、冰川、AMD、油藏环境等,一直是地质微生物研究领域的热点。极端环境往往具有迥异于普通环境的特殊微生物群落组成,从极端环境中挖掘未培养微生物,并揭示其生态角色,是我们理解地质微生物及其生态过程的重要组成部分。以热泉为例,基于全球地热系统微生物16S rRNA基因高通量测序结果表明,热泉环境孕育着大量的微生物“暗物质”[113],在门一级水平达到16.1%,纲一级水平达34.0%,目一级水平达42.1%,科一级水平高达46.9%。我们课题组在前期工作中从滇藏热泉生境中分离并描述了许多微生物新物种[114-115],极大地拓展了高温微生物资源。同时基于16S rRNA基因利用高通量测序,对滇藏部分热泉微生物群落组成进行了分析[116-119],结果发现部分热泉中未培养微生物的丰度在门一级别可高达30%左右。通过与DOE-JGI等单位合作,利用宏基因组结合单细胞基因组技术,从热泉环境中发现1个高温中性热泉环境特有的潜在细菌新门Candidate “Kryptonia”,由于该类群的16S rRNA基因与现有细菌通用引物存在错配,所以一直未被探测到,基因组代谢潜能分析发现该类群微生物营异养生长,存在营养缺陷,需要与其他微生物共生生长[88]。后续通过对云南地区5个热泉宏基因组的深度分析,还探测到其他的未培养微生物,其中包含5个潜在的新门(图 1),对它们的代谢潜能及其在热泉重要生源元素循环中所起的作用,还有待进一步研究。
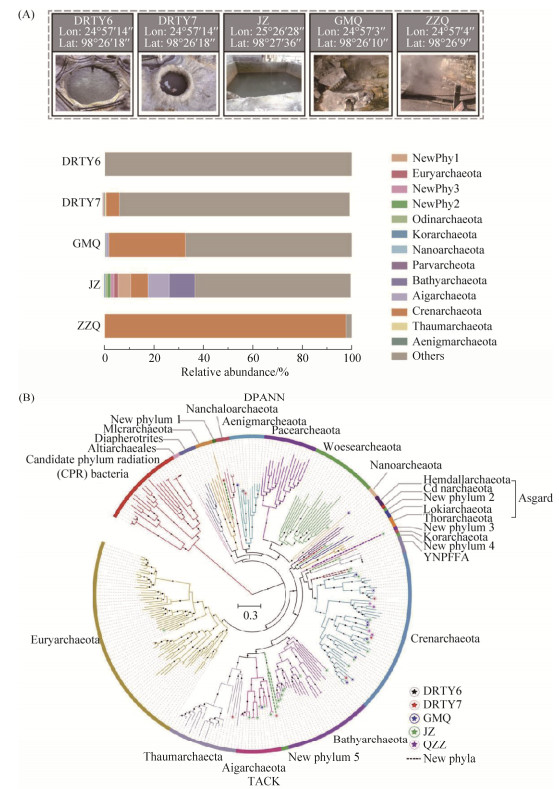 |
| 图 1 基于云南腾冲5个热泉宏基因组分析热泉微生物多样性(A)和系统发育关系(B) Figure 1 The microbial diversity (A) and phylogenetic relationships (B) in 5 hot springs located in Tengchong, Yunnan, based on the metagenomic analysis. |
| 图选项 |
致谢: 特别感谢中山大学生命科学学院花正双博士和陈林兴博士在本文撰写过程中所提供的帮助!
References
| [1] | Falkowski PG, Fenchel T, Delong EF. The microbial engines that drive Earth's biogeochemical cycles. Science, 2008, 320(5879): 1034-1039. DOI:10.1126/science.1153213 |
| [2] | Winterberg H. Zur methodik der bakterienz?hlung. Zeitschrift für Hygiene und Infektionskrankheiten, 1898, 29(1): 75-93. |
| [3] | Staley JT, Konopka A. Measurement of in situ activities of nonphotosynthetic microorganisms in aquatic and terrestrial habitats. Annual Review of Microbiology, 1985, 39(1): 321-346. DOI:10.1146/annurev.mi.39.100185.001541 |
| [4] | Woese CR, Fox GE. Phylogenetic structure of the prokaryotic domain:the primary kingdoms. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74(11): 5088-5090. DOI:10.1073/pnas.74.11.5088 |
| [5] | Marcy Y, Ouverney C, Bik EM, L?sekann T, Ivanova N, Martin HG, Szeto E, Platt D, Hugenholtz P, Relman DA, Quake SR. Dissecting biological "dark matter" with single-cell genetic analysis of rare and uncultivated TM7 microbes from the human mouth. Proceedings of the National Academy of Sciences of the United States of America, 2007, 104(29): 11889-11894. DOI:10.1073/pnas.0704662104 |
| [6] | Rinke C, Schwientek P, Sczyrba A, Ivanova NN, Anderson IJ, Cheng JF, Darling A, Malfatti S, Swan BK, Gies EA, Dodsworth JA, Hedlund BP, Tsiamis G, Sievert SM, Liu WT, Eisen JA, Hallam SJ, Kyrpides NC, Stepanauskas R, Rubin EM, Hugenholtz P, Woyke T. Insights into the phylogeny and coding potential of microbial dark matter. Nature, 2013, 499(7459): 431-437. DOI:10.1038/nature12352 |
| [7] | Zhou J, Bruns MA, Tiedje JM. DNA recovery from soils of diverse composition. Applied and Environmental Microbiology, 1996, 62(2): 316-322. |
| [8] | Costea PI, Zeller G, Sunagawa S, Pelletier E, Alberti A, Levenez F, Tramontano M, Driessen M, Hercog R, Jung FE, Kultima JR, Hayward MR, Coelho LP, Allen-Vercoe E, Bertrand L, Blaut M, Brown JRM, Carton T, Cools-Portier S, Daigneault M, Derrien M, Druesne A, de Vos WM, Finlay BB, Flint HJ, Guarner F, Hattori M, Heilig H, Luna RA, van Hylckama Vlieg J, Junick J, Klymiuk I, Langella P, Le Chatelier E, Mai V, Manichanh C, Martin JC, Mery C, Morita H, O'Toole PW, Orvain C, Patil KR, Penders J, Persson S, Pons N, Popova M, Salonen A, Saulnier D, Scott KP, Singh B, Slezak K, Veiga P, Versalovic J, Zhao LP, Zoetendal EG, Ehrlich SD, Dore J, Bork P. Towards standards for human fecal sample processing in metagenomic studies. Nature Biotechnology, 2017, 35: 1069-1076. |
| [9] | Li DH, Luo RB, Liu CM, Leung CM, Ting HF, Sadakane K, Yamashita H, Lam TW. MEGAHIT v1.0:A fast and scalable metagenome assembler driven by advanced methodologies and community practices. Methods, 2016, 102: 3-11. DOI:10.1016/j.ymeth.2016.02.020 |
| [10] | Nurk S, Meleshko D, Korobeynikov A, Pevzner PA. metaSPAdes:a new versatile metagenomic assembler. Genome Research, 2017, 27(5): 824-834. DOI:10.1101/gr.213959.116 |
| [11] | Bankevich A, Nurk S, Antipov D, Gurevich AA, Dvorkin M, Kulikov AS, Lesin VM, Nikolenko SI, Pham S, Prjibelski AD, Pyshkin AV, Sirotkin AV, Vyahhi N, Tesler G, Alekseyev MA, Pevzner PA. SPAdes:a new genome assembly algorithm and its applications to single-cell sequencing. Journal of Computational Biology, 2012, 19(5): 455-477. DOI:10.1089/cmb.2012.0021 |
| [12] | Namiki T, Hachiya T, Tanaka H, Sakakibara Y. MetaVelvet:an extension of Velvet assembler to de novo metagenome assembly from short sequence reads. Nucleic Acids Research, 2012, 40(20): e155. DOI:10.1093/nar/gks678 |
| [13] | Imelfort M, Parks D, Woodcroft BJ, Dennis P, Hugenholtz P, Tyson GW. GroopM:an automated tool for the recovery of population genomes from related metagenomes. PeerJ, 2014, 2: e603. DOI:10.7717/peerj.603 |
| [14] | Wu YW, Tang YH, Tringe SG, Simmons BA, Singer SW. MaxBin:an automated binning method to recover individual genomes from metagenomes using an expectation-maximization algorithm. Microbiome, 2014, 2(1): 26. DOI:10.1186/2049-2618-2-26 |
| [15] | Kang DD, Froula J, Egan R, Wang Z. A robust statistical framework for reconstructing genomes from metagenomic data. bioRxiv, 2014, doi: 10.1101/011460. |
| [16] | Nielsen HB, Almeida M, Juncker AS, Rasmussen S, Li JH, Sunagawa S, Plichta DR, Gautier L, Pedersen AG, Le Chatelier E, Pelletier E, Bonde I, Nielsen T, Manichanh C, Arumugam M, Batto JM, Quintanilha dos Santos MB, Blom N, Borruel N, Burgdorf KS, Boumezbeur F, Casellas F, Doré J, Dworzynski P, Guarner F, Hansen T, Hildebrand F, Kaas RS, Kennedy S, Kristiansen K, Kultima JR, Léonard P, Levenez F, Lund O, Moumen B, Le Paslier D, Pons N, Pedersen O, Prifti E, Qin JJ, Raes J, S?rensen S, Tap J, Tims S, Ussery DW, Yamada T, Renault P, Sicheritz-Ponten T, Bork P, Wang J, Brunak S, Ehrlich SD. Identification and assembly of genomes and genetic elements in complex metagenomic samples without using reference genomes. Nature Biotechnology, 2014, 32(8): 822-828. DOI:10.1038/nbt.2939 |
| [17] | Dick GJ, Andersson AF, Baker BJ, Simmons SL, Thomas BC, Yelton AP, Banfield JF. Community-wide analysis of microbial genome sequence signatures. Genome Biology, 2009, 10(8): R85. DOI:10.1186/gb-2009-10-8-r85 |
| [18] | Alneberg J, Bjarnason BS, de Bruijn I, Schirmer M, Quick J, Ijaz UZ, Loman NJ, Andersson AF, Quince C. CONCOCT: clustering contigs on coverage and composition. arXiv preprint arXiv: 13124038, 2013. |
| [19] | Strous M, Kraft B, Bisdorf R, Tegetmeyer HE. The binning of metagenomic contigs for microbial physiology of mixed cultures. Frontiers in Microbiology, 2012, 3: 410. |
| [20] | Sieber CMK, Probst AJ, Sharrar A, Thomas BC, Hess M, Tringe SG, Banfield JF. Recovery of genomes from metagenomes via a dereplication, aggregation, and scoring strategy. bioRxiv, 2017, doi: 10.1101/107789. |
| [21] | Parks DH, Imelfort M, Skennerton CT, Hugenholtz P, Tyson GW. CheckM:assessing the quality of microbial genomes recovered from isolates, single cells, and metagenomes. Genome Research, 2015, 25(7): 1043-1055. DOI:10.1101/gr.186072.114 |
| [22] | Tyson GW, Chapman J, Hugenholtz P, Allen EE, Ram RJ, Richardson PM, Solovyev VV, Rubin EM, Rokhsar DS, Banfield JF. Community structure and metabolism through reconstruction of microbial genomes from the environment. Nature, 2004, 428(6978): 37-43. DOI:10.1038/nature02340 |
| [23] | Anantharaman K, Brown CT, Hug LA, Sharon I, Castelle CJ, Probst AJ, Thomas BC, Singh A, Wilkins MJ, Karaoz U, Brodie EL, Williams KH, Hubbard SS, Banfield JF. Thousands of microbial genomes shed light on interconnected biogeochemical processes in an aquifer system. Nature Communications, 2016, 7: 13219. DOI:10.1038/ncomms13219 |
| [24] | Stepanauskas R. Single cell genomics:an individual look at microbes. Current Opinion in Microbiology, 2012, 15(5): 613-620. DOI:10.1016/j.mib.2012.09.001 |
| [25] | Streets AM, Zhang X, Cao C, Pang Y, Wu X, Xiong L, Yang L, Fu Y, Zhao L, Tang F, Huang Y. Microfluidic single-cell whole-transcriptome sequencing. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(19): 7048-7053. DOI:10.1073/pnas.1402030111 |
| [26] | Zhang Q, Wang TT, Zhou Q, Zhang P, Gong YH, Gou HL, Xu J, Ma B. Development of a facile droplet-based single-cell isolation platform for cultivation and genomic analysis in microorganisms. Scientific Reports, 2017, 7: 41192. DOI:10.1038/srep41192 |
| [27] | Jiang CY, Dong LB, Zhao JK, Hu XF, Shen CH, Qiao YX, Zhang XY, Wang YP, Ismagilov RF, Liu SJ, Du WB, Parales RE. High-throughput single-cell cultivation on microfluidic streak plates. Applied and Environmental Microbiology, 2016, 82(7): 2210-2218. DOI:10.1128/AEM.03588-15 |
| [28] | Lasken RS. Single-cell genomic sequencing using multiple displacement amplification. Current Opinion in Microbiology, 2007, 10(5): 510-516. DOI:10.1016/j.mib.2007.08.005 |
| [29] | Schmieder R, Edwards R. Fast identification and removal of sequence contamination from genomic and metagenomic datasets. PLoS One, 2011, 6(3): e17288. DOI:10.1371/journal.pone.0017288 |
| [30] | Eren AM, Esen ?C, Quince C, Vineis JH, Morrison HG, Sogin ML, Delmont TO. Anvi'o:an advanced analysis and visualization platform for 'omics data'. PeerJ, 2015, 3(358): e1319. |
| [31] | Tennessen K, Andersen E, Clingenpeel S, Rinke C, Lundberg DS, Han J, Dangl JL, Ivanova N, Woyke T, Kyrpides N, Pati A. ProDeGe:a computational protocol for fully automated decontamination of genomes. The ISME Journal, 2016, 10(1): 269-272. DOI:10.1038/ismej.2015.100 |
| [32] | Dodsworth JA, Blainey PC, Murugapiran SK, Swingley WD, Ross CA, Tringe SG, Chain PSG, Scholz MB, Lo CC, Raymond J, Quake SR, Hedlund BP. Single-cell and metagenomic analyses indicate a fermentative and saccharolytic lifestyle for members of the OP9 lineage. Nature Communications, 2013, 4: 1854. DOI:10.1038/ncomms2884 |
| [33] | Peng Y, Leung HCM, Yiu SM, Chin FYL. IDBA-UD:a de novo assembler for single-cell and metagenomic sequencing data with highly uneven depth. Bioinformatics, 2012, 28(11): 1420-1428. DOI:10.1093/bioinformatics/bts174 |
| [34] | Konstantinidis KT, Ramette A, Tiedje JM. The bacterial species definition in the genomic era. Philosophical Transactions of the Royal Society B:Biological Sciences, 2006, 361(1475): 1929-1940. DOI:10.1098/rstb.2006.1920 |
| [35] | Lasken RS. Genomic sequencing of uncultured microorganisms from single cells. Nature Reviews Microbiology, 2012, 10(9): 631-640. DOI:10.1038/nrmicro2857 |
| [36] | Walker A. Adding genomic 'foliage' to the tree of life. Nature Reviews Microbiology, 2014, 12(2): 78-79. DOI:10.1038/nrmicro3203 |
| [37] | Blainey PC. The future is now:single-cell genomics of bacteria and archaea. FEMS Microbiology Reviews, 2013, 37(3): 407-427. DOI:10.1111/1574-6976.12015 |
| [38] | Pelletier E, Kreimeyer A, Bocs S, Rouy Z, Gyapay G, Chouari R, Rivière D, Ganesan A, Daegelen P, Sghir A, Cohen GN, Medigue C, Weissenbach J, Le Paslier D. "Candidatus cloacamonas acidaminovorans":genome sequence reconstruction provides a first glimpse of a new bacterial division. Journal of Bacteriology, 2008, 190(7): 2572-2579. DOI:10.1128/JB.01248-07 |
| [39] | Elkins JG, Podar M, Graham DE, Makarova KS, Wolf Y, Randau L, Hedlund BP, Brochier-Armanet C, Kunin V, Anderson I, Lapidus A, Goltsman E, Barry K, Koonin EV, Hugenholtz P, Kyrpides N, Wanner G, Richardson P, Keller M, Stetter KO. A korarchaeal genome reveals insights into the evolution of the Archaea. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(23): 8102-8107. DOI:10.1073/pnas.0801980105 |
| [40] | Erkel C, Kube M, Reinhardt R, Liesack W. Genome of rice cluster Ⅰ archaea-the key methane producers in the rice rhizosphere. Science, 2006, 313(5785): 370-372. DOI:10.1126/science.1127062 |
| [41] | Lücker S, Wagner M, Maixner F, Pelletier E, Koch H, Vacherie B, Rattei T, Damsté JSS, Spieck E, Le Paslier D, Daims H. A Nitrospira metagenome illuminates the physiology and evolution of globally important nitrite-oxidizing bacteria. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(30): 13479-13484. DOI:10.1073/pnas.1003860107 |
| [42] | Duan YP, Zhou LJ, Hall DG, Li WB, Doddapaneni H, Lin H, Liu L, Vahling CM, Gabriel DW, Williams KP, Dickerman A, Sun YJ, Gottwald T. Complete genome sequence of citrus huanglongbing bacterium, 'Candidatus Liberibacter asiaticus' obtained through metagenomics. Molecular Plant-Microbe Interactions, 2009, 22(8): 1011-1020. DOI:10.1094/MPMI-22-8-1011 |
| [43] | Nunoura T, Takaki Y, Kakuta J, Nishi S, Sugahara J, Kazama H, Chee GJ, Hattori M, Kanai A, Atomi H. Insights into the evolution of Archaea and eukaryotic protein modifier systems revealed by the genome of a novel archaeal group. Nucleic Acids Research, 2010, 39(8): 3204-3223. |
| [44] | Takami H, Noguchi H, Takaki Y, Uchiyama I, Toyoda A, Nishi S, Chee G-J, Arai W, Nunoura T, Itoh T, Hattori M, Takai K. A deeply branching thermophilic bacterium with an ancient acetyl-CoA pathway dominates a subsurface ecosystem. PLoS One, 2012, 7(1): e30559. DOI:10.1371/journal.pone.0030559 |
| [45] | van de Vossenberg J, Woebken D, Maalcke WJ, Wessels HJC, Dutilh BE, Kartal B, Janssen-Megens EM, Roeselers G, Yan J, Speth D, Gloerich J, Geerts W, van der Biezen E, Pluk W, Francoijs KJ, Russ L, Lam P, Malfatti SA, Tringe SG, Haaijer SCM, Op den Camp HJM, Stunnenberg HG, Amann R, Kuypers MMM, Jetten MSM. The metagenome of the marine anammox bacterium 'Candidatus Scalindua profunda' illustrates the versatility of this globally important nitrogen cycle bacterium. Environmental Microbiology, 2013, 15(5): 1275-1289. DOI:10.1111/emi.2013.15.issue-5 |
| [46] | Bertin PN, Heinrich-Salmeron A, Pelletier E, Goulhen-Chollet F, Arsene-Ploetze F, Gallien S, Lauga B, Casiot C, Calteau A, Vallenet D, Bonnefoy V, Bruneel O, Chane-Woon-Ming B, Cleiss-Arnold J, Duran R, Elbaz-Poulichet F, Fonknechten N, Giloteaux L, Halter D, Koechler S, Marchal M, Mornico D, Schaeffer C, Smith AAT, van Dorsselaer A, Weissenbach J, Médigue C, Le Paslier D. Metabolic diversity among main microorganisms inside an arsenic-rich ecosystem revealed by meta-and proteo-genomics. The ISME Journal, 2011, 5(11): 1735-1747. DOI:10.1038/ismej.2011.51 |
| [47] | Flowers JJ, He SM, Malfatti S, Del Rio TG, Tringe SG, Hugenholtz P, McMahon KD. Comparative genomics of two 'Candidatus Accumulibacter' clades performing biological phosphorus removal. The ISME Journal, 2013, 7(12): 2301-2314. DOI:10.1038/ismej.2013.117 |
| [48] | Ghai R, Mizuno CM, Picazo A, Camacho A, Rodriguez-Valera F. Metagenomics uncovers a new group of low GC and ultra-small marine Actinobacteria. Scientific Reports, 2013, 3: 2471. DOI:10.1038/srep02471 |
| [49] | Lynch RC, Darcy JL, Kane NC, Nemergut DR, Schmidt SK. Metagenomic evidence for metabolism of trace atmospheric gases by high-elevation desert Actinobacteria. Frontiers in Microbiology, 2014, 5: 698. |
| [50] | Mondav R, Woodcroft BJ, Kim EH, McCalley CK, Hodgkins SB, Crill PM, Chanton J, Hurst GB, VerBerkmoes NC, Saleska SR, Hugenholtz P, Rich VI, Tyson GW. Discovery of a novel methanogen prevalent in thawing permafrost. Nature Communications, 2014, 5: 3212. |
| [51] | Hua ZS, Han YJ, Chen LX, Liu J, Hu M, Li SJ, Kuang JL, Chain PSG, Huang LN, Shu WS. Ecological roles of dominant and rare prokaryotes in acid mine drainage revealed by metagenomics and metatranscriptomics. The ISME Journal, 2015, 9(6): 1280-1294. DOI:10.1038/ismej.2014.212 |
| [52] | Zhalnina KV, Dias R, Leonard MT, de Quadros PD, Camargo FA, Drew JC, Farmerie WG, Daroub SH, Triplett EW. Genome sequence of Candidatus Nitrososphaera evergladensis from group Ⅰ. 1b enriched from Everglades soil reveals novel genomic features of the ammonia-oxidizing archaea. PLoS One, 2014, 9(7): e101648. DOI:10.1371/journal.pone.0101648 |
| [53] | Hug LA, Baker BJ, Anantharaman K, Brown CT, Probst AJ, Castelle CJ, Butterfield CN, Hernsdorf AW, Amano Y, Ise K, Suzuki Y, Dudek N, Relman DA, Finstad KM, Amundson R, Thomas BC, Banfield JF. A new view of the tree of life. Nature Microbiology, 2016, 1: 16048. DOI:10.1038/nmicrobiol.2016.48 |
| [54] | Parks DH, Rinke C, Chuvochina M, Chaumeil P-A, Woodcroft BJ, Evans PN, Hugenholtz P, Tyson GW. Recovery of nearly 8, 000 metagenome-assembled genomes substantially expands the tree of life. Nature Microbiology, 2017, 2: 1533-1542. DOI:10.1038/s41564-017-0012-7 |
| [55] | Paez-Espino D, Eloe-Fadrosh EA, Pavlopoulos GA, Thomas AD, Huntemann M, Mikhailova N, Rubin E, Ivanova NN, Kyrpides NC. Uncovering Earth's virome. Nature, 2016, 536(7617): 425-430. DOI:10.1038/nature19094 |
| [56] | Blainey PC, Mosier AC, Potanina A, Francis CA, Quake SR. Genome of a low-salinity ammonia-oxidizing archaeon determined by single-cell and metagenomic analysis. PLoS One, 2011, 6(2): e16626. DOI:10.1371/journal.pone.0016626 |
| [57] | He XS, McLean JS, Edlund A, Yooseph S, Hall AP, Liu SY, Dorrestein PC, Esquenazi E, Hunter RC, Cheng GH, Nelson KE, Lux R, Shi WY. Cultivation of a human-associated TM7 phylotype reveals a reduced genome and epibiotic parasitic lifestyle. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(1): 244-249. DOI:10.1073/pnas.1419038112 |
| [58] | McLean JS, Lombardo M-J, Badger JH, Edlund A, Novotny M, Yee-Greenbaum J, Vyahhi N, Hall AP, Yang Y, Dupont CL, Ziegler MG, Chitsaz H, Allen AE, Yooseph S, Tesler G, Pevzner PA, Friedman RM, Nealson KH, Venter JC, Lasken RS. Candidate phylum TM6 genome recovered from a hospital sink biofilm provides genomic insights into this uncultivated phylum. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(26): E2390-E2399. DOI:10.1073/pnas.1219809110 |
| [59] | Youssef NH, Blainey PC, Quake SR, Elshahed MS. Partial genome assembly for a candidate division OP11 single cell from an anoxic spring (Zodletone Spring, Oklahoma). Applied and Environmental Microbiology, 2011, 77(21): 7804-7814. DOI:10.1128/AEM.06059-11 |
| [60] | Fullerton H, Moyer CL. Comparative single-cell genomics of Chloroflexi from the Okinawa Trough deep-subsurface biosphere. Applied and Environmental Microbiology, 2016, 82(10): 3000-3008. DOI:10.1128/AEM.00624-16 |
| [61] | Swan BK, Martinez-Garcia M, Preston CM, Sczyrba A, Woyke T, Lamy D, Reinthaler T, Poulton NJ, Masland EDP, Gomez ML, Sieracki ME, DeLong EF, Herndl GJ, Stepanauskas R. Potential for chemolithoautotrophy among ubiquitous bacteria lineages in the dark ocean. Science, 2011, 333(6047): 1296-1300. DOI:10.1126/science.1203690 |
| [62] | Hongoh Y, Sharma VK, Prakash T, Noda S, Taylor TD, Kudo T, Sakaki Y, Toyoda A, Hattori M, Ohkuma M. Complete genome of the uncultured Termite Group 1 bacteria in a single host protist cell. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(14): 5555-5560. DOI:10.1073/pnas.0801389105 |
| [63] | Engel P, Stepanauskas R, Moran NA. Hidden diversity in honey bee gut symbionts detected by single-cell genomics. PLoS Genetics, 2014, 10(9): e1004596. DOI:10.1371/journal.pgen.1004596 |
| [64] | Dupont CL, Rusch DB, Yooseph S, Lombardo MJ, Richter RA, Valas R, Novotny M, Yee-Greenbaum J, Selengut JD, Haft DH, Halpern AL, Lasken RS, Nealson K, Friedman R, Craig Venter J. Genomic insights to SAR86, an abundant and uncultivated marine bacterial lineage. The ISME Journal, 2012, 6(6): 1186-1199. DOI:10.1038/ismej.2011.189 |
| [65] | Nobu MK, Narihiro T, Rinke C, Kamagata Y, Tringe SG, Woyke T, Liu WT. Microbial dark matter ecogenomics reveals complex synergistic networks in a methanogenic bioreactor. The ISME Journal, 2015, 9(8): 1710-1722. DOI:10.1038/ismej.2014.256 |
| [66] | Mason OU, Hazen TC, Borglin S, Chain PSG, Dubinsky EA, Fortney JL, Han J, Holman HYN, Hultman J, Lamendella R, Mackelprang R, Malfatti S, Ttephanieom LM, Tringe SG, Woyke T, Zhou JZ, Rubin EM, Jansson JK. Metagenome, metatranscriptome and single-cell sequencing reveal microbial response to Deepwater Horizon oil spill. The ISME Kournal, 2012, 6(9): 1715-1727. DOI:10.1038/ismej.2012.59 |
| [67] | Yu FQ, Blainey PC, Schulz F, Woyke T, Horowitz MA, Quake SR. Microfluidic-based mini-metagenomics enables discovery of novel microbial lineages from complex environmental samples. bioRxiv, 2017, doi: 10.1101/114496. |
| [68] | Turnbaugh PJ, Ley RE, Hamady M, Fraser-Liggett CM, Knight R, Gordon JI. The human microbiome project:exploring the microbial part of ourselves in a changing world. Nature, 2007, 449(7164): 804-810. DOI:10.1038/nature06244 |
| [69] | Gilbert JA, Jansson JK, Knight R. The Earth Microbiome project:successes and aspirations. BMC Biology, 2014, 12(1): 69. DOI:10.1186/s12915-014-0069-1 |
| [70] | Wu DY, Hugenholtz P, Mavromatis K, Pukall R, Dalin E, Ivanova NN, Kunin V, Goodwin L, Wu M, Tindall BJ, Hooper SD, Pati A, Lykidis A, Spring S, Anderson IJ, D'Haeseleer P, Zemla A, Singer M, Lapidus A, Nolan M, Copeland A, Han C, Chen F, Cheng JF, Lucas S, Kerfeld C, Lang E, Gronow S, Chain P, Bruce D, Rubin EM, Kyrpides NC, Klenk HP, Eisen JA. A phylogeny-driven genomic encyclopaedia of Bacteria and Archaea. Nature, 2009, 462(7276): 1056-1060. DOI:10.1038/nature08656 |
| [71] | Baker BJ, Dick GJ. Omic approaches in microbial ecology:charting the unknown. Microbe, 2013, 8: 353-360. |
| [72] | Liu SJ, Shi WY, Zhao GP. China microbiome initiative:opportunity and challenges. Bulletin of Chinese Academy of Sciences, 2017, 32(3): 241-250. (in Chinese) 刘双江, 施文元, 赵国屏. 中国微生物组计划:机遇与挑战. 中国科学院院刊, 2017, 32(3): 241-250. |
| [73] | Dubilier N, McFall-Ngai M, Zhao LP. Microbiology:Create a global microbiome effort. Nature, 2015, 526(7575): 631-634. DOI:10.1038/526631a |
| [74] | Bowers RM, Kyrpides NC, Stepanauskas R, Harmon-Smith M, Doud D, Reddy TBK, Schulz F, Jarett J, Rivers AR, Eloe-Fadrosh EA, Tringe SG, Ivanova NN, Copeland A, Clum A, Becraft ED, Malmstrom RR, Birren B, Podar M, Bork P, Weinstock GM, Garrity GM, Dodsworth JA, Yooseph S, Sutton G, Gl?ckner FO, Gilbert JA, Nelson WC, Hallam SJ, Jungbluth SP, Ettema TJG, Tighe S, Konstantinidis KT, Liu WT, Baker BJ, Rattei T, Eisen JA, Hedlund B, McMahon KD, Fierer N, Knight R, Finn R, Cochrane G, Karsch-Mizrachi I, Tyson GW, Rinke C, Genome Standards Consortium, Lapidus A, Meyer F, Yilmaz P, Parks DH, Eren AM, Schriml L, Banfield JF, Hugenholtz P, Woyke T. Minimum information about a single amplified genome (MISAG) and a metagenome-assembled genome (MIMAG) of bacteria and archaea. Nature Biotechnology, 2017, 35(8): 725-731. DOI:10.1038/nbt.3893 |
| [75] | Lloyd KG, Schreiber L, Petersen DG, Kjeldsen KU, Lever MA, Steen AD, Stepanauskas R, Richter M, Kleindienst S, Lenk S, Schramm A, J?rgensen BB. Predominant archaea in marine sediments degrade detrital proteins. Nature, 2013, 496(7444): 215-218. DOI:10.1038/nature12033 |
| [76] | Kozubal MA, Romine M, deM Jennings R, Jay ZJ, Tringe SG, Rusch DB, Beam JP, McCue LA, Inskeep WP. Geoarchaeota:a new candidate phylum in the Archaea from high-temperature acidic iron mats in Yellowstone National Park. The ISME Journal, 2013, 7(3): 622-634. DOI:10.1038/ismej.2012.132 |
| [77] | Zaremba-Niedzwiedzka K, Caceres EF, Saw JH, B?ckstr?m D, Juzokaite L, Vancaester E, Seitz KW, Anantharaman K, Starnawski P, Kjeldsen KU, Stott MB, Nunoura T, Banfield JF, Schramm A, Baker BJ, Spang A, Ettema TJG. Asgard archaea illuminate the origin of eukaryotic cellular complexity. Nature, 2017, 541(7637): 353-358. DOI:10.1038/nature21031 |
| [78] | Hugenholtz P, Pitulle C, Hershberger KL, Pace NR. Novel division level bacterial diversity in a Yellowstone hot spring. Journal of Bacteriology, 1998, 180(2): 366-376. |
| [79] | Spang A, Saw JH, J?rgensen SL, Zaremba-Niedzwiedzka K, Martijn J, Lind AE, van Eijk R, Schleper C, Guy L, Ettema TJ. Complex archaea that bridge the gap between prokaryotes and eukaryotes. Nature, 2015, 521(7551): 173-179. DOI:10.1038/nature14447 |
| [80] | Huber H, Hohn MJ, Rachel R, Fuchs T, Wimmer VC, Stetter KO. A new phylum of Archaea represented by a nanosized hyperthermophilic symbiont. Nature, 2002, 417(6884): 63-67. DOI:10.1038/417063a |
| [81] | Ghai R, Pa?i? L, Fernández AB, Martin-Cuadrado AB, Mizuno CM, McMahon KD, Papke RT, Stepanauskas R, Rodriguez-Brito B, Rohwer F, Sánchez-Porro C, Ventosa A, Rodríguez-Valera F. New abundant microbial groups in aquatic hypersaline environments. Scientific Reports, 2011, 1: 135. DOI:10.1038/srep00135 |
| [82] | Castelle CJ, Wrighton KC, Thomas BC, Hug LA, Brown CT, Wilkins MJ, Frischkorn KR, Tringe SG, Singh A, Markillie LM, Taylor RC, Williams KH, Banfield JF. Genomic expansion of domain archaea highlights roles for organisms from new phyla in anaerobic carbon cycling. Current Biology, 2015, 25(6): 690-701. DOI:10.1016/j.cub.2015.01.014 |
| [83] | Baker BJ, Comolli LR, Dick GJ, Hauser LJ, Hyatt D, Dill BD, Land ML, VerBerkmoes NC, Hettich RL, Banfield JF. Enigmatic, ultrasmall, uncultivated Archaea. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(19): 8806-8811. DOI:10.1073/pnas.0914470107 |
| [84] | Seitz KW, Lazar CS, Hinrichs KU, Teske AP, Baker BJ. Genomic reconstruction of a novel, deeply branched sediment archaeal phylum with pathways for acetogenesis and sulfur reduction. The ISME Journal, 2016, 10(7): 1696-1705. DOI:10.1038/ismej.2015.233 |
| [85] | Vanwonterghem I, Evans PN, Parks DH, Jensen PD, Woodcroft BJ, Hugenholtz P, Tyson GW. Methylotrophic methanogenesis discovered in the archaeal phylum Verstraetearchaeota. Nature Microbiology, 2016, 1: 16170. DOI:10.1038/nmicrobiol.2016.170 |
| [86] | Wrighton KC, Thomas BC, Sharon I, Miller CS, Castelle CJ, VerBerkmoes NC, Wilkins MJ, Hettich RL, Lipton MS, Williams KH, Long PE, Banfield JF. Fermentation, hydrogen, and sulfur metabolism in multiple uncultivated bacterial phyla. Science, 2012, 337(6102): 1661-1665. DOI:10.1126/science.1224041 |
| [87] | Wrighton KC, Castelle CJ, Wilkins MJ, Hug LA, Sharon I, Thomas BC, Handley KM, Mullin SW, Nicora CD, Singh A, Lipton MS, Long PE, Williams KH, Banfield JF. Metabolic interdependencies between phylogenetically novel fermenters and respiratory organisms in an unconfined aquifer. The ISME Journal, 2014, 8(7): 1452-1463. DOI:10.1038/ismej.2013.249 |
| [88] | Eloe-Fadrosh EA, Paez-Espino D, Jarett J, Dunfield PF, Hedlund BP, Dekas AE, Grasby SE, Brady AL, Dong HL, Briggs BR, Li WJ, Goudeau D, Malmstrom R, Pati A, Pett-Ridge J, Rubin EM, Woyke T, Kyrpides NC, Ivanova NN. Global metagenomic survey reveals a new bacterial candidate phylum in geothermal springs. Nature Communications, 2016, 7: 10476. DOI:10.1038/ncomms10476 |
| [89] | Sekiguchi Y, Ohashi A, Parks DH, Yamauchi T, Tyson GW, Hugenholtz P. First genomic insights into members of a candidate bacterial phylum responsible for wastewater bulking. PeerJ, 2015, 3: e740. DOI:10.7717/peerj.740 |
| [90] | Dojka MA, Hugenholtz P, Haack SK, Pace NR. Microbial diversity in a hydrocarbon-and chlorinated-solvent-contaminated aquifer undergoing intrinsic bioremediation. Applied and Environmental Microbiology, 1998, 64(10): 3869-3877. |
| [91] | Di Rienzi SC, Sharon I, Wrighton KC, Koren O, Hug LA, Thomas BC, Goodrich JK, Bell JT, Spector TD, Banfield JF, Ley RE. The human gut and groundwater harbor non-photosynthetic bacteria belonging to a new candidate phylum sibling to Cyanobacteria. Elife, 2013, 2: e01102. |
| [92] | Ettwig KF, Butler MK, Le Paslier D, Pelletier E, Mangenot S, Kuypers MMM, Schreiber F, Dutilh BE, Zedelius J, de Beer D, Gloerich J, Wessels HJCT, van Alen T, Luesken F, Wu ML, van de Pas-Schoonen KT, Op den Camp HJM, Janssen-Megens EM, Francoijs KJ, Stunnenberg H, Weissenbach J, Jetten MSM, Strous M. Nitrite-driven anaerobic methane oxidation by oxygenic bacteria. Nature, 2010, 464(7288): 543-548. DOI:10.1038/nature08883 |
| [93] | Siegl A, Kamke J, Hochmuth T, Piel J, Richter M, Liang CG, Dandekar T, Hentschel U. Single-cell genomics reveals the lifestyle of Poribacteria, a candidate phylum symbiotically associated with marine sponges. The ISME Journal, 2011, 5(1): 61-70. DOI:10.1038/ismej.2010.95 |
| [94] | Rheims H, Rainey FA, Stackebrandt E. A molecular approach to search for diversity among bacteria in the environment. Journal of Industrial Microbiology, 1996, 17(3/4): 159-169. |
| [95] | Wang ZP, Guo F, Liu LL, Zhang T. Evidence of carbon fixation pathway in a bacterium from candidate phylum SBR1093 revealed with genomic analysis. PLoS One, 2014, 9(10): e109571. DOI:10.1371/journal.pone.0109571 |
| [96] | Kantor RS, Wrighton KC, Handley KM, Sharon I, Hug LA, Castelle CJ, Thomas BC, Banfield JF. Small genomes and sparse metabolisms of sediment-associated bacteria from four candidate phyla. mBio, 2013, 4(5): e00708-13. |
| [97] | Wilson MC, Mori T, Rückert C, Uria AR, Helf MJ, Takada K, Gernert C, Steffens UAE, Heycke N, Schmitt S, Rinke C, Helfrich EJN, Brachmann AO, Gurgui C, Wakimoto T, Kracht M, Crüsemann M, Hentschel U, Abe I, Matsunaga S, Kalinowski J, Takeyama H, Piel J. An environmental bacterial taxon with a large and distinct metabolic repertoire. Nature, 2014, 506(7486): 58-62. DOI:10.1038/nature12959 |
| [98] | Wagner M, Nielsen PH, Loy A, Nielsen JL, Daims H. Linking microbial community structure with function:fluorescence in situ hybridization-microautoradiography and isotope arrays. Current Opinion in Biotechnology, 2006, 17(1): 83-91. DOI:10.1016/j.copbio.2005.12.006 |
| [99] | Behrens S, L?sekann T, Pett-Ridge J, Weber PK, Ng WO, Stevenson BS, Hutcheon ID, Relman DA, Spormann AM. Linking microbial phylogeny to metabolic activity at the single-cell level by using enhanced element labeling-catalyzed reporter deposition fluorescence in situ hybridization (EL-FISH) and NanoSIMS. Applied and Environmental Microbiology, 2008, 74(10): 3143-3150. DOI:10.1128/AEM.00191-08 |
| [100] | Neufeld JD, Murrell JC. Witnessing the last supper of uncultivated microbial cells with Raman-FISH. The ISME Journal, 2007, 1(4): 269-270. DOI:10.1038/ismej.2007.55 |
| [101] | Xavier M, Peter KW, Jennifer PR. Taxon-specific C/N relative use efficiency for amino acids in an estuarine community. FEMS Microbiology Ecology, 2013, 83(2): 402-412. DOI:10.1111/j.1574-6941.12000.x |
| [102] | Temme K, Zhao D, Voigt CA. Refactoring the nitrogen fixation gene cluster from Klebsiella oxytoca. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(18): 7085-7090. DOI:10.1073/pnas.1120788109 |
| [103] | Murray RG, Schleifer KH. Taxonomic notes:a proposal for recording the properties of putative taxa of procaryotes. International Journal of Systematic Bacteriology, 1994, 44(1): 174-176. DOI:10.1099/00207713-44-1-174 |
| [104] | Oren A. A plea for linguistic accuracy-also for Candidatus taxa. International Journal of Systematic and Evolutionary Microbiology, 2017, 67(4): 1085-1094. DOI:10.1099/ijsem.0.001715 |
| [105] | Whitman WB. Genome sequences as the type material for taxonomic descriptions of prokaryotes. Systematic and Applied Microbiology, 2015, 38(4): 217-222. DOI:10.1016/j.syapm.2015.02.003 |
| [106] | Konstantinidis KT, Rosselló-Móra R, Amann R. Uncultivated microbes in need of their own taxonomy. The ISME Journal, 2017, 11(11): 2399-2406. DOI:10.1038/ismej.2017.113 |
| [107] | Shleeva MO, Kudykina YK, Vostroknutova GN, Suzina NE, Mulyukin AL, Kaprelyants AS. Dormant ovoid cells of Mycobacterium tuberculosis are formed in response to gradual external acidification. Tuberculosis, 2011, 91(2): 146-154. DOI:10.1016/j.tube.2010.12.006 |
| [108] | Nichols D, Cahoon N, Trakhtenberg EM, Pham L, Mehta A, Belanger A, Kanigan T, Lewis K, Epstein SS. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Applied and Environmental Microbiology, 2010, 76(8): 2445-2450. DOI:10.1128/AEM.01754-09 |
| [109] | Lewis K. Antibiotics from the microbial dark matter. The FASEB Journal, 2017, 31(1 Supplement): Abstract Number: 257. 2. |
| [110] | Lackner G, Peters EE, Helfrich EJN, Piel J. Insights into the lifestyle of uncultured bacterial natural product factories associated with marine sponges. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(3): E347-E356. DOI:10.1073/pnas.1616234114 |
| [111] | Ling LL, Schneider T, Peoples AJ, Spoering AL, Engels I, Conlon BP, Mueller A, Sch?berle TF, Hughes DE, Epstein S, Jones M, Lazarides L, Steadman VA, Cohen DR, Felix CR, Fetterman KA, Millett WP, Nitti AG, Zullo AM, Chen C, Lewis K. A new antibiotic kills pathogens without detectable resistance. Nature, 2015, 517(7535): 455-459. DOI:10.1038/nature14098 |
| [112] | Woyke T, Rubin EM. Searching for new branches on the tree of life. Science, 2014, 346(6210): 698-699. DOI:10.1126/science.1258871 |
| [113] | Hedlund BP, Murugapiran SK, Alba TW, Levy A, Dodsworth JA, Goertz GB, Ivanova N, Woyke T. Uncultivated thermophiles:current status and spotlight on 'Aigarchaeota'. Current Opinion in Microbiology, 2015, 25: 136-145. DOI:10.1016/j.mib.2015.06.008 |
| [114] | Zhou EM, Yang LL, Song ZQ, Yu TT, Nie GX, Ming H, Zhou Y, Tang SK, Li WJ. Thermocatellispora tengchongensis gen. nov., sp. nov., a new member of the family Streptosporangiaceae. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(Pt 10): 2417-2423. DOI:10.1099/ijs.0.036897-0 |
| [115] | Zhou EM, Yu TT, Liu L, Ming H, Yin YR, Dong L, Tseng M, Nie GX, Li WJ. Geothermomicrobium terrae gen. nov., sp. nov., a novel member of the family Thermoactinomycetaceae. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(Pt 9): 2998-3004. DOI:10.1099/ijs.0.059766-0 |
| [116] | Song ZQ, Wang FP, Zhi XY, Chen JQ, Zhou EM, Liang F, Xiao X, Tang SK, Jiang HC, Zhang CL, Dong HL, Li WJ. Bacterial and archaeal diversities in Yunnan and Tibetan hot springs, China. Environmental Microbiology, 2013, 15(4): 1160-1175. DOI:10.1111/emi.2013.15.issue-4 |
| [117] | Wang S, Dong HL, Hou WG, Jiang HC, Huang QY, Briggs BR, Huang LQ. Greater temporal changes of sediment microbial community than its waterborne counterpart in Tengchong hot springs, Yunnan Province, China. Scientific Reports, 2014, 4: 7479. |
| [118] | Wang S, Hou WG, Dong HL, Jiang HC, Huang LQ, Wu G, Zhang CL, Song ZQ, Zhang Y, Ren HL, Zhang J, Zhang L. Control of temperature on microbial community structure in hot springs of the Tibetan Plateau. PLoS One, 2013, 8(5): e62901. DOI:10.1371/journal.pone.0062901 |
| [119] | Hou WG, Wang S, Dong HL, Jiang HC, Briggs BR, Peacock JP, Huang QY, Huang LQ, Wu G, Zhi XY, Li WJ, Dodsworth JA, Hedlund BP, Zhang CL, Hartnett HE, Dijkstra P, Hungate BA. A comprehensive census of microbial diversity in hot springs of Tengchong, Yunnan Province China using 16S rRNA gene pyrosequencing. PLoS One, 2013, 8(1): e53350. DOI:10.1371/journal.pone.0053350 |
