王博1,2, 陈坚1,3, 堵国成1,4, 方芳1,2


1.江南大学生物工程学院, 工业生物技术教育部重点实验室, 江苏 无锡 214122;
2.江南大学食品科学与技术国家重点实验室, 江苏 无锡 214122;
3.江南大学粮食发酵工艺与技术国家工程实验室, 江苏 无锡 214122;
4.江南大学糖化学与生物技术教育部重点实验室, 江苏 无锡 214122
收稿日期:2018-01-10;修回日期:2018-03-28;网络出版日期:2018-05-29
基金项目:国家自然科学基金(31771955)
*通信作者:方芳, Tel:+86-510-85918307, Fax:+86-510-85918309, E-mail:ffang@jiangnan.edu.cn
摘要:[目的]对酱油酱醪来源的嗜盐四联球菌进行分类,并比较菌株的生理特性。[方法]采用多位点序列分型和糖类代谢聚类分析相结合的方法对菌株进行分类,通过菌株对环境耐受性分析研究菌株的生理特性。[结果]酱醪来源的12株嗜盐四联球菌被分为两个类群,菌株所属类别与其分离的原生环境存在一定的关联性。嗜盐四联球菌C25、C33、C3、R55耐高盐及高糖能力均较强,菌株C1、R44和R23耐高盐能力较强。虽然嗜盐四联球菌普遍不耐酸,但是菌株C33和R44在酸性培养体系(pH 5.0)中仍能正常生长。较低的培养温度(15℃和25℃)对嗜盐四联球菌的生长有显著抑制。[结论]采用基因型与表型结合的方式成功将酱醪来源的嗜盐四联球菌归为2个类群,不同类群的菌株在耐高渗等生理特性上具有差异性。
关键词: 酱油 嗜盐四联球菌 多位点序列分型 糖类代谢 胁迫
Classification and characteristics of Tetragenococcus halophilus derived from moromi
Bo Wang1,2, Jian Chen1,3, Guocheng Du1,4, Fang Fang1,2


1.Key Laboratory of Industrial Biotechnology, Ministry of Education, School of Biotechnology, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
2.State Key Laboratory of Food Science and Technology, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
3.National Engineering Laboratory for Cereal Fermentation Technology, Jiangnan University, Wuxi 214122, Jiangsu Province, China;
4.The Key Laboratory of Carbohydrate Chemistry and Biotechnology, Ministry of Education, Jiangnan University, Wuxi 214122, Jiangsu Province, China
Received 10 January 2018; Revised 28 March 2018; Published online 29 May 2018
*Corresponding author: Fang Fang, Tel: +86-510-85918307; Fax: +86-510-85918309; E-mail: ffang@jiangnan.edu.cn
Supported by the National Natural Science Foundation of China (31771955)
Abstract: [Objective]This study aims to classify Tetragenococcus halophilus strains from soy sauce moromi mash using taxonomy analysis, and to characterize these strains.[Methods]T. halophilus strains were classified using multilocus sequence typing combined with cluster analysis of their carbohydrates metabolisms. Physiological characteristics of T. halophilus strains were studied by analyzing their tolerance to stress.[Results]Twelve T. halophilus strains of soy sauce mash origin were classified into two groups. The cluster or group type of individual strain is correlated with the environment where it derived from. T. halophilus strains C25, C33, C3 and R55 tolerated osmotic stress (high concentrations of sugar and salt), whereas strains R44, C1 and R23 exhibited better resistance to high concentrations of salt. Growth of stains C33 and R44 in acidic medium (pH 5.0) was not significantly inhibited though T. halophilus generally grows poorly under acidic conditions. Growth of T. halophilus strains was significantly inhibited when grown at low temperatures (15℃ and 25℃).[Conclusion]T. halophilus strains from soy sauce moromi mash origin were classified into two groups, using the methods based on genotype and phenotype categorizing. Strains from different groups exhibit differences in physiological characteristics.
Keywords: soy sauce Tetragenococcus halophilus multilocus sequence typing carbohydrate metabolism stress
乳酸菌在发酵食品生产过程中具有提高产品风味、质量和缩短发酵周期的作用。嗜盐四联球菌(Tetragenococcus halophilus)是一类耐高渗和嗜盐的乳酸菌,广泛存在于酱油、腌制食品和豆制品等发酵食品中[1]。嗜盐四联球菌可耐受高盐且具有多种蛋白酶活性,常用于提高发酵食品和调味品中总氨基酸含量和鲜味氨基酸的含量[2-3]。研究证实,将嗜盐四联球菌和酵母菌结合作为发酵剂补充在发酵食品生产过程中,可显著地提高发酵食品中酮类、醛类、醇类等多种风味物质的含量[4-6]。嗜盐四联球菌对发酵食品中的风味物质具有较为重要的贡献,尤其可使食品中具有明显增香效果的呋喃酮、糠醛和糠醇等物质的含量显著增加[7-8]。此外,嗜盐四联球菌还具有降低发酵食品中氨(胺)类有害物质的潜力,如减少酱油中氨基甲酸乙酯前体瓜氨酸的积累,降低发酵食品中生物胺含量等[9-11]。与其他广泛用于食品发酵乳酸菌具有良好的研究基础不同,目前关于嗜盐四联球菌系统分类、遗传特性和生理生化特性方面的研究基础较为薄弱,也缺乏相应的基因操作工具,对其在食品中的推广应用造成了一定影响。
乳酸菌中很多菌属的菌株多样性的特点较为显著。因此研究者为了系统研究菌株的遗传特性和生理特性,已开发并应用多种分子分型技术对乳酸菌进行分类,包括随机扩增DNA多态性分析(Randomly amplified polymorphic DNA,RAPD)、限制性酶切片段长度多态性分析(Restriction fragment length polymorphism,RFLP)、扩增片段长度多态性分析(Amplified fragment length polymorphism,AFLP)和多位点序列分型(Multilocus sequence typing,MLST)等[12-14]。其中RAPD、RFLP、AFLP等方法是通过凝胶电泳条带图谱的差异来分析菌株DNA序列差异,其结果难以在不同的研究间进行分析和比较[15]。MLST是通过基因测序技术分析不同菌株基因组中看家基因序列上的差异来对菌株进行分类。该方法具有稳定性好、简便快捷、重复性和分辨率更好的优势,现在已经广泛用于微生物菌株的种间或株间分类[16]。此外,微生物的生理生化特性分析如糖类代谢表型、产酶表型等也常用于菌株的分类研究。用这类表型分析方法获得的结果与基因型分类法获得的结果存在一定程度上的差异,因此也是用于微生物鉴定和分类的一种方式和重要补充[17]。
发酵食品的生产体系对生产菌株可能是胁迫环境,具有较强的耐受环境胁迫能力是菌株在发酵食品中进行推广应用的重要特性[18]。因此,在微生物分类的基础上对菌株进行适应生长或应用环境相关特性的研究,对于菌株的工业化应用和生产工艺优化具有重要的意义。
很多菌属的乳酸菌具有大的种间多样性特点[19],菌株的遗传和生理特性通常与菌株来源有一定的关联[20]。前期研究表明,酱醪来源的嗜盐四联球菌在基因型和物质代谢特性上有较大的区别[21]。因此,通过系统分类和特性分析,可以系统了解不同来源或同一来源的嗜盐四联球菌的生物学特性、耐受环境胁迫能力以及物质代谢特点,对于拓展嗜盐四联球菌在发酵食品中的应用具有重要意义。
1 材料和方法 1.1 菌株 嗜盐四联球菌(R23、R21、R44、R55、R8、R18、R9、R13、C25、C33、C3、C1)分离自高盐稀态的酱醪(R,强化鲁氏接合酵母工艺;C,不添加酵母工艺)。嗜盐四联球菌JCM 5888为模式菌,来源于腌鱼,购自中国普通微生物菌种保藏管理中心(CGMCC)。
1.2 培养基 种子培养基:含有100 g/L NaCl的MRS培养基(OXOID);高渗透压培养基:不含葡萄糖的MRS培养基(g/L,酵母膏5.0,牛肉膏5.0,胰蛋白胨5.0,七水合硫酸镁0.2,一水合硫酸锰0.05,硫酸铁0.4,柠檬酸三铵2.0,碳酸钙0.1,吡哆醛-5-磷酸0.05,磷酸氢二钾2.0,吐温80 1 mL),添加3 mol/L蔗糖溶液调节最终糖度分别为45 °Bx、48 °Bx、50 °Bx、55 °Bx、60 °Bx,糖度由手持式折光仪(TD-004,常州三丰仪器科技有限公司)测定。
1.3 嗜盐四联球菌多位点序列分型(MLST)分析
1.3.1 目的基因的选择与引物合成: 多位点序列分型选择基因需要尽量分布于整个基因组,大小适中,能够反映基因组变化情况,国际研究中通常选择单拷贝看家基因的保守序列进行分析[16]。本研究根据该原则从嗜盐四联球菌基因组的不同位置选取rpoB、recA、dnaA、uvrC、dnaK、pyrG、clpX和groEL这8个单拷贝的看家基因,作为MLST分析的靶基因(图 1)。靶基因及其编码产物信息见表 1。
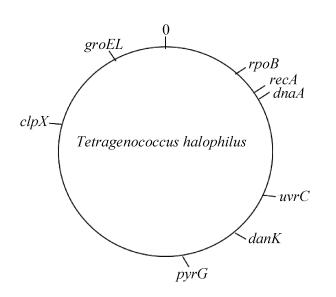 |
| 图 1 靶基因在嗜盐四联球菌基因组上的位点分布 Figure 1 Distribution of target genes on the genome of T. halophilus. |
| 图选项 |
表 1. 用于MLST分析的靶基因信息 Table 1. Target genes for MLST analysis
| Genes | Products |
| clpX | ATP-dependent Clp protease ATP-binding subunit ClpX |
| dnaA | Chromosomal replication initiator protein |
| dnaK | Molecular chaperone DnaK |
| groEL | Chaperonin GroEL |
| pyrG | CTP synthase |
| recA | Recombination protein RecA |
| rpoB | DNA-directed RNA polymerase subunit beta |
| uvrC | Excinuclease ABC subunit C |
表选项
使用软件Primer Premier 5设计靶基因引物(表 2),引物均由生工生物工程(上海)股份有限公司合成。
表 2. 用于MLST分析的引物 Table 2. Primers used for MLST analysis
| Primers | Sequences (5′→3′) | Genes | Amplicon/bp |
| clpX1 | TAGGACAAGATGAGGCT | clpX | 697 |
| clpX2 | ACAGGCAAACGACCGAT | ||
| dnaA1 | TATGATGAGCGGAGAAG | dnaA | 579 |
| dnaA2 | TGTGGCAATGTAGGGAT | ||
| dnaK1 | GAACTATTGTTTGGAGAAG | dnaK | 717 |
| dnaK2 | TGAGTCATTGAAATAAGCA | ||
| groEL1 | ACTTCTGGGGCTAATC | groEL | 692 |
| groEL2 | GAATCGCCAGCACCTT | ||
| pyrG1 | CTTAGGCATTCAACCC | pyrG | 586 |
| pyrG2 | AATCACATTTCTGGCA | ||
| recA1 | TAGATGTCGCATTAGGAG | recA | 622 |
| recA2 | GGGGGAGCAACTTTATTT | ||
| rpoB1 | ACAGATACATTGACACCG | rpoB | 715 |
| rpoB2 | CTTTACATAACAGAGCAG | ||
| uvrC1 | TGGGGCAGAGTATTTCGG | uvrC | 720 |
| uvrC2 | CGTTCTTGTGCTCGTTCG |
表选项
1.3.2 目的基因扩增与测序: 将菌株接种至种子培养基中,30 ℃静置培养。使用细菌DNA提取试剂盒(美国Omega Bio-Tek公司)提取嗜盐四联球菌基因组。基因的扩增(50 μL):2×Pfu DNA聚合酶25 μL,ddH2O 22 μL,上下游引物各1 μL (10 μmol/L),模板1 μL (50 ng/μL)。PCR扩增条件:95 ℃ 5 min;95 ℃ 30 s,56 ℃ 30 s,72 ℃ 1 min,30个循环;72 ℃ 10 min。PCR产物送至生工生物工程(上海)股份有限公司进行双向测序。
1.3.3 MLST分析及系统发育树构建: 利用DNASTAR软件中的SeqMan对测序获得的单个基因序列进行比对和拼接,通过MEGA 5.0比对不同菌株的看家基因序列,对嗜盐四联球菌进行分型组分析。
将每株嗜盐四联球菌的看家基因按照rpoB-recA-dnaA-uvrC-dnaK-pyrG-clpX-groEL的顺序连接成一个序列(总长度为4687 bp),利用MEGA 5.0软件中的邻接法(Neighbor-joining method)构建系统发育树。
1.4 嗜盐四联球菌糖类及其衍生物代谢分析 采用API? 50 CH试剂条(法国梅里埃公司)对嗜盐四联球菌利用49种糖类及其衍生物(多元醇、糖醛酸)的发酵情况进行分析。具体操作按照说明书进行。
1.5 嗜盐四联球菌耐高渗透压能力比较 将嗜盐四联球菌种子液分别以1%的接种量接种至糖度45 °Bx、48 °Bx、50 °Bx、55 °Bx和60 °Bx的高渗透压培养基中,30 ℃静置培养60 h,通过测定OD600 (OD600高于0.8的菌液用相同培养基稀释至OD600介于0.2-0.8之间)比较菌株耐高渗透压的能力。
1.6 寡肽酶活性测定 取2 mL嗜盐四联球菌培养液(109 CFU/mL)离心收集菌体,随后用0.2 mol/L的PBS缓冲液(pH 7.0)洗涤,使用液氮研磨法对菌体进行破壁,加1 mL PBS缓冲液重悬菌体,离心取上清液,作为寡肽酶粗酶液。取等量菌液离心获得菌体并在105 ℃下干燥至恒重,测定细胞干重。
寡肽酶活性测定采用改良的茚三酮显色法[22]。以二肽(Leu-Pro)和三肽(Leu-Leu-Leu)为底物,将20 μL上述粗酶液与60 μL反应液(0.2 mol/L PBS缓冲液,pH 7.0)和20 μL底物(10 mmol/L的Leu-Pro、5 mmol/L的Leu-Leu-Leu)混合,30 ℃下反应2 h,用茚三酮显色法在570 nm下检测游离氨基酸生成量。每分钟释放1 μmol的Leu定义为一个酶活力单位。
1.7 嗜盐四联球菌耐胁迫能力分析 将嗜盐四联球菌分别接种至含有100、180、220、250 g/L NaCl的MRS培养基中,30 ℃静置培养60 h,并使用分光光度计测定OD600,用于分析菌株对盐的耐受能力。
嗜盐四联球菌耐酸性能力分析采用对数后期(OD600=1.5)细胞。将离心收集的菌体垂悬于pH 5.0和pH 5.5的MRS (100 g/L NaCl)培养基中,在30 ℃静置培养5 h,采用的活菌数计算相对生长率,相对生长率按公式(1)计算。
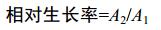 | 公式(1) |
嗜盐四联球菌耐受较低温度能力分析:将在30 ℃培养至对数后期(OD600=1.5)的嗜盐四联球菌分别转至15 ℃和25 ℃下静置培养5 h,采用的活菌数计算相对生长率,相对生长率按公式(2)计算。
 | 公式(2) |
2 结果和分析 2.1 嗜盐四联球菌MLST聚类分析 通过对分离自不同酱油生产工艺酱醪的84株嗜盐四联球菌进行精氨酸代谢和相关基因的初步分析发现,这些菌株在精氨酸代谢和基因型等特性上具有一定差异[21]。因此,本研究从中选取了包含不同特性和来源的12株嗜盐四联球菌代表性菌株进行进一步研究。为了对这些嗜盐四联球菌进行株间比较,我们首先开展了对菌株的分类分析。
基于基因序列的MLST的菌株分类,通常采用两种方法:一是分析等位基因与序列型(Sequence typing,ST),二是基于等位基因序列变化进行分析[23]。对嗜盐四联球菌的分类采用第二种方法,选择通过分析8个单拷贝的看家基因(rpoB、recA、dnaA、uvrC、dnaK、pyrG、clpX和groEL)的序列变化对菌株进行了分类。根据分型组的分类依据,如果进行比较的2个菌株基因组中的8个看家基因中有5个基因相同时,认为这2个菌株属于同一个分型组(Group)[23]。依据此方法,初步将12株酱醪来源的嗜盐四联球菌和1株模式菌分成7个分型组,酱醪来源的嗜盐四联球菌分布在6个分型组,与模式菌不同组(表 3)。
表 3. 嗜盐四联球菌分型组分析 Table 3. The typing group of T. halophilus
| Groups | Group 1 | Group 2 | Group 3 | Group 4 | Group 5 | Group 6 | Group 7 |
| Strains | R23, R21, R8, R13 | R18, R44, R55 | C33, C3 | R9 | C25 | C1 | JCM 5888T |
| T: T. halophilus type strain. | |||||||
表选项
在分型组分析的基础上,采用基因序列比对,进一步对嗜盐四联球菌进行精准聚类分析。利用MEGA 5.0软件将每株嗜盐四联球菌的8个看家基因序列连接为1个DNA序列,并用邻接法构建了聚类分析树。由图 2可知,嗜盐四联球菌被分为2个类群,每个类群中各自含有2个亚群。酱醪来源的7个嗜盐四联球菌(大多数来源于强化鲁氏接合酵母的高盐稀态酱油工艺)与模式菌是Ⅰ型,其余5株嗜盐四联球菌为Ⅱ型,与模式菌分型关系较近的是菌株C25。表 3的结果是用分型组方法初步对酱醪来源的嗜盐四联球菌菌株进行分组,这一分析结果是对较为相似菌株的初步分组,并不能说明菌株间差异大小或在进化树上的距离。图 2所示结果为采用邻接(Neighbor- Joining)法对相关菌株进行的聚类分析,此分类更为准确和精细,因此结果更为可靠。通过比较两种聚类的分析结果发现:两种分析方法除了对菌株R44分类有差异外,对其他菌株的分类有较好的一致性(如R23与R21、R13和R8较为相似,R18与R55较为相似,C33与C3较为相似,C25与C33和C3存在差异等)。此外,通过分型分析发现,嗜盐四联球菌的菌株分型与其来源有一定关联性,来源于相同工艺酱醪的菌株大多被聚类为同一类群。
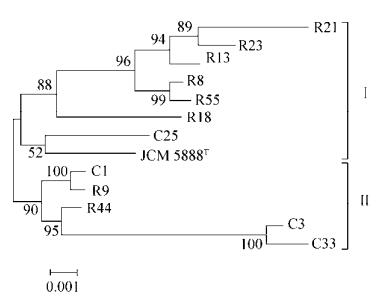 |
| 图 2 嗜盐四联球菌聚类分析 Figure 2 Clustering analysis of T. halophilus strains. A joint sequence of eight house-keeping genes (rpoB, recA, dnaA, uvrC, dnaK, pyrG, clpX and groEL) of T. halophilus was used to construct the neighbor-joining tree by using MEGA 5.0. T: T. halophilus type strain. |
| 图选项 |
2.2 嗜盐四联球菌糖类发酵特性的比较 微生物利用糖类碳源的发酵特性也经常作为菌株间分类的依据。本研究采用API? 50 CH试剂条考察了嗜盐四联球菌对49种糖类及其衍生物的利用情况,结果见图 3。嗜盐四联球菌可利用进行发酵的糖类及其衍生物的种类在20个左右,可发酵核糖、葡萄糖、果糖、甘露糖等,不能利用木糖、鼠李糖、棉子糖、木糖醇等。此外,从添加酵母酱油生产工艺的酱醪中分离的菌株50%可利用阿拉伯糖,而不添加酵母工艺的酱醪中分离得到的菌株都可以利用阿拉伯糖。根据菌株对糖类的发酵特性对菌株进行聚类分析,发现酱醪来源的嗜盐四联球菌可分为3类:R23是第一类,R9和R55是第2类,其他菌株与模式菌分在第三类。在利用糖类物质发酵这一特性上R23与其他菌株的不同之处在于:R23可利用甲基-α-吡喃葡萄糖苷,其他菌株不能;其他菌株都利用D-半乳糖和N-乙酰葡萄糖胺,R23则不能。第二类的菌株R9和R55不能发酵苦杏仁苷、熊果苷、水杨苷、纤维二糖和龙胆二糖产酸。
 |
| 图 3 基于糖类发酵特性的嗜盐四联球菌聚类分析 Figure 3 Clustering of T. halophilus based on their carbohydrate fermentative profiles. Black, positive reaction, fermentative; grey, partially fermentative; white, negative reaction, non-fermentative. GLY-5KG, abbreviations for sugars and their derivatives, represent glycerol-potassium 5-ketogluconate. |
| 图选项 |
细菌对于糖类物质的利用主要依赖菌株所具有的完整代谢途径和功能性转运蛋白,糖类代谢表现型上的差异往往反映了部分代谢相关基因在不同生态位上的进化,具体表现为基因的插入与缺失[24-25]。Roling等利用RAPD和糖类利用相结合的方法对来自酱油的嗜盐四联球菌进行了种间分类。结果显示,依据RAPD分类和糖类利用特性分类得到的菌株分型与来源有一定关联性,并且从不强化酵母工艺的酱醪中分离到的菌株都可利用阿拉伯糖,而从强化酵母工艺的酱醪中分离到的60%的菌株不利用阿拉伯糖[26]。与MLST依据看家基因保守序列差异进行分类的方法不同,利用糖发酵特性对菌株进行分类涉及代谢途径较多且受基因转录调控影响,更能体现菌株间的整体差异性或类似性。因此分子分型结果和表型数据在对菌株分类上有一致的方面,但也会存在一定的差异[27-28]。采用分子分型与表型结合的菌株分类方法可更加全面和客观反映菌株间的系统发育关系。本研究中采用的两种方式对酱醪来源的嗜盐四联球菌菌株分类并不完全相同,但结果显示两种分类方式之间仍具有相似性:分离自不添加酵母酱油发酵工艺条件下酱醪中的大多数菌株聚类为同一类群(C3、C33、C1、C25),它们对有些糖(阿拉伯糖、苦杏仁苷、纤维二糖)的代谢特性与添加酵母工艺条件酱醪来源的菌株(R23、R55等)有显著区别;R44在分子分型和表现型上与菌株C3和C33较接近。
2.3 嗜盐四联球菌耐高渗透压能力的比较 嗜盐四联球菌是可耐受较高渗透压的菌株,也常存在和应用于高渗透压培养体系。由于T. halophilus菌株具有耐高渗的特性,Justé尝试了用此特性并结合RAPD和糖类代谢结果分析将T. halophilus分为2个亚种:在糖浓度为69 °Bx (浓缩果汁)培养体系中可生长的Tetragenococcus halophilus subsp. flandriensis (分离自糖浆),和在低于65 °Bx糖浓度培养体系中生长的Tetragenococcus halophilus subsp. halophilus (分离自发酵鱼类和发酵豆制品)[29]。为了解酱醪来源的T.halophilus菌株的耐渗透压特性是否与其分类具有关联性,考察了酱醪来源的12株T.halophilus耐渗透压的能力。由表 4可知,酱醪来源的12株T.halophilus可在糖度为45 °Bx的培养体系中生长,其中C25耐高渗能力最强,可在糖度为50 °Bx的培养体系中生长;来源于不添加酵母酱油工艺酱醪的大多数菌株(C25、C33、C3)耐高渗能力(耐受48 °Bx糖度)强于来源于添加酵母酱油工艺酱醪的菌株(R23、R21、R44等)。虽然来源于酱油酱醪的T. halophilus不能在高于50 °Bx糖浓度(葡萄糖)培养体系中生长,与文献报道的存在差距,但这可能与培养体系中糖的种类有关。对酱醪来源的T.halophilus的MLST分类结果显示,菌株C3和C33较为相似,菌株R8和R55较为相似,而C25与其他菌株均存在一定的差异。对T.halophilus耐高渗能力的分析结果表明,菌株耐受高渗透压的能力与菌株来源的原生体系和菌株的分类存在一定的关联性。
表 4. 嗜盐四联球菌耐高渗透压能力比较 Table 4. Growth of T. halophilus under osmotic stress
| Brix | C25 | C33 | C3 | R55 | R18 | R21 | R8 | R23 | R44 | C1 | R13 | R9 |
| 45 °Bx | + | + | + | + | + | + | + | + | + | + | + | + |
| 48 °Bx | + | + | + | + | + | + | + | - | - | - | - | - |
| 50 °Bx | + | - | - | - | - | - | - | - | - | - | - | - |
| C25-R9: T. halophilus strains; +: growth; -: no growth. | ||||||||||||
表选项
2.4 嗜盐四联球菌产寡肽酶活性比较 氨基酸含量和种类是酱油的重要理化指标,对酱油的品质和风味有重要影响。T. halophilus菌株在基因组和糖类代谢上的差异说明它们合成蛋白酶、肽酶的能力也可能存在不同。了解T. halophilus合成蛋白酶的特性,可以为选择合适的菌株用于发酵提供参考依据。通过考察嗜盐四联球菌产寡肽酶的能力发现,菌株产三肽酶的能力高于产二肽酶的能力,前者是后者的2.1-8.7倍(图 4)。菌株C1合成三肽酶的水平最高,达到11.3 kU/g DCW。C25和R18合成三肽酶和二肽酶的能力相对较高。
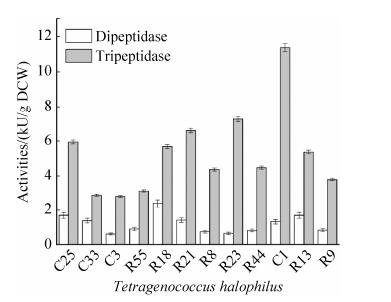 |
| 图 4 嗜盐四联球菌产寡肽酶能力分析 Figure 4 Production of oligopeptidase by T. halophilus strains. |
| 图选项 |
酱醪来源的T. halophilus合成三肽酶的能力接近乳杆菌的水平,但是它们合成二肽酶能力则普遍低于乳杆菌[22]。此外,前期研究表明,酱醪来源的T. halophilus合成氨肽酶的能力高于乳酸乳球菌和干酪乳杆菌[21]。以上结果说明,T. halophilus可以合成寡肽酶和氨肽酶等蛋白酶,这一特性对于提高酱油中氨基酸的含量和种类具有重要作用。
2.5 嗜盐四联球菌抗胁迫能力比较
2.5.1 菌株耐盐能力的比较: 嗜盐四联球菌是耐盐乳酸菌,可在含有100 g/L盐的培养体系中生长。高盐稀态酱油的酱醪盐度通常为150-200 g/L,T. halophilus对高盐的耐受性直接影响它们在酱油发酵过程中的作用与功能。通过考察T. halophilus对高盐的耐受性发现,培养体系中NaCl浓度高于和等于180 g/L时,菌浓低于对照培养条件下(100 g/L NaCl)的菌浓,当NaCl浓度为180 g/L和220 g/L时T.halophilus均可生长(OD600 > 1),NaCl浓度达到250 g/L时大多数T.halophilus生长被显著抑制。菌株R55的耐盐性最好,在250 g/L NaCl培养体系中仍可生长(OD600 > 1)(图 5)。
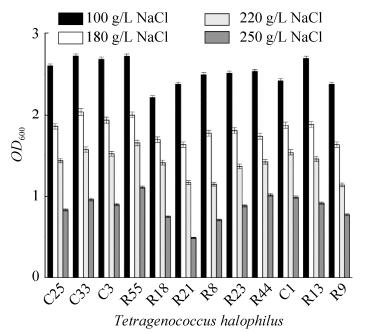 |
| 图 5 嗜盐四联球菌的耐盐能力比较 Figure 5 Tolerance of T. halophilus strains to salt stress. |
| 图选项 |
细菌通常可以通过两种途径来减少高盐、高糖等高渗透压对细胞的损害:一是调节胞内Na+和K+浓度,二是合成或吸收相容性溶质来平衡较高的渗透压,例如氨基酸、糖类及衍生物等物质[30-31]。研究证明嗜盐四联球菌可通过多种方法提高对高盐环境的耐受性:一是转运及合成甘氨酸甜菜碱作为调节渗透压的物质;二是合成热休克蛋白提高菌株适应性;三是提高编码转运蛋白基因的表达量,通过转运氨基酸、离子等物质调节渗透压[31-33]。酱醪来源的T. halophilus对渗透压的耐受能力存在差异:菌株C25、C33、C3、R55可以耐受高盐及高糖两种高渗透环境,R44、C1和R23可耐受高盐。
2.5.2 菌株耐酸性比较: 本研究初步考察嗜盐四联球菌在不同条件下的生长情况时发现,菌株最适生长环境偏中性,也不耐低温。因此,考察菌株耐受酸性和低温环境特性时,对这2个特性相近的菌株只选择代表菌株,最终选取C33、R23、C25、R21和R44这5株代表性菌株进行研究。
酱油发酵过程中,酱醪pH通常为4.7-5.5,对T.halophilus的生长不利。通过比较T.halophilus在酸性条件下的生长情况发现,酸性环境对T. halophilus的生长有较大影响。在5株考察的菌株中,R23、C25和R21的耐酸性较差,它们在pH 5.0下的相对生长率分别是对照的32%、27%和39%;C33和R44的耐酸性优于其他菌株,它们在pH 5.0下的相对生长率是对照的65% (图 6)。嗜盐四联球菌的耐酸机制目前还不十分清楚。但有研究表明,嗜盐四联球菌在酸胁迫条件下细胞膜中不饱和脂肪酸含量升高,其与氨基酸转化生成NH3和ATP相关途径的酶的表达量也有提高,因此可能存在与其他乳酸菌类似的抵御酸胁迫的相关机制[34-37]。
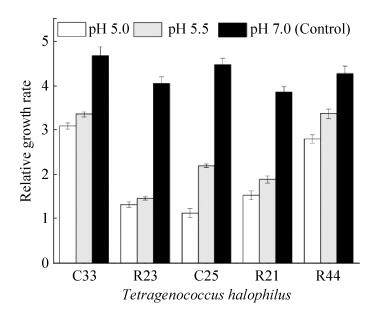 |
| 图 6 嗜盐四联球菌耐酸能力比较 Figure 6 Tolerance of T. halophilus strains to acidic stress. Relative growth rate, the ratio of viable cells numbers (after acid stress treatment vs before treatment). |
| 图选项 |
2.5.3 嗜盐四联球菌在较低温度下的生长情况: 强化鲁氏接合酵母的高盐稀态酱油生产工艺中,在发酵前期(0-20 d)需要将发酵温度控制在15 ℃,菌株对低温的耐受能力也是考察其应用价值的重要因素。对选取的5株T. halophius在低温条件下的生长情况进行比较。结果表明,低温条件对T. halophilus的生长有显著影响,温度降低以后所有菌株的生长都受到显著抑制(图 7)。其中菌株C25和R23在15 ℃培养条件下的相对生长率是对照的22%;菌株C33和R44在低温下的生长略优于其他3株T. halophilus,它们在15 ℃培养条件下的相对生长率分别是对照的34%和37%。研究表明,乳酸菌可通过提高细胞膜中不饱和脂肪酸含量、合成冷激蛋白(Cold-shock proteins,CSPs)等方式应对低温的影响[38]。
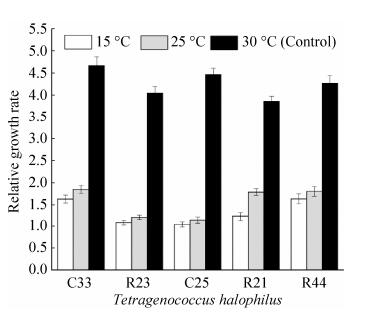 |
| 图 7 嗜盐四联球菌在低温条件下的生长情况 Figure 7 The growth of T. halophilus in low temperature environment. Relative growth rate, the ratio of viable cells numbers (after low temperature treatment vs before treatment). |
| 图选项 |
3 结论 本研究通过采用多位点序列分型(MLST)与糖类代谢相结合的方法,将来源于酱醪的12株嗜盐四联球菌分为2个类群。由于分类原理不同,依据糖类代谢特性对嗜盐四联球菌进行分类的结果与本研究采用的MLST分类结果存在一些差异,但总体上采用两种方法分析获得的结果是有一致性的。例如,两种分类结果均显示分类特征与菌株的原生环境(酱醪发酵工艺)存在一定的相关性,即相同工艺酱醪分离得到的菌株多数可被聚类为相近的类群。由于基因序列突变与基因演化的速度大于表型改变的速度,基因序列上的改变不一定导致表型发生变化。此外,MLST分类结果受选择基因及基因在基因组上分布的影响,不如糖代谢特性分类结果确定和稳定,但对于菌株的系统分类是有一定参考意义的,是用于菌株分类的重要辅助方法。如果需要对菌株进行精确分类和区分,可在比较基因组分析的基础上,验证菌株的相关生理和生化特性。酱醪来源的嗜盐四联球菌在生理特性上存在差异,但分离自不添加酵母工艺条件酱醪的一类菌株大多具有更强的高渗透压耐受能力。例如,菌株C25、C33、C3、R55耐受高盐及高糖能力均较强,R44、C1和R23耐高盐能力较强。虽然嗜盐四联球菌普遍不耐酸且在低温下生长被抑制,但是分离自酱醪的嗜盐四联球菌C33和R44在酸性培养体系(pH 5.0)中仍能正常生长,在较低温度下(15 ℃和25 ℃)的生长情况也略优于其他菌株。通过系统分类和耐受环境胁迫能力分析,较为系统地阐述了嗜盐四联球菌与酱油发酵相关的生物学特性及应用特性,研究结果可以为嗜盐四联球菌在发酵食品中应用提供理论依据和参考。
References
| [1] | Ogasawara M, Yamada Y, Egi M. Taste enhancer from the long-term ripening of miso (soybean paste). Food Chemistry, 2006, 99(4): 736-741. DOI:10.1016/j.foodchem.2005.08.051 |
| [2] | Yongsawatdigul J, Rodtong S, Raksakulthai N. Acceleration of Thai fish sauce fermentation using proteinases and bacterial starter cultures. Journal of Food Science, 2007, 72(9): M382-M390. DOI:10.1111/jfds.2007.72.issue-9 |
| [3] | Udomsil N, Rodtong S, Tanasupawat S, Yongsawatdigul J. Proteinase-producing halophilic lactic acid bacteria isolated from fish sauce fermentation and their ability to produce volatile compounds. International Journal of Food Microbiology, 2010, 141(3): 186-194. DOI:10.1016/j.ijfoodmicro.2010.05.016 |
| [4] | Wu CD, Liu CL, He GQ, Huang J, Zhou RQ. Characterization of a multiple-stress tolerance Tetragenococcus halophilus and application as starter culture in Chinese horsebean-chili-paste manufacture for quality improvement. Food Science and Technology Research, 2013, 19(5): 855-864. DOI:10.3136/fstr.19.855 |
| [5] | Harada R, Yuzuki M, Ito K, Shiga K, Bamba T, Fukusaki E. Influence of yeast and lactic acid bacterium on the constituent profile of soy sauce during fermentation. Journal of Bioscience and Bioengineering, 2016, 123: 203-208. |
| [6] | Nosaka S, Miyazawa M. Characterization of volatile components and odor-active compounds in the oil of edible mushroom Boletopsis leucomelas. Journal of Oleo Science, 2014, 63(6): 577-583. DOI:10.5650/jos.ess13215 |
| [7] | Cui RY, Zheng J, Wu CD, Zhou RQ. Effect of different halophilic microbial fermentation patterns on the volatile compound profiles and sensory properties of soy sauce moromi. European Food Research and Technology, 2014, 239(2): 321-331. DOI:10.1007/s00217-014-2225-9 |
| [8] | He GQ, Huang J, Liang R, Wu CD, Zhou RQ. Comparing the differences of characteristic flavour between natural maturation and starter culture for Mucor-type Douchi. International Journal of Food Science and Technology, 2016, 51(5): 1252-1259. DOI:10.1111/ijfs.13077 |
| [9] | Liao DY, Fang F, Chen J, Du GC, Liu L. Isolation and arginine metabolism detection of a Tetragenococcus halophilus strain. Journal of Food Science and Biotechnology, 2016, 35(1): 48-53. (in Chinese) 廖淡宜, 方芳, 陈坚, 堵国成, 刘龙. 嗜盐四联球菌的分离及其精氨酸代谢. 食品与生物技术学报, 2016, 35(1): 48-53. DOI:10.3969/j.issn.1673-1689.2016.01.007 |
| [10] | Udomsil N, Rodtong S, Choi YJ, Hua YL, Yongsawatdigul J. Use of Tetragenococcus halophilus as a starter culture for flavor improvement in fish sauce fermentation. Journal of Agricultural and Food Chemistry, 2011, 59(15): 8401-8408. DOI:10.1021/jf201953v |
| [11] | Kuda T, Izawa Y, Ishii S, Takahashi H, Torido Y, Kimura B. Suppressive effect of Tetragenococcus halophilus, isolated from fish-nukazuke, on histamine accumulation in salted and fermented fish. Food Chemistry, 2012, 130(3): 569-574. DOI:10.1016/j.foodchem.2011.07.074 |
| [12] | Delgado S, Mayo B. Phenotypic and genetic diversity of Lactococcus lactis and Enterococcus spp. strains isolated from Northern Spain starter-free farmhouse cheeses. International Journal of Food Microbiology, 2004, 90(3): 309-319. DOI:10.1016/S0168-1605(03)00323-4 |
| [13] | Pál K, Szén O, Kiss A, Naár Z. Comparison and evaluation of molecular methods used for identification and discrimination of lactic acid bacteria. Journal of the Science of Food and Agriculture, 2012, 92(9): 1931-1936. DOI:10.1002/jsfa.5564 |
| [14] | Fusco V, Quero GM, Chieffi D, Franz CMAP. Identification of Lactobacillus brevis using a species-specific AFLP-derived marker. International Journal of Food Microbiology, 2016, 232: 90-94. DOI:10.1016/j.ijfoodmicro.2016.06.002 |
| [15] | Wang ZQ, Qiu SF, Wang Y, Sun YS, Song HB. Progress in research on multilocus sequence typing technique. Bulletin of the Academy of Military Medical Sciences, 2010, 34(1): 76-79. (in Chinese) 王中强, 邱少富, 王勇, 孙岩松, 宋宏斌. 多位点序列分型技术及其研究进展. 军事医学科学院院刊, 2010, 34(1): 76-79. DOI:10.3969/j.issn.1674-9960.2010.01.021 |
| [16] | Bilhère E, Lucas PM, Claisse O, Lonvaud-Funel A. Multilocus sequence typing of Oenococcus oeni:detection of two subpopulations shaped by intergenic recombination. Applied and Environmental Microbiology, 2009, 75(5): 1291-1300. DOI:10.1128/AEM.02563-08 |
| [17] | Charteris WP, Kelly PM, Morelli L, Collins JK. Quality control Lactobacillus strains for use with the API 50CH and API ZYM systems at 37 degrees C. Journal of Basic Microbiology, 2001, 41(5): 241-251. DOI:10.1002/1521-4028(200110)41:5<241::AID-JOBM241>3.0.CO;2-2 |
| [18] | Gunde-Cimerman N, Oren A, Plemenita? A. Adaptation to Life at High Salt Concentrations in Archaea, Bacteria, and Eukarya. Netherlands: Springer, 2005. |
| [19] | Drosinos EH, Paramithiotis S, Kolovos G, Tsikouras I, Metaxopoulos I. Phenotypic and technological diversity of lactic acid bacteria and staphylococci isolated from traditionally fermented sausages in Southern Greece. Food Microbiology, 2007, 24(3): 260-270. DOI:10.1016/j.fm.2006.05.001 |
| [20] | Poga?i? T, Mancini A, Santarelli M, Bottari B, Lazzi C, Neviani E, Gatti M. Diversity and dynamic of lactic acid bacteria strains during aging of a long ripened hard cheese produced from raw milk and undefined natural starter. Food Microbiology, 2013, 36(2): 207-215. DOI:10.1016/j.fm.2013.05.009 |
| [21] | Liao DY. Isolation and characterization of Tetragenococcus halophilus strains from moromi. Master Dissertation of Jiangnan University, 2015. (in Chinese) 廖淡宜. 酱醪中嗜盐四联球菌的分离及特性研究. 江南大学硕士学位论文, 2015. http://journals.im.ac.cn/html/actamicrocn/2018/10/%20//cdmd.%20cnki.%20com.%20cn/Article/CDMD-10295-1015430137.%20htm |
| [22] | Francavilla R, De Angelis M, Rizzello CG, Cavallo N, Dal Bello F, Gobbetti M. Selected probiotic Lactobacilli have the capacity to hydrolyze gluten peptides during simulated gastrointestinal digestion. Applied and Environmental Microbiology, 2017, 83(14): e00376-17. DOI:10.1128/AEM.00376-17 |
| [23] | Yu J. Multilocus sequence typing of Streptococcus thermophilus from traditional fermented dairy products in China, Russia and Mongolia. Doctoral Dissertation of Inner Mongolia Agricultural University, 2013. (in Chinese) 于洁. 中国、俄罗斯和蒙古国地区传统发酵乳制品中嗜热链球菌的多位点序列分型研究. 内蒙古农业大学博士学位论文, 2013. http://journals.im.ac.cn/html/actamicrocn/2018/10/%20//cdmd.%20cnki.%20com.%20cn/Article/CDMD-10129-1013249787.%20htm |
| [24] | Douillard FP, Ribbera A, Kant R, Pietil? TE, J?rvinen HM, Messing M, Randazzo CL, Paulin L, Laine P, Ritari J, Caggia C, L?hteinen T, Brouns SJJ, Satokari R, von Ossowski I, Reunanen J, Palva A, de Vos WM. Comparative genomic and functional analysis of 100Lactobacillus rhamnosus strains and their comparison with strain GG. PLoS Genetics, 2013, 9(8): e1003683. DOI:10.1371/journal.pgen.1003683 |
| [25] | Kankainen M, Paulin L, Tynkkynen S, von Ossowski I, Reunanen J, Partanen P, Satokari R, Vesterlund S, Hendrickx APA, Lebeer S, de Keersmaecker SCJ, Vanderleyden J, H?m?l?inen T, Laukkanen S, Salovuori N, Ritari J, Alatalo E, Korpela R, Mattila-Sandholm T, Lassig A, Hatakka K, Kinnunen KT, Karjalainen H, Saxelin M, Laakso K, Surakka A, Palva A, Salusj?rvi T, Auvinen P, de Vos WM. Comparative genomic analysis of Lactobacillus rhamnosus GG reveals pili containing a human-mucus binding protein. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(40): 17193-17198. DOI:10.1073/pnas.0908876106 |
| [26] | Roling W, van Verseveld HW. Characterization of Tetragenococcus halophila populations in Indonesian soy mash (Kecap) fermentation. Applied and Environmental Microbiology, 1996, 62(4): 1203-1207. |
| [27] | Li Y, Raftis E, Canchaya C, Fitzgerald GF, van Sinderen D, O'Toole PW. Polyphasic analysis indicates that Lactobacillus salivarius subsp. salivarius and Lactobacillus salivarius subsp. salicinius do not merit separate subspecies status. International Journal of Systematic and Evolutionary Microbiology, 2006, 56(10): 2397-2403. DOI:10.1099/ijs.0.64426-0 |
| [28] | Raftis EJ, Salvetti E, Torriani S, Felis GE, O'Toole PW. Genomic diversity of Lactobacillus salivarius. Applied and Environmental Microbiology, 2011, 77(3): 954-965. DOI:10.1128/AEM.01687-10 |
| [29] | Justé A, van Trappen S, Verreth C, Cleenwerck I, de Vos P, Lievens B, Willems KA. Characterization of Tetragenococcus strains from sugar thick juice reveals a novel species, Tetragenococcus osmophilus sp. nov., and divides Tetragenococcus halophilus into two subspecies, T. halophilus subsp. halophilus subsp. nov. and T. halophilus subsp. flandriensis subsp. nov. International Journal of Systematic and Evolutionary Microbiology, 2011, 62(1): 129-137. |
| [30] | Cánovas D, Vargas C, Calderón MI, Ventosa A, Nieto JJ. Characterization of the genes for the biosynthesis of the compatible solute ectoine in the moderately halophilic bacterium Halomonas elongata DSM 3043. Systematic and Applied Microbiology, 1998, 21(4): 487-497. DOI:10.1016/S0723-2020(98)80060-X |
| [31] | Liu LC, Si LF, Meng X, Luo L. Comparative transcriptomic analysis reveals novel genes and regulatory mechanisms of Tetragenococcus halophilus in response to salt stress. Journal of Industrial Microbiology & Biotechnology, 2015, 42(4): 601-616. |
| [32] | Robert H, Le Marrec C, Blanco C, Jebbar M. Glycine betaine, carnitine, and choline enhance salinity tolerance and prevent the accumulation of sodium to a level inhibiting growth of Tetragenococcus halophila. Applied and Environmental Microbiology, 2000, 66(2): 509-517. DOI:10.1128/AEM.66.2.509-517.2000 |
| [33] | Sugimoto S, Saruwatari K, Higashi C, Tsuruno K, Matsumoto S, Nakayama J, Sonomoto K. In vivo and in vitro complementation study comparing the function of DnaK chaperone systems from halophilic lactic acid bacterium Tetragenococcus halophilus and Escherichia coli. Bioscience, Biotechnology, and Biochemistry, 2008, 72(3): 811-822. DOI:10.1271/bbb.70691 |
| [34] | Denich TJ, Beaudette LA, Lee H, Trevors JT. Effect of selected environmental and physico-chemical factors on bacterial cytoplasmic membranes. Journal of Microbiological Methods, 2003, 52(2): 149-182. DOI:10.1016/S0167-7012(02)00155-0 |
| [35] | He GQ, Feng X, Wu CD, Huang J, Zhou RQ. Influence of multiple stresses on the survival and cellar constituent of Tetragenococcus halophilus CGMCC 3792. Science and Technology of Food Industry, 2016, 37(5): 182-186. (in Chinese) 何桂强, 冯笑, 吴重德, 黄均, 周荣清. 多重胁迫对嗜盐四联球菌CGMCC 3792存活率及细胞成分的影响. 食品工业科技, 2016, 37(5): 182-186. |
| [36] | Teixeira JS, Seeras A, Sanchez-Maldonado AF, Zhang CG, Su MS, G?nzle MG. Glutamine, glutamate, and arginine-based acid resistance in Lactobacillus reuteri. Food Microbiology, 2014, 42: 172-180. DOI:10.1016/j.fm.2014.03.015 |
| [37] | He GQ, Wu CD, Huang J, Zhou RQ. Acid tolerance response of Tetragenococcus halophilus:A combined physiological and proteomic analysis. Process Biochemistry, 2015, 51(2): 213-219. |
| [38] | Shao YY, Chen X, Yang M, Wu L, Zhang HP. Anti-freezing property and mechanism of lactic acid bacteria. Microbiology China, 2010, 37(2): 274-279. (in Chinese) 邵玉宇, 陈霞, 杨梅, 乌兰, 张和平. 乳酸菌的抗冷冻性及耐受机理. 微生物学通报, 2010, 37(2): 274-279. |
