李恒, 唐双焱


中国科学院微生物研究所, 中国科学院微生物生理与代谢工程重点实验室, 北京 100101
收稿日期:2017-03-24;修回日期:2017-04-22;网络出版日期:2017-04-26
基金项目:国家自然科学基金(21472234)
*通信作者:唐双焱, Tel:+86-10-64806140;E-mail:tangsy@im.ac.cn
摘要:糖类是自然界数量最多的一类有机化合物,生物对其降解利用是最重要的反应之一。碳水化合物酶是具有降解、修饰和生成糖苷键功能的一大类酶。由于高分子糖类可溶性低,其糖苷键难以触及,因此其被酶作用的效率相对较低。碳水化合物结合结构域能特异性结合多糖底物,对提升糖类底物的酶催化效率起着关键的作用。本文从碳水化合物结合结构域的家族类型、结构类型、结构与功能关系以及与催化结构域的关系几个方面进行了综述,对阐明碳水化合物结合结构域与碳水化合物识别机制,进而将其广泛应用于生物和医疗领域具有重要意义。
关键词: 碳水化合物结合结构域 碳水化合物酶 多糖 底物特异性
Carbohydrate-binding modules:assisted polysaccharide recognition
Heng Li, Shuang-Yan Tang


CAS Key Laboratory of Microbial Physiological and Metabolic Engineering, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China
Received 24 March 2017; Revised 22 April 2017; Published online 26 April 2017
*Corresponding author: Shuang-Yan Tang, Tel:+86-10-64806140;E-mail:tangsy@im.ac.cn
Supported by the National Natural Science Foundation of China (21472234)
Abstract: In nature, the biological degradation of carbohydrates is one of the most important reactions. Carbohydrate-active enzymes are enzymes involved in the synthesis, degradation and modification of carbohydrates. Due to the insolubility of some high-molecular weight polysaccharide substrates, the catalytic efficiency of some carbohydrate-active enzymes is low. Carbohydrate-binding modules (CBM) specifically bind carbohydrate substrates and improve the catalytic efficiency of carbohydrate-active enzymes by elevating the substrate concentrations around the catalytic modules. This review summarizes the knowledge of CBM, including their nomenclature and classification, the relationship between CBM structures and functions, and so on. Furthermore, some applications of CBM in biological and medical engineering are also presented.
Key words: carbohydrate binding module (CBM) carbohydrate-active enzyme polysaccharide substrate specificity
糖类是自然界中广泛分布的一类重要有机化合物,地球上生物量干重的50%以上是由葡萄糖的聚合物构成的[1]。糖类是光合作用的最终产物,是所有消费者的主要能量物质,因此,在自然界物质和能量的循环利用中起到举足轻重的作用。糖类主要以多糖的形式存在,包括纤维素、几丁质、淀粉、糖原类等物质。这些多糖由于分子量成千上万[2],属于不溶性多糖,难以被生物降解。纤维素结合结构域(Cellulose-binding domain,CBD)是最早发现的碳水化合物结合结构域,最初,纤维素结合结构域根据蛋白序列的相似性被划分为不同的类型,如Ⅰ型、Ⅱ型CBD[3-5]。随着对碳水化合物水解酶的深入研究,在半纤维素降解酶和其他水解酶中也陆续发现了类似结构,它们组成酶的一个独立结构域,没有催化活性,但能够促进催化活性结构域与碳水化合物的结合,被统称为碳水化合物结合结构域(Carbohydrate-bindingmodule,CBM)[6]。CBM根据蛋白序列的相似性被分为不同的家族,至2017年3月,共有81个确定的CBM家族(http://www.cazy.org/)[7]。根据其不同的底物结合特异性,不同的CBM能特异识别纤维素、几丁质、葡聚糖、木聚糖、菊粉、甘露聚糖、半乳聚糖、淀粉或糖原类物质,此外还有一些CBM能结合细胞表面的多糖。CBM的多样性使得其在促进酶与底物结合(尤其是不溶性底物)、底物特异识别、以及酶稳定性等方面都有重要作用。因此总结当前CBM结构与功能的相关知识,对促进碳水化合物结合结构域的深入研究,推动其广泛应用都有重要意义。
1 CBM命名和分类 CBM家族目前有81个,它们的命名依据是其所属家族和物种来源。直接以其所属家族命名是最简单的情况,例如CBM家族4来自于菌株Acholeplasma brassicae O502,可以称其为CBM4。为了阐述得更清楚,可以将其物种来源加入命名中,称为AbCBM4或者AbO502CBM4。有的水解酶中只有1个CBM,有的水解酶中可能有多个串联的CBM,一般从N-端开始命名,例如CBM4-1是第1个CBM,CBM4-2是第2个CBM,以此类推。在当前的81个CBM家族中共包含有95621种CBM,此外还有300余种未分类的CBM。近年来,许多CBM的晶体结构已被报道,使得我们能够深入理解其生物学功能。但是,部分CBM的研究尚不透彻,有的只是在序列水平,尚需进一步的功能验证。最大的5个CBM家族依次是家族50、48、5、13和32 (图 1)。CBM家族50是最大的CBM家族,共有37000多种CBM,主要来源于细菌和真菌的糖苷水解酶类。该家族的CBM序列长度一般约50个氨基酸左右,主要识别几丁质、肽聚糖等,也被称作LysM结构域。例如,酵母Kluyveromyces lactis中有一种几丁质酶含有LysM结构域,通过识别酵母细胞壁上的几丁质并将其水解,可以抑制酵母细胞的生长[8]。又比如,在孢子萌发过程中,蜡样芽孢杆菌Bacilluscereus ATCC 10876中的肽聚糖溶解酶SleL,利用其LysM结构域,识别孢子外壁并进一步水解孢壁,起始其孢子萌发[9]。第二大的CBM家族48包含15000多种CBM,其序列长度一般约为100个氨基酸,主要来自糖苷水解酶,多为普鲁兰酶、异淀粉酶、分支酶等等[10, 11]。家族13的CBM的序列长度一般为150个氨基酸,它的主要特点是结构域能够形成三叶草结构。例如链霉菌Streptomyces lividans中的木聚糖酶XlnA的CBM的三叶草结构中有3个底物结合位点[12]。纤维素酶也是CBM的一个重要来源,CBM家族1、2、3、6、8、11、13、16、17、63和64中都有部分CBM被证实特异结合纤维素[13-17]。还有许多小的CBM家族,多数是来源于细菌中的酶类。
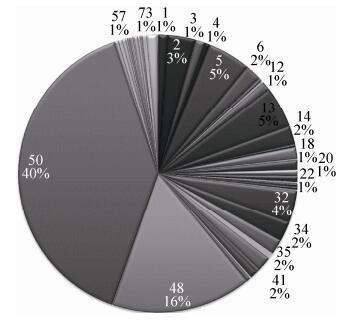 |
| 图 1 81个CBM家族的分布 Figure 1 Overview of 81 CBM family. |
| 图选项 |
2 CBM的结构 在当前的CBM家族中,部分家族种类稀少尚无深入的研究,图 2展示了已知的44个CBM家族的序列长度分布情况。其中80%的CBM长度在50-100个氨基酸之间。CBM家族13的序列虽然比较长,但其实其内部含有3段重复序列。由于大多数CBM并不太长,为了满足更强的生物功能,生物体会采取在一个酶中使用多个CBM的策略,例如,黄枝孢霉中的效应蛋白Ecp6含有3个CBM[18]。长度最短的CBM为40个氨基酸左右,包括CBM家族1和18,它们分别识别纤维素和几丁质。由于序列很短只能形成简单的结构,如CBM家族1中来源于里氏木霉Trichoderma reesei的内切葡聚糖酶的CBM只有38个氨基酸(PDB:4BMF),它能够通过二硫键形成一个简单的平面,并利用平面上方的多个芳香族氨基酸与纤维素相互作用[19]。CBM74是已知CBM家族中分子量最大的,该家族是2016年在革兰氏阳性菌Microbacterium aurum B8.A的α淀粉酶MaAmyA中发现的。CBM74不仅能够结合直链淀粉和支链淀粉,而且能够结合淀粉颗粒,促进α淀粉酶MaAmyA对淀粉颗粒的降解[20-22]。
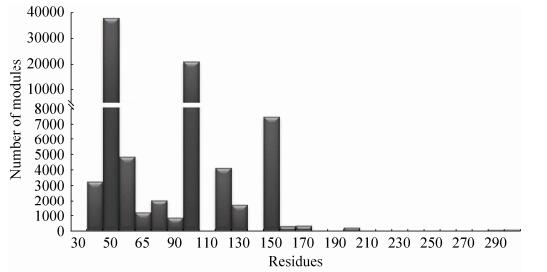 |
| 图 2 CBM序列长度的分布情况 Figure 2 Number of residues in different CBMs. |
| 图选项 |
长度比较短的CBM,其形成的蛋白结构也比较简单,多数都是β折叠,或者β折叠基础上形成的β-Sandwich折叠(CBM2,3,4,6,9,15,17,22,31-36,48,51等)、β-Trefoil折叠(CBM13,42) 和Hevein折叠(CBM18),此外,还有一些不规则的独特结构[23-25]。在自然界中CBM在酶的许多部位都出现过,无论是N-端、C-端还是中间部位,CBM都能够行使功能,有研究者移动CBM在水解酶中的相对位置,发现并不影响其与底物的结合,说明CBM是一个独立的结构域[26-28]。甚至,有时CBM能够独立作为一个蛋白质存在,不含有催化结构域。例如嗜热裂孢菌Thermobifida fusca中的蛋白E7和E8,E7是只包含有一个CBM的独立蛋白,而E8则含有多个CBM,它们都能结合几丁质但是都没有催化活性。但它们都能够提高几丁质酶的活性[29]。
3 CBM的结构与功能的关系 另一种依据结构和功能的分类方法将CBM分为3种类型:表面结合型CBM (A型)、链式结合型CBM (B型)和小分子糖结合型CBM (C型)。这种分类方法相对于CBM家族分类更加接近于CBM的实际功能(图 3)。A型包括CBM家族1、3、5、10等,能够结合不溶性、结晶的底物。一般情况下,当底物溶解分散在溶液中时,CBM可以全面接触到该化合物。而对于不溶性和结晶的底物,CBM只能接触到其表面,此时,底物对于CBM来说是一个巨大的平面,只有它的结合位点位于表面才能结合,这也是水解酶难以降解不溶性和结晶化合物的原因。A型CBM正是符合这种特点的结构域,这种CBM的结合位点往往分布在一个平面或者接近平面,这种平整的结构刚好可以与不溶性和结晶的底物互补[30],如图 3-A所示,A型CBM与下方的底物只有一个面的接触。鉴于其特异性,A型CBM能够结合不溶性或结晶的底物,对可溶性底物不亲和或者亲和力差。已经证明芳香族氨基酸残基对于A型CBM与底物的结合至关重要,例如,对于里氏木霉Trichoderma reesei的CBM1-Y5A,人们发现其芳香族氨基酸残基的数量以及它们在结合平面的空间排布对其结合能力都十分关键[31]。
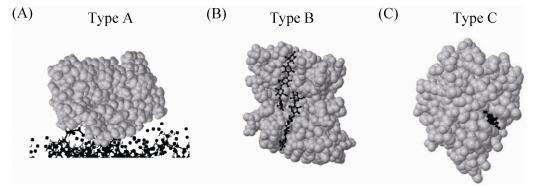 |
| 图 3 根据与底物的结合方式对CBM分类 Figure 3 Interactions of substrates and different types of CBMs. A: Schematic diagram of Type A CBM; B: TypeB CBM from CfCBM4-1[32]; C: Type C CBM from TmCBM[35]. |
| 图选项 |
与A型CBM不同,许多B型CBM的晶体结构显示,其蛋白往往含有一个凹槽或者裂缝,不同的CBM有不同程度深浅的凹槽[32-34]。例如图 3中的B型CBM (PBD:1GU3) 来源于粪碱纤维单胞菌Cellulomonas fimi Cel9B,属于CBM家族4,全称为CfCBM4-1,它采取β-jelly卷曲折叠的方式在蛋白中部形成一条凹槽,能很好地结合β-1, 4寡糖(黑色所示)[32]。B型CBM的结合效率被证明是与底物的聚合度紧密相关的,往往对长链的糖比短链或者单糖有更强的结合效率,因此被称为链式结合型CBM。
C型CBM优先结合一糖、二糖和三糖,因此被称作小分子糖结合型CBM。更准确地说,它不仅能结合小糖,而且能结合多糖的末端糖基[35]。图 3所示的C型CBM来源于海栖热袍菌Thermotogamaritima的木聚糖酶10A,其结合位点位于蛋白表面,大小和深度恰好适合容纳1个纤维二糖(黑色所示),通过2个色氨酸残基与纤维二糖紧密结合[35]。C型CBM最早因凝集素为人所知,凝集素广泛存在于动物、植物和微生物中,它们可以结合溶液中游离的糖,或者能识别糖蛋白和脂蛋白中,甚至是细胞膜上复杂的碳水化合物结构。一种凝集素含有多个CBM,具有专一结合某一种特异性糖基的能力[36-38]。C型CBM由于只结合单糖、寡糖或者多糖末端糖基,因此其与B型CBM相比并没有凹槽存在,其结合位点更加集中。CBM家族9、13、14、18等都属于C型CBM。
值得一提的是,不同类型的CBM可以同时存在于一个蛋白质中,例如,混合纤维弧菌Cellvibriomixtus Cel5A的CBM6同时包含独立的B型和C型结构域[39-40]。对CBM进行分类可以加深对其特征的认识,但当前的分类方式可能并不能囊括自然界中所有CBM的功能类型。
4 CBM功能与应用 CBM的主要功能就是提高酶的催化效率。一般其通过结合碳水化合物底物拉近酶与底物的距离,有效增加了酶附近的底物浓度。为了增强CBM的功能,多个相同或者不同的CBM可以同时存在于同一酶蛋白中。如上文所述有些CBM还能单独构成蛋白,它们的作用机制尚需进一步的研究。一般来说,不同的催化结构域具有不同的底物特异性[41];由于CBM结合底物具有特异性,同样的催化结构域如果与不同的CBM融合可能会降解不同类型的碳水化合物。另一方面,某些高温菌的CBM具有较高的稳定性,属于热稳定性结构域。有研究表明热稳定性结构域与不稳定的蛋白结构域融合可以提高后者的热稳定性[42-44]。因此在酶分子改造中,利用稳定的CBM可以提高单一催化结构域的稳定性和最适温度等特性[45]。但并不是所有的结构域都能相互融合而不影响其功能,而且将CBM与催化活性结构域融合时使用的连接肽也可能影响酶稳定性。
利用CBM结合底物的特异性,以及CBM的相对独立性和分子量小的特点,亦可开发CBM作为蛋白纯化标签。由于纤维素等多糖价格低廉,用其制作吸附柱具有巨大的价格优势。同样,利用CBM这些特点还可以将其用于固定化酶技术。在医学上,利用CBM特异性结合不溶性底物的特点,可以将其用于靶向治疗,设计新的识别位点,使CBM能够结合特异的细胞表面多糖,定向输送药物。
5 展望 由CBM的分类可知,其来源非常广泛,造成了CBM序列和结构上的多样性。使得其在促进酶与底物结合、底物特异识别等方面都有不同的作用。本质上,CBM的结构决定了其功能,不同的CBM中结合位点所在的位置有所不同,导致三种不同的CBM与底物的结合方式,进而发挥出不同功能和作用。此外,CBM本身的特性也会对整个蛋白产生相应影响,比如稳定性和耐热性。
随着各种碳水化合物酶的深入研究,新型的CBM还将不断被发现,更多的CBM结构与功能将逐步被阐明。CBM的功能具有巨大的应用前景。研究与改造CBM对于阐明蛋白质与碳水化合物识别机制,进而广泛将其应用于酶分子改造和药物开发都具有重要意义。CBM也在大量工业用酶包括纤维素酶、木聚糖酶及淀粉酶中起着关键作用。
References
| [1] | 王镜岩. 生物化学:上册. 北京: 高等教育出版社, 2002. |
| [2] | 南京大学化学系有机化学教研室. 有机化学:下册. 北京: 高等教育出版社, 1965. |
| [3] | Tomme P, Van Tilbeurgh H, Pettersson G, Van Damme J, Vandekerckhove J, Knowles J, Teeri T, Claeyssens M. Studies of the cellulolytic system of Trichoderma reesei QM 9414:analysis of domain function in two cellobiohydrolases by limited proteolysis. European Journal of Biochemistry, 1988, 170(3): 575-581. DOI:10.1111/ejb.1988.170.issue-3 |
| [4] | Gilkes NR, Warren RA, Miller Jr RC, Kilburn DG. Precise excision of the cellulose binding domains from two Cellulomonas fimi cellulases by a homologous protease and the effect on catalysis. The Journal of Biological Chemistry, 1988, 263(21): 10401-10407. |
| [5] | Tomme P, Warren RAJ, Miller Jr RC, Kilburn DG, Gilkes NR. Cellulose-binding domains:classification and properties//Saddler JN, Penner MH. Enzymatic Degradation of Insoluble Carbohydrates. ACS Symposium Series. Washington, DC:American Chemical Society, 1996:142-163. http://pubs.acs.org/doi/abs/10.1021/bk-1995-0618.ch010 |
| [6] | Rodríguez-Sanoja R, Oviedo N, Escalante L, Ruiz B, Sánchez S. A single residue mutation abolishes attachment of the CBM26 starch-binding domain from Lactobacillus amylovorus α-amylase. Journal of Industrial Microbiology & Biotechnology, 2009, 36(3): 341-346. |
| [7] | Cantarel BL, Coutinho PM, Rancurel C, Bernard T, Lombard V, Henrissat B. The carbohydrate-active enzymes database (CAZy):an expert resource for glycogenomics. Nucleic Acids Research, 2009, 37(S1): D233-D238. |
| [8] | Stark MJR, Boyd A. The killer toxin of Kluyveromyces lactis:characterization of the toxin subunits and identification of the genes which encode them. The EMBO Journal, 1986, 5(8): 1995-2002. |
| [9] | üstok FI, Chirgadze DY, Christie G. Structural and functional analysis of SleL, a peptidoglycan lysin involved in germination of Bacillus spores. Proteins Structure, Function, and Bioinformatics, 2015, 83(10): 1787-1799. DOI:10.1002/prot.24861 |
| [10] | Woo EJ, Lee S, Cha H, Park JT, Yoon SM, Song HN, Park KH. Structural insight into the bifunctional mechanism of the glycogen-debranching enzyme TreX from the archaeon Sulfolobus solfataricus. Journal of Biological Chemistry, 2008, 283(42): 28641-28648. DOI:10.1074/jbc.M802560200 |
| [11] | Sim L, Beeren SR, Findinier J, Dauvillée D, Ball SG, Henriksen A, Palcic MM. Crystal structure of the Chlamydomonas starch debranching enzyme isoamylase ISA1 reveals insights into the mechanism of branch trimming and complex assembly. Journal of Biological Chemistry, 2014, 289(33): 22991-23003. DOI:10.1074/jbc.M114.565044 |
| [12] | Notenboom V, Boraston AB, Williams SJ, Kilburn DG, Rose DR. High-resolution crystal structures of the lectin-like xylan binding domain from Streptomyces lividans xylanase 10A with bound substrates reveal a novel mode of xylan binding. Biochemistry, 2002, 41(13): 4246-4254. DOI:10.1021/bi015865j |
| [13] | Angelov A, Loderer C, Pompei S, Liebl W. Novel family of carbohydrate-binding modules revealed by the genome sequence of Spirochaeta thermophila DSM 6192. Applied and Environmental Microbiology, 2011, 77(15): 5483-5489. DOI:10.1128/AEM.00523-11 |
| [14] | Kerff F, Amoroso A, Herman R, Sauvage E, Petrella S, Filée P, Charlier P, Joris B, Tabuchi A, Nikolaidis N, Cosgrove DJ. Crystal structure and activity of Bacillus subtilis YoaJ (EXLX1), a bacterial expansin that promotes root colonization. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(44): 16876-16881. DOI:10.1073/pnas.0809382105 |
| [15] | Viegas A, Sardinha J, Freire F, Duarte DF, Carvalho AL, Fontes CMGA, Rom?o MJ, Macedo AL, Cabrita EJ. Solution structure, dynamics and binding studies of a family 11 carbohydrate-binding module from Clostridium thermocellum (CtCBM11). Biochemical Journal, 2013, 451(2): 289-300. DOI:10.1042/BJ20120627 |
| [16] | van Bueren AL, Morland C, Gilbert HJ, Boraston AB. Family 6 carbohydrate binding modules recognize the non-reducing end of β-1, 3-linked glucans by presenting a unique ligand binding surface. Journal of Biological Chemistry, 2005, 280(1): 530-537. DOI:10.1074/jbc.M410113200 |
| [17] | Mandelman D, Belaich A, Belaich JP, Aghajari N, Driguez H, Haser R. X-ray crystal structure of the multidomain endoglucanase Cel9G from Clostridium cellulolyticum complexed with natural and synthetic cello-oligosaccharides. Journal of Bacteriology, 2003, 185(14): 4127-4135. DOI:10.1128/JB.185.14.4127-4135.2003 |
| [18] | Sánchez-Vallet A, Saleem-Batcha R, Kombrink A, Hansen G, Valkenburg DJ, Thomma BP, Mesters JR. Fungal effector Ecp6 outcompetes host immune receptor for chitin binding through intrachain LysM dimerization. eLife, 2013, 2: e00790. |
| [19] | Mattinen ML, Linder M, Drakenberg T, Annila A. Solution structure of the cellulose-binding domain of endoglucanase I from Trichoderma reesei and its interaction with cello-oligosaccharides. European Journal of Biochemistry, 1998, 256(2): 279-286. DOI:10.1046/j.1432-1327.1998.2560279.x |
| [20] | Valk V, van der Kaaij RM, Dijkhuizen L. Characterization of the starch-acting MaAmyB enzyme from Microbacterium aurum B8.A representing the novel subfamily GH13_42 with an unusual, multi-domain organization. Scientific Reports, 2016, 6: 36100. DOI:10.1038/srep36100 |
| [21] | Valk V, Eeuwema W, Sarian FD, van der Kaaij RM, Dijkhuizen L. Degradation of granular starch by the bacterium Microbacterium aurum strain B8.A involves a modular α-amylase enzyme system with FNIII and CBM25 domains. Applied and Environmental Microbiology, 2015, 81(19): 6610-6620. DOI:10.1128/AEM.01029-15 |
| [22] | Valk V, van Bueren AL, van der Kaaij RM, Dijkhuizen L. Carbohydrate-binding module 74 is a novel starch-binding domain associated with large and multidomain α-amylase enzymes. The FEBS Journal, 2016, 283(12): 2354-2368. DOI:10.1111/febs.13745 |
| [23] | Andersen NH, Cao BL, Rodriguez-Romero A, Arreguin B. Hevein:NMR assignment and assessment of solution-state folding for the agglutinin-toxin motif. Biochemistry, 1993, 32(6): 1407-1422. DOI:10.1021/bi00057a004 |
| [24] | Nakamura T, Mine S, Hagihara Y, Ishikawa K, Ikegami T, Uegaki K. Tertiary structure and carbohydrate recognition by the chitin-binding domain of a hyperthermophilic chitinase from Pyrococcus furiosus. Journal of Molecular Biology, 2008, 381(3): 670-680. DOI:10.1016/j.jmb.2008.06.006 |
| [25] | Kaus K, Lary JW, Cole JL, Olson R. Glycan specificity of the Vibrio vulnificus hemolysin lectin outlines evolutionary history of membrane targeting by a toxin family. Journal of Molecular Biology, 2014, 426(15): 2800-2812. DOI:10.1016/j.jmb.2014.05.021 |
| [26] | Mamo G, Hatti-Kaul R, Mattiasson B. Fusion of carbohydrate binding modules from Thermotoga neapolitana with a family 10 xylanase from Bacillus halodurans S7. Extremophiles, 2007, 11(1): 169-177. DOI:10.1007/s00792-006-0023-4 |
| [27] | Shin ES, Yang MJ, Jung KH, Kwon EJ, Jung JS, Park SK, Kim J, Yun HD, Kim H. Influence of the transposition of the thermostabilizing domain of Clostridium thermocellum xylanase (XynX) on xylan binding and thermostabilization. Applied and Environmental Microbiology, 2002, 68(7): 3496-3501. DOI:10.1128/AEM.68.7.3496-3501.2002 |
| [28] | Ali E, Zhao GS, Sakka M, Kimura T, Ohmiya K, Sakka K. Functions of family-22 carbohydrate-binding module in Clostridium thermocellum Xyn10C. Bioscience, Biotechnology, and Biochemistry, 2005, 69(1): 160-165. DOI:10.1271/bbb.69.160 |
| [29] | Moser F, Irwin D, Chen SL, Wilson DB. Regulation and characterization of Thermobifida fusca carbohydrate-binding module proteins E7 and E8. Biotechnology and Bioengineering, 2008, 100(6): 1066-1077. DOI:10.1002/(ISSN)1097-0290 |
| [30] | Santos CR, Paiva JH, Sfor?a ML, Neves JL, Navarro RZ, Cota J, Akao PK, Hoffmam ZB, Meza AN, Smetana JH, Nogueira ML, Polikarpov I, Xavier-Neto J, Squina FM, Ward RJ, Ruller R, Zeri AC, Murakami MT. Dissecting structure-function-stability relationships of a thermostable GH5-CBM3 cellulase from Bacillus subtilis 168. Biochemical Journal, 2012, 441(1): 95-104. DOI:10.1042/BJ20110869 |
| [31] | Mattinen ML, Kontteli M, Kerovuo J, Drakenberg T, Annila A, Linder M, Reinikainen T, Lindeberg G. Three-dimensional structures of three engineered cellulose-binding domains of cellobiohydrolase I from Trichoderma reesei. Protein Science, 1997, 6(2): 294-303. |
| [32] | Boraston AB, Nurizzo D, Notenboom V, Ducros V, Rose DR, Kilburn DG, Davies GJ. Differential oligosaccharide recognition by evolutionarily-related β-1, 4 and β-1, 3 glucan-binding modules. Journal of Molecular Biology, 2002, 319(5): 1143-1156. DOI:10.1016/S0022-2836(02)00374-1 |
| [33] | von Schantz L, H?kansson M, Logan DT, Walse B, ?sterlin J, Nordberg-Karlsson E, Ohlin M. Structural basis for carbohydrate-binding specificity-a comparative assessment of two engineered carbohydrate-binding modules. Glycobiology, 2012, 22(7): 948-961. DOI:10.1093/glycob/cws063 |
| [34] | Alahuhta M, Xu Q, Bomble YJ, Brunecky R, Adney WS, Ding SY, Himmel ME, Lunin VV. The unique binding mode of cellulosomal CBM4 from Clostridium thermocellum cellobiohydrolase A. Journal of Molecular Biology, 2010, 402(2): 374-387. DOI:10.1016/j.jmb.2010.07.028 |
| [35] | Notenboom V, Boraston AB, Kilburn DG, Rose DR. Crystal structures of the family 9 carbohydrate-binding module from Thermotoga maritima xylanase 10A in native and ligand-bound forms. Biochemistry, 2001, 40(21): 6248-6256. DOI:10.1021/bi0101704 |
| [36] | Goldstein IJ, Hughes RC, Monsigny M, Osawa T, Sharon N. What should be called a lectin?. Nature, 1980, 285(5760): 66. |
| [37] | Sharon N, Lis H. Lectins as cell recognition molecules. Science, 1989, 246(4927): 227-234. DOI:10.1126/science.2552581 |
| [38] | Van Damme EJM, Peumans WJ, Barre A, Rougé P. Plant lectins:a composite of several distinct families of structurally and evolutionary related proteins with diverse biological roles. Critical Reviews in Plant Sciences, 1998, 17(6): 575-692. DOI:10.1080/07352689891304276 |
| [39] | Pires VMR, Henshaw JL, Prates JAM, Bolam DN, Ferreira LMA, Fontes CMGA, Henrissat B, Planas A, Gilbert HJ, Czjzek M. The crystal structure of the family 6 carbohydrate binding module from Cellvibrio mixtus endoglucanase 5A in complex with oligosaccharides reveals two distinct binding sites with different ligand Specificities. Journal of Biological Chemistry, 2004, 279(20): 21560-21568. DOI:10.1074/jbc.M401599200 |
| [40] | Henshaw JL, Bolam DN, Pires VMR, Czjzek M, Henrissat B, Ferreira LMA, Fontes CMGA, Gilbert HJ. The family 6 carbohydrate binding module CmCBM6-2 contains two ligand-binding sites with distinct specificities. Journal of Biological Chemistry, 2004, 279(20): 21552-21559. DOI:10.1074/jbc.M401620200 |
| [41] | Liang CN, Zhang Y, Jia Y, Wang WZ, Li YH, Lu SK, Jin JM, Tang SY. Engineering a carbohydrate-processing transglycosidase into glycosyltransferase for natural product glycodiversification. Scientific Reports, 2016, 6: 21051. DOI:10.1038/srep21051 |
| [42] | Ribeiro LF, Furtado GP, Lourenzoni MR, Costa-Filho AJ, Santos CR, Nogueira SCP, Betini JA, Polizeli MDLTM, Murakami MT, Ward RJ. Engineering bifunctional laccase-xylanase chimeras for improved catalytic performance. Journal of Biological Chemistry, 2011, 286(50): 43026-43038. DOI:10.1074/jbc.M111.253419 |
| [43] | Furtado GP, Ribeiro LF, Lourenzoni MR, Ward RJ. A designed bifunctional laccase/β-1, 3-1, 4-glucanase enzyme shows synergistic sugar release from milled sugarcane bagasse. Protein Engineering Design & Selection, 2013, 26(1): 15-23. |
| [44] | Pierre B, Labonte JW, Xiong TN, Aoraha E, Williams A, Shah V, Chau E, Helal KY, Gray JJ, Kim JR. Molecular determinants for protein stabilization by insertional fusion to a thermophilic host protein. Chembiochem, 2015, 16(16): 2392-2402. DOI:10.1002/cbic.v16.16 |
| [45] | Yeom SJ, Han GH, Kim M, Kwon KK, Fu YY, Kim H, Lee H, Lee DH, Jung H, Lee SG. Controlled aggregation and increased stability of β-glucuronidase by cellulose binding domain fusion. PLoS ONE, 2017, 12(1): e0170398. DOI:10.1371/journal.pone.0170398 |
