谭超1,2, 谭华荣1, 张集慧1


1.中国科学院微生物研究所, 微生物资源前期开发国家重点实验室, 北京 100101;
2.三峡大学第一临床医学院, 湖北 宜昌 443002
收稿日期:2016-05-04;修回日期:2016-05-25;网络出版日期:2016-06-13
基金项目:国家“973计划”(2015CB150600);国家自然科学基金(31571281)
*通信作者:张集慧, Tel:+86-10-64807467;E-mail:zhang.jihui@im.ac.cn
摘要: 盐霉素(salinomycin)是由白色链霉菌(Streptomyces albus)产生的一元羧酸聚醚类抗生素,具有较强的抗革兰氏阳性菌和杀灭球虫的作用,而且对环境污染也较低;此外,还能特异性地抑制多种肿瘤细胞及肿瘤干细胞的生长,具有多重作用靶点,有望成为抗肿瘤的特效药。为提高盐霉素的产量,人们采用传统诱变技术和现代分子遗传学手段,对盐霉素产生菌进行了改造,获得了高产菌株;同时,通过对盐霉素化学结构进行修饰或者通过药物载体和联合用药等,增强了其活性和靶向性、减少了毒副作用。本文对盐霉素产生菌的改造策略、药物靶向性提高和活性优化等研究进展进行综述,并对今后的研究热点进行展望。
关键词: 盐霉素 菌株改造 生物活性 抗肿瘤 结构修饰 药物靶向性
Enhancement of salinomycin production and its activity optimization-A review
Chao Tan1,2, Huarong Tan1, Jihui Zhang1


1.State Key Laboratory of Microbial Resources, Institute of Microbiology, Chinese Academy of Sciences, Beijing 100101, China;
2.The First Clinical Medical College, Three Gorges University, Yichang 443002, Hubei Province, China
Received 04 May 2016; Revised 25 May 2016; Published online 13 June 2016
*Corresponding author: Jihui Zhang, Tel:+86-10-64807467;E-mail:zhang.jihui@im.ac.cn
Supported by the Grants from the Ministry of Science and Technology of China (2015CB150600) and by the National Natural Science Foundation of China (31571281)
Abstract: Salinomycin is a monocarboxylic acid polyether antibiotics produced by Streptomyces albus. It has strong inhibiting and killing activity against most gram-positive bacteria and various coccidiums with low adverse impact on environment. In addition, salinomycin can specifically inhibit the growth of a variety of cancer cells and cancer stem cells via targeting to multiple sites, and is a promising anti-tumor drug candidate. To obtain high yield salinomycinproducing strain, conventional mutation techniques and modern molecular genetic methods have been used. Meanwhile, bioactivity and selectivity of salinomycin could be improved by modifying the chemical structure and changing drug delivery methods. Here, we summarize the key strategies for enhancing salinomycin production and review the progresses in optimizing its drug activity and targeting properties. The future research focus is also addressed.
Key words: salinomycin strain improvement bioactivity antitumor structure modification drug targeting property
盐霉素(salinomycin)是由白色链霉菌(Streptomyces albus)产生的一种聚醚类离子载体抗生素(图 1-A),由乙酸、丙酸和丁酸分别提供的酰基缩合而成的三元螺环结构,其醚氧原子位于四氢吡喃或者四氢呋喃环中,可以螯合Na+、K+等单价金属离子,并携带这些离子进入细胞,打破膜两侧离子电势平衡,促使生物膜破裂,最终导致细胞凋亡[1]。其生物合成基因簇(约106 kb)已被克隆,其中有21个基因与盐霉素的生物合成密切相关,还有上游4个(sal N,O,P,Q)和下游5个(orf 1-5)基因可能与调控和底物供给相关[2-3](图 1-B)。盐霉素对革兰氏阳性菌、真菌、病毒及疟原虫都有很好的杀灭作用,能有效地预防及控制禽畜肠道的球虫生长,并有催肥增重的效果,因此被广泛应用于养殖业[4],近期的研究揭示它在抗肿瘤方面也具有特殊的疗效。尽管白色链霉菌产生的盐霉素已被大规模工业化生产,但为了降低成本,对盐霉素产生菌进行合理改造、进一步提高产量、并对其化学结构和生物活性进行优化、增强药物靶向性、减少毒副作用和降低耐药性等,这些依然是企业界非常感兴趣的问题,这些问题的解决对推动盐霉素更加广泛的应用和生产具有重要的意义。
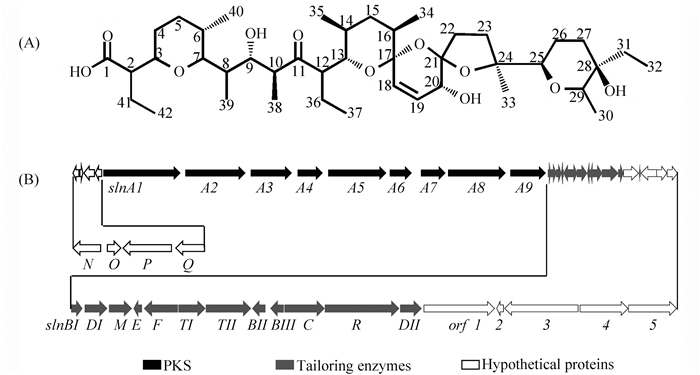 |
| 图 1. 盐霉素的化学结构(A)及其生物合成基因簇的排布(B) Figure 1. Chemical structure (A) and biosynthetic gene cluster (B) of salinomycin (Adapted from [2] and [3]). |
| 图选项 |
1 盐霉素产生菌的改良策略 盐霉素具有独特的生物活性和应用前景,获得盐霉素高产菌株,提高发酵水平,对盐霉素的开发和应用具有重要意义。早期主要采取物理或者化学诱变等随机突变的方式获得高产菌株,但现代基因工程、代谢工程和合成生物学等手段已成为菌株定向改良的主要发展方向(图 2)。
 |
| 图 2. 抗生素产生菌的改造策略 Figure 2. Strategies of strain improvement for enhancing antibiotics production. Nc-RNA:non-coding RNA; random mutagenesis and rational engineering are shaded in light blue and light brown, respectively. |
| 图选项 |
1.1 随机突变 基因定点突变是带有目的性的,如可以把某个碱基或某段DNA序列进行改变或删除;而随机突变是随机的,也就是说什么碱基或DNA序列发生了改变是不能预测的,只能通过大量筛选,得到目的菌株后进行基因组的DNA序列分析才能确定。
1.1.1 物理和化学诱变 传统的菌株改良方式主要是用物理诱变(紫外线、激光、X射线和γ射线等)和化学诱变方式(烷化剂、核酸碱基类似物及亚硝基胍和叠氮化钠等诱变剂)对链霉菌孢子或者原生质体进行交替处理,从而获得性能优良的菌株,由于其简便易行,已经被普遍接受和采纳。如贾永峰等[5]对S. albus的原生质体进行紫外诱变,并通过高通量方法来筛选突变株,使盐霉素的产量提高了69.7%;刘朋等[6]用常规化学试剂甲基磺酸乙酯诱变处理S. albus,使盐霉素的产量提高了38%。低能氮离子注入是一种新兴的多效应诱变技术,兼有物理和化学诱变的特性以及分子蚀刻作用等,使突变率更高、突变谱更广。武利勤等[7]用该技术对S. albus进行诱变,使盐霉素产量提高了41%。尽管物理和化学诱变有好的效果,但由于抗生素的生物合成是一个很复杂的过程,涉及到基因簇中多个结构基因和调控基因编码的酶和调控蛋白以及一些相关辅因子的参与,因此采用更多的方法和策略进行诱变将有望达到预期的效果。
1.1.2 核糖体工程 核糖体工程的概念由Ochi等提出,是指以核糖体的各组成部分为作用靶点(大、小S亚基和核糖体RNA),单独或者组合使用抗生素对菌株进行亚致死量的抗性筛选,致使核糖体蛋白编码基因发生突变,从而改变核糖体对蛋白质的合成能力,获得抗生素高产菌株[8];此外,核糖体功能的变化会诱导全局性调控因子ppGpp的合成,后者可作用于RNA聚合酶,使其对靶基因的选择性发生改变,从而启动链霉菌孢子分化和次级代谢的产生,而抗生素可能通过诱导该酶的突变,使菌株产生类似于ppGpp介导的应答反应,调节次级代谢产物的合成,所以核糖体工程是针对翻译元件和转录元件的调控方式[9]。常用的抗生素主要有链霉素、庆大霉素、氯霉素、霸龙霉素、新霉素、林可霉素和利福平等。如Tamehiro等[10]对S. albus进行链霉素、庆大霉素和利福平单独诱变时,其盐霉素的产量增产不明显,而通过这3种抗生素组合诱变选育,可使产量提高到2.3倍;吴健等[11]首先对S. albus进行紫外线诱变和紫外线复合氯化锂诱变、然后以棕榈油完全替代豆油改善发酵条件、再用链霉素和利福平等抗生素对其核糖体进行突变,使突变株产生的盐霉素产量提高了3倍。因此,核糖体工程与其他突变方式结合,更有效地提高了抗生素的生物合成能力,也是菌株改造的重要策略之一。
1.1.3 基因组重排 基因组重排(Genome shuffling)是对整个微生物的基因组重新进行组合排列、无需基因组和代谢途径信息的技术。其主要过程为:通过随机诱变产生突变库,并在设定的筛选条件下去获得所期望的正向突变菌株;再将不同正突变株的原生质体进行迭代式重组和融合,实现全基因组水平的随机组合,使优良性状快速富集,从而获得产量较高的菌株。同时该技术也可提高子代菌株的遗传多样性[12]。如Wang[13]和Du[14]等分别对活跃链霉菌(Streptomyces actuosus)及筑波链霉菌(Streptomyces tsukubaensis)进行基因组重排后,其产物诺西肽(Nosiheptide)及普乐可复(FK506)分别可达9倍和11倍以上;而Jin等[15]对刺糖多孢菌(Saccharopolyspora spinosa)进行紫外和亚硝基胍随机突变后,再对高产菌株进行4轮基因组重排,发现其代谢产物多杀菌素(Spinosad)的产量可提高到2倍,而同时进行的4轮常规筛选后,产量仅提高了42%。此外,Zhang等[12]也认为,2轮的基因组重排可以达到传统随机突变20轮才能实现的效果,且后者需要做的实验数量为前者的40倍。可见,基因组重排可对不同随机突变产生的链霉菌菌株进行全基因组重排,将优良遗传信息快速富集,大大缩短筛选轮次和周期,可望在盐霉素产生菌的改造中发挥重要作用。 1.2 基因的遗传操作在菌株改造中的应用 随着越来越多链霉菌基因组信息的发布、抗生素生物合成途径和调控机制的解析,可以通过基因的遗传操作(代谢工程和合成生物学等),更有效地理性改造菌株(图 2)。
1.2.1 关键结构基因及基因簇的高表达 在抗生素的生物合成过程中,一些酶促催化反应构成了合成的限速步骤,通过增加这些酶编码基因的拷贝数或者进行强启动子置换,使关键结构基因高表达或者对关键酶进行定点突变、优化酶活性,从而提高产物合成效率。此外,将整个基因簇装载在多拷贝质粒上或者整合到染色体中,以此来增加拷贝数,也是一条有效的途径。例如Murrell等[16]将烯二炔类抗肿瘤抗生素C-1027生物合成基因簇中的关键基因sgcA1在球孢链霉菌(Streptomyces globisporus)中倍增,使C-1027的产量明显提高;Du等[17]采用ΦBT1整合酶介导的克隆策略,将达托霉素和放线紫红素生物合成基因簇克隆到多拷贝质粒pKC1139上,增加了这些抗生素的产量;Liao等[18]将尼可霉素生物合成基因簇整合到圈卷产色链霉菌(Streptomyces ansochromogenes)中使其拷贝数增加一倍,使尼可霉素的产量提高4倍。在盐霉素生物合成基因簇中,SlnA1-A9是典型的I型聚酮合酶,其余12个基因编码的蛋白与聚酮链的环化、修饰以及产物外排等有关:如SlnC是环氧化酶,与环氧化水解酶SlnBII和SlnBIII共同负责聚酮链的环氧化和醚键形成等[2],是盐霉素合成的关键步骤,将这些酶的编码基因整合到染色体上(或在游离质粒上独立高表达),有助于提高盐霉素的产量[2]。而Yin等[19]采用了同源重组技术,通过3轮直接PCR和拼接,获得106 kb的盐霉素生物合成基因簇,这为盐霉素生物合成基因簇的表达和倍增奠定了基础。
1.2.2 调控基因的有效控制 抗生素生物合成基因簇的表达通常受到簇内调控基因编码的调控子(Cluster situated regulator, CSR)和(或者)全局性调控子(Global regulator, GR)的有序控制(激活或阻遏),在阐明它们对抗生素合成调控机制的基础上,对这些基因进行高表达或者敲除,从而可精细地调节抗生素的生物合成。例如Li等[20]发现多氧霉素生物合成基因簇中polR是正调控基因,在可可链霉菌(Streptomyces cacaoi)中增加一个拷贝可使其产量提高3倍;Smanski等[21]阻断普特拉链霉菌(Streptomyces platensis)平板霉素生物合成负调控基因ptmR1后,其平板霉素(platensimycin)和平板素(platencin)的产量提高约100倍;Li等[22]对始旋链霉菌中普那霉素II的生物合成基因簇进行改造,敲除负调控基因papR5同时高表达正调控基因papR4和papR6,而构建了组合突变菌株,然后再将体外重构的基因簇导入该突变株中,使普那霉素II的产量提高5倍。可见调控基因的改造对提高抗生素产量具有事半功倍的效果。盐霉素产生菌中,slnR是盐霉素合成的正调控基因[2],本实验室将slnR基因整合到野生型菌株的染色体上,使拷贝数增加一倍,发现盐霉素产量有所增加(未发表),而用组成型启动子置换原有启动子,可望使盐霉素产量提高更多。 全局性调控基因在许多链霉菌的生长发育和次级代谢产物合成中起重要的作用。例如,adpA是链霉菌的一种多效调控基因,同时它受到γ-丁酸内酯类信号分子受体ArpA及其配体组成的信号系统的调节,在抗生素合成中形成多效级联调控网络[23]。而其他全局性调控基因如四磷酸鸟苷(ppGpp)合成酶基因relA[24]、与发育分化中的孢子形成和抗生素的生物合成密切相关的wblA[25]、全局性链霉菌分化负调控基因nsdA、nsdB等[26]都高度保守的存在于多种链霉菌中,与许多抗生素的生物合成密切相关。如Higo等[23]在灰色链霉菌(Streptomyces griseus)中适量过表达AdpA,可使链霉素(Streptomycin)提前产生及产量提高;Wang等[27]阻断冰城链霉菌(Streptomyces bingchengensis)的nsdA基因,可使米尔贝霉素(Milbemycins)和南昌霉素(Nanchangmycin)产量分别提高到1.5和9.0倍;Woo等[28]阻断wblA的基因,可使委内瑞拉链霉菌(Streptomyces venezuelae)中的苦霉素(Picromycin)产量提高到3.5倍。虽然这些基因在盐霉素合成中的作用尚不明确,但是我们经同源性比对发现,产盐霉素的S. albus DSM41328中relA、nsdA以及wblA与天蓝色链霉菌中的同源基因一致性为83%-89%,极有可能是盐霉素菌株改造的新靶点。
1.2.3 抗生素外排蛋白的高表达 在链霉菌中,通常存在抗生素外排泵蛋白,负责将合成的产物及时输出细胞,避免对自身的毒性。因此提高外排蛋白的表达,及时降低胞内抗生素浓度,可有效减少反馈抑制,促使基因簇超量表达。例如Ostash等[29]阻断加纳链霉菌(Streptomyces ghanaensis)默诺霉素(moenomycin)生物合成基因簇中的外排基因moeP5moe X5时,其代谢产物默诺霉素明显降低;而Qiu等[30]高表达阿维链霉菌(Streptomyces avermitilis)阿维菌素(avermectin)生物合成基因簇中的外排基因avtAB,可使阿维菌素的产量提高1倍。刘静等[31]对S. albus的外排基因slnTI和slnTII一起阻断时,盐霉素的产量下降明显,而对二者串联超量表达时,盐霉素的产量可提高14.6%。因此,超量表达盐霉素产生菌的外排蛋白基因,对提高其产量可能也有一定的作用。
1.2.4 前体合成和代谢流的调节 链霉菌中次级代谢产物合成的前体大多与初级代谢密切相关,增加前体供应,提高前体的合成效率,可以有效地提高抗生素产量。而控制代谢流的走向,阻断非目标代谢产物的合成,也有助于目标抗生素的合成[32]。盐霉素是聚酮类化合物,它与其他聚酮类化合物的生物合成共享前体底物丙二酰辅酶A、甲基丙二酰辅酶A和乙基丙二酰辅酶A,因此阻断其他PKS模块,有利于前体物质进入盐霉素合成途径中[33]。如单独阻断盐霉素产生菌的基因组中其他7个PKS/PKS-NRPS模块,盐霉素产量都有显著提高;而在PKS-NRPS-2及PKS-6的双阻断突变株中,虽然盐霉素产量未进一步提高,乙基丙二酰辅酶A含量也较低,但是却积累了大量的丙二酰辅酶A和甲基丙二酰辅酶A。由于巴豆酰-CoA还原酶基因(ccr)是乙基丙二酰辅酶A主要合成酶的编码基因,为了提高乙基丙二酰辅酶A的合成效率,上海交通大学白林泉教授及其同事等在S. albus中,通过调节聚酮添加物的供给以及在PKS-NRPS-2及PKS-6双阻断突变株中过表达ccr,使盐霉素的产量提高了近10倍[33]。可见前体合成及代谢流的改变对盐霉素的生物合成有重要的影响。
1.2.5 基因簇异源表达 选择不同的异源宿主,将目的生物合成基因簇转化其中,进行异源表达不但可激活隐性基因簇,还可解除原有宿主菌中负调控因子或相关阻遏物(Repressor)的影响,同时在宿主菌中敲除了一些非必需次级代谢基因簇,减少了跟外源基因簇进行前体物质的竞争,有利于目的基因簇的表达[34]。Gomez等[35]在S. coelicolor中敲除放线紫红素、十一烷基灵菌红素、钙依赖抗生素和coelimycin的生物合成基因簇,再将rpoB和rpsL进行突变,高效表达了氯霉素、纺锤菌素等抗生素的生物合成基因簇。关于盐霉素生物合成基因簇的异源表达,Yin等[19]在S. coelicolor中进行了初步尝试,所克隆的基因簇可以在该菌中进行表达,但是产量较低,表达系统尚需要进一步优化,而选用不同的宿主菌和进行宿主菌改造是常用的策略。在S. avermitilis中筛除冗余内源基因簇I型PKS,pladienolides的产量是改造前的20倍[36];S. albus J1074的基因组序列是目前相对最短的,通常不产生生物活性代谢产物,拥有attB和假attB的ΦC1整合位点,可以接收外源基因并将其整合到染色体上[37]。因此,可选择合适的宿主菌对盐霉素基因簇进行表达,筛选出表达效率最高的宿主,从而提高盐霉素的产量。 总之,传统的诱变育种是盐霉素菌株选育的常规手段,而近年来盐霉素产生菌的基因组测序已经完成,生物合成途径得以解析,菌株改造正在朝定向进化和理性改造方面发展,除以上提到的代谢工程之外,其他理性改造策略也可能将会在盐霉素菌株中发挥重要作用。如非编码RNA (non-coding RNA, ncRNA),通常发挥多种功能,其中有些起重要的调控作用。通过碱基配对,识别并调控靶标mRNA的翻译或稳定性,是菌株适应环境压力和代谢调节的重要调控因子[38]。通过RNA-seq等发现在链霉菌中存在sRNA (small RNA),asRNA (antisense RNA)以及cutoRNA (convergent un-translated overlapping RNA)等多种ncRNA,很多位于抗生素生物合成基因簇中,通过表观遗传的方式影响附近基因的表达和翻译,构成了链霉菌生长发育和代谢的另一个重要调控系统[38-40]。目前盐霉素产生菌中ncRNA对抗生素生物合成的影响研究不多,但是由于其存在的广泛性和功能多样性,随着对调控机制的深度解析,ncRNA将可能成为盐霉素产生菌改造的另一个新靶点。而合成生物学是很有潜力的抗生素产生菌优化的新方法,其在代谢工程的基础上,利用生物、化学、工程和计算机等多种学科知识,优化设计生物的生命系统以及代谢通路,并构建与合成、代谢、调控以及信号系统等功能相关的生物模块,或者引入新的功能基因模块,进而组装人工生物系统,使目的产物超量合成[41]。目前已经构建了一些超级底盘细胞[37],以及人工合成了具有普遍适用性的优化后的强启动子库和RBS元件库[42-43],可望用于盐霉素生物合成基因簇中相关元件的置换以及异源表达,提高盐霉素的生物合成效率。这些新的调控机制和改造策略将为盐霉素产生菌改造提供新思路。 2 盐霉素的活性优化 盐霉素具有广谱的抗菌、抗球虫活性,是一种在国内外畜禽养殖中广泛使用的农用抗生素;同时细胞和动物体内实验都显示盐霉素对多种肿瘤细胞和肿瘤干细胞具有很强的杀伤作用,是一个很有应用前景的新一代抗肿瘤候选药物。但是盐霉素的进一步应用和开发却面临很大挑战:长期用药可诱导耐药菌株的出现,而盐霉素的药物选择性较低,呈现较显著的毒副作用,阻碍了临床研究的进行。因此,在获得高产菌株以满足市场需求的同时,还需要不断开发结构多样的新型盐霉素衍生物去应对药物耐受性问题,而如何提高盐霉素的生物活性、增强其药物作用的靶向性和改善药学特征,是保障临床研究成功的一个重要环节。
2.1 盐霉素的衍生物及活性研究 化学结构修饰是改变药物活性的主要方式,可以通过基因工程、化学合成等技术获得结构多样的衍生物。如Jiang等[44]阻断S. albus中的slnM基因,可以得到3种盐霉素的衍生物,但抗肿瘤能力和抗菌活性要弱于原盐霉素(图 3-A、B、C);Huang等[45-46]采用半合成的方式,对盐霉素C20位进行脱羟基、酰化或者C18-C19加氢还原,得到5种衍生物(图 3-D、E、F),其对乳腺癌肿瘤干细胞的杀伤作用均优于盐霉素;Huczyński和Antoszczak等将氟尿苷[47](图 3-G1)和水飞蓟宾[48](图 3-G2)分别与盐霉素结合形成复合物,其效果强于这3种药物的单独使用,对正常细胞的毒副作用更低,且前者对耐甲氧西林金黄色葡萄球菌(MRSA)也有很好的抑菌效果;而在C1位的羟基上分别连接5种不同的L-氨基酸甲基酯(图 3-H),发现对白血病的细胞增殖及耐阿霉素的结肠癌细胞均有很好的杀伤作用,毒副作用也比常规化疗药要低[49];但是在羧基上进行单取代的卤化或硝基化N-苄胺酰胺化,衍生出12种新化合物(图 3-I),却发现这些衍生物的抗肿瘤能力和抗菌活性均弱于原盐霉素[50]。
 |
| 图 3. 代表性盐霉素衍生物的化学结构 Figure 3. Chemical structures of representative salinomycin derivatives. A–F: Salinomycin derivatives with modifications at C17–20; G–I: salinomycin derivatives with modifications at carboxyl group. |
| 图选项 |
综上所述,目前尚未获得选择性和活性都显著高于原盐霉素的衍生物,可能是由于修饰位点单一、衍生物种类和数量有限等因素造成的。但随着对盐霉素生物合成基因功能的研究,敲除个别结构基因可能产生更多新的衍生物;而将不同PKS模块进行组合,可合成多种PKS骨架及结构多样的终产物;或者在基因组中引入负责合成其他特殊功能基团的基因(簇),经组合生物合成得到杂合化合物,可能兼具多种生物活性特征。如Menzella等[51]将8个不同聚酮类化合物PKS模块进行拼接得到了154个新的组合,其中有一半左右的组合都合成了新的化合物。此外计算机辅助药物设计可帮助理性设计化合物,增加衍生物的种类,构成化合物文库,通过高通量筛选,获得对不同细菌和肿瘤细胞靶向性都提高的新化合物。
2.2 药物载体对盐霉素活性的影响 不同药物剂型和用药方式对改善药物的靶向性和利用率有重要影响。T??l?[52]与Ni[53]等分别用聚乳酸-羟基乙酸共聚物将盐霉素封装成2种不同的纳米颗粒,前者能诱导人胶质母细胞瘤细胞凋亡,后者可选择性的杀死骨肉瘤细胞;Mao等[54]用iRGD (internalizing Arg-Gly-Asp peptide)与盐霉素形成纳米胶束,发现对肝癌细胞及干细胞具有很好的活性;Daman等[55]用载盐霉素的聚乙二醇-聚乳酸聚合物胶束对耐吉西他滨的胰腺癌细胞进行小鼠试验,发现不仅促进了癌细胞的死亡和凋亡,而且动物的生存率提高;Yao等[56]在单壁碳纳米管中装载盐霉素、壳聚糖及透明质酸形成复合物,可以特异性穿透并进入胃癌及干细胞胞内,对细胞的凋亡有很强的促进作用。可见通过纳米技术携带盐霉素,能实现药物缓释、专一性增强的目的。根据相似的作用原理,相信在抗革兰氏阳性菌、球虫等应用中,也可能提高盐霉素的细胞渗透性和靶向性,降低毒副作用。除了这些辅助试剂,细胞穿膜肽(cell penetrating peptides CPPs)作为一种广泛使用的药物载体,其独特的生物活性及效应[57],可能会在盐霉素应用中发挥更好的功能,成为另一种有效的辅助载体。
2.3 盐霉素与其他药物的协同作用 联合用药可以发挥药物协同作用从而提高疗效,是临床疾病治疗的常规措施之一。Hermawan等[58]用盐霉素与阿霉素联合使用,能抑制乳腺癌耐药基因MDR1和BCRP1的表达,提高细胞对阿霉素的敏感性及疗效;Zou等[59]用盐霉素与吉非替尼联合用药,可诱导结直肠癌细胞凋亡;Zhang等[60]发现盐霉素处理后的鼻咽癌细胞,对辐射的敏感性提高,有望作为鼻咽癌放疗辐射敏化剂;Jangamreddy等[61]用盐霉素分别与2-氟-2-脱氧-D-葡萄糖、2-脱氧-D-葡萄糖联合处理喉癌细胞,可增加喉癌细胞的Caspase程序性凋亡,并降低对盐霉素的保护性自噬;Kai等[62]发现组蛋白脱乙酰酶抑制剂LBH589和盐霉素能很好地协同抑制三阴性乳腺癌(triple-negative breast cancer TNBC)细胞,诱导其死亡,且没有明显的毒副作用;Xiao等[63]发现二甲双胍和盐霉素联合使用,对非小细胞肺癌及干细胞有很好的杀伤作用。可见,联合用药是提高盐霉素的药理活性的有效途径。
3 盐霉素的应用研究 高产菌株的获得为盐霉素的应用提供了前提和保障,而盐霉素化学结构的修饰及活性优化是提高盐霉素药理活性、减少毒副作用、改善药学特征的重要手段,这些进展为盐霉素在农业和医疗(如抗肿瘤的临床研究)中的广泛应用创造了必要条件。
3.1 盐霉素在农业上的应用 现代养殖主要采用集约化的养殖模式,其环境差、密度高、动物易患病及大量死亡,而在动物饲料里加入抗生素可有效控制动物疾病及死亡,还可促进机体生长发育,提高动物生长速度,使养殖户获利。而在追求产量及提高经济效益的同时,盲目的用药导致动物体内产生多种耐药基因,且多数抗生素在动物体内并未完全吸收或代谢,而又直接排泄出体外,其排泄物作为肥料广泛施用,又随之渗透至土壤及地表水造成严重污染,导致环境微生物产生了抗药性,同时这些微生物和水源中残留的抗生素又经农作物吸收和富集进入食物链,对动物和人体构成潜在的威胁[64]。
盐霉素具有较强的抗革兰氏阳性菌和杀灭球虫作用,作为离子载体型抗生素,其抗菌机制独特,所以不易产生交叉抗药性,在各种禽畜的粪便中检出率较低,在富营养水域降解速率快、残留量低,对微生物重污染区域能起到净化及改善的作用[4]。目前盐霉素主要作为饲料添加剂使用,1993年经我国农业部批准作为鸡球虫抑制剂和饲料添加剂,2002年是被欧盟允许使用的4种动物饲料抗生素之一,同时盐霉素在使用剂量上需严格加以控制,防止其滥用[4]。因此,盐霉素作为一种“绿色”环保抗生素,在欧美等国际市场上需求量大,据预测,我国动物饲养用盐霉素每年的缺口在11000吨左右,未来具有可观的经济收益和市场前景,而高产菌株的获得是解决市场需求的必要手段。同时本实验室发现盐霉素对临床多重耐药菌株(如金黄色葡萄球菌、表皮葡萄球菌等)具有很好的抑制作用(未发表),其是否对那些罕见的“超级耐药菌”也同样有效,将是我们今后研究的重要方向。
3.2 盐霉素在抗肿瘤上的应用 近年来,各种生态环境的污染日益严重,2012年全世界肿瘤新发及死亡人数约为1400万和820万,其中中国为306万和220万,约占全球的20%和25%;但2015年中国肿瘤新发及死亡病例约有429.2万和281.4万,说明我国肿瘤的新生及死亡率已呈明显上升趋势[65],如何控制恶性肿瘤的发生已成为我国的首要任务,同时也是世界性难题。
目前,抗肿瘤药物种类繁多,紫杉醇及其结构类似物是临床应用非常广泛的经典抗肿瘤药,早期的来源主要依赖于植物中提取,后来微生物发酵获得前体后,再进行半合成大大减少了林木砍伐,缓解了环境破坏。但是研究表明,盐霉素的抗肿瘤细胞活性比紫杉醇高近100倍,且作用范围非常广泛,因此在肿瘤疾病的治疗中有很大的应用潜力[66]。盐霉素不仅能抑制肺癌、白血病、骨肉瘤等多种肿瘤细胞及干细胞的生长和转移,还能通过多种途径诱导细胞凋亡,明显提高患者的生存率。其机制主要为:(1)?穿透细胞膜进入肿瘤细胞及CSCs,使Na+/K+失去平衡,使K+从细胞质和线粒体流出,让细胞内的pH降低,抑制DNA合成,促使细胞凋亡[1, 67];(2)?下调肿瘤细胞及CSCs细胞周期调控因子Cyclin D1、STAT3、ALDH1及Skp2的表达,促进细胞凋亡[46, 67];(3)?诱导癌细胞及CSCs氧化应激反应,增加细胞中的ROS水平,从而诱导细胞凋亡[59];(4)?激活AMPK,下调肿瘤细胞及肿瘤干细胞周期的相关癌基因表达,使细胞自噬性死亡[60-61, 68];(5)?通过对Wnt/β-catenin信号途径中相关蛋白及Wnt/β-catenin信号途径的靶基因及蛋白表达抑制来实现对肿瘤细胞及CSCs增殖及转移的抑制作用[69];(6)?抑制Hedgehog信号通路的激活,使该途径中的相关蛋白PTCH、Smo、Gli1、Gli2表达下调,从而进一步抑制其下游靶基因c-myc、bcl-2及转录因子Snail的表达,促使肿瘤细胞凋亡[70];(7)?下调外排泵、多重耐药性(multiple drug resistance, MDR)等基因的表达水平,降低细胞的耐药性,增强相关化疗药物对细胞的敏感性,提高药物对细胞的杀伤作用[58, 71]。
鉴于盐霉素在抗肿瘤细胞方面的重要作用,2010年研究人员对一些转移性乳腺癌、转移性卵巢癌、转移性头颈部鳞状上皮癌患者进行隔日静脉注射盐霉素200-250 μg/kg,共3周的临床性治疗研究,发现原发肿瘤及转移瘤有部分消退的迹象,且没有传统化疗药物的副作用[72]。Resham等[73]进行了初步前期临床研究,表明盐霉素在小鼠体内经肝代谢,降解速度快,内在清除率和血浆蛋白结合率都比较高。盐霉素的抗肿瘤作用大多还停留在细胞和动物体内实验阶段,系统的临床研究亟待进行。
4 展望 盐霉素是一种在养殖业和医学中有着重要价值的抗生素,具有抗菌谱广、低残留等特点,可以缓解抗生素滥用和环境污染等棘手问题;同时盐霉素还有突出的抗肿瘤细胞活性,有望成为新一代抗肿瘤药物。为满足正在扩大的市场需求、降低工业成本,需要不断对盐霉素产生菌进行优化改造,以提高产量。目前传统的诱变育种正在朝定向进化和理性改造方面发展,代谢工程开始逐渐在盐霉素菌株改造中得到应用,而合成生物学以及新的调控靶点(全局性调控因子、ncRNA)等将有可能开启对盐霉素生物合成和调控的新领域。此外,为了应对病原菌耐药性的出现、解决盐霉素药物选择性低的问题,对盐霉素结构和功能进行优化成为另一个研究热点:通过化学合成、基因工程等手段,可获得结构多样的盐霉素衍生物,并从中筛选活性高、毒副作用低的绿色、环境友好的新型抗生素;而药物载体以及联合用药策略可有效增强药物对肿瘤细胞的药理活性和靶向性。这些都将为扩大盐霉素适用范围、开展盐霉素的临床研究提供重要前提和保障,进而促进盐霉素在农业(如养殖业)和医药(如肿瘤治疗)等领域中发挥更重要的作用。
致谢: 值此论文付梓之际,感谢中国科学院微生物研究所的田宇清副研究员和牛国清副研究员在论文写作过程中提出的宝贵建议。
参考文献
| [1] | Zhou S, Wang FF, Wong ET, Fonkem E, Hsieh TC, Wu JM, Wu EX. Salinomycin:a novel anti-cancer agent with known anti-coccidial activities.Current Medicinal Chemistry, 2013, 20(33): 4095–4101DOI:10.2174/15672050113109990199. |
| [2] | Jiang CY, Wang HG, Kang QJ, Liu J, Bai LQ. Cloning and characterization of the polyether salinomycin biosynthesis gene cluster of Streptomyces albus XM211.Applied and Environmental Microbiology, 2012, 78(4): 994–1003DOI:10.1128/AEM.06701-11. |
| [3] | Yurkovich ME, Tyrakis PA, Hong H, Sun YH, Samborskyy M, Kamiya K, Leadlay PF. A late-stage intermediate in salinomycin biosynthesis is revealed by specific mutation in the biosynthetic gene cluster.ChemBioChem, 2012, 13(1): 66–71DOI:10.1002/cbic.201100590. |
| [4] | Miyazaki Y, Shibuya M, Sugawara H, Kawaguchi O, Hirose C, Nagatsu J, Esum S. Salinomycin, a new polyether antibiotic.The Journal of Antibiotics, 1974, 27(11): 814–821DOI:10.7164/antibiotics.27.814. |
| [5] | Jia YF, Liang JG, Wang YH, Hao YY, Chu J. High-throughput screening of high salinomycin-producing strains with UV treatment on protoplasts.Chinese Journal of Pharmaceuticals, 2013, 44(6): 560–564(in Chinese).贾永峰, 梁剑光, 王永红, 郝玉有, 储炬. 原生质体紫外诱变高通量选育盐霉素高产菌.中国医药工业杂志, 2013, 44(6): 560–564. |
| [6] | Liu P, Hang HF, Chu XH, Guo MJ, Chu J, Zhuang YP, Zhang SL. Screening of high-salinomycin-yield mutants of Streptomyces albus by self-resistance methodology.Chinese Journal of Antibiotics, 2012, 37(2): 101–104(in Chinese).刘朋, 杭海峰, 储消和, 郭美锦, 储炬, 庄英萍, 张嗣良. 盐霉素自身抗性高产突变菌株筛选.中国抗生素杂志, 2012, 37(2): 101–104. |
| [7] | Wu LQ, Miao FX, Gu HK, Shang HZ. Breeding of high-yield salinomycin-producing Streptomyces albus strain by low energy N+ion beam irradiation.Journal of Anhui Agricultural Sciences, 2012, 40(28): 13734–13735(in Chinese).武利勤, 苗凤香, 顾海科, 尚宏忠. 低能离子注入诱变选育盐霉素高产菌株.安徽农业科学, 2012, 40(28): 13734–13735. |
| [8] | Ochi K, Hosaka T. New strategies for drug discovery:activation of silent or weakly expressed microbial gene clusters.Applied Microbiology and Biotechnology, 2013, 97(1): 87–98DOI:10.1007/s00253-012-4551-9. |
| [9] | Wang GJ, Hosaka T, Ochi K. Dramatic activation of antibiotic production in Streptomyces coelicolor by cumulative drug resistance mutations.Applied and Environmental Microbiology, 2008, 74(9): 2834–2840DOI:10.1128/AEM.02800-07. |
| [10] | Tamehiro N, Hosaka T, Xu J, Hu HF, Otake N, Ochi K. Innovative approach for improvement of an antibiotic-overproducing industrial strain of Streptomyces albus.Applied and Environmental Microbiology, 2003, 69(11): 6412–6417DOI:10.1128/AEM.69.11.6412-6417.2003. |
| [11] | Wu J, Dai GF, Li ZW, Zhang XH, Huo YP. The breeding of high yield salinomycin strains of Streptomyces albus through antibiotic-resistant strains screening.Journal of Zhengzhou University (Natural Science Edition), 2005, 37(4): 92–95(in Chinese).吴健, 戴桂馥, 李宗伟, 张晓红, 霍裕平. 利用抗药性选育盐霉素高产菌株.郑州大学学报(理学版), 2005, 37(4): 92–95. |
| [12] | Zhang YX, Perry K, Vinci VA, Powell K, Stemmer WPC, del Cardayré SB. Genome shuffling leads to rapid phenotypic improvement in bacteria.Nature, 2002, 415(6872): 644–646DOI:10.1038/415644a. |
| [13] | Wang QL, Zhang D, Li YD, Zhang FM, Wang C, Liang XL. Genome shuffling and ribosome engineering of Streptomyces actuosus for high-yield nosiheptide production.Applied Biochemistry and Biotechnology, 2014, 173(6): 1553–1563DOI:10.1007/s12010-014-0948-5. |
| [14] | Du WJ, Huang D, Xia ML, Wen JP, Huang M. Improved FK506 production by the precursors and product-tolerant mutant of Streptomyces tsukubaensis based on genome shuffling and dynamic fed-batch strategies.Journal of Industrial Microbiology&Biotechnology, 2014, 41(7): 1131–1143. |
| [15] | Jin ZH, Xu B, Lin SZ, Jin QC, Cen PL. Enhanced production of spinosad in Saccharopolyspora spinosa by genome shuffling.Applied Biochemistry and Biotechnology, 2009, 159(3): 655–663DOI:10.1007/s12010-008-8500-0. |
| [16] | Murrell JM, Liu W, Shen B. Biochemical characterization of the SgcA1α-D-glucopyranosyl-1-phosphate thymidylyltransferase from the enediyne antitumor antibiotic C-1027 biosynthetic pathway and overexpression of sgcA1 in Streptomyces globisporus to improve C-1027 production.Journal of Natural Products, 2004, 67(2): 206–213DOI:10.1021/np0340403. |
| [17] | Du DY, Wang L, Tian YQ, Liu H, Tan HR, Niu GQ. Genome engineering and direct cloning of antibiotic gene clusters via phage ΦBT1 integrase-mediated site-specific recombination in Streptomyces.Scientific Reports, 2015, 5: 8740DOI:10.1038/srep08740. |
| [18] | Liao GJ, Li JE, Li L, Yang HH, Tian YQ, Tan HR. Cloning, reassembling and integration of the entire nikkomycin biosynthetic gene cluster into Streptomyces ansochromogenes lead to an improved nikkomycin production.Microbial Cell Factories, 2010, 9(1): 6DOI:10.1186/1475-2859-9-6. |
| [19] | Yin J, Hoffmann M, Bian XY, Tu Q, Yan F, Xia LQ, Ding XZ, Stewart AF, Müller R, Fu J, Zhang YM. Direct cloning and heterologous expression of the salinomycin biosynthetic gene cluster from Streptomyces albus DSM41398 in Streptomyces coelicolor A3(2).Scientific Reports, 2015, 5: 15081DOI:10.1038/srep15081. |
| [20] | Li R, Xie ZJ, Tian YQ, Yang HH, Chen WQ, You DL, Liu G, Deng ZX, Tan HR. polR, a pathway-specific transcriptional regulatory gene, positively controls polyoxin biosynthesis in Streptomyces cacaoi subsp.asoensis.Microbiology, 2009, 155(6): 1819–1831DOI:10.1099/mic.0.028639-0. |
| [21] | Smanski MJ, Peterson RM, Rajski SR, Shen B. Engineered Streptomyces platensis strains that overproduce antibiotics platensimycin and platencin.Antimicrobial Agents and Chemotherapy, 2009, 53(4): 1299–1304DOI:10.1128/AAC.01358-08. |
| [22] | Li L, Zhao YW, Ruan LJ, Yang S, Ge M, Jiang WH, Lu YH. A stepwise increase in pristinamycin Ⅱ biosynthesis by Streptomyces pristinaespiralis through combinatorial metabolic engineering.Metabolic Engineering, 2015, 29: 12–25DOI:10.1016/j.ymben.2015.02.001. |
| [23] | Higo A, Horinouchi S, Ohnishi Y. Strict regulation of morphological differentiation and secondary metabolism by a positive feedback loop between two global regulators AdpA and BldA in Streptomyces griseus.Molecular Microbiology, 2011, 81(6): 1607–1622DOI:10.1111/j.1365-2958.2011.07795.x. |
| [24] | Gomez-Escribano JP, Martín JF, Hesketh A, Bibb MJ, Liras P. Streptomyces clavuligerus relA-null mutants overproduce clavulanic acid and cephamycin C:negative regulation of secondary metabolism by (p) ppGpp.Microbiology, 2008, 154(3): 744–755DOI:10.1099/mic.0.2007/011890-0. |
| [25] | Rabyk M, Ostash B, Rebets Y, Walker S, Fedorenko V. Streptomyces ghanaensis pleiotropic regulatory gene wblAgh influences morphogenesis and moenomycin production.Biotechnology Letters, 2011, 33(12): 2481–2486DOI:10.1007/s10529-011-0728-z. |
| [26] | Zhang L, Li WC, Zhao CH, Chater KF, Tao MF. NsdB, a TPR-like-domain-containing protein negatively affecting production of antibiotics in Streptomyces coelicolor A3(2).Acta Microbiologica Sinica, 2007, 47(5): 849–854. |
| [27] | Wang XJ, Guo SL, Guo WQ, Xi D, Xiang WS. Role of nsdA in negative regulation of antibiotic production and morphological differentiation in Streptomyces bingchengensis.The Journal of Antibiotics, 2009, 62(6): 309–313DOI:10.1038/ja.2009.33. |
| [28] | Woo MW, Nah HJ, Choi SS, Kim ES. Pikromycin production stimulation through antibiotic down-regulatory gene disruption in Streptomyces venezuelae.Biotechnology and Bioprocess Engineering, 2014, 19(6): 973–977DOI:10.1007/s12257-014-0407-8. |
| [29] | Ostash B, Doud E, Walker S. ABC transporter genes from Streptomyces ghanaensis moenomycin biosynthetic gene cluster:roles in antibiotic production and export.Archives of Microbiology, 2012, 194(11): 915–922DOI:10.1007/s00203-012-0827-9. |
| [30] | Qiu JF, Zhuo Y, Zhu DQ, Zhou XF, Zhang LX, Bai LQ, Deng ZX. Overexpression of the ABC transporter AvtAB increases avermectin production in Streptomyces avermitilis.Applied Microbiology and Biotechnology, 2011, 92(2): 337–345DOI:10.1007/s00253-011-3439-4. |
| [31] | Liu J, Jiang CY, Zhang BC, Bai LQ. Involvement of ABC transporter genes slnTI and slnTⅡ in salinomycin biosynthesis.Microbiology China, 2014, 41(1): 58–66(in Chinese).刘静, 姜春艳, 张部昌, 白林泉. ABC转运蛋白基因slnTI和slnTⅡ与盐霉素生物合成的相关性.微生物学通报, 2014, 41(1): 58–66. |
| [32] | Ren J, Cui YQ, Zhang F, Cui H, Ni XP, Chen F, Li L, Xia HZ. Enhancement of nystatin production by redirecting precursor fluxes after disruption of the tetramycin gene from Streptomyces ahygroscopicus.Microbiological Research, 2014, 169(7/8): 602–608. |
| [33] | Lu CY, Zhang XJ, Jiang M, Bai LQ. Enhanced salinomycin production by adjusting the supply of polyketide extender units in Streptomyces albus.Metabolic Engineering, 2016, 35: 129–137DOI:10.1016/j.ymben.2016.02.012. |
| [34] | Liu G, Chater KF, Chandra G, Niu GQ, Tan HR. Molecular regulation of antibiotic biosynthesis in Streptomyces.Microbiology and Molecular Biology Reviews, 2013, 77(1): 112–143DOI:10.1128/MMBR.00054-12. |
| [35] | Gomez-Escribano JP, Bibb MJ. Engineering Streptomyces coelicolor for heterologous expression of secondary metabolite gene clusters.Microbial Biotechnology, 2011, 4(2): 207–215DOI:10.1111/j.1751-7915.2010.00219.x. |
| [36] | Komatsu M, Uchiyama T, ?mura S, Cane DE, Ikeda H. Genome-minimized Streptomyces host for the heterologous expression of secondary metabolism.Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(6): 2646–2651DOI:10.1073/pnas.0914833107. |
| [37] | Baltz RH. Genetic manipulation of secondary metabolite biosynthesis for improved production in Streptomyces and other actinomycetes.Journal of Industrial Microbiology&Biotechnology, 2016, 43(2/3): 343–370. |
| [38] | Moody MJ, Young RA, Jones SE, Elliot MA. Comparative analysis of non-coding RNAs in the antibiotic-producing Streptomyces bacteria.BMC Genomics, 2013, 14(1): 558DOI:10.1186/1471-2164-14-558. |
| [39] | Pánek J, Bobek J, Mikulík K, Basler M, Vohradsky J. Biocomputational prediction of small non-coding RNAs in Streptomyces.BMC Genomics, 2008, 9(1): 217DOI:10.1186/1471-2164-9-217. |
| [40] | Vockenhuber MP, Heueis N, Suess B. Identification of metE as a second target of the sRNA scr5239 in Streptomyces coelicolor.PLoS One, 2015, 10(3): e0120147DOI:10.1371/journal.pone.0120147. |
| [41] | Endy D. Foundations for engineering biology.Nature, 2005, 438(7067): 449–453DOI:10.1038/nature04342. |
| [42] | Bai CX, Zhang Y, Zhao XJ, Hu YL, Xiang SH, Miao J, Lou CB, Zhang LX. Exploiting a precise design of universal synthetic modular regulatory elements to unlock the microbial natural products in Streptomyces.Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(39): 12181–12186DOI:10.1073/pnas.1511027112. |
| [43] | Sohoni SV, Fazio A, Workman CT, Mijakovic I, Lantz AE. Synthetic promoter library for modulation of actinorhodin production in Streptomyces coelicolor A3(2).PLoS One, 2014, 9(6): e99701DOI:10.1371/journal.pone.0099701. |
| [44] | Jiang CY, Qi Z, Kang QJ, Liu J, Jiang M, Bai LQ. Formation of the Δ18, 19 double bond and bis (spiroacetal) in salinomycin is atypically catalyzed by SlnM, a methyltransferase-like enzyme.Angewandte Chemie International Edition, 2015, 54(31): 9097–9100DOI:10.1002/anie.201503561. |
| [45] | Huang XL, Borgstr?m B, M?nsson L, Persson L, Oredsson S, Hegardt C, Strand D. Semisynthesis of SY-1 for investigation of breast cancer stem cell selectivity of C-ring-modified salinomycin analogues.ACS Chemical Biology, 2014, 9(7): 1587–1594DOI:10.1021/cb5002153. |
| [46] | Huang XL, Borgstr?m B, Kempengren S, Persson L, Hegardt C, Strand D, Oredsson S. Breast cancer stem cell selectivity of synthetic nanomolar-active salinomycin analogs.BMC Cancer, 2016, 16(1): 675DOI:10.1186/s12885-016-2727-x. |
| [47] | Huczyński A, Antoszczak M, Kleczewska N, Lewandowska M, Maj E, Stefańska J, Wietrzyk J, Janczak J, Celewicz L. Synthesis and biological activity of salinomycin conjugates with floxuridine.European Journal of Medicinal Chemistry, 2015, 93: 33–41DOI:10.1016/j.ejmech.2015.01.045. |
| [48] | Antoszczak M, Klejborowska G, Kruszyk M, Maj E, Wietrzyk J, Huczyński A. Synthesis and antiproliferative activity of silybin conjugates with salinomycin and monensin.Chemical Biology&Drug Design, 2015, 86(6): 1378–1386. |
| [49] | Antoszczak M, Sobusiak M, Maj E, Wietrzyk J, Huczyński A. Synthesis and antiproliferative activity of new bioconjugates of salinomycin with amino acid esters.Bioorganic&Medicinal Chemistry Letters, 2015, 25(17): 3511–3514. |
| [50] | Antoszczak M, Maj E, Napiórkowska A, Stefańska J, Augustynowicz-Kope? E, Wietrzyk J, Janczak J, Brzezinski B, Huczyński A. Synthesis, anticancer and antibacterial activity of salinomycin N-benzyl amides.Molecules, 2014, 19(12): 19435–19459DOI:10.3390/molecules191219435. |
| [51] | Menzella HG, Reid R, Carney JR, Chandran SS, Reisinger SJ, Patel KG, Hopwood DA, Santi DV. Combinatorial polyketide biosynthesis by de novo design and rearrangement of modular polyketide synthase genes.Nature Biotechnology, 2005, 23(9): 1171–1176DOI:10.1038/nbt1128. |
| [52] | T??l? Ayd?n RS, Kaynak G, Gümü?derelio?lu M. Salinomycin encapsulated nanoparticles as a targeting vehicle for glioblastoma cells.Journal of Biomedical Materials Research Part A, 2016, 104(2): 455–464DOI:10.1002/jbm.a.35591. |
| [53] | Ni MZ, Xiong M, Zhang XC, Cai GP, Chen HW, Zeng QM, Yu ZC. Poly (lactic-co-glycolic acid) nanoparticles conjugated with CD133 aptamers for targeted salinomycin delivery to CD133+ osteosarcoma cancer stem cells.International Journal of Nanomedicine, 2015, 10(1): 2537–2554. |
| [54] | Mao XL, Liu JJ, Gong ZR, Zhang H, Lu Y, Zou H, Yu Y, Chen Y, Sun ZG, Li W, Li BH, Gao J, Zhong YQ. iRGD-conjugated DSPE-PEG2000 nanomicelles for targeted delivery of salinomycin for treatment of both liver cancer cells and cancer stem cells.Nanomedicine, 2015, 10(17): 2677–2695DOI:10.2217/nnm.15.106. |
| [55] | Daman Z, Montazeri H, Azizi M, Rezaie F, Ostad SN, Amini M, Gilani K. Polymeric micelles of PEG-PLA copolymer as a carrier for salinomycin against gemcitabine-resistant pancreatic cancer.Pharmaceutical Research, 2015, 32(11): 3756–3767DOI:10.1007/s11095-015-1737-8. |
| [56] | Yao HJ, Zhang YG, Sun L, Liu Y. The effect of hyaluronic acid functionalized carbon nanotubes loaded with salinomycin on gastric cancer stem cells.Biomaterials, 2014, 35(33): 9208–9223DOI:10.1016/j.biomaterials.2014.07.033. |
| [57] | Dinca A, Chien WM, Chin MT. Intracellular delivery of proteins with cell-penetrating peptides for therapeutic uses in human disease.International Journal of Molecular Sciences, 2016, 17(2): 263DOI:10.3390/ijms17020263. |
| [58] | Hermawan A, Wagner E, Roidl A. Consecutive salinomycin treatment reduces doxorubicin resistance of breast tumor cells by diminishing drug efflux pump expression and activity.Oncology Reports, 2016, 35(3): 1732–1740. |
| [59] | Zou ZZ, Nie PP, Li YW, Hou BX, Li R, Shi XP, Ma ZK, Han BW, Luo XY. Synergistic induction of apoptosis by salinomycin and gefitinib through lysosomal and mitochondrial dependent pathway overcomes gefitinib resistance in colorectal cancer.Oncotarget, 2015DOI:10.18632/oncotarget.5628. |
| [60] | Zhang YQ, Zuo Y, Guan ZF, Lu WD, Xu Z, Zhang H, Yang Y, Yang ML, Zhu HC, Chen XC. Salinomycin radiosensitizes human nasopharyngeal carcinoma cell line CNE-2 to radiation.Tumor Biology, 2016, 37(1): 305–311DOI:10.1007/s13277-015-3730-6. |
| [61] | Jangamreddy JR, Jain MV, Hallbeck AL, Roberg K, Lotfi K, ?os MJ. Glucose starvation-mediated inhibition of salinomycin induced autophagy amplifies cancer cell specific cell death.Oncotarget, 2015, 6(12): 10134–10145DOI:10.18632/oncotarget. |
| [62] | Kai M, Kanaya N, Wu SV, Mendez C, Nguyen D, Luu T, Chen S. Targeting breast cancer stem cells in triple-negative breast cancer using a combination of LBH589 and salinomycin.Breast Cancer Research and Treatment, 2015, 151(2): 281–294DOI:10.1007/s10549-015-3376-5. |
| [63] | Xiao ZG, Sperl B, Ullrich A, Knyazev P. Metformin and salinomycin as the best combination for the eradication of NSCLC monolayer cells and their alveospheres (cancer stem cells) irrespective of EGFR, KRAS, EML4/ALK and LKB1 status.Oncotarget, 2014, 5(24): 12877–12890. |
| [64] | Hao RZ, Zhao RT, Qiu SF, Wang LG, Song HB. Antibiotics crisis in China.Science, 2015, 348(6239): 1100–1101. |
| [65] | Chen WQ, Zheng RS, Baade PD, Zhang SW, Zeng HM, Bray F, Jemal A, Yu XQ, He J. Cancer statistics in China, 2015.CA:A Cancer Journal for Clinicians, 2016, 66(2): 115–132DOI:10.3322/caac.21338. |
| [66] | Gupta PB, Onder TT, Jiang GZ, Tao K, Kuperwasser C, Weinberg RA, Lander ES. Identification of selective inhibitors of cancer stem cells by high-throughput screening.Cell, 2009, 138(4): 645–659DOI:10.1016/j.cell.2009.06.034. |
| [67] | Antoszczak M, Huczyński A. Anticancer activity of polyether ionophore-salinomycin.Anti-cancer Agents in Medicinal Chemistry, 2015, 15(5): 575–591DOI:10.2174/1871520615666150101130209. |
| [68] | Zhu LQ, Zhen YF, Zhang Y, Guo ZX, Dai J, Wang XD. Salinomycin activates AMP-activated protein kinase-dependent autophagy in cultured osteoblastoma cells:a negative regulator against cell apoptosis.PLoS One, 2013, 8(12). |
| [69] | Lu WY, Li YH. Salinomycin suppresses LRP6 expression and inhibits both Wnt/β-catenin and mTORC1 signaling in breast and prostate cancer cells.Journal of Cellular Biochemistry, 2014, 115(10): 1799–1807DOI:10.1002/jcb.24850. |
| [70] | Fu YZ, Yan YY, He M, Xiao QH, Yao WF, Zhao L, Wu HZ, Yu ZJ, Zhou MY, Lv MT, Zhang SS, Chen JJ, Wei MJ. Salinomycin induces selective cytotoxicity to MCF-7 mammosphere cells through targeting the hedgehog signaling pathway.Oncology Reports, 2016, 35(2): 912–922. |
| [71] | Kim KY, Kim SH, Yu SN, Park SK, Choi HD, Yu HS, Ji JH, Seo YK, Ahn SC. Salinomycin enhances doxorubicin-induced cytotoxicity in multidrug resistant MCF-7/MDR human breast cancer cells via decreased efflux of doxorubicin.Molecular Medicine Reports, 2015, 12(2): 1898–1904. |
| [72] | Naujokat C, Steinhart R. Salinomycin as a drug for targeting human cancer stem cells.Journal of Biomedicine and Biotechnology, 2012, 2012: 950658. |
| [73] | Resham K, Patel PN, Thummuri D, Guntuku L, Shah V, Bambal RB, Naidu VGM. Preclinical drug metabolism and pharmacokinetics of salinomycin, a potential candidate for targeting human cancer stem cells.Chemicoε, 2015, 240: 146–152. |
