韩永和1

 , 王珊珊2
, 王珊珊2 1.南京大学环境学院, 污染控制与资源化研究国家重点实验室, 江苏 南京 210046;
2.福建师范大学泉港石化研究院, 福建 泉州 362801
收稿日期:2015-07-13;修回日期:2015-08-27;网络出版日期:2016-06-04
资助课题:江苏省博士研究生创新项目(KYLX_0052);泉州市科技计划项目(2015Z105)
通信作者:Tel:+86-25-89680633;E-mail:hanyonghe0423@163.com
摘要: 砷是一种无处不在的有毒类金属,其强致癌性引起了人类的广泛关注。在自然环境中,砷的转化存在物理化学过程和生物过程,其中微生物介导的砷转化是环境砷行为的主要影响因素。微生物的耐砷特性与砷吸收、氧化还原、甲基化、区隔化和外排等过程密切相关。砷在微生物体内的转运转化主要与砷解毒有关,但某些微生物可利用氧化还原过程产生的能量以维持其生长需求。本文综述了微生物介导的砷吸收、转化、区隔化和外排机制,这对阐明砷的地球化学循环过程及指导砷污染土壤和水体修复、阻控农作物砷吸收等方面具有重要意义。
关键词: 砷 癌症 微生物 污染 生物地球化学循环
Heterologous Expressionand Characterization of a Thermostable Acyl-CoA Synthetase
Yonghe Han1

 , Shanshan Wang2
, Shanshan Wang2 1.State Key Laboratory of Pollution Control and Resource Reuse, School of the Environment, Nanjing University, Nanjing 210046, Jiangsu Province, China;
2.Quangang Research Institute of Petrochemical Technology, Fujian Normal University, Quanzhou 362801, Fujian Province, China
Supported by the Program for Postgraduates Research and Innovation in Jiangsu Province (KYLX_0052) and by the Quanzhou Projects in the Science and Technology Program (2015Z105)
Abstract: As a ubiquitous metalloid in the environment, arsenic (As) has attracted concerns by humans due to its strong carcinogenic property. Environmental As fate is often affected by physico-chemical and biological factors, of which microbe-mediated arsenic transformation predominates. Arsenic uptake, redox, methylation, sequestration and efflux have been verified to be involved in As resistance in microbes. In some microbes, As transformation contributes to detoxification, while some can obtain the energy for their growth and reproduction from this process. Here we reviewed the mechanisms of microbe-mediated As uptake, transformation, sequestration and efflux, which may help elucidate As geochemical cycling and provide the methods to remediate As-contaminated soils and waters, and to reduce As uptake by crops.
Key words: arsenic cancer microbes pollution biogeochemical cycling
砷(arsenic,As)是国际癌症研究组织(International Agency for Research on Cancer,IARC)最早确认的I类致癌物。砷在自然界中无处不在,其地壳丰度排第20位,在海水和人体中分别排第14位和第12位[1]。环境中的平均砷浓度为5 mg/kg,分布于560多种矿物中[如毒砂(FeAsS)、雄黄(As4S4)和雌黄(As2S3)等,以毒砂为主][2]。地球化学过程如矿物风化、火山喷发和森林火灾等会导致自然源砷矿物的释放,该过程是导致土壤砷(0.8-1.7×103 t)和大气砷(1.7-2.5×109 t)积累的主要原因[1]。此外,采矿、矿物冶炼、煤的燃烧,杀虫剂、除草剂、灭鼠剂、木材防腐剂,饲料加工、玻璃制造和染料中砷的添加等人为活动也是砷污染的重要原因[1-2]。
环保部和国土资源部2014年对全国土壤污染状况的调查显示,我国土壤镉、汞、砷、铅和铬5大重点监控重金属的点位超标率在1%-7%之间,砷超标率为3%[3]。农作物砷富集和地下水砷污染是导致人体砷暴露的主要途径[2, 4]。Jia等[4]和Zhang等[5]研究表明,我国农田土壤中砷的形态转化与微生物的氧化还原及甲基化作用密切相关。这些抗砷菌主要包括γ-Proteobacteria和δ-Proteobacteria,还包括ε-Proteobacteria、β-Proteobacteria和厚壁菌门(Firmicutes)中的芽孢杆菌(Bacillus sp.)[4-5]。最近的研究指出,微生物介导的铁锰氧化物及氢氧化物的还原与释放是导致地下水砷污染的主要原因[6]。Sforna等[7]认为,砷的微生物代谢起源于27亿年前,包括微生物细胞内AsV的还原和AsⅢ的甲基化、砷酸盐异化还原菌(dissimilatory arsenate-respiring prokaryotes,DARPs)和两种亚砷酸盐氧化菌——异养亚砷酸盐氧化菌(heterotrophic arsenite oxidizers,HAOs)和化能无机自养亚砷酸盐氧化菌(chemolitho-autotrophic arsenite oxidizers,CAOs)在细胞周质中对砷的氧化还原作用[8]以及AsⅢ的外排作用[9],如图1所示。由此可见,研究微生物耐砷机制对阐明砷的地球化学循环过程及阻控砷在农作物中的富集具有重要意义[1]。本文就微生物耐砷机理及其对砷的地球化学循环作用作一简要综述。
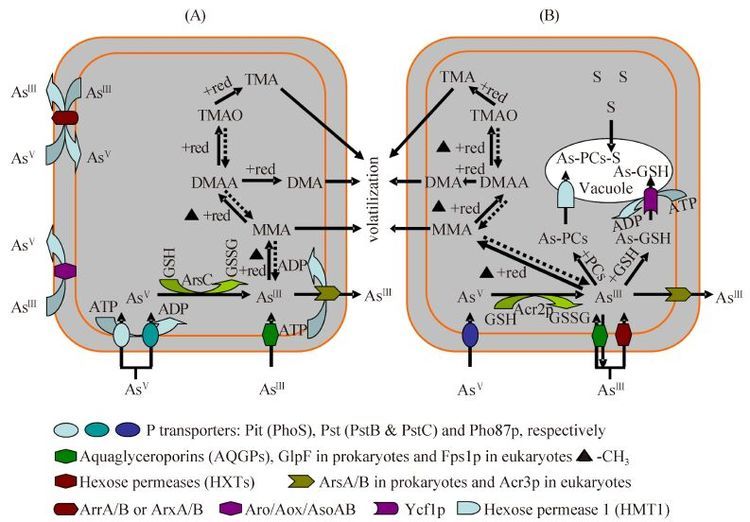 |
| 图 1. 原核微生物(A)和真核微生物(B)中砷的吸收与代谢示意图 Figure 1. Schematic diagram of arsenic uptake and metabolisms in prokaryotes (A) and eukaryotes (B). Solid lines and dotted lines indicate the major pathways and minor pathways of arsenic uptake and/or metabolisms respectively. As, arsenic; AsⅢ, arsenite; AsV, arsenate; ATP, adenosine triphosphate; ADP, adenosine diphosphate; GSH, reduced glutathione; ArsC/Acr2p, arsenate reductase; GSSG, oxidized glutathione; red, reduction; MMA, methylarsine; DMA, dimethylarsine; DMAA, dimethylarsinoylacetate; TMAO, trimethylarsine oxide; TMA, trimethylarsine; S, sulfur; PCs, phytochelatins. |
| 图选项 |
1 微生物对砷的吸收 AsV是磷的化学相似物,微生物中AsV的吸收可能与磷通道蛋白有关。20世纪70年代,Medveczky和Rosenberg[10]首次开展了微生物中AsV吸收的相关研究。结果表明,大肠杆菌AsV抗性缺失突变体也失去了对磷的吸收功能,说明磷通道蛋白可能参与了AsV的吸收。磷通道蛋白对磷和AsV都具有高亲和能力,原核生物中主要包括2类:Pit(PhoS)和Pst(PstB和PstC)(图1-A),前者是主要的通道蛋白且通常不能转运其他含氧阴离子[9, 11]。然而,原核生物中负责转运AsⅢ的水-甘油通道蛋白(aquaglyceroporins,AQGPs)GlpF直到1997年才得到确认,其最初定义的功能是负责SbⅢ的吸收[12]。与原核生物类似,真核生物对AsV和AsⅢ的吸收也是通过磷转运通道和AQGPs实现的(图1-B);不同的是,真核生物中参与AsV吸收的关键蛋白是Pho87,而Fps1p,Hxt1-5,Hxt7和Hxt9是AsⅢ转运通道蛋白[9]。
2 AsV 的还原机理 2.1 细胞内 ArsC/Acr2p 还原酶介导的 AsV 还原 几十亿年前,地球的极端还原环境促使微生物首先对AsⅢ产生了抗性,氧气的出现要求微生物对AsV的存在做出适应性调整。显然,将AsV转化为AsⅢ并排出体外是一种进化意义上经济有效的代谢途径。目前的研究认为,原核和真核微生物都存在将AsV还原为AsⅢ并将其排出体外的解毒机制(图1)。在原核生物中,AsV还原酶ArsC可由2种操纵子表达,第一种包含5个基因(arsRDABC),第二种包含3个基因(arsRBC)[13]。arsRDABC操纵子主要存在于革兰氏阴性细菌如大肠杆菌(Escherichia coli)的质粒上,而后者主要存在于革兰氏阳性细菌如金黄色葡萄球菌(Staphylococcus aureus)的质粒或者染色体上。其中,arsR基因编码ArsR/SmtB家族的反式作用阻遏蛋白进而对操纵子进行转录调控,arsB编码AsⅢ外排蛋白,而arsA和arsD负责编码2种附加蛋白——ArsA:受AsⅢ激活的ATP酶和ArsD:另一种类金属响应的转录阻遏蛋白[13]。真核微生物的AsV还原酶Acr2p则由arr基因编码。
根据AsV还原酶基因不同的进化顺序,其编码的AsV还原酶被分为3个家族[14]:第一类以谷氧还蛋白和谷胱甘肽(Grx-GSH)为还原剂,主要存在于以大肠杆菌、小肠结肠炎耶尔森氏菌(Yersinia enterocolitica)和流感嗜血杆菌(Haemophilusin fluenzae)等为代表的革兰氏阴性细菌的染色体和质粒上;第二类以硫氧还蛋白(Trx)为还原剂,主要存在于以金黄色葡萄球菌为代表的革兰氏阳性细菌及以枯草芽孢杆菌(Bacillus subtilis)和恶臭假单胞菌(Pseudomonas putida)为代表的革兰氏阴性细菌的染色体和质粒上。这类还原酶与小分子蛋白磷酸酪氨酸磷酸酶存在相似性;第三类是到目前为止发现的唯一一类存在于真菌中的AsV还原酶Acr2p,它介导的AsV还原由谷氧还蛋白作为还原剂且与包含细胞分化周期蛋白25a(cell division cycle 25a,Cdc25a)的磷酸酪氨酸磷酸酶存在相似性。
由ArsC/Acr2p介导的AsV还原是最普遍的1种砷解毒机制,它们在有氧或无氧条件下都可发生[9]。Mukhopadhyay和Rosen[14]认为,这2种酶与磷酸酶共用的是1个进化谱系,因此对理解哺乳动物中AsV的还原机制有启发意义。
2.2 细胞周质ArrA还原酶介导的AsV还原 与细胞内ArsC不同的是,原核微生物细胞周质的AsV呼吸还原酶(arsenate respiratory reductase,ArrA)与能量代谢有关,且只在厌氧条件下发挥作用[9]。
原核微生物如砷酸产金菌(Chrysiogenes arsenatis)在AsV还原的过程中,能以AsV为最终电子受体,并以醋酸盐为呼吸电子供体进行厌氧呼吸[15]。经鉴定,催化这个过程的酶是砷呼吸还原酶ArrAB,它是1种位于细胞周质的异质二聚体蛋白,由分子量为87 kDa (ArrA)和29 kDa (ArrB)的大小亚基组成。序列分析表明,ArrA是1种与二甲基亚砜(DMSO)还原酶相似的Mo/Fe蛋白,ArrB含有1个[4Fe-4S]簇,电子从[4Fe-4S]簇被传递给ArrA亚基的钼辅因子[9]。对革兰氏阳性菌或嗜盐嗜碱芽孢杆菌中AsV的呼吸还原,其最初的研究对象是Bacillus selenitireducens[16]。B. selenitireducens和C. arsenatis的AsV呼吸还原酶的差异在于,前者位于细胞周质中,而后者定位于细胞周质的膜上(图1-A)。此外,前者的还原酶分子量比后者大,它是由110 kDa的ArrA大亚基和34 kDa的ArrB小亚基构成的。另一种研究较早的AsV呼吸还原微生物是希瓦氏菌属细菌(Shewanella sp. ANA-3)[17]。该菌的大亚基ArrA(95 kDa)也与DMSO还原酶很相似,小亚基ArrB的分子量是25.7 kDa;进一步研究表明,只当ArrA和ArrB共同表达时,才有AsV还原酶活性,每一个Arr由一个Mo原子,一个含4个S的蝶呤型辅因子和4-5个[4Fe-4S]组成[18]。有趣的是,ANA-3不仅能进行ArrA的呼吸还原,还能发挥ArsC的还原作用。研究表明,在厌氧条件下,该菌的ArrA转录表达可被浓度低至nM的AsV或者AsⅢ激活;在生长的早期阶段,ANA-3倾向于利用AsV还原过程产生的能量进行生长,但AsⅢ的累积会诱导ars操纵子的表达以增强微生物的砷抗性[17]。
近来研究表明,Desulfurispirillum indicum[19]、Denitrovibrio acetiphilus[20]和盐碱湖生菌(Nitrincola sp. DAR-403)[21]等微生物中也存在ArrA还原酶。此外,Fisher和Hollibaugh[22]发现,莫诺湖(Mono lake)存在兼具AsV呼吸还原和AsⅢ氧化功能的微生物,该菌的16S rRNA序列与B. selenitireducens亲缘关系最近。类似的现象也存在于某些光合自养微生物中[23]。到目前为止,共有30多种存在arrA基因的细菌得到了详细研究[24]。虽然含有arrA基因的微生物多在厌氧环境如湖底及沉积物中被检测到,Jia等[4]和Zhang等[5]在水稻根际土中克隆到了该基因,说明arrA基因的表达可能并非严格厌氧。
3 AsⅢ的氧化机理 3.1 AroA氧化酶介导的AsⅢ氧化 自然界中广泛存在AsⅢ氧化微生物,主要包括异养微生物和化能/光合自养微生物。目前研究最透彻的AsⅢ氧化酶是AroAB(即AoxAB或AsoAB)[25],该酶定位于细胞周质中(图1-A)。以粪产碱杆菌(Alcaligenes faecalis)为例,其AroAB分子量为100 kDa,大催化亚基AroA有825个氨基酸残基,该残基包裹着Mo核,Mo核与2个蝶呤辅因子和1个[3Fe-4S]簇结合,其结构与钼酶家族的DMSO还原酶和Arr还原酶有相似之处。小亚基AroB约含134个残基,其中Rieske型[2Fe-2S]簇与cytb复合物的Rieske蛋白结构域及双加氧酶相似[9]。
根据AsⅢ代谢方式不同,砷氧化微生物可分为2类:AsⅢ氧化供能进行化能自养生长(CAOs)及单纯氧化解毒(HAOs)以提高砷抗性,前者编码的砷氧化酶基因为aro,而后者为aox或者aso[25]。根据电子供体/受体的差异,这些微生物又可被分为4类:一类以O2为电子受体,AsⅢ为电子供体,并以氧化过程产生的能量固定CO2进行生长;另一类主要在厌氧环境下以NO3-、SO42-和ClO3-等为电子受体,以AsⅢ为电子供体进行产能生长。例如,Santini等[26]从金矿筛选到9种微生物,其中4种能够以O2为电子受体,以AsⅢ为电子供体并以含CO2的重碳酸盐为唯一碳源进行化能生长;另外5种属于单纯的砷氧化解毒微生物,分别属于f-Proteobacteria和g-Proteobacteria。作为典型的土壤微生物,农杆菌(Agrobacterium tumefaciens)中的AsⅢ氧化酶基因也得到了深入的研究[27]。有趣的是,该菌的AsⅢ氧化酶基因对AsⅢ的化学相似物SbⅢ也有氧化功能[28];第三类是依赖于Se的厌氧氧化微生物,如Bacillus sp. ML-SRAO[22]。该菌能以SeV或AsV为电子受体进行AsⅢ氧化代谢生长,且存在ArrA呼吸还原酶(见3.2节)。除以上3类AsⅢ的化能自养代谢外,Hoeft等[29]还发现了同时具备在好氧和厌氧条件下AsⅢ氧化功能的微生物Alkalilimnicola ehrlichii MHLE-1T,该菌可在好氧条件下以O2为电子受体进行异养生长,且能以NO3-为电子受体进行化能自养生长。在化能自养生长过程中,MHLE-1T可在核酮糖1,5二磷酸羧化酶作用下通过卡尔文循环实现对无机碳的固定。然而,aroA基因并未在MHLE-1T中被检测到,而是与arrA还原酶基因很相似[30-31]。因此,MHLE-1T中的AsⅢ氧化酶基因可能是一种与arrA高度相似的新型基因,或者表明ArrA酶具有双重功能(详见下文)。
此外,常见的砷氧化菌还有嗜热泉生古细菌(Aeropyrum pernix)、超嗜热古菌(Sulfolobus tokodaii)和Chloroflexus aurantiacus等[24],这些细菌在加利福利亚的莫诺湖等湖底的泉口大量存在,说明砷氧化还原细菌在砷的地球化学循环过程中起着重要作用[8]。
3.2 ArxA 氧化酶介导的 AsⅢ 氧化 如前所述,A. ehrlichii MHLE-1T存在2种AsⅢ氧化功能,但基因组分析显示,该菌缺失了传统的AsⅢ氧化酶基因(aox),却存在2个编码类arr还原酶基因的操纵子[31]。早期的研究还发现,MHLE-1T虽然有arrA-like和arsC基因,却不能还原溶液中的AsV,也不能降低其浓度[29]。类似的情况也发生在A. ehrlichii PHS-1和Ectothiorhodospira shaposhnikovii HS-1中[23]。Zargar等[30]对MHLE-1T的mlg_0216和mlg_2426基因进行了深入研究,前者被证实与AsⅢ的氧化有关系,后者的功能有待进一步研究(但确定与AsⅢ氧化无关)。为此,作者将此基因命名为arxA,其编码的AsⅢ氧化酶ArxA只能在化能自养条件下与NO3-的还原共同发挥作用。类似地,Zargar等[32]也在PHS-1中发现了arxA的存在,该基因的表达受到AsⅢ的严格调控。序列分析显示,PHS-1和MHLE-1T的arxA(及其上下游)基因非常相似。
Richey等[31]还发现,A. oremlandii和Shewanella sp. ANA-3也存在ArxA氧化酶。事实上,ANA-3存在ArrA还原酶功能(2.2节),说明ArxA可能是1种氧化还原双功能酶(biochemically reversible enzyme)。Fisher和Hollibaugh[22]在B. selenitireducens ML-SRAO中发现了类似的现象——该菌同时具备AsV还原和AsⅢ氧化功能,但其序列与arrA只有部分相似。最近,Hamamura等[21]又在蒙古库苏古勒湖(Khovsgol)发现了1株包含与MHLE-1T的arxA基因序列相近的微生物Halomonas sp. ANAO-440。基于以上结论,那些具备AsⅢ氧化功能与类arrA基因片段的微生物所包含的氧化酶为ArxA,而非AroA;此外,有些微生物能够以此作为AsⅢ氧化和AsV还原的双向功能蛋白[31]。鉴于ArrA和AroA都属于DMSO家族还原酶,而ArsC属于通用型硫氰酸酶(rhodanase)家族成员,这3种酶的进化顺序可能是:ArsC→ArrA→AroA。近些年发现的ArxA型AsⅢ氧化酶则很可能在ArrA→AroA的进化中起过渡作用或者ArxA在ArsC之后出现并进一步进化成ArrA和AroA。然而,目前的研究仍无法回答以上的哪种模式是确实存在的,抑或兼而有之。结合MHLE-1T中存在mlg_2426基因但表达的蛋白并未表现出AsⅢ氧化酶功能的事实[30],arxA基因很有可能比arrA和aroA更早出现,但在进化过程中逐渐丢失了mlg_2426基因的功能,转而由mlg_0216主导AsⅢ的氧化调控。事实上,某些微生物和ANA-3甚至存在ArsC还原、ArrA还原和ArxA氧化的三重功能[17, 31],说明这几种酶的进化顺序很可能是:ArsC→ArrA→ArxA→AroA。
无论如何,以上研究都说明了微生物砷转化功能的多样性、普遍性和古老性,这也为砷地球化学循环的微生物作用及研究指明了方向。
4 AsⅢ的甲基/去甲基化、隔离及外排 人类在18世纪曾广泛使用含砷染料的墙纸,导致了多次砷中毒事件。之后的研究发现砷中毒可能与墙纸发出的大蒜味气体有关,该气体被意大利物理学家Bartolomeo Gosio确认为甲基砷[33]。进一步研究发现,一些真菌如大毛霉(Mucor mucedo [sic])和Penicillium brevicaule是产生甲基砷的“元凶”,此后灰绿曲霉(Aspergillus glaucus)、Aspergillus virens、分枝状毛霉(Mucor ramosus)、棉铃红粉病菌(Cephalothecium roseum)和Sterigmatocystis ochracea等具有砷甲基化功能的真菌相继被发现[34]。直到1935年,Challenger和Higginbottom开始研究真菌短帚霉(Scopulariopsis brevicaulis)的砷甲基化过程,它包括AsV→AsⅢ和AsⅢ→As(CH3)n的一系列还原和甲基化,即Challenger通路[35]。随着研究的深入与拓展,包括细菌、古菌、真菌、藻类、动物和人类在内的AsⅢ甲基化逐渐得到确认[1, 2, 36](图1)。
基因组测序表明,真菌中存在编码AsⅢ甲基转移酶(arsenite-S-adenosylmethyltransferase,ArsM)的基因,这些基因往往与其他编码砷解毒蛋白的基因毗连[36]。在甲基化过程中,S-腺苷甲硫氨酸(S-adenosylmethionine,SAM)充当甲基供体的角色,但具体的传递过程仍不清楚[37]。AsⅢ甲基化的机理研究最早以哺乳动物为对象,目前已报道的类似于哺乳动物(包括哺乳动物)甲基化酶AS3MT的序列超过250条,其中细菌>160条,古菌>25条,真菌>17条(NCBI数据库)。以沼泽红假单胞菌(Rhodopseudomonas palustris)为例,其arsM基因的表达使大肠杆菌表现出了明显的砷抗性;在GSH和SAM存在的情况下,经纯化的ArsM能将AsⅢ转化为毒性小100倍的二甲基砷(dimethylarsinic acid,DMAV)和小1000倍的二甲基砷氧化物(DMAO),但不存在毒性更强的一甲基砷(monomethylarsonic acid,MMAsⅢ或MMAsV)[1, 9, 37]。因此,微生物介导的砷甲基化机理及其对砷地球化学循环的影响有待进一步研究。
AsⅢ在真菌中的解毒不仅仅是甲基化,还能在钙调因子Ycf1p(类似于人体耐药相关蛋白MRP1的1种ATP酶)催化下以AsⅢ-GSH形式被泵入液泡中隔离[38]。作为1种重金属解毒的重要诱导肽,植物螯合蛋白(phytochelatins,PCs)在植物和一些真菌中广泛存在[39]。早期研究表明,粟酒裂殖酵母(Schizosaccharomyces pombe)的PCs在Cd2+、Cu2+和Zn2+等重金属解毒中发挥了重要作用[39]。实际上,真菌的PCs对重金属的解毒存在普遍意义,包括对AsⅢ的解毒[11]。AsⅢ经PCs络合成As-PCs复合物后,在ABC转液泡膜蛋白HMT1作用下被隔离于液泡中,并与硫形成稳定的As-PCs-S复合物(图1-B)[11]。不同的是,细菌并不存在砷的细胞内隔离,而以砷的能量代谢、甲基化和外排为主(图1-A)。AsⅢ的外排在细菌中由ArsB(AsⅢ:H+-抑制剂)或ArsA/B(AsⅢ ATP酶,ArsA有加强作用)介导完成,而真菌由Acr3p介导完成(图1)[9-11]。有趣的是,Maciaszczyk-Dziubinsk等[38]研究表明,在酿酒酵母(Saccharomyces cerevisiae)中,AsⅢ的外排不是由Acr3p单独完成的,AsⅢ的吸收通道蛋白Fps1p也参与其中(图1),说明Fps1p是一种运输AsⅢ的双向通道蛋白。
水生微生物对砷的甲基化也是水体砷循环的重要推动者[41]。早在2001年,Geiszinger等[42]就发现齿缘墨角藻(Fucus serratus)能在100 μg/L的AsV溶液中生存高达19周,其产物包括少量的AsⅢ和MMAV,以及大量的DMAV和砷糖(arsenic sugar,AsS)。之后,Lehr等[43]报道了生活于美国黄石公园热泉中的嗜热砷甲基化红藻,从而揭示了砷甲基化微生物在进化史上的重要性。值得关注的是,有些微生物不存在arsM基因,却能将甲基砷去甲基化(图1)。例如,分歧杆菌(Mycobacterium neoaurum)可将2种类型的甲基胂酸(MMAsⅢ和MMAsV)去甲基化,产生AsⅢ(43%)和AsV (27%)[44]。有研究表明,微生物介导的砷去甲基化可能存在不同微生物间的协同作用[45]。例如,伯克霍尔德氏菌属(Burkholderia sp. MR1)和恶臭假单胞菌(Pseudomonas puditaKT2440)可吸收MMAsV并将其还原成毒性更强的MMAsⅢ。MMAsⅢ被Burkholderia排出体外后又被链霉菌(Streptomyces sp. MD1)吸收,完成去甲基化后以AsⅢ形式被排出细胞[45]。由此可见,环境中砷的甲基化和去甲基化是一个动态过程,且去甲基化可能需要不同微生物通过2步完成。
5 总结和展望 微生物砷代谢是一种古老的耐砷机制,这与环境中砷的广泛存在有关。虽然环境中的砷总量不会减少,微生物介导的砷转化会显著地改变砷的环境归趋与生物有效性,这种特性是生物界普遍存在的一种对环境的重塑(remodel)过程[1]。现有研究证实,微生物对砷的耐受并非通过阻断其进入细胞内,而是通过吸收、转化、隔离和外排等一系列过程实现砷解毒[9]。此外,某些微生物还可在砷的转化过程中获得能量用于生长与繁殖[23, 36]。到目前为止,大量砷氧化、还原和甲基化微生物得到了分离与研究,但环境中存在大量未可培养微生物,因其对微生物介导的砷转化路径也存在很多认识上的不足。今后的研究必须克服砷解毒功能基因引物覆盖面不全的问题,同时,有必要通过高通量测序技术或者单细胞测序技术等挖掘出更多的基因信息,从整体(环境角度)到局部(甚至是单个细胞)认识微生物抗砷基因的多样性及其差异。
鉴于砷对人体健康存在致癌风险,如何更有效地实现水体和土壤砷修复仍是我国环境保护中必须重点关注的问题。微生物是土壤砷转化的重要推动者,围绕基因工程手段构建出耐砷微生物并将其应用于土壤修复是今后的研究重点,这方面的工作目前已初见成效[46-47]。另一方面,微生物介导的砷还原会提高砷的生物有效性,进而对人类生产、生活带来不利影响,例如地下水砷污染[6];未来的研究可围绕兼性厌氧氧化微生物在这方面的应用,以期在抑制AsV还原的同时结合物理化学吸附法提高砷的去除效率。在农作物砷阻控方面,砷氧化和甲基化微生物活动可直接或间接“钝化”砷,例如通过氧化作用提高水稻根表铁膜对砷的吸附以降低作物砷富集或通过甲基化作用降低土壤砷浓度,进而保障食品安全[4-5]。综上所述,今后的研究必须进一步挖掘微生物耐砷性机制,并将其与环境污染修复、食品安全和人体健康关联起来,为揭示微生物-砷地球化学循环的具体过程及其在砷污染阻控和降低人体砷暴露风险中的应用提供理论与技术参考。
参考文献
| [1] | Zhu YG, Yoshinaga M, Zhao FJ, Rosen BP. Earth abides arsenic biotransformations. Annual Review of Earth and Planetary Sciences, 2014, 42(1): 443-467. |
| [2] | Bowell RJ, Alpers CN, Jamieson HE, Nordstrom DK, Majzlan J. The environmental geochemistry of arsenic—an overview. Reviews in Mineralogy and Geochemistry, 2014, 79(1): 1-16. |
| [3] | Zhao FJ, Ma YB, Zhu YG, Tang Z, McGrath SP. Soil contamination in China: current status and mitigation strategies. Environmental Science & Technology, 2015, 49(2): 750-759. |
| [4] | Jia Y, Huang H, Chen Z, Zhu YG. Arsenic uptake by rice is influenced by microbe-mediated arsenic redox changes in the rhizosphere. Environmental Science & Technology, 2014, 48(2): 1001-1007. |
| [5] | Zhang SY, Zhao FJ, Sun GX, Su JQ, Yang XR, Li H, Zhu YG. Diversity and abundance of arsenic biotransformation genes in paddy soils from Southern China. Environmental Science & Technology, 2015, 49(7): 4138-4146. |
| [6] | Osborne TH, McArthur JM, Sikdar PK, Santini JM. Isolation of an arsenate-respiring bacterium from a redox front in an arsenic-polluted aquifer in West Bengal, Bengal Basin. Environmental Science & Technology, 2015, 49(7): 4193-4199. |
| [7] | Sforna MC, Philippot P, Somogyi A, van Zuilen MA, Medjoubi K, Schoepp-Cothenet B, Nitschke W, Visscher PT. Evidence for arsenic metabolism and cycling by microorganisms 2. 7 billion years ago. Nature Geoscience, 2014, 7(11): 811-815. |
| [8] | Oremland RS, Stolz JF. The ecology of arsenic. Science, 2003, 300(5621): 939-944. |
| [9] | Bhattacharjee H, Rosen BP. Arsenic metabolism in prokaryotic and eukaryotic microbes//Nies DH, Silver S. Molecular Microbiology of Heavy Metals. Berlin, Germany: Springer-Verlag, 2007. |
| [10] | Medveczky N, Rosenberg H. Phosphate transport in Escherichia coli. Biochimica et Biophysica Acta (BBA)-Biomembranes, 1971, 241(2): 494-506. |
| [11] | Tsai SL, Singh S, Chen W. Arsenic metabolism by microbes in nature and the impact on arsenic remediation. Current Opinion in Biotechnology, 2009, 20(6): 659-667. |
| [12] | Sanders OI, Rensing C, Kuroda M, Mitra B, Rosen BP. Antimonite is accumulated by the glycerol facilitator GlpF in Escherichia coli. Journal of Bacteriology, 1997, 179(10): 3365-3367. |
| [13] | Patel PC, Goulhen F, Boothman C, Gault AG, Charnock JM, Kalia K, Lloyd JR. Arsenate detoxification in a Pseudomonad hypertolerant to arsenic. Archives of Microbiology, 2007, 187(3): 171-183. |
| [14] | Mukhopadhyay R, Rosen BP. Arsenate reductases in prokaryotes and eukaryotes. Environmental Health Perspectives, 2002, 110(S5): 745-748. |
| [15] | Krafft T, Macy JM. Purification and characterization of the respiratory arsenate reductase of Chrysiogenes arsenatis. European Journal of Biochemistry, 1998, 255(3): 647-653. |
| [16] | Afkar E, Lisak J, Saltikov C, Basu P, Oremland RS, Stolz JF. The respiratory arsenate reductase from Bacillus selenitireducens strain MLS10. FEMS Microbiology Letters, 2003, 226(1): 107-112. |
| [17] | Saltikov CW, Newman DK. Genetic identification of a respiratory arsenate reductase. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(19): 10983-10988. |
| [18] | Malasarn D, Keeffe JR, Newman DK. Characterization of the arsenate respiratory reductase from Shewanella sp. strain ANA-3. Journal of Bacteriology, 2008, 190(1): 135-142. |
| [19] | Rauschenbach I, Yee N, H?ggblom MM, Bini E. Energy metabolism and multiple respiratory pathways revealed by genome sequencing of Desulfurispirillum indicum strain S5. Environmental Microbiology, 2011, 13(6): 1611-1621. |
| [20] | Denton K, Atkinson MM, Borenstein SP, Carlson A, Carroll T, Cullity K, DeMarsico C, Ellowitz D, Gialtouridis A, Gore R. Identification of a possible respiratory arsenate reductase in Denitrovibrio acetiphilus, a member of the phylum Deferribacteres. Archives of Microbiology, 2013, 195(9): 661-670. |
| [21] | Hamamura N, Itai T, Liu YT, Reysenbach AL, Damdinsuren N, Inskeep WP. Identification of anaerobic arsenite-oxidizing and arsenate-reducing bacteria associated with an alkaline saline lake in Khovsgol, Mongolia. Environmental Microbiology Reports, 2014, 6(5): 476-482. |
| [22] | Fisher JC, Hollibaugh JT. Selenate-dependent anaerobic arsenite oxidation by a bacterium from Mono Lake, California. Applied and Environmental Microbiology, 2008, 74(9): 2588-2594. |
| [23] | Kulp TR, Hoeft SE, Asao M, Madigan MT, Hollibaugh JT, Fisher JC, Stolz JF, Culbertson CW, Miller LG, Oremland RS. Arsenic (Ⅲ) fuels anoxygenic photosynthesis in hot spring biofilms from Mono Lake, California. Science, 2008, 321(5891): 967-970. |
| [24] | Stolz JF, Basu P, Oremland RS. Microbial arsenic metabolism: new twists on an old poison. Microbe, 2010, 5(2): 53-59. |
| [25] | Sultana M, Vogler S, Zargar K, Schmidt AC, Saltikov C, Seifert J, Schl?mann M. New clusters of arsenite oxidase and unusual bacterial groups in enrichments from arsenic-contaminated soil. Archives of Microbiology, 2012, 194(7): 623-635. |
| [26] | Santini JM, Sly LI, Wen AM, Comrie D, de Wulf-Durand P, Macy JM. New arsenite-oxidizing bacteria isolated from Australian gold mining environments-phylogenetic relationships. Geomicrobiology Journal, 2002, 19(1): 67-76. |
| [27] | Wang Q, Qin D, Zhang SZ, Wang L, Li JX, Rensing C, McDermott TR, Wang GJ. Fate of arsenate following arsenite oxidation in Agrobacterium tumefaciens GW4. Environmental Microbiology, 2015, 17(6): 1926-1940. |
| [28] | Li JX, Wang Q, Li MS, Yang BR, Shi MM, Guo W, McDermott TR, Rensing C, Wang GJ. Proteomics and genetics for identification of a bacterial antimonite oxidase in Agrobacterium tumefaciens. Environmental Science & Technology, 2015, 49(10): 5980-5989. |
| [29] | Hoeft SE, Blum JS, Stolz JF, Tabita FR, Witte B, King GM, Santini JM, Oremland RS. Alkalilimnicola ehrlichii sp. nov., a novel,arsenite-oxidizing haloalkaliphilic gammaproteobacterium capable of chemoautotrophic or heterotrophic growth with nitrate or oxygen as the electron acceptor. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(3): 504-512. |
| [30] | Zargar K, Hoeft S, Oremland R, Saltikov CW. Identification of a novel arsenite oxidase gene, arxA, in the haloalkaliphilic, arsenite-oxidizing bacterium Alkalilimnicola ehrlichii strain MLHE-1. Journal of Bacteriology, 2010, 192(14): 3755-3762. |
| [31] | Richey C, Chovanec P, Hoeft SE, Oremland RS, Basu P, Stolz JF. Respiratory arsenate reductase as a bidirectional enzyme. Biochemical and Biophysical Research Communications, 2009, 382(2): 298-302. |
| [32] | Zargar K, Conrad A, Bernick DL, Lowe TM, Stolc V, Hoeft S, Oremland RS, Stolz J, Saltikov CW. ArxA, a new clade of arsenite oxidase within the DMSO reductase family of molybdenum oxidoreductases. Environmental Microbiology, 2012, 14(7): 1635-1645. |
| [33] | Gosio B. Action of microphytes on solid compounds of arsenic: a recapitulation. Science, 1892, 19(472): 104-106. |
| [34] | Wang PP, Sun GX, Jia Y, Meharg AA, Zhu YG. A review on completing arsenic biogeochemical cycle: microbial volatilization of arsines in environment. Journal of Environmental Sciences, 2014, 26(2): 371-381. |
| [35] | Challenger F. Biological methylation. Chemical Review, 1945, 36(3): 315-361. |
| [36] | Ye J, Rensing C, Rosen BP, Zhu YG. Arsenic biomethylation by photosynthetic organisms. Trends in Plant Science, 2012, 17(3): 155-162. |
| [37] | Qin J, Rosen BP, Zhang Y, Wang GJ, Franke S, Rensing C. Arsenic detoxification and evolution of trimethylarsine gas by a microbial arsenite S-adenosylmethionine methyltransferase. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(7): 2075-2080. |
| [38] | Ghosh M, Shen J, Rosen BP. Pathways of As (Ⅲ) detoxification in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(9): 5001-5006. |
| [39] | Ha SB, Smith AP, Howden R, Dietrich WM, Bugg S, O'Connell MJ, Goldsbrough PB, Cobbett CS. Phytochelatin synthase genes from Arabidopsis and the yeast Schizosaccharomyces pombe. The Plant Cell, 1999, 11(6): 1153-1163. |
| [40] | Maciaszczyk-Dziubinsk E, Migdal I, Migocka M, Bocer T, Wysocki R. The yeast aquaglyceroporin Fps1p is a bidirectional arsenite channel. FEBS Letters, 2010, 584(4): 726-732. |
| [41] | Maguffin SC, Kirk MF, Daigle AR, Hinkle SR, Jin QS. Substantial contribution of biomethylation to aquifer arsenic cycling. Nature Geoscience, 2015, 8(4): 290-293. |
| [42] | Geiszinger A, Goessler W, Pedersen SN, Francesconi KA. Arsenic biotransformation by the brown macroalga Fucus serratus. Environmental Toxicology and Chemistry, 2001, 20(10): 2255-2262. |
| [43] | Lehr CR, Frank SD, Norris TB, D'Imperio S, Kalinin AV, Toplin JA, Castenholz RW, McDermott TR. Cyanidia (Cyanidiales) population diversity and dynamics in an acid-sulfate-chloride spring in Yellowstone National Park. Journal of Phycology, 2007, 43(1): 3-14. |
| [44] | Lehr CR, Polishchuk E, Radoja U, Cullen WR. Demethylation of methylarsenic species by Mycobacterium neoaurum. Applied Organometallic Chemistry, 2003, 17(11): 831-834. |
| [45] | Yoshinaga M, Cai Y, Rosen BP. Demethylation of methylarsonic acid by a microbial community. Environmental Microbiology, 2011, 13(5): 1205-1215. |
| [46] | Visioli G, D'Egidio S, Sanangelantoni AM. The bacterial rhizobiome of hyperaccumulators: future perspectives based on omics analysis and advanced microscopy. Frontiers in Plant Science, 2015, 5: 752. |
| [47] | Chen J, Sun GX, Wang XX, de Lorenzo V, Rosen BP, Zhu YG. Volatilization of arsenic from polluted soil by Pseudomonas putida engineered for expression of the arsM arsenic (Ⅲ) S-adenosine methyltransferase gene. Environmental Science & Technology, 2014, 48(17): 10337-10344. |
