 , 徐慧敏1,2, 苏芮1, 沈华东1, 何肖微1,3, 曾巾2, 余钟波1, 赵大勇1,3
, 徐慧敏1,2, 苏芮1, 沈华东1, 何肖微1,3, 曾巾2, 余钟波1, 赵大勇1,3

1. 河海大学水文水资源与水利工程科学国家重点实验室, 全球变化与水循环国际合作联合实验室, 南京 210098;
2. 中国科学院南京地理与湖泊研究所湖泊与环境国家重点实验室, 南京 210008;
3. 河海大学水利学科专业实验教学中心, 南京 210098
收稿日期: 2019-06-25; 修回日期: 2019-08-05; 录用日期: 2019-08-05
基金项目: 国家重点研发计划(No.2016YFC0402710);国家自然科学基金(No.31730013,41671078)
作者简介: 丁浩(1994-), 男, E-mail:ding-hao@hhu.edu.cn
通讯作者(责任作者): 赵大勇, E-mail:dyzhao@hhu.edu.cn
摘要: 若尔盖湿地作为中国最大的泥炭沼泽区,是生物地球化学循环的重要场所.本文以若尔盖湿地的花湖为研究对象,采集0~47 cm的沉积物样品,通过实时荧光定量PCR(qPCR)技术,探究沉积物中氨氧化(amoA)和反硝化(nirS、nirK、nosZ clade I)功能基因丰度的垂向分布特征,及其对环境因子的响应.结果表明:花湖沉积物中古菌amoA基因丰度在垂向分布上呈下降趋势,而nirS基因丰度呈上升趋势;古菌和细菌的amoA基因丰度相近,nirS基因丰度则远高于nirK基因,且氨氧化功能基因丰度整体上比反硝化功能基因低1~2个数量级.总氮(TN)、总磷(TP)、氨态氮(NH4+-N)、硝态氮(NO3--N)和亚硝态氮(NO2--N)与古菌amoA基因丰度均呈显著正相关关系(p < 0.05),而与nirS基因丰度呈显著负相关关系(p < 0.05).这两种功能基因明显受到花湖沉积物中不同形式氮素浓度的影响与限制.通过研究花湖沉积物氨氧化与反硝化功能基因的垂向分布特征及其对环境的响应,可为深入了解高原湖泊沉积物中的氮循环机理提供参考.
关键词:花湖沉积物氨氧化反硝化垂向分布环境响应qPCR
Vertical distribution and environmental response of the abundance of ammonia-oxidizing and denitrifying functional genes in sediments of Huahu Lake in Zoige
DING Hao1
 , XU Huimin1,2, SU Rui1, SHEN Huadong1, HE Xiaowei1,3, ZENG Jin2, YU Zhongbo1, ZHAO Dayong1,3
, XU Huimin1,2, SU Rui1, SHEN Huadong1, HE Xiaowei1,3, ZENG Jin2, YU Zhongbo1, ZHAO Dayong1,3

1. State Key Laboratory of Hydrology-Water Resources and Hydraulic Engineering, Joint International Research Laboratory of Global Change and Water Cycle, Hohai University, Nanjing 210098;
2. State Key Laboratory of Lake Science and Environment, Nanjing Institute of Geography and Limnology, Chinese Academy of Sciences, Nanjing 210008;
3. The Experimental Teaching Center of Water Resources of Hohai University, Hohai University, Nanjing 210098
Received 25 June 2019; received in revised from 5 August 2019; accepted 5 August 2019
Abstract: As the largest peat bog in China,the Zoige wetland is an important place for biogeochemical cycles. In this study,sediment samples at depth of 0~47 cm were collected in Huahu Lake located in the Zoige wetland. The vertical distribution of abundance of ammonia-oxidizing (amoA) and denitrifying (nirS,nirK,nosZ clade I) functional genes in sediments were explored by real-time fluorescence quantitative PCR (qPCR) technology. The mechanisms underlying their responses to environmental factors were also studied here. The results showed that the abundance of archaea-amoA gene declined with the depth,whereas the abundance of nirS gene increased. The abundance of archaea-amoA gene kept similar level as bacteria-amoA gene,but the abundance of nirS gene was much higher than that of nirK gene. Generally,the abundance of ammonia-oxidizing functional gene was 1~2 orders of magnitude lower than that of denitrifying functional gene. The abundance of archaea-amoA gene showed significant positive correlations (p < 0.05) with total nitrogen (TN),total phosphorus (TP),ammonia-nitrogen (NH4+-N),nitrate-nitrogen (NO3--N),and nitrite-nitrogen (NO2--N),whereas the abundance of nirS gene was significantly negatively correlated with these environmental factors (p < 0.05). Therefore,the two functional genes were obviously affected and restricted by the concentration of different forms of nitrogen in sediments of Huahu Lake. It can provide reference for further understanding the nitrogen cycles in sediments of plateau lakes by studying the vertical distribution characteristics of ammonia-oxidizing and denitrifying functional genes in sediments of Huahu Lake and their responses to the environment.
Keywords: Huahu Lakesedimentammonia-oxidationdenitrificationvertical distributionenvironmental responseqPCR
1 引言(Introduction)随着人类活动的频繁干扰, 全球氮循环严重失衡, 引发了温室气体排放、水体富营养化与酸化等一系列环境问题(Dag et al., 1997).作为氮循环的中心环节(Prosser, 1990), 氨氧化作用可以将NH4+氧化为NO2-;反硝化作用则是氮循环的枢纽(王洋等, 2006), 能将NO3-和NO2-逐步还原为N2.这两个过程是降低活性氮, 维护氮循环平衡的重要环节, 也一直是氮循环研究中的热点.氨氧化作用主要由氨氧化古菌(AOA)和氨氧化细菌(AOB)中的功能基因氨单加氧酶(amoA)完成(Beman et al., 2006; Kuypers et al., 2018).而将NO2-还原为NO则是反硝化作用的标志性过程(Braker et al., 2011), 这一反应主要由亚硝酸还原酶nirS和nirK执行(Ullah et al., 2011).氧化亚氮还原酶(nosZ clade I, 以下简称nosZI)能够执行反硝化作用的最后一步反应, 将N2O转化为N2, 这是减少温室气体N2O的主要途径(Kuypers et al., 2018).
湖泊作为生态系统中的一个“汇”(Kalff, 2002), 是氮循环过程的重要场所(王洋等, 2006).一部分汇入湖泊水体中的物质, 会以沉积物为载体进行迁移和转化(李志建, 2011), 并对其中的微生物产生影响.花湖是若尔盖高寒湿地的第二大湖泊, 该地区是中国最大的泥炭沼泽区(费世民等, 2006), 且位于生物地球化学循环的重要场所——青藏高原.若尔盖湿地曾受环境变化影响(Li et al., 2014)和人类活动干扰(Chen et al., 2014; Shen et al., 2016)而发生退化, 后又经过保护修复(Li et al., 2016), 因而生态系统发生过较大变化, 具有较高的研究价值.目前对该地区元素循环的研究主要集中在碳元素上(Shen et al., 2016), 关注于氮循环的研究较为稀少.已有研究表明, 氮元素的平衡状况会影响到碳循环的稳定性(王绍强等, 2008), 因此在该区域开展氮循环(如氨氧化和反硝化作用)相关研究具有重要意义.与此同时, 有研究报道, 湖泊沉积物中氮、磷等营养元素及微生物群落组成(赵兴青等, 2008)均呈现一定的垂向分布特征, 但是功能微生物是否存在垂向分布特征仍不够清楚.因此, 通过研究湖泊沉积物中氨氧化和反硝化功能基因与环境因子的垂向分布特征关系, 可以完善对于湖泊沉积物不同深度中生物地球化学循环过程的理解.
本研究通过实时荧光定量PCR(qPCR)技术, 以参与氨氧化作用和反硝化作用的关键功能基因(amoA、nirS、nirK、nosZI)为标记, 分析其在花湖沉积物中的垂向分布特征.并结合沉积物理化性质, 分析研究氨氧化与反硝化功能基因对环境因子的响应, 以期为深入了解高原湖泊沉积物中的氮循环机理提供参考.
2 材料与方法(Materials and methods)2.1 研究区域与样品采集本研究的沉积物样品来自位于若尔盖湿地的花湖(102.48°E, 33.55°N), 海拔约3434 m.2015年7月, 使用重力采样器采集沉积物样柱, 样柱长47 cm.现场切分沉积物样柱, 每1 cm厚度分为一个样品, 将其放入无菌袋中密封保存, 共47个样品.分装后的样品置于保温箱冷藏, 运回实验室后放在-20 ℃冷冻保存.
2.2 理化指标测定使用冷冻干燥机(ALPHA 1-2, CHRIST, 德国)将沉积物样品进行冻干处理.干燥至恒重后, 将样品磨细, 保存在-20 ℃条件下, 留待后续实验使用.本研究测定的理化指标为pH、烧失量(LOI)、总磷(TP)、总氮(TN)、氨态氮(NH4+-N)、硝态氮(NO3--N)和亚硝态氮(NO2--N), 其中测LOI、TN和TP时每份样品设置3组平行.
取约5 g沉积物样品, 用0.5 mol · L-1 KCl溶液浸提, 振荡混匀后在浸提液呈混浊状态下使用pH计(PHB-4, 上海雷磁)测定pH;过滤浸提液, 取滤液, 使用连续流动分析仪(San++, SKALER, 荷兰)测定NH4+-N、NO3--N和NO2--N浓度.LOI通过马弗炉550 ℃煅烧前后的质量差计算得到(朱广伟等, 2004), 用来表征样品的有机质含量.测总磷和总氮需先将样品进行消解, 然后根据Rice等的方法进行测定(Rice et al., 2012).
2.3 DNA提取与qPCR扩增结合测得的理化指标数据, 选取沉积物中深度为0~6、7~10、12~13、15~16、18~19、21~22、25~26、29~30、33~34、37~38、40~41、44~45和46~47 cm共20组样品进行DNA提取, 用于后续功能基因分析.称取0.25 g沉积物样品, 使用PowerSoil DNA提取试剂盒(MoBio Laboratory, Solana Beach, CA)进行DNA提取和纯化(Wu et al., 2006).
将提取的DNA分别对细菌16S rRNA、古菌16S rRNA、amoA、nirS、nirK和nosZI基因进行PCR扩增, 各类基因所使用的引物信息和退火温度见表 1.qPCR扩增体系均为20 μL:10 μL TB Green(Takara, 日本), 7.4 μL ddH2O, 正、反向引物各0.8 μL, 1 μL DNA模板(样品)或已知浓度质粒(标曲).每份样品都设置两组平行, 并设阴性对照组.体系配好并离心后, 使用PCR仪IQ5 Thermocycler(RG65HD, Corbett, 澳大利亚)进行定量PCR扩增.扩增反应程序为:①95 ℃预变性5 min; ②95 ℃变性, 退火, 72 ℃延伸, 共循环45次;③72 ℃延伸10 min, 4 ℃保存.根据测得的质粒DNA浓度, 通过公式(1)(Whelan et al., 2003)换算得到基因拷贝数.扩增结果的标准曲线需要具有可行度和线性关系(回归方程可决系数R2> 0.99), 且扩增效率为0.92~1.05(胡万婷等, 2017).
表 1(Table 1)
| 表 1 本研究使用的qPCR信息 Table 1 Information of qPCR used in this study | ||||||||||||||||||||||||||||||||||||||||||||||||
表 1 本研究使用的qPCR信息 Table 1 Information of qPCR used in this study
| ||||||||||||||||||||||||||||||||||||||||||||||||
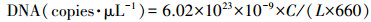 | (1) |
2.4 数据处理与分析使用软件SigmaPlot 12.5将沉积物理化指标及功能基因数据绘制成折线图.利用软件R 3.5.3中stats软件包的“hclust”函数, 针对理化因子数据进行聚类分析, 并制作聚类图.
选用双变量Spearman相关性分析法, 利用软件SPSS 22.0进行统计分析, 来研究沉积物氨氧化和反硝化功能基因对环境因子的响应.
3 结果与讨论(Results and discussion)3.1 沉积物理化因子垂向分布特征花湖沉积物pH为8.04~8.45(图 1a), 表明花湖沉积物呈弱碱性的特征, 整体上pH高于周边土壤(7.42 ± 0.40)(赵娜娜等, 2019).NO2--N含量为0.21~0.60 mg · kg-1(图 1c), 且0~8 cm处的NO2--N含量明显较高, 9 cm以下NO2--N含量呈现较为平稳且低含量的趋势;NO3--N含量变化范围为113.5~657.6 mg · kg-1(图 1d), 明显高于NO2--N和NH4+-N含量, 并且在0~8、9~24以及24~47 cm这3个不同的深度范围之间有明显差异, 整体呈垂向下降趋势;NH4+-N含量则在1.78~27.14 mg · kg-1之间变化(图 1e), 垂向变化趋势与NO2--N类似.各深度沉积物的LOI指标在7.0%~29.2%之间波动变化, 但能明显观测到30 cm以下的LOI指标呈现上升趋势(图 1b).TN含量的变化范围在2.03~4.13 g · kg-1, 其中0~9 cm和39~47 cm样品中的TN含量较为接近, 而9~39 cm样品中的TN含量则较低(图 1g).TP含量在0.07~0.19 g · kg-1之间波动, 在16 cm以上呈现垂向上升趋势, 而在16~34 cm之间呈下降趋势, 34 cm之下呈缓慢上升趋势(图 1f).花湖沉积物中TN、TP和有机质含量都低于湖泊附近土壤中(Gu et al., 2012; Wu et al., 2015; 青烨等, 2015).
图 1(Fig. 1)
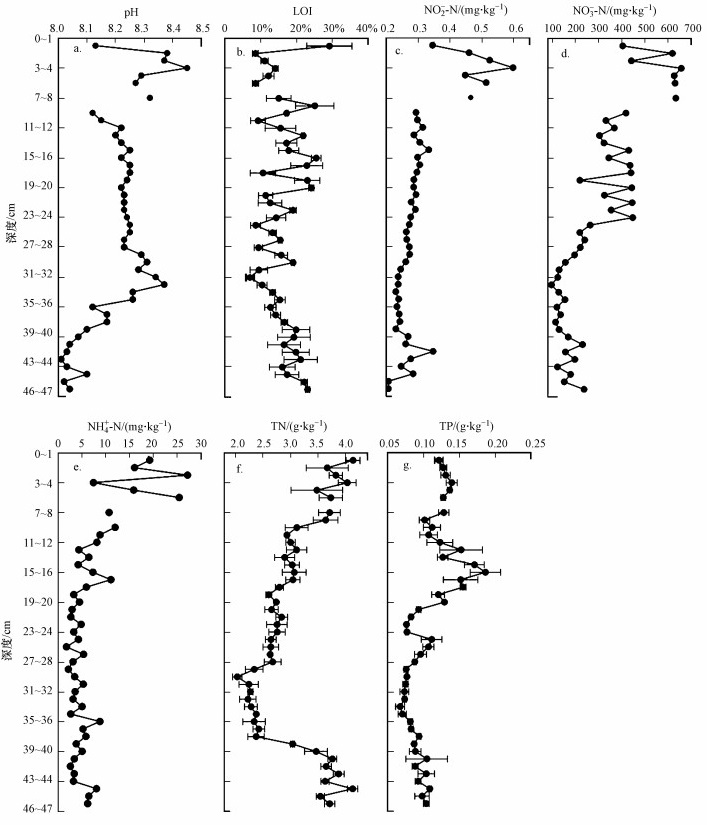 |
| 图 1 沉积物理化因子垂向分布 (TN:总氮, TP:总磷, NH4+-N:氨态氮, NO3--N:硝态氮, NO2--N:亚硝态氮, LOI:烧失量) Fig. 1Vertical distribution of physicochemical factors in sediments (TN: total nitrogen, TP: total phosphorus, NH4+-N: ammonia-nitrogen, NO3--N: nitrate-nitrogen, NO2--N: nitrite-nitrogen, LOI: loss on ignition) |
根据理化因子得到的聚类结果(图 2)显示, 花湖沉积物上层1~8 cm较为接近, 可归为一类;中层9~20 cm和20~38 cm可归为第2类;下层38~47 cm可归为第3类.这3类中, 中层与下层的聚类结果更为相近.结合沉积物理化指标的垂向变化趋势, 上层、中层和下层的NO3--N含量和pH值逐层递减;而NH4+-N和NO2--N含量在中层和下层数值相近, 明显低于上层.
图 2(Fig. 2)
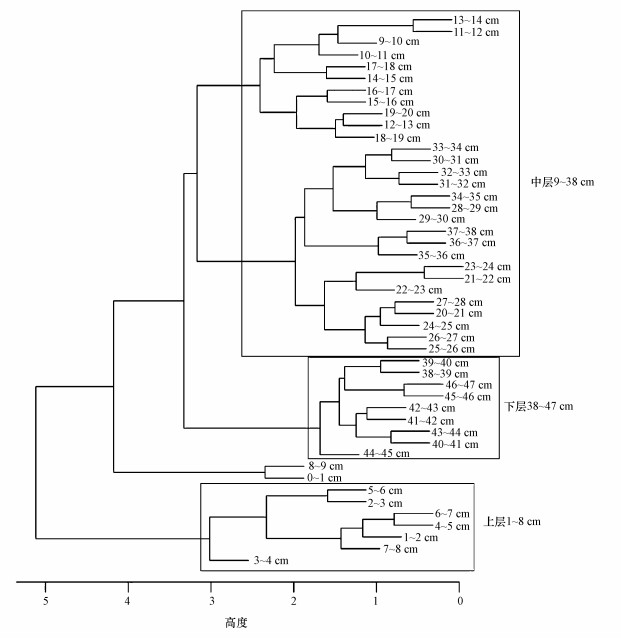 |
| 图 2 沉积物理化因子聚类分析 Fig. 2Cluster analysis of physicochemical factors in sediments |
3.2 氧化功能基因丰度垂向分布特征花湖沉积物冻干样品中, 古菌amoA基因丰度的均值为10.53 ×105 copies · g-1, 细菌amoA基因丰度均值为8.05 ×105 copies · g-1, 两者整体上基本持平(图 3a、3b).古菌和细菌amoA基因在上层沉积物中的丰度高于中层和下层, 而中层和下层中丰度相近.随着沉积物所处深度加深, 古菌amoA基因丰度呈现出降低的趋势, 且变化趋于平缓;而细菌amoA基因丰度升降趋势不够明显, 上层沉积物中有所波动, 中层和下层则比较平缓.在0~3 cm沉积物中, 古菌amoA基因丰度高过细菌amoA, 这个深度以下则是细菌amoA基因丰度更高.而从相对丰度来看, 古菌amoA则明显高于细菌amoA(图 3c、3d).
图 3(Fig. 3)
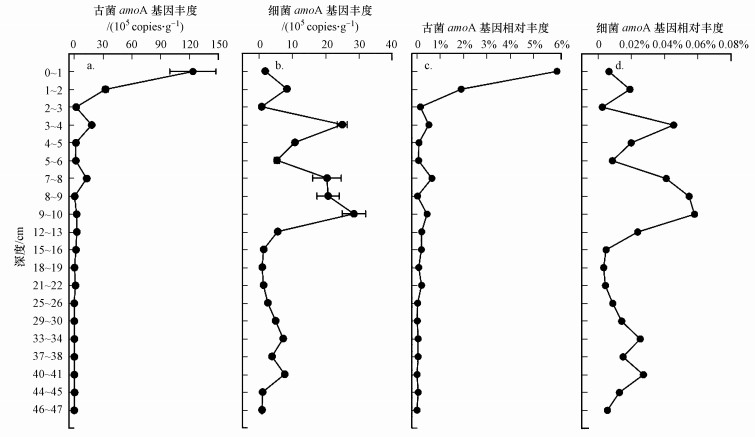 |
| 图 3 沉积物中氨氧化功能基因(amoA)丰度及其相对丰度(分别相对于古菌或细菌16S rRNA基因丰度)的垂向分布 Fig. 3The vertical distribution of abundance and relative abundance (divided by the abundance of archaeal/bacterial 16S rRNA gene, respectively) of the ammonia-oxidizing functional genes (amoA) in sediments |
尽管在许多环境中, 古菌amoA丰度都比细菌amoA高, 但也存在两者丰度相近, 甚至古菌amoA丰度更高的情况(贺纪正等, 2009; 2013; 杨柳燕等, 2016).这是因为多种环境因子都能直接影响到古菌和细菌中amoA基因的丰度(贺纪正等, 2009; 杨柳燕等, 2016).NO3--N和NO2--N含量过高会抑制氨氧化过程(杨柳燕等, 2016), 与该区域的湿地土壤相比(Gu et al., 2012), 花湖沉积物中NO3--N含量明显过高, 这或许是影响古菌和细菌中amoA基因丰度的重要原因.而NH4+-N含量对决定古菌amoA与细菌amoA基因丰度间的高低关系有重要作用(Di et al., 2009; Verhamme et al., 2011), AOA比AOB更能适应NH4+-N含量低的环境, 但当NH4+-N含量升高时, AOB的增长速率更快.而相对于周边的牧区湿地土壤(Zheng et al., 2014), 花湖沉积物中NH4+-N含量则较高一些.同时pH也会影响到古菌amoA与细菌中amoA间的丰度高低(杨柳燕等, 2016), 酸性条件下细菌amoA活性受到抑制, 而古菌amoA受到的影响却不大, 这或许是部分酸性环境中古菌amoA丰度高的原因.花湖沉积物是弱碱性环境, 在这个pH范围内, AOA和AOB都能正常生长, 一定程度上降低了AOA的优势.较高的NH4+-N含量和呈弱碱性的环境, 这或许是造成花湖沉积物中古菌amoA和细菌amoA丰度相近的原因.
3.3 反硝化功能基因丰度垂向分布特征与氨氧化功能基因相比, 反硝化功能基因丰度处在较高的水平, 这或许是部分因为弱碱性环境有利于反硝化微生物生长(张淑楠等, 2015).沉积物冻干样品中nirS基因丰度变化范围在1.75×107~29.22 ×107 copies · g-1之间, 整体上随深度加深呈上升趋势(图 4b).一般而言, 在一定范围内随着沉积物深度加深, 溶解氧会降低(杨旭楠等, 2013), 有研究表明nirS基因偏好低氧浓度的环境(Yuan et al., 2012; Gu et al., 2017), 这或许是花湖沉积物中nirS基因丰度垂向升高的原因.nirK基因丰度的垂向分布略有波动, nosZI基因丰度在中层略高于上层和下层, 但整体上两者随深度变化的趋势不是很明显, nirK基因丰度在3.32×105~25.60 ×105 copies · g-1之间(图 4a), nosZI则在0.10×107~5.64 ×107 copies · g-1之间变化(图 4c).从整体上看, 花湖沉积物中, 这3种参与反硝化作用的功能基因丰度在中层要高于上层和下层.nirS基因在细菌16S rRNA中的相对丰度随沉积物深度增加亦呈上升趋势(图 4e);nosZI基因的相对丰度变化趋势不明显(图 4f);而nirK除了46~47 cm沉积物中的相对丰度较高之外, 整体趋势也无明显变化(图 4d).
图 4(Fig. 4)
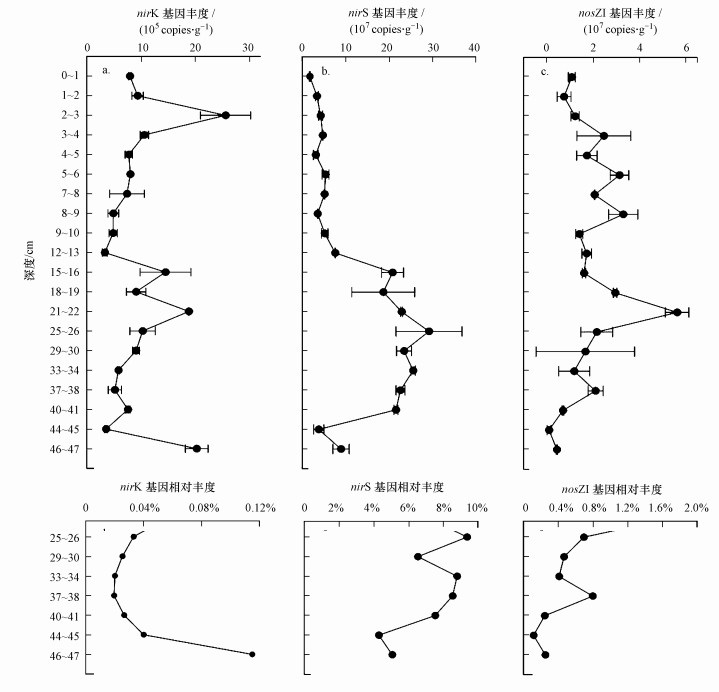 |
| 图 4 沉积物中反硝化功能基因(nirK、nirS、nosZI)丰度与相对丰度(相对于细菌16S rRNA基因丰度)的垂向分布 Fig. 4The vertical distribution of abundance and relative abundance (divided by the abundance of bacterial 16S rRNA gene) of the denitrifying functional genes (nirK、nirS、nosZI) in sediments |
有研究表明, 沉积物中nirS反硝化细菌比nirK反硝化细菌丰度高(Mosier et al., 2010), 本研究的结果跟此结论相符.可以推断, 花湖沉积物中携带nirS基因的细菌在将NO2-还原为NO的过程中占优势地位.不同生境中, nirS基因与nirK基因丰度的高低情况有所差异(杨亚东等, 2018), 因为这两种功能基因的主要驱动因子不同(Philippot et al., 2009).花湖沉积物中, 垂向分布上nirS基因丰度有明显的上升趋势, nirK基因丰度则没有, 而沉积物含氧量一般是垂向降低(杨旭楠等, 2013).且nirS基因更偏好厌氧状态(Yuan et al., 2012; Gu et al., 2017), 溶解氧含量可能是影响花湖沉积物中nirS基因和nirK基因丰度高低的重要因素.
3.4 氨氧化与反硝化功能基因丰度与环境因子的关系细菌16S rRNA、AOA和nirS基因的丰度受到多项理化因子的显著影响, 且都与NH4+-N、NO3--N和NO2--N含量有较高的相关性(表 2).细菌16S rRNA丰度跟pH、NH4+-N、NO3--N和NO2--N呈显著正相关, 跟LOI呈显著负相关(p < 0.05).古菌amoA基因丰度跟TN、TP、NH4+-N、NO3--N和NO2--N均呈显著正相关(p < 0.05), 但是细菌amoA则与本研究中的理化因子无显著关系.郑有坤等也发现, 若尔盖湿地中AOA与NH4+-N和NO3--N含量显著相关(p < 0.05)(Zheng et al., 2014), 这与本研究结果一致;nirS基因则跟TN、TP、NH4+-N、NO3--N和NO2--N均呈现显著负相关(p < 0.05), 然而nirK和nosZI与本研究中的理化因子并无显著关系.从基因丰度和环境因子间的相关性可以发现, 花湖沉积物中TN、TP、NH4+-N、NO3--N和NO2--N等因子的浓度升高时, 古菌amoA基因的丰度会随之升高, 而nirS基因的丰度则会下降.在不同生境中, TN、NH4+-N和NO3--N等因子都曾被发现与古菌amoA基因丰度呈显著正相关(杨柳燕等, 2016), 而NO3--N浓度大于2 mmol · L-1时会抑制反硝化速率(Senbayram et al., 2012).携带古菌amoA基因的微生物多为自养型, 主要以无机氮作为氮源;而携带nirS基因的微生物多为异养型, 通过代谢有机氮来获取能量(Kuypers et al., 2018).有研究表明, 有机肥对nirS基因丰度的提高效果比无机肥更显著(李晨华等, 2014).因此, 在一定范围内NH4+-N、NO3--N和NO2--N等无机氮含量升高时, 古菌amoA基因丰度会升高, 而nirS基因丰度则会一定程度下降.同时, nirS基因丰度在中层要高于上层和下层, 而中层沉积物的TN明显低于上层和下层.相关性分析结果也显示TN与nirS基因丰度显著负相关(p < 0.05).此外, 花湖沉积物中细菌16S rRNA丰度跟TN、TP含量关系不显著, 说明这两者不是细菌丰度的限制性条件.古菌16S rRNA丰度则跟TN和NO3--N显著相关(p < 0.05).
表 2(Table 2)
| 表 2 沉积物中氨氧化与反硝化功能基因丰度与理化因子间的Spearman相关性分析 Table 2 Spearman correlation analysis between abundance of ammonia-oxidizing and denitrifying functional genes and physicochemical factors in sediments | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 2 沉积物中氨氧化与反硝化功能基因丰度与理化因子间的Spearman相关性分析 Table 2 Spearman correlation analysis between abundance of ammonia-oxidizing and denitrifying functional genes and physicochemical factors in sediments
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
花湖沉积物中, 细菌16S rRNA丰度((3.59 ×109 ± 1.36 ×109) copies · g-1)比古菌((1.75×108 ± 0.81 ×108) copies · g-1)高, nirS基因丰度比nirK基因高, 而AOA基因丰度虽然跟AOB接近, 但其在古菌16S rRNA中的相对丰度却比AOB在细菌16S rRNA中的高.而NH4+-N、NO3--N和NO2--N含量与细菌16S rRNA、nirS基因和古菌amoA基因的丰度均有显著相关性(p < 0.05).可以推测, 花湖沉积物中参与氨氧化和反硝化作用的优势菌群丰度受到环境因子的影响, TN、TP、NH4+-N、NO3--N和NO2--N是花湖沉积物中影响氨氧化和反硝化作用的重要驱动力.
4 结论(Conclusions)1) 花湖沉积物中的古菌amoA基因丰度在垂向分布上呈降低趋势, 而细菌amoA基因丰度并无明显的垂向变化.总体上, 古菌与细菌amoA基因丰度相近, 且比反硝化功能基因的整体丰度水平低了1~2个数量级.
2) 花湖沉积物中的nirS基因丰度随着深度加深呈现升高趋势, 且基因丰度明显高于nirK基因和nosZI基因, 是花湖沉积物中反硝化作用的重要驱动力.
3) 花湖沉积物中, 古菌amoA和nirS基因丰度与环境因子的相关性更显著.其中, TN、TP、NH4+-N、NO3--N和NO2--N与古菌amoA基因丰度均呈显著正相关(p < 0.05), 而与nirS基因丰度呈显著负相关(p < 0.05).这两种功能基因明显受到花湖沉积物中不同形式氮素浓度的影响与限制.
参考文献
| Beman J M, Francis C A. 2006. Diversity of ammonia-oxidizing archaea and bacteria in the sediments of a hypernutrified subtropical estuary:Bahia del Tobari, Mexico[J]. Applied and Environmental Microbiology, 72(12): 7767–7777.DOI:10.1128/AEM.00946-06 |
| Braker G, Conrad R. 2011. Diversity, structure, and size of N2O-producing microbial communities in soils-what matters for their functioning?[J]. Advances in Applied Microbiology, 75: 33–70.DOI:10.1016/B978-0-12-387046-9.00002-5 |
| Chen K, Yang X, Chen J. 2014. Ecological compensation policy of zoige plateauwetlands[J]. Wetland Science, 12(4): 419–423. |
| DagO Hessen, HenriksenA, HindarA, 等. 1997. 人类对氮循环的影响:从地区远景看全球问题[J]. AMBIO-人类环境杂志, 1997, 26(05): 314–317. |
| Di H J, Cameron K C, Shen J P, et al. 2009. Nitrification driven by bacteria and not archaea in nitrogen-rich grassland soils[J]. Nature Geoscience, 2: 621.DOI:10.1038/ngeo613 |
| Francis C A, Roberts K J, Beman J M, et al. 2005. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences, 102(41): 14683–14688.DOI:10.1073/pnas.0506625102 |
| 费世民, 崔丽娟, 何亚平, 等. 2006. 若尔盖高寒湿地生态系统定位站的背景研究——若尔盖高寒湿地研究概述[J]. 四川林业科技, 2006, 27(2): 21–29.DOI:10.3969/j.issn.1003-5508.2006.02.005 |
| Gu Y F, Wang Y Y, Xiang Q J, et al. 2017. Implications of wetland degradation for the potential denitrifying activity and bacterial populations with nirS genes as found in a succession in Qinghai-Tibet plateau, China[J]. European Journal of Soil Biology, 80: 19–26.DOI:10.1016/j.ejsobi.2017.03.005 |
| Hallin S, Lindgren P E. 1999. PCR detection of genes encoding nitrite reductase in denitrifying bacteria[J]. Applied and Environmental Microbiology, 65(4): 1652–1657. |
| 贺纪正, 张丽梅. 2009. 氨氧化微生物生态学与氮循环研究进展[J]. 生态学报, 2009, 29(1): 406–415.DOI:10.3321/j.issn:1000-0933.2009.01.049 |
| 贺纪正, 张丽梅. 2013. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98–108. |
| 胡万婷, 唐千, 孙伟, 等. 2017. 水体中蓝藻水华分解产甲烷动态过程研究[J]. 中国环境科学, 2017, 37(2): 702–710. |
| Kalff J. 2002. Limnology:inland water ecosystems[M]. Upper Saddle River, New Jersey: Prentice Hall. |
| Kloos K, Mergel A, R?sch C, et al. 2001. Denitrification within the genus Azospirillum and other associative bacteria[J]. Functional Plant Biology, 28(9): 991–998.DOI:10.1071/PP01071 |
| Kuypers M M M, Marchant H K, Kartal B. 2018. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 16(5): 263–276.DOI:10.1038/nrmicro.2018.9 |
| Li B Q, Yu Z B, Liang Z M, et al. 2014. Effects of Climate Variations and Human Activities on Runoff in the Zoige Alpine Wetland in the Eastern Edge of the Tibetan Plateau[J]. Journal of Hydrologic Engineering, 19(5): 1026–1035.DOI:10.1061/(ASCE)HE.1943-5584.0000868 |
| 李晨华, 张彩霞, 唐立松, 等. 2014. 长期施肥土壤微生物群落的剖面变化及其与土壤性质的关系[J]. 微生物学报, 2014, 54(3): 319–329. |
| Li G M, Gan Q, Wu X P, et al. 2016. The quantitative analysis of space-time distribution and the evaluation of ecological load of land cover-a case study of Zoige wetland national nature reserve[C]. International Conference on Geoinformatics. Galway, Irelandhttps://www.researchgate.net/publication/308989313_The_quantitative_analysis_of_space-time_distribution_and_the_evaluation_of_ecological_load_of_land_cover_-a_case_study_of_Zoige_Wetland_National_Nature_Reserve |
| 李志建. 2011.南四湖沉积物内源污染翻译规律与水质响应的关系研究[D].济南: 山东大学. 7-8http://cdmd.cnki.com.cn/Article/CDMD-10422-1011170779.htm |
| Michotey V, Méjean V, Bonin P. 2000. Comparison of methods for quantification of cytochrome cd 1-denitrifying bacteria in environmental marine samples[J]. Applied and Environmental Microbiology, 66(4): 1564–1571.DOI:10.1128/AEM.66.4.1564-1571.2000 |
| Mosier A C, Francis C A. 2010. Denitrifier abundance and activity across the San Francisco Bay estuary[J]. Environmental Microbiology Reports, 2(5): 667–676.DOI:10.1111/j.1758-2229.2010.00156.x |
| Muyzer G, Hottentr?ger S, Teske A, et al. 1996. Denaturing gradient gel electrophoresis of PCR-amplified 16S rDNA. A new molecular approach to analyze the genetic diversity of mixed microbial communities[J]. Molecular Microbial Ecology Manual 3.4.4, Dordrecht, The Netherlands:Kluwer Academic Publishers: 1–23. |
| Ovre?s L, Forney L, Daae F L, et al. 1997. Distribution of bacterioplankton in meromictic Lake Saelenvannet, as determined by denaturing gradient gel electrophoresis of PCR-amplified gene fragments coding for 16S rRNA[J]. Applied and Environmental Microbiology, 63(9): 3367–3373. |
| Philippot L, ?uhel J, Saby N P A, et al. 2009. Mapping field-scale spatial patterns of size and activity of the denitrifier community[J]. Environmental Microbiology, 11(6): 1518–1526.DOI:10.1111/j.1462-2920.2009.01879.x |
| Prosser J I. 1990. Autotrophic nitrification in bacteria[J]. Advances in Microbial Physiology, 30: 125–181.DOI:10.1016/S0065-2911(08)60112-5 |
| 青烨, 孙飞达, 李勇, 等. 2015. 若尔盖高寒退化湿地土壤碳氮磷比及相关性分析[J]. 草业学报, 2015, 24(3): 38–47. |
| Rice E W, Baird R B, Eaton A D, et al. 2012. Standard methods for the examination of water and wastewater[S]. Washington D C: American Public Health Association(APHA) |
| Rotthauwe J H, Witzel K P, Liesack W. 1997. The ammonia monooxygenase structural gene amoA as a functional marker:molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 63(12): 4704–4712. |
| Senbayram M, Chen R, Budai A, et al. 2012. N2O emission and the N2O/(N2O+N2) product ratio of denitrification as controlled by available carbon substrates and nitrate concentrations[J]. Agriculture, Ecosystems & Environment, 147: 4–12. |
| Shen G, Xu B, Jin Y, et al. 2016. Advances in studies of wetlands in Zoige Plateau[J]. Geography and Geo-Information Science, 32(4): 76. |
| Stahl D A, Flesher B, Mansfield H R, et al. 1988. Use of phylogenetically based hybridization probes for studies of ruminal microbial ecology[J]. Applied and Environmental Microbiology, 54(5): 1079–1084. |
| Throb?ck I N, Enwall K, Jarvis ?, et al. 2004. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE[J]. FEMS Microbiol Ecology, 49(3): 401–417.DOI:10.1016/j.femsec.2004.04.011 |
| Ullah S, Moore T R. 2011. Biogeochemical controls on methane, nitrous oxide, and carbon dioxide fluxes from deciduous forest soils in eastern Canada[J]. Journal of Geophysical Research:Biogeosciences, 116(G3): 1–15. |
| Verhamme T D, Prosser J I, Nicol G W. 2011. Ammonia concentration determines differential growth of ammonia-oxidising archaea and bacteria in soil microcosms[J]. The ISME Journal, 5(6): 1067–1071.DOI:10.1038/ismej.2010.191 |
| 王绍强, 于贵瑞. 2008. 生态系统碳氮磷元素的生态化学计量学特征[J]. 生态学报, 2008, 28(8): 3937–3947.DOI:10.3321/j.issn:1000-0933.2008.08.054 |
| 王洋, 刘景双, 孙志高, 等. 2006. 湿地系统氮的生物地球化学研究概述[J]. 湿地科学, 2006, 4(4): 311–320.DOI:10.3969/j.issn.1672-5948.2006.04.013 |
| Whelan J A, Russell N B, Whelan M A. 2003. A method for the absolute quantification of cDNA using real-time PCR[J]. Journal of Immunological Methods, 278(1/2): 261–269. |
| Wu L S, Feng S, Nie Y Y, et al. 2015. Soil cellulase activity and fungal community responses to wetland degradation in the Zoige Plateau, China[J]. Journal of Mountain Science, 12(2): 471–482.DOI:10.1007/s11629-014-3183-1 |
| Wu Q L, Zwart G, Schauer M, et al. 2006. Bacterioplankton community composition along a salinity gradient of sixteen high-mountain lakes located on the Tibetan Plateau, China[J]. Applied and Environmental Microbiology, 72(8): 5478–5485.DOI:10.1128/AEM.00767-06 |
| 杨柳燕, 王楚楚, 孙旭, 等. 2016. 淡水湖泊微生物硝化反硝化过程与影响因素研究[J]. 水资源保护, 2016, 32(1): 12–22. |
| 杨旭楠, 林兴锐, 符诗雨, 等. 2013. 感潮河流沉积物中溶解氧对硝化细菌垂向分布的影响[J]. 中山大学学报(自然科学版), 2013, 52(05): 91–96. |
| 杨亚东, 宋润科, 马俊永, 等. 2018. 长期氮磷不同施用量对土壤细菌、硝化与反硝化微生物数量的影响[J]. 中国农业大学学报, 2018, 23(09): 81–88. |
| Yuan Q, Liu P, Lu Y. 2012. Differential responses of nirK- and nirS-carrying bacteria to denitrifying conditions in the anoxic rice field soil[J]. Environmental Microbiology Reports, 4(1): 113–122.DOI:10.1111/j.1758-2229.2011.00311.x |
| Yun J L, Zhuang G Q, Ma A Z, et al. 2012. Community structure, abundance, and activity of methanotrophs in the Zoige Wetland of the Tibetan Plateau[J]. Microbial Ecology, 63(4): 835–843.DOI:10.1007/s00248-011-9981-x |
| 张淑楠, 王心义, 夏大平. 2015. 初始pH值对微生物反硝化的影响[J]. 环境工程, 2015, 33(S1): 67–71. |
| 赵娜娜, 宁宇, 马骅, 等. 2019. 若尔盖湿地土壤特性空间变化研究[J]. 水资源与水工程学报, 2019, 30(1): 232–240. |
| 赵兴青, 杨柳燕, 尹大强, 等. 2008. 不同空间位点沉积物理化性质与微生物多样性垂向分布规律[J]. 环境科学, 2008, 29(12): 3537–3545.DOI:10.3321/j.issn:0250-3301.2008.12.041 |
| Zheng Y, Wang X, Gu Y, et al. 2014. Diversity of ammonia-oxidizing archaea in Tibetan Zoige plateau wetland[J]. Acta microbiologica Sinica, 54(9): 1090–1096. |
| 朱广伟, 秦伯强, 高光, 等. 2004. 灼烧对沉积物烧失量及铁、磷测定的影响[J]. 分析试验室, 2004, 23(9): 72–76.DOI:10.3969/j.issn.1000-0720.2004.09.022 |
