 , 李琦1, 郭楚玲1,2
, 李琦1, 郭楚玲1,2
 , 汪涵1, 曾宇飞1, 卢桂宁1,2, 党志1,2
, 汪涵1, 曾宇飞1, 卢桂宁1,2, 党志1,21. 华南理工大学环境与能源学院, 广州 510006;
2. 工业聚集区污染控制与生态修复教育部重点实验室, 广州 510006
收稿日期: 2019-02-20; 修回日期: 2019-05-03; 录用日期: 2019-05-03
基金项目: 国家自然科学基金(No.41330639,41720104004);广东省自然科学基金(No.2018A030313918)
作者简介: 丁翠(1993-), 女, E-mail:840015968@qq.com
通讯作者(责任作者): 郭楚玲, E-mail:clguoscut@hotmail.com
摘要: 为探究酸性矿山废水(AMD)对稻田土壤微生物群落结构的影响,通过取自矿区受AMD污染和未受污染的稻田土壤进行微宇宙灌溉模拟实验,研究了AMD污染过程中土壤理化性质和微生物群落的变化,同时建立环境条件变化引起土壤微生物群落结构改变的相关性关系.结果表明,受AMD污染的土壤中SO42-、Cd、Zn含量显著上升,土壤酸化且土壤中细菌群落的多样性下降;而恢复清洁水灌溉可提高土壤细菌群落的多样性,有利于修复AMD的污染.采用高通量测序技术分析了不同处理稻田土壤中微生物群落在门和属分类水平上的相对丰度分布变化,冗余分析(RDA)表明,土壤pH和重金属(Pb、Cu)含量是影响稻田土壤微生物群落结构的主要环境因子.研究结果不仅有助于进一步揭示AMD污染、土壤因子与土壤微生物群落的相互关系,同时可为恢复AMD污染农业土壤提供理论依据.
关键词:AMD稻田土壤高通量测序微生物
Responses of microbial communities in paddy soils to the intrusion of acid mine drainage
DING Cui1
 , LI Qi1, GUO Chuling1,2
, LI Qi1, GUO Chuling1,2
 , WANG Han1, ZENG Yufei1, LU Guining1,2, DANG Zhi1,2
, WANG Han1, ZENG Yufei1, LU Guining1,2, DANG Zhi1,2 1. School of Environment and Energy, South China University of Technology, Guangzhou 510006;
2. The Key Lab of Pollution Control and Ecosystem Restoration in Industry Clusters, Ministry of Education, Guangzhou 510006
Received 20 February 2019; received in revised from 3 May 2019; accepted 3 May 2019
Abstract: In this study, laboratory simulation experiment was performed to evaluate the effect of AMD intrusion on paddy soil microbial communities. The alteration of microbial communities in long-term AMD-polluted and unpolluted soils under continuous AMD irrigating was comprehensively studied. Meanwhile, soil physicochemical properties were also regularly measured to determine their correlation with soil microbial community. The results indicated that AMD intrusion significantly increased the content of SO42-, Cd and Zn in soil, acidified the soil and decreased the diversity of bacterial community. While clear water irrigation was beneficial to the remediation of AMD contaminated soils and increased the diversity of bacterial community. A comprehensive survey of taxa distribution on the phylum and genus level of microbial community has been investigated by high-throughput sequencing technique. Results of RDA analysis showed that soil pH and heavy mental (Pb and Cu) content were the key factors influencing the community structure. Results also elucidate the relationship among AMD pollution, soil factors and soil microbial communities, and are useful for developing strategies to remediate AMD pollution in agriculture soil.
Keywords: AMDpaddy soilhigh-throughput sequencingmicrobial community
1 引言(Introduction)酸性矿山废水(AMD)是在矿山开发和利用过程中大量的硫化矿物暴露于地表, 与空气和水接触, 并在微生物的共同作用下氧化形成的废水, 以低pH、富含高浓度硫酸根离子和重金属为主要特点(Baker et al., 2003; Cravotta, 2008).AMD的排放会污染矿区周边环境的地表水、地下水和土壤, 表现为酸化协同重金属污染, 进而对人类健康和周围生态系统构成威胁(Tarras-Wahlberg et al., 2001; Lin et al., 2005; Navarro et al., 2008).在一些矿区, 可能由于水灾或干旱季节缺乏水源和人们对污染认识的不足, 受到AMD污染的河流会被作为农业生产灌溉用水.研究表明, AMD进入农田土壤会造成土地功能退化、生物多样性下降及影响农作物的生产和品质安全等诸多问题(Li et al., 2009; Zhuang et al., 2009a; 2009b), 导致环境灾害区的出现, 例如, 横石河下游被称为“癌症村”的上坝村.横石河位于广东韶关大宝山矿区, 大宝山是一座以露天开采为主的特大型多金属硫化物矿山, 悠久的采矿历史形成了大量的AMD, 未经处理的AMD从拦泥坝溢出流入横石河, 导致横石河流域几十公里范围内遭受严重污染(Chen et al., 2015).横石河是上坝村农业灌溉水源, 由此造成土壤酸化、重金属和硫酸根含量较高, 以及大米中重金属含量严重超标, 这也是导致村民癌症、皮肤病、肝病多发的主要原因(周建民等, 2004; 许超等, 2007; 郑堃等, 2018).对受AMD污染的土壤进行恢复和治理是我国当前生态环境保护所面临的紧迫任务, 而利用微生物进行生物处理被认为是最经济、最环保的方法之一.
土壤微生物是土壤生态系统的重要组成部分, 驱动着土壤中大部分生物地球化学过程的发生, 包括有机质分解、有害物质降解、土壤质量改善及营养元素的转化和循环等, 对保持土壤生产力和作物生长至关重要(Brookes, 1995; Tiedje, 1988; Rillig et al., 2006; van der Heijden et al., 2008; Schnitzer et al., 2011).而且土壤微生物参数, 如微生物多样性和群落组成及其变化是土壤质量和健康状况的敏感指标(Kennedy et al., 1995; Visser et al., 2009).了解AMD对土壤的污染特征, 认识AMD污染土壤中微生物群落组成和变化规律, 是开展AMD污染土壤修复工作的必要前提.因此, 研究AMD污染对土壤微生物群落的影响就显得非常重要, 在一定程度上有助于制定污染农田的生物修复策略.基于此, 本文通过对取自广东大宝山矿区已受AMD污染和未受污染的稻田土壤进行AMD和清洁水灌溉的微宇宙模拟实验, 研究AMD污染过程中土壤理化性质和微生物群落的变化, 建立环境条件与土壤微生物群落的相关性关系, 以了解AMD污染对土壤微生物的生物学效应, 从而为改善土壤质量、提高土壤肥力和恢复AMD污染农业土壤提供理论依据.
2 材料与方法(Materials and methods)2.1 研究区概况和样品采集土壤采集区域位于广东省韶关市大宝山矿区(24°34′28″N, 113°43′42″E), 该区域属于亚热带季风气候, 湿润的气候和丰沛的雨水非常有利于该区AMD的形成.于2017年11月在大宝山矿区采集TP和S8两个不同地点的稻田土壤, 其中, TP点(24°29′23″N, 113°49′21″E)稻田土壤采样地未受到AMD污染, 而S8点(24°27′48"N, 113°48′35"E)稻田土壤取自上坝村, 长期受到AMD的污染.于水稻收获后采集稻田土壤, 选定地块, 根据地形和耕作习惯采用对角线采样法布置3个采样点, 土壤采集深度为0~20 cm, 去除植物根系、石块等杂物, 放在无菌密封袋中保存, 并置于-4 ℃的便携式冰箱中以保证微生物群落的稳定, 尽快运回实验室.AMD现场采集于储存AMD的拦泥坝(24°30′58″N, 113°43′28″E), 运回实验室用作灌溉水, 其主要参数为:pH=2.67, SO42-、Fe2+、Cr、Ni、Cu、Zn、Cd、Pb浓度分别为5300、488.11、0.024、0.486、4.011、122.29、0.301、0.015 mg·L-1.
2.2 实验处理土柱灌溉微宇宙实验共设4个不同的处理:AMD灌溉TP土壤(TP+AMD);AMD灌溉S8土壤(S8+AMD);去离子水灌溉TP土壤(TP+CW)和S8土壤(S8+CW)作为对照.所用柱子直径10 cm, 高40 cm, 加入土壤至20 cm高, 然后加入灌溉水(AMD或清洁水)至30 cm处, 实验过程中保持灌溉水一直处于土壤上方10 cm高度.实验周期为189 d, 期间共采集土壤13次, 烘干后测定理化性质, 采集原始土壤及17、95、189 d的土壤, 冷冻保存用于DNA提取.每次从土柱取3个样品进行土壤理化因子和微生物群落的测定.
2.3 土壤理化性质测定烘干的土壤研磨过筛去除根系、石子等.灌溉水pH使用便携式pH计测定;在干土与1 mol·L-1 KCl溶液土液比为1:2.5时测定土壤pH;土壤有机质采用重铬酸钾外加热法测定;土壤中Fe2+含量采用邻菲罗啉分光光度法在510 nm波长处分析测定(UV-2550, SHIMADZU);SO42-含量是在水土比为5:1时使用离子色谱仪(DIONEX ICS-1500, Sunnyvale, USA)测定;土壤四酸(盐酸、硝酸、氢氟酸和高氯酸)消解后使用原子吸收光谱仪测定重金属总量(Cr、Cd、Cu、Pb、Ni、Zn).
2.4 土壤DNA提取、PCR扩增和高通量测序参照MoBio DNA试剂提取盒(MoBio Laboratories, Carlsbad, CA, USA)建议的步骤, 分别提取各个土壤样品中的DNA, 利用1%琼脂糖凝胶电泳检测DNA的完整性、纯度和浓度.利用细菌通用引物515F(5′-CCGGACTACVSGGGTATCTAAT-3′)和806R (5′-GTGCCAGCMGCCGCGGTAA-3′), 以基因组DNA为模板进行PCR扩增.设置反应条件为:94 ℃预变性5 min;94 ℃变性30 s, 52 ℃退火30 s, 72 ℃延伸30 s, 30个循环;72 ℃延伸10 min.每个样本进行3个重复, 并将同一样本的PCR产物进行混合, 用1%琼脂糖凝胶电泳检测PCR产物的片段长度和浓度, 主带长度在正常范围内的样品可用于进一步的实验.使用EZNA Gel Extraction Kit (Omega, USA)凝胶回收试剂盒回收PCR混合产物, TE缓冲液洗脱回收目标DNA片段.本研究的测序和生物信息服务在广州美格生物科技有限公司完成.按照NEBNext? UltraTM DNA Library Prep Kit for Illumina? (New England Biolabs, USA)标准流程进行建库操作, 使用Illumina Hiseq 2500平台对构建的扩增子文库进行PE250测序.
2.5 数据处理及统计分析使用FASTQ软件对原始双端测序数据进行质量筛查, 随后利用FLASH软件(v1.2.7, http://ccb.jhu.edu/software/FLASH/)对通过质量初筛的双端序列根据重叠碱基进行配对并拼接成一条完整的目的片段, 同时对reads的质量和merge的效果进行质控过滤.最后, 经过去嵌合体、singleton OTU及污染OTU后得到用于分析的有效数据(No. of seqs).所有土壤样品共获得1589934条有效序列.在97%的相似度水平下对有效序列进行OTUs(Operational Taxonomic Units)聚类及物种注释以获得每个样品的OTUs的分布及物种信息, 基于以上信息进行样品物种丰度及多样性等分析.同时, 结合环境因子利用Canoco 5.0进行冗余分析(RDA)来研究土壤细菌群落和环境因子之间的关系.通过Galaxy在线分析平台(http://huttenhower.sph.harvard.edu/galaxy/)上传属水平的OTU表格, 按分析流程进行LEfSe分析, 并将分析结果可视化后导出.
3 结果与讨论(Results and discussion)3.1 灌溉后土壤理化因子的变化3.1.1 基本理化因子的变化通过分析AMD灌溉和清洁水灌溉土壤理化因子的变化结果发现(图 1), AMD灌溉提高了土壤中SO42-的含量, 降低了土壤pH, 而清洁水灌溉提高了土壤pH, 降低了土壤中SO42-的含量.本实验中AMD的pH值较低, 灌溉过程中基本维持在3左右.S8点的土壤因长期受到AMD污染本身酸度较高, 清水灌溉过程中会缓慢释放大量酸, 一段时间后灌溉水pH才缓慢上升;TP点土壤是南方酸性土壤, AMD污染使其土壤酸化, 由于土壤的缓冲能力, 土壤pH变化不是很剧烈.OM的变化可能与土壤的性质、深度有关, 总体来说呈下降趋势, 这是因为一方面在土壤微生物的作用下分解为简单的无机化合物;另一方面, 灌溉水可将土壤有机质中可溶性的物质洗出, 造成OM含量减少.实验测得的SO42-为水溶态(WS), 也是AMD污染带入土壤中硫的主要形态, 具有高活性和易迁移性, 进入土壤后会发生吸附和同化作用, 清水灌溉促进了土壤中SO42-进入土壤溶液(杨成方等, 2016).不同处理土壤中亚铁含量有较大幅度升高, 这可能是因为淹水条件下的厌氧环境有利于三价铁的还原.
图 1(Fig. 1)
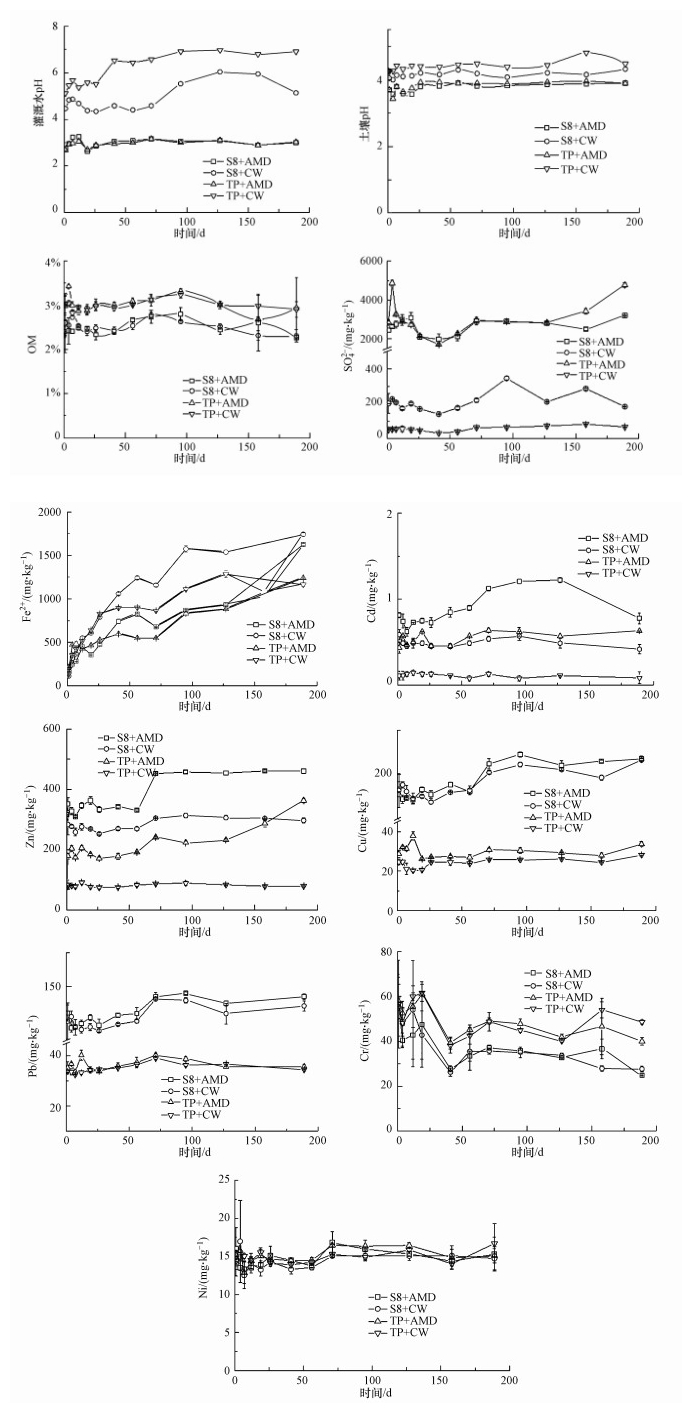 |
| 图 1 土壤理化性质的变化 Fig. 1The alteration of soil physicochemical properties |
3.1.2 土壤中重金属含量的变化AMD灌溉提高了土壤中Cd、Zn的含量, 对Pb、Cu无明显富集作用.而邓冬梅等(2016)对受AMD污染一年的水稻土分析发现, 污染土壤中Cu富集明显, 但Pb、Zn和Cd无明显富集.这主要是因为不同来源的AMD性质差异较大, 本研究所使用的AMD中Zn、Cd的浓度显著较高, 而Pb、Cu浓度较低;其次, 农作物的吸收富集作用也有一定影响.AMD中Cr、Ni浓度较低, 在土壤中没有出现明显累积.S8和TP点土壤在受到AMD污染过程中出现不同程度的Cd累积, 可能与污染时间及土壤自身性质的差异有关.清水灌溉使得S8点土壤中部分Cd被淋溶出来进入灌溉水中, 土壤中总Cd含量降低, 而Cu、Pb、Zn含量并没有减少, 可能是由于Cd的迁移能力大于Pb、Zn、Cu (雷鸣等, 2007).
3.2 AMD污染对土壤中微生物群落多样性的影响为了比较不同处理中细菌群落的多样性, 采用不同的Alpha多样性指数进行分析, 通常计算的参数包括Observed species指数、Chao1指数、Shannon指数、Simpson指数及PD whole tree等.在97%分类水平上, 利用Mothur软件计算各处理189 d时的Alpha多样性参数值(表 1).通过比较可知, 各组的丰富度和多样性排列为S8+AMD < S8+CW、TP+AMD < TP+CW, 可见AMD污染降低了土壤中细菌群落的多样性, 降低程度与受AMD污染程度有关, 恢复清洁水灌溉后多样性会缓慢提高.
表 1(Table 1)
| 表 1 土壤微生物群落Alpha多样性指数统计表 Table 1 The Alpha diversity index of soil bacterial community in different irrigation systems | |||||||||||||||||||||||||||||||||||
表 1 土壤微生物群落Alpha多样性指数统计表 Table 1 The Alpha diversity index of soil bacterial community in different irrigation systems
| |||||||||||||||||||||||||||||||||||
3.3 稻田土壤微生物群落分析4个不同处理的原始土壤及17、95、189 d土壤中细菌群落在门水平上的相对丰度分布如图 3所示, 本文选取土壤中相对丰度排名前10的物种进行展示.土壤细菌群落在门分类水平上具有较高的多样性, 均达到43个门以上.优势门以变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、酸杆菌门(Acidobacteria)、浮霉菌门(Planctomycetes)、疣微菌门(Verrucomicrobia)为主, 相对丰度总和约占58%~81%.平均而言, 变形菌门(Proteobacteria)在所有土壤样品中占有最高的丰度, 为27%.各门类在不同处理、不同时间土壤细菌群落中所占比例各有不同, 并随时间发生一定的变化.189 d后, 受AMD污染的S8和TP点土壤中Proteobacteria、Verrucomicrobia、OD1、Planctomycetes、Bacteroidetes的相对丰度分别低于清洁水灌溉的S8和TP点土壤中的相对丰度, 而Acidobacteria、Chloroflexi、Gemmatimonadetes、Chlorobi、Nitrospirae的相对丰度高于清洁水灌溉的S8和TP点土壤中的相对丰度.
图 2(Fig. 2)
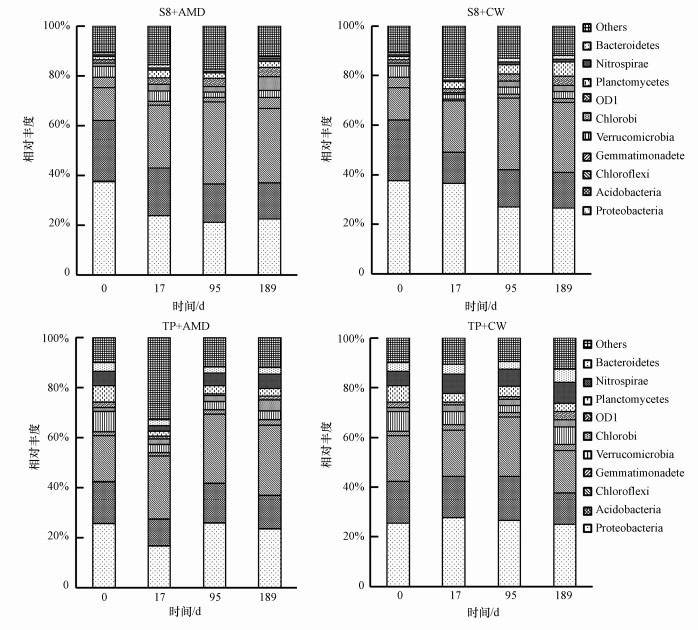 |
| 图 2 不同灌溉处理稻田土壤中细菌在门水平上的优势物种相对丰度分布 Fig. 2The relative abundance of dominant phyla in different irrigation systems |
图 3展示了灌溉189 d时土壤中优势属的相对丰度分布, 不同灌溉处理的稻田土壤主要以Anaerolinea、Anaeromyxobacter、Candidatus Solibacter、Geobacter为主.结果显示, 土壤中存在较多的铁硫循环相关的属, 如Acinetobacter、Anaeromyxobacter、Citrobacter、Clostridium、Desulfosporosinus、GOUTA19、Geobacter、Pseudomonas (Akagi et al., 1962; Caccavo et al., 1994; Childers et al., 2002; Sanford etal., 2002; Treude et al., 2003; Lopes et al., 2014).其中, Geobacter为常见的异化铁还原菌, 在Fe(Ⅲ)丰富且可供微生物利用时成为铁还原的主力军(Lovley et al., 2004).Anaeromyxobacter具备厌氧铁还原的能力, 形成孢子的特性使它能够在酸性环境中生长(Petrie et al., 2003).虽然Desulfosporosinus被认为是硫酸盐还原菌, 菌株常从酸性沉积物中分离出来(Sánchez-Andrea et al., 2015), 但研究还发现Desulfosporosinus也具备还原Fe3+的能力(Bertel et al., 2012).因此, 三价铁的微生物还原作用是导致土壤中亚铁含量升高的主要原因.从Acinetobacter属分离的一些菌株能够在厌氧条件下氧化Fe2+, 对重金属和pH有一定的耐受性(Paul et al., 2012; Su et al., 2015; Kushwaha et al., 2017).Citrobacter、Pseudomonas、Clostridium、GOUTA19是硫代谢菌, 其中, Citrobacter和Pseudomonas的菌株多从AMD环境中分离出来, Citrobacter和Pseudomonas也是AMD处理修复过程中的重要微生物(Bhattacharya et al., 2006; Qiu et al., 2009; Maharana et al., 2016; Sajjad et al., 2016; Zhang et al., 2017).受AMD污染的S8和TP组中耐酸属Acinetobacter、Anaerolinea、Citrobacter、Desulfosporosinus的相对丰度分别高于清洁水灌溉的S8和TP组, 而中性属Anaeromyxobacter、Geobacter在清洁水灌溉的S8和TP组中较丰富.微生物群落结构与所处的环境密切相关, AMD进入稻田土壤改变土壤的理化性质从而对微生物群落产生影响, 能够适应AMD污染环境的细菌成为优势类群, 不能适应的细菌则逐渐减少或者消失.
图 3(Fig. 3)
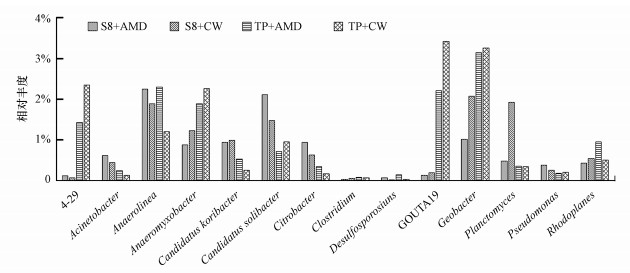 |
| 图 3 不同灌溉处理稻田土壤中细菌在属水平上的优势物种相对丰度分布 Fig. 3The relative abundance of dominant genera in different irrigation systems |
3.4 AMD污染对稻田土壤微生物群落结构的影响本研究采用LEfSe来分析AMD污染对稻田土壤细菌菌群结构的影响, 并将组间具有显著差异的分类单元(biomarker)通过LDA值分布柱状图和进化分支图两种形式来展示(图 4).图 4中只展示了LDA值大于3.5的物种, 可以看出, TP点土壤不同灌溉组间具有更多的差异物种.对于TP点不同灌溉组间, 在门和纲的水平上, Firmicutes、Chloroflexi、Gammaproteobacteria、Clostridia、Acidobacteriia更多地富集在TP+AMD组土壤中, 而Planctomycetes、Betaproteobacteria、Deltaproteobacteria、Bacteroidetes、Nitrospirae、Nitrospira富集在TP+CW土壤中.在属水平上, Desulfosporosinus、Anaerolinea在TP+AMD组中占据主要地位, 而Anaeromyxobacter、Geobacter、GOUTA19更多地存在于对照组.Firmicutes能够产生芽孢的特性使其可以抵抗脱水和极端环境, Clostridia是Firmicutes典型的纲(Galperin, 2013).Proteobacteria是环境中最丰富的类群, 广泛分布在各种生态中, 在酸性环境中也常被检测到, 其中, Gammaproteobacteria和Alphaproteobacteria对低pH环境显示出很强的适应性(Kuang et al., 2013), Deltaproteobacteria和Betaproteobacteria这两类菌纲都常见于稻田土壤生态系统中.Acidobacteriia在酸性环境中生长较好(Eichorst et al., 2007). Desulfosporosinus为铁硫还原菌, 菌株大多从酸性环境中分离出来(Casiot et al., 2005; Bertel et al., 2012).Planctomycetes、Bacteroidetes广泛分布在环境中(Woese, 1987; Fernandez-Gomez et al., 2013).对于S8不同灌溉组间, Solibacteres、Candidatus Solibacter富集在S8+AMD组, 而Phycisphaerae、Acidobacteria-6、Planctomycetes、Alphaproteobacteria、Deltaproteobacteria、Proteobacteria和Geobacter在S8+CW组中丰度较高.Wang等(2018)也检测到Candidatus Solibacter在长期受到AMD污染的稻田土壤中占优势地位.
图 4(Fig. 4)
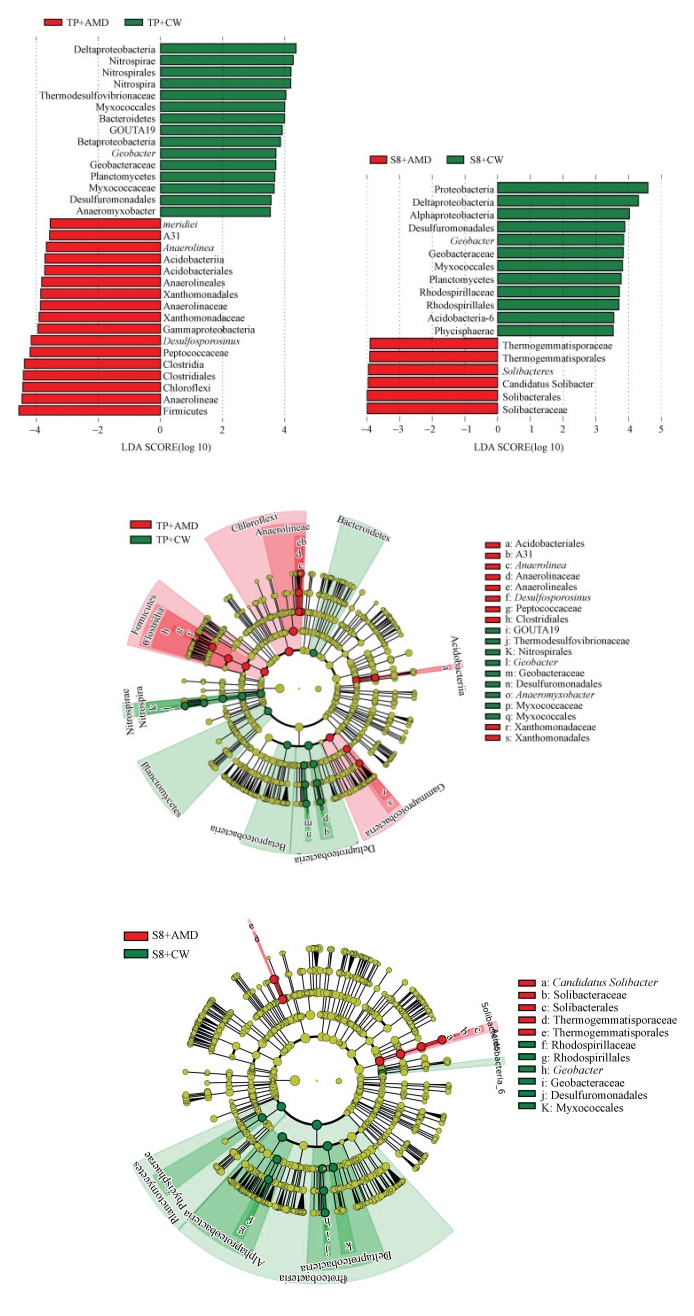 |
| 图 4 不同灌溉组间群落差异的LEfSe分析 Fig. 4LEfSe analysis identified the bacteria taxa with significant differences between AMD and clear water irrigated soil (Only the taxa with LDA value higher than 3.5 were presented) |
3.5 稻田土壤中微生物群落与环境因子的相关性分析采用CANOCO 5.0软件将土壤中相对丰度大于1%的优势属与土壤pH、有机质、SO42-等环境因子进行冗余分析(RDA), 结果见图 5.第一轴特征值为53.76%, 包含了大部分的土壤环境信息和微生物群落信息, 可以较好地解释各环境因子及样本之间的关系.结果表明, 土壤pH、重金属(Pb、Cu)含量对不同灌溉处理的稻田土壤微生物群落结构的影响最大, Cu和Pb的影响几乎完全一致, 可能是Cu与Pb的化学性质相似造成的.很多研究表明, pH是影响土壤细菌群落结构的关键因素(Lauber et al., 2008; Lauber et al., 2009), 而Wang等(2018)对大宝山沿河流域稻田土壤中微生物群落调查研究时也发现重金属(Pb、Cu)是重要的影响因素.
图 5(Fig. 5)
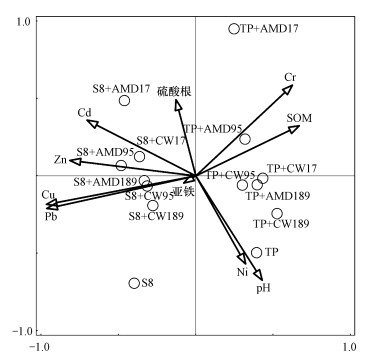 |
| 图 5 属水平群落结构与环境因子的RDA分析 Fig. 5RDA ordination diagram of genus and measured soil parameters |
4 结论(Conclusions)本研究采用实验室土柱模拟实验系统研究了AMD污染稻田土壤后细菌菌群结构和土壤理化性质的变化, 使用高通量测序技术对连续189 d AMD灌溉和清水灌溉的TP(正常土壤)和S8(受污染)土壤中细菌群落的结构和多样性进行了研究, 结果发现, AMD污染显著降低了细菌多样性, 改变了土壤理化性质和细菌群落结构.不同灌溉处理的土壤以变形菌门(Proteobacteria)为主, 存在较多的铁硫代谢属, 这些菌的存在对土壤中铁硫元素的循环转化有重要影响.受污染土壤采用干净水灌溉后会增加土壤细菌群落的多样性, 有助于土壤的修复.
参考文献
| Akagi J M, Campbell L L. 1962. Studies on thermophilic sulfate-reducing bacteria Ⅲ.: Adenosine triphosphate-sulfurylase of Clostridium nigrificans and Desulfovibrio desulfuricans[J]. Journal of Bacteriology, 84(6): 1194–1201. |
| Baker B J, Banfield J F. 2003. Microbial communities in acid mine drainage[J]. FEMS Microbiology Ecology, 44: 139–152.DOI:10.1016/S0168-6496(03)00028-X |
| Bertel D, Peck J, Quick T J, et al. 2012. Iron transformations induced by an acid-tolerant Desulfosporosinus species[J]. Applied and Environmental Microbiology, 78(1): 81–88.DOI:10.1128/AEM.06337-11 |
| Bhattacharya J, Islam M, Cheong Y W. 2006. Microbial growth and action:implications for passive bioremediation of acid mine drainage[J]. Mine Water and the Environment, 25(4): 233–240.DOI:10.1007/s10230-006-0138-y |
| Bolger A M, Lohse M, Usadel B. 2014. Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 30: 2114–2120.DOI:10.1093/bioinformatics/btu170 |
| Brookes P C. 1995. The use of microbial parameters in monitoring soil pollution by heavy metals[J]. Biology and Fertility of Soils, 19: 269–279.DOI:10.1007/BF00336094 |
| Caccavo F, Lonergan D J, Lovley D R, et al. 1994. Geobacter sulfurreducens sp.nov., a hydrogen-and acetate-oxidizing dissimilatory metal-reducing microorganism[J]. Applied and Environmental Microbiology, 60(10): 3752–3759. |
| Casiot C, Lebrun S, Morin G, et al. 2005. Sorption and redox processes controlling arsenic fate and transport in a stream impacted by acid mine drainage[J]. Science of the Total Environment, 347(1/3): 122–130. |
| Chen M, Lu G, Guo C, et al. 2015. Sulfate migration in a river affected by acid mine drainage from the Dabaoshan mining area, South China[J]. Chemosphere, 119: 734–743.DOI:10.1016/j.chemosphere.2014.07.094 |
| Childers S E, Ciufo S, Lovley D R. 2002. Geobacter metallireducens accesses insoluble Fe(Ⅲ) oxide by chemotaxis[J]. Nature, 416: 767–769.DOI:10.1038/416767a |
| Cravotta Ⅲ C A. 2008. Dissolved metals and associated constituents in abandoned coal-mine discharges, Pennsylvania, USA.Part 1:Constituent quantities and correlations[J]. Applied Geochemistry, 23(2): 166–202.DOI:10.1016/j.apgeochem.2007.10.011 |
| 邓冬梅, 邱玉龙, 王银杏, 等. 2016. 酸性矿石废水短期污染对水稻土的影响[J]. 土壤, 2016, 48(4): 754–761. |
| Eichorst S A, Breznak J A, Schmidt T M. 2007. Isolation and characterization of soil bacteria that define Terriglobus gen.nov., in the phylum Acidobacteria[J]. Applied and Environmental Microbiology, 73(8): 2708–2717.DOI:10.1128/AEM.02140-06 |
| Fernandez-Gomez B, Richter M, Schuler M, et al. 2013. Ecology of marine bacteroidetes:A comparative genomics approach[J]. ISME Journal, 7(5): 1026–1037.DOI:10.1038/ismej.2012.169 |
| Galperin M Y.2013.Genome diversity of spore-forming Firmicutes[J].Microbiology Spectrum, 1(2): TBS-0015-2012 |
| Kennedy A C, Smith K L. 1995. Soil microbial diversity and the sustainability of agricultural soils[J]. Plant and Soil, 170(1): 75–86.DOI:10.1007/BF02183056 |
| Kuang J L, Huang L N, Chen L X, et al. 2013. Contemporary environmental variation determines microbial diversity patterns in acid mine drainage[J]. ISME Journal, 7(5): 1038.DOI:10.1038/ismej.2012.139 |
| Kushwaha A, Rani R, Kumar S, et al. 2017. A new insight to adsorption and accumulation of high lead concentration by exopolymer and whole cells of lead-resistant bacterium Acinetobacter junii L.Pb1 isolated from coal mine dump[J]. Environmental Science and Pollution Research, 24(11): 10652–10661.DOI:10.1007/s11356-017-8752-8 |
| Lauber C L, Hamady M, Knight R, et al. 2009. Pyrosequencing-based assessment of soil pH as a predictor of soil bacterial community structure at the continental scale[J]. Applied and Environment Microbiology, 75: 5111–5120.DOI:10.1128/AEM.00335-09 |
| Lauber C L, Strickland M S, Bradford M A, et al. 2008. The influence of soil properties on the structure of bacterial and fungal communities across land-use types[J]. Soil Biology & Biochemistry, 40: 2407–2415. |
| 雷鸣, 廖柏寒, 秦普丰, 等. 2007. 矿区污染土壤Pb、Cd、Cu和Zn的形态分布及其生物活性的研究[J]. 生态环境, 2007, 16(3): 807–811.DOI:10.3969/j.issn.1674-5906.2007.03.020 |
| Lin C, Tong X, Lu W, et al. 2005. Environmental impacts of surface mining on mined lands, affected streams and agricultural lands in the Dabaoshan Mine region, southern China[J]. Land Degradation & Development, 16: 463–474. |
| Li Y T, Becquer T, Dai J, et al. 2009. Ion activity and distribution of heavy metals in acid mine drainage polluted subtropical soils[J]. Environmental Pollution, 157: 1249–1257.DOI:10.1016/j.envpol.2008.11.050 |
| Lopes A R, Manaia C M, Nunes O C. 2014. Bacterial community variations in an alfalfa-rice rotation system revealed by 16S rRNA gene 454-pyrosequencing[J]. FEMS microbiology ecology, 87(3): 650–663.DOI:10.1111/1574-6941.12253 |
| Lovley D R, Holmes D E, Nevin K P. 2004. Dissimilatory Fe(Ⅲ) and Mn(Ⅳ) reduction[J]. Microbiology and Molecular Biology Reviews, 55(2): 259–287. |
| Macaskie L E, Dean A C. 1984. Cadmium accumulation by a Citrobacter sp[J]. Journal of General Microbiology, 130: 53–62. |
| Maharana J K, Sahu G, Agrawal P, et al. 2016. Microbial characterization and phylogenetic analysis of a chemolithotrophic bacterial strain isolated from acid mine drainage[J]. International Journal of New Technology and Research, 2(5): 8–15. |
| Navarro M C, Perez-Sirvent C, Martinez-Sanchez M J, et al. 2008. Abandoned mine sites as a source of contamination by heavy metals:A case study in a semi-arid zone[J]. Journal of Geochemical Exploration, 96: 183–193.DOI:10.1016/j.gexplo.2007.04.011 |
| Paul M L, Samuel J, Chandrasekaran N, et al. 2012. Comparative kinetics, equilibrium, thermodynamic and mechanistic studies on biosorption of hexavalent chromium by live and heat killed biomass of Acinetobacter junii VITSUKMW2, an indigenous chromite mine isolate[J]. Chemical Engineering Journal, 187: 104–113.DOI:10.1016/j.cej.2012.01.106 |
| Petrie L, North N N, Dollhopf S L, et al. 2003. Enumeration and characterization of iron(Ⅲ)-reducing microbial communities from acidic subsurface sediments contaminated with uranium(Ⅵ)[J]. Applied and Environmental Microbiology, 69(12): 7467–7479.DOI:10.1128/AEM.69.12.7467-7479.2003 |
| Puranik P R, Paknikar K M. 1999. Biosorption of lead, cadmium, and zinc by Citrobacter strain MCM B-181:characterization studies[J]. Biotechnology Progress, 15: 228–237.DOI:10.1021/bp990002r |
| Qiu R, Zhao B, Liu J, et al. 2009. Sulfate reduction and copper precipitation by a Citrobacter sp.isolated from a mining area[J]. Journal of Hazardous Materials, 164(2/3): 1310–1315. |
| Rillig M C, Mummey D L. 2006. Mycorrhizas and soil structure[J]. New Phytologist, 171(1): 41–53.DOI:10.1111/j.1469-8137.2006.01750.x |
| Sajjad W, Bhatti T M, Hasan F, et al. 2016. Characterization of sulfur-oxidizing bacteria isolated from acid mine drainage and black shale samples[J]. Pakistan Journal of Botany, 48(3): 1253–1262. |
| Sánchez-Andrea I, Stams A J M, Hedrich S, et al. 2015. Desulfosporosinus acididurans sp.nov.:an acidophilic sulfate-reducing bacterium isolated from acidic sediments[J]. Extremophiles, 19(1): 39–47.DOI:10.1007/s00792-014-0701-6 |
| Sanford R A, Cole J R, Tiedje J M. 2002. Characterization and description of Anaeromyxobacter dehalogenans gen.nov., sp.nov., an aryl-halorespiring facultative anaerobic myxobacterium[J]. Applied and Environmental Microbiology, 68(2): 893–900.DOI:10.1128/AEM.68.2.893-900.2002 |
| Schnitzer S A, Klironomos J N, HilleRisLambers J, et al. 2011. Soil microbes drive the classic plant diversity-productivity pattern[J]. Ecology, 92: 296–303.DOI:10.1890/10-0773.1 |
| Sharma J, Fulekar M H. 2009. Potential of Citrobacter freundii for bioaccumulation of heavy metal-copper[J]. Biology and Medicine, 1(3): 7–14. |
| Soman C, Li D, Wander M M, et al. 2017. Long-term fertilizer and crop-rotation treatments differentially affect soil bacterial community structure[J]. Plant and Soil, 413: 145–159.DOI:10.1007/s11104-016-3083-y |
| Su J F, Zheng S C, Huang T L, et al. 2015. Simultaneous removal of Mn(Ⅱ) and nitrate by the manganese-oxidizing bacterium Acinetobacter sp.SZ28 in anaerobic conditions[J]. Geomicrobiology Journal, 33(7): 586–591. |
| Tarras-Wahlberg N H, Flachier A, Lane S N, et al. 2001. Environmental impacts and metal exposure of aquatic ecosystems in rivers contaminated by small scale gold mining:the Puyango River basin, southern Ecuador[J]. Science of the Total Environment, 278: 239–261.DOI:10.1016/S0048-9697(01)00655-6 |
| Tiedje J M. 1988. Ecology of denitrification and dissimilatory nitrate reduction to ammonium[J]. Biology of Anaerobic Microorganisms, 717: 179–244. |
| Treude N, Rosencrantz D, Liesack W, et al. 2003. Strain FAc12, a dissimilatory iron-reducing member of the Anaeromyxobacter subgroup of Myxococcales[J]. FEMS Microbiology Ecology, 44(2): 261–269.DOI:10.1016/S0168-6496(03)00048-5 |
| Van Der Heijden M G A, Bardgett R D, Van Straalen N M. 2008. The unseen majority:soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems[J]. Ecology Letters, 11(3): 296–310.DOI:10.1111/j.1461-0248.2007.01139.x |
| Visser S, Parkinson D. 2009. Soil biological criteria as indicators of soil quality:Soil microorganisms[J]. American Journal of Alternative Agriculture, 7: 33–37. |
| Wang H, Zeng Y, Guo C, et al. 2018. Bacterial, archaeal, and fungal community responses to acid mine drainage-laden pollution in a rice paddy soil ecosystem[J]. Science of the Total Environment, 616-617: 107–116.DOI:10.1016/j.scitotenv.2017.10.224 |
| Woese C R. 1987. Bacterial evolution[J]. Microbiological Reviews, 51: 221. |
| 许超, 夏北成, 秦建桥, 等. 广东大宝山矿山下游地区稻田土壤的重金属污染状况的分析与评价[J]. 农业环境科学学报, 26(增刊): 549–553. |
| 杨成方.2016.金属硫化物矿区稻田土壤中硫素的迁移转化及次生硫酸盐矿物中重金属的溶出机制[D].广州: 华南理工大学 |
| Zhang H, Li M, Yang Z, et al. 2017. Isolation of a non-traditional sulfate reducing-bacteria Citrobacter freundii sp.and bioremoval of thallium and sulfate[J]. Ecological Engineering, 102: 397–403.DOI:10.1016/j.ecoleng.2017.02.049 |
| 郑堃, 任宗玲, 覃小泉, 等. 2018. 韶关工矿区水稻土和稻米中重金属污染状况及风险评价[J]. 农业环境科学学报, 2018, 37(5): 915–925. |
| 周建民, 党志, 司徒粤, 等. 2004. 大宝山矿区周围土壤重金属污染分布特征研究[J]. 农业环境科学学报, 2004, 23(6): 1172–1176.DOI:10.3321/j.issn:1672-2043.2004.06.033 |
| Zhuang P, McBride M B, Xia H, et al. 2009a. Health risk from heavy metals via consumption of food crops in the vicinity of Dabaoshan mine, South China[J]. Science of the Total Environment, 407: 1551–1561.DOI:10.1016/j.scitotenv.2008.10.061 |
| Zhuang P, Zou B, Li N Y, et al. 2009b. Heavy metal contamination in soils and food crops around Dabaoshan mine in Guangdong, China:implication for human health[J]. Environmental Geochemistry and Health, 31: 707–715.DOI:10.1007/s10653-009-9248-3 |
