 , 雷宝坤2, 张丹1, 毛昆明1, 付斌2, 陈安强2
, 雷宝坤2, 张丹1, 毛昆明1, 付斌2, 陈安强2
 , 王蓉1
, 王蓉1

1. 云南农业大学资源与环境学院, 昆明 650201;
2. 云南省农业科学院农业环境资源研究所, 昆明 650205
收稿日期: 2019-03-28; 修回日期: 2019-05-31; 录用日期: 2019-05-31
基金项目: 国家自然科学基金(No.41661048,41401248,41701105);云南省科技人才和平台计划(No.2019HB033)
作者简介: 崔荣阳(1993-), 男, E-mail:cuirongyang@163.com
通讯作者(责任作者): 陈安强, E-mail:chaq163@163.com
王蓉, E-mail:17254044@qq.com
摘要: 为揭示湖泊近岸浅层地下水升降对菜地土壤剖面硝化与反硝化功能微生物基因丰度的影响,以洱海湖滨带菜地土壤剖面为研究对象,通过模拟地下水升降过程,分析了水位升高(S1)、水位降低(S2)及落干(S3)过程中土壤剖面AOA-amoA、AOB-amoA、nirK、nirS、nosZ基因丰度的变化特征,探讨了功能基因与土壤环境因子的耦合关系.结果表明:S3阶段的土壤剖面AOA-amoA和AOB-amoA基因丰度显著高于S1和S2;S1阶段的土壤剖面nirK、nirS、nosZ基因丰度均显著高于S2和S3.AOA-amoA基因丰度显著高于AOB-amoA基因丰度,nirS基因丰度显著高于nirK、nosZ基因丰度;不同取样时期的土壤剖面AOA-amoA、AOB-amoA、nirK、nirS、nosZ基因丰度均表现为A层> B层> C层> D层.水位升降对土壤剖面AOA-amoA、AOB-amoA、nirK、nirS、nosZ基因丰度有显著影响,且AOA-amoA和nirS基因对水位升降更敏感,分别在硝化与反硝化作用中占主导地位;pH、有机碳(SOC)、全氮(TN)为功能基因AOA-amoA、AOB-amoA的环境驱动因子,而功能基因nirK、nirS、nosZ的环境驱动因子为土壤含水量(W)、铵态氮(NH4+-N)、硝态氮(NO3--N)、TN、SOC、pH.该研究结果可为揭示浅层地下水升降过程中菜地土壤剖面氮素循环的微生物学机制提供科学依据.
关键词:浅层地下水位升降土壤剖面amoAnirKnirSnosZ
Effects of shallow groundwater fluctuations on the abundances of nitrification and denitrification microbes in the soil profile of vegetable field
CUI Rongyang1,2
 , LEI Baokun2, ZHANG Dan1, MAO Kunming1, FU Bin2, CHEN Anqiang2
, LEI Baokun2, ZHANG Dan1, MAO Kunming1, FU Bin2, CHEN Anqiang2
 , WANG Rong1
, WANG Rong1

1. College of Resources and Environment Science, Yunnan Agricultural University, Kunming 650201;
2. Institute of Agricultural Environmental Resources Research, Yunnan Academy of Agricultural Sciences, Kunming 650205
Received 28 March 2019; received in revised from 31 May 2019; accepted 31 May 2019
Abstract: In order to reveal the effects of shallow groundwater fluctuations on the abundances of functional genes associated with nitrification and denitrification microbes in soil, vegetable field soil in the lakeside area of the Erhai Lake was taken as the research object, and the variation of abundances of AOA-amoA, AOB-amoA, nirK, nirS and nosZ in soil profiles ware analyzed during rising (S1), falling (S2) of water level and drying (S3) by simulating the groundwater table. The coupling relationships between functional genes and soil environmental factors were discussed. The results show that the abundances of AOA-amoA and AOB-amoA in soil profiles in the stage S3 were significantly higher than that in the stages S1 and S2. The abundances of nirK, nirS and nosZ in the soil profile in the stage S1 were significantly higher than those in the stages S2 and S3. The gene abundance of AOA-amoA was significantly higher than that of AOB-amoA. The gene abundance of nirS was significantly higher than that of nirK and nosZ. The abundances of AOA-amoA, AOB-amoA, nirK, nirS and nosZ in different soil layers in different sampling periods were as follows: A layer >B layer>C layer>D layer. The rising and falling of water table had significant effects on the gene abundances of AOA-amoA, AOB-amoA, nirK, nirS and nosZ in soil profile. The AOA-amoA and nirS weredominant genes in the nitrification and denitrification, respectively, and were more sensitive to the fluctuation of water level. pH, organic carbon (SOC) and total nitrogen (TN) were the main environmental driving factors of the AOA-amoA and AOB-amoA, and the main environmental driving factors of the nirK, nirS and nosZ are soil moisture content (W), ammonium nitrogen (NH4+-N), nitrate nitrogen (NO3--N), TN, SOC, pH. The results of this study provide a scientific basis for revealing the microbial mechanism of nitrogen cycling process in the soil profile of vegetable fields under the groundwater table fluctuations.
Keywords: shallow groundwater fluctuationsoil profileamoAnirKnirSnosZ
1 引言(Introduction)陆地生态系统中土壤微生物数量庞大、分布广泛, 是系统中物质能量流动和养分元素循环的重要驱动者(Xia et al., 2010).土壤氮素循环是生物地球化学循环的重要过程, 影响氮素转化的关键步骤硝化和反硝化作用均由相关微生物驱动(Molina et al., 2007).氨氧化是硝化作用的限速步骤, 主要由氨氧化古菌(AOA)和氨氧化细菌(AOB)的氨单加氧酶(amoA)、完全氨氧化菌(Comammox)驱动完成(Kowalchuk et al., 2001; Arp et al., 2003).NO2·还原为NO的过程是反硝化作用的标志性反应(陈晨等, 2017), 相应的亚硝酸盐还原酶(Nir)是执行该步骤的限速酶, 分为cdl-亚硝酸盐还原酶和Cu-亚硝酸盐还原酶, 其编码基因nirK和nirS的丰度常被用来表征及预测N2O的释放(Morales et al., 2010).氧化亚氮还原酶控制着N2O还原为N2的过程, 是反硝化作用的最后关键环节, 此酶的编码基因nosZ也是研究反硝化微生物的功能标志基因(Thomson et al., 2012).
近年来, 蔬菜种植过程中超量施氮和频繁灌溉造成了氮素在剖面土壤中累积明显, 氮素利用率低且损失严重(王朝辉等, 2002), 而硝化/反硝化耦合过程是土壤氮素损失的最主要驱动(Bao et al., 2012), 土壤含水量变化决定着硝化或反硝化过程及速率(Wu et al., 2015), 一般认为, 50%~70%田间持水量的土壤水分是硝化作用的最佳水分状态(钱琛等, 2010), 而土壤反硝化作用则会随着土壤水分的增加而活跃(Szukics et al., 2010).土壤含水量又影响着硝化或反硝化功能微生物丰度的变化, narG和nosZ基因丰度会随着田面水落干过程的推移而快速增加(卢静等, 2014).此外, 地下水波动对土壤微生物丰度也有较大影响.孙翼飞等(2017)通过模拟水位下降与刈割发现, 两者交互显著影响土壤微生物AOA-amoA、AOB-amoA基因丰度, 且nirS基因丰度显著高于nirK;Tian等(2015)也发现表层土壤中AOA-amoA基因丰度随水位下降而下降, Liu等(2015)也得出三峡消落带无论水位上涨之前还是之后, AOA的基因丰度始终高于AOB.可见, 无论田面水淹水-落干及水位的波动过程均会影响土壤含水量变化, 从而改变着硝化/反硝化微生物功能基因丰度.
洱海湖滨区多年种植蔬菜, 使得菜地土壤中氮素累积严重, 加之该区浅层地下水位浅, 且季节性波动大, 平均水位波幅在10~90 cm左右.水位波动影响着土壤剖面的水分含量和氧化还原环境, 进而影响着参与硝化/反硝化微生物功能基因丰度.目前, 关于影响硝化/反硝化微生物功能基因丰度的研究主要集中于施肥(陈晨等, 2017)、灌溉(陈意超等, 2018)等农艺措施及动物(展鹏飞等, 2018)、人为干扰(孙翼飞等, 2018)等, 对于地下水升降过程中土壤剖面硝化/反硝化微生物功能基因丰度变化的研究鲜见报道.为探究菜地剖面土壤氮转化主要微生物功能基因对浅层地下水升降的响应机制, 本研究以洱海湖滨带菜地剖面土壤为研究对象, 通过模拟地下水升降过程, 并结合实时荧光定量qPCR技术研究了浅层地下水升降过程中硝化/反硝化主要微生物功能基因丰度在土壤剖面中的变化, 揭示了因水位升降引起的土壤环境因子对微生物功能基因丰度的影响, 以期为认识浅层地下水位升降下土壤微生物对菜地土壤剖面氮素循环过程的驱动机制, 减少土壤剖面氮素流失对浅层地下水污染提供科学依据.
2 材料与方法(Materials and methods)2.1 供试土壤样品和采集供试土壤取自云南省大理州下关市洱海湖滨区大庄村蔬菜地(100°12′31″ E, 25°40′14″ N), 海拔为1966 m.气候类型为亚热带高原季风性气候, 年平均降水量1100 mm, 主要集中在6—10月, 约占年降水量的85%~90%, 年平均气温13~20 ℃.土壤类型为水稻土, 属湖积冲积土, 稻田自20世纪80年代中后期开始种植蔬菜, 一年可种3季或以上.取样土壤发生层分为4层, 耕作层(0~30 cm)、犁底层(30~40 cm)、潴育层(40~60 cm)和潜育层(>60 cm), 土壤剖面理化性质见表 1.取样时选取具有代表性的点挖取土壤剖面, 根据土壤发生层分布分层取剖面土壤样, 每层剖面挖的土独立保存, 并立即带回实验室进行土体填充.
表 1(Table 1)
| 表 1 土壤剖面基本理化性质 Table 1 Basic physical and chemical properties of soil profiles | |||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 1 土壤剖面基本理化性质 Table 1 Basic physical and chemical properties of soil profiles
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||
2.2 试验设计试验装置设盛土池(长120 cm、宽57 cm、高110 cm)和位于盛土池两侧的供水池、出水池(长20 cm、宽57 cm、高110 cm)以及水位控制装置.水体由供水池底部入水孔进入盛土池, 出水池和盛土池中间的隔板上设有出水孔, 盛土池中的水通过这些出水孔流入出水池, 超出设计水位的水经出水池安装的水位龙头排出, 整个试验水位由出水池的水位龙头控制, 制作装置的材料为厚1.6 cm的有机玻璃.盛土池底部填充约1 cm厚的石英砂, 底部及两侧用4层孔径为2 mm的尼龙网覆盖, 防止土壤渗漏和确保水流流动, 整个装置四周进行避光处理.然后进行土体构建, 首先去除土样中大的石块, 根据自然土层剖面分布及各层高度和土壤密度进行分层填充, 填充深度为90 cm, 土体构建完成后, 静置2个月, 让构建的土体结构及生化环境充分稳定.
根据对该区域地下水NH4+-N和NO3--N浓度两年的平均监测数据, 设定加入水的NO3--N和NH4+-N浓度分别为15 mg·L-1和0.3 mg·L-1, 主要用KNO3、(NH4)2SO4和去离子水配置.试验开始前, 根据土壤发生层分布, 先用小型土钻取构建好土体的剖面样(编号为A、B、C和D四层), 用于化学指标测定(表 1).设计最高水位为80 cm, 试验从2017年6月中旬开始, 2018年3月结束.试验开始时在供水池中加入配置好的水溶液, 通过入水孔进入中间的盛土池, 通过出水孔进入出水池, 直到水位达到设定的最高水位时, 出水池的水经水位龙头排出.同时要设定好入水孔和出水龙头的水流速率, 确保最高水位保持不变, 连续通水保持最高水位至8月中旬, 这时取高水位剖面土样(S1), 之后慢慢调节水位下降, 直至11月中旬盛土池不再有水渗出, 这时取低水位剖面土样(S2), 然后等待剖面土体落干至2018年3月, 取落干时的剖面样(S3).每个剖面用小型土钻取3个土壤样, 各分两份, 一份存储在-80 ℃冰箱用于微生物功能基因测定, 其余存储于4 ℃冰箱用于分析土壤理化指标.
2.3 硝化/反硝化微生物功能基因和土壤理化性质测定称取0.25 g冻干土, 根据土壤基因组DNA提取试剂盒PowerSoilTM Total DNA Isolation(Mo Bio Laboratories, Inc., San Diego, CA, USA)所提供的操作指南提取土壤微生物总DNA, 并溶解于无菌水后储存在-20 ℃冰箱中待用.
土壤硝化/反硝化主要微生物功能基因定量PCR扩增引物序列和反应条件如表 2所示, 定量测定在iCycler iQ5荧光定量PCR仪(Bio-Red Laboratories, Inc., USA)上进行.采用SYBR GREEN法, 25 μL反应体系中含有12.5 μL的2×SYBR Premix Ex TaqTM(Takara Biotechnology, Japan), 上下游引物各0.5 μL(浓度10 μmoL·L-1)及DNA模板2 μL, 其余用灭菌水补足, 各基因均在72 ℃收集反应荧光信号.然后使用10倍连续稀释提取的质粒DNA测定的浓度构建标准曲线, 计算目的基因和内参基因引物的扩增效率(85%~100%, R2≥0.99), 最后得出目的基因拷贝数.
表 2(Table 2)
| 表 2 定量PCR扩增引物序列及反应条件 Table 2 Amplification primer sequences and reaction condition of quantitative PCR | ||||||||||||||||||||||||
表 2 定量PCR扩增引物序列及反应条件 Table 2 Amplification primer sequences and reaction condition of quantitative PCR
| ||||||||||||||||||||||||
土壤铵态氮(NH4+-N)、硝态氮(NO3--N)采用KCI溶液浸提, 连续流动分析仪测定;pH(土水比1:5)值采用pH计测定;有机质(SOM)、有机碳(SOC)采用重铬酸钾容量法测定;全氮(TN)采用半微量凯氏定氮法测定;土壤含水率(W)采用衡重法测定, 样品先在105 ℃烘16 h;机械组成采用比重计法测定.
2.4 数据处理分析试验数据用Office 2007进行整理, SPSS 20.0进行差异显著性检测及相关性分析, 数据作图在Origin9.0中完成, 并采用CANOCO 5.0软件进行冗余度分析.
3 结果与分析(Results and analysis)3.1 土壤剖面硝化微生物功能基因丰度的变化浅层地下水升降过程中土壤剖面的AOA-amoA和AOB-amoA基因丰度差异较大(图 1), 土壤剖面AOA-amoA和AOB-amoA基因丰度变化范围分别为(0.45~4.53)×106 copies·g-1和(0.57~64.77)×104 copies·g-1, AOA-amoA基因丰度显著(p<0.01)高于AOB-amoA.不同取样时间相同土壤剖面层的AOA-amoA和AOB-amoA基因丰度差异较大, S3阶段的各土壤剖面层AOA-amoA和AOB-amoA基因丰度均显著(p<0.01)高于S1和S2, S1和S2阶段的基因丰度无显著(p>0.05)差异.相同取样时间不同土壤剖面层的AOA-amoA和AOB-amoA基因丰度也有较大差异, 同一取样时间中A、B层的AOA-amoA和AOB-amoA基因丰度均显著(p<0.01)高于C、D层, 其中A层AOB-amoA基因丰度均显著(p<0.05)高于B层, 而A层和B层的AOA-amoA基因丰度及C层和D层中的两种基因丰度无显著(p>0.05)差异.整体来看, S3阶段的AOA-amoA和A、B层的AOB-amoA基因丰度较S1和S2大幅增加, 且随着土层深度的增加AOA-amoA和AOB-amoA基因丰度均降低.
图 1(Fig. 1)
 |
| 图 1 土壤剖面中AOA-amoA (a)、AOB-amoA (b)功能基因丰度 Fig. 1Abundances of functional genes of AOA-amoA and AOB-amoA in soil profiles |
3.2 土壤剖面反硝化微生物功能基因丰度的变化图 2表明了3种反硝化微生物功能基因丰度在不同水位升降期及剖面层均存在较大差异, nirK、nirS和nosZ基因丰度变化范围分别为(1.14~9.19)×106 copies·g-1、(1.18~18.07)×106 copies·g-1和(0.14~4.85)×106 copies·g-1, nirS基因丰度显著(p<0.01)高于nirK、nosZ基因丰度.不同取样时间相同土壤剖面层的反硝化功能基因丰度随水位的下降而降低, S1阶段的各土壤剖面层的nirK、nirS、nosZ基因丰度均显著(p<0.01)高于S2、S3, S1阶段的nirK、nirS、nosZ基因丰度较S2和S3下降较大.相同取样时间不同土壤剖面层反硝化功能基因丰度随土层深度的增加呈下降趋势, 且A、B层的nirK、nirS、nosZ基因丰度显著(p<0.05)高于C、D层, 除nosZ基因丰度在A、B层之间有显著(p<0.05)差异外, 其余基因丰度在A、B层和C、D层之间均不显著(p>0.05).
图 2(Fig. 2)
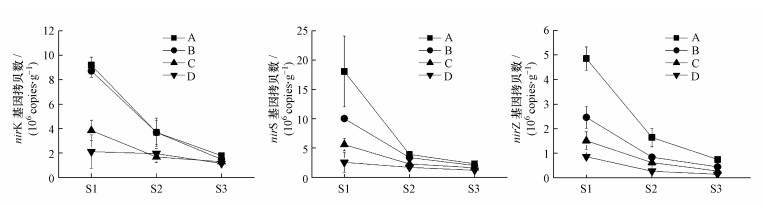 |
| 图 2 土壤剖面中nirK、nirS、nosZ功能基因丰度 Fig. 2Abundance of functional genes of nirK、nirS and nosZ in soil profiles |
3.3 土壤硝化/反硝化微生物功能基因丰度与环境因子的相关性将硝化/反硝化微生物功能基因丰度与土壤环境因子进行RDA分析, 结果(图 3)表明, 第一排序轴解释了43.19%的硝化/反硝化微生物功能基因丰度变化量, 第二排序轴解释了38.83%的变化量, 两个排序轴共解释了82.02%的变化量.所有的环境因子解释了82.9%的功能基因丰度变化信息, 贡献率大小依次为W>TN>pH>NH4+-N>SOC>NO3--N, 其中, W、TN、NH4+-N和pH显著(p<0.05)解释了硝化/反硝化微生物功能基因丰度的变化.图 3和表 3也表明, nirK、nirS、nosZ基因丰度与W、NH4+-N、NO3--N、TN、SOC呈显著正相关关系, 与pH呈显著负相关关系;AOA-amoA、AOB-amoA基因丰度与pH、SOC、TN呈显著正相关关系, 与W呈显著负相关关系.RDA分析也表明土壤环境因子和微生物功能基因呈现明显的土层和水位变化的差异性分布.
图 3(Fig. 3)
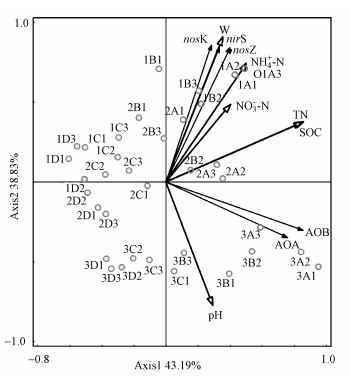 |
| 图 3 土壤硝化/反硝化微生物功能基因丰度与环境因子的RDA分析 Fig. 3RDA analysis of functional genes abundance and environmental factors of soil nitrification and denitrification |
表 3(Table 3)
| 表 3 土壤硝化/反硝化微生物功能基因丰度与环境因子的相关性分析 Table 3 Correlation analysis of functional genes abundance and environmental factors of soil nitrification and denitrification | ||||||||||||||||||||||||||||||||||||||||||||||||
表 3 土壤硝化/反硝化微生物功能基因丰度与环境因子的相关性分析 Table 3 Correlation analysis of functional genes abundance and environmental factors of soil nitrification and denitrification
| ||||||||||||||||||||||||||||||||||||||||||||||||
4 讨论(Discussion)4.1 浅层地下水升降引起的土壤剖面硝化/反硝化微生物功能基因丰度差异分析氨氧化作用在硝化作用中占主导地位, 因此AOA-amoA和AOB-amoA成为硝化作用的标志性功能基因.秦燕(2016)对牧草施肥、切根研究草原土壤得出AOA-amoA和AOB-amoA基因丰度分别位于107~108和104~105, 不同放牧强度下AOA-amoA和AOB-amoA基因丰度达109~1010和106~107, 远高于本研究所得出的基因丰度范围, 说明硝化微生物功能基因对不同土地利用方式及环境因素的响应不同, 水位的升降可能抑制AOA-amoA和AOB-amoA基因发挥活性.S3阶段的各土壤剖面层AOA-amoA和AOB-amoA基因丰度显著高于S1和S2, S2阶段的基因丰度稍高于S1但无显著差异, 说明氨氧化微生物功能基因对水位升降的响应较为敏感, 研究结果与孙翼飞等(2017)、钢迪嘎等(2017)一致, 主要原因:①S3阶段土壤含水量较低, 较适合氨氧化微生物生长, 只有在适宜的水分条件下AOA-amoA和AOB-amoA基因才会变得活跃;②土壤水分对土壤氮矿化速率影响较大, 相对干燥更有利于氮素矿化(Hagedorn et al., 1997), 本研究中, 水位下降及落干3个月后, S3阶段的土壤含水率较低, 促进AOA和AOB的活性;③AOA和AOB为好氧微生物, 而水分通过控制土壤中O2的扩散间接影响好氧微生物活性, 水位下降及落干提高了土壤通气性, 增加土壤孔隙度, 从而减小了O2扩散阻力, 使土壤从缺氧环境过渡为兼氧或好氧环境.同时, 随着土层深度的增加, AOA-amoA和AOB-amoA基因丰度均降低, 这可能与土壤通气性、土壤质地及有机碳含量有关, 随着土层深度增加,O2扩散阻力越大, 缺氧环境也越恶劣, 不同土壤质地的土壤通气性、透水性不同, 从而土壤水分和反应底物浓度也不同;加之随着土层深度的增加有机碳含量变得越低, 越不利于AOA和AOB活性提高, 与岳中辉等(2007)、张建贵等(2017)研究结果一致.S3阶段的AOA-amoA和A、B层的AOB-amoA基因丰度大幅增加, 进一步说明水位升降显著影响着硝化微生物功能基因丰度;S3阶段的C、D层AOB-amoA基因丰度增幅并不明显, 主要是因为土壤中有机碳含量较低所致.此外, AOA-amoA基因丰度显著高于AOB-amoA基因丰度, 主要与供试土壤pH处于5.13~6.56之间呈酸性有关, 与Shen等(2012)、Zhang等(2012)证实的AOA是酸性土壤中起主导作用的结果一致.这可能是因为AOA对水分的响应较AOB灵敏(Bustamante et al., 2012), 导致AOA-amoA基因丰度远高于AOB-amoA.
土壤氮的反硝化作用过程复杂, 参与的微生物及功能基因较多, 其主要受nirK、nirS、nosZ等功能基因驱动.本研究结果显示nirK、nirS、nosZ基因丰度均处于106~107之间, 与大多数****研究结果并不一致(刘若萱等, 2014; 孙翼飞等, 2017), 可能是土壤水分、有机碳含量和土壤机械组成差异较大所致.刘若萱等(2014)研究表明, nirK、nirS功能基因丰度随水分的增加而增加, 反硝化作用也随水分的增加而增加(Szukics et al., 2010), 本研究中S1阶段的土壤剖面层nirK、nirS、nosZ基因丰度均显著高于S2、S3, 研究结果与刘若萱等(2014)基本一致.这与土壤水分和底物浓度密切相关, 反硝化作用在厌氧或兼氧条件下普遍发生, 淹水过程便为反硝化微生物创造了厌氧生长的有利条件, 由S1阶段过渡到S3阶段, 也就是缺氧环境过渡到好氧环境及底物浓度降低的过程, 且农业土壤中O2含量对反硝化微生物有明显的抑制作用;通过Pearson与RDA分析也表明, NH4+-N、NO3--N与nirK、nirS、nosZ基因丰度均呈显著正相关, 本研究用水中含NH4+-N、NO3--N, 这就提供了充足的反应底物, 增加了nirK、nirS、nosZ基因丰度, 且反硝化微生物功能基因有一个最适宜水位, 当水位进一步下降时, 则不利于其活动.Mergel等(2001)研究发现反硝化微生物功能基因在0~5 cm土层最高, 并随深度增加而降低, 本研究也发现nirK、nirS、nosZ基因丰度也随土层深度的增加呈下降趋势.这可能与底物浓度、有机碳含量及土壤机械组成密切有关, 随着土层深度的增加, 土壤中NH4+-N、NO3--N、SOC均呈下降趋势, 有机质、土壤机械组成的差异导致各土层保水保肥等能力也不同, 由本研究各土层有机质、机械组成可知, 随着土层深度的增加土壤保水保肥能力均降低, 导致底物浓度也降低, 以致于nirK、nirS、nosZ基因丰度降低.相比于S1, S2和S3阶段的nirK、nirS、nosZ基因丰度大幅下降, 说明水位升降显著影响着土壤反硝化微生物功能基因丰度.本研究中nirS基因丰度显著高于nirK、nosZ基因丰度, 与孙翼飞等(2017)、Zheng等(2014)、Ligi等(2014)研究结果一致, 说明nirS基因对水位升降及水分变化响应更敏感, 且在反硝化作用中占有主导地位, 不同的反硝化微生物功能基因对环境因素响应也不同.
4.2 土壤硝化/反硝化微生物功能基因丰度与环境因子的关系土壤硝化与反硝化微生物功能基因丰度受土壤中多环境因子的影响, RDA和相关性分析(图 3和表 3)表明, AOA-amoA、AOB-amoA、nirK、nirS、nosZ基因丰度与W、NH4+-N、NO3--N、TN、SOC和pH均存在不同程度的相关性, 且W、TN、pH为主要环境驱动因子.Hayden等(2010)研究表明, 影响AOA-amoA、AOB-amoA基因丰度的环境驱动因子为pH、AP、SOC、NO3--N和TN, 杨丹等(2015)、谢开治等(2016)也得出AOA-amoA、AOB-amoA基因丰度与土壤pH、SOC、TN、NH4+-N、NO3--N呈显著正相关;但也有研究(Shen et al., 2012)指出, 在区域尺度上AOA-amoA、AOB-amoA基因丰度与NH4+-N、NO3--N含量均无明显相关关系.本研究结果中AOA-amoA、AOB-amoA基因丰度仅与土壤pH、SOC、TN呈显著正相关, 而与NH4+-N、NO3--N无显著相关性, 这可能是因为两种功能基因的生长更易于受有机氮而非无机氮的刺激(贺纪正等, 2013), 此外, 除区域土壤环境及理化性质有较大影响外, 不同土地利用类型土壤中AOA-amoA和AOB-amoA对环境条件变化响应也不同, 从而综合导致AOA-amoA、AOB-amoA基因丰度与NH4+-N、NO3--N无显著相关性.反硝化微生物功能基因丰度受pH、O2含量、W、SOC和养分状况等土壤因子的影响(Bulow et al., 2010), 本研究结果显示, nirK、nirS、nosZ基因丰度与W、NH4+-N、TN、SOC呈显著正相关, 研究结果与孙翼飞等(2018)、解开治等(2016)的结果一致.此外, 本研究中nirK、nirS、nosZ基因丰度与pH呈显著负相关, 与NO3--N含量呈显著正相关, 与解开治等(2016)研究结果并不一致.这可能是由于反硝化微生物功能基因对pH的响应较为敏感, 只有在适宜的pH值范围内才适合生长, 而本研究中土壤pH偏酸性, 从而对反硝化微生物功能基因起到抑制作用;Wallenstein等(2006)、Dandie等(2008)研究指出, 土壤反硝化微生物功能基因丰度并不受NO3--N浓度的影响, 但也有****(Bremer et al., 2007; 贺纪正等, 2013)认为, NO3--N浓度是土壤反硝化微生物功能基因丰度的主要影响因子, 而国内众多****对反硝化单个功能基因丰度与NO3--N浓度的相关性研究也得出两者呈显著正相关.由此可推测, 造成研究结果不一致的原因可能与外界环境因素和土壤类型(Di et al., 2010; 崔培媛, 2016)及两者的交互作用等有关, 因此对于NO3--N浓度对反硝化微生物功能基因丰度的影响有待进一步研究.
5 结论(Conclusions)1) 水位升降对土壤剖面硝化微生物功能基因AOA-amoA、AOB-amoA和反硝化微生物功能基因nirK、nirS、nosZ丰度有显著影响, AOA-amoA和AOB-amoA基因丰度随水位下降而增加, nirK、nirS、nosZ基因丰度随水位下降而降低;不同取样时期土壤剖面硝化和反硝化功能基因丰度均表现为A层>B层>C层>D层.
2) 土壤pH、有机碳(SOC)、全氮(TN)为土壤硝化微生物功能基因AOA-amoA、AOB-amoA的环境驱动因子;反硝化微生物功能基因nirK、nirS、nosZ的环境驱动因子为W、NH4+-N、NO3--N、TN、SOC、pH.
参考文献
| Arp D J, Stein L Y. 2003. Metabolism of inorganic N compounds by ammonia-oxidizing bacteria[J]. Critical Reviews in Biochemistry and Molecular Biology, 38(6): 471–495.DOI:10.1080/10409230390267446 |
| Avrahami S, Conrad R, Braker G. 2003. Effect of ammonium concentration on N2O release and on the community structure of ammonia oxidizers and denitrifiers[J]. Applied and Environmental Microbiology, 69(5): 5685–5692. |
| Bao Q, Ju X, Gao B, et al. 2012. Response of nitrous oxide and corresponding bacteria to managements in an agricultural soil[J]. Soil Science Society of America Journal, 76(1): 130.DOI:10.2136/sssaj2011.0152 |
| Braker G, Ayala-Del-Rio H L, Devol A H, et al. 2001. Community structure of denitrifiers, bacteria, and archaea along redox gradients in pacific northwest marine sediments by terminal restriction fragment length polymorphism analysis of amplified nitrite reductase (nirS) and 16S rRNA genes[J]. Applied and Environmental Microbiology, 67(4): 1893–1901.DOI:10.1128/AEM.67.4.1893-1901.2001 |
| Bustamante M, Verdejo V, Zú?iga C, et al. 2012. Comparison of water availability effect on ammonia-oxidizing bacteria andarchaea in microcosms of a Chilean semiarid soil[J]. Frontiers in Microbiology, 3(3): 282. |
| Bulow S E, Francis C A, Jackson G A, et al. 2010. Sediment denitrifier community composition and nirS gene expression investigated with functional gene microarrays[J]. Environmental Microbiology, 10(11): 3057–3069. |
| Bremer C, Braker G, Matthies D, et al. 2007. Impact of plant functional group, plant species, and sampling time on the composition of nirK-type denitrifier communities in soil[J]. Applied and Environmental Microbiology, 73(21): 6876–6884.DOI:10.1128/AEM.01536-07 |
| 陈晨, 许欣, 毕智超, 等. 2017. 生物炭和有机肥对菜地土壤N2O排放及硝化、反硝化微生物功能基因丰度的影响[J]. 环境科学学报, 2017, 37(5): 1912–1920. |
| 陈意超, 李伏生, 李烙布. 2018. 不同灌溉方式和尿素猪粪比例对稻田氮素转化相关微生物活性的影响[J]. 华南农业大学学报, 2018, 39(1): 31–39. |
| 崔培媛. 2016.施肥、硝化抑制剂和温度对农田土壤N2O释放的影响及其微生物驱动机制[D].北京: 中国农业科学院 |
| Dandie C E, Burton D L, Zebarth B J, et al. 2008. Changes in denitrifier community abundance over time in an agricultural fied and their relationship with denitrification activity[J]. Applied and Environmental Microbiology, 74(19): 5997–6005.DOI:10.1128/AEM.00441-08 |
| Di H J, Cameron K C, Shen J P, et al. 2010. A lysimeter study of nitrate leaching from grazed grassland as affected by a nitrification inhibitor, dicyandiamide, and relationships with ammonia oxidizing bacteria and archaea[J]. Soil Use & Management, 25(4): 454–461. |
| 钢迪嘎, 齐维晓, 刘会娟, 等. 2017. 水位变化对消落带氨氧化微生物丰度和多样性的影响[J]. 环境科学学报, 2017, 37(5): 1615–1622. |
| Hagedorn F, Steiner K G, Sekayange L, et al. 1997. Effects of rainfall pattern on nitrogen mineralization and leaching in green manure experiment in South Rwanda[J]. Plant & Soil, 195: 365–375. |
| Hayden H L, Drake J, Imhof M, et al. 2010. The abundance of nitrogen cycle genes amoA and nifH depends on land-uses and soil types in South-Eastern Australia[J]. Soil Biology & Biochemistry, 42(10): 1774–1783. |
| 贺纪正, 张丽梅. 2013. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98–108. |
| Kowalchuk G A, Stephen J R. 2001. Ammonia-oxidizing bacteria: a model for molecular microbial ecology[J]. Annual Review of Microbiology, 55(1): 485.DOI:10.1146/annurev.micro.55.1.485 |
| 刘若萱, 贺纪正, 张丽梅. 2014. 稻田土壤不同水分条件下硝化/反硝化作用及其功能微生物的变化特征[J]. 环境科学, 2014, 1(11): 4275–4283. |
| 卢静, 刘金波, 盛荣, 等. 2014. 短期落干对水稻土反硝化微生物丰度和N2O释放的影响[J]. 应用生态学报, 2014, 25(10): 2879–2884. |
| Liu S, Hu B L, He Z F, et al. 2015. Ammonia-oxidizing archaea have better adaptability in oxygenated/hypoxic alternant conditions compared to ammonia-oxidizing bacteria[J]. Applied Microbiology and Biotechnology, 99(20): 8587–8596.DOI:10.1007/s00253-015-6750-7 |
| Ligi T, Truu M, Truu J, et al. 2014. Effects of soil chemical characteristics and water regime on denitrification genes (nirS, nirK, and nosZ) abundances in a created riverine wetland complex[J]. Ecological Engineering, 72: 47–55.DOI:10.1016/j.ecoleng.2013.07.015 |
| Molina V, Ulloa O, Farias L, et al. 2007. Ammonia-oxidizing beta-proteobacteria from the oxygen minimum zone off northern Chile[J]. Applied and Environmental Microbiology, 73(11): 3547–3555.DOI:10.1128/AEM.02275-06 |
| Morales S E, Cosart T, Holben W E. 2010. Bacterial gene abundances as indicators of greenhouse gas emission in soils[J]. The Isme Journal, 4(6): 799–808.DOI:10.1038/ismej.2010.8 |
| Mergel A, Schmitz O, Mallmann T, et al. 2001. Relative abundance of denitrifying and dinitrogen-fixing bacteria in layers of a forest soil[J]. FEMS Microbiology Ecology, 36(1): 33–42.DOI:10.1111/j.1574-6941.2001.tb00823.x |
| 钱琛, 蔡祖聪. 2010. 利用方式对红壤硝化作用的水分效应的影响[J]. 环境科学, 2010, 31(10): 2417–2422. |
| 秦燕. 2016.半干旱羊草草原土壤氮素转化与关键微生物特性研究[D].北京: 中国农业科学院 |
| Rotthauwe J H, Witzel K P, Liesack W. 1997. The ammonia monooxygenase structural gene amoA as a functional marker: molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 63(12): 4704–4712. |
| Szukics U, Abell G C J, H?dl V, et al. 2010. Nitrifiers and denitrifiers respond rapidly to changed moisture and increasing temperature in a pristine forest soil[J]. Fems Microbiology Ecology, 72(3): 395–406.DOI:10.1111/j.1574-6941.2010.00853.x |
| 孙翼飞, 沈菊培, 张翠景, 等. 2017. 模拟水位下降与刈割对高寒湿地土壤氨氧化与反硝化微生物的影响[J]. 农业环境科学学报, 2017, 36(11): 2356–2364.DOI:10.11654/jaes.2017-0589 |
| 孙翼飞, 沈菊培, 张翠景, 等. 2018. 不同放牧强度下土壤氨氧化和反硝化微生物的变化特征[J]. 生态学报, 2018, 38(8): 2874–2883. |
| Scala D J, Kerkhof L J. 2000. Horizontal heterogeneity of denitrifying bacterial communication in sediments by terminal restriction fragment length polymorphism analysis[J]. Applied and Environmental Microbiology, 66(5): 1980–1986.DOI:10.1128/AEM.66.5.1980-1986.2000 |
| Shen J P, Zhang L M, Di H J, et al. 2012. A review of ammonia-oxidizing bacteria and archaea in Chinese soil[J]. Frontiers in Microbiology, 3: 296. |
| Thomson A J, Giannopoulos G, Pretty J, et al. 2012. Biological sources and sinks of nitrous oxide and strategies to mitigate emissions[J]. Philosophical Transactions of the Royal Society of London, 367(1593): 57–68. |
| Tian J, Shu C, Chen H, et al. 2015. Response of archaeal communities to water regimes under simulated warming and drought conditions in Tibetan Plateau wetlands[J]. Journal of Soils and Sediments, 15(1): 179–188.DOI:10.1007/s11368-014-0978-1 |
| Francis C A, Roberts K J, Beman M, et al. 2005. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. PNAS, 102(41): 14683–14688.DOI:10.1073/pnas.0506625102 |
| 王朝辉, 宗志强, 李生秀, 等. 2002. 蔬菜的硝态氮累积及菜地土壤的硝态氮残留[J]. 环境科学, 2002, 23(3): 79–83. |
| Wu L S, Feng S, Nie Y Y, et al. 2015. Soil cellulase activity and fungal community responses to wetland degradation in the Zoige Plateau, China[J]. Journal of Mountain Science, 12(2): 471–482.DOI:10.1007/s11629-014-3183-1 |
| Wallenstein M D, Myrold D D, Voytek F M. 2006. Environmental controls on denitrifying communities and denitrification rates: insights from molecular methods[J]. Ecological Applications, 16(6): 2143–2152.DOI:10.1890/1051-0761(2006)016[2143:ECODCA]2.0.CO;2 |
| Xia J, Niu S, Wan S. 2010. Response of ecosystem carbon exchange to warming and nitrogen addition during two hydrologically contrasting growing seasons in a temperate steppe[J]. Global Change Biology, 15(6): 1544–1556. |
| 解开治, 徐培智, 蒋瑞萍, 等. 2016. 有机无机肥配施提升冷浸田土壤氮转化相关微生物丰度和水稻产量[J]. 植物营养与肥料学报, 2016, 22(5): 1267–1277. |
| 岳中辉, 张兴义, 许景刚, 等. 2007. 黑土硝酸还原酶活性的分布特征[J]. 东北农业大学学报, 2007, 38(6): 767–770.DOI:10.3969/j.issn.1005-9369.2007.06.012 |
| 杨丹, 余旋, 刘旭, 等. 2015. 栽培模式对沙棘人工林土壤微生物群落结构和参与氮循环功能基因的影响[J]. 应用生态学报, 2015, 26(12): 3634–3640. |
| 展鹏飞, 肖德荣, 闫鹏飞, 等. 2018. 藏猪扰动作用下的高寒草甸土壤退化特征及微生物群落结构变化[J]. 环境科学, 2018, 39(4): 1840–1850. |
| 张建贵, 将永梅, 姚拓, 等. 2017. 不同管理措施对高寒草甸土壤微生物氮素生理群数量影响研究[J]. 草业学报, 2017, 26(8): 65–73. |
| Zhang L M, Hu H W, Shen J P, et al. 2012. Ammonia-oxidizing archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. The ISME Journal, 6(5): 1032–1045.DOI:10.1038/ismej.2011.168 |
| Zheng Y, Yang W, Hu H W, et al. 2014. Ammonia oxidizers and denitrifiers in response to reciprocal elevation translocation in an alpine meadow on the Tibetan Plateau[J]. Journal of Soils and Sediments, 14(6): 1189–1199.DOI:10.1007/s11368-014-0867-7 |
