中国科学院动物研究所张勇研究组及合作者以黑腹果蝇和人类这两个内含子分布和特性迥异的系统为模型深入探讨了上述两个问题,相关成果于2021年12月31日在Nature Ecology & Evolution(NEE)杂志发表,论文标题为“Dosage sensitivity and exon shuffling shape the landscape of polymorphic duplicates in Drosophila and humans”(论文链接:https://dx.doi.org/10.1038/s41559-021-01614-w)。
在本研究中,该团队基于自己实验室产出的6个果蝇品系的重测序数据,鉴定了270个多态性重复基因;同时,利用人类数据库GTEx鉴定了964个重复基因(图1)。根据基因重复发生区段,研究人员将重复基因分为完整重复基因、部分重复基因和内含子重复基因(图2A)。果蝇和人类中数量最多的分别是部分重复基因和内含子区重复基因,随机模拟显示这一分布差异可以被不同物种基因组外显子、内含子的相对比例和重复区间与基因的相对长度分布所解释。
研究人员通过转录组数据分析,发现三类重复基因中只有完整重复基因的表达显著上调(图2B)。蛋白复合体成员基因在完整重复基因中相对较少(图2C)。X染色体连锁剂量补偿基因是另一类剂量敏感基因,果蝇中X染色体的完整重复基因在雄性和雌性脂肪体中表达水平相当(图2D),暗示果蝇的X染色体剂量补偿体系可以容忍基因拷贝数上升。与果蝇不同,人类X染色体编码的完整重复基因倾向于性别特异表达,无需剂量补偿。
不完整重复可介导嵌合基因的形成。为检测嵌合基因,研究人员根据重复断点的位置,将嵌合基因分为6类。由于果蝇基因密度高,其基因融合(5’-3’,5’-5’,3’-3’)的比例均高于人类;反之由于人类基因较长,其内部重复的比例更高(图3A上)。研究人员通过转录本拼接发现果蝇和人类分别有66.7%和26.5%的嵌合基因能活跃转录(图3A下)。在6类嵌合基因中,5’-嵌合基因因其携带启动子区域重复,往往更容易被转录(图3A下)。值得注意的是,基因内部重复导致外显子洗牌而改变母基因的编码区,即该基因往往进化出新可变剪切形式(图3B):一种是只包含一个重复单元的与祖先基因相同的短转录本,一种是包含两个重复单元的长转录本。基于测序读长深度和qPCR的相对定量方法发现,生物体往往主要表达短转录本(图3C),这可能是个体维持重复发生之前基因结构的巧妙途径。与果蝇相比,人类基因组中内含子数量多且长度较长,其基因内部重复更多的使用位于内含子的断点,导致更多的维持原读码框的融合蛋白基因的产生。
最后,研究人员以提前终止密码子(受负选择)和同义突变(受中性选择)为参照物开展分析,发现物种中完整重复基因和部分重复基因均受到强烈的负选择,暗示它们带来的剂量上升或新的嵌合结构通常都是有害的(图4)。值得注意的是,果蝇里内含子重复也受负选择,而人类内含子重复其选择压力更接近中性;这可能是因为后者其内含子长度更大,对小的突变不敏感。
整体来看,该工作通过深入分析两个差异较大的动物体系,系统展示了剂量敏感性和外显子洗牌过程如何塑造重复基因演化的最早期蓝图。该工作不仅将新基因起源的两个主要机制即基因重复和外显子洗牌融合到一起 [8];同时也说明Gilbert 43年前提出的两个核心概念相辅相成 [7],即可变剪切是基因内部重复发生的前提,使得更复杂的蛋白结构可以在不影响此前基因结构的前提下演化出来。
张勇研究员长期致力于重复机制导致的新基因起源研究,发现新基因不仅推动了人脑发育过程的演化(Genome Research 2019),也参与了人类特异的红细胞发育调控网络(Developmental Cell 2021);NEE这项最新成果和其早前LTR逆转座子及DNA转座子(Genome Research 2016、Nature Communications 2021)介导基因重复的工作则一起组成了该团队关于突变机制如何影响重复基因演化的系列性探索。本项研究由中国科学院动物研究所、中国科学院北京基因组研究所、中国中医科学院中药研究所等机构合作完成,动物所张勇研究员为通讯作者。动物所博士生张丹、中药研究所博士后冷梁为本研究共同第一作者,动物所陈春燕、黄家蔚、张雅琼、袁昊和马辰宇和基因组所陈华研究员等共同协作。该研究得到了国家重点研发计划(2018YFC1406902和2019YFA0802600)、中国科学院(DBS-LY-SM005, XBZG-ZDSYS-201913和XDPB17)、国家自然科学基金(31771410和31970565)、北京脑科学与类脑研究中心开放研究项目等的资助。
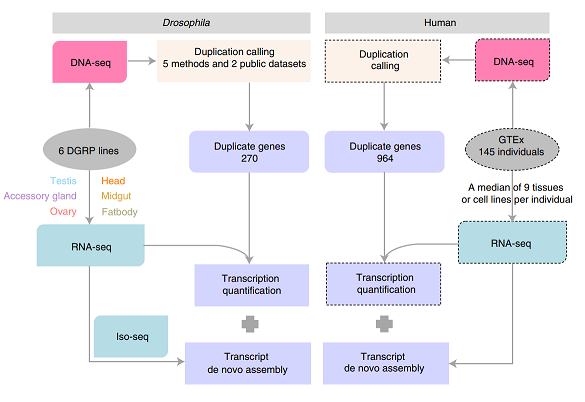
图1. 项目总体设计图。果蝇的重复基因数据集来源于对自产重测序数据的分析,人类的重复基因数据集基于公开数据GTEx。转录组测序用于检测重复基因的表达水平和基因结构的变化。
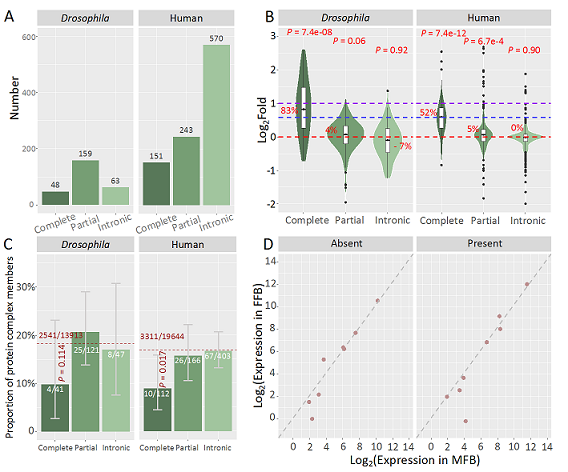
图2. 重复基因表达差异的分布和剂量效应。A)不同种类重复基因的数量分布;B)不同种类重复基因转录水平改变幅度的分布;C)不同种类重复基因中蛋白复合体基因的比例,红色虚线为蛋白复合体基因在所有蛋白编码基因中的比例;D)8个X染色体连锁的完整重复基因在基因重复存在个体和基因重复缺失个体中的表达分布,虚线对角线表示在雌性脂肪体和雄性脂肪体中基因的表达相等。
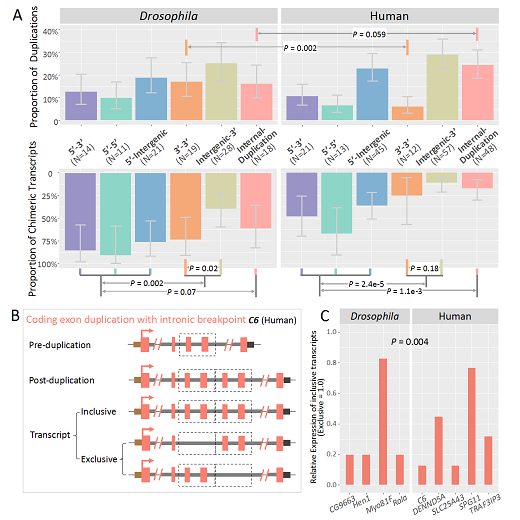
图3. 嵌合基因和不完整重复基因的分布及特点。A)根据断点位置预测的嵌合基因(上)和基于转录本拼接验证的活跃转录嵌合基因(下)比例;B)人类基因内部重复示例。C6基因中间的2个外显子发生串联重复。转录本拼接结果显示存在两种转录本:包含两个重复单元的长转录本(上)和只包含一个单元的短转录本(下);C)相比之下,长转录本表达水平较低。
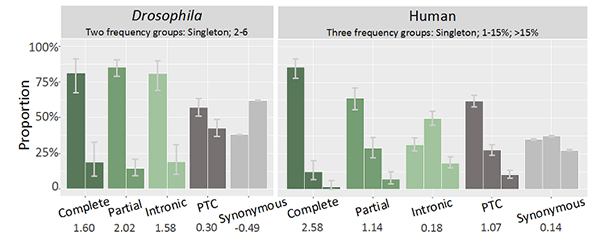
图4. 不同类型重复基因频率的分布。果蝇和人类不同重复基因、提前终止子和同义突变的等位基因频率。数字表示偏度值(skewness)。
参考文献:
1. Muller, H.J., Bar Duplication. Science, 1936. 83(2161): p. 528-30.
2. Ohno, S., Evolution by gene duplication. 1970: London: George Alien & Unwin Ltd. Berlin, Heidelberg and New York: Springer-Verlag.
3. Zhang, J., Evolution by gene duplication: an update. Trends in ecology & evolution, 2003. 18(6): p. 292-298.
4. Innan, H. and F. Kondrashov, The evolution of gene duplications: classifying and distinguishing between models. Nature Reviews Genetics, 2010. 11(2): p. 97-108.
5. Chen, S.D., B.H. Krinsky, and M.Y. Long, New genes as drivers of phenotypic evolution. Nature Reviews Genetics, 2013. 14(9): p. 645-660.
6. Kuzmin, E., J.S. Taylor, and C. Boone, Retention of duplicated genes in evolution. Trends in Genetics, 2021.
7. Gilbert, W., Why Genes in Pieces. Nature, 1978. 271(5645): p. 501-501.
8. Long, M.Y., et al., New Gene Evolution: Little Did We Know. Annual Review of Genetics, 2013. 47: p. 307-333.
| 附件下载: | |
