中国科学院动物研究所魏辅文院士团队联合西北工业大学王文教授团队、浙江大学周琦教授团队及国内众多科研和保护机构测序组装了具有祖先核型的獐(2n=70),小麂(2n=46),雌性和雄性黑麂(2n=8/9)的染色体水平基因组,以及黑麂最近缘物种贡山麂的草图基因组。全基因组比对分析证实染色体融合是导致麂属染色体数目在种间剧烈变化的主要重排类型。经过比较基因组分析发现黑麂和贡山麂在1.44百万年前产生分化。种群历史动态分析发现黑麂、赤麂和贡山麂在聂聂雄拉冰期期间种群数量明显降低,而同时期小麂种群数量没有下降,研究推测由冰期引发的遗传漂变和地理隔离加速麂属物种染色体融合的固定(图1)。
研究阐明了由一个复杂重复序列结构介导的染色体串联融合机制。该重复序列结构包含一个短的端粒类似序列,两侧有回文序列,再两侧分别是鹿科I类和IV类卫星序列(图2)。研究比较了獐、小麂和黑麂这三种染色体数目呈阶梯变化的物种基因组三维构象,发现尽管染色体数目差异巨大,但是它们在染色体区室(compartment A/B)层面相差甚小,表明染色体剧烈演化对这些物种大尺度三维构象的影响很小。另外,研究通过比较neo-X和neo-Y倒位区的多种序列特征,发现在neo-Y倒位区聚集了大量染色质构象的转换(compartment switch),且这些转换已经影响了其内部基因表达的水平。相比之下,拓扑相关域则在neo-Y倒位区内仍然保持稳定。这些结果提示早期Y染色体在大尺度染色质结构上可能发生了基因表达的改变,随后才跟随基因的退化和剂量补偿效应的出现。
研究成果以“Molecular mechanisms and topological consequences of drastic chromosomal rearrangements of muntjac deer”为题于2021年11月25日在Nature Communications杂志在线发表。中科院动物研究所魏辅文院士、西北工业大学王文教授和浙江大学周琦教授为共同通讯作者;西北工业大学博士生殷源、中科院动物研究所樊惠中助理研究员、西北工业大学博士生周博童、中科院动物研究所胡义波研究员、华大基因范广益博士、武汉菲沙基因周帆、中科院昆明细胞库王金焕和佴文惠研究员为共同第一作者。该项目得到了国家自然科学基金、中科院B类战略先导专项、西北工业大学专项经费、钱江源国家公园和生态环境部生物多样性调查、观测与评估等项目的支持。
全文链接:https://www.nature.com/articles/s41467-021-27091-0
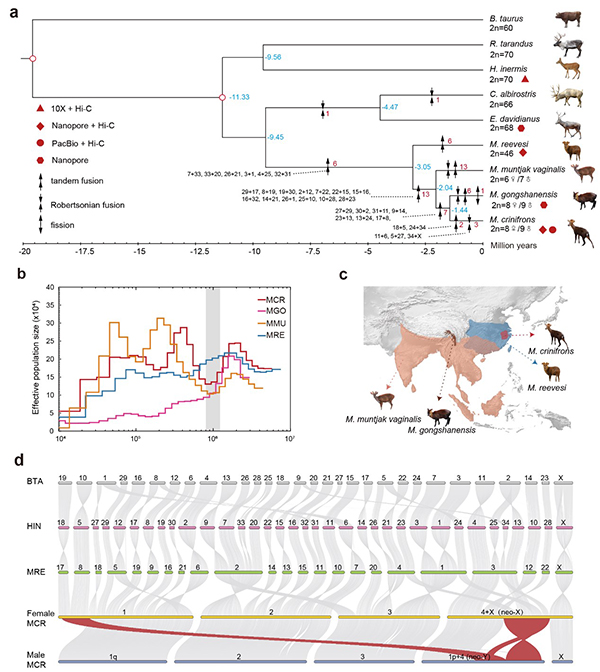
图1. 麂属物种系统发育关系、种群历史动态、物种分布及基因组共线性。a, 麂属物种系统发育关系。箭头表示染色体重排事件,蓝色数字表示物种分歧时间;b, 麂属物种种群历史动态(MCR:黑麂、MGO:贡山麂、MMU: 赤麂、MRE: 小麂);c, 麂属物种分布图;d, 牛、獐、小麂、雌性和雄性黑麂基因组共线性图谱。
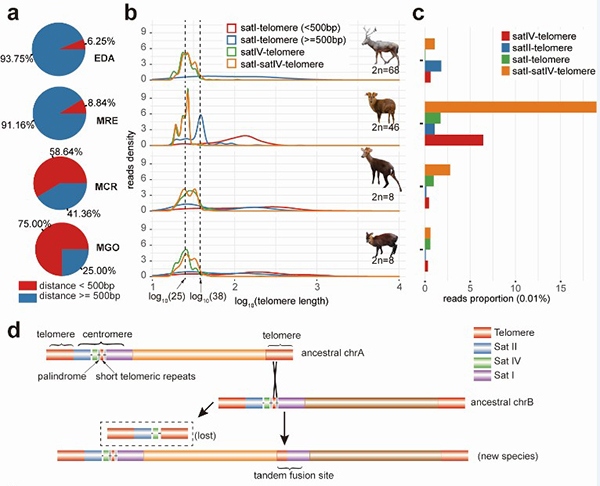
图2. 麂属动物染色体融合的分子基础。a, 一类卫星序列和端粒序列距离大于或小于500bp的两种三代reads在各物种内的比例(EDA: 獐、MRE: 小麂、MCR:黑麂、MGO:贡山麂);b, 各物种内,包含不同卫星序列和端粒序列的三代reads 随端粒序列长度的密度分布;c, 包含不同卫星序列和端粒序列的三代reads在各物种内所占的数量比例;d, 推测的短端粒类似序列介导染色体融合的分子机制。
| 附件下载: | |
