

中国科学院大学材料科学与光电技术学院, 北京 100049
2018年3月30日 收稿; 2018年5月2日 收修改稿
基金项目: 国家自然科学基金(21274164)和国家重点基础研究(973)项目(2014CB931900)资助
通信作者: 李晓毅, E-mail:lixy@ucas.ac.cn
摘要: 细胞色素c是一种广泛存在在细胞线粒体中的富电子蛋白。它在与纳米材料相互作用的过程中引发自由基的生成,扩大纳米材料的生物毒性。为了在分子尺度上理解其与纳米材料的相互作用,通过分子动力学模拟研究细胞色素c与富勒烯衍生物体系(富勒醇、三丙二酸富勒烯)。分析吸附后体系的能量、均方回转半径、接触原子数以及细胞色素c中的Fe原子到富勒烯衍生物质量中心的距离,进而揭示细胞色素c与富勒烯衍生物相互作用机理。
关键词: 细胞色素c富勒醇三丙二酸富勒烯分子动力学模拟
Adsorption interaction between cytochrome c and fullerene derivatives
ZHANG Yujie, LI Xiaoyi


College of Materials Science and Opto-electronics Technology, University of Chinese Academy of Sciences, Beijing 100049, China
Abstract: Cytochrome c is an electron-rich protein that widely exists in cell mitochondria. In the interaction process with nanomaterials, it generates free radicals and amplifies the biological toxicity of nanomaterials. In order to understand its interaction with nanomaterials, molecular dynamics simulations are conducted on cytochrome c and fullerene derivative systems including fullerene trimalonate and fullerol. We have studied the interaction energy, the mean radius of gyration, the number of contacting atoms, and the distance from the Fe atom in cytochrome c to the mass center of the fullerene derivative. As a result, mechanism of the interaction between cytochrome c and fullerene derivatives is revealed at the atomic dimension.
Keywords: cytochrome cfullerolfullerene trimalonatemolecular dynamics simulation
蛋白质吸附是基础而重大的问题。当外来的纳米粒子进入细胞和血浆,首先会发生蛋白质的吸附[1]。纳米粒子能够穿过细胞膜进入细胞,在器官中聚集并引发毒性反应[2]。因此研究蛋白质的吸附对于揭示纳米粒子的生物效应是必不可少的。同时,蛋白质吸附与一系列生物传感器件紧密联系,如药物运输和癌症治疗等[3-7]。因此,基于安全性和潜在应用等方面的考虑,蛋白质的吸附具有十分重要的意义。
细胞色素c是生命中一种重要的水溶性氧化还原蛋白,分子内部包含一个血红素(中心卟啉铁),广泛存在原核生物和真核生物细胞的线粒体内膜上。细胞色素c中心的血红素是电子转移的核心,在细胞能量消耗、生长和分化中有着重要的作用[8-9]。
在过去的30年,富勒烯及其衍生物在多个领域具有广泛的应用,包括神经保护、抗氧化、抗菌和抗病毒活性[10-11]。富勒烯的球状结构具有强烈的非极化特性,可以形成容易穿过细胞膜的脂质体系[12]。在生物技术中,它已被广泛应用于药物运输和生物传感器等领域。其衍生物富勒醇和三丙二酸富勒烯是通过在富勒烯的碳上引入羟基和羧基而得到,其官能团分别为羟基和羧基,从而提高富勒烯的水溶性。
本文主要利用分子动力学模拟,研究细胞色素c和富勒醇(分子式为C60(OH)24)[13]、三丙二酸富勒烯(分子式为C60(C(COOH)2)3的吸附作用,为研究二者之间进一步的物理化学反应提供基础。
1 计算方法体系中的蛋白质为马心细胞色素c(Cytc,1HRC), 其初始结构是从蛋白库中获得。富勒烯衍生物是由Materials Studio软件搭建。体系中的水分子模型为TIP3P,体系到水盒子边界的距离为1 nm。为了模拟生理环境,体系中添加Na+和Cl-, 浓度为0.15 mol/L。采用CHARMM[14]力场,所用的软件为NAMD[15]。模拟过程是在NPT(1 atm, 300 K)系综下进行的。温度和压强分别用Berendsen恒温和恒压方法保持稳定。整个模拟采用周期性边界条件。
2 结果与讨论蛋白质的结构决定蛋白质的生物活性和生物功能。为了度量蛋白质的结构,本文分析相互作用能、均方根偏差(root mean square deviation, RMSD)、均方回转半径(radius of gyration, Rg)随吸附模拟时间的变化曲线。其中,RMSD为蛋白质在这个时刻的构象与初始构象进行对比,使之与初始构象的重心重合,然后计算每个对应原子的坐标的差值,把差值平方再平均,最后开根号。它表示蛋白质结构随时间的演化。Rg为蛋白分子内的每个原子到蛋白质质心距离的平方的平均值,表示蛋白质在空间的伸展程度。如图 1(a)所示,P1(细胞色素c与富勒醇)和P2(细胞色素c与三丙二酸富勒烯)的相互作用能随着模拟时间的变化,一直稳定在某一个值左右,没有大幅度的变化,说明体系处于稳定的状态,同时也说明15 ns的分子动力学模拟对这两个体系也是足够的。从图 1(b)可以看出,两个体系在15 ns的模拟时间内,细胞色素c的RMSD值先处于上下波动的状态,最后逐渐趋于稳定,说明体系结构已经达到平衡。该结果与均方回转半径得到的结果一致。细胞色素c的结晶回转半径为1.324 nm,在水溶液中为1.333 nm,细胞色素c在吸附富勒烯衍生物之后,所有的回转半径都在1.3~1.4 nm之间,P1的平均回转半径为1.312 nm,P2的平均回转半径为1.314 nm,如表 1所示。表明细胞色素c结构的稳定性。
Fig. 1
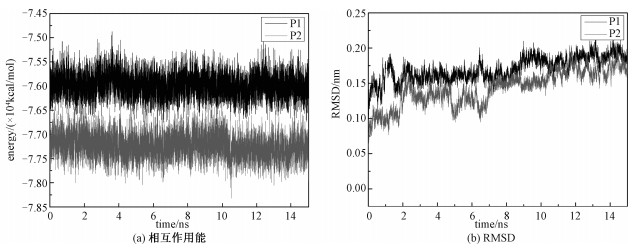 | Download: JPG larger image |
| 图 1 相互作用能及RMSD随时间的变化 Fig. 1 Temporal evolution of the interaction energy (a) and RMSD (b) 图 1 相互作用能及RMSD随时间的变化 Fig. 1 Temporal evolution of the interaction energy (a) and RMSD (b) --> | |
Table 1
| 表 1 体系的均方回转半径、距离和接触原子数Table 1 Average value of Rg, distance, and number of contacting atoms | ||||||||||||||||
除吸附能外,均方回转半径、距离的变化和接触原子数也可以表明体系的吸附构象。距离指细胞色素c中的Fe原子到富勒醇和三丙二酸富勒烯质心间的距离。接触原子数是指距离细胞色素c表面0.5 nm范围内的富勒醇和三丙二酸富勒烯的原子数。由表 1可知,富勒醇与细胞色素c的距离更近,并且与蛋白质的接触原子数也会更多。这主要是由于富勒醇中的羟基官能团的个数比较多,并在富勒烯表面分布均匀,相比于三丙二酸富勒烯而言,富勒醇具有更高的水溶性,在水溶液中可以更稳定地存在,同时,富勒醇中的羟基与蛋白质残基中氨基形成的氢键也远多于三丙二酸富勒烯中的羧基与氨基所形成的氢键,从而大大加强了富勒醇与蛋白的相互作用。因此,富勒醇与细胞色素c的相互作用更强,可以更稳定地吸附在细胞色素c的表面,图 2为稳定后的轨迹截图。
Fig. 2
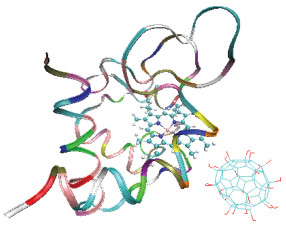 | Download: JPG larger image |
| 图 2 体系P1在15 ns时的轨迹截图 Fig. 2 Snapshot of system P1 at 15 ns 图 2 体系P1在15 ns时的轨迹截图 Fig. 2 Snapshot of system P1 at 15 ns --> | |
3 结论本文通过分子动力学模拟的方法研究富勒醇和三丙二酸富勒烯与细胞色素c的吸附过程,通过相互作用能、均方回转半径、均方根偏差、接触原子数等研究吸附后的结构。结果表明,细胞色素c的结构没有发生很大的改变。此外,模拟结果也表明,富勒醇与细胞色素c的因含有较多的氢键数而具有更强的相互作用,更容易对生物体产生影响。
本文的计算结果是在中国科学院计算机网络信息中心超级计算中心的“元”超级计算机上得到的。
参考文献
| [1] | Gunawan C, Im M, Marquis C P, et al. Nanoparticle-protein corona complexes govern the biological fates and functions of nanoparticles[J]. Journal of Materials Chemistry B, 2014, 2(15): 2060-2083. DOI:10.1039/c3tb21526a |
| [2] | Shang L, Yang L X, Seiter J, et al. Nanoparticles interacting with proteins and cells:a systematic study of protein surface charge effects[J]. Advanced Materials Interfaces, 2014, 1(2): 1-10. |
| [3] | Fischer T, Agarwal A, Hess H. A smart dust biosensor powered by kinesin motors[J]. Nature Nanotechnology, 2009, 4(3): 162-166. DOI:10.1038/nnano.2008.393 |
| [4] | Im H, Huang X J, Gu B, et al. A dielectric-modulated field-effect transistor for biosensing[J]. Nature Nanotechnology, 2007, 2(7): 430-434. DOI:10.1038/nnano.2007.180 |
| [5] | Orosco M M, Pacholski C, Sailor M J. Real-time monitoring of enzyme activity in a mesoporous silicon double layer[J]. Nature Nanotechnology, 2009, 4(4): 255-258. DOI:10.1038/nnano.2009.11 |
| [6] | Graslund S, Nordlund P, Weigelt J, et al. Protein production and purification[J]. Nature Methods, 2008, 5(2): 135-146. DOI:10.1038/nmeth.f.202 |
| [7] | Thingholm T E, Jorgensen T J D, Jensen O N, et al. Highly selective enrichment of phosphorylated peptides using titanium dioxide[J]. Nature Protocols, 2006, 1(4): 1929-1935. DOI:10.1038/nprot.2006.185 |
| [8] | Bushnell G W, Louie G V, Brayer G D. High-resolution three-dimensional structure of horse heart cytochrome c[J]. Journal of Molecular Biology, 1990, 213(2): 585-595. |
| [9] | Steven J M. Cytochrome c as an experimental model protein[J]. Metallomic, 2011, 3(4): 319-322. DOI:10.1039/c0mt00089b |
| [10] | Bosi S, Tatiana D R, Spalluto G, et al. Fullerene derivatives:an attractive tool for biological applications[J]. European Journal of Medicine Chemistry, 2003, 38(11): 913-923. |
| [11] | Rajagopalan M, Oh I. Fullerenol-based electroactive artificial muscles utilizing biocompatible polyetherimide[J]. ACS Nano, 2011, 5(3): 2248-2256. DOI:10.1021/nn103521g |
| [12] | Montellano A, Tatiana D R, Biancob A, et al. Fullerene C60 as a multifunctional system for drug and gene delivery[J]. Nanoscale, 2011, 3(10): 4035-4041. DOI:10.1039/c1nr10783f |
| [13] | Maciel C, Fileti E E, Rivelino R. Assessing the solvation mechanism of C60(OH)24 in aqueous solution[J]. Chemical Physics Letters, 2001, 507(4): 244-247. |
| [14] | MacKerell A D, Bashford D, Bellott M, et al. All-atom empirical potential for molecular modeling and dynamics studies of proteins[J]. Journal of Physical Chemistry B, 1998, 102(18): 3586-3616. DOI:10.1021/jp973084f |
| [15] | Phillips J C, Braun R, Wang W, et al. Scalable molecular dynamics with namd[J]. Journal of Computional Chemistry, 2005, 26(16): 1781-1802. DOI:10.1002/(ISSN)1096-987X |
