0 引言
【研究意义】生物量是生态系统中重要的理化参数之一[1],是评估植被生命活动的一个重要指标[2],并且对长势监测和作物估产有重要意义[3]。传统的生物量估算方法不仅费时费力、破坏性大,且难以实现生物量的大范围监测[4]。近年来,随着遥感技术的发展,高光谱遥感技术以其准确、快速、大尺度、非破坏性等优势已广泛应用于植被生物量的遥感估测中[2]。【前人研究进展】已有许多研究者[5,6,7]基于植被光谱特征,利用植被指数及其衍生光谱指数等对植株生物量进行了估测[8],并取得了一定的进展。SHIBAYAMA等[9]利用差值和比值植被指数构建了水稻生物量的估算模型,精度R2高达0.93。侯学会等[10]基于多种植被指数构建了生物量估算模型,研究发现,基于红边位置的指数模型估算冬小麦生物量的精度最高,决定系数R2=0.83。刘琼阁等[11]以TM遥感影像为数据源,基于波段及波段组合构建了森林生物量的偏最小二乘回归模型,估算精度较高,决定系数为0.72。然而,仅利用光谱特征进行生物量估测,在高植被密度条件下,植被指数会表现出对冠层生物量变化不敏感,即出现所谓的“饱和”现象[12],在识别地物及理化参数反演中容易出现偏差,影响反演的精度和效果。除光谱信息外,纹理信息作为另一重要遥感信息,是物体的一种固有属性,不容易受到外界环境的影响,反映的是图像灰度性质及其空间关系,能够提供地物的几何信息,扩大了基于原始影像亮度的空间信息辨识度,能够改善单一光谱信息源反演时存在的饱和问题,一定程度上提升理化参数反演的潜力[13]。对于纹理特征在定量反演中的应用,许多****进行了相关研究。GU等[14]对于植被覆盖度进行了研究,发现植被指数和纹理特征的结合可以有效的反演植被覆盖度。SARKER等[15]对森林生物量进行了研究,发现植被指数和纹理特征共同反演生物量的效果较好。曹庆先等[16]基于TM影像的纹理和光谱特征构建的生物量多元回归模型,可以高效的进行红树林生物量的估算,为红树林湿地生物量的估算提供了一种新方法。牧其尔等[17]以RapidEye多光谱影像为数据源,选取4种典型植被指数,并提取了不同植被指数的灰度共生矩阵纹理特征值,构建了植被指数、纹理特征各自与生物量的多元回归模型,研究发现,纹理特征估测生物量的能力高于单一的植被指数。【本研究切入点】目前,研究者多基于纹理特征与光谱特征的多元回归模型进行生物量估算研究,并发现结合纹理与光谱信息进行生物量反演的效果更好。然而,从饱和问题出发,联系饱和现象与估算生物量的能力之间的关系,将光谱信息与纹理信息有机结合进行生物量反演的研究较为少见。【拟解决的关键问题】为进一步探究植被指数的饱和问题,本文尝试将对生物量敏感的纹理特征加入到植被指数当中,考虑各纹理指标随着叶面积指数(LAI)增大的变化情况不同,采用将植被指数与纹理指标相乘或相除两种形式构建一种“图-谱”融合指标,探究“图-谱”融合指标的抗饱和能力及估测生物量的能力,以实现结合光谱信息和纹理信息的冬小麦生物量遥感估测,同时发挥光谱信息与纹理信息的优势,从而更好的反演植被生物量,为生物量定量反演提供一种新思路。1 材料与方法
1.1 试验区概况
试验于2014—2015年冬小麦生长季,在北京市昌平区小汤山国家精准农业研究示范基地(40°00′—40°21′N,116°34′—117°00′E,海拔高度36 m)进行。试验区气候温和,四季分明,属于典型的暖温带半湿润大陆季风气候。前茬作物为玉米,试验土壤类型为潮土,土壤中0—30 cm土层中硝态氮含量为3.16—14.82 mg·kg-1,全氮含量为1.0—1.2 g·kg-1,有机质含量为15.8—20.0 g·kg-1,有效磷含量为3.14—21.18 mg·kg-1,速效钾含量为86.83—120.62 mg·kg-1。试验设计为品种、氮肥、水分的正交试验(图1)。冬小麦品种分别为京9843(J9843)和中麦175(ZM175);氮肥(尿素)施用量包括0(N1)、195 kg·hm-2(N2)、390 kg·hm-2(N3)和585 kg·hm-2(N4)4个水平;水分包括雨养(W1,0)、正常灌水(W2,192 mm)和2倍正常灌水(W3,384 mm)3个水平。试验共有48个小区,16个小区为一组,重复3次。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图1研究区概况及试验小区分布
-->Fig. 1Location of study area and experiment design
-->
1.2 数据获取
1.2.1 地面数据获取 试验于冬小麦挑旗期(2015年4月26日)进行,试验数据主要为地上部生物量数据和叶面积指数。在室外调查固定样方2行×1m(行距15 cm),进行群体密度调查,并在各小区选取具有代表性的20个小麦植株作为样本。获取的样本分器官处理,测定鲜重后放入纸袋中,置于烘箱105℃杀青30 min,然后设置温度到75℃烘干至恒重(约24—48 h),测定各器官干重,最终通过群体密度和样本干重,计算得到单位面积上的冬小麦生物量。
总生物量=(叶干重+茎干重+穗干重)/取样茎数×亩茎数 (1)
在试验小区样方内选取20个具有代表性的小麦作为样本,将获取的样本茎叶分离,选取样本中具有代表性的叶片,采用手持式叶面积激光仪CI-203(CID Inc.,USA)测定其叶片面积,求和计算出样本叶片总面积,最后通过测定的群体密度,计算出冬小麦的叶面积指数。
叶面积指数(LAI)= 样方叶片总面积/样方面积 (2)
1.2.2 高光谱数据获取 本研究以八旋翼电动无人机为近地飞行平台搭载UHD185成像光谱仪(Cubert,德国)(图2),选择晴朗无云天气,在中午12:00—13:00对小麦试验田进行航拍,飞行高度为50 m。获取空间分辨率为0.012 m的小汤山冬小麦挑旗期(2015年4月26日)一景高光谱影像。
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图2搭载高光谱传感器(UHD185)的无人机平台
-->Fig. 2UAV platform with hyperspectral sensor (UHD 185)
-->
为消除成像过程中传感器性能、大气传输等因素的影响,集成高光谱分辨率和高空间分辨率为一体的遥感影像,本文对原始影像进行数据预处理。主要包括:利用无人机自带Cubert-Pilot软件(Cubert,德国)进行影像融合,利用Agisoft PhotoScan软件(Agisoft LLC,俄罗斯)对融合后影像进行图像拼接[18],利用ENVI软件(Exelis Visual Information Solutions,美国)进行辐射校正,大气校正、裁剪[19]。
1.3 高光谱影像光谱分析
根据前人研究及相关参考文献,选取了10个与生物量相关的植被指数对冬小麦生物量进行估算(表1)。Table 1
表1
表1本文用到的植被指数
Table 1Vegetation index and its formulas in this study
| 植被指数 Vegetation index | 计算公式 Formulas |
|---|---|
| 归一化差值植被指数Normalized difference vegetation index, NDVI | (R802-R658)/(R802+R658) |
| 差值植被指数Difference vegetation index, DVI | R802-R658 |
| 比值植被指数Ratio vegetation index, RVI | R802/R658 |
| 土壤调整植被指数Soil adjusted vegetation index, SAVI | (R802-R658)/( R802+R658+0.5)(1+0.5) |
| 最优土壤调整植被指数 Optimized soil adjusted vegetation index, OSAVI | (1+0.16)( R802-R658)/ ( R802+R658+0.16) |
| 再归一化差值植被指数 Ratio difference vegetation index, RDVI | ($\sqrt{DVI \times NDVI}$) |
| 归一化绿波段差值植被指数Green normalized difference vegetation index, GNDVI | (R802-R514)/( R802+R514) |
| 增强型植被指数 Enhanced vegetation index, EVI | 2.5(R802-R658)/( R802+6R658-7R490+1) |
| 修正土壤调整植被指数 Modified soil adjusted vegetation index, MSAVI | [(2R802+1)-$\sqrt{(2R_{802}+1)^{2}-8DVI }$]/2 |
| 修正三角植被指数2 Modified triangular vegetation index 2, MTVI2 | $frac{1.5[1.2(R_{802}-R_{514})-2.5(R_{658}-R_{514})]}{\sqrt{(2R_{802}+1)^{2}-6(R_{802}-5\sqrt{R_{658}})-0.5}}$ |
新窗口打开
1.4 高光谱影像纹理分析
1.4.1 波段筛选 通过原始波段反射率与生物量的相关性分析,筛选出3个与生物量相关性较大的波段,并将其作为纹理特征分析波段。1.4.2 纹理特征提取 灰度共生矩阵法[20]是1973 年由HARALICK提出的目前应用最广的纹理提取方法之一,因其具有旋转不变性和多尺度特性,且计算复杂度小,受到了较多的关注[21]。在ENVI 5.3中,利用灰度共生矩阵方法(GLCM)对所筛选的3个波段,进行0°、45°、90°、135° 4个方向的8个纹理特征的提取(表2),对不同方向的纹理特征进行平均,得到各波段的8个纹理特征。然后在ArcGIS 10.3.1(Esri,美国)中,对于各波段的纹理特征影像分别进行感兴趣区域划定,提取出所划区域的纹理值,并将该值作为这个小区的纹理特征值。
Table 2
表2
表2研究用到的纹理特征及其计算公式
Table 2Texture features and its formulas in this study
| 纹理特征 Texture feature | 公式 Formula |
|---|---|
| 均值 Mean (mean) | 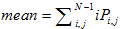 |
| 方差 Variance (var) | 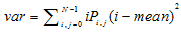 |
| 同质性 Homogeneity (hom) | 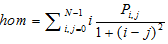 |
| 对比度 Contrast (con) | 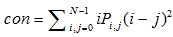 |
| 差异性 Dissimilarity (dis) | 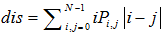 |
| 熵 Entropy (ent) | 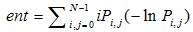 |
| 二阶距 Second Moment (sm) |  |
| 相关性 Correlation (corr) |  |
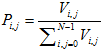 ,式中Vi, j表示第i行第j列的像元亮度值,N表示纹理分析时的窗口大小
,式中Vi, j表示第i行第j列的像元亮度值,N表示纹理分析时的窗口大小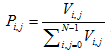 , where Vi, j represents the pixel brightness value of the (i, j)th element, and N represents the window size of the texture analysis
, where Vi, j represents the pixel brightness value of the (i, j)th element, and N represents the window size of the texture analysis新窗口打开
1.4.3 纹理特征与生物量的相关性分析 利用SPSS 22.0软件(IBM,USA),采用皮尔逊相关分析法分析纹理特征与冬小麦地上生物量之间的相关性,筛选出与生物量相关性较大的纹理特征构建生物量估算模型。采用多元逐步回归法,基于所筛选纹理特征构建生物量估算模型。
1.5 饱和性分析及“图-谱”融合指标的建立
植被指数在植被覆盖度较大时易过早饱和,尝试将与生物量相关的纹理特征加入到植被指数当中,考虑各纹理指标随着LAI增大的变化情况不同,采用将植被指数与纹理指标相乘或相除两种形式,构建“图-谱”融合指标(表3),并通过饱和点位置的变化来探究“图-谱”融合指标的抗饱和能力。Table 3
表3
表3本研究用到的“图-谱”融合指标
Table 3'image and spectrum' fusion index in this study
| “图-谱”融合指标类型 Type of 'image and spectrum' fusion index | “图-谱”融合指标 'image and spectrum' fusion index |
|---|---|
| VI/Texture | VI/ent658, VI/dis658, VI/con658, VI/mean658, VI/dis514, VI/con514, VI/var514, VI/mean514 |
| VI×Texture | VI×sm658, VI×hom658, VI×hom514, VI×con802, VI×mean802, VI×dis802 |
新窗口打开
1.6 模型构建及数据分析
本研究中,选取挑旗期2个重复试验的数据进行模型构建(样本量n=32),该生育期的另一重复试验数据进行模型验证(n=16)。最后根据建模精度(R2作为评判标准)和验证精度(RMSE作为评判标准)来评判模型的拟合效果。一般来说,R2越高,RMSE越低,建模与验证精度越高,依此选出最佳生物量模型。2 结果
2.1 基于光谱指数的生物量估算
2.1.1 植被指数与生物量的相关性分析 对选取的10个植被指数和生物量的相关性进行分析(表4)。由表中可以得到,植被指数与生物量之间的相关性均达到极显著水平(P<0.01),其中与生物量相关性最大的是GNDVI,相关系数为0.776,最小是DVI,相关系数为0.547;相关性由大到小依次为:GNDVI、NDVI、RVI、OSAVI、MTVI2、MSAVI、RDVI、SAVI、EVI、DVI。Table 4
表4
表4植被指数和生物量的相关性分析
Table 4Correlation analysis between vegetation index and biomass
| 植被指数 Vegetation index | 相关系数(r) Correlation coefficient(r) | 植被指数 Vegetation index | 相关系数(r) Correlation coefficient (r) | |
|---|---|---|---|---|
| RVI | 0.737** | SAVI | 0.645** | |
| MTVI2 | 0.683** | RDVI | 0.647** | |
| MSAVI | 0.653** | OSAVI | 0.709** | |
| EVI | 0.607** | NDVI | 0.762** | |
| DVI | 0.547** | GNDVI | 0.776** |
新窗口打开
2.1.2 基于植被指数的生物量估算模型 采用多元逐步回归法,基于10个植被指数建立生物量回归模型(图3),同时基于植被指数的回归模型得到冬小麦生物量的结果分布(图4)。图3中可以看出,生物量模型精度为R2=0.69,RMSE=874.25 kg·hm-2,拟合效果较好,表明利用植被指数估算冬小麦生物量是可行的。同时发现,在生物量大于7 000 kg·hm-2时,部分预测值略低于实测值,可能是植被初期生长的过程中,随着叶片逐渐增大,出现了轻微的饱和现象。由图4中可以看出,不施氮处理(N1)和0.5倍常规施氮处理(N2)的冬小麦生物量较小,多分布在5 100 kg·hm-2以下,部分小区的冬小麦生物量分布在5 100—6 300 kg·hm-2,分布范围较大;而常规施氮处理(N3)和1.5倍常规施氮处理 (N4)的冬小麦生物量相对较大,多分布在5 100—7 500 kg·hm-2。雨养处理下(W1),冬小麦生物量分布在5 100 kg·hm-2以下;在正常水(W2)及2倍正常水(W3)处理下,冬小麦的生物量较大,生物量多集中在5 100—7 500 kg·hm-2;但是W2与W3处理之间的生物量差异并不很明显。能够看出,冬小麦生物量随着施氮水平的提高,以及水分处理水平的提升,整体呈逐步增加的趋势,但是,生物量的分布范围较大,具备的规律性较差,可能是植被指数进行生物量反演的过程中受到了饱和现象的影响,因此,仅利用光谱特征进行生物量反演并不能十分准确的反映出实际生物量的分布情况,存在一定的局限。
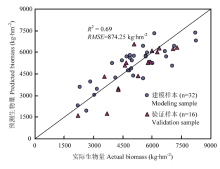 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图3多个植被指数的生物量模型预测值与实际值对比
-->Fig. 3The relationship between actual biomass and predicted biomass of biomass estimation model by multiple vegetation indices
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图4基于植被指数的冬小麦生物量反演结果
-->Fig. 4Biomass inversion results of winter wheat by vegetation index
-->
2.2 基于纹理特征的生物量估算
2.2.1 纹理特征波段筛选 原始波段反射率与生物量的相关性分析如图5所示,由图中看出,454—726 nm波段光谱反射率与生物量呈负相关,730—882 nm波段区间呈正相关,其中分别在514 nm(r = -0.697),658 nm(r = -0.775)处形成两个波谷,在802 nm(r =0.565)波段处形成高反射平台,因此最终选择与生物量相关性较大的3个波段,绿波段(514 nm)、红波段(658 nm)以及近红外波段 (802 nm)作为纹理特征分析波段。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图5光谱反射率与生物量的相关性曲线图
-->Fig. 5Correlation curve between spectral reflectance and biomass
-->
2.2.2 基于纹理特征的生物量估算模型 经过对比不同窗口的纹理特征后发现,本文所采用的高光谱影像在3×3窗口下的分辨率最适合进行纹理特征分析,因此,选取3×3窗口下,与生物量相关性较好的纹理特征,即mean514(514 nm波段处的mean特征)、var514、hom514、con514、dis514、mean658、ent658、sm658、hom658、con658、dis658、mean802、con802、dis802这些纹理特征作为生物量的估测指标,对于冬小麦生物量的估算有积极意义。
采用多元逐步回归法,考虑前文所筛选的全部纹理特征(mean514、var514、hom514、con514、dis514、mean658、ent658、sm658、hom658、con658、dis658、mean802、con802、dis802),构建生物量回归模型(图6),同时基于纹理特征的回归模型得到冬小麦生物量的结果分布(图7)。图6中可以看出,生物量模型精度为R2=0.71,RMSE=828.87 kg·hm-2,与光谱特征构建的生物量模型(R2=0.69,RMSE=874.25 kg·hm-2)相比,估算模型精度R2有所提高,均方根误差减小,拟合效果较好,可能原因是纹理特征的模型中含有丰富的纹理信息,一定程度上弥补了光谱信息的不足,缓解了光谱特征反演时存在的饱和以及“同物异谱,同谱异物”现象,从而提高了模型的拟合精度。由图7中可以看出,不施氮处理(N1)和0.5倍常规施氮处理(N2)的冬小麦生物量较小,多分布在3 900—6 300 kg·hm-2,部分小区的冬小麦生物量分布在3 900 kg·hm-2以下;而常规施氮处理(N3)和1.5倍常规施氮处理(N4)的冬小麦生物量相对较大,多分布在5 100—7 500 kg·hm-2。雨养处理下(W1),冬小麦生物量分布在5 100 kg·hm-2以下;在正常水(W2)及2倍正常水(W3)处理下,冬小麦的生物量较大,生物量多集中在5 100— 7 500 kg·hm-2,相比之下,W3处理比W2处理下的生物量更大,冬小麦长势更好一些。整体上,不同处理之间的生物量差异及规律能够较好的呈现出来,相比基于植被指数的冬小麦生物量反演结果要更加具有规律性,更加贴近实际情况。因此,基于纹理特征的生物量模型的拟合效果较好,能较准确地反演冬小麦地上生物量,具有一定的应用价值。
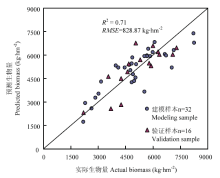 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图6多个纹理特征的生物量模型预测值与实际值对比
-->Fig. 6The relationship between actual biomass and predicted biomass of biomass estimation model by multiple texture features
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图7基于纹理特征的冬小麦生物量反演结果
-->Fig. 7Biomass inversion results of winter wheat based on texture feature
-->
2.3 基于“图-谱”融合指标的生物量估算
2.3.1 “图-谱”融合指标的饱和性分析 在利用植被指数估测生物量时,发现当生物量超过一定量时,出现部分预测值低于实测值的现象。主要原因可能是随着植被生长,叶片逐渐增大,植被覆盖度逐渐增加,使得植被指数对于生物量变化不再敏感,造成了光谱的饱和现象。因此,可通过分析光谱指标随LAI增大的变化情况来分析各光谱指标的饱和性。不同光谱指数随LAI的变化情况如图所示(图8—图11)。本研究对14种“图-谱”融合指标都进行了饱和性探究,图8—图11仅列出了部分“图-谱”融合指标(VI,VI×sm658,VI×con802,VI×mean802)随着LAI 的变化情况,但是分析时仍对其他的“图-谱”融合指标饱和性进行分析,最终的结果分析包括所有的“图-谱”融合指标。 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图8各植被指数随LAI的变化情况
-->Fig. 8The relationship between vegetation index and LAI
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图9“图-谱”融合指标VI×sm658随LAI的变化情况
-->Fig. 9The relationship between 'image and spectrum' fusion index (VI×sm658) and LAI
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图10“图-谱”融合指标VI×con802随LAI的变化情况
-->Fig. 10The relationship between 'image and spectrum' fusion index (VI×con802) and LAI
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图11“图-谱”融合指标VI×mean802随LAI的变化情况
-->Fig. 11The relationship between 'image and spectrum' fusion index (VI×mean802) and LAI
-->
对比植被指数与“图-谱”融合指标的饱和点位置可以发现(图8—图11),大部分植被指数在LAI=5的时候出现饱和现象;“图-谱”融合指标VI×sm658、VI/ent658、VI/dis658、VI/con658、VI/dis514、VI/ con514、VI/var514、VI×con802、VI×dis802随LAI增大几乎呈线性趋势递增,或是先逐步增大后趋于稳定,均在LAI>5后出现饱和现象,延后了饱和点位置,其抗饱和能力有所提高;但是“图-谱”融合指标VI/mean514、VI×hom514、VI/mean658、VI×hom658、VI×mean802 随着LAI的增大,先逐步增大后略微下降,LAI在5以内或大约等于5 时出现了饱和点,其饱和现象并没有得到改善。
2.3.2 “图-谱”融合指标与生物量的相关性分析 将植被指数、“图-谱”融合指标分别与生物量进行相关性分析(表5)。结果表明,所有指标与生物量的相关性都达到了极显著水平(P<0.01),与生物量相关性相对较大的2种“图-谱”融合指标为VI×con802和VI×dis802,其中,相关性最大的“图-谱”融合指标是RVI×con802(r=0.815)。对比植被指数和生物量之间的相关性,VI×sm658,VI/ent658,VI×hom658,VI/dis658,VI/con658,VI/dis514,VI×hom514,VI/con514,VI/var514,VI×con802,VI×dis802中大部分指标与生物量的相关性都有所提高;而VI/mean658,VI/mean514,VI×mean802中大多指标的相关性没有提高,反而有所下降。
Table 5
表5
表5植被指数和“图-谱”融合指标与生物量的相关性分析
Table 5Correlation analysis between vegetation index, 'image and spectrum' fusion index and biomass
| “图-谱”融合指标 'image and spectrum' fusion index | RVI | MTVI2 | MSAVI | EVI | DVI | SAVI | RDVI | OSAVI | NDVI | GNDVI |
|---|---|---|---|---|---|---|---|---|---|---|
| VI | 0.758** | 0.729** | 0.706** | 0.671** | 0.622** | 0.699** | 0.701** | 0.745** | 0.775** | 0.782** |
| VI×sm658 | 0.759** | 0.772** | 0.770** | 0.763** | 0.742** | 0.774** | 0.774** | 0.783** | 0.781** | 0.782** |
| VI/ ent658 | 0.747** | 0.776** | 0.777** | 0.777** | 0.766** | 0.780** | 0.779** | 0.779** | 0.771** | 0.769** |
| VI×hom658 | 0.759** | 0.747** | 0.734** | 0.712** | 0.673** | 0.736** | 0.738** | 0.765** | 0.778** | 0.781** |
| VI/ dis658 | 0.746** | 0.774** | 0.775** | 0.774** | 0.764** | 0.778** | 0.777** | 0.777** | 0.771** | 0.769** |
| VI/ con658 | 0.745** | 0.772** | 0.774** | 0.772** | 0.763** | 0.776** | 0.775** | 0.776** | 0.770** | 0.769** |
| VI/ mean658 | 0.628** | 0.656** | 0.657** | 0.660** | 0.664** | 0.654** | 0.653** | 0.647** | 0.640** | 0.637** |
| VI/ dis514 | 0.744** | 0.774** | 0.774** | 0.774** | 0.765** | 0.771** | 0.769** | 0.760** | 0.739** | 0.729** |
| VI×hom514 | 0.760** | 0.759** | 0.750** | 0.732** | 0.694** | 0.753** | 0.754** | 0.774** | 0.773** | 0.769** |
| VI/ con514 | 0.734** | 0.764** | 0.764** | 0.765** | 0.762** | 0.759** | 0.757** | 0.747** | 0.727** | 0.719** |
| VI/ var514 | 0.728** | 0.756** | 0.756** | 0.757** | 0.755** | 0.749** | 0.747** | 0.737** | 0.719** | 0.711** |
| VI×con802 | 0.815** | 0.801** | 0.791** | 0.775** | 0.757** | 0.783** | 0.785** | 0.789** | 0.784** | 0.776** |
| VI/ mean514 | 0.658** | 0.681** | 0.679** | 0.682** | 0.694** | 0.665** | 0.662** | 0.646** | 0.625** | 0.617** |
| VI×meann802 | 0.677** | 0.604** | 0.585** | 0.567** | 0.543** | 0.580** | 0.581** | 0.601** | 0.624** | 0.615** |
| VI×dis802 | 0.814** | 0.814** | 0.802** | 0.786** | 0.769** | 0.785** | 0.786** | 0.773** | 0.737** | 0.704** |
新窗口打开
因此,最终筛选抗饱和能力强,且与生物量相关性相对较大的“图-谱”融合指标作为生物量的估测指标,即VI×sm658,VI/ent658, VI/dis658, VI/con658,VI/dis514, VI/con514,VI/var514,VI×con802,VI×dis802。
2.3.3 基于“图-谱”融合指标的生物量估算模型 采用多元逐步回归法,基于所筛选的“图-谱”融合指标构建生物量估算模型(图12),同时基于“图-谱”融合指标的回归模型得到冬小麦生物量的结果分布(图13)。图12可以看出,生物量模型精度为R2 =0.81,RMSE=826.02 kg·hm-2,与纹理特征构建的生物量估算模型(R2=0.71,RMSE=828.87 kg·hm-2)和植被指数构建的生物量估算模型(R2=0.69,RMSE=874.25 kg·hm-2)相比,“图-谱”融合指标构建的生物量估算模型精度R2明显提高,均方根误差较小,拟合效果最好。由图13中可以看出,不施氮处理(N1)和0.5倍常规施氮处理(N2)的冬小麦生物量较小,多分布在2 000—5 900 kg·hm-2;而常规施氮处理(N3)和1.5倍常规施氮处理(N4)的冬小麦生物量相对较大,多分布在4 600—7 200 kg·hm-2。雨养处理下(W1),冬小麦生物量分布在5 900 kg·hm-2以下;在正常水(W2)及2倍正常水(W3)处理下,冬小麦的生物量较大,生物量多集中在4 600—7 200 kg·hm-2。不同水氮处理间的冬小麦生物量值分布存在明显的阶梯性,而每个处理水平内的冬小麦生物量分布较集中,能够很好的表征冬小麦生物量。
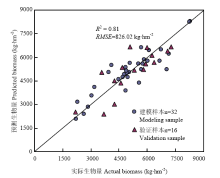 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图12多个“图-谱”融合指标的生物量模型预测值与实际值对比
-->Fig. 12The relationship between actual biomass and predicted biomass of biomass estimation model by multiple 'image and spectrum' fusion indices
-->
 显示原图|下载原图ZIP|生成PPT
显示原图|下载原图ZIP|生成PPT图13基于“图-谱”融合指标的冬小麦生物量反演结果
-->Fig. 13Biomass inversion results of winter wheat based on 'image and spectrum' fusion index
-->
结果表明,融合光谱信息与纹理信息的“图-谱”融合指标,综合考虑了纹理特征及光谱特征对生物量的贡献性。敏感的植被指数对于不同生物量下的光谱特征有很好的表征,而丰富的纹理信息减弱了光谱特征存在的饱和问题,抗饱和能力得到明显改善,因此,其生物量回归模型估算精度提高,相比单一纹理特征、单一光谱特征的生物量模型,纹理与光谱共同反演冬小麦地上生物量的效果最好。
3 讨论
前人多利用光谱信息对生物量进行估算,取得了一定的进展,但是基于光谱信息进行生物量反演,在植被覆盖度较大时易出现饱和问题。纹理特征应用于分类研究中,研究者[22,23,24]发现加入纹理特征后分类精度有了明显的提升;而在森林结构参数反演的研究中[25,26,27],纹理特征能够反映树木的冠层结构及异质性,引入纹理特征估算结构参数,能够取得较好的估算效果。基于纹理特征进行生物量反演的研究,李明诗等[28]发现,少数纹理特征ME(均值)、VA(方差)在森林生物量估算中是有效的。前人多是直接选取对生物量敏感的纹理及光谱特征,同时基于所选纹理和光谱特征构建多元回归模型进行生物量估算,而本研究从饱和问题出发,利用无人机获取的高光谱影像,提取其光谱特征与纹理特征,主要探究了光谱与纹理信息相结合的“图-谱”融合指标的抗饱和能力和估测生物量的能力。研究发现,相比植被指数,本文所构建的“图-谱”融合指标VI×sm658,VI/ent658,VI/dis658,VI/con658, VI/dis514,VI/con514,VI/var514,VI×con802,VI×dis802延后了对LAI响应的饱和点位置,其抗饱和能力有所提升,同时,对比植被指数与生物量的相关性发现,这些指标与生物量的相关性也有所提高,因此选取具有抗饱和特性并且对生物量敏感的指标,探究其反演生物量的效果。最终结果表明,基于“图-谱”融合指标构建的生物量估算模型精度明显提高,主要是因为“图-谱”融合指标考虑了光谱特征与纹理特征两者共同对生物量的贡献性,敏感的植被指数对于不同生物量下的光谱特征有了很好的表征,而丰富的纹理信息提供了作物的空间特征,减弱了单一植被指数反演时存在的饱和现象,一定程度上弥补了光谱信息的不足,“图-谱”融合指标结合了两者的优势,提高了模型的拟合精度。本文的研究结果发现,相比单一光谱特征、单一纹理特征,纹理与光谱共同反演冬小麦地上生物量的效果最佳,与前人[16-17, 28]的相关研究结果一致。然而,前人基于纹理特征的生物量反演,主要应用在红树林[14]、绿洲沙漠植被[15]、森林[29,30]等方面,对于冬小麦的生物量反演研究较为少见。同时,本研究对于饱和问题具有一定的针对性。本文尝试从提升光谱特征的抗饱和能力出发,联系抗饱和能力与估算生物量能力之间的关系,探究了不同指标的生物量估算能力,为冬小麦生物量反演提供了一种新思路。同时,拓宽了纹理特征在长势监测、产量估测等方面的应用前景。本研究基于“图-谱”融合指标构建的生物量模型精度提高,但是均方根误差并没有明显改善,可能的原因是,研究仅利用了冬小麦挑旗期的高光谱影像及地面实测数据,所使用的样本量较少,对所构建模型的通用性可能会有一定影响,未来的相关研究应增加试验生育期、品种,拓展模型应用范围,从而提高结果的普适性和稳定性。另外,目前存在的纹理特征提取方法多是针对单波段影像,不适用于包含多波段的高光谱影像,而本文是通过原始波段反射率与生物量的相关性分析,筛选得到纹理特征分析波段,考虑因素不够全面,对于最终估算模型的构建可能会产生一定的影响,因此,还需要进一步的研究,探究出一种更适用高光谱影像波段筛选的方法,从而更加合理有效地利用高光谱影像的波段信息。
4 结论
本文基于无人机高光谱影像,对光谱信息和纹理信息估测冬小麦地上部生物量的能力及其饱和性进行了分析,主要获得以下结论:(1)相比植被指数,“图-谱”融合指标VI×sm658,VI/ent658, VI/dis658, VI/con658,VI/dis514, VI/con514,VI/var514,VI×con802 ,VI×dis802延后了LAI的饱和点位置,其抗饱和能力明显提高,而“图-谱”融合指标VI/mean658,VI/mean514,VI×mean802 ,VI×hom658,VI×hom514的抗饱和性并没有得到提升。
(2)对比植被指数与生物量的相关性(除NDVI,GNDVI外)发现,“图-谱”融合指标VI×sm658,VI/ent658,VI×hom658,VI/dis658, VI/con658,VI/dis514,VI×hom514,VI/con514,VI/var514,VI×con802,VI×dis802中大部分指标与生物量的相关性都明显提高,而VI/mean658,VI/mean514,VI×mean802中大多指标与生物量的相关性没有提高,反而有所下降。
(3)与植被指数相比,所有抗饱和能力提高的“图-谱”融合指标,其与生物量的相关性也明显增强;抗饱和性没有得到改善的“图-谱”融合指标(除VI×hom658,VI×hom514外),其与生物量的相关性并没有增大,反而有所减小。同时,各指标与生物量的相关性大小和抗饱和能力的高低是成正比的。
(4)相比单一光谱特征、纹理特征,将纹理特征与光谱特征相结合的“图-谱”融合指标估算小麦生物量的能力最强,模型精度(R2 =0.81)明显高于植被指数(R2 =0.69)、纹理特征(R2 =0.71)构建的生物量模型,表明融合光谱与纹理信息共同反演生物量的效果较好,具有一定的优势。
(责任编辑 杨鑫浩)
The authors have declared that no competing interests exist.
