为了进一步从基因组上揭示其超亲遗传抗性机理,该研究利用抗性品种构建了细菌染色体文库(BAC),通过筛选Chr11 和Chr21两条染色体上和抗性基因簇相连或者同源的分子标记(图1)确定BAC克隆并对其进行测序和基因功能注释。因为目前发表的棉花基因组都是感病品种,所以该研究是首次利用抗性品种对抗性基因簇区间进行测序,通过比对与感病参考基因组TM1之间的差异揭示了Chr11抗性基因簇区间序列比对上的比例远远低于Chr21;通过比对TM1物理图谱和由棉花抗根结线虫自交重组群体构成的遗传图谱发现两条染色体之间的分子标记重新定位(relocation)(图1);这些研究潜在的解释了两条染色体之间的差异。与Chr21相比,Chr11的序列中包含更多的抗性(R)蛋白拷贝数(图2)及其抗性基因附近具有更多的转座子(transposable elements),由于转座子在作物基因重组中起到重要作用,该研究说明转座子很可能是导致超亲遗传抗性的重要因素;此外,抗性基因的NB-ARC区域鉴定的独特片段序列的缺失以及不同拷贝数的LRR结构域(图3)揭示了这些R基因序列的差异也可能是导致基因复杂的重组而产生超亲抗性的重要原因之一。该研究为植物超亲遗传研究提供了模式系统;进一步对这些抗性基因和转座子的功能研究不仅能够更多的揭示抗性基因的遗传进化,而且为培育新的抗性品种奠定坚实的理论基础。
该研究发表在国际期刊Frontiers in Plant Sciences。王从丽研究员是第一作者,Philip Roberts教授是通讯作者。该研究得到国家自然科学基金项目资助。
文章信息:Wang C, Ulloa M, Nichols RL and Roberts PA (2020) Sequence Composition of Bacterial Chromosome Clones in a Transgressive Root-Knot Nematode Resistance Chromosome Region in Tetraploid Cotton. Front. Plant Sci. 11:574486. https://doi.org/10.3389/fpls.2020.574486
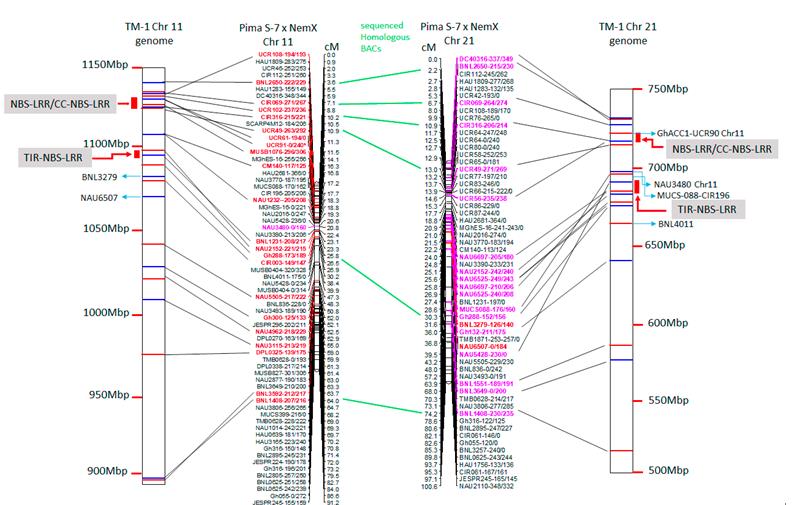
图1:棉花TM1物理图谱和RIL(PimaS-7xNemX)群体形成的遗传图谱比对。红色的SSR分子标记表示被定位到TM1-Chr11,粉色的SSR表示被定位到TM1-Chr21。
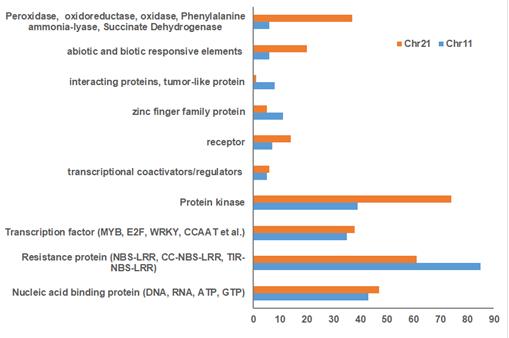
图2:基于棉花Unigene数据库对Chr11抗性基因簇和Chr21染色体的同源区域的应激反应元件(stress response elements)的比较
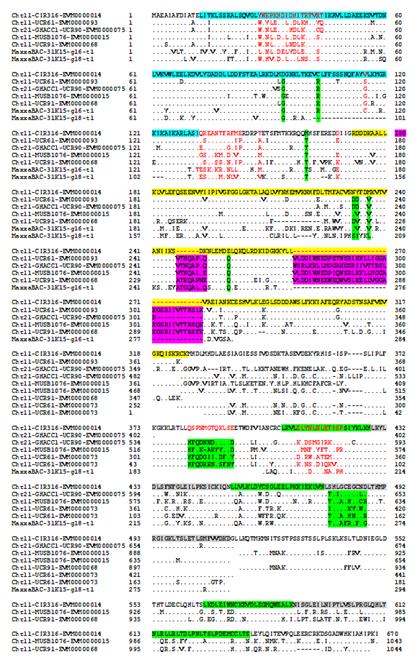
图3:Chr11抗性基因蛋白(CC)-NBS-LRR 与Chr21和感病品种中的同源抗性蛋白比较 (首行蓝色代表CC结构域,黄色代表NB-ARC结构域,绿色和灰色代表LRR结构域)
