
 , 万雯3, 崔古贞4, 官志忠1,2, 齐晓岚1,2, 禹文峰1,2
, 万雯3, 崔古贞4, 官志忠1,2, 齐晓岚1,2, 禹文峰1,2 1. 贵州医科大学 地方病与少数民族疾病教育部重点实验室,贵州 贵阳 550004;
2. 贵州省医学分子生物学重点实验室,贵州 贵阳 550004;
3. 贵州医科大学 病理学教研室,贵州 贵阳 550004;
4. 贵州医科大学 基础医学院 微生物教研室,贵州 贵阳 550004
收稿日期:2019-05-02;接收日期:2019-07-10;网络出版时间:2019-10-30
基金项目:国家自然科学基金(Nos. 31560318,31601012,31760318,U1812403),贵州省科技计划项目黔科合平台人才(No. [2017]5652)资助
摘要:艰难梭菌Clostridioes difficile是一种革兰氏阳性、产芽孢、专性厌氧细菌,是医院相关性腹泻的主要病原体。近年来,随着强毒株的出现(如核糖体027型),其流行性与致死率逐年上升,因此对艰难梭菌生理、生化特征及致病机制的研究受到广泛重视。艰难梭菌生理、生化特征及致病机制研究又以建立其稳定、高效的基因编辑方法为必要前提。借助基因编辑工具,研究者可以扰动艰难梭菌核心生物学过程,在分子水平研究其分子致病机制。如ClosTron技术在艰难梭菌毒素A (Toxin A)和毒素B (Toxin B)与其致病力关系的研究中起到了关键作用。文中以时间为主线综述了艰难梭菌基因编辑技术的发展历程和最新进展,并对艰难梭菌基因编辑技术未来的研究方向进行展望。
关键词:艰难梭菌艰难梭菌感染基因编辑基因功能研究致病机制
Advances in Clostridioes difficile genome editing
Wei Hong1,2

 , Wen Wan3, Guzhen Cui4, Zhizhong Guan1,2, Xiaolan Qi1,2, Wenfeng Yu1,2
, Wen Wan3, Guzhen Cui4, Zhizhong Guan1,2, Xiaolan Qi1,2, Wenfeng Yu1,2 1. Key Laboratory of Endemic and Ethnic Diseases, Ministry of Education, Guizhou Medical University, Guiyang 550004, Guizhou, China;
2. Key Laboratory of Medical Molecular Biology, Guizhou Province, Guiyang 550004, Guizhou, China;
3. Department of Pathology, Guizhou Medical University, Guiyang 550004, Guizhou, China;
4. Department of Microbiology, School of Basic Medical Sciences, Guizhou Medical University, Guiyang 550004, Guizhou, China
Received: May 2, 2019; Accepted: July 10, 2019; Published: October 30, 2019
Supported by: National Natural Science Foundation of China (Nos. 31560318, 31601012, 31760318, U1812403), Guizhou Science and Technology Project (Qiankehepintairencai No. [2017]5652)
Corresponding author: Wei Hong. Tel: +86-851-86752814; E-mail: hongwei@gmc.edu.cn.
Abstract: Clostridioes difficile is a Gram-positive, spore-forming, obligate anaerobic bacterium, and the main cause of hospital-associated diarrhea. In recent years, with the presence of virulent strains (i.e., ribosome type 027), the prevalence and mortality events have increased. Thus, studies on physiological and biochemical characteristics, and pathogenic mechanisms of C. difficile have been performed. The development of efficient and stable genome-editing methods for C. difficile is urgent for the dissection of its physiological and pathogenic mechanism. For example, ClosTron technology plays a key role in study of the relationship between C. difficile toxins (Toxin A and Toxin B) and its pathogenicity. This article reviews the history, recent progress and future prospects of C. difficile genome-editing technologies.
Keywords: Clostridioes difficileClostridioes difficile infection (CDI)genome editinggene function researchpathogenic mechanism
1953年Hall与O’Toole首次从新生儿粪便样本中分离到艰难梭菌Clostridioes difficile[1]。艰难梭菌是一种革兰氏阳性、产芽孢、专性厌氧棒状杆菌。20世纪70年代末,研究者确认艰难梭菌导致20%-25%的抗生素相关性腹泻[2]。艰难梭菌感染(C. difficile infection,CDI)的主要症状有腹泻、腹痛、发烧、中毒性巨结肠、肠道穿孔等[3]。从2000年开始,北美、欧洲、亚洲CDI发病率和致死率明显增加[4]。如2002年艰难梭菌强毒株的出现(核糖体BI/NAP1/027型,同时产生毒素A、毒素B和二元毒素),导致CDI在欧洲大面积暴发,其致病率与致死率显著提高[3, 5]。如今,在北美和欧洲,每10 000例住院病人中,就有7例CDI患者。CDI已成为医院获得性感染中的首要疾病[6-7]。在全球CDI防控形势严峻的前提下,2013年美国疾病预防与控制中心将艰难梭菌与抗碳青霉烯的肠杆菌及耐药性淋病奈瑟菌并列为3种最紧急的多重耐药病原微生物[8-9]。
随着CDI致病率的逐年提高,研究人员想要通过建立艰难梭菌基因编辑系统,以研究其生理、生化特征和致病机制。2003年,Roberts等在艰难梭菌中建立的基于Tn916的反义RNA干扰技术,揭示了细胞壁结合蛋白66 (Cell wall protein 66,Cwp66)对艰难梭菌的细胞黏附能力的贡献[10]。2007年,Heap等基于乳酸球菌(Lactococcus lactis) ltrB基因(Ll.ltrB)二型内含子建立了ClosTron基因失活方法。应用该方法Heap等揭示了spo0A (The master regulator of sporulation)、selC (Spore cortex-lytic enzymes)基因在艰难梭菌芽孢产生中的调控作用[11-13],验证了毒素A与毒素B都具有细胞毒性和致病能力[14],修正了毒素A不能单独引起肠炎的结论[15]。2010年,基于Himar1随机突变技术,发现了cspBA基因对于艰难梭菌芽孢产生具有贡献[16]。2012年,基于尿嘧啶合成缺陷突变株(ΔpyrF)的基因敲除方法的建立,修正了tcdC负调控毒素基因表达的假说[17]。2013年,基于ΔpyrE基因的非等长同源臂偶联等位交换(Allele-coupled exchange,ACE)技术的建立,使得cwp84基因的功能得以揭示[18-19]。除此之外,tcdE基因与毒素基因表达调控关系的阐明[20]、鞭毛蛋白编码基因fliC在艰难梭菌中的多基因调控功能的阐明[21],以及艰难梭菌疫苗的研发[22],都是以基因编辑技术的建立为前提。由此可见,艰难梭菌基因编辑技术的建立对于研究其生理、生化特征和致病机制至关重要。
艰难梭菌基因编辑技术经历了反义RNA干扰技术、ClosTron技术、转座子随机基因失活技术、ACE技术和CRISPR-Cas技术等不同发展阶段(图 1)。文中以时间为主线,综述艰难梭菌基因编辑技术研究进展并展望未来的发展方向。
 |
| 图 1 艰难梭菌基因编辑技术的不同发展阶段[10-11, 16, 19, 23-30] Fig. 1 Different developing stages of genetic-engineering methods of C. difficile[10-11, 16, 19, 23-30]. |
| 图选项 |
1 艰难梭菌基因编辑的前提条件(2002- 2004年)实现艰难梭菌基因编辑需3个前提条件:1)基因组序列测定完成[31];2)建立可在大肠杆菌-艰难梭菌中穿梭复制的质粒系统;3)突破限制性内切酶屏障,建立外源质粒转化方法,详述如下。
1.1 基因组序列信息基因组序列信息包含:基因序列、基因注释和基因排列方式等信息,是基因编辑技术建立的重要前提。2006年,Mohammed等发表了艰难梭菌630菌株(C. difficile 630,CD 630)的全基因组序列[31],该研究发现CD630基因组中包含大量可移动的基因元件(11%),这些可移动基因元件可能与抗生素抗性、菌株毒力、与宿主相互作用等机制具有密切关系。2015年,Eijk等测定了CD630ΔErm (红霉素敏感)菌株的全基因组序列,该菌株基因组与CD 630菌株共有71个位点不同,包括8个删除突变、10个插入突变和50个碱基替换突变等不同突变形式[32]。CD630与CD630ΔErm菌株基因组序列的公布,为后续基因编辑技术的开发奠定了基础。
1.2 建立可在大肠杆菌-艰难梭菌中穿梭复制的质粒系统2002年,Purdy等发现了CD630内源质粒pCD6,该质粒大小为6 829 bp,GC含量为24.5%,包含5个开放阅读框(Open reading frame,orf):orfA、orfB、orfC、orfD和orfE (图 2)[23, 33]。orfA大小为3 994 bp,编码复制蛋白,其下游有一段重复序列,可驱动质粒在CD630中复制。orfB编码未知功能肽段且不是复制必需的。orfC和ofrD也位于复制区域,但是功能未知。orfE编码长度为187个氨基酸的蛋白质,与胞壁酰五肽羧肽酶有相似性,推测参与细胞壁的生物合成。Purdy等使用pCD6复制子(orfA)构建了pMTL9301质粒,该质粒在艰难梭菌中的转化效率较高(5.7×10-5转化子/质粒供体)。连续传代实验表明,pMTL9301在艰难梭菌中可以稳定复制。在无抗生素的培养基中连续32次传代后,仅有8%的克隆丢失质粒。相比之下,pMTL9401 (pB102复制子)在同样的培养条件下,有96%的菌体丢失了质粒[23]。因此pCD6复制子被用于构建大肠杆菌-艰难梭菌穿梭质粒[34]。该质粒可在大肠杆菌(CA434)协助质粒(Helper plasmid)的帮助下,通过结合转移的方式进入艰难梭菌中,并可在其中稳定复制[23]。
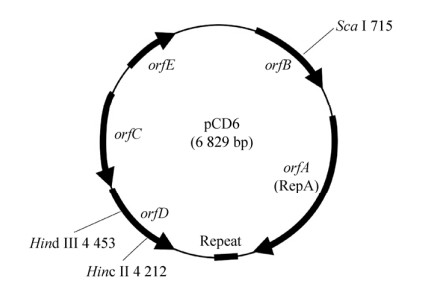 |
| 图 2 pCD6质粒图谱 Fig. 2 Plasmid map of pCD6. |
| 图选项 |
1.3 突破限制性内切酶屏障,建立外源质粒转化方法限制性酶修饰系统(Restriction/modification system,RM系统)是一种微生物应对外源DNA入侵的防御机制[35]。RM系统由限制性内切酶和DNA甲基化酶组成。DNA甲基化酶通过对自身DNA的甲基化,保护其免受自身限制性内切酶切割,否则则被限制性内切酶降解。实现外源DNA转化艰难梭菌的前提条件是突破其固有的RM系统,可以通过两种方式实现:1)质粒转化之前,在体外修饰或删除质粒中的限制性内切酶切割位点,从而保护外源质粒不受内源性的限制性内切酶切割[23];如pMTL9301质粒在转化CD3和CD6菌株之前,质粒上的CdiⅠ位点被同义突变,从而避免了CD3和CD6菌株的RM系统对质粒的切割[23];2)失活宿主菌株内部的RM系统。如在解纤维梭菌Clostridium cellulolyticum中敲除ccel2866基因(该基因编码MspⅠ限制性内切酶)[36];在丙酮丁醇梭菌Clostridium acetobutylicum ATCC 824和DSM 1731菌株中失活CA_1502基因后(编码Ⅱ型限制性内切酶),外源质粒转化效率显著提高[37, 38]。在巴氏梭菌24 (Clostridium pasteurianum)菌株中采用类似的方法,也获得了较高的外源DNA转化效率[39]。
不同艰难梭菌的限制性酶修饰系统具有明显的差异,如在CD6菌株中存在两种RM系统:Sau96Ⅰ/M.Sau96Ⅰ(5′-GGNMCC-3′)与MboⅠ/M. MboⅠ(5′-GMATC-3′),而CD 3只有Ⅱ型限制性内切酶CdiⅠ,该酶识别并切割5′-CATCG-3′序列。另外,CD630菌株中并未发现RM屏障[23]。随着基因组测序和分析技术的进步,出现了计算机程序辅助的RM系统预测方法,如REBASE (http://rebase.neb.com/rebase/)网站预测了常见微生物的RM系统编码信息。研究者可以通过该网站查询某菌株RM系统信息,从而有针对性地失活其RM系统。综上所述,在测定了艰难梭菌全基因组序列、获得了艰难梭菌复制元件和揭示了艰难梭菌不同菌株的RM系统后,基本具备了建立艰难梭菌基因编辑方法的3个前提。
2 基于Tn916的反义RNA技术(2003年)2003年Roberts等利用来源于枯草芽孢杆菌的Tn916转座子,携带一段反义RNA编码序列下调cwp66基因的表达。Cwp66蛋白含有两个结构域:N端与枯草芽孢杆菌Bacillus subtilis CwlB (Peptidoglycan recognition domain)具有同源性;C端结构域定位在细胞膜的表面[40],因此推测其与艰难梭菌细胞黏附功能有关。Tn916是一种广宿主结合转移元件,并为宿主提供四环素抗性(TcR)决定基因[41]。该转座子可在破伤风梭菌Clostridium tetani[42]和丙酮丁醇梭菌Clostridium acetobutylicum[36]基因组中跳跃,且跳跃位点具有明显的偏好性[43]。利用该特性,Roberts等首先克隆并改造了Tn916转座子,在其中加入了cwp66基因反义RNA表达框。将该改造后的转座子作为供体结合转移至CD630菌株,从而尝试降低cwp66基因的表达量(图 3)[10]。然而,该研究并没有显著降低Cwp66蛋白的表达和改变艰难梭菌对细胞的粘附能力,作者推测可能的原因是:1) Cwp66蛋白本身可能并不参与艰难梭菌与细胞的黏附过程;2)基于Tn916的反义RNA并未发挥作用。除此之外,基于Tn916转座子的反义RNA技术还具有如下局限性:1) Tn916在基因组中的插入位点具有较强的偏好性,使得该技术并不适合用于随机突变和理性靶向突变;2)该技术产生的反义RNA的量对靶基因表达的调节能力有限。
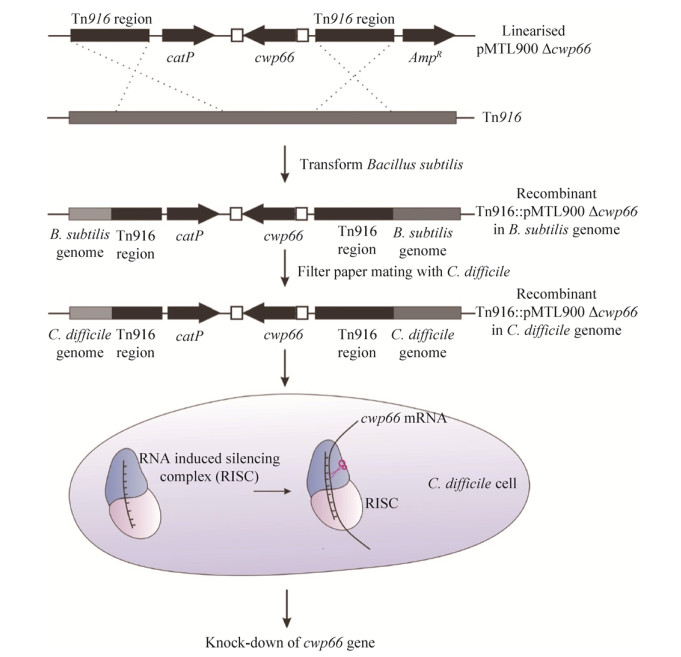 |
| 图 3 基于Tn916的反义RNA敲降cwp66基因表达[10] Fig. 3 Knock down cwp66 gene expression in C. difficile using Tn916 transposon[10]. |
| 图选项 |
3 ClosTron技术(2007年)2007年,梭菌基因编辑的先驱Minton研究组,Heap等将来源于乳酸球菌ltrB基因的Ll.ltrB二型内含子与穿梭质粒融合,开发了首个能够在梭菌中通用的基因失活工具——ClosTron,该系统借助二型内含子可重编码的“归巢(Retrohoming)”效应[44-45],成功地在艰难梭菌、丙酮丁醇梭菌和生孢梭菌C. sporogenes等菌株中实现了靶向基因失活(图 4)[12, 24-27]。该研究组在接下来的工作中进一步改进了ClosTron系统,如在ClosTron质粒上加入了转座激活筛选基因(Retrotransposition- activated marker,RAM),从而提高了突变株的筛选效率[46];建立了免费的引物设计网站http://clostron.com,提高打靶效率;在质粒载体构建过程中加入蓝白斑筛选标记,从而提高载体构建和筛选的效率[25]。
 |
| 图 4 ClosTron系统的自我剪接及“归巢”[48-49] Fig. 4 Self splicing and "retrohoming" of group Ⅱ intron[48-49]. |
| 图选项 |
ClosTron技术可在10-14 d内快速获得靶基因失活突变株,因此极大地提高了梭菌属细菌突变株构建效率。该技术目前仍是艰难梭菌突变株构建的主要工具,并被深度开发为艰难梭菌必需基因检测手段[47]。2010年Minton研究组发表了使用ClosTron技术对艰难梭菌毒素基因的研究结果,他们发现Toxin A和Toxin B都具有细胞毒性,并都能够单独在仓鼠模型中诱导肠道疾病的发生[15]。
ClosTron系统的核心元件二型内含子(GroupⅡintron)是具有催化活性的核酶,其剪接机制与Ⅰ型内含子的剪接相似,也需要形成一个套索的中间体,通过5′-2′磷酸二酯键将剪接的位点连接到一起。与Ⅰ型内含子不同的是Ⅱ型内含子具有自我剪接的功能(核酶活性),不需要剪接体和核小RNA (snRNA)的参与,也不需要ATP供能[48]。
ClosTron系统借助Ⅱ型内含子“归巢”效应,即具有核酶活性Ⅱ型内含子RNA在IEP (Intron encoded protein)的协助下,插入DNA链靶位点的过程,实现任意靶基因的插入失活。来源于乳酸球菌的Ll.ltrBⅡ型内含“归巢”过程分为两个阶段(图 4)。第一阶段,Ⅱ型内含子RNA前体“自我拼接”形成套索RNA和重组的外显子(图 4A)[49];第二阶段,套索RNA在逆转录酶(RT)辅助下“归巢”至靶DNA位点[50](图 4)。
综上所述,Ⅱ型内含子由具有核酶催化活性的Ⅱ型内含子RNA和IEP组成。Ⅱ型内含子RNA“自我拼接”成套索RNA,套索RNA与IEP形成核糖核蛋白复合体(RNPs),RNPs通过“归巢”效应特异性地插入、失活靶基因,该过程可以通过重新编码内含子识别位点,实现打靶位点的“重编程”[51]。
4 基于Himar1C9的艰难梭菌随机失活工具(2010年)如前文所述,Tn916转座子被用于艰难梭菌基因编辑[10],然而,Tn916转座子识别位点特异性高,突变体库丰度低,不适合改造成基因组随机失活工具[16]。来源于角蝇Haematobia irrtans的Himar1C9转座子,属于mariner/Tc1转座子家族[52-53],靶向TA二核苷酸识别位点[54],可以在多种微生物基因组中活跃跳跃[55-56]。其转座过程如图 5所示,DNA双链被转座子切割出2 nt粘性末端,产生3′-羟基。切割产生的转座子可在染色体中跳跃,插入基因组中其他TA位点[54]。Mimar19转座子靶序列识别范围广,且相对严谨,因此构建突变株单插入率高达98.3%;不仅如此,Himar1可自我催化完成转座全过程,无需其他蛋白辅助,便于打靶载体构建。因此,Cartman等将Himar1C9改造成了艰难梭菌随机突变工具,并应用该工具建立了艰难梭菌R20291突变体库。对该突变体库的筛选,获得了芽孢复苏蛋白酶基因cspBA突变株和嘧啶生物合成营养缺陷型pyrB基因突变株。
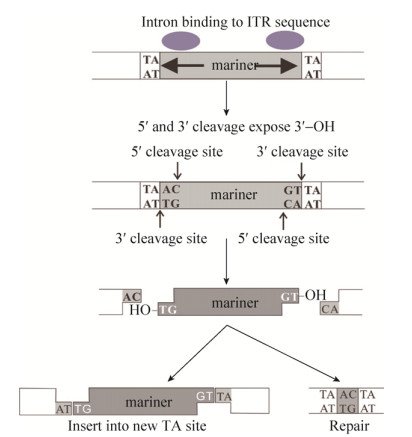 |
| 图 5 mariner/Tc1转座子家族转座模型[16] Fig. 5 Transposition mechanism of mariner/Tc1 family transposon[16]. |
| 图选项 |
5 基于codA基因的精确基因失活系统(2012年)Cartman等将来源于大肠杆菌的胞嘧啶脱氨基酶基因(Cytosine deaminase gene,codA)作为负筛选标记,在艰难梭菌中建立了无痕基因编辑系统。CodA蛋白催化胞嘧啶脱氨基形成尿嘧啶。该酶底物特异性不高,也可以催化无毒嘧啶类似物5-氟胞嘧啶(5-fluorocytosine,5-FC)为具有较强细胞毒性的5-氟尿嘧啶(5-fluorouracil,5-FU)。5-FU在尿嘧啶磷酸生物合成转移酶(Uracil phosphoribosyltransferase,EC 2.4.2.9)的催化下,经过下游反应不可逆地抑制胸腺嘧啶合成酶的活性(核苷酸生物合成的关键酶),并错误地将5-FU引入DNA和RNA中,导致宿主死亡。利用CodA可将无毒5-FC转化为较强细胞毒性的5-FU的特性,将codA基因与尿素磷酸转移酶(UPP)基因共表达,作为反向筛选标记,可实现艰难梭菌无痕的靶向基因敲除(图 6)。有趣的是,单交换的艰难梭菌在BHIS平板上形成的菌落因比未发生单交换菌株形成的菌落大,可以直观地通过菌落的大小判断是否发生单交换,因此该质粒骨架也被形象地称为“假自杀”质粒[18]。
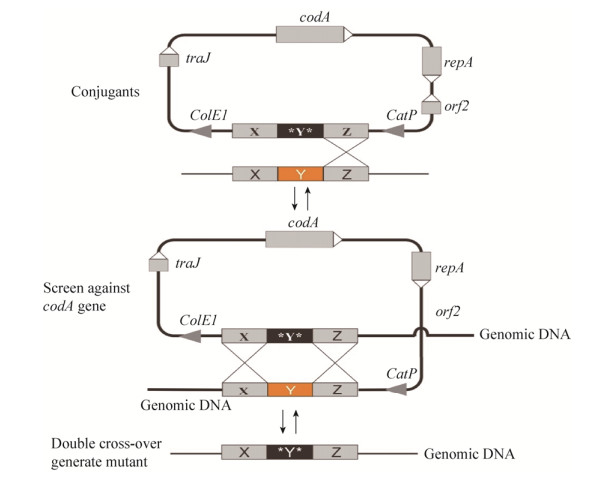 |
| 图 6 基于codA基因的无痕基因失活过程[18] Fig. 6 Scar-free gene knock-out system based on codA gene[18]. |
| 图选项 |
6 基于非等长同源臂偶联等位交换的艰难梭菌精确、无痕基因编辑技术(2013年)2012年Heap等发表了非等长同源臂偶联等位交换(Allele-coupled exchange,ACE)基因编辑方法[57]。该方法基于ΔpyrE突变株(乳清酸磷酸核糖基转移酶突变株,orotate phosphoribosyltransferasemutant)和非等长同源臂偶联等位交换,可通过不同长度同源臂精确控制单、双交换发生的次序,从而实现高效、无痕基因敲除。pyrE基因是嘧啶从头生物合成的必需基因。基于pyrE基因可实现双向筛选:1)该基因缺失,会造成突变株无法从头合成尿嘧啶,导致细菌死亡;2)该基因存在,可将5-FU掺入DNA和RNA分子中,导致细菌死亡。换言之,pyrE基因缺失突变株不能在尿嘧啶限制性培养基中生长,而含有pyrE基因的菌株不能在含有5-氟乳清酸(5-fluoroorotic acid,5-FOA,可转化为5-FU)的培养基中生长。因此,pyrE基因可作为正向(含有pyrE生存)和负向(含有pyrE基因死亡)筛选标记[57]。ACE方法对于不能直接转化自杀质粒、难于遗传改造的菌株(如艰难梭菌、热纤梭菌等)具有重要意义,可以极大地加速突变的筛选速度,且不会像ClosTron技术一样在基因组中引入外源DNA片段,因此可减少极性效应的产生和提高基因突变株染色体的稳定性。
使用ACE方法构建pyrE基因突变株的过程如图 7A所示,pyrE敲除载体包含CatP、ColE1、TraJ、RepH复制蛋白,以及pyrE基因上游同源臂(LHA,约300 bp)、LacZα片段和下游同源臂(RHA,约1 200 bp)。该质粒转化艰难梭菌后,在氯霉素培养基中筛选发生单交换的菌株(图 7A)。单交换菌株扩增后再涂布在5-FOA平板上,发生单交换菌株因含有pyrE基因被淘汰(负筛选)。发生双交换的菌株因pyrE基因缺失而对5-FOA具有抗性,即获得ΔpyrE突变株。ΔpyrE突变株因对5-FOA具有抗性且在尿嘧啶限制性培养基中不能生长,用作后续基因敲除的底盘细胞。
 |
| 图 7 基于ACE的艰难梭菌精确、无痕基因编辑技术[19] Fig. 7 Precise, scar-free genome editing system based on ACE in C. difficile[19]. |
| 图选项 |
以ΔpyrE突变株作为底盘细胞,cwp84突变株构建过程如图 7B所示。敲除载体包含300 bp上游同源臂、携带突变的cwp84基因和1 200 bp下游同源臂,另外还包含来源于产孢梭菌的pyrE基因(与CD630菌株pyrE基因仅有65%同源性,可降低同源重组概率,图 7B)。该载体转化进入CD630后,首先在氯霉素培养基中筛选单交换菌株。由于未发生单交换的质粒会造成营养负担,因此在氯霉素平板上形成小菌落。相反,发生单交换的菌落,因为抗生素基因已经整合进入基因组,营养负担小,形成较大菌落。挑取发生单交换的大菌落涂布于含有5-FOA的平板上,单交换菌株因携带外源pyrE基因,所以对5-FOA敏感,因此发生双交换的ΔpyrEΔcwp84菌株能够生存下来。
在获得目标基因的突变株后,pyrE基因被自身的启动子或fdx强启动子回补至艰难梭菌基因组中,以确保突变株的鉴定在尿嘧啶合成充分的情况下进行(图 7)。利用ACE技术,作者在艰难梭菌菌株中成功敲除了spo0A、cwp84 (Cysteine protease)、mtlD (Mannitol-1-phosphate)基因[19]。Δspo0A产芽孢能力比野生型菌株低104倍,Δcwp84菌株对S-layer protein A (SlpA)蛋白切割效率急剧降低,ΔmtlD丧失了甘露醇利用能力[19]。
7 CRISPR-Cas9系统(2017-2018年)串联规律间隔短回文序列(Cluster regularly interspaced short palindromic repeats,CRISPR)和CRISPR相关蛋白(CRISPR-associated protein,Cas)组成的CRISPR-Cas系统是一种RNA和Cas蛋白介导的细菌和古菌免疫系统[58]。最近几年,来源于化脓性链球菌Streptococcus pyogenes的CRISRPR-Cas9元件被广泛应用于原核生物和真核生物的精确基因编辑[58-72]。CRISPR-Cas9系统有2个主要的组分:1) Cas9效应蛋白;2)用于指导Cas9蛋白识别靶位点的crRNA (CRISPRRNA)和tracrRNA (trans-activating crRNA)。Cas9效应蛋白与crRNA、tracrRNA组成蛋白质核酸复合体(RNP)能够特异性的识别PAM序列(Protospacer adjacent motif,PAM,5′-NGG-3′)前的20 nt核苷酸,并在Cas9核酸内切酶的作用下产生双链DNA平末端缺口(Double-strand break,DSB)。为了简化CRISPR-Cas9系统,Mali等将crRNA和tracrRNA整合在一起,形成单一嵌合型导向RNA (Single chimeric guide RNA,sgRNA),sgRNA同样具有指导Cas9蛋白识别靶位点的功能[73]。在真核生物中,DSB缺口可以通过非同源性末端连接系统修复(Nonhomologous end-joining repair pathway,NHEJ),并在修复过程中会产生插入/删除突变,从而导致基因编码的蛋白质读码框发生移码突变,造成靶基因的失活。然而,在大多原核生物中,由于缺乏NHEJ酶系,CRISPR-Cas9系统在基因组双链中产生DSB后,微生物细胞不能自主修复,产生DNA复制缺陷,导致宿主细菌死亡。当基因打靶质粒提供DSB切割位点两侧同源臂时,断裂基因组发生依赖同源重组的基因修复,(Homology-directed repair,HDR),其结果为:1)细菌得以存活;2)依赖同源臂,引入DNA突变(插入、缺失、删除等)[74]。
艰难梭菌中的CRISPR-Cas9系统由McAllister等于2017年建立。该系统使用脱水四环素诱导启动子(tetR)驱动密码子优化的Cas9基因的表达,使用改造后的谷氨酸脱氢酶(gdh)启动子启动sgRNA表达,靶基因(selD)同源臂被放在结合转移元件traJ和tetR启动子之间(图 8)。该系统对selD基因(Selenophosphate synthetase)的敲除效率为20%-50%,ΔselD突变株生长速度极度降低,缺失了将硒元素螯合进入硒蛋白的能力[28]。CRISPR-Cas9技术的应用,克服了ClosTron基因失活时插入二型内含子而产生的极性效应(基因插入引起的上、下游基因表达变化)和ACE方法需要先构建ΔermΔpyrE底盘细胞,敲除靶基因后再回补pyrE基因的繁琐过程,是目前艰难梭菌基因编辑工具中最简单、高效的方法。
 |
| 图 8 CRISPR-Cas9技术敲除艰难梭菌Spo0A基因和敲入PpFbFpm基因[29] Fig. 8 Knock-out Spo0A gene and knock-in PpFbFpm gene in C. difficile by using CRISPR-Cas9 system[29]. |
| 图选项 |
2018年,本课题组(王邵花、洪伟等)独立构建了一套CRISPR-Cas9系统,该系统以乳糖诱导启动子启动Cas9蛋白的表达,以小RNA启动子(sRNAP)启动sgRNA的表达。使用该质粒系统实现了CD630菌株的spo0A基因的敲除(图 8A)和厌氧绿色荧光蛋白PpFbFpm基因的敲入(图 8B),效率分别为:80%和100%[29]。表型分析结果表明,Δspo0A突变株产生芽孢的能力急剧下降,PpFbFpm基因插入突变株在荧光显微镜下可见明显的绿色荧光信号[29]。
8 CRISPR-Cpf1系统(2018年)与CRISPR-Cas9系统相似,CRISPR-Cpf1系统也是基于核酸内切酶的细菌免疫系统。CRISPR-Cpf1系统来源于Francisella nocidida U112菌株[70],与Cas9相比CRISPR-Cpf1具有以下独特的特点:1) Cpf1核酸酶仅由单一crRNA导向,不需要tracrRNA参与;2) Cpf1核酸内切酶识别富含T的PAM序列(5′-TTTN-3′),更适合低GC含量微生物基因编辑(如艰难梭菌);3) Cpf1在远离PAM序列端,将DNA双链切割为粘性末端[65]。
我们研究组将CRISPR-Cpf1系统开发成了高效艰难梭菌基因编辑“工具箱”[30]。该“工具箱”中的核心“工具”为CRISPR-Cpf1打靶质粒(图 9),其组成如下:Cpf1基因在乳糖诱导启动子(iLacP)的启动下表达,cpf1基因下游紧跟一个终止子(thlT);以小RNA启动子(Small RNA promoter,sRNAP)启动crRNA基因的表达。根据CRISPR-Cpf1系统仅需crRNA无需tracrRNA的特点,我们提出了一步组装法(One step assembly, OSA)。该方法将sRNAP::crRNA与靶基因上、下游同源臂同时组装,从而将基因打靶质粒的构建周期缩短至原来的一半,仅用两天时间就可以获得基因打靶载体。除此之外,我们还为OSA方法开发了靶位点寻找和突变引物设计软件(OSAprimer finder,OPF),可以在1分钟内设计好构建打靶载体所需的引物。OSA方法与OPF工具结合,极大地提高了艰难梭菌的基因编辑效率[30]。
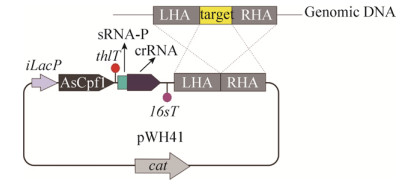 |
| 图 9 使用CRISPR-Cpf1技术实现在艰难梭菌基因编辑[30] Fig. 9 Achieve genome editing in C. difficile using CRISPR-Cpf1 system[30]. |
| 图选项 |
我们使用以上建立的CRISPR-Cpf1“工具箱”,针对CD630菌株fur (ferric uptake regulator)、tetM (tetracycline resistance protein)、ermB1/2 (erythromycin resistance protein 1/2)、cwp66、tcdA (Toxin A)、phiCD630-2 (CD630 phage-2)基因进行了靶向基因敲除,基因敲除效率从25%-100%不等。并首次在CD 630菌株中实现了使用同一载体同时编辑两个基因(cwp66和tcdA)。另外,CRISPR-Cpf1技术还实现了长达49 kb的phiCD630-2基因座敲除,这是目前艰难梭菌中最长的靶基因敲除成功案例。另外,基于该系统的打靶质粒可以通过连续传代的方式丢失,因此能够实现抗性基因的回收,从而实现使用同一筛选标记连续敲除不同基因(如tetM和erm1/2基因)。综上所述,我们开发的CRISPR-Cpf1“工具箱”可以极大加速艰难梭菌基因编辑进程,为揭示艰难梭菌生理生化特征、致病机制和开发对抗CDI的新疗法提供技术支持[30]。
9 基因编辑系统展望本文综述了艰难梭菌基因编辑技术的发展历程和最新进展。2002-2004年之间,艰难梭菌限制性内切酶系统得以揭示、克隆了pCD6复制子、实现了外源质粒的结合转移,这些方法的建立、元件的获得,为艰难梭菌基因编辑技术奠定了基础。艰难梭菌基因编辑技术经历了反义RNA、随机转座子、ClosTron技术、ACE技术、CRISPR-Cas技术等不同阶段[10-11, 16, 19, 25, 29, 30]。实现了从随机基因失活到靶向基因失活、从低效率基因失活到高效基因失活的技术进步(表 1)。随着基因编辑技术的发展,艰难梭菌毒力基因在CDI发生过程中的作用[14]、孢子产生机制(spo0A)[12]、肠壁黏附机制(cwp66,cwp2)[10, 30, 40, 75]、抗生素抗性基因功能(tetM,ermB)等得以阐明[30]。由此可见快速、高效、准确的基因编辑技术,在艰难梭菌致病机制、生理、生化特征的研究过程中起到了核心作用,在未来可能进一步影响CDI的防治措施和策略的制定,为更好地预防和控制CDI感染提供理论支持。
表 1 艰难梭菌中不同基因编辑方法比较Table 1 Comparison of different genome editing methods in Clostridioes difficile
| Items | Random transposon | Antisense RNA | ClosTron | ACE | CRISPR-Cas9 | CRISPR-Cpf1 |
| Polar effect | Yes | No | Yes | No | No | No |
| Defective chassis cell | Yes | No | Yes | Yes | No | No |
| Selection marker | Non-recyclable | Non-recyclable | Recyclable | Recyclable | Recyclable | Recyclable |
| Mutant | Insertion | Knock down | Insertion | Clean | Clean | Clean |
| Work load | Light | Light | Light | Heavy | Medium | Medium |
| Targeting site | Random | Specific | Specific | Specific | Specific | Specific |
| Off-target efficiency | High | High | High | Low | Low | Low |
| Transformation efficiency | High | High | High | Medium | Low | Low |
| References | [16] | [10] | [11] [24-27] | [19] | [28-29] | [30] |
表选项
除了外源性CRISPR-Cas系统,艰难梭菌内源性CRISPR-Cas也是研究的热点。在217株艰难梭菌基因组中,内源性IB型(type IB)CRISPR- Cas系统存在于保守的基因组位点[76]。深度转录组测序发现CD630菌株Ⅰ型CRISPR系统(typeⅠ)具有高度的转录活性[77]。Boudry等的研究阐明了在CD630和R20291菌株中的ⅠB型CRISPR系统的作用机制,并在体外验证了该系统的活性[78]。CD630和R20291分别编码12个和13个可能的CRISPR阵列,其中5个定位于原噬菌体(Prophage)区域。转录组数据表明,在实验室条件下,CD630和R20291分别有12个和9个CRISPR阵列都处于活性转录状态。体外实验表明该系统在大肠杆菌中具有双链DNA靶向切割活性。由此可见,艰难梭菌内源性CRISPR-Cas系统具有改造成为基因编辑工具的潜力。“绑架”艰难梭菌内源性CRISPR-Cas系统用于其基因编辑,无需外源表达Cas9/Cpf1效应蛋白,因此可极大地降低打靶载体的分子量,提高外源质粒的转化效率,从而提高基因编辑的成功率。使用ⅠB型CRISPR系统完成基因编辑已有先例,如张杰等使用酪丁酸梭菌Clostridium tyrobutyricum内源性的CRISPR系统构建的基因编辑工具,编辑酪丁酸梭菌产溶剂代谢通路,获得了目前为止最高的丙酮-丁醇-乙醇产量[79]。由此可见,在艰难梭菌中建立内源CRISPR系统也很有可能进一步提高艰难梭菌基因编辑效率。
CRISPR-Cas系统在基因组中产生的DSB有两种修复方式,一种是HDR,一种是NHEJ。在大多数原核生物中,没有NHEJ修复方式。Yang等在耻垢分枝杆菌Mycobacterium smegmatis中测试了14种Cas效应蛋白,发现CRISPR-FnCpf1_cg系统能够在该菌株中实现NHEJ型的基因编辑[80]。NHEJ型DSB修复具有修复速度快、载体构建简单等优点(无需同源臂)。但是NHEJ的实现需要宿主中具备NHEJ支持酶系统。遗憾的是,艰难梭菌并不具备NHEJ酶系。在艰难梭菌中外源表达NHEJ支持酶系统以实现NHEJ型DBS修复,可能进一步提高其基因编辑的效率。
CRISPR-Cas系统在艰难梭菌中的成功建立,极大地加快了艰难梭菌基因编辑效率。该系统具有高效、无痕、操作简单等诸多优点,也赋予了基因编辑超高的灵活性。王义等使用CRISPR-Cas9系统在拜氏梭菌中实现了单碱基编辑[71],在基因组编辑的精度上有了较大提升,对于精确改变核心基因的功能具有重要意义。目前,单碱基编辑在艰难梭菌中尚未见报道,可能是未来研究的重要方向。
目前艰难梭菌基因编辑方法的研究还有以下不足:1)艰难梭菌转化效率较低。实现外源基因的转化是基因编辑的最重要的前提条件,然而目前艰难梭菌仅适用于转化效率较低的转化方法。因此,进一步提高艰难梭菌外源质粒转化效率,可以提升其基因编辑效率,如开发高效的电转化方法等。2) CRISPR-Cas系统在艰难梭菌中是否存在脱靶现象尚未阐明。有报道称,Cas9蛋白在微生物中具有毒性[74],毒性的产生可能与Cas9异常脱靶有关。在未来的研究中,利用生物信息学技术分析打靶严谨位点的特征,从而设计出更加精确的“基因剪刀”是CRISPR-Cas系统需要解决的技术难题之一。目前,针对艰难梭菌中的CRISPR-Cas9脱靶的问题还没有深入研究。因此,借鉴其他物种中解决CRISPR系统脱靶问题的方法,进一步完善和改进该系统是未来重要的研究方向。3)艰难梭菌高通量基因编辑方法尚待开发。目前在艰难梭菌中的基因编辑技术主要针对单基因和最多两个基因的编辑[74]。有报道称,使用转座子携带的dCas9系统,可以实现同时在多个物种中、调控多个基因的表达[81]。这种高通量基因编辑方式,对于研究病原微生物对抗生素的耐药性及治疗“超级”细菌的感染患者提供了新的思路。可能在不久的将来,面对耐药艰难梭菌,也许我们可以首先通过该方法,改变其对抗生素的敏感性,从而使用现有的抗生素有效地根除CDI。
综上所述,艰难梭菌的基因编辑系统在过去的20年间取得了长足的进步。这些进步推动了艰难梭菌生理、生化特征、致病机制、治疗及防控的研究。随着以上机制的阐明,相信在不久的将来,临床医生可以更好地预防和治疗CDI。
参考文献
| [1] | Hall IC, O'Toole E. Intestinal flora in new-born infants: with a description of a new pathogenic anaerobe, Bacillus difficilis. Am J Dis Child, 1935, 49(2): 390-402. DOI:10.1001/archpedi.1935.01970020105010 |
| [2] | Collins DA, Elliott B, Riley TV. Molecular methods for detecting and typing of Clostridium difficile. Pathology, 2015, 47(3): 211-218. DOI:10.1097/PAT.0000000000000238 |
| [3] | He M, Miyajima F, Roberts P, et al. Emergence and global spread of epidemic healthcare-associated Clostridium difficile. Nat Genet, 2013, 45(1): 109-113. DOI:10.1038/ng.2478 |
| [4] | Tang CJ, Cui LB, Xu YQ, et al. The incidence and drug resistance of Clostridium difficile infection in Mainland China: a systematic review and meta-analysis. Sci Rep, 2016, 6: 37865. DOI:10.1038/srep37865 |
| [5] | Freeman J, Bauer MP, Baines SD, et al. The changing epidemiology of Clostridium difficile infections. Clin Microbiol Rev, 2010, 23(3): 529-549. DOI:10.1128/CMR.00082-09 |
| [6] | Kelly CP, LaMont JT. Clostridium difficile—more difficult than ever. N Engl J Med, 2008, 359(18): 1932-1940. DOI:10.1056/NEJMra0707500 |
| [7] | Lessa FC, MuY, Bamberg WM, et al. Burden of Clostridium difficile infection in the United States. N Engl J Med, 2015, 372(9): 825-834. DOI:10.1056/NEJMoa1408913 |
| [8] | Abdullatif VN, Noymer A. Clostridium difficile infection: an emerging cause of death in the twenty-first century. Biodemography Soc Biol, 2016, 62(2): 198-207. DOI:10.1080/19485565.2016.1172957 |
| [9] | Centers for Disease Control and Prevention. Antibiotic resistance threats in the United States. Atlanta: Centers for Disease Control and Prevention, 2013. |
| [10] | Roberts AP, Hennequin C, Elmore M, et al. Development of an integrative vector for the expression of antisense RNA in Clostridium difficile. J Microbiol Methods, 2003, 55(3): 617-624. DOI:10.1016/S0167-7012(03)00200-8 |
| [11] | Heap JT, Pennington OJ, Cartman ST, et al. The ClosTron: a universal gene knock-out system for the genus Clostridium. J Microbiol Methods, 2007, 70(3): 452-464. DOI:10.1016/j.mimet.2007.05.021 |
| [12] | Burns DA, Heap JT, Minton NP. Clostridium difficile spore germination: an update. Res Microbiol, 2010, 161(9): 730-734. DOI:10.1016/j.resmic.2010.09.007 |
| [13] | Burns DA, Heap JT, Minton NP. SleC is essential for germination of Clostridium difficile spores in nutrient-rich medium supplemented with the bile salt taurocholate. J Bacteriol, 2010, 192(3): 657-664. DOI:10.1128/JB.01209-09 |
| [14] | Kuehne SA, Cartman ST, Heap JT, et al. The role of toxin A and toxin B in Clostridium difficile infection. Nature, 2010, 467(7316): 711-713. DOI:10.1038/nature09397 |
| [15] | Lyras D, O'Connor JR, Howarth PM, et al. Toxin B is essential for virulence of Clostridium difficile. Nature, 2009, 458(7242): 1176-1179. DOI:10.1038/nature07822 |
| [16] | Cartman ST, Minton NP. A mariner-based transposon system for in vivo random mutagenesis of Clostridium difficile. Appl Environ Microbiol, 2010, 76(4): 1103-1109. DOI:10.1128/AEM.02525-09 |
| [17] | Cartman ST, Kelly ML, Heeg D, et al. Precise manipulation of the Clostridium difficile chromosome reveals a lack of association between the tcdC genotype and toxin production. Appl Environ Microbiol, 2012, 78(13): 4683-4690. DOI:10.1128/AEM.00249-12 |
| [18] | Ehsaan M, Kuehne SA, Minton NP. Clostridium difficile genome editing using pyrE alleles. Methods Mol Biol, 2016, 1476: 35-52. |
| [19] | Ng YK, Ehsaan M, Philip S, et al. Expanding the repertoire of gene tools for precise manipulation of the Clostridium difficile genome: allelic exchange using pyrE alleles. PLoS ONE, 2013, 8(2): e56051. DOI:10.1371/journal.pone.0056051 |
| [20] | Olling A, Seehase S, Minton NP, et al. Release of TcdA and TcdB from Clostridium difficile cdi 630 is not affected by functional inactivation of the tcdE gene. Microb Pathog, 2012, 52(1): 92-100. DOI:10.1016/j.micpath.2011.10.009 |
| [21] | Barketi-Klai A, Monot M, Hoys S, et al. The flagellin fliC of Clostridium difficile is responsible for pleiotropic gene regulation during in vivo infection. PLoS ONE, 2014, 9(5): e96876. DOI:10.1371/journal.pone.0096876 |
| [22] | Donald RGK, Flint M, Kalyan N, et al. A novel approach to generate a recombinant toxoid vaccine against Clostridium difficile. Microbiology, 2013, 159(7): 1254-1266. |
| [23] | Purdy D, O'Keeffe TAT, Elmore M, et al. Conjugative transfer of clostridial shuttle vectors from Escherichia coli to Clostridium difficile through circumvention of the restriction barrier. Mol Microbiol, 2002, 46(2): 439-452. DOI:10.1046/j.1365-2958.2002.03134.x |
| [24] | Heap JT, Cartman ST, Kuehne SA, et al. ClosTron-targeted mutagenesis. Methods Mol Biol, 2010, 646: 165-182. |
| [25] | Heap JT, Kuehne SA, Ehsaan M, et al. The ClosTron: mutagenesis in Clostridium refined and streamlined. J Microbiol Methods, 2010, 80(1): 49-55. DOI:10.1016/j.mimet.2009.10.018 |
| [26] | Kuehne SA, Heap JT, Cooksley CM, et al. ClosTron-mediated engineering of Clostridium. Methods Mol Biol, 2011, 765: 389-407. |
| [27] | Kuehne SA, Minton NP. ClosTron-mediated engineering of Clostridium. Bioengineered, 2012, 3(4): 247-254. |
| [28] | McAllister KN, Bouillaut L, Kahn JN, et al. Using CRISPR-Cas9-mediated genome editing to generate C. difficile mutants defective in selenoproteins synthesis. Sci Rep, 2017, 7(1): 14672. DOI:10.1038/s41598-017-15236-5 |
| [29] | Wang S, Hong W, Dong S, et al. Genome engineering of Clostridium difficile using the CRISPR-Cas9 system. Clin Microbiol Infect, 2018, 24(10): 1095-1099. DOI:10.1016/j.cmi.2018.03.026 |
| [30] | Hong W, Zhang J, Cui GZ, et al. Multiplexed CRISPR-Cpf1-mediated genome editing in Clostridium difficile toward the understanding of pathogenesis of C. difficile infection. ACS Synth Biol, 2018, 7(6): 1588-1600. DOI:10.1021/acssynbio.8b00087 |
| [31] | Sebaihia M, Wren BW, Mullany P, et al. The multidrug-resistant human pathogen Clostridium difficile has a highly mobile, mosaic genome. Nat Genet, 2006, 38(7): 779-786. DOI:10.1038/ng1830 |
| [32] | van Eijk E, Anvar SY, Browne HP, et al. Complete genome sequence of the Clostridium difficile laboratory strain 630Δerm reveals differences from strain 630, including translocation of the mobile element CTn5. BMC Genomics, 2015, 16(1): 31. DOI:10.1186/s12864-015-1252-7 |
| [33] | Swinfield TJ, Jannière L, Ehrlich SD, et al. Characterization of a region of the Enterococcus faecalis plasmid pAMβ1 which enhances the segregational stability of pAMβ1-derived cloning vectors in Bacillus subtilis. Plasmid, 1991, 26(3): 209-221. |
| [34] | Minton N, Carter G, Herbert M, et al. The development of Clostridium difficile genetic systems. Anaerobe, 2004, 10(2): 75-84. DOI:10.1016/j.anaerobe.2003.11.003 |
| [35] | Lesiak JM, Liebl W, Ehrenreich A. Development of an in vivo methylation system for the solventogen Clostridium saccharobutylicum NCP 262 and analysis of two endonuclease mutants. J Biotechnol, 2014, 188: 97-99. DOI:10.1016/j.jbiotec.2014.07.005 |
| [36] | Cui GZ, Hong W, Zhang J, et al. Targeted gene engineering in Clostridium cellulolyticum H10 without methylation. J Microbiol Methods, 2012, 89(3): 201-208. DOI:10.1016/j.mimet.2012.02.015 |
| [37] | Dong HJ, Zhang YP, Dai ZJ, et al. Engineering Clostridium strain to accept unmethylated DNA. PLoS ONE, 2010, 5(2): e9038. DOI:10.1371/journal.pone.0009038 |
| [38] | Croux C, Nguyen NPT, Lee J, et al. Construction of a restriction-less, markerless mutant useful for functional genomic and metabolic engineering of the biofuel producer Clostridium acetobutylicum. Biotechnol Biofuels, 2016, 9: 23. DOI:10.1186/s13068-016-0432-2 |
| [39] | Pyne ME, Moo-Young M, Chung DA, et al. Development of an electrotransformation protocol for genetic manipulation of Clostridium pasteurianum. Biotechnol Biofuels, 2013, 6: 50. DOI:10.1186/1754-6834-6-50 |
| [40] | Waligora AJ, Hennequin C, Mullany P, et al. Characterization of a cell surface protein of Clostridium difficile with adhesive properties. Infect Immun, 2001, 69(4): 2144-2153. DOI:10.1128/IAI.69.4.2144-2153.2001 |
| [41] | Mullany P, Wilks M, Tabaqchali S. Transfer of Tn916 and Tn916ΔE into Clostridium difficile: demonstration of a hot-spot for these elements in the C. difficile genome. FEMS Microbiol Lett, 1991, 79(2/3): 191-194. |
| [42] | Volk WA, Bizzini B, Jones KR, et al. Inter- and intrageneric transfer of Tn916 between Streptococcus faecalis and Clostridium tetani. Plasmid, 1988, 19(3): 255-259. |
| [43] | Bertram J, Dürre P. Conjugal transfer and expression of streptococcal transposons in Clostridium acetobutylicum. Arch Microbiol, 1989, 151(6): 551-557. DOI:10.1007/BF00454874 |
| [44] | Guo HT, Karberg M, Long M, et al. Group Ⅱ introns designed to insert into therapeutically relevant DNA target sites in human cells. Science, 2000, 289(5478): 452-457. DOI:10.1126/science.289.5478.452 |
| [45] | Minton NP, Ehsaan M, Humphreys CM, et al. A roadmap for gene system development in Clostridium. Anaerobe, 2016, 41: 104-112. DOI:10.1016/j.anaerobe.2016.05.011 |
| [46] | Zhong J, Karberg M, Lambowitz AM. Targeted and random bacterial gene disruption using a group Ⅱ intron (targetron) vector containing a retrotransposition-activated selectable marker. Nucleic Acids Res, 2003, 31(6): 1656-1664. DOI:10.1093/nar/gkg248 |
| [47] | Walker DJF, Heap JT, Winzer K, et al. A genetic assay for gene essentiality in Clostridium. Anaerobe, 2016, 42: 40-43. DOI:10.1016/j.anaerobe.2016.07.007 |
| [48] | Mohr G, Hong W, Zhang J, et al. A targetron system for gene targeting in thermophiles and its application in Clostridium thermocellum. PLoS ONE, 2013, 8(7): e69032. DOI:10.1371/journal.pone.0069032 |
| [49] | Lambowitz AM, Zimmerly S. Group Ⅱ introns: mobile ribozymes that invade DNA. Cold Spring Harb Perspect Biol, 2011, 3(8): a003616. |
| [50] | Smith D, Zhong J, Matsuura M, et al. Recruitment of host functions suggests a repair pathway for late steps in group Ⅱ intron retrohoming. Genes Dev, 2005, 19(20): 2477-2487. DOI:10.1101/gad.1345105 |
| [51] | Lambowitz AM, Zimmerly S. Group Ⅱ introns: mobile ribozymes that invade DNA. Cold Spring Harb Perspect Biol, 2011, 3(8): a003616. |
| [52] | Hartl DL, Lohe AR, Lozovskaya ER. Modern thoughts on an ancyent marinere: function, evolution, regulation. Annu Rev Genet, 1997, 31: 337-358. DOI:10.1146/annurev.genet.31.1.337 |
| [53] | Plasterk RHA, Izsvák Z, Ivics Z. Resident aliens: the Tc1/mariner superfamily of transposable elements. Trends Genet, 1999, 15(8): 326-332. DOI:10.1016/S0168-9525(99)01777-1 |
| [54] | Lampe DJ, Churchill ME, Robertson HM. A purified mariner transposase is sufficient to mediate transposition in vitro. EMBO J, 1996, 15(19): 5470-5479. DOI:10.1002/j.1460-2075.1996.tb00930.x |
| [55] | Gao LY, Groger R, Cox JS, et al. Transposon mutagenesis of Mycobacterium marinum identifies a locus linking pigmentation and intracellular survival. Infect Immun, 2003, 71(2): 922-929. DOI:10.1128/IAI.71.2.922-929.2003 |
| [56] | Cao M, Bitar AP, Marquis H. A mariner-based transposition system for Listeria monocytogenes. Appl Environ Microbiol, 2007, 73(8): 2758-2761. DOI:10.1128/AEM.02844-06 |
| [57] | Heap JT, Ehsaan M, Cooksley CM, et al. Integration of DNA into bacterial chromosomes from plasmids without a counter-selection marker. Nucleic Acids Res, 2012, 40(8): e59. DOI:10.1093/nar/gkr1321 |
| [58] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278. DOI:10.1016/j.cell.2014.05.010 |
| [59] | Woolston BM, Emerson DF, Currie DH, et al. Rediverting carbon flux in Clostridium ljungdahlii using CRISPR interference (CRISPRi). Metab Eng, 2018, 48: 243-253. DOI:10.1016/j.ymben.2018.06.006 |
| [60] | Xu RF, Qin RY, Li H, et al. Generation of targeted mutant rice using a CRISPR-Cpf1 system. Plant Biotechnol J, 2017, 15(6): 713-717. DOI:10.1111/pbi.12669 |
| [61] | Wasels F, Jean-Marie J, Collas F, et al. A two-plasmid inducible CRISPR/Cas9 genome editing tool for Clostridium acetobutylicum. J Microbiol Methods, 2017, 140: 5-11. DOI:10.1016/j.mimet.2017.06.010 |
| [62] | Wang SH, Dong S, Wang PX, et al. Genome editing in Clostridium saccharoperbutylacetonicum N1-4 with the CRISPR-Cas9 system. Appl Environ Microbiol, 2017, 83(10): e00233-17. |
| [63] | Moreno-Mateos MA, Fernandez JP, Rouet R, et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nat Commun, 2017, 8: 2024. DOI:10.1038/s41467-017-01836-2 |
| [64] | Liu Q, Gao RR, Li JG, et al. Development of a genome-editing CRISPR/Cas9 system in thermophilic fungal Myceliophthora species and its application to hyper-cellulase production strain engineering. Biotechnol Biofuels, 2017, 10: 1. DOI:10.1186/s13068-016-0693-9 |
| [65] | Kim H, Kim ST, Ryu J, et al. CRISPR/Cpf1-mediated DNA-free plant genome editing. Nat Commun, 2017, 8: 14406. DOI:10.1038/ncomms14406 |
| [66] | Jiang Y, Qian FH, Yang JJ, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum. Nat Commun, 2017, 8: 15179. DOI:10.1038/ncomms15179 |
| [67] | Nissim L, Perli SD, Fridkin A, et al. Multiplexed and programmable regulation of gene networks with an integrated RNA and CRISPR/Cas toolkit in human cells. Mol Cell, 2014, 54(4): 698-710. DOI:10.1016/j.molcel.2014.04.022 |
| [68] | Zetsche B, Heidenreich M, Mohanraju P, et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array. Nat Biotechnol, 2017, 35(1): 31-34. DOI:10.1038/nbt.3737 |
| [69] | Joung J, Konermann S, Gootenberg JS, et al. Genome-scale CRISPR-Cas9 knockout and transcriptional activation screening. Nat Protoc, 2017, 12(4): 828-863. DOI:10.1038/nprot.2017.016 |
| [70] | Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038 |
| [71] | Shmakov S, Abudayyeh OO, Makarova KS, et al. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Mol Cell, 2015, 60(3): 385-397. DOI:10.1016/j.molcel.2015.10.008 |
| [72] | Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508 |
| [73] | Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826. DOI:10.1126/science.1232033 |
| [74] | Wang Y, Zhang ZT, Seo SO, et al. Bacterial genome editing with CRISPR-Cas9: deletion, integration, single nucleotide modification, and desirable "clean" Mutant Selection in Clostridium beijerinckii as an example. ACS Synth Biol, 2016, 5(7): 721-732. DOI:10.1021/acssynbio.6b00060 |
| [75] | Bradshaw WJ, Kirby JM, Roberts AK, et al. Cwp2 from Clostridium difficile exhibits an extended three domain fold and cell adhesion in vitro. FEBS J, 2017, 284(17): 2886-2898. DOI:10.1111/febs.14157 |
| [76] | Andersen JM, Shoup M, Robinson C, et al. CRISPR diversity and microevolution in Clostridium difficile. Genome Biol Evol, 2016, 8(9): 2841-2855. DOI:10.1093/gbe/evw203 |
| [77] | Soutourina OA, Monot M, Boudry P, et al. Genome-wide identification of regulatory RNAs in the human pathogen Clostridium difficile. PLoS Genet, 2013, 9(5): e1003493. DOI:10.1371/journal.pgen.1003493 |
| [78] | Boudry P, SemenovaE, Monot M, et al. Function of the CRISPR-Cas system of the human pathogen Clostridium difficile. mBio, 2015, 6(5): e01112-15. |
| [79] | Zhang J, Zong WM, Hong W, et al. Exploiting endogenous CRISPR-Cas system for multiplex genome editing in Clostridium tyrobutyricum and engineer the strain for high-level butanol production. Metab Eng, 2018, 47: 49-59. DOI:10.1016/j.ymben.2018.03.007 |
| [80] | Sun BB, Yang JJ, Yang S, et al. A CRISPR-cpf1-assisted non-homologous end joining genome editing system of Mycobacterium smegmatis. Biotechnol J, 2018, 13(9): 1700588. DOI:10.1002/biot.201700588 |
| [81] | Peters JM, Koo BM, Patino R, et al. Enabling genetic analysis of diverse bacteria with Mobile-CRISPRi. Nat Microbiol, 2019, 4(2): 244-250. DOI:10.1038/s41564-018-0327-z |
