

温州医科大学 眼视光学院 眼视光学和视觉科学国家重点实验室,浙江 温州 325027
收稿日期:2018-10-17;接收日期:2019-01-17
基金项目:国家自然科学基金(No.81201181), 浙江省科技项目(No.2017C37176)资助
摘要:成簇规律间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)系统在近年来得到越来越广泛的应用。相比传统的基因组编辑技术,CRISPR/Cas系统具有编辑效率高、特异性强、花费低、对实验技术要求低等优势。其中Ⅱ型和Ⅴ型CRISPR/Cas系统分别仅需要单个Cas9蛋白和单个Cpf1蛋白作为切割双链DNA的工具,因此特别受到研究者们的青睐。目前CRISPR/Cas9技术已成功应用于斑马鱼、小鼠、人类细胞等真核生物的基因组编辑中,并取得一系列重要成果,但在细菌领域进行的相关研究并不多。文中将简单描述CRISPR/Cas系统及其作用机制,重点介绍该系统的优化以及近年来其在细菌学领域所取得的进展。
关键词:CRISPR/Cas系统细菌基因组编辑优化
Application and optimization of CRISPR/Cas system in bacteria
Junhao Fu, Fayu Yang, Haihua Xie, Feng Gu


State Key Laboratory of Ophthalmology and Optometry, School of Ophthalmology and Optometry, Wenzhou Medical University, Wenzhou 325027, Zhejiang, China
Received: October 17, 2018; Accepted: January 17, 2019
Supported by: National Natural Science Foundation of China (No. 81201181), Science and Technology Projects of Zhejiang Province (No. 2017C37176)
Corresponding author: Feng Gu. Tel: +86-577-88831367; E-mail: gufenguw@gmail.com.
Abstract: Clustered regular interspaced short palindromic repeats (CRISPR) system has been widely used in recent years. Compared with traditional genome editing technology, CRISPR/Cas system has notable advantages, including high editing efficiency, high specificity, low cost and the convenience for manipulation. Type Ⅱ and Ⅴ CRISPR/Cas system only requires a single Cas9 protein or a single Cpf1 protein as effector nucleases for cutting double-stranded DNA, developed as genome editing tools. At present, CRISPR/Cas9 technology has been successfully applied to the genome editing of eukaryotes such as zebrafish, mice and human cells, whereas limited progress has been made in the genome editing of bacteria. In our review, we describe CRISPR/Cas system, its mechanism and summarize the optimization and progress of genome editing in bacteria.
Keywords: CRISPR/Cas systembacteriagenome editingoptimization
CRISPR作为强大的基因组编辑工具,目前在人类细胞[1]、植物[2-4]、动物[5-7]等真核生物中得到了很好的应用并取得了举世瞩目的成果。但是在细菌领域所开展的相关研究并不多。这可能是:1)细菌中基因操作的技术相对成熟高效;2)在大部分细菌中不存在非同源末端连接(Non- homologous end joining,NHEJ)修复机制,因此不能对Cas9蛋白诱导产生的DNA双链断裂进行有效修复,使得以细菌自身内源性基因为靶标的DNA编辑对其造成致死性的损伤,从而限制了CRISPR/Cas技术在细菌学领域的应用[8]。本文综述了CRISPR/Cas系统作用机制,介绍了该系统的优化及其在细菌领域的应用进展,以期为细菌基因组编辑研究提供参考。
1 CRISPR/Cas系统的介绍CRISPR/Cas系统广泛分布于几乎所有的古菌(约90%)和多数细菌(约50%)中[9]。CRISPR/Cas基因座由一系列编码Cas蛋白的基因和一个CRISPR重复间隔序列组成[10] (图 1)。典型的CRISPR重复间隔序列由一段前导序列、一系列短的高度保守的正向重复序列和间隔序列顺序排列组成[11]。关于CRISPR/Cas系统的详细分类等内容,因为已经有多篇相关综述介绍,本文将不作详细介绍,具体请参考相关文献[12-13]。
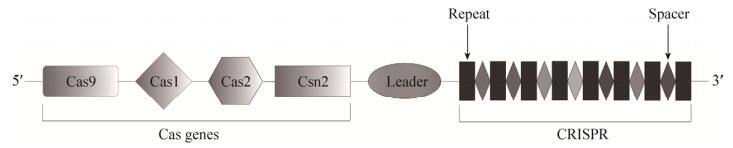 |
| 图 1 CRISPR基因座示意图 Fig. 1 Diagram of CRISPR locus. |
| 图选项 |
细菌CRISPR/Cas系统在抵御外来核酸入侵时所产生的特异性防御过程都可大致分为3个阶段:适应阶段;表达阶段;干扰阶段[14]。但不同类型系统之间也存在一定差异。以CRISPR/Cas9系统为例,当外源核酸首次入侵包含CRISPR/Cas9系统的细菌时,在Cas1和Cas2蛋白的作用下被当作间隔序列并整合到CRISPR阵列中的两段重复序列之间,由此细菌对该外源序列产生记忆;当再次入侵时,细菌的CRISPR免疫系统开始转录Cas9 mRNA、pre-crRNA、tracrRNA,接着tracrRNA与pre-crRNA的部分序列通过碱基互补配对形成复合物,Cas9蛋白稳定复合物并与之形成复合体,再由RNase Ⅲ剪切该复合体产生有活性的Cas9-crRNA-tracrRNA核糖核蛋白复合物;最后成熟的crRNA与入侵的外源DNA通过碱基互补配对结合,再由核糖核蛋白复合物通过识别合适的PAM(Protospacer adjacent motif)序列定位切割的片段,并在Cas9的作用下将其剪切[15-16]。
2 CRISPR/Cas系统在细菌中的应用2.1 对细菌基因组的编辑研究表明,DNA结合蛋白Ku和连接酶LigD是进行非同源末端连接(NHEJ)修复必不可少的成分,但NHEJ相关蛋白仅存在于厚壁菌门、变形菌门、放线菌门等少数菌种中,因此在应用CRISPR/Cas系统编辑细菌的基因组时,需要格外考虑对双链切口的修复,以防止细菌的死亡[17]。在细菌中修复切口的方式包括同源重组(Homologous recombination,HR)修复和NHEJ修复。因此有研究者将这两种方式联合CRISPR/Cas系统对细菌的基因组进行编辑。
2013年,Jiang等将CRISPR/Cas9技术与HR共同应用于基因组编辑[18]。发生同源重组的细菌因Cas9不能继续切割靶基因得以存活,未发生同源重组的细菌则被持续切割而死亡。利用同样的策略在拜氏梭菌[19-20]、放线菌[21-24]、解纤维梭菌[25]等菌种中均能成功实现基因编辑。白仲虎团队利用相似的原理,在谷氨酸棒杆菌中引入HR修复的模板,对Cas9产生的DSB(Double-strand break)进行修复,实现了高效的基因缺失。另外,点突变和基因插入的效率可分别达到100%和66.7%[26]。同时,孙际宾团队开发了一种CRISPR/Cas9介导的单链DNA (Single-stranded DNA,ssDNA)重组工程,可以在谷氨酸棒杆菌的基因组中精确地引入小的修饰和单核苷酸改变,效率超过了80.0%[27]。谷氨酸棒杆菌作为微生物细胞工厂,已被用于各种氨基酸的工业生产,因此这些研究为提高谷氨酸棒杆菌的生产力提供了工具。随后,Penewit等在金黄色葡萄球菌中开发了重组工程和CRISPR/Cas9介导的反选择条件系统,能够在金黄色葡萄球菌基因组中有效和精确地设计点突变和大的单基因缺失[28]。
2014年,科学家在CRISPR系统中引入来自缺陷的RAC前噬菌体的RecBCD重组系统,在罗伊氏乳杆菌中成功地进行基因组编辑[29]。2015年,在CRISPR系统中引入来自λ噬菌体的λred同源重组系统也成功应用于大肠杆菌的基因组编辑中[30]。2016年,Bassalo等同样利用λred同源重组系统,不仅提升了HR修复效率,而且提高了CRISPR系统对大肠杆菌基因组的编辑能力[31]。
除了Cas9蛋白的应用,Cpf1蛋白也被成功地作为细菌基因组编辑的工具,如杨晟团队选用另一种效应蛋白——FnCpf1 (新凶手弗朗西斯菌Francisella novicida Cpf1,也叫Cas12a),并结合ssDNA重组系统,成功地构建了抗L-脯氨酸反馈抑制的高产菌株[32]。
上述研究均利用了HR进行基因组编辑,然而,用这种方法进行编辑时,往往要经过一个相对复杂的DNA编辑模板构建过程,并且在大规模基因组编辑方面也会受到一定的限制。因此,有****通过引入NHEJ相关蛋白来实现简单而有效的基因组编辑。目前,利用NHEJ进行基因组编辑的研究并不多,Tong等通过共表达Cas9蛋白和LigD蛋白,利用NHEJ途径在链霉菌中实现了有效的基因组编辑[22]。随后,祁庆生团队开发了CRISPR-Cas9辅助的非同源末端连接(CA-NHEJ)策略,该策略首先将Cas9以及来自结核分枝杆菌的保守型原核NHEJ相关蛋白转入大肠杆菌,然后将sgRNA表达质粒通过电转方式引入宿主菌,含有导入的NHEJ相关蛋白的宿主菌能够修复DSB,使其在Cas9的切割中存活,并在靶位点产生突变,而野生的宿主菌则被淘汰[33]。同样,赵国屏团队在大肠杆菌中引入Cas9及耻垢分枝杆菌的NHEJ系统,实现了快速的基因失活或片段删除。并且通过进一步的设计,该系统可进行连续的基因失活或DNA片段删除[34]。因此通过引入DNA结合蛋白Ku和连接酶LigD来补救细菌的NHEJ修复途径,可以有效地提升CRISPR系统的编辑效果。
然而,在进行HR修复或NHEJ修复时,都是在靶基因座处引入DSB作为基因校正的第一步,这很容易导致不希望的突变。因此新开发的单碱基编辑系统很好地弥补了这一缺点。季泉江团队便把该策略引入了金黄色葡萄球菌,他们设计了Cas9切口酶(nCas9)和胞苷脱氨酶(APOBEC1)的融合体,通过过早产生终止密码子使基因失活,从而在金黄色葡萄球菌中进行快速有效的遗传操作,加速细菌生理学的研究[35]。同时,该课题组在假单胞菌属物种中也实现了高效的C→T碱基编辑[36]。刘正飞团队利用相似的策略在大肠杆菌中实现了C→T转换[37]。郑平团队、孙际宾团队和王猛团队合作,在谷氨酸棒杆菌中开发了多元自动化碱基编辑方法,同样实现了C→T转换,且进行单、双和三基因座编辑时,效率分别高达100%、87.2%和23.3%[38]。
2.2 CRISPR/Cas系统的筛选优化对于细菌来说,外源性基因通常指从外部引入的其他物种的DNA或人工合成的DNA。一方面,包含CRISPR/Cas系统的细菌,能够利用自身内源性的Cas蛋白对引入的外源核酸进行特异性剪切。该过程是细菌针对噬菌体等外源物质的入侵形成的一套适应性免疫防御机制[15]。正是以该机制为基础,经过人工改造形成了新型的基因组编辑工具——CRISPR/Cas系统。
另一方面,通过引入外源CRISPR/Cas系统,实现对外源基因的剪切。该方法通常应用于对细菌基因组的编辑,用以菌株构建、基因功能研究以及对CRISPR/Cas系统的优化[19-25]。而在对外源基因进行编辑的应用中,****们通常将其作为筛选优化的系统[39-40]。以下将从不同效应蛋白筛选优化系统的建立,进一步阐述基因组编辑在该方面的应用。
2.2.1 基于细菌的SpCas9突变体的筛选优化目前,使用最为广泛的SpCas9 (酿脓链球菌Streptococcus pyogenes Cas9)为野生型,它主要识别的PAMs为NGG (N为A/T/C/G),这导致了SpCas9的应用仅限于包含NGG的序列[15, 18]。Kleinstiver等利用基于细菌的阳性选择试验,筛选能够识别新型PAMs的SpCas9突变体[39]。该实验包含了两种外源性质粒:编码诱导型毒性基因和靶位点的报告质粒(氨苄抗性);Cas9/sgRNA的编码质粒(氯霉素抗性)。将PAM相互作用域(PI)突变的Cas9/sgRNA质粒文库电转到含有报告质粒的感受态BW25141 (λDE3)中,经过活化后涂布在含有诱导剂和氯霉素的LB固体培养基上,若Cas9实现对报告质粒的切割,就会导致毒性基因的丢失,因此细菌就能存活(表 1)。存活的克隆再进行基于人类细胞的EGFP失活试验,最终Kleinstiver等获得了能够识别NGA PAMs的Cas9突变体VQR(D1135V/R1335Q/T1337R)和EQR (D1135E/R1335Q/T1337R),以及能够识别NGCG PAMs的Cas9突变体VRER (D1135V/G1218R/ R1335E/T1337R)。
表 1 CRISPR/Cas系统筛选优化策略Table 1 CRISPR/Cas system screening optimization strategy
| Name | SpCas9 | SaCas9 | Lb/AsCpf1 | SpCas9 |
| Selection system | Positive selection | Positive selection | Negative selection | Positive selection |
| Cas9 plasmid | Include SpCas9, sgRNA and chloramphenicol resistance gene | Include SaCas9, sgRNA and chloramphenicol resistance gene | Include Cpf1, crRNA and chloramphenicol resistance gene | Include ω-dCas9 and phage genes |
| Target plasmid | Include an inducible toxic genes and target site | Include an inducible toxic genes and target site | Include target site and ampicillin resistance gene | Include target site, gene Ⅲ and sgRNA |
| Inefficient or no cleavage | Bacterial death | Bacterial death | Bacterial survival | Gene Ⅲ expression and phage propagation are limited |
| Efficient or cleavage | Bacterial survival | Bacterial survival | Bacterial death | Gene Ⅲ expression and phage propagation |
| Results | VQR; EQR; VRER | KKH | RR; RVR | xCas9 |
表选项
在此基础上,David Liu等利用PACE (Phage-assisted continuous evolution)系统对SpCas9进行了进一步的筛选优化(表 1)[40]。研究者将宿主菌液持续地流过含有SP (Selection phages)的反应池,其中宿主菌含有诱导突变发生的质粒MP (Mutagenesis plasmid)和激活后能表达gene Ⅲ (噬菌体存活的必需基因)的质粒AP (Accessory plasmid);SP可表达ω-dCas9 (无剪切活性的Cas9连接着细菌RNA聚合酶的ω亚基)。当宿主菌液流过SP时,在MP的作用下引起DNA突变,由于宿主菌液的流速比宿主菌细胞分裂快,因此宿主菌不会累积突变;相反若宿主菌液的流速比SP的生命周期慢,SP就会累积突变。SP中不同的突变,就产生了dCas9的突变体文库。若ω-dCas9突变体能够识别并结合到AP (含有PAM的靶序列)上,ω亚基就能将细菌RNA聚合酶募集到靶序列下游的geneⅢ序列前,从而诱导geneⅢ的表达。由于geneⅢ是噬菌体感染所必需的,因此SP上的dCas9突变体与AP上的靶序列结合的越多,geneⅢ表达越多,噬菌体就可以更多地扩增。最终,有活性的dCas9突变体被保留下来,无活性的dCas9突变体被淘汰。David Liu等利用该策略获得了xCas9,此突变体不仅可以识别包括NG、GAA和GAT在内的广泛的PAM序列,而且具有更强的特异性[40]。
2.2.2 基于细菌的SaCas9突变体的筛选优化SaCas9是一种来自金黄色葡萄球菌Staphylococcus aureus的Cas9酶类,其编码序列具有比SpCas9小的特点,因此更适合用于腺相关病毒的包装,展现了其在基因治疗中巨大的潜力[41]。天然的SaCas9识别的PAM序列为NNGRRT (N为A/T/C/G;R为A/G),预计在基因组上每32个碱基会出现一次[42]。为了拓宽SaCas9在基因组上的识别范围,Kleinstiver等首先对SaCas9进行改造,研究者基于其之前对SpCas9突变体的筛选,采用相同的策略,将研究方向转向了SaCas9 (表 1)[42]。该实验首先根据Cas9直系同源物的蛋白质结构域比对,预测了SaCas9的PI结构域;然后对PI进行随机突变,构建了SaCas9突变体文库;再利用基于细菌的阳性选择试验和基于人类细胞的EGFP失活试验确定目标SaCas9突变体。最终研究者成功地筛选出了名为KKH SaCas9的突变体,该突变体在包含NNNRRT PAMs的人类内源性靶位点上显示出强大的基因组编辑活性,从而将SaCas9的靶向范围提高了2–4倍。
2.2.3 基于细菌的Cpf1突变体的筛选优化Cpf1属于2类Ⅴ型CRISPR效应蛋白,与Cas9不同,Cpf1同时具有DNA和RNA内切酶活性,因此不需要RNase Ⅲ参与对pre-crRNA的加工;此外,Cas9剪切产生平末端,而Cpf1剪切产生粘性末端,因此可促进靶基因通过NHEJ方式插入靶位点[43]。野生型的Cpf1能识别富含胸腺嘧啶(T)的PAM序列,如AsCpf1 (氨基酸球菌Acidaminococcus sp. BV3L6 Cpf1)和LbCpf1 (毛螺科菌Lachnospiraceae bacterium ND2006 Cpf1)识别的PAM序列为TTTN (N为A/T/C/G),FnCpf1识别的PAM序列为KYTV(K为G/T,Y为T/C,V为A/C/G)[44]。
为了进一步提高Cpf1的靶向范围,Gao等利用细菌筛选可以识别更多PAM的Cpf1 (AsCpf1和LbCpf1)突变体,从而拓宽Cpf1的靶向范围(表 1)[45]。该研究首先构建了表达crRNA和Cpf1突变体(大部分为单个氨基酸突变)的质粒文库,且质粒上带有氯霉素抗性基因;然后用携带氨苄青霉素抗性基因和带有突变PAM的靶位点的第2个质粒进行转化,若Cpf1突变体成功识别突变的PAM且完成对靶序列的切割,就会导致氨苄青霉素抗性的丢失,结果菌落就不能在含有氨苄的平板上存活;最后将存活的菌株与Cpf1突变体文库进行比较,确定需要的突变体。Gao等利用该方法成功筛选到了RR (S542R/K607R)和RVR (S542R/K548V/N552R)两种突变体,它们能分别识别TYCY和TATV(Y为T/C,V为A/C/G) PAMs,且表现出增强的活性。
2.2.4 单碱基编辑系统的优化目前,科学家们主要开发了两种单碱基编辑系统。第一种可以将C·G碱基对转变为T·A碱基对,称为CBE (Cytidine base editing)[46-49];第二种可以将A·T碱基对转变为G·C碱基对,称为ABE (Adenine base editing)[49-52]。关于ABE对应的腺嘌呤脱氨酶,研究人员通过定向进化和蛋白质工程化改造技术对来自大肠杆菌的脱氨酶TadA进行改造,并开发了一套可以利用细菌对TadA酶进行进化的方法:首先修改抗生素抗性基因的关键位点,使得细菌抗生素抗性失效,作为一种选择质粒;然后构建与dCas9融合的突变体TadA基因的质粒文库,只有TadA酶作用于DNA并将突变位点修正后才能使得抗生素抗性基因恢复正常功能,细菌才能存活[50]。最终,研究人员获得了理想的脱氨酶TadA突变体。ABE目前在定点的编辑(如压缩到只编辑1个碱基)、编辑活性、编辑窗口(扩大编辑窗口)等方面仍然需要强化。
2.3 对细菌基因表达的调控对于某些高度保守且难以敲除或置换的靶基因,常常可通过调控基因的表达水平来达到研究的目的。其中,dCas9 (dead Cas9)在这方面已发展为一种有效的工具。dCas9是Cas9的一种突变体,它在Cas9的两个切割结构域(RuvC和HNH)产生点突变(D10A和H840A),导致Cas9失去对靶序列的剪切作用,但保留了与DNA的结合能力[53]。dCas9通过结合到靶基因的阅读框内或启动子区,阻断转录的延伸或干扰RNA聚合酶(RNAP)与启动子的结合,从而参与基因表达的调控[54-56]。由于具有与RNA干扰技术(RNAi)类似的作用,因此该技术又被称为CRISPR干扰技术(CRISPR interference,CRISPRi)[56]。有****发现,当采用阻断转录延伸的方式时,dCas9结合到非模板链可以达到更好的抑制效果[55-57]。目前,更多的研究是采用干扰转录起始的方式,当dCas9与靶基因的启动子结合,可竞争RNAP在其上的结合位置,从而抑制转录的起始。研究发现,采用后者方式抑制效果更为显著[56, 58]。2013年,Bikard等将dCas9应用于肺炎链球菌,发现dCas9可将β-半乳糖苷酶的表达量下调14倍[54]。2015年,Choudhary等利用dCas9鉴定分枝杆菌中某些重要基因的功能[59]。2016年,Singh等利用dCas9抑制分枝杆菌中某些功能性基因的表达,从而观察分枝杆菌表型上的变化[60]。2018年,Vigouroux等揭示指导RNA和靶标之间的互补性水平控制RNA聚合酶从靶标“踢出”dCas9并完成转录的速率,并且利用这种机制精确且稳健地减少细菌基因的表达[61]。
此外,dCas9还可用于上调基因的表达。将转录激活因子与dCas9融合后,能够显著提升基因的转录水平。该策略已成功应用于真核生物,但在细菌领域的相关报道并不多[62-66]。Bikard等将该策略应用于大肠杆菌,实现了基因的激活[54]。该研究将RNA聚合酶的ω亚基分别与dCas9的C-端和N-端融合,结果发现,当融合蛋白的结合位点处于弱启动子的转录起始位点上游96 nt时,对基因的激活效果最佳。2018年,Dong等在大肠杆菌中开发了一种改进的细菌基因激活工具包[67]。他们通过CRISPR/Cas系统鉴定了几种能够有效激活基因表达的蛋白质,最后使用最有效的激活剂SoxS,在激活一个目的基因的同时可以用CRISPRi抑制不同的靶基因,而且可以用驱动CRISPR/Cas系统组件的诱导型启动子控制整个多基因表达过程。但相比于dCas9对基因的抑制程度,基因激活仍有很大的提升空间。
3 CRISPR/Cas系统总结与展望CRISPR/Cas系统作为新型的第3代基因组编辑技术,与传统的锌指核酸酶(Zinc-finger nucleases,ZFN)技术和类转录激活因子效应物核酸酶(Transcription activator-like effector nucleases,TALEN)技术相比,具有省时省力、设计简单、编辑效率高、特异性强、对实验技术要求低等优点。但是随着深入的研究,该系统的缺陷也逐渐暴露,如具有较高的脱靶效应[68-69]。因此,目前的许多研究都在致力于降低CRISPR/Cas系统的脱靶效应[70]。
除了脱靶效应问题,CRISPR/Cas系统对PAM的需求也限制了其更为广泛的应用。因此,研究人员便主张对现有的Cas蛋白进行改造,以扩大其对PAM的识别范围。如上文所诉的各类Cas蛋白突变体,均在不同程度上扩大了野生型Cas蛋白的识别范围。虽然目前CRISPR/Cas系统对PAM的需求已大大降低,但它仍有可改造的空间。
另外,相比真核生物,CRISPR/Cas系统在细菌及其他原核生物中的应用比较少。细菌等原核生物缺乏NHEJ修复所需的元件,可能是制约其应用的主要因素[8, 22]。科学家除了通过添加NHEJ修复所需元件来降低基因组编辑的毒性外,还有****在对蓝细菌进行基因组编辑时,发现FnCpf1产生的细胞毒性比Cas9低得多[71]。因此,在对不同种类原核生物进行基因组编辑时,效应蛋白的选择可能很关键。同时,单碱基编辑系统的发现为CRISPR/Cas系统在细菌中的应用提供了新的途径。理论上,任何细菌都可以利用单碱基编辑系统来进行操作。单碱基编辑系统的逐渐成熟将为细菌等原核生物的基因组编辑提供更大的操作空间。
对于CRISPR/Cas系统来说,虽然已经取得了很多研究成果,但它仍有很大的提升空间。Cas蛋白突变体的筛选、CRISPR系统保真性的研究、单碱基编辑系统的改善、基因表达调控的应用等都需要更深入的挖掘。另一方面,由于细菌具有经济、易培养、生长迅速等优点,因此可以大大缩短研究周期和减少开支,展现了其在CRISPR系统研究中的巨大潜力。在不断的探索之下,CRISPR/Cas系统将会发展成为更加完善的基因组编辑工具,同时也期待它能在科学研究、临床应用以及工农业生产中带来更大的突破。
参考文献
| [1] | Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells.eLife, 2013, 2: e00471.DOI: 10.7554/eLife.00471 |
| [2] | Ma XL, Zhang QY, Zhu QL, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in Monocot and Dicot Plants.Mol Plant, 2015, 8(8): 1274–1284.DOI: 10.1016/j.molp.2015.04.007 |
| [3] | Wang YP, Cheng X, Shan QW, et al. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew.Nat Biotechnol, 2014, 32(9): 947–951.DOI: 10.1038/nbt.2969 |
| [4] | Zhao YP, Zhang CS, Liu WW, et al. An alternative strategy for targeted gene replacement in plants using a dual-sgRNA/Cas9 design.Sci Rep, 2016, 6: 23890.DOI: 10.1038/srep23890 |
| [5] | Wu MM, Wei CH, Lian ZX, et al. Rosa26-targeted sheep gene knock-in via CRISPR-Cas9 system.Sci Rep, 2016, 6: 24360.DOI: 10.1038/srep24360 |
| [6] | Liao HK, Gu Y, Diaz A, et al. Use of the CRISPR/Cas9 system as an intracellular defense against HIV-1 infection in human cells.Nat Commun, 2015, 6: 6413.DOI: 10.1038/ncomms7413 |
| [7] | Yin H, Xue W, Chen SD, et al. Genome editing with Cas9 in adult mice corrects a disease mutation and phenotype.Nat Biotechnol, 2014, 32(6): 551–553.DOI: 10.1038/nbt.2884 |
| [8] | Vercoe RB, Chang JT, Dy RL, et al. Cytotoxic chromosomal targeting by CRISPR/Cas systems can reshape bacterial genomes and expel or remodel pathogenicity islands.PLoS Genet, 2013, 9(4): e1003454.DOI: 10.1371/journal.pgen.1003454 |
| [9] | Makarova KS, Wolf YI, Alkhnbashi OS, et al. An updated evolutionary classification of CRISPR-Cas systems.Nat Rev Microbiol, 2015, 13(11): 722–736.DOI: 10.1038/nrmicro3569 |
| [10] | Doudna JA, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9.Science, 2014, 346(6213): 1258096.DOI: 10.1126/science.1258096 |
| [11] | Amitai G, Sorek R. CRISPR-Cas adaptation: insights into the mechanism of action.Nat Rev Microbiol, 2016, 14(2): 67–76.DOI: 10.1038/nrmicro.2015.14 |
| [12] | Li C, Cao WG. Advances in CRISPR/Cas9-mediated gene editing.Chin J Biotech, 2015, 31(11): 1531–1542.(in Chinese). 李聪, 曹文广. CRISPR/Cas9介导的基因编辑技术研究进展.生物工程学报, 2015, 31(11): 1531-1542. |
| [13] | Zhan CS, Xia XY. Research progress of CRISPR-Cas9 system for gene therapy.Chin J Biotech, 2016, 32(7): 861–869.(in Chinese). 詹长生, 夏小雨. 基于CRISPR-Cas9技术的基因治疗研究进展.生物工程学报, 2016, 32(7): 861-869. |
| [14] | Mohanraju P, Makarova KS, Zetsche B, et al. Diverse evolutionary roots and mechanistic variations of the CRISPR-Cas systems.Science, 2016, 353(6299): aad5147.DOI: 10.1126/science.aad5147 |
| [15] | Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.Science, 2012, 337(6096): 816–821.DOI: 10.1126/science.1225829 |
| [16] | Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ.Nature, 2011, 471(7340): 602–607.DOI: 10.1038/nature09886 |
| [17] | Bowater R, Doherty AJ. Making ends meet: repairing breaks in bacterial DNA by non-homologous end-joining.PLoS Genet, 2006, 2(2): e8.DOI: 10.1371/journal.pgen.0020008 |
| [18] | Jiang Y, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems.Nat Biotechnol, 2013, 31(3): 233–239.DOI: 10.1038/nbt.2508 |
| [19] | Wang Y, Zhang ZT, Seo SO, et al. Markerless chromosomal gene deletion in Clostridium beijerinckii using CRISPR/Cas9 system.J Biotechnol, 2015, 200: 1–5.DOI: 10.1016/j.jbiotec.2015.02.005 |
| [20] | Wang Y, Zhang ZT, Seo SO, et al. Bacterial genome editing with CRISPR-Cas9: deletion, integration, single nucleotide modification, and desirable "Clean" mutant selection in Clostridium beijerinckii as an example.ACS Synth Biol, 2016, 5(7): 721–732.DOI: 10.1021/acssynbio.6b00060 |
| [21] | Huang H, Zheng GS, Jiang WH, et al. One-step high-efficiency CRISPR/Cas9-mediated genome editing in Streptomyces.Acta Biochim Biophys Sin, 2015, 47(4): 231–243.DOI: 10.1093/abbs/gmv007 |
| [22] | Tong YJ, Charusanti P, Zhang LX, et al. CRISPR-Cas9 based engineering of Actinomycetal genomes.ACS Synth Biol, 2015, 4(9): 1020–1029.DOI: 10.1021/acssynbio.5b00038 |
| [23] | Zeng H, Wen SS, Xu W, et al. Highly efficient editing of the actinorhodin polyketide chain length factor gene in Streptomyces coelicolor M145 using CRISPR/Cas9-CodA(sm) combined system.Appl Microbiol Biotechnol, 2015, 99(24): 10575–10585.DOI: 10.1007/s00253-015-6931-4 |
| [24] | Cobb RE, Wang YJ, Zhao MH. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system.ACS Synth Biol, 2015, 4(6): 723–728.DOI: 10.1021/sb500351f |
| [25] | Xu T, Li YC, Shi Z, et al. Efficient genome editing in Clostridium cellulolyticum via CRISPR-Cas9 nickase.Appl Environ Microbiol, 2015, 81(13): 4423–4431.DOI: 10.1128/AEM.00873-15 |
| [26] | Peng F, Wang XY, Sun Y, et al. Efficient gene editing in Corynebacterium glutamicum using the CRISPR/Cas9 system.Microb Cell Fact, 2017, 16: 201.DOI: 10.1186/s12934-017-0814-6 |
| [27] | Liu J, Wang Y, Lu YJ, et al. Development of a CRISPR/Cas9 genome editing toolbox for Corynebacterium glutamicum.Microb Cell Fact, 2017, 16(1): 205.DOI: 10.1186/s12934-017-0815-5 |
| [28] | Penewit K, Holmes EA, Mclean K, et al. Efficient and scalable precision genome editing in staphylococcus aureus through conditional recombineering and CRISPR/Cas9-mediated counterselection.mBio, 2018, 9(1): e00067–18. |
| [29] | Oh JH, van Pijkeren JP. CRISPR-Cas9-assisted recombineering in Lactobacillus reuteri.Nucleic Acids Res, 2014, 42(17): e131.DOI: 10.1093/nar/gku623 |
| [30] | Pyne ME, Moo-Young M, Chung DA, et al. Coupling the CRISPR/Cas9 system with Lambda red recombineering enables simplified chromosomal gene replacement in Escherichia coli.Appl Environ Microbiol, 2015, 81(15): 5103–5114.DOI: 10.1128/AEM.01248-15 |
| [31] | Bassalo MC, Garst AD, Halweg-Edwards AL, et al. Rapid and efficient one-step metabolic pathway integration in E. coli.ACS Synth Biol, 2016, 5(7): 561–568.DOI: 10.1021/acssynbio.5b00187 |
| [32] | Jiang Y, Qian FH, Yang JJ, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum.Nat Commun, 2017, 8: 15179.DOI: 10.1038/ncomms15179 |
| [33] | Su TY, Liu FP, Gu PF, et al. A CRISPR-Cas9 assisted non-homologous end-joining strategy for one-step engineering of bacterial genome.Sci Rep, 2016, 6: 37895.DOI: 10.1038/srep37895 |
| [34] | Zheng X, Li SY, Zhao GP, et al. An efficient system for deletion of large DNA fragments in Escherichia coli via introduction of both Cas9 and the non-homologous end joining system from Mycobacterium smegmatis.Biochem Biophys Res Commun, 2017, 485(4): 768–774.DOI: 10.1016/j.bbrc.2017.02.129 |
| [35] | Gu TN, Zhao SQ, Pi YS, et al. Highly efficient base editing in Staphylococcus aureus using an engineered CRISPR RNA-guided cytidine deaminase.Chem Sci, 2018, 9(12): 3248–3253.DOI: 10.1039/C8SC00637G |
| [36] | Chen WZ, Zhang Y, Zhang YF, et al. CRISPR/Cas9-based genome editing in Pseudomonas aeruginosa and cytidine deaminase-mediated base editing in Pseudomonas species.iScience, 2018, 6: 222–231.DOI: 10.1016/j.isci.2018.07.024 |
| [37] | Zheng K, Wang Y, Li N, et al. Highly efficient base editing in bacteria using a Cas9-cytidine deaminase fusion.Commun Biol, 2018, 1: 32.DOI: 10.1038/s42003-018-0035-5 |
| [38] | Wang Y, Liu Y, Liu J, et al. MACBETH: Multiplex automated Corynebacterium glutamicum base editing method.Metab Eng, 2018, 47: 200–210.DOI: 10.1016/j.ymben.2018.02.016 |
| [39] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities.Nature, 2015, 523(7561): 481–485.DOI: 10.1038/nature14592 |
| [40] | Hu JH, Miller SM, Geurts MH, et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity.Nature, 2018, 556(7699): 57–63.DOI: 10.1038/nature26155 |
| [41] | Ran FA, Cong L, Yan WX, et al. In vivo genome editing using Staphylococcus aureus Cas9.Nature, 2015, 520(7546): 186–191.DOI: 10.1038/nature14299 |
| [42] | Kleinstiver BP, Prew MS, Tsai SQ, et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition.Nat Biotechnol, 2015, 33(12): 1293–1298.DOI: 10.1038/nbt.3404 |
| [43] | Zetsche B, Gootenberg JS, Abudayyeh OO, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-cas system.Cell, 2015, 163(3): 759–771.DOI: 10.1016/j.cell.2015.09.038 |
| [44] | Tu MJ, Lin L, Cheng YL, et al. A 'new lease of life': FnCpf1 possesses DNA cleavage activity for genome editing in human cells.Nucleic Acids Res, 2017, 45(19): 11295–11304.DOI: 10.1093/nar/gkx783 |
| [45] | Gao LY, Cox DBT, Yan WX, et al. Engineered Cpf1 variants with altered PAM specificities.Nat Biotechnol, 2017, 35(8): 789–792.DOI: 10.1038/nbt.3900 |
| [46] | Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage.Nature, 2016, 533(7603): 420–424.DOI: 10.1038/nature17946 |
| [47] | Nishida K, Arazoe T, Yachie N, et al. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems.Science, 2016, 353(6305): aaf8729.DOI: 10.1126/science.aaf8729 |
| [48] | Ma YQ, Zhang JY, Yin WJ, et al. Targeted AID-mediated mutagenesis (TAM) enables efficient genomic diversification in mammalian cells.Nat Methods, 2016, 13(12): 1029–1035.DOI: 10.1038/nmeth.4027 |
| [49] | Hua K, Tao XP, Zhu JK. Expanding the base editing scope in rice by using Cas9 variants.Plant Biotechnol J, 2018.DOI: 10.1111/pbi.12993 |
| [50] | Gaudelli NM, Komor AC, Rees HA, et al. Programmable base editing of A·T to G·C in genomic DNA without DNA cleavage.Nature, 2017, 551(7681): 464–471.DOI: 10.1038/nature24644 |
| [51] | Hua K, Tao XP, Yuan FT, et al. Precise A·T to G·C base editing in the Rice genome.Mol Plant, 2018, 11(4): 627–630.DOI: 10.1016/j.molp.2018.02.007 |
| [52] | Yan F, Kuang YJ, Ren B, et al. Highly efficient A·T to G·C base editing by Cas9n-guided tRNA adenosine deaminase in Rice.Mol Plant, 2018, 11(4): 631–634.DOI: 10.1016/j.molp.2018.02.008 |
| [53] | Cheng AW, Wang HY, Yang H, et al. Multiplexed activation of endogenous genes by CRISPR-on, an RNA-guided transcriptional activator system.Cell Res, 2013, 23(10): 1163–1171.DOI: 10.1038/cr.2013.122 |
| [54] | Bikard D, Jiang WY, Samai P, et al. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system.Nucleic Acids Res, 2013, 41(15): 7429–7437.DOI: 10.1093/nar/gkt520 |
| [55] | Jinek M, East A, Cheng A, et al. RNA-programmed genome editing in human cells.eLife, 2013, 2: e00471.DOI: 10.7554/eLife.00471 |
| [56] | Qi LS, Larson MH, Gilbert LA, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.Cell, 2013, 152(5): 1173–1183.DOI: 10.1016/j.cell.2013.02.022 |
| [57] | Cleto S, Jensen JVK, Wendisch VF, et al. Corynebacterium glutamicum metabolic engineering with CRISPR interference (CRISPRi).Acs Synth Biol, 2016, 5(5): 375–385.DOI: 10.1021/acssynbio.5b00216 |
| [58] | Rath D, Amlinger L, Hoekzema M, et al. Efficient programmable gene silencing by Cascade.Nucleic Acids Res, 2015, 43(1): 237–246.DOI: 10.1093/nar/gku1257 |
| [59] | Choudhary E, Thakur P, Pareek M, et al. Gene silencing by CRISPR interference in mycobacteria.Nat Commun, 2015, 6: 6267.DOI: 10.1038/ncomms7267 |
| [60] | Singh AK, Carette X, Potluri LP, et al. Investigating essential gene function in Mycobacterium tuberculosis using an efficient CRISPR interference system.Nucleic Acids Res, 2016, 44(18): e143.DOI: 10.1093/nar/gkw625 |
| [61] | Vigouroux A, Oldewurtel E, Cui L, et al. Tuning dCas9's ability to block transcription enables robust, noiseless knockdown of bacterial genes.Mol Syst Biol, 2018, 14(3): e7899.DOI: 10.15252/msb.20177899 |
| [62] | Perez-Pinera P, Kocak DD, Vockley CM, et al. RNA-guided gene activation by CRISPR-Cas9-based transcription factors.Nat Methods, 2013, 10(10): 973–976.DOI: 10.1038/nmeth.2600 |
| [63] | Maeder ML, Linder SJ, Cascio VM, et al. CRISPR RNA-guided activation of endogenous human genes.Nat Methods, 2013, 10(10): 977–979.DOI: 10.1038/nmeth.2598 |
| [64] | Sander JD, Joung JK. CRISPR-Cas systems for editing, regulating and targeting genomes.Nat Biotechnol, 2014, 32(4): 347–355.DOI: 10.1038/nbt.2842 |
| [65] | Chakraborty S, Ji H, Kabadi AM, et al. A CRISPR/ Cas9-based system for reprogramming cell lineage specification.Stem Cell Rep, 2014, 3(6): 940–947.DOI: 10.1016/j.stemcr.2014.09.013 |
| [66] | Gilbert LA, Larson MH, Morsut L, et al. CRISPR-mediated modular RNA-Guided regulation of transcription in eukaryotes.Cell, 2013, 154(2): 442–451.DOI: 10.1016/j.cell.2013.06.044 |
| [67] | Dong C, Fontana J, Patel A, et al. Synthetic CRISPR-Cas gene activators for transcriptional reprogramming in bacteria.Nat Commun, 2018, 9(1): 2489. |
| [68] | Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering.Cell, 2014, 157(6): 1262–1278.DOI: 10.1016/j.cell.2014.05.010 |
| [69] | Pattanayak V, Lin S, Guilinger JP, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity.Nat Biotechnol, 2013, 31(9): 839–843.DOI: 10.1038/nbt.2673 |
| [70] | Haeussler M, Concordet JP. Genome editing with CRISPR-Cas9: can it get any better?.J Genet Genom, 2016, 43(5): 239–250.DOI: 10.1016/j.jgg.2016.04.008 |
| [71] | Ungerer J, Pakrasi HB. Cpf1 is a versatile tool for CRISPR genome editing across diverse species of Cyanobacteria.Sci Rep, 2016, 6: 39681.DOI: 10.1038/srep39681 |
