
 , 李玉花
, 李玉花

东北林业大学 生命科学学院,黑龙江 哈尔滨 150040
收稿日期:2018-07-17;接收日期:2018-08-29;网络出版时间:2018-10-18
基金项目:中央高校基本科研业务费专项(No.2572016BA06), 黑龙江省应用技术研究与开发计划(No.GY2016ZB0097), 国家林业局“948”项目(No.2014-4-60)资助
摘要:代谢工程作为通过引入外源合成途径或改造优化代谢网络,进行高附加值的天然代谢产物生物合成的技术,已经得到广泛应用。但随着目标合成产物的结构日渐复杂,构建多基因的从头合成途径造成宿主生物代谢失衡与中间产物对宿主细胞产生毒害作用等一系列问题发生的可能性也随之增加。为解决这些问题合成支架策略应运而生,合成支架将途径酶共定位以提高局部酶和代谢物的浓度,来增强代谢通量并限制中间产物与宿主细胞环境间的相互作用,成为生物催化和合成生物学研究的热点之一。尽管由核酸、蛋白质构成的合成支架策略已经应用于多种代谢物的异源合成,并取得了不同程度的成功,但合成支架的精确组装仍然是一项艰巨的任务。文中详细介绍了合成支架技术的研究现状,详细阐述了合成支架技术的原理和实例,并初步探讨了其应用前景。
关键词:代谢工程合成支架途径酶共定位生物催化合成生物学
Research progress on synthetic scaffold in metabolic engineering – a review
Xue Yin, Chen Liang, Yue Feng, He Zhang, Yu Wang

 , Yuhua Li
, Yuhua Li

College of Life Sciences, Northeast Forestry University, Harbin 150040, Heilongjiang, China
Received: July 17, 2018; Accepted: August 29, 2018; Published: October 18, 2018
Supported by: The Fundamental Research Funds for the Central Universities (No. 2572016BA06), Heilongjiang Province Applied Technology Research and Development Plan (No. GY2016ZB0097), the State Bureau of Forestry 948 Project (No. 2014-4-60)
Corresponding author: Yu Wang. Tel: +86-451-82191783; E-mail: wangyu@nefu.edu.cn;
Yuhua Li. Tel/Fax: +86-451-82191733; E-mail: lyhshen@126.com.
Abstract: Metabolic engineering is a powerful tool to increase many valuable metabolites through enhancing endogenous pathways or introducing exogenous pathways from other organisms. As the complexity of the targeted structure increases, many problems arise when the host suffers from flux imbalance and some toxic effects. An emerging approach to solve these problems is the use of synthetic scaffolds to co-localize key enzymes and metabolites of the synthetic pathways, enhance the metabolic flux and limit the interaction between intermediate products in the host cell. Although many scaffolds made of proteins and nucleic acids have been explored and applied to a variety of research to the heterogeneous synthesis of multiple metabolites, success is rather limited. The precise assembly of synthetic scaffolds remains a difficult task. In this review, we summarized the application of synthetic scaffolds in metabolic engineering, and outlined the main principle of scaffold designs, then highlighted the current challenges in their application.
Keywords: metabolic engineeringsynthetic scaffoldpathway enzyme co-localizationbiocatalysissynthetic biology
近年来,以环境友好型方式实现从廉价可再生的简单化合物催化合成高附加值的复杂化合物的代谢工程迅猛发展,在未来有望成为合成化学的替代品[1-5]。然而,与生物体内的天然合成途径相比,外源性合成途径并不适用于合成各种各样的目标产物。在宿主自身代谢环境中,目标产物的过度生产对其自身生存是很少有利或必需的[6]。随着目标合成产物的结构日渐复杂,在构建多基因的从头合成途径过程中,一些问题也随之而来,例如,途径代谢物被内源性反应转移或通过分泌而丧失,中间产物的快速扩散和降解或对宿主自身有毒害作用,底物利用率低,宿主代谢失衡影响自身生长活力等,最终导致目标产物产量降低[6]。为解决这些问题,研究人员模仿自然代谢控制机制,将途径酶在空间上组织形成多酶复合体,通过将途径酶共定位来提高途径酶和代谢物的局部浓度,形成底物通道,从而提高途径酶转化效率,减少中间产物的积累,降低与宿主生物中其他组分的交叉反应等[7]。
1985年Bulow等受到天然多功能蛋白的启发,第一次尝试将大肠杆菌Escherichia coli的β-半乳糖苷酶的3′端与半乳糖激酶的5′端融合表达,成功构建了一个能催化由2个单独酶催化完成的2个顺序反应的双功能酶[8]。之后,该研究团队又将来自荧光假单胞菌Pseudomonas fluorescens的β-半乳糖激酶和半乳糖脱氢酶融合表达,将乳糖水解为半乳糖后,再将半乳糖氧化为乳酸,并且融合后的双功能酶与游离酶相比,反应效率提高了1.5–2.4倍[9]。该策略通过基因融合的方法将酶在空间上拉近,产生底物通道作用,实现催化效率的提高,已经广泛地应用到其他研究中[10-13]。尽管多酶融合表达是一种极简单且高效的多酶组装策略,但在实际应用中仍然存在一些不确定因素和限制,例如,蛋白质的错误折叠或形成包涵体表达,导致酶催化活力降低甚至丧失等[14-16]。而且该策略不易应用于两个反应以上的代谢途径,并且由于融合蛋白的固定比例表达而不能平衡途径酶的化学计量数。为了能使途径酶化学计量达到平衡状态,减少中间产物的积累,提高酶催化效率,合成生物学家采用更加模块化的方法,使用合成生物支架的策略使途径酶在空间上共定位,顺序催化代谢反应[17]。与融合蛋白策略不同的是,合成支架是利用不同相互作用结构域或物理螯合的方式使途径酶共定位的一个非催化亚单位,通过控制合成支架中结合结构域的位置和比例对途径酶的化学计量数进行优化平衡,能够灵活地应用于多个酶催化的代谢途径[18]。
合成支架具有调节途径酶达到最优催化效率的巨大潜在优势,推动了近些年生物学家对合成支架的开发利用。合成支架多是截取从自然界发现的具有相互作用的生物元件构建而成,特别是基于蛋白质的支架[19]。而近年来,随着核酸纳米技术的发展,研究人员除了蛋白支架外,还开发出利用核酸作为合成支架的策略[20-24]。本文基于合成支架的最新研究进展,对其在合成生物学中解决代谢通量平衡发挥重要功能的原理进行详细阐述,并对其应用前景作了初步探讨。
1 蛋白支架在生物进化过程中,存在许多利用酶复合物提高代谢途径通量的自然实例。色氨酸合酶就是其中具有代表性的例子,其由线性排列的4个亚基αββα组成,通过形成分子内隧道,使中间产物直接从一个酶亚基的活性中心传递到下一个酶亚基的活性中心,减少中间产物的扩散或被细胞内其他组分降解,从而提高催化反应速率,类似的酶还有氨甲酰磷酸合酶和谷氨酰胺磷酸核糖基焦磷酸酰胺基转移酶[25-28]。而另一个典型的例子就是厌氧纤维素分解菌的天然纤维小体,由纤维素酶、半纤维素酶等水解酶以及蛋白支架构成[29-30]。其中,蛋白支架是由包含重复序列的粘连蛋白(Cohesion)组成的一个非催化亚单位,含有重复序列的锚定蛋白(Dockerin)与酶相连,通过cohesin- dockerin的相互作用将纤维素酶、半纤维素酶等水解酶固定实现多酶复合体的组装,使多种水解酶与底物紧密接触,提高底物局部浓度并确保酶的正确化学计量和顺序,从而最大限度发挥酶的协同作用。因此,由蛋白支架组装的多酶复合体的催化效率比游离的可溶性酶的催化效率高。
受到这种天然蛋白支架的启发,研究人员构建人工合成的蛋白支架来提高酶的催化效率[31-33]。蛋白支架是截取天然蛋白质具有相互作用的结构域(即受体结构域)融合表达构建而成,通过将相应蛋白配体与途径酶融合表达,利用蛋白质-蛋白质相互作用将途径酶固定在蛋白支架上,然后通过调节受体结构域的比例和顺序,平衡相关途径酶的化学计量数,从而实现代谢途径通量的增强。Tsai等利用特异的cohesin-dockerin相互作用在酿酒酵母Saccharomyces cerevisiae细胞表面组装外切葡聚糖酶、内切葡聚糖酶和β-葡萄糖苷酶的多酶级联反应,实现了纤维素水解与乙醇生产相结合,乙醇的产量相对于通过游离酶混合物催化方式高出2.6倍以上[31] (图 1)。类似的,在甲醇氧化脱氢生成二氧化碳的反应过程中,利用蛋白支架将反应过程中的3种NAD+依赖的脱氢酶组装在酵母细胞表面,催化多酶级联反应,形成底物通道,使NADH的产率提高了5倍[33]。
 |
| 图 1 酵母细胞表面组装的功能化纤维小体[31] Fig. 1 Functional assembly of minicellulosomes on the yeast cell surface[31]. |
| 图选项 |
在硫磺矿硫化叶菌Sulfolobus solfataricus中发现的环状的异源三聚体的细胞增殖核抗原(Proliferating cell nuclear antigen,PCNA)也被用作体外多酶组装的蛋白支架,将假单孢氧还蛋白还原酶(Putidaredoxin reductases,PdR)、假单孢氧还蛋白(Putidaredoxin,PdX)和细胞色素P450 (Cytochrome P450,P450cam)的N末端分别与PCNA的3个亚基的C末端融合,得到融合蛋白PCNA1-PdR、PCNA2-PdX和PCNA3-P450cam,在E. coli中表达纯化后,利用PCNA 3个亚基间的相互作用通过体外混合的方式实现3个酶的组装,产生更为有效的电子传递系统[34]。随后,又通过结构信息分析在PCNA亚基间特异地引入二硫键,使蛋白支架间的结合更为稳定,相比于非共价键结合形成多酶复合物的方式使得P450cam反应效率增强[35]。
除了上述用于体外反应途径的蛋白支架之外,体内增强代谢通量应用最多的人工蛋白支架是Dueber等截取后生动物信号蛋白相互作用域融合表达构建而成的蛋白支架(GBD)x-(SH3)y-(PDZ)z[36] (图 2A)。他们将甲羟戊酸途径中的乙酰乙酰-辅酶A硫解酶(Acetoacetyl-CoA thiolase,AACT)、羟甲基戊二酸-辅酶A合成酶(Hydroxy-methylglutaryl-CoA synthase,HMGS)、羟基-甲基戊二酰-辅酶A还原酶(Hydroxymethylglutaryl-CoA reductase,HMGR)分别与3种多肽GBD、SH3、PDZ的配体融合,然后将这3种融合蛋白与其可以特异性结合的蛋白支架在E. coli中共同表达。使用阻遏子平衡蛋白支架和酶的表达水平,通过调节蛋白支架的比例和顺序对酶的化学计量数进行优化,最终当蛋白支架比例x:y:z为1:2:2时,甲羟戊酸的产量与无支架的酶催化相比提高77倍。该课题组还将此蛋白支架应用于葡萄糖到葡萄糖酸的反应途径中,且当蛋白支架比例x:y:z为1:4:4时,葡萄糖酸的产量提高5倍[37]。随后,Agapakis等利用该支架使产氢气反应速率提高3倍[38],Baek等[39]利用该支架共定位丁酸合成途径中的酶,使丁酸产量提高3倍。并且,蛋白支架(GBD)x-(SH3)y-(PDZ)z还被成功地应用于白藜芦醇在真核生物S. cerevisiae的生物合成途径中,通过对途径酶进行组装,最终白藜芦醇的产量与组装前相比提高5倍,与融合蛋白策略相比提高2.7倍[40]。除此之外,研究人员还将单独的支架蛋白及其配体分别与途径酶融合表达,催化组装2个酶的级联反应,达到了提高目标产物产量的目的[41-44]。尽管蛋白支架提高途径代谢通量的确切机制尚不清楚,但据推测,由于酶的寡聚作用,使其可能形成大型复合物,类似于天然存在的代谢物微区,复合物中生成的中间体在扩散之前就很快被复合物中的酶所消耗[17] (图 2B)。蛋白支架(GBD)x-(SH3)y-(PDZ)z的实际应用不仅证明了其普遍适用性,还显示出不同合成途径优化的最佳支架结构比例会有所不同,这对如何合理设计具有预期功能的蛋白支架提出挑战。
 |
| 图 2 蛋白支架(GBD)x-(SH3)y-(PDZ)z介导多酶组装示意图[17, 36] Fig. 2 Schematic diagram of protein scaffold (GBD)x-(SH3)y-(PDZ)z-mediated multi-enzyme assembly[17, 36]. (A) The synthetic scaffolds were constructed with three modular protein-protein interaction domains (GBD, SH3, and PDZ), which recruit three mevalonate biosynthetic enzymes (AACT, HMGS, and HMGR) C-terminally tagged with peptide ligands specific for these interaction domains[36]. Local concentrations of intermediate and enzymes are elevated to improve reaction fluxes and balance kinetic parameters, in part by changing the number of repeats of these interaction domains (x, y, z) [36]. (B) Enzymes with oligomeric structures could potentially bind multiple scaffolds, resulting in enzyme-scaffold complexes[17]. |
| 图选项 |
亲和体affibody分子由金黄色葡萄球菌Staphylococcus aureus蛋白A的免疫球蛋白结合区域衍化而来,含有58个氨基酸残基,其相对分子质量小、折叠速度快、特异性亲和力很高,是一种类似于抗体的亲和配体[45]。Tippmann等利用affibody分子来标记途径酶,抗独特型affibody作为基础来设计蛋白支架,利用affibody-抗独特型与affibody间高特异性亲和力结合使途径酶共定位,提高反应速率[46]。在S. cerevisiae体内利用该蛋白支架体系共定位法尼基二磷酸合酶和法尼烯合酶,使法尼烯产量提高135%。通过对affibody分子的受体结合部位进行随机重组构建噬菌体展示文库,可以产生许多不同的高亲和力的affibody分子,其能够与相应的蛋白质分子(即抗独特型affibody)特异性结合。因此,由affibody分子和抗独特型affibody为基础构建的蛋白支架有望应用于涉及更多酶促级联反应的代谢途径。
应用蛋白支架提高代谢通量,可以从以下几个方面入手:1)在支架配体与途径酶融合表达时,二者之间需引入一段接头序列(Linker,即连接肽),多选用柔性Linker (如(GerSer)n),降低支架配体与途径酶之间的空间位阻,更有利于融合蛋白各个结构域的正确折叠。2)考虑到支架配体与途径酶融合表达后,是否会对途径酶的表达及活性造成影响,可以对其酶动力参数进行鉴定。3)在构建含有多个重复的配体结合结构的蛋白支架时,可以利用含有相同粘性末端的限制性内切酶进行设计连接[47]。4)将蛋白支架应用于体内代谢途径时,除了对蛋白支架比例和位置进行优化,还可以采用诱导型启动子或阻遏子对途径酶及蛋白支架的表达量进行控制,从而减轻宿主的代谢负担[36, 40]。
2 核酸基支架尽管合成蛋白支架在介导多酶级联反应、改善代谢通量等方面的研究取得了一定进展,但随着目标代谢产物的结构越来越复杂,途径酶的种类及分子量的增加,导致含有多个重复的配体结合结构域的蛋白支架可能会难以表达。而近年来,随着核酸纳米技术的发展,核酸分子作为遗传信息的载体,具有以下优势:1)具有碱基互补配对原则,并且有了一定深入的研究[48]。2)通过杂交(DNA-DNA/DNA-RNA/RNA-RNA)或锌指蛋白(Zinc-finger protein,ZFP) DNA结合结构域或类转录激活因子(Transcription activator-like effectors,TALEs)的DNA结合结构域,可以提供DNA/RNA-蛋白质的相互作用。3)核酸分子短链可折叠成各种结构或组装成二聚体或多聚体构件[49],从而可以合成各种具有特定的可编程的三维空间结构[50-52]。4)在纳米级精度下核酸的二级和三级结构可以用计算机模拟[53-55],而一些计算机工具的开发也促进了核酸纳米结构的合理设计[56-58]。所以,研究人员开发了基于核酸分子构建合成支架,对途径酶进行组装的新技术[59-60]。
2.1 DNA支架DNA支架主要分为两种类型,其一是用含有与DNA支架互补的特异核苷酸序列对途径酶进行化学修饰,然后利用碱基互补配对原则使途径酶结合到DNA支架上,以提高酶促反应速率,但这种类型的DNA支架只能应用于体外反应途径;其二是将途径酶与含有DNA结合结构域的蛋白进行融合表达,通过DNA结合结构域将途径酶固定在DNA支架上,实现途径酶的共定位,其主要应用于体内代谢途径。目前,已经有许多成功的范例将DNA支架应用于多酶体系的组装。
2009年,Wilner等利用滚环扩增技术(Rolling circle amplification,RCA)合成具有特异序列的单链DNA,并且用含有与特异序列互补的DNA寡核苷酸以共价连接的方式分别对葡萄糖氧化酶(Glucose oxidase,GOx)和辣根过氧化物酶(Horseradish peroxidase,HRP)进行化学修饰,体外利用碱基互补配对使GOx和HRP结合到单链DNA支架上,形成双酶复合物激活级联反应,而在没有DNA支架存在的情况下则不能激活双酶级联反应[61] (图 3A)。这证明了DNA作为分子支架将酶有序组装的可行性。然而,尽管此方法用于多酶组装是可行的,但是单链DNA缺乏刚性可能导致支架的折叠,为了增加一维DNA支架的刚性,该研究团队还设计了二维DNA支架。预先设计一组具有部分互补序列的单链DNA形成六边形结构,其具有10 bp突出的特异DNA寡核苷酸序列,可以与带有特异序列互补的DNA寡核苷酸序列修饰的GOx和HRP通过碱基互补配对,附着到两个不同的六边形上[62]。当酶间距由4个六边形(~33 nm)缩短为2个六边形(~13 nm)时,观察到产物的量有显著增加,研究表明这种酶催化效率的提高不仅是由于酶间距缩短使局部中间产物浓度升高而实现,还是由于中间产物在酶表面扩散受限而导致,而二维-DNA折纸结构也观察到了相似的结果[22, 62] (图 3B)。随后,Fu等利用可编程的DNA纳米结构构建了具有NAD+辅酶摆动臂的DNA双交联拼贴支架,体外介导了6-磷酸葡萄糖脱氢酶(Glucose-6-phosphate dehydrogenase,G6pDH)和苹果酸脱氢酶(Malic dehydrogenase,MDH)的两步脱氢级联反应[21] (图 4)。通过利用DNA纳米结构的可编程性,可以对酶间距、位置及化学计量数进行调节,来获得最优结果[63]。还有其他的大的超分子DNA支架和DNA折纸同样可以用来介导多酶级联反应,来加快其反应速率[64-67]。但在体外将特异的寡聚核苷酸序列与途径酶的赖氨酸残基共价连接的化学修饰技术,成本较高并且会对酶的活性造成损坏,降低了这些策略的可行性与应用的普遍性。
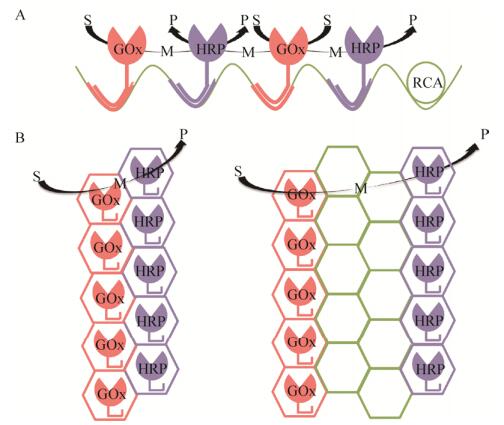 |
| 图 3 多维DNA支架结构示意图[61-62] Fig. 3 Schematic diagram of multi-dimensional DNA scaffolds[61-62]. (A) Linear one-dimensional single-stranded DNA scaffold synthesized using the rolling circle amplification (RCA). The two enzymes (GOx and HRP) were chemically conjugated with oligonucleotides to bind onto specific sites on the linear DNA scaffold[61]. (B) Assembly of the GOx and HRP enzymes on the two-hexagon and four-hexagon two-dimensional DNA scaffolds through strands with partial complimentarity[62]. |
| 图选项 |
 |
| 图 4 辅酶摆动臂-DNA双交联拼贴支架结构示意图[21] Fig. 4 Schematic diagram of coenzyme swinging arm/DNA double-crossover tile scaffold[21]. The NAD+-modified swinging arm is positioned halfway between two the dehydrogenases (G6pDH and MDH), facilitating the transfer of hydrides. |
| 图选项 |
而如何将DNA支架的体外模型应用于体内环境又对研究人员提出了一项新的挑战。对于体内应用来说,任何化学修饰的使用都不切实际。为避免化学修饰技术的使用,研究人员开始利用核酸结合蛋白与核苷酸序列特异性结合的特征实现对途径酶的组装。锌指蛋白(ZFP)具有特异性识别核苷酸序列并与之特异结合的能力[68],Conrado等将ZFP与质粒DNA支架结合使用,实现了在E. coli体内的合成代谢途径的增强[69]。通过将相应ZFP与途径酶基因融合,然后在ZFP的DNA结合结构域与质粒DNA支架相应锌指结合位点的驱动下,途径酶就会结合到DNA支架的相应位置上,从而介导多酶级联反应,实现合成代谢途径的增强。该支架的优势在于可以通过改变锌指结合位点的间距、数量和顺序对途径酶的组装进行优化。质粒DNA支架介导的合成代谢途径具有更多的代谢物生成与普遍性应用,使白藜芦醇、1, 2-丙二醇、甲羟戊酸的产量分别提高5倍、4.6倍、2.5倍[24] (图 5),L-苏氨酸的生产时间缩短50%以上[69]。除此之外,ZFP与DNA支架的结合使用还可以介导纤维素水解转化为其他化合物的反应[70-71]。质粒DNA支架具有可以容纳多个互作基序和可变间距、无需考虑溶解性等问题的优势,但却需要多个锌指结构域(约90–120个氨基酸残基)对途径酶进行修饰,并且细胞内DNA支架的最大浓度受到最大质粒拷贝数的限制。
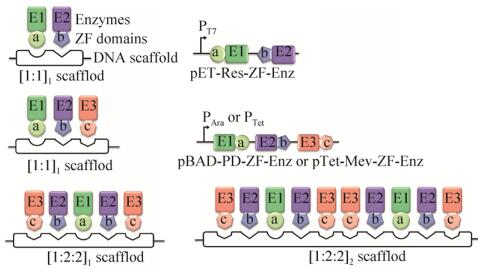 |
| 图 5 锌指蛋白-质粒DNA支架介导的大肠杆菌代谢途径中的酶组装[24] Fig. 5 Zinc finger protein-plasmid DNA scaffold assisted assembly of metabolic pathways in E. coli[24]. Schematic of different scaffold arrangements used for the two- or three-enzyme pathways producing resveratrol, 1, 2-propanediol or mevalonate. E1, E2 and E3 are the biosynthetic pathway enzymes, a, b and c are Zinc finger domains, respectively. Scaffolds are plasmids with different zinc finger binding sites. |
| 图选项 |
与ZFP相比,以类转录激活因子(TALEs)为基础的DNA结合模块具有更高的序列特异性,较低的非靶位点结合能力和细胞毒性,并且设计操作更简便。Zhu等利用TALE构建了人工TALE-DNA支架,体内将途径酶与TALE基因融合,通过TALE的DNA结合结构域与DNA支架特异性结合,形成多酶复合体,用于增强E. coli中异源合成吲哚乙酸(Indole-3-acetic acid,IAA)途径[72]。TALE是植物致病菌黄单胞菌Xanthomonas的一类天然细菌效应因子,与原核细胞具有实质相容性,因此该支架更适用于整合到原核生物体内。此外,TALE-DNA支架相较于其他支架体系还具有更高的稳定性,不易被降解或与细胞内其他组分发生相互作用。
2.2 RNA支架非编码RNA (Non-coding RNA,ncRNA)也同样可以作为构建合成支架的材料,其可以在细胞内大量表达,且在细胞核外稳定存在。但由于具有高度特异性结合亲和力的RNA结合结构域较少,所以RNA支架很难采用与DNA支架相同的构建策略。因此,研究人员选用RNA适配体作为途径酶的结合位点,利用构建顺序对称RNA元件的策略在细胞内建立RNA的等温线组装方法,构建了基于具有二聚化和聚合结构域的RNA的离散型、一维和二维RNA支架,并已经用于体内经由[FeFe]-氢化酶和铁氧还蛋白催化的产氢途径的优化[23] (图 6)。通过将与RNA适配体靶蛋白融合的[FeFe]-氢化酶和铁氧还蛋白招募到具有RNA适配体的离散型、一维和二维RNA支架上,使E. coli内产氢量比无支架菌株有不同程度的增加,分别为4倍、11倍和48倍。这一结果显示,随着RNA支架维度的增加,其级联反应速率加快所产生的产物浓度呈指数级增加。
 |
| 图 6 RNA支架介导的途径酶的组装[23] Fig. 6 RNA scaffold-mediated pathway enzyme assembly[23]. Pathway enzyme A and B scaffolded onto discrete, 1D, and 2D RNA assemblies. |
| 图选项 |
同样的RNA支架策略也被用来优化在E. coli中异源表达酰基-ACP还原酶(Acyl-ACP reductase,AAR)和醛去甲基化加氧酶(Aldehyde deformylating oxygenase,ADO)生产十五烷的代谢途径[73]。与无支架菌株相比,二维RNA支架使十五烷产量提高2.4倍。研究人员发现,RNA适配体茎环长度的改变会引起AAR与ADO活性位点空间方向的变化,从而影响代谢途径的产量,说明了利用RNA支架使途径酶共定位时,酶活性中心的相对空间取向也十分重要。迄今为止,使用体内RNA支架策略优化了更为复杂的4个酶催化的琥珀酸代谢途径,使其代谢通量提高了88%,也进一步说明了该支架的实用性。该支架相较于DNA支架来说,不仅可以优化酶的化学计量数,控制途径酶的间距和顺序,还可以控制酶的空间方向[20]。而作为一种特异性识别分子,RNA适配体具有稳定性高、靶目标广泛等优点,由此构建的RNA支架可以扩展到更复杂的代谢途径中使用。
3 总结与展望在代谢工程领域,合成代谢途径本质上是多种来源的具有不同催化活性的酶整合在一起,获得能高产目标产物的工程菌株。为此,研究人员采用多种优化策略,如密码子优化[74]、启动子工程[75]、核糖体结合位点优化[76]等,对代谢途径中途径酶的表达水平进行优化,并取得了一定的成功。合成支架策略作为补充或平行的方法,提供了模块化且高度灵活的组装工具,已经在代谢途径中广泛应用,通过翻译后共定位途径酶来调节其化学计量数、距离和空间取向,达到改善代谢通量的目的。表 1总结了近年来在合成支架研究领域的主要研究进展。
表 1 不同类型合成支架的总结Table 1 A summary of the different synthetic scaffolds
| Strategy | Application | Building material | in vivo/in vitro | References |
| Fusion enzymes | Synthesis | Protein | in vivo/in vitro | [9-13] |
| Cohesin-dockerin scaffold | Synthesis | Protein | in vitro | [29-33] |
| PCNA | Synthesis | Protein | in vitro | [34-35] |
| (GBD)x-(SH3)y-(PDZ)z scaffold | Synthesis | Protein | in vivo | [36-40] |
| PDZ: PDZlig | Synthesis | Protein | in vivo/in vitro | [41-43] |
| SH3: SH3lig | Synthesis | Protein | in vivo | [44] |
| Affibody-Anti-idiotype affibody | Synthesis | Protein | in vivo | [46] |
| Single-stranded DNA scaffold | Synthesis | DNA oligonucleotides | in vitro | [61] |
| Hexagonal DNA scaffold | Synthesis | DNA oligonucleotides | in vitro | [62] |
| 2D DNA origami | Synthesis | DNA oligonucleotides | in vitro | [22] |
| Coenzyme swinging arm/DNA double-crossover tile scaffold | Synthesis | DNA oligonucleotides | in vitro | [21] |
| DNA origami scaffold | Protein/Enzyme assembly | DNA oligonucleotides | in vitro | [64] |
| Halo-tag conjugation of nucleotides to proteins | Synthesis | Modified oligonucleotides | in vitro | [65-66] |
| Zinc finger proteins/plasmid DNA scaffolds | Synthesis | dsDNA | in vivo/in vitro | [24, 70-71] |
| Transcription activator-like effectors/dsDNA scaffolds | Synthesis | dsDNA | in vivo | [74] |
| RNA scaffolding with aptamers | Synthesis | RNA oligonucleotides | in vivo | [20, 23, 73] |
表选项
虽然,合成支架策略在实际应用过程中取得了一定进展,但仍存在一些挑战需要不断地探索前进,例如如何在确保酶活性不受影响的情况下精确地控制途径酶的间距和排列,如何挖掘具有更高特异性的合成支架相关的生物学元件,以及如何实现该技术在工业上的广泛应用等。为克服这些挑战,就必须更精确地了解合成支架的作用机制。合理设计具有组装多酶功能的新型合成支架是一个复杂且涉及多学科的课题,其中包括生物物理、生物化学、计算生物学以及蛋白质工程和核酸纳米技术等。相信随着合成生物学理论和技术的快速发展,在不久的将来会开发出更高效的工程工具,用来优化天然产物合成代谢体系,从而实现复杂天然产物的工程化生产。
参考文献
| [1] | Zhou YJ, Buijs NA, Zhu ZW, et al. Production of fatty acid-derived oleochemicals and biofuels by synthetic yeast cell factories.Nat Commun, 2016, 7: 11709.DOI: 10.1038/ncomms11709 |
| [2] | Zhuang Y, Yang GY, Chen XH, et al. Biosynthesis of plant-derived ginsenoside Rh2 in yeast via repurposing a key promiscuous microbial enzyme.Metab Eng, 2017, 42: 25–32.DOI: 10.1016/j.ymben.2017.04.009 |
| [3] | Yan X, Fan Y, Wei W, et al. Production of bioactive ginsenoside compound K in metabolically engineered yeast.Cell Res, 2014, 24(6): 770–773.DOI: 10.1038/cr.2014.28 |
| [4] | Galanie S, Thodey K, Trenchard IJ, et al. Complete biosynthesis of opioids in yeast.Science, 2015, 349(6252): 1095–1100.DOI: 10.1126/science.aac9373 |
| [5] | Li YR, Li SJ, Thodey K, et al. Complete biosynthesis of noscapine and halogenated alkaloids in yeast.Proc Natl Acad Sci USA, 2018: 201721469.DOI: 10.1073/pnas.1721469115 |
| [6] | Nicolaou SA, Gaida SM, Papoutsakis ET. A comparative view of metabolite and substrate stress and tolerance in microbial bioprocessing: from biofuels and chemicals, to biocatalysis and bioremediation.Metab Eng, 2010, 12(4): 307–331.DOI: 10.1016/j.ymben.2010.03.004 |
| [7] | Conrado RJ, Varner JD, Delisa MP. Engineering the spatial organization of metabolic enzymes: mimicking nature's synergy.Curr Opin Biotechnol, 2008, 19(5): 492–499.DOI: 10.1016/j.copbio.2008.07.006 |
| [8] | Bülow L, Ljungcrantz P, Mosbach K. Preparation of a soluble bifunctional enzyme by gene fusion.Nat Biotechnol, 1985, 3(9): 821–823.DOI: 10.1038/nbt0985-821 |
| [9] | Ljungcrantz P, Carlsson H, Mansson MO, et al. Construction of an artificial bifunctional enzyme, beta-galactosidase/galactose dehydrogenase, exhibiting efficient galactose channeling.Biochemistry, 1989, 28(22): 8786–8792.DOI: 10.1021/bi00448a016 |
| [10] | Orita I, Sakamoto N, Kato N, et al. Bifunctional enzyme fusion of 3-hexulose-6-phosphate synthase and 6-phospho-3-hexuloisomerase.Appl Microbiol Biotechnol, 2007, 76(2): 439–445. |
| [11] | Zhao FL, Bai P, Liu T, et al. Optimization of a cytochrome P450 oxidation system for enhancing protopanaxadiol production in Saccharomyces cerevisiae.Biotechnol Bioeng, 2016, 113(8): 1787–1795.DOI: 10.1002/bit.v113.8 |
| [12] | Bakkes PJ, Riehm JL, Sagadin T, et al. Engineering of versatile redox partner fusions that support monooxygenase activity of functionally diverse cytochrome P450s.Sci Rep, 2017, 7(1): 9570. |
| [13] | Han JY, Song JM, Seo SH, et al. Ty1-fused protein-body formation for spatial organization of metabolic pathways in Saccharomyces cerevisiae.Biotechnol Bioeng, 2018, 115(3): 694–704.DOI: 10.1002/bit.v115.3 |
| [14] | Chang HC, Kaiser CM, Hartl FU, et al. De novo folding of GFP fusion proteins: high efficiency in eukaryotes but not in bacteria.J Mol Biol, 2005, 353(2): 397–409.DOI: 10.1016/j.jmb.2005.08.052 |
| [15] | Netzer WJ, Hartl FU. Recombination of protein domains facilitated by co-translational folding in eukaryotes.Nature, 1997, 388(6640): 343–349.DOI: 10.1038/41024 |
| [16] | Christensen T, Amiram M, Dagher S, et al. Fusion order controls expression level and activity of elastin-like polypeptide fusion proteins.Protein Sci, 2009, 18(7): 1377–1387.DOI: 10.1002/pro.157 |
| [17] | Lee H, Deloache WC, Dueber JE. Spatial organization of enzymes for metabolic engineering.Metab Eng, 2012, 14(3): 242–251.DOI: 10.1016/j.ymben.2011.09.003 |
| [18] | Siu KH, Chen RP, Sun Q, et al. Synthetic scaffolds for pathway enhancement.Curr Opin Biotechnol, 2015, 36: 98–106.DOI: 10.1016/j.copbio.2015.08.009 |
| [19] | Whitaker WR, Dueber JE. Metabolic pathway flux enhancement by synthetic protein scaffolding.Methods Enzymol, 2011, 497: 447–468.DOI: 10.1016/B978-0-12-385075-1.00019-6 |
| [20] | Delebecque CJ, Silver PA, Lindner AB. Designing and using RNA scaffolds to assemble proteins in vivo.Nat Protoc, 2012, 7(10): 1797–1807.DOI: 10.1038/nprot.2012.102 |
| [21] | Fu JL, Yang YR, Johnson-Buck A, et al. Multi-enzyme complexes on DNA scaffolds capable of substrate channelling with an artificial swinging arm.Nat Nanotechnol, 2014, 9(7): 531–536.DOI: 10.1038/nnano.2014.100 |
| [22] | Fu JL, Liu MH, Liu Y, et al. Interenzyme substrate diffusion for an enzyme cascade organized on spatially addressable DNA nanostructures.J Am Chem Soc, 2012, 134(12): 5516–5519.DOI: 10.1021/ja300897h |
| [23] | Delebecque CJ, Lindner AB, Silver PA, et al. Organization of intracellular reactions with rationally designed RNA assemblies.Science, 2011, 333(6041): 470–474.DOI: 10.1126/science.1206938 |
| [24] | Conrado RJ, Wu GC, Boock JT, et al. DNA-guided assembly of biosynthetic pathways promotes improved catalytic efficiency.Nucleic Acids Res, 2012, 40(4): 1879–1889.DOI: 10.1093/nar/gkr888 |
| [25] | Hyde CC, Ahmed SA, Padlan EA, et al. Three-dimensional structure of the tryptophan synthase α2β2 multienzyme complex from Salmonella typhimurium.J Biol Chem, 1988, 263(33): 17857–17871. |
| [26] | Thoden JB, Holden HM, Wesenberg G, et al. Structure of carbamoyl phosphate synthetase: a journey of 96 ? from substrate to product.Biochemistry, 1997, 36(21): 6305–6316.DOI: 10.1021/bi970503q |
| [27] | Krahn JM, Kim JH, Burns MR, et al. Coupled formation of an amidotransferase interdomain ammonia channel and a phosphoribosyltransferase active site.Biochemistry, 1997, 36(37): 11061–11068.DOI: 10.1021/bi9714114 |
| [28] | Miles EW, Rhee S, Davies DR. The molecular basis of substrate channeling.J Biol Chem, 1999, 274(18): 12193–12196.DOI: 10.1074/jbc.274.18.12193 |
| [29] | Bayer EA, Belaich JP, Shoham Y, et al. The cellulosomes: multienzyme machines for degradation of plant cell wall polysaccharides.Ann Rev Microbiol, 2004, 58(1): 521–554.DOI: 10.1146/annurev.micro.57.030502.091022 |
| [30] | Doi RH, Kosugi A. Cellulosomes: plant-cell-wall-degrading enzyme complexes.Nat Rev Microbiol, 2004, 2(7): 541–551.DOI: 10.1038/nrmicro925 |
| [31] | Tsai SL, Oh J, Singh S, et al. Functional assembly of minicellulosomes on the Saccharomyces cerevisiae cell surface for cellulose hydrolysis and ethanol production.Appl Environ Microbiol, 2009, 75(19): 6087–6093.DOI: 10.1128/AEM.01538-09 |
| [32] | You C, Myung S, Zhang YHP. Facilitated substrate channeling in a self-assembled trifunctional enzyme complex.Angew Chem Int Ed, 2012, 51(35): 8787–8790.DOI: 10.1002/anie.v51.35 |
| [33] | Liu F, Banta S, Chen W. Functional assembly of a multi-enzyme methanol oxidation cascade on a surface-displayed trifunctional scaffold for enhanced NADH production.Chem Commun, 2013, 49(36): 3766–3768.DOI: 10.1039/c3cc40454d |
| [34] | Hirakawa H, Nagamune T. Molecular assembly of P450 with ferredoxin and ferredoxin reductase by fusion to PCNA.Chembiochem, 2010, 11(11): 1517–1520.DOI: 10.1002/cbic.v11:11 |
| [35] | Hirakawa H, Kakitani A, Nagamune T. Introduction of selective intersubunit disulfide bonds into self-assembly protein scaffold to enhance an artificial multienzyme complex's activity.Biotechnol Bioeng, 2013, 110(7): 1858–1864.DOI: 10.1002/bit.24861 |
| [36] | Dueber JE, Wu GC, Malmirchegini GR, et al. Synthetic protein scaffolds provide modular control over metabolic flux.Nat Biotechnol, 2009, 27(8): 753–759.DOI: 10.1038/nbt.1557 |
| [37] | Moon TS, Dueber JE, Shiue E, et al. Use of modular, synthetic scaffolds for improved production of glucaric acid in engineered E. coli.Metab Eng, 2010, 12(3): 298–305.DOI: 10.1016/j.ymben.2010.01.003 |
| [38] | Agapakis CM, Ducat DC, Boyle PM, et al. Insulation of a synthetic hydrogen metabolism circuit in bacteria.J Biol Eng, 2010, 4: 3.DOI: 10.1186/1754-1611-4-3 |
| [39] | Baek JM, Mazumdar S, Lee SW, et al. Butyrate production in engineered Escherichia coli with synthetic scaffolds.Biotechnol Bioeng, 2013, 110(10): 2790–2794.DOI: 10.1002/bit.24925 |
| [40] | Wang YC, Yu O. Synthetic scaffolds increased resveratrol biosynthesis in engineered yeast cells.J Biotechnol, 2012, 157(1): 258–260.DOI: 10.1016/j.jbiotec.2011.11.003 |
| [41] | Gao X, Yang S, Zhao CC, et al. Artificial multienzyme supramolecular device: highly ordered self-assembly of oligomeric enzymes in vitro and in vivo.Angew Chem Int Ed Engl, 2015, 53(51): 14027–14030. |
| [42] | Zhao CC, Gao X, Liu XB, et al. Enhancing biosynthesis of a ginsenoside precursor by self-assembly of two key enzymes in Pichia pastoris.J Agric Food Chem, 2016, 64(17): 3380–3385.DOI: 10.1021/acs.jafc.6b00650 |
| [43] | Yang ZW, Gao X, Xie H, et al. Enhanced itaconic acid production by self-assembly of two biosynthetic enzymes in Escherichia coli.Biotechnol Bioeng, 2016, 114(2): 457–462. |
| [44] | Somasundaram S, Eom GT, Hong SH. Efficient malic acid production in Escherichia coli using a synthetic scaffold protein complex.Appl Biochem Biotechnol, 2017, 184(4): 1308–1318. |
| [45] | Zhang L, Lu Y, Zhong YQ. Affibody molecules: a new class of ligands with high affinity.J Int Pharm Res, 2012, 39(2): 127–131.(in Chinese). 张磊, 鲁莹, 钟延强. Affibody分子:一类具有高度亲和力的新配体.国际药学研究杂志, 2012, 39(2): 127-131. |
| [46] | Tippmann S, Anfelt J, David F, et al. Affibody Scaffolds improve sesquiterpene production in Saccharomyces cerevisiae.ACS Synth Biol, 2016, 6(1): 19–28. |
| [47] | Shetty RP, Endy D, Knight Jr TF. Engineering biobrick vectors from biobrick parts.J Biol Eng, 2008, 2: 5.DOI: 10.1186/1754-1611-2-5 |
| [48] | Jaeger L, Chworos A. The architectonics of programmable RNA and DNA nanostructures.Curr Opin Struc Biol, 2006, 16(4): 531–543.DOI: 10.1016/j.sbi.2006.07.001 |
| [49] | Rothemund PW. Folding DNA to create nanoscale shapes and patterns.Nature, 2006, 440(7082): 297–302.DOI: 10.1038/nature04586 |
| [50] | Douglas SM, Dietz H, Liedl T, et al. Self-assembly of DNA into nanoscale three-dimensional shapes.Nature, 2010, 459(7245): 414–418. |
| [51] | Han DR, Pal S, Nangreave J, et al. DNA origami with complex curvatures in three-dimensional space.Science, 2011, 332(6027): 342–346.DOI: 10.1126/science.1202998 |
| [52] | Zhang F, Jiang SX, Wu SY, et al. Complex wireframe DNA origami nanostructures with multi-arm junction vertices.Nat Nanotechnol, 2015, 10(9): 779–784.DOI: 10.1038/nnano.2015.162 |
| [53] | Zuker M. Mfold web server for nucleic acid folding and hybridization prediction.Nucleic Acids Res, 2003, 31(13): 3406–3415.DOI: 10.1093/nar/gkg595 |
| [54] | Zadeh JN, Steenberg CD, Bois JS, et al. NUPACK: Analysis and design of nucleic acid systems.J Comput Chem, 2011, 32(1): 170–173.DOI: 10.1002/jcc.v32:1 |
| [55] | Andronescu M, Aguirre-Hernandez R, Condon A, et al. RNAsoft: a suite of RNA secondary structure prediction and design software tools.Nucleic Acids Res, 2003, 31(13): 3416–3422.DOI: 10.1093/nar/gkg612 |
| [56] | Douglas SM, Marblestone AH, Teerapittayanon S, et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno.Nucleic Acids Res, 2009, 37(15): 5001–5006.DOI: 10.1093/nar/gkp436 |
| [57] | Castro CE, Kilchherr F, Kim DN, et al. A primer to scaffolded DNA origami.Nat Methods, 2011, 8(3): 221–229.DOI: 10.1038/nmeth.1570 |
| [58] | Williams S, Lund K, Lin CX, et al. Tiamat: a three-dimensional editing tool for complex DNA structures//Goel A, Simmel FC, Sosík P, eds. DNA Computing. New York: Springer Berlin Heidelberg, 2008, 5347: 90-101. |
| [59] | Pinheiro AV, Han DR, Shih WM, et al. Challenges and opportunities for structural DNA nanotechnology.Nat Nanotechnol, 2011, 6(12): 763–772.DOI: 10.1038/nnano.2011.187 |
| [60] | Linko V, Dietz H. The enabled state of DNA nanotechnology.Curr Opin Biotechnol, 2013, 24(4): 555–561.DOI: 10.1016/j.copbio.2013.02.001 |
| [61] | Wilner OI, Shimron S, Weizmann Y, et al. Self-assembly of enzymes on DNA scaffolds: en route to biocatalytic cascades and the synthesis of metallic nanowires.Nano Lett, 2009, 9(5): 2040–2043.DOI: 10.1021/nl900302z |
| [62] | Wilner OI, Weizmann Y, Gill R, et al. Enzyme cascades activated on topologically programmed DNA scaffolds.Nat Nanotechnol, 2009, 4(4): 249–254.DOI: 10.1038/nnano.2009.50 |
| [63] | Fu JL, Yang YR, Dhakal S, et al. Assembly of multienzyme complexes on DNA nanostructures.Nat Protoc, 2016, 11(11): 2243–2273.DOI: 10.1038/nprot.2016.139 |
| [64] | Numajiri K, Yamazaki T, Kimura M, et al. Discrete and active enzyme nanoarrays on DNA origami scaffolds purified by affinity tag separation.J Am Chem Soc, 2010, 132(29): 9937–9939.DOI: 10.1021/ja104702q |
| [65] | Erkelenz M, Kuo CH, Niemeyer CM. DNA-mediated assembly of cytochrome P450 BM3 subdomains.J Am Chem Soc, 2011, 133(40): 16111–16118.DOI: 10.1021/ja204993s |
| [66] | Sun Q, Chen W. HaloTag mediated artificial cellulosome assembly on a rolling circle amplification DNA template for efficient cellulose hydrolysis.Chem Commun, 2016, 52(40): 6701–6704.DOI: 10.1039/C6CC02035F |
| [67] | Kou BB, Chai YQ, Yuan YL, et al. PtNPs as scaffolds to regulate interenzyme distance for construction of efficient enzyme cascade amplification for ultrasensitive electrochemical detection of MMP-2.Anal Chem, 2017, 89(17): 9383–9387.DOI: 10.1021/acs.analchem.7b02210 |
| [68] | Negi S, Imanishi M, Matsumoto M, et al. New redesigned zinc-finger proteins: design strategy and its application.Cheminform, 2008, 39(30): 3236–3249. |
| [69] | Lee JH, Jung SC, Le MB, et al. Improved production of L-threonine in Escherichia coli by use of a DNA scaffold system.Appl Environ Microbiol, 2013, 79(3): 774–782.DOI: 10.1128/AEM.02578-12 |
| [70] | Chen Q, Yu S, Myung N, et al. DNA-guided assembly of a five-component enzyme cascade for enhanced conversion of cellulose to gluconic acid and H2O2.J Biotechnol, 2017, 263: 30–35.DOI: 10.1016/j.jbiotec.2017.10.006 |
| [71] | Sun Q, Madan B, Tsai SL, et al. Creation of artificial cellulosomes on DNA scaffolds by zinc finger protein-guided assembly for efficient cellulose hydrolysis.Chem Commun, 2014, 50(12): 1423–1425.DOI: 10.1039/C3CC47215A |
| [72] | Zhu LY, Qiu XY, Zhu LY, et al. Spatial organization of heterologous metabolic system in vivo based on TALE.Sci Rep, 2016, 6: 26065.DOI: 10.1038/srep26065 |
| [73] | Sachdeva G, Garg A, Godding D, et al. In vivo co-localization of enzymes on RNA scaffolds increases metabolic production in a geometrically dependent manner.Nucleic Acids Res, 2014, 42(14): 9493–9503.DOI: 10.1093/nar/gku617 |
| [74] | Shao M, Sha Z, Zhang X, et al. Efficient androst-1, 4-diene-3, 17-dione production by co-expressing 3-ketosteroid-Δ1-dehydrogenase and catalase in Bacillus subtilis.J Appl Microbiol, 2017, 122(1): 119–128.DOI: 10.1111/jam.2017.122.issue-1 |
| [75] | Jones JA, Toparlak ?D, Koffas MAG. Metabolic pathway balancing and its role in the production of biofuels and chemicals.Curr Opin Biotechnol, 2015, 33: 52–59.DOI: 10.1016/j.copbio.2014.11.013 |
| [76] | Ahmadi MK, Pfeifer BA. Recent progress in therapeutic natural product biosynthesis using Escherichia coli.Curr Opin Biotechnol, 2016, 42: 7–12.DOI: 10.1016/j.copbio.2016.02.010 |
