
 , 卢元2
, 卢元2

1. 沈阳师范大学 生命科学学院,辽宁 沈阳 110034;
2. 清华大学 化学工程系,北京 100084
收稿日期:2019-06-22;接收日期:2019-08-07
基金项目:国家自然科学基金(Nos. 21706144, 21878173),北京市自然科学基金(No. 2192023)资助
作者简介:卢元 清华大学化学工程系助理教授、博士生导师。课题组研究的焦点在于发展和应用前沿合成生物学技术,结合多学科交叉手段,突破天然生命体系的限制,高效合成和精准改造复杂蛋白质、非天然蛋白质等生物大分子,以解决生物制造、人类健康等领域最具挑战性的科学与工程难题。目前已在Proc Natl Acad Sci USA等杂志上发表50多篇高影响学术论文,拥有5项国际授权专利、12项国内授权专利。研究成果得到了相关领域国内国际同行专家的广泛关注和高度评价。更多信息请浏览课题组网站:http://LuLab.org.
摘要:无细胞合成生物系统,能够在体外完成生命转录翻译过程,因体系灵活开放、便于控制、表达周期短、高耐受性等特点,可表达细胞系统难以表达的蛋白质。随着无细胞生物传感和体系冻干技术的不断发展,其在医药健康领域的应用不断拓展。本文综述了无细胞合成生物学在按需生物医药合成和便携式医疗检测等医药健康领域的研究进展,该体系的进一步发展有潜力实现更复杂后修饰蛋白质药物的合成、可丰富无细胞生物传感器类型并提高其灵敏性。无细胞合成生物学作为新兴工程策略,未来必将更好地应用于高通量医药蛋白质筛选、新型病原体的检测等医药健康领域。
关键词:无细胞合成生物学医药蛋白质便携式检测无细胞生物传感器
Cell-free synthetic biology: an emerging strategy torevolutionize the biomedical industry
Yingying Liu1,2, Ning Bu1

 , Yuan Lu2
, Yuan Lu2

1. College of Life Sciences, Shenyang Normal University, Shenyang 110034, Liaoning, China;
2. Department of Chemical Engineering, Tsinghua University, Beijing 100084, China
Received: June 22, 2019; Accepted: August 7, 2019
Supported by: National Natural Science Foundation of China (Nos. 21706144, 21878173), Beijing Natural Science Foundation (No.2192023)
Corresponding author: Ning Bu. E-mail: buning@sohu.com;
Yuan Lu. Tel/ Fax: +86-10-62780127; E-mail: yuanlu@tsinghua.edu.cn.
Abstract: Cell-free synthetic biology system can perform biological transcription and translation process in vitro. Because of its advanced features, such as flexible openness, easy control, short expression time and high tolerance to cytotoxicity, this systemhas been successfully used to synthesize proteins that are difficult to express in cells. With the continuous development of cell-free biosensing technology and the lyophilization technology, its applications have widely expanded into many biomedical fields. This review discusses the current research progress of cell-free synthetic biology system in on-demand biopharmaceutical synthesis, portable diagnostics, and others. Further development of the system can lead to even more complicated synthesis of therapeutic proteins with post-translational modifications and evolution of different cell-free biosensors with high sensitivity. Cell-free synthetic biology as an emerging engineering strategy can be a better means applied to high-throughput screening of pharmaceutical proteins, detection of new pathogens, and other important health-care fields in the future.
Keywords: cell-free synthetic biologypharmaceutical proteinportable detectioncell-free biosensors
疾病严重威胁人类的健康和生存,也是阻碍社会经济发展的重要原因之一,因此能够快速准确地诊断和治疗疾病,对人类健康尤为重要。威胁人类生存的几种重大疾病比如癌症和肿瘤发病率逐年递增并且向着年轻化发展[1],其诊断和治疗是研究人员一直未能攻克的难题。以手术和放化疗为主的传统疗法虽然正随着生物技术的发展而进步,但依然有很大的副作用,迫切需要研究者们在治疗手段方面有所创新和突破。另外近年来,全球生态环境不断变化,全球蚊媒病毒病呈上升趋势[2],许多病毒性、细菌性和寄生虫感染疾病在临床上难以区别,对其早期的确诊在医疗上具有重大的防控意义。飞速发展的现代医学虽能够控制某些传染病的传播,但是大多数情况下,很多传染病依然潜伏在人们身边,特别是贫穷地区和体弱人群易受感染[3]。这就迫切需要人们将疾病治疗推广到贫困地区并且研究更快速有效的检测和治疗方法来帮助人类克服这些重大疾病。
为了更好地克服这些疾病带来的危害,近几年来,蛋白质、多肽类药物[4]凭借极高的靶向特异性、高活性和低毒性的特点,在临床上的应用越来越广泛。有许多销售额排名靠前的药物都是蛋白质药物,如曲妥珠单抗[5]、阿达木单抗[6]、甘精胰岛素[7]和肺炎球菌13价结合疫苗[8]等。目前,研究者们利用哺乳动物细胞制备蛋白质药物取得了很大进展,并且随着生物技术与合成生物学的快速发展,许多基因工程药物和重组药物被合成,许多新药被发现,丰富了蛋白质药物的种类并提高了疾病治愈的几率。但是目前蛋白质药物70%都是由中国仓鼠卵巢细胞CHO生产[9],细胞内表达的蛋白质药物生长周期长导致筛选过程慢,甚至有些蛋白质药物由于对宿主细胞有毒性作用[10],无法在细胞内产生,还有一些蛋白质药物在偏远地区和环境恶劣缺少资源的地区无法合成,在运输过程中也存在失活现象。因此,解决细胞毒性、快速生产并且稳定运输是蛋白质药物研究的重点。
早期传染病病原体诊断方法正在不断进步,随着生物技术的不断发展,基于生物传感器的检测手段成为主流趋势,特别是即时检验(Point of care testing,POCT)[11]诊断技术,因特异性强、灵敏度高等特点得到快速发展。其生物敏感元件通常包括生物分子酶、抗原、抗体等,细胞、细胞器或组织等。主要的生物传感器类型有酶催化法、酶联免疫法、全细胞传感器、金属纳米颗粒传感器和微流控生物芯片传感器等。现有这些传感器虽然反应快速,灵敏度高,但是仍然存在一些缺点,如酶的稳定性低、全细胞生物传感器的生物安全问题、纳米材料和微流控传感器制作成本高且相对复杂等[12-13]。这就需要探索出稳定性高、相对简单并且能够解决生物安全问题的新型检测生物传感器。
为了解决上述目前疾病检测和治疗手段存在的问题,在蛋白质药物的生产和体外检测领域需要突破活细胞的限制,以便更灵活便捷地生产蛋白质药物和进行体外检测。因此,有研究者开始使用无细胞蛋白质合成系统(Cell-free protein synthesis,CFPS)[14]进行蛋白质药物合成和基于无细胞体系的生物传感器的研究。CFPS是以mRNA或DNA为模板,利用细胞提取物中的底物和能源物质,在体外完成蛋白质合成翻译及后修饰过程。CFPS体系开放、便于控制表达过程、较短的表达周期,并且可耐受细胞毒性和表达细胞难表达蛋白质[14]。近年来,随着冻干技术的发展,冻干无细胞体系可以维持其活性长达一年之久,而且储存的无细胞试剂可以在不到两周的时间内生产出高滴度的疫苗。另外,基于无细胞体系的生物传感器[15]也凭借体系的灵活开放,可以实现低成本、高灵敏度、低消耗的快速体外诊断。本文综述了无细胞蛋白质合成系统近几年来在医药蛋白质、医药健康方面的研究进展与应用。并进一步展望了该技术存在的重大挑战和未来的重要发展方向。
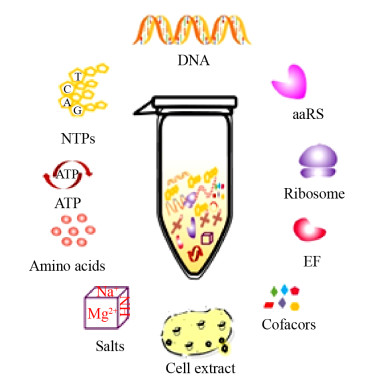
1 无细胞蛋白质合成系统1.1 无细胞体系概述CFPS通过利用微生物、动物、植物细胞的提取物并补充底物和能量,以外源mRNA或者DNA为模板,在体外完成转录、翻译、折叠、后修饰和能量代谢过程,如图 1所示。随着CFPS体系的发展,CFPS已经解决了膜蛋白、非天然蛋白质、抗体片段、类病毒颗粒的表达问题和纸上检测的问题。无细胞技术发展始于1907年,德国化学家Eduard Buchner使用破碎后的酵母细胞进行酒精发酵,从而发现了酒精发酵中底物酶系作用,证实了无需活细胞仍然可以发生生物反应[16]。1961年美国生物学家Heinrich Matthaei和Marshall Warren Nirenberg利用大肠杆菌无细胞体系,将只含有尿嘧啶的mRNA成功翻译出一条只有苯丙氨酸的多肽,并首次发现了蛋白质翻译时三联密码子UUU对应苯丙氨酸。自此,CFPS技术得到快速发展。
 |
| 图 1 无细胞蛋白质合成系统 Fig. 1 Cell-free protein synthesis system. |
| 图选项 |
目前,在直接控制转录和翻译的基础上,研究者们构建和发展了两种主要类型的无细胞蛋白质合成系统。一种是基于提取物的系统;另一种是纯化后的体系。理论上,任何生物体都可以作为无细胞体系中细胞提取物的来源, 但最常见的细胞提取物来源是大肠杆菌、小麦胚芽、酵母细胞[17]、兔网织红细胞和昆虫细胞等。随着研究者的不断探索,近年来一些其他来源的提取物也开始被研究,包括链霉菌属物种[18]、弧菌[19]、枯草芽孢杆菌[20]、醋酸杆菌[21]、中国仓鼠卵巢细胞CHO[22]和哺乳动物细胞[23-24]等。常用原核生物提取物来自大肠杆菌,因为大肠杆菌易培养、成本低、蛋白质表达量高[25]。但是由于原核体系的折叠和翻译机制,大肠杆菌无细胞系统仍然有其固有的局限性。而真核无细胞体系,对真核蛋白的翻译后修饰和正确折叠等方面占据优势。如酵母细胞提取物在创建功能性糖基化蛋白质方面取得了进展[26]。此外,小麦胚芽无细胞体系还能够构建在生物科学和医学领域具有巨大的潜力的蛋白质微阵列[27-28]。另一种纯化后的体系包括从大肠杆菌中纯化的一系列翻译组分,包括起始因子、延伸因子(Elongation factors,EFs)、释放因子(Release factors,RFs)、核糖体回收因子、20个氨酰-tRNA合成酶(Aminoacyl tRNA synthetase,aaRS)、蛋氨酸-tRNA转化酶、核糖体等。如Shimizu等开发了一个由36种参与转录和翻译的酶组成,以及高度纯化的核糖体的完全重组的无细胞系统,称为PURE体系[29]。因此,PURE体系的蛋白质合成反应完全缺乏蛋白酶和核酸酶,减少了表达模板等DNA元件的损失,是研究生物学的一个非常强大和基础的研究工具。然而,由于PURE体系的成本高,因此以粗提物为基础的CFPS仍然是研究的首选。
为了进行DNA转录和蛋白质合成,除了细胞粗提物外,系统还需要补充其他基本成分,包括DNA模板、RNA聚合酶、提供能量的底物、氨基酸、核苷三磷酸盐(Nucleoside triphosphate,NTPs)、tRNAs、辅因子和盐等。能够激活细胞提取物中促进蛋白质合成的通路是CFPS研究的关键。目前,许多研究都集中在刺激中枢代谢以促进高水平CFPS,而不是由磷酸烯醇丙酮酸(Phosphoenolpyruvate,PEP)或类似化合物驱动的昂贵的一步磷酸化反应。Calhoun等证明葡萄糖可以促进蛋白质合成[30-31],并且他们还利用单磷酸核苷(Nucleoside monophosphate,NMPs)取代核苷三磷酸(NTPs),在保持高蛋白质产量的同时,进一步降低了能源成本[30, 32]。还有研究人员利用聚合碳水化合物,如麦芽糊精[33]和可溶性淀粉或糖原[34]作为能量底物以降低能源成本。
另外,体系中的基因模板是影响无细胞系统中的稳定性和限制表达效率的关键因素。传统无细胞体系使用质粒作为基因表达模板,系统较稳定,但是存在制作质粒耗时较长,不能用于高通量蛋白表达等问题。所以近年来,以线性PCR产物直接作为表达模板得到了研究者的青睐,有研究者以单克隆抗体的线性PCR产物通过无细胞体系合成单克隆抗体[35]。但由于线性表达模板较质粒相比更加脆弱,易被细胞提取物中的核酸酶降解,于是Wu等利用特殊引物将线性片段制作成了类似于质粒的环状DNA分子,从而提高目的基因模板抵御核酸外切酶的能力[36]。还有研究者以细胞提取物为研究对象提高体系稳定性,Ahn等对体系中mRNA结构进行改造,使之更稳定,并且利用敲除了编码核酸内切酶E基因的菌株制备细胞提取物,提高了基于PCR产物的CFPS的生产力,为高通量蛋白质生产提供更便捷的可能[37]。
除了CFPS体系内物质对它的限制,在反应过程中产生的副产物的积累,也可能对CFPS产生限制。Spirin等在大肠杆菌和真核无细胞系统中引入了连续无细胞蛋白质合成的概念[38],使用连续交换或双层系统,因为被动扩散可以补充底物和去除副产物。连续补加大大延长了CFPS反应时间并提高了蛋白质产量[39]。
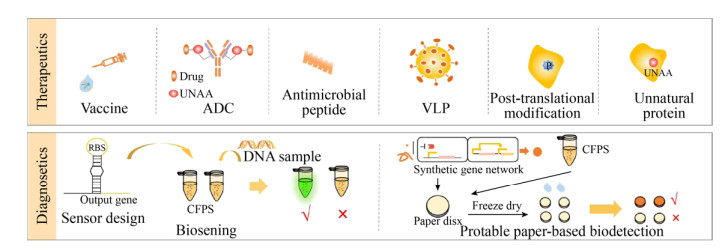
1.2 无细胞体系的优势近几年来,无细胞合成体系经过优化得到了快速发展。CFPS在以下几个方面具有重要优势。1)体系开放,可以耐受细胞毒性蛋白质;可产生难表达蛋白质,如具有细胞毒性的硒代蛋氨酸[40]和膜蛋白[41]等。2)反应迅速,且真核CFPS体系有丰富的蛋白质翻译后修饰能力;有研究组可以在24 h内就可以分别利用真核和原核的CFPS系统产生重组人红细胞生成素(Recombinant human erythropoietin rhEPO)和重组人粒细胞巨噬细胞集落刺激因子(Recombinant human granulocyte macrophage colony-stimulating factor,rhGMCSF)[42]。3)便于调控反应过程[14],利于非天然氨基酸与蛋白质的位点特异性结合,实现非天然蛋白质的合成[43]。4)基于CFPS的生物传感器操作简便,能够解决生物安全性问题,产生丰富类型的传感器。5)结合冻干技术的纸上CFPS可以实现反应的便携式,打破冷链运输问题;储存的无细胞试剂可以快速生产出高滴度的疫苗,例如,用于癌症治疗的活性核糖核酸酶onconase也可以通过冻干CFPS系统生产[44]。因为这些优势,CFPS在表达重组蛋白质药物、后修饰药物、非天然蛋白质药物、便携式生物传感器以及体外诊断等表现出巨大应用潜力。
2 无细胞体系在医药健康领域的应用2.1 无细胞体系合成医药蛋白2.1.1 疫苗接种疫苗被认为是预防传染性疾病最有效的的手段。通过接种有效的疫苗,已经消灭了天花、牛瘟等重要人和动物传染病。目前用于人类疾病防治的疫苗根据技术特点可分为传统疫苗和新型疫苗[45]。传统疫苗具有安全高效和免疫谱广的特点,主要包括减毒活疫苗和灭活疫苗;新型疫苗相比于传统疫苗来说,具有更好的安全性和稳定性,主要类型是基因工程疫苗[46]。目前疫苗大多由不同种类的细胞产生,如真核细胞体系可以产生糖基化疫苗[47],但是一些疫苗的生长制作周期长,无法与抗原变异速度匹配,对宿主细胞有毒性作用并且也存在运输导致失活现象[48]。因此,在疫苗的生产领域需要一个具有突破性的体系,而CFPS体系可以满足疫苗合成的灵活、快速、低成本并且耐受细胞毒性的需求,甚至冻干后的体系也可以在短时间内产生高滴度的疫苗[49],能够打破传统冷链的运输方式。
无细胞原核系统大肠杆菌体系为快速生产疫苗提供了一个快速设计合成平台。Kanter等开发了一种可产生高产量的生物活性粒细胞-巨噬细胞集落刺激因子(GM-CSF)的无细胞蛋白质表达系统,产生的蛋白质可以作为小鼠B细胞淋巴瘤的疫苗,并进一步提高蛋白质的溶解度和生物活性[49]。Lu等描述了一种基于大肠杆菌的CFPS方法,用于从2009年H1N1大流行毒株中提取HA茎域三聚体,他们的方法为广泛的保护性疫苗提供了广阔的前景,同时也展示了一种独特的方法来生产复杂多聚体蛋白质的单个结构域[50]。另外,真核无细胞系统能够表达出更复杂的疫苗,如一直存在重组蛋白质难以表达问题的疟疾蛋白已经可以在CFPS体系内成功表达。Tsuboi等利用小麦胚芽无细胞系统成功表达了3种疟疾蛋白,并成功在小鼠体内表现出良好的免疫原性,表明无细胞体系有发现疟疾候选疫苗方面的潜力[51]。另外,有一类疫苗需要按照患者的需求而制备出适合患者的个性化疫苗,通常需要从患者受影响的细胞中分离出核酸,继而表达出蛋白质用作抗原引起免疫反应。尤其是在晚期癌症的诊断中,受有效治疗时间的限制,快速和随需应变的蛋白质生成至关重要,无细胞体系有望成为这种快速随需应变的生产平台。
2.1.2 抗体抗体是指抗原刺激机体的免疫系统后免疫细胞产生的、可与相应抗原发生特异性结合的具有保护作用的免疫球蛋白,是生物目标的关键检测元件,也是疾病治疗、诊断、研究应用的重要工具[52]。近年来,随着生物技术的发展,抗体解决了免疫原性问题,并且能够作为靶向载体输送药物以及作为免疫疗法激活免疫应答[53]。目前重组抗体已经成为重要的治疗性蛋白,主要通过哺乳动物细胞系进行生产,但是体内表达的生长周期限制了下一步筛选的速率,后续的纯化过程也有一定难度,并且一些复杂抗体仍很难合成[54]。因此,抗体的合成需要一个更快速简便的生产体系。而CFPS凭借其反应迅速、纯化简单并且真核无细胞体系能够满足复杂抗体的后修饰需要,成为合成抗体的新兴体系[55]。
目前,CFPS已经成功地用于抗体和抗体片段的合成。如Patel等利用大肠杆菌无细胞体系创造了含有炔丙基甘氨酸的萤光素酶变体以及携带pAzF的scFv与IM9蛋白的融合构建体,而且证明了用其检测有独特表面标记的细胞的可行性[56]。原核无细胞体系仍是重组抗体乃至全长抗体的主要来源。1997年,Ryabova等首次在基于大肠杆菌的无细胞体系中合成scFv抗体片段[57]。不久,Merk等使用类似的无细胞体系,通过添加还原性和氧化性谷胱甘肽、蛋白质二硫键异构酶(PDI)和伴侣蛋白,成功合成了两个抗溶菌酶scFv抗体片段[58]。并且已经证明,在合成scFv抗体片段时,添加还原性和氧化性谷胱甘肽、PDI和分子伴侣蛋白可以提高目标蛋白的溶解度和抗原结合活性[59]。此外,为了对复杂分子的折叠提供一个更合适的环境,真核细胞无翻译系统也已被用于合成功能二硫键蛋白,包括scFv和Fab抗体片段。虽然真核生物的无细胞翻译系统与大肠杆菌相比效率较低,但它们为合成可溶性全长蛋白[60]、复杂蛋白和需要翻译后修饰的蛋白提供了更好的基础[61]。Stech等利用中国仓鼠卵巢(CHO)无细胞体系展示了复杂抗体的合成,特别是免疫球蛋白G (IgG)和单链可变片段Fc融合(scFv-Fc)[62]。Takeda等利用小麦无细胞蛋白质合成系统和脂质体技术,利用双层透析法和生物素化脂质体相互作用试验(BiLIA)两种方法制备抗G蛋白耦联受体(G-protein-coupled receptors,GPCR)单克隆抗体[63]。小麦胚芽细胞无蛋白质合成系统也被用于制备可溶性高、免疫完整的中东呼吸综合征冠状病毒核衣壳蛋白(Middle East respiratory syndrome coronavirus,MERS-CoV)单克隆抗体。
此外,双特异性抗体是近年来在肿瘤学、炎症性疾病和感染性疾病治疗领域中出现的一个有前景的研究领域。双特异性抗体的双重目标识别能力使新的治疗假设得以验证,Xu等使用无细胞表达系统(Xpress CF)在多个支架中产生(Knobs- into-holes,KIH)双特异性抗体的方法,实现高效的KIH生产[64]。
2.1.3 抗菌肽抗菌肽(Antimicrobial peptides,AMPs)是多细胞生物先天免疫防御系统的重要组成部分[65]。与抗生素的药效和作用方式的不同,抗菌肽没有药物屏蔽作用,热稳定性好,AMPs可作为传统抗生素的替代品备受关注[66]。然而天然抗菌肽来源有限、化学合成成本高、纯化困难、难以保证活性稳定性,基因重组成为合成AMPs的新方法,其中主要以大肠杆菌细胞体系最为常见[67]。但是大部分抗菌肽在基因工程生产中对宿主都有害,而且对于许多抗菌肽的作用机制还需要进一步研究,这就需要一个能够相对迅速且耐受毒性的体系来大量合成抗菌肽,为进一步改造抗菌肽打下基础。不同提取物来源的CFPS可以耐受毒性并满足多种抗菌肽的快速生产,利于对抗菌肽作用机制的研究及改造抗菌肽[68]。
虽然许多抗菌肽通过破坏细胞膜来裂解细胞,但有一类特殊的非裂解肽,称为富含脯氨酸抗菌肽(PrAMPs),作用于细胞内的目标核糖体,分布于所有生物体内。其中吡咯霉素是一种富含脯氨酸的抗菌肽(PrAMP),通过与伴侣蛋白DnaK (Chaperone protein DnaK)特异性结合,以剂量依赖的方式杀死敏感物种。Taniguchi等为了阐明吡咯霉素的作用机制,使用大肠杆菌无细胞体系,比较了吡咯霉素的添加量对总基因表达过程的影响,与常规抗生素相比较,吡咯霉素对GFP合成的这种抑制作用不是发生在转录阶段,而是发生在翻译阶段,这与链霉素对GFP合成的抑制作用类似[69]。
2.1.4 类病毒颗粒类病毒颗粒(Virus like particles,VLPs)是具有自组装能力的多亚基蛋白质复合物,由于缺乏病毒遗传物质,因此不具传染性,也无法复制,是一种相比减毒或灭活病毒的传统疫苗更安全的新兴疫苗[70]。VLP在疫苗、药物传递、基因治疗和材料科学等方面的应用受到广泛关注[71]。大多数VLP通过细胞体系产生,相比于细菌和酵母体系,昆虫细胞表达系统因其生长速度快、可大规模培养并且具有与哺乳动物细胞相似的翻译后修饰能力等优点而得到广泛应用[72-73];但是昆虫体系的最大问题就是去活、清除和提纯的下游工艺中的操作难点以及在宿主细胞的特定环境中运输稳定性问题。而且在细胞内产生VLP会受到难以控制的环境和VLP毒性的影响,细胞无法正常生长。由于自组装效率低,特别是对于复杂的VLPs,它们应用往往受到设计和生产上的阻碍[74]。因此,还需要建立新的能够耐受毒性、纯化简便的操作系统方法,用于筛选VLP的自组装和稳定因素。CFPS系统可以方便地用于高通量筛选,加快VLP的开发,是一个有吸引力的能够灵活表达VLP蛋白并进行体外组装的平台[75]。
有研究者已经成功在大肠杆菌和酵母无细胞体系中成功生产VLP。同样,含有有毒中间体A2蛋白和非天然氨基酸的VLP也已在无细胞体系中成功制备[76]。Cerqueira等已经建立了一个无细胞系统来研究人类乳头瘤病毒16型(HPV16)的组装[77]。其中,原核无细胞体系可以在数小时之内组装起VLP,2007年Bundy等第一次使用基于原核细胞的体外转录翻译系统产生病毒样颗粒,并且以高产量形式生产MS2噬菌体表面蛋白VLP和C末端截短的乙肝核心蛋白VLP,而且这些VLP与体内产生的VLP具有相似的特性[78]。Sheng等利用大肠杆菌的CFPS系统可以在4 h内合成并组装人诺如病毒(Human noroviruses,HuNoVs)[79]。Chien等将在大肠杆菌中产生的神经坏死病毒(Nervous necrosis virus,NNV)重组衣壳蛋白,后经过无细胞系统自组装形成点带石斑鱼神经坏死病毒(Orange-spotted grouper NNV,OSGNNV) VLP,保留了原病毒的结构特征且具有较强的免疫原性和较高的疫苗应用潜力,与细胞内组装的VLPs相比更安全[80]。
另外,有些VLP还具有药物传递载体作用,如豇豆绿斑病毒(Cowpea chlorotic mottle virus,CCMV)在外表面附有标签以促进它的药物传递载体作用和构建产分子结构,还需要控制衣壳内的RNA含量,Garmann等通过一种高效的无细胞体系合成CCMV颗粒,可以用于封装任何来源的异种RNA[81]。
2.1.5 后修饰药物翻译后修饰(Post-translational modification,PTM)指蛋白质在翻译后的化学修饰,其在调节蛋白质活性、结构和功能等方面发挥着重要的作用[82],能够使蛋白质的结构变得复杂,功能更加完善,目前已经发现了20种常见的蛋白质翻译后修饰类型。很多医药蛋白都具有不同形式的后修饰,在蛋白质翻译后修饰中的研究中,获得大量位点专一性蛋白质是关键问题[83]。目前常见的翻译后修饰蛋白质的生物学合成方法主要利用真核表达体系、酶学催化和非天然氨基酸法。生物学方法在早期产生了很多后修饰蛋白,但是由于蛋白质后修饰在整个细胞生命活动周期中存在着时空的调控,所以,对于同一种蛋白质来说,不同时期存在不同的后修饰类型。而对于位点的精准性,研究者会采用将非天然氨基酸以特定位点结合到蛋白质上[84]。另外酶催化法的效率低并且种类少[85]。因此,为了更精确地合成翻译后修饰,就需要利用一个能提高位点精准度、效率高的体系。而近几年随着CFPS体系的发展,已经能够将非天然氨基酸位点特异性地与蛋白质结合,并且CFPS的真核体系能够实现许多种类的后修饰[86]。
糖基化是主要的PTM类型之一,是药物蛋白开发中非常重要的领域,对重组蛋白质治疗药物的生产尤为重要,因为糖基化不当,会对治疗药物的治疗活性或循环半衰期产生不利影响[87]。2012年,Guarino等首次在原核无细胞体系合成N-链糖蛋白[88]。关于另一种糖基化蛋白疟疾蛋白,之前使用原核和真核细胞表达的疟疾蛋白均糖基化不佳,通常可溶性差,折叠不当,导致免疫原性降低。而基于HeLa细胞的CFPS却可以体外表达糖基化的疟疾蛋白[23]。此外利用优化大肠杆菌菌株,获得具有选择性糖基化成分的细胞提取物,其中包括寡糖转移酶(OSTs)和脂联寡糖(LLOs)[89],也可以实现糖基化。另一种方式,在CFPS中添加微粒体CHO也可以促进蛋白质糖基化,如Gurramkonda等利用CHO无细胞系统生产重组促红细胞生成素(EPO),使EPO的表达纯化率提高了一倍[90]。
磷酸化是磷酸基团与蛋白质的可逆结合,也是自然界中最重要的PTMs之一。近年来无细胞体系为测量和研究位点特异性磷酸化作出了巨大贡献,Oza等通过原核CFPS体外合成高活性的双磷酸化人MEK1激酶,这也是首次证明了由原核CFPS合成活性的MEK1,而不需要融合蛋白来实现可溶性蛋白的表达[91]。
无细胞系统凭借其开放性还能促进蛋白质折叠,使其非常适合合成含二硫键的蛋白质,以及进行结构和功能分析。Matsuda等开发了基于大肠杆菌细胞裂解液的CFPS,成功合成了毫克数量级的功能性具有二硫键的高等真核蛋白牛胰蛋白酶抑制剂(BPTI)和人溶菌酶C (LYZ)[92]。
此外,无细胞体系还能产生N端生物素化蛋白质库(BPL),与患者血清抗体反应作为检测自体抗原的快速筛选方法[93]。
2.1.6 非天然蛋白质药物非天然氨基酸(Unnatural amino acids, UNAAs)[94]在蛋白质合成中的嵌入是强大的生物合成工具,利用含有特殊侧链的非天然氨基酸取代天然氨基酸可以提供多种具有新的理化性质和生物学功能的蛋白质。非天然氨基酸大多数是在细胞内通过抑制终止密码子,利用氨酰tRNA合成酶/tRNA对来实现将非天然氨基酸的嵌入[95]。这就需要维持细胞的生长状态,但是过程繁琐导致产量低,且一些UNAA不易穿过细胞膜,还会对细胞产生毒性,抑制细胞生长,后续的纯化步骤也较繁琐,这些都限制了UNAA的嵌入效率。UNAA嵌入蛋白质中需要打破活细胞体系的限制,因此,越来越多的研究使用CFPS体系进行非天然蛋白质的合成,因为CFPS没有细胞膜的屏障,能够提高UNAA的利用率,在配体蛋白相互作用、生物治疗等方面具有广阔的应用前景[96-97]。
目前,已经有很多研究成功利用CFPS实现了UNAA的嵌入和标记。Ugwumba等将荧光非天然氨基酸7-(羟基香豆素-4-酰基)乙基甘氨酸(Hco)标记西尼罗河病毒非结构蛋白酶,可以鉴定新的蛋白质抑制剂从而发现新药物[55]。在开发抗体-药物偶联物(Antibody-drug conjugate,ADC)的过程中,抗癌药物与针对特定肿瘤标记物的抗体偶联,通过无细胞系统将UNAA位点特异性地结合到抗体中,为药物的控制和稳定附着提供了一个位点[98]。在最近的一项研究中,Cho等将非天然氨基酸对乙酰苯丙氨酸(pAcF)加入到人类生长因子(hGH)基因序列的不同位置,从而允许其位点特异性地与聚乙二醇(PEG)结合。将另外一类非天然D型氨基酸加入蛋白质或多肽中也有一些研究,Katoh等使用工程tRNAs和优化的翻译因子能够将十个连续的D-Ser引入一个肽段中,还表达了由4、5个连续的D型氨基酸组成的大环肽[99],这些研究为镜像药物研制奠定了基础。
 |
| 图 2 无细胞蛋白质合成系统在医药健康领域的应用 Fig. 2 Application of cell-free protein synthesis system in the field of medicine and health. |
| 图选项 |
2.2 无细胞体系与诊断2.2.1 无细胞生物传感器生物传感器是发展生物医药健康技术必不可少的一种高效灵敏的检测与监控方法,同时也是分子水平的微量、快速分析方法。目前生物传感器的类型主要包括酶催化法、酶联免疫法、全细胞传感器、金属纳米颗粒传感器和微流控生物芯片传感器等[100-102],反应快速,灵敏度高。但是以酶作为识别元件的传感器,由于酶的价格较昂贵且稳定性不够,其应用受到一定限制;纳米颗粒及微流控传感体系制作复杂且成本高;全细胞生物传感器会造成生物安全问题。而无细胞体系能够行使生物传感器的功能,而且没有生物安全问题。在无细胞体系下的生物传感器能像全细胞传感器一样接收外界样品中输入信号,经过一系列蛋白合成的生化反应后,输出可被检测的光信号、电信号等。
有些研究人员在转录水平上开发了无细胞生物传感器,这些基于转录的生物传感器在不存在目标分析物时,用来抑制指示蛋白质的DNA的转录[103]。Pellinen等提出了一种以萤火虫荧光素酶为报告基因的四环素和汞诱导启动子为模型的、用于检测特定小分子配体诱导转录的无细胞生物传感器[15]。除了在转录水平上使用CFPS系统作为生物传感器外,在翻译阶段进行传感也是一种很有前途的方法。主要包括用于检测蛋白质翻译所需化学成分的传感器和利用翻译激活控制的传感器,其最直接的方法是检测转录和翻译所必需的分子,如氨基酸、核糖核苷酸以及Mg离子等。到目前为止,这种方法仅在翻译水平上用于氨基酸检测。Jang等利用缺乏氨基酸的CFPS,对外源氨基酸产生荧光信号,以检测某种氨基酸,其荧光强度与氨基酸浓度呈线性正比。该方法的检测限低至100 nmol/L,已成功应用于生物样品中与疾病相关氨基酸的定量检测中[104]。除了上述转录和翻译水平的CFPS生物传感器外,CFPS系统还可以利用翻译后的蛋白质折叠过程作为生物传感机制。Salehi等提出一个通过无细胞体系表达变构融合蛋白并通过比色反应直接检测化学物质与人体雌激素受体β (hERβ)的快速适应便携式体外检测生物传感器平台[94]。这种传感器的传感步骤是由蛋白质折叠调控触发的。
有些蛋白质则是通过CFPS来生产而用作生物传感,Kaiser利用CFPS合成了3种嗅觉受体蛋白hOR17-4、mOR23和mS51,是获得大量嗅觉受体设计仿生传感器的一个里程碑变化[105]。此外,为避免一些有毒无机离子和毒蛋白的毒性作用,Zhang等构建了5种不同的无细胞生物传感器来检测体系中的有毒无机离子砷和汞、以及有机分子3OC12-HSL、pC-HSL和苯甲酸。3OC12-HSL和pC-HSL都属于酰化高丝氨酸内酯(Acyl-homoserine lactone,AHL),是一种群体感应信号分子存在于许多囊性纤维化患者的痰、尿和血液中。因此,无细胞生物传感器是有效的疾病标志物的检测方法[106]。Davies等构建了包含多种病毒抗原的无细胞蛋白质微阵列,用于检测病毒免疫后的人或动物血清,从而发现强免疫原性的蛋白质用于预防接种的候选或者作为诊断指标[107]。
此外,虽然CFPS生物传感器主要使用廉价的大肠杆菌为基础的系统[104, 108],但随着开发出更复杂的生物传感器,其他非大肠杆菌为基础的CFPS生物传感器,包括农杆菌[109]、酵母[17]、小麦[110]和哺乳动物系统可能成为必要的系统。随着冻干技术的升级,冻干无细胞生物传感器更便携,而且满足了偏远环境恶劣地区应用冻干体系进行生物传感的需求。挑战仍然存在,并且需要更多的工程来实现许多应用所需的敏感性。
2.2.2 便携式检测现代医疗越来越依赖于生物疗法,偏远地区和恶劣环境中资源稀缺,病原体的诊断以及合成个性化的医疗蛋白较困难,因此在偏远和恶劣环境中检测和治疗的能力将是非常重要的。商业上可用的无细胞转录翻译系统(细菌或哺乳动物)被冻干在纸上或其他多孔基质上,以创建稳定的遗传调控网络,该网络可在室温下长期保存,并通过水合作用激活,Smith等提出了制备大肠杆菌无细胞冻干体系的方法,通过直接提取冻干和粉末能源系统的方法使无细胞粉末体系更加稳定和高密度存储和运输[111]。
基于纸上和其他多孔材料的冻干CFPS不仅克服冷库的限制、偏远地区的生物疗法和疫苗生产,还可以实现便携的体外诊断功能。Pardee等已经应用了基于纸上的冻干CFPS来快速检测埃博拉病毒和寨卡病毒[112-113]。Ma等通过基于纸张的CFPS、等温扩增和同源体的病毒富集来检测临床样本中的诺如病毒[114]。Jang等在利用个人血糖仪分析氨基酸的过程中利用无细胞合成转化酶生成葡萄糖,再利用个人血糖仪对氨基酸进行定量,其结果的准确性可与标准的高效液相色谱法相媲美[115]。
冻干CFPS还可以作为环境检测的便携工具,Gr?we等构建一个基于纸上的无细胞生物传感器用于水质评估,分别采用MerR和BlcR作为无细胞反应的转录调节剂进行检测离子Hg(Ⅱ)和小分子羟丁酸(GHB),输出基于绿色荧光蛋白(sfGFP)的表达信号,并且通过使用简单的滤光器和智能手机就能解决野外荧光检测的问题[116]。无细胞生物传感器有很大的应用潜力,还有待进一步开拓。
3 结论与展望医药健康与人类息息相关,在开发过程中往往需要进行工程设计以获得最佳的功能和理化性质。CFPS的开放、灵活、便携、可控等特点使之在蛋白质药物开发方面具有重大优势,实现了难表达医药蛋白、非天然药物蛋白、复杂后修饰蛋白等医药蛋白的快速高效合成。并且冻干CFPS体系可以解决疫苗运输和偏远地区对蛋白质合成需求等问题。虽然CFPS系统在各方面的快速发展,已解决了合成效率、合成成本、难表达蛋白质合成、高通量蛋白筛选、蛋白质后修饰等主要问题,但仍然存在一些挑战,比如缺乏低成本且高效的真核生物CFPS系统,以更好合成更复杂的真核蛋白质。CFPS系统虽然可以对蛋白质进行简单的翻译后修饰,但如何实现一些成熟药物蛋白质所需的极其复杂的修饰,还需要不断地探索。未来无细胞合成生物学的发展需要进一步发挥其灵活性和高通量潜力,应用于大规模高通量功能性医药蛋白筛选和不断发展以应对新型病原体的检测与治疗;并进一步利用其开放性特点,融合先进生物学、材料学、化学、物理学、计算机学等学科优势,以发挥其在医学领域巨大潜力,继续推动其在医药健康领域的发展和广泛应用。
参考文献
| [1] | Berg JW. The incidence of multiple primary cancers. I. Development of further cancers in patients with lymphomas, leukemias, and myeloma. J Natl Cancer Inst, 1967, 38(5): 741-752. |
| [2] | Hubálek Z. Mosquito-borne viruses in Europe. Parasitol Res, 2008, 103(1): 29-43. |
| [3] | Mabey D, Peeling RW, Ustianowski A, et al. Tropical infectious diseases: diagnostics for the developing world. Nat Rev Microbiol, 2004, 2(3): 231-240. DOI:10.1038/nrmicro841 |
| [4] | Cao S, Xu S, Wang H, et al. Nanoparticles: Oral delivery for protein and peptide drugs. Mol Ther, 2019, 20(5): 190. |
| [5] | Barok M, Joensuu H, Isola J. Trastuzumab emtansine: mechanisms of action and drug resistance. Breast Cancer Res, 2014, 16(2): 209. |
| [6] | Bartelds GM, Wijbrandts CA, Nurmohamed M, et al. Clinical response to adalimumab: relationship to anti-adalimumab antibodies and serum adalimumab concentrations in rheumatoid arthritis. Ann Rheum Dis, 2007, 66(7): 921-926. DOI:10.1136/ard.2006.065615 |
| [7] | Rosenstock J, Park G, Zimmerman J, et al. Basal insulin glargine (HOE 901) versus NPH insulin in patients with type 1 diabetes on multiple daily insulin regimens. US Insulin Glargine (HOE 901) Type 1 Diabetes Investigator Group. Diabetes care, 2000, 23(8): 1137-1142. DOI:10.2337/diacare.23.8.1137 |
| [8] | Moore MR, Link-Gelles R, Schaffner W, et al. Effect of use of 13-valent pneumococcal conjugate vaccine in children on invasive pneumococcal disease in children and adults in the USA: analysis of multisite, population-based surveillance. Lancet Infect Dis, 2015, 15(3): 301-309. DOI:10.1016/S1473-3099(14)71081-3 |
| [9] | Bosques CJ, Collins BE, Meador Ⅲ JW, et al. Chinese hamster ovary cells can produce galactose-α-1, 3-galactose antigens on proteins. Nat Biotechnol, 2010, 28(11): 1153. DOI:10.1038/nbt1110-1153 |
| [10] | Dumon-Seignovert L, Cariot G, Vuillard L. The toxicity of recombinant proteins in Escherichia coli: a comparison of overexpression in BL21 (DE3), C41 (DE3), and C43 (DE3). Protein Expression Purif, 2004, 37(1): 203-206. DOI:10.1016/j.pep.2004.04.025 |
| [11] | Luppa PB, Müller C, Schlichtiger A, et al. Point-of-care testing (POCT): Current techniques and future perspectives. TrAC, Trends Anal Chem, 2011, 30(6): 887-898. DOI:10.1016/j.trac.2011.01.019 |
| [12] | Zhang X, Ju H, Wang J. Electrochemical sensors, biosensors and their biomedical applications. Vol. Academic Press, 2011. |
| [13] | Cho H, Daniel T, Buechler YJ, et al. Optimized clinical performance of growth hormone with an expanded genetic code. Proc Natl Acad Sci, 2011, 108(22): 9060-9065. DOI:10.1073/pnas.1100387108 |
| [14] | Carlson ED, Gan R, Hodgman CE, et al. Cell-free protein synthesis: applications come of age. Biotechnol Adv, 2012, 30(5): 1185-1194. DOI:10.1016/j.biotechadv.2011.09.016 |
| [15] | Pellinen T, Huovinen T, Karp M. A cell-free biosensor for the detection of transcriptional inducers using firefly luciferase as a reporter. Anal Biochem, 2004, 330(1): 52-57. |
| [16] | Buchner E. Cell-free fermentation. Nobel Lecture, 1907, 103-120. |
| [17] | Gan R, Jewett MC. A combined cell-free transcription-translation system from Saccharomyces cerevisiae for rapid and robust protein synthe. Biotechnol J, 2014, 9(5): 641-651. DOI:10.1002/biot.201300545 |
| [18] | Li J, Wang H, Kwon YC, et al. Establishing a high yielding streptomyces-based cell-free protein synthesis system. Biotechnol Bioeng, 2017, 114(6): 1343-1353. DOI:10.1002/bit.26253 |
| [19] | Des Soye BJ, Davidson SR, Weinstock MT, et al. Establishing a high-yielding cell-free protein synthesis platform derived from Vibrio natriegens. ACS Synth Biol, 2018, 7(9): 2245-2255. DOI:10.1021/acssynbio.8b00252 |
| [20] | Kelwick R, Webb AJ, MacDonald JT, et al. Development of a Bacillus subtilis cell-free transcription-translation system for prototyping regulatory elements. Metab Eng, 2016, 38: 370-381. DOI:10.1016/j.ymben.2016.09.008 |
| [21] | Ullah MW, Ul-Islam M, Khan S, et al. Innovative production of bio-cellulose using a cell-free system derived from a single cell line. Carbohydr Polym, 2015, 132: 286-294. DOI:10.1016/j.carbpol.2015.06.037 |
| [22] | Br?del AK, Sonnabend A, Kubick S. Cell-free protein expression based on extracts from CHO cells. Biotechnol Bioeng, 2014, 111(1): 25-36. |
| [23] | Yadavalli R, Sam-Yellowe T. HeLa Based cell free expression systems for expression of Plasmodium rhoptry proteins. J Visualized Exp, 2015(100): e52772. |
| [24] | Br?del AK, Sonnabend A, Roberts LO, et al. IRES-mediated translation of membrane proteins and glycoproteins in eukaryotic cell-free systems. PLoS ONE, 2013, 8(12): e82234. DOI:10.1371/journal.pone.0082234 |
| [25] | Swartz J. Developing cell-free biology for industrial applications. J Ind Microbiol Biotechnol, 2006, 33(7): 476-485. DOI:10.1007/s10295-006-0127-y |
| [26] | Laukens B, De Wachter C, Callewaert N. Engineering the Pichia pastoris N-glycosylation pathway using the GlycoSwitch technology//Glyco-Engineering. Humana Press, New York, NY, 2015: 103–122. |
| [27] | Zhu H, Snyder M. Protein chip technology. Curr Opin Chem Biol, 2003, 7(1): 55-63. DOI:10.1016/S1367-5931(02)00005-4 |
| [28] | Merkel JS, Michaud GA, Salcius M, et al. Functional protein microarrays: just how functional are they?. Curr Opin Biotechnol, 2005, 16(4): 447-452. DOI:10.1016/j.copbio.2005.06.007 |
| [29] | Shimizu Y, Inoue A, Tomari Y, et al. Cell-free translation reconstituted with purified components. RSC Adv, 2001, 19(8): 751. |
| [30] | Calhoun KA, Swartz JR. An economical method for cell-free protein synthesis using glucose and nucleoside monophosphates. Biotechnol Prog, 2005, 21(4): 1146-1153. |
| [31] | Calhoun KA, Swartz JR. Energizing cell-free protein synthesis with glucose metabolism. Biotechnol Bioeng, 2005, 90(5): 606-613. DOI:10.1002/bit.20449 |
| [32] | Jewett MC, Calhoun KA, Voloshin A, et al. An integrated cell-free metabolic platform for protein production and synthetic biology. Mol Syst Biol, 2008, 4(1): 220. |
| [33] | Wang Y, Zhang YHP. Cell-free protein synthesis energized by slowly-metabolized maltodextrin. BMC Biotechnol, 2009, 9(1): 58. |
| [34] | Kim DM, Swartz JR. Regeneration of adenosine triphosphate from glycolytic intermediates for cell-free protein synthesis. Biotechnol Bioeng, 2001, 74(4): 309-316. |
| [35] | Ojima-Kato T, Hashimura D, Kojima T, et al. In vitro generation of rabbit anti-Listeria monocytogenes monoclonal antibody using single cell based RT-PCR linked cell-free expression systems. J Immunol Methods, 2015, 427: 58-65. DOI:10.1016/j.jim.2015.10.001 |
| [36] | Wu PSC, Ozawa K, Lim SP, et al. Cell-free transcription/translation from PCR-amplified DNA for high-throughput NMR studies. Angew Chem Int Ed, 2007, 46(18): 3356-3358. DOI:10.1002/anie.200605237 |
| [37] | Ahn JH, Chu HS, Kim TW, et al. Cell-free synthesis of recombinant proteins from PCR-amplified genes at a comparable productivity to that of plasmid-based reactions. Biochem Biophys Res Commun, 2005, 338(3): 1346-1352. DOI:10.1016/j.bbrc.2005.10.094 |
| [38] | Spirin AS, Baranov VI, Ryabova LA, et al. A continuous cell-free translation system capable of producing polypeptides in high yield. Science, 1988, 242(4882): 1162-1164. DOI:10.1126/science.3055301 |
| [39] | Endo Y, Sawasaki T. Cell-free expression systems for eukaryotic protein production. Curr Opin Biotechnol, 2006, 17(4): 373-380. DOI:10.1016/j.copbio.2006.06.009 |
| [40] | Kigawa T, Yamaguchi-Nunokawa E, Kodama K, et al. Selenomethionine incorporation into a protein by cell-free synthesis. J Struct Funct Genomics, 2002, 2(1): 29-35. DOI:10.1023/A:1013203532303 |
| [41] | Klammt C, L?hr F, Sch?fer B, et al. High level cell-free expression and specific labeling of integral membrane proteins. Eur J Biochem, 2004, 271(3): 568-580. |
| [42] | Sullivan CJ, Pendleton ED, Sasmor HH, et al. A cell-free expression and purification process for rapid production of protein biologics. Biotechnol J, 2016, 11(2): 238-248. |
| [43] | Noren CJ, Anthony-Cahill SJ, Griffith MC, et al. A general method for site-specific incorporation of unnatural amino acids into proteins. Science, 1989, 244(4901): 182-188. DOI:10.1126/science.2649980 |
| [44] | Salehi ASM, Smith MT, Bennett AM, et al. Cell-free protein synthesis of a cytotoxic cancer therapeutic: Onconase production and a just-add-water cell-free system. Anal Chem, 2016, 11(2): 274-281. |
| [45] | Horwitz MA, Harth G. A new vaccine against tuberculosis affords greater survival after challenge than the current vaccine in the guinea pig model of pulmonary tuberculosis. Infect Immun, 2003, 71(4): 1672-1679. DOI:10.1128/IAI.71.4.1672-1679.2003 |
| [46] | Vaughan A, Wang R, Kappe SHI. Genetically engineered, attenuated whole-cell vaccine approaches for malaria. Hum Vaccines, 2010, 6(1): 107-113. DOI:10.4161/hv.6.1.9654 |
| [47] | Wadle A, Mischo A, Strahl S, et al. NY-ESO-1 protein glycosylated by yeast induces enhanced immune responses. Yeast, 2010, 27(11): 919-931. DOI:10.1002/yea.1796 |
| [48] | Zaffran M, Vandelaer J, Kristensen D, et al. The imperative for stronger vaccine supply and logistics systems. Vaccine, 2013, 31: B73-B80. DOI:10.1016/j.vaccine.2012.11.036 |
| [49] | Kanter G, Yang J, Voloshin A, et al. Cell-free production of scFv fusion proteins: an efficient approach for personalized lymphoma vaccines. Blood, 2007, 109(8): 3393-3399. DOI:10.1182/blood-2006-07-030593 |
| [50] | Lu Y, Welsh JP, Swartz JR. Production and stabilization of the trimeric influenza hemagglutinin stem domain for potentially broadly protective influenza vaccines. Proc Natl Acad Sci USA, 2014, 111(1): 125-130. DOI:10.1073/pnas.1308701110 |
| [51] | Tsuboi T, Takeo S, Iriko H, et al. Wheat germ cell-free system-based production of malaria proteins for discovery of novel vaccine candidates. Infect Immun, 2008, 76(4): 1702-1708. DOI:10.1128/IAI.01539-07 |
| [52] | Padlan EA. Anatomy of the antibody molecule. Molecular Immunol, 1994, 31(3): 169-217. |
| [53] | Lerner RA. Tapping the immunological repertoire to produce antibodies of predetermined specificity. Nature, 1982, 299(5884): 592. DOI:10.1038/299592a0 |
| [54] | Birch JR, Racher AJ. Antibody production. Adv Drug Delivery Rev, 2006, 58(5/6): 671-685. |
| [55] | Zimmerman ES, Heibeck TH, Gill A, et al. Production of site-specific antibody-drug conjugates using optimized non-natural amino acids in a cell-free expression system. Nat Rev Microbiol, 2014, 25(2): 351-361. |
| [56] | Patel KG, Ng PP, Kuo CC, et al. Cell-free production of Gaussia princeps luciferase-antibody fragment bioconjugates for ex vivo detection of tumor cells. Biochem Biophys Res Commun, 2009, 390(3): 971-976. DOI:10.1016/j.bbrc.2009.10.087 |
| [57] | Ryabova LA, Desplancq D, Spirin AS, et al. Functional antibody production using cell-free translation: effects of protein disulfide isomerase and chaperones. Nat Biotechnol, 1997, 15(15): 79-84. |
| [58] | Merk H, Stiege W, Tsumoto K, et al. Cell-free expression of two single-chain monoclonal antibodies against lysozyme: effect of domain arrangement on the expression. J Biochem, 1999, 125(2): 328-333. DOI:10.1093/oxfordjournals.jbchem.a022290 |
| [59] | Jiang XP, Ookubo Y, Fujii I, et al. Expression of Fab fragment of catalytic antibody 6D9 in an Escherichia coli in vitro coupled transcription/translation system. FEBS Lett, 2002, 514(2/3): 290-294. |
| [60] | Hillebrecht JR, Chong S. A comparative study of protein synthesis in in vitro systems: from the prokaryotic reconstituted to the eukaryotic extract-based. Title BMC Biotechnol, 2008, 8(1): 58. DOI:10.1186/1472-6750-8-58 |
| [61] | Tarui H, Murata M, Tani I, et al. Establishment and characterization of cell-free translation/glycosylation in insect cell (Spodoptera frugiperda 21) extract prepared with high pressure treatment. Appl Microbiol Biotechnol, 2001, 55(4): 446-453. DOI:10.1007/s002530000534 |
| [62] | Stech M, Nikolaeva O, Thoring L, et al. Cell-free synthesis of functional antibodies using a coupled in vitro transcription-translation system based on CHO cell lysates. Sci Rep, 2017, 7(1): 12030. |
| [63] | Takeda H, Ogasawara T, Ozawa T, et al. Production of monoclonal antibodies against GPCR using cell-free synthesized GPCR antigen and biotinylated liposome-based interaction assay. Sci Rep, 2015, 5: 11333. DOI:10.1038/srep11333 |
| [64] | Xu Y, Lee J, Tran C, et al. Production of bispecific antibodies in "knobs-into-holes" using a cell-free expression system. Taylor & Francis, 2015, 231-242. |
| [65] | Zasloff M. Antimicrobial peptides of multicellular organisms. Nature, 2002, 415(6870): 389. DOI:10.1038/415389a |
| [66] | Reddy KVR, Yedery RD, Aranha C. Aranha. Antimicrobial peptides: premises and promises. Int J Antimicrob Agents, 2004, 24(6): 536-547. DOI:10.1016/j.ijantimicag.2004.09.005 |
| [67] | Li Y. Recombinant production of antimicrobial peptides in Escherichia coli: a review. Protein Expression Purif, 2011, 80(2): 260-267. DOI:10.1016/j.pep.2011.08.001 |
| [68] | Chen H, Xu Z, Xu N, et al. Efficient production of a soluble fusion protein containing human beta-defensin-2 in E. coli cell-free system. J Biotechnol, 2005, 115(3): 307-315. |
| [69] | Taniguchi M, Ochiai A, Kondo H, et al. Pyrrhocoricin, a proline-rich antimicrobial peptide derived from insect, inhibits the translation process in the cell-free Escherichia coli protein synthesis system. J Biosci Bioeng, 2016, 121(5): 591-598. DOI:10.1016/j.jbiosc.2015.09.002 |
| [70] | Galarza JM, Latham T, Cupo A. Virus-like particle (VLP) vaccine conferred complete protection against a lethal influenza virus challenge. Viral Immunol, 2005, 18(1): 244-251. |
| [71] | Rold?o A, Mellado MCM, Castilho LR, et al. Virus-like particles in vaccine development. Title Expert Rev Vaccines, 2010, 9(10): 1149-1176. DOI:10.1586/erv.10.115 |
| [72] | Cruz PE, Cunha A, Peixoto CC, et al. Optimization of the production of virus-like particles in insect cells. Abbreviated Title Biotechnol Bioeng, 1998, 60(4): 408-418. DOI:10.1002/(SICI)1097-0290(19981120)60:4<408::AID-BIT2>3.0.CO;2-Q |
| [73] | Maranga L, Braz?o TF, Carrondo MJT. Virus-like particle production at low multiplicities of infection with the baculovirus insect cell system. Biotechnol Bioeng, 2003, 84(2): 245-253. |
| [74] | Hopper JE, Bostian KA, Rowe LB, et al. Translation of the L-species dsRNA genome of the killer-associated virus-like particles of Saccharomyces cerevisiae. Title J Biol Chem, 1977, 252(24): 9010-9017. |
| [75] | Hodgman CE, Jewett MC. Cell-free synthetic biology: thinking outside the cell. Metab Eng, 2012, 14(3): 261-269. |
| [76] | Smith MT, Varner CT, Bush DB, et al. The incorporation of the A2 protein to produce novel Qβ virus-like particles using cell-free protein synthesis. Biotechnol Progr, 2012, 28(2): 549-555. DOI:10.1002/btpr.744 |
| [77] | Cerqueira C, Pang YYS, Day PM, et al. A cell-free assembly system for generating infectious human papillomavirus 16 capsids implicates a size discrimination mechanism for preferential viral genome packaging. J Virol, 2016, 90(2): 1096-1107. DOI:10.1128/JVI.02497-15 |
| [78] | Bundy BC, Franciszkowicz MJ, Swartz JR. Escherichia coli-based cell-free synthesis of virus-like particles. Biotechnol Bioeng, 2008, 100(1): 28-37. |
| [79] | Sheng J, Lei S, Lu Y, et al. Cell-free protein synthesis of norovirus virus-like particles. RSC Adv, 2017, 7(46): 28837-28840. DOI:10.1039/C7RA03742B |
| [80] | Chien MH, Wu SY, Lin CH. Oral immunization with cell-free self-assembly virus-like particles against orange-spotted grouper nervous necrosis virus in grouper larvae, Epinephelus coioides. Vet Immunol. Immunopathol, 2018, 197: 69–75. |
| [81] | Garmann RF, Knobler CM, Gelbart WM. Protocol for efficient cell-free synthesis of cowpea chlorotic mottle virus-like particles containing heterologous RNAs. New York NY, 2018, 249-265. |
| [82] | Mann M, Jensen ON. Proteomic analysis of post-translational modifications. Nat Biotechnol, 2003, 21(3): 255-261. DOI:10.1038/nbt0303-255 |
| [83] | Foley TL, Burkart MD. Site-specific protein modification: advances and applications. Curr Opin Chem Biol, 2007, 11(1): 12-19. DOI:10.1016/j.cbpa.2006.11.036 |
| [84] | Goerke AR, Swartz JR. High-level cell-free synthesis yields of proteins containing site-specific non-natural amino acids. Biotechnol Bioeng, 2009, 102(2): 400-416. DOI:10.1002/bit.22070 |
| [85] | Zemella A, Thoring L, Hoffmeister C, et al. Cell-free protein synthesis: Pros and cons of prokaryotic and eukaryotic systems. Chem Bio Chem, 2015, 16(17): 2420-2431. DOI:10.1002/cbic.201500340 |
| [86] | Tarui H, Imanishi S, Hara T. A novel cell-free translation/glycosylation system prepared from insect cells. J Biosci Bioeng, 2000, 90(5): 508-514. DOI:10.1016/S1389-1723(01)80031-1 |
| [87] | Guarino C, DeLisa MP. A prokaryote-based cell-free translation system that efficiently synthesizes glycoproteins. Glycobiology, 2011, 22(5): 596-601. |
| [88] | Jaroentomeechai T, Stark JC, Natarajan A, et al. Single-pot glycoprotein biosynthesis using a cell-free transcription-translation system enriched with glycosylation machinery. Nat. Commun, 2018, 9(1): 2686. |
| [89] | Gurramkonda C, Rao A, Borhani S, et al. Improving the recombinant human erythropoietin glycosylation using microsome supplementation in CHO cell-free system. Biotechnol Bioeng, 2018, 115(5): 1253-1264. DOI:10.1002/bit.26554 |
| [90] | Oza JP, Aerni HR, Pirman NL, et al. Robust production of recombinant phosphoproteins using cell-free protein synthesis. Nat Commun, 2015, 6: 8168. DOI:10.1038/ncomms9168 |
| [91] | Matsuda T, Watanabe S, Kigawa T. Cell-free synthesis system suitable for disulfide-containing proteins. J Struct Funct Genomics, 2013, 431(2): 296-301. |
| [92] | Matsuoka K, Komori H, Nose M, et al. Simple screening method for autoantigen proteins using the N-terminal biotinylated protein library produced by wheat cell-free synthesis. J Proteome Res, 2010, 9(8): 4264-4273. DOI:10.1021/pr9010553 |
| [93] | Ugwumba IN, Ozawa K, de la Cruz L, et al. Using a genetically encoded fluorescent amino acid as a site-specific probe to detect binding of low-molecular- weight compounds. Assay Drug Dev Technol, 2011, 9(1): 50-57. DOI:10.1089/adt.2010.0306 |
| [94] | Wang L. Genetically encoding new bioreactivity. New Biotechnol, 2017, 38: 16-25. DOI:10.1016/j.nbt.2016.10.003 |
| [95] | Gao W, Bu N, Lu Y. Recent advances in cell-free unnatural protein synthesis. Chin J Biotech, 2018, 34(9): 1371-1385 (in Chinese). 高伟, 卜宁, 卢元. 无细胞体系非天然蛋白质合成研究进展. 生物工程学报, 2018, 34(9): 1371-1385. |
| [96] | Albayrak C, Swartz JR. Cell-free co-production of an orthogonal transfer RNA activates efficient site-specific non-natural amino acid incorporation. Nucl Acids Res, 2013, 41(11): 5949-5963. DOI:10.1093/nar/gkt226 |
| [97] | Br?cker MJ, Ho J ML, Church GM, et al. Recoding the genetic code with selenocysteine. Angew Chem, 2014, 53(1): 319-323. |
| [98] | Katoh T, Tajima K, Suga H. Consecutive elongation of D-amino acids in translation. Cell Chem Biol, 2017, 24(1): 46-54. DOI:10.1016/j.chembiol.2016.11.012 |
| [99] | Yang YH, Kim TW, Park SH, et al. Cell-free Escherichia coli-based system to screen for quorum-sensing molecules interacting with quorum receptor proteins of Streptomyces coelicolor. Appl Environ Microbiol, 2009, 75(19): 6367-6372. DOI:10.1128/AEM.00019-09 |
| [100] | Turner APF. Biosensors: sense and sensibility. Chemical Society Reviews, 2013, 42(8): 3184-3196. DOI:10.1039/c3cs35528d |
| [101] | Zhu C, Yang G, Li H, et al. Electrochemical sensors and biosensors based on nanomaterials and nanostructures. Anal Chem, 2014, 87(1): 230-249. |
| [102] | Lafleur JP, Joensson A, Senkbeil S, et al. Recent advances in lab-on-a-chip for biosensing applications. Biosens Bioelectron, 2016, 76: 213-233. DOI:10.1016/j.bios.2015.08.003 |
| [103] | Jang YJ, Lee KH, Yoo TH, et al. Complementary cell-free translational assay for quantification of amino acids. Anal Chem, 2017, 89(18): 9638-9642. DOI:10.1021/acs.analchem.7b01956 |
| [104] | Salehi ASM, Yang SO, Earl CC, et al. Biosensing estrogenic endocrine disruptors in human blood and urine: a RAPID cell-free protein synthesis approach. Toxicol Appl Pharmacol, 2018, 345: 19-25. DOI:10.1016/j.taap.2018.02.016 |
| [105] | Kaiser L, Graveland-Bikker J, Steuerwald D, et al. Efficient cell-free production of olfactory receptors: detergent optimization, structure, and ligand binding analyses. Proc Natl Acad Sci USA, 2008, 105(41): 15726-15731. DOI:10.1073/pnas.0804766105 |
| [106] | Zhang P, Feng H, Yang J, et al. Detection of inorganic ions and organic molecules with cell-free biosensing systems. J Biotechnol, 2019. |
| [107] | Davies DH, Liang X, Hernandez JE, et al. Profiling the humoral immune response to infection by using proteome microarrays: high-throughput vaccine and diagnostic antigen discovery. Proc Natl Acad Sci USA, 2005, 102(3): 547-552. DOI:10.1073/pnas.0408782102 |
| [108] | Salehi ASM, Shakalli Tang MJ, Smith MT, et al. Cell-free protein synthesis approach to biosensing hTRβ-specific endocrine disruptors. Anal Chem, 2017, 89(6): 3395-3401. DOI:10.1021/acs.analchem.6b04034 |
| [109] | Kawaguchi T, Chen YP, Norman RS, et al. Rapid screening of quorum-sensing signal N-acyl homoserine lactones by an in vitro cell-free assay. Appl Environ Microbiol, 2008, 74(12): 3667-3671. DOI:10.1128/AEM.02869-07 |
| [110] | Endo Y, Sawasaki T. High-throughput, genome-scale protein production method based on the wheat germ cell-free expression system. Biotechnol Adv, 2003, 21(8): 695-713. DOI:10.1016/S0734-9750(03)00105-8 |
| [111] | Smith MT, Berkheimer SD, Werner CJ, et al. Lyophilized Escherichia coli-based cell-free systems for robust, high-density, long-term storage. J. Bioprocess Biotechnol, 2014, 56(4): 186-193. |
| [112] | Pardee K, Green AA, Ferrante T, et al. based synthetic gene networks. Cell, 2014, 159(4): 940-954. DOI:10.1016/j.cell.2014.10.004 |
| [113] | Pardee K, Green AA, Takahashi MK, et al. Rapid, low-cost detection of Zika virus using programmable biomolecular components. Cell, 2016, 165(5): 1255-1266. DOI:10.1016/j.cell.2016.04.059 |
| [114] | Ma D, Shen L, Wu K, et al. Low-cost detection of norovirus using paper-based cell-free systems and synbody-based viral enrichment. Synth Biol, 2018, 3(1): ysy018. |
| [115] | Jang YJ, Lee KH, Yoo TH, et al. Interfacing a personal glucose meter with cell-free protein synthesis for rapid analysis of amino acids. Anal Chem, 2019, |
| [116] | Gr?we A, Dreyer A, Vornholt T, et al. A paper-based, cell-free biosensor system for the detection of heavy metals and date rape drugs. PLoS ONE, 2019, 14(3): e0210940. DOI:10.1371/journal.pone.0210940 |
