雍康1,2, 罗正中2, 骆巧3, 杨庆稳2, 张传师2, 张勇1

 , 曹随忠3
, 曹随忠3

1. 甘肃农业大学动物医学院, 甘肃 兰州 730070;
2. 重庆三峡职业学院动物科技学院, 重庆 万州 404155;
3. 四川农业大学动物医学院, 四川 成都 611130
收稿日期:2020-09-13;修回日期:2020-10-29;网络出版日期:2020-11-11
基金项目:甘肃省引导科技创新专项(GSCXZX-2019-1);甘肃省教育厅高校科技项目(2018C-15)
*通信作者:张勇, Tel/Fax: +86-931-7631229, E-mail: zhangyong@gsau.edu.cn;
曹随忠, Tel/Fax: +86-28-86296382, E-mail: suizhongcao@126.com.
摘要:[目的] 本试验旨在测定产后健康奶牛和罹患真胃左方变位(left displacement of the abomasum,LDA)奶牛粪便中微生物菌群的变化,以期探讨LDA发生与菌群的关联性,评估其对机体代谢的潜在影响。[方法] 采用16S rDNA高通量测序技术,测定10头健康奶牛(Health)、10头真胃左方变位奶牛(LDA)粪便中微生物16S rDNA V3-V4区序列,并对菌群群落结构和多样性进行比较,分析其与LDA发生的相关性。[结果] 多样性分析表明,Health组与LDA组奶牛的粪便中微生物多样性和群落组成存在较大的差异,其中LDA奶牛粪便微生物具有较高的物种丰度和种群差异性。对门、科、属3个分类水平上最大丰度排名前10的物种进行分析发现,LDA奶牛疣微菌门、蓝菌门、变形菌门、梭杆菌门、拟杆菌科、p-2534-18B5菌科、艰难杆菌科、梭杆菌科、螺旋菌属、5-7N15菌属和梭杆菌属的丰度显著升高(P < 0.05),而螺旋体门、TM7菌门、瘤胃球菌科、理研菌科、密螺旋体属的丰度显著下降(P < 0.05)。功能预测分析表明,LDA奶牛碳水化合物代谢和脂质代谢通路相关功能基因显著上调,而遗传信息处理相关功能基因显著下调。[结论] 本试验研究了健康奶牛和LDA奶牛粪便微生物的变化,为LDA的致病机理、早期诊断提供了理论依据与研究基础。
关键词:奶牛LDA粪便微生物16S rDNA高通量测序技术
Revealing the impact of left displacement of the abomasum on fecal microbes of dairy cows by 16S rDNA amplicon sequencing technology
Kang Yong1,2, Zhengzhong Luo2, Qiao Luo3, Qingwen Yang2, Chuanshi Zhang2, Yong Zhang1

 , Suizhong Cao3
, Suizhong Cao3

1. College of Veterinary Medicine, Gansu Agricultural University, Lanzhou 730070, Gansu Province, China;
2. College of Animal Science and Technology, Chongqing Three Gorges Vocational College, Chongqing 404100, China;
3. College of Veterinary Medicine, Sichuan Agricultural University, Chengdu 611130, Sichuan Province, China
Received: 13 September 2020; Revised: 29 October 2020; Published online: 11 November 2020
*Corresponding author: Yong Zhang, Tel/Fax: +86-931-7631229; E-mail: zhangyong@gsau.edu.cn;
Suizhong Cao, Tel/Fax: +86-28-86296382; E-mail: suizhongcao@126.com.
Foundation item: Supported by the Gansu Flying Scholar Special Appointment Professor Plan and Scientific Research Projects of Colleges (GSCXZX-2019-1) and by the Universities of Gansu Provincial Education Department (2018C-15)
Abstract: [Objective] Our study aims to measure the changes of microbial flora in feces of healthy postpartum cows and cows suffering from left displacement of the abomasum (LDA), so as to explore the relevance between LDA occurrence and the flora, and assess its potential impact on body metabolism. [Methods] We used 16S rDNA High-Throughput Sequencing technology to determine the 16S rDNA V3-V4 region sequence of microorganisms in the feces of 10 healthy cows and 10 cows with LDA. [Results] The diversity analysis indicated that there were significant differences in microbial diversity and community composition between healthy group and LDA group, and the fecal microbes of the LDA cows had higher species abundance and population differences. The analysis of the top 10 species with the largest abundance at the level of phyla, family, and genus showed that the abundances of Verrucomicrobia, Cyanobacteria, Proteobacteria, Fusobacteria, Bacteroides, p-2534-18B5, Mogibacteriaceae, Fusobacteriaceae, Oscillospira, 5-7N15 and Faecococcus in LDA cows increased significantly (P < 0.05), while the abundances of Spirochetes, TM7, Ruminococcaceae, Rikenellaceae and Treponema decreased significantly (P < 0.05). Functional prediction analysis showed that the functional genes related to carbohydrate metabolism and lipid metabolism pathway in cows with LDA were greatly up-regulated, while those related to genetic information processing were significantly down regulated. [Conclusion] Our findings confirm that the changes of fecal microorganisms in dairy cows before and after LDA, and provide a foundation for a deeper understanding of the pathogenesis and early diagnosis of LDA.
Keywords: dairy cowsLDAfecal microorganisms16S rDNA high-throughput sequencing
真胃左方变位(left displacement of the abomasum,LDA)是一种产后高产奶牛的常发疾病,主要发生在泌乳期的第1个月[1]。每头LDA患病奶牛会给牧场带来超过700美元的经济损失,包括直接损失(矫正、用药、牛奶丢弃等)和间接损失(生产和繁殖性能下降、淘汰率增加等),且这一损失随着LDA发病率增加而增加[2-3]。LDA可根据产后奶牛食欲减退、产奶量下降、在左腹部叩诊配合听诊出现钢管音进行诊断[1]。
动物肠道微生态系统与动物体的健康状况、营养代谢、免疫功能、疾病发生等密切相关[4-5]。动物肠道菌群参与了宿主多种代谢途径的调控,产生了相互作用的宿主和微生物代谢轴、宿主和微生物信号轴及免疫炎症轴,这些生理轴连接着肠道、肝脏、肌肉和大脑等多个机体器官[6]。前期研究表明,肠道菌群失调可能是引起奶牛乳腺炎的原因之一[7],且酮病奶牛与健康奶牛肠道微生物区系存在显著差异[8]。乳房炎、酮病、LDA多发生在奶牛围产期,在致病机理方面可互为因果[3, 9]。目前,有关肠道微生物在LDA的致病方面的研究报道较少。为了更深入了解罹患LDA奶牛肠道菌群结构的变化情况,本试验采用Illumina MiSeq测序平台,分别测定健康和真胃左方变位奶牛粪便中微生物16S rDNA V3–V4区序列,并对其群落结构和多样性进行比较分析,以期为揭示LDA的发病机理提供数据支撑。
1 材料和方法 1.1 试验动物的选择 本试验于2020年1月至2020年3月在四川某奶牛场进行,选择泌乳天数、胎次、体重、月龄相近的健康奶牛(Health组)和真胃左方变位奶牛(LDA组)各10头(表 1),LDA的诊断方法参考“Diagnosis, treatment and control of left displaced abomasum in cattle”[1]。所有奶牛统一饲养管理,奶牛日粮组成和营养成分见表 2。
表 1. 各组奶牛的胎次、月龄和泌乳天数 Table 1. Parity, month age and days in milk of cows in each group
| Groups | Quantity/head | Parity | Month age | Days in milk |
| Health | 10 | 2.00±0.71 | 33.78±6.48 | 18.22±1.99 |
| LDA | 10 | 1.89±0.79 | 32.67±7.71 | 17.78±3.38 |
表选项
表 2. 围产期奶牛日粮组成和营养成分(干物质基础,%) Table 2. Composition and nutrition levels of the diets for transition dairy cows (DM basis, %)
| Item | Peri-parturient period | Post-parturient period |
| Ingredients | ||
| Corn silage | 20.50 | 16.52 |
| Corn | 8.30 | 20.14 |
| Soybean meal | 12.50 | 4.86 |
| Wheat meal | 3.50 | 4.88 |
| Oat hay | 45.60 | – |
| Rapeseed meal | 3.60 | – |
| Cottonseed meal | 3.50 | – |
| Premix1) | 2.10 | – |
| Choline | 0.40 | – |
| Alfalfa hay | – | 22.63 |
| Premix2) | – | 0.60 |
| Extruded soybean | – | 7.54 |
| Brewer's grains | – | 5.51 |
| Cottonseed | – | 10.19 |
| Fat powder | – | 0.99 |
| NaCl | – | 0.74 |
| Calcium phosphate | – | 1.49 |
| Calcium powder | – | 0.59 |
| NaHCO3 | – | 0.79 |
| Molasses | – | 2.53 |
| Total | 100.00 | 100.00 |
| Nutrition levels3) | ||
| Dry matter | 50.17 | 50.36 |
| NEL (Mcal/kg) | 1.39 | 1.71 |
| Crude protein | 16.88 | 18.14 |
| Neutral detergent fiber | 42.44 | 30.3 |
| Acid detergent fiber | 26.87 | 20.68 |
| Calcium | 0.35 | 1.03 |
| Phosphorus | 0.36 | 0.69 |
| 1): The premix provided the following per kg of the diet: VA 600 KIU, VD3 150 KIU, VE 6600 IU, Cu 2525 mg, Fe 4150 mg, Mn 4200 mg, Zn 10025 mg, Co 60 mg, moisture < 10%. 2): The premix provided the following per kg of the diet: VA 1404 KIU, VD3 351 KIU, VE 7020 IU, biotin 140 mg, beta-carotene 23.4 mg, Cu 3240 mg, Zn 16200 mg, Mn 3240 mg, Se 95 mg, Co 48 mg, I 216 mg, moisture < 10%. 3): Net energy of lactation was calculated value, while the others were measured values. | ||
表选项
1.2 样本采集 直肠采集Health组和LDA组奶牛粪便样本5 g,装入冻存管,编号后立即投入液氮中保存待测。
1.3 样本检测 将样本送至上海中科新生命生物科技有限公司进行检测,测序过程如下:首先通过粪便组基因DNA提取试剂盒提取样本DNA,且在1%琼脂糖凝胶电泳检测合格后,使用通用引物338F (5′-ACTCCTACGGGAGGCAGCAG-3′)与806R (5′-GGACTACHVGGGTWTCTAAT-3′)对样本16S rDNA的V3–V4区域进行PCR扩增,并构建PE扩增文库。使用Illumina Miseq PE 2500平台进行测序。Miseq测序得到的双端测序数据去除barcode与primer拼接,并进一步去除嵌合体、短序列得到clean tags。在97%相似度条件下,利用qiime (v1.8.0)软件进行可分类操作单元(operational taxonomic units,OTUs)聚类和物种注释。将OUTs代表序列与相应微生物数据库进行比对,得到各样本的物种分类信息与各水平注释信息。同时,获得基于聚类结果的多样性分析与基于注释结果的各分类水平物种组成信息。
1.4 数据处理和分析 采用相似性分析(ANOSIM)方法计算组间未加权Unifrac距离;采用单向方差分析(ANOVA)比较Alpha多样性;采用SPSS 19.0统计软件比较奶牛肠道微生物不同分类水平组成;采用STAMP软件检测不同丰度的KEGG;上述分析作图软件为“vegan” R package;P < 0.05表示差异显著,P < 0.01表示差异极显著。
2 结果和分析 2.1 粪便细菌微生物序列及多样性分析
2.1.1 数据预处理统计及质控: 本试验共获得原始Tags序列1803187条,除去低质量及嵌合体后,最终用于后续分析的有效Tags序列为1401000条,平均每个样本得到70050条高质量序列,其中Q20和Q30平均值分别为98%和93%。本次检测的样本序列数以及质量良好,满足后续分析要求。
2.1.2 各组样品中物种的均匀度和丰富度: 稀释曲线可直接反映测序数据量的合理性,也可间接反映样品中物种的丰富程度;当曲线趋向平坦时,表明测序数据量合理。由图 1-A可见,随着有效序列测序深度的增加,稀释曲线呈现先急速上升随后趋于平缓,表明样本测序数据量合理,测序质量良好,具有一定的深度和代表性。Rank-abundance曲线可用来解释多样性的2个方面,即物种丰度和物种均匀度。图 1-B显示,随着Rank值的增加,丰度减少的趋势逐渐趋于平稳,表明样品中物种的均匀度和丰富度较好。
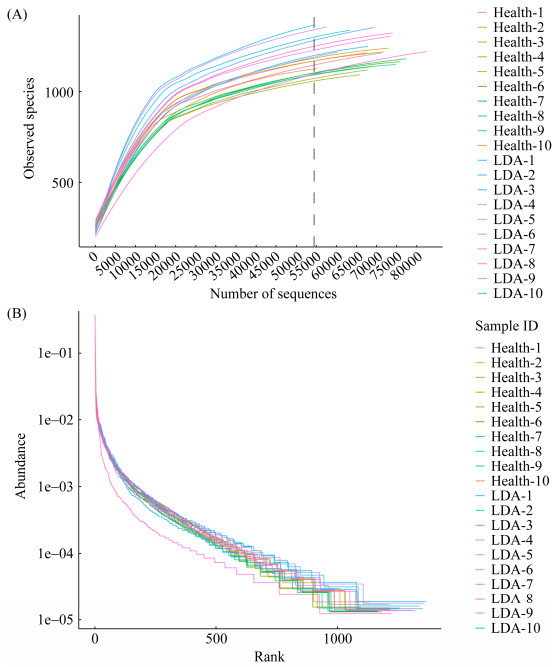 |
| 图 1 样品中物种的均匀度和丰富度分析 Figure 1 Analysis of uniformity and richness of species in samples. A: rarefaction curve can directly reflect the rationality of the amount of sequencing data and indirectly reflect the abundance of species in the sample; B: rank-abundance curve can be used to explain species abundance and species uniformity. |
| 图选项 |
2.1.3 细菌微生物OTUs比较: Illumina MiSeq平台测序结果获得后,对所有序列按97%的相似度进行可分类操作单元聚类,对样品进行OTUs统计后,Health组、LDA组分别共获得1765个、1951个OTUs,Health组和LDA组样品共享OTUs为1681个(图 2)。
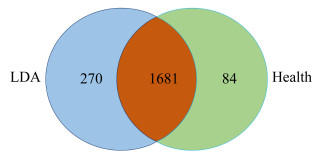 |
| 图 2 Health组和LDA组奶牛粪便微生物基因OTUs分布结果 Figure 2 OTUs distribution results of fecal microbial genes in Health group and LDA group. |
| 图选项 |
2.1.4 微生物群落Alpha多样性分析: Alpha多样性用于分析样品内的微生物群落多样性。Shannon和Simpson为菌群多样性指数;Chao1和ACE为菌群丰度指数。由图 3可知,LDA组的Chao1、ACE、Shannon、Simpson指数均高于Health组,差异极显著(P < 0.01)。
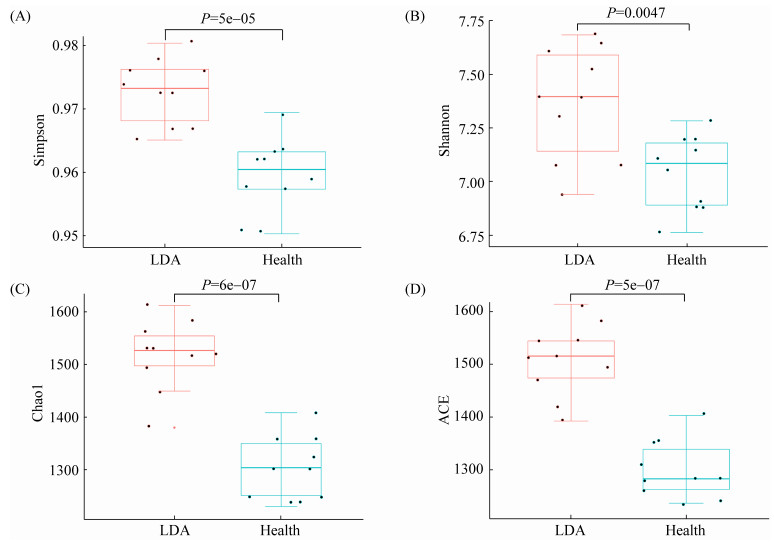 |
| 图 3 Health组和LDA组奶牛粪便微生物Alpha多样性指数 Figure 3 Alpha diversity index of fecal microorganisms in Health group and LDA group. A–D: boxplots of bacterial alpha diversities evaluated by Simpson's diversity index, Shannon diversity index, Chao1 index and ACE index. Different numbers in the picture denoted a P value of different alpha diversity index among groups; each boxplot represent the median, interquartile range, minimum, and maximum values. |
| 图选项 |
2.1.5 微生物群落Beta多样性分析: Beta多样性主要分析样本与样本间微生物群落组成的相似性,本试验通过非度量多维尺度法(non-metric multidimensional scaling,NMDS)与主坐标分析(principal co-ordinates analysis,PCoA)用以分析样本Beta多样性。由图 4可直观看到,两组粪便微生物结构组成有差异。基于Bray-Curtis算法,Adonis分析显示各组肠道菌群的组间差异大于组内差异,分析结果如图 5所示。
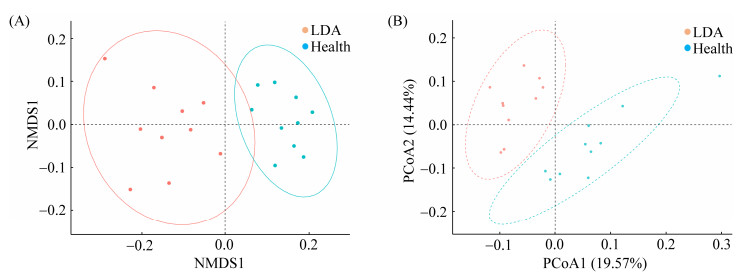 |
| 图 4 Health组和LDA组奶牛粪便微生物Beta多样性指数 Figure 4 Beta diversity index of fecal microorganisms in Health group and LDA group. A: scatterplot from NMDS analysis based on Bray-Curtis and unweight UniFrac distance in bacterial communities; B: scatterplot from PCoA analysis based on Bray-Curtis and unweight UniFrac distance in bacterial communities. |
| 图选项 |
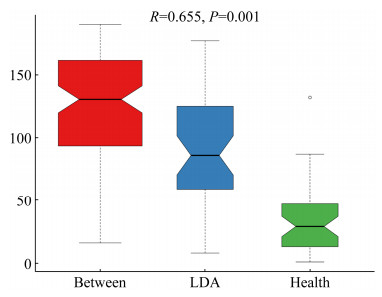 |
| 图 5 Anosim分析各组间群落结构差异 Figure 5 Anosim analyzes the differences in community structure between groups. Between is the rank obtained by sorting the distance values between the two sets of samples. The R value is between (–1, 1), and the R value is greater than 0, indicating that the difference between groups is significant, and P < 0.05 indicates statistical significance. |
| 图选项 |
2.2 Health组和LDA组粪便微生物结构组成分析 在不同分类水平上对测序所得有效序列进行物种注释,共得到19个门、36个纲、57个目、87个科、137个属。以下主要从门、科、属这3个分类水平上最大丰度排名前10的物种进行分析。
2.2.1 门分类学水平: 基于门分类水平研究发现(图 6-A),厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)为各组粪便中检测到的主要菌门,其中厚壁菌门相对丰度最高(Health组:73.98%,LDA组:72.99%);接着是拟杆菌门,其相对丰度在Health组中为21.18%,LDA组中为21.80%。软壁菌门(Tenericutes)、变形菌门(Proteobacteria)、螺旋体门(Spirochaetes)、疣微菌门(Verrucomicrobia)、放线菌门(Actinobacteria)、蓝菌门(Cyanobacteria)、TM7菌门、梭杆菌门(Fusobacteria)在Health组的相对丰度分别为2.30%、0.58%、0.93%、0.06%、0.37%、0.04%、0.21%、0.03%,在LDA组的相对丰度分别为1.61%、1.51%、0.39%、0.95%、0.07%、0.27%、0.07%、0.14%。与Health组相比,LDA组中疣微菌门、蓝菌门、梭杆菌门、变形菌门的丰度显著升高(P < 0.05),螺旋体门、TM7菌门的丰度显著下降(P < 0.05)。
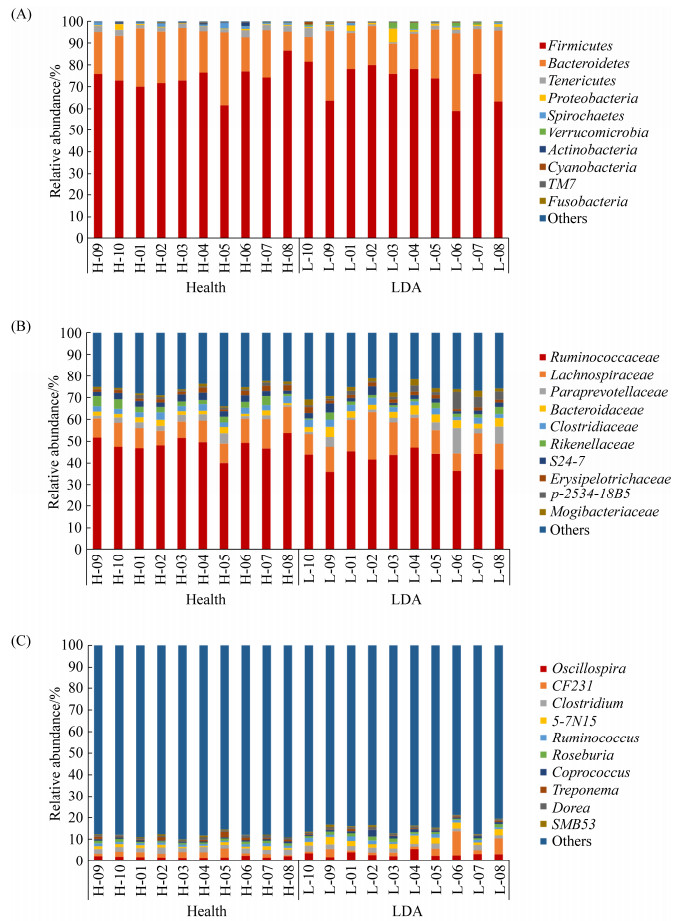 |
| 图 6 Health组和LDA组基于不同分类学水平物种组成分析柱状图 Figure 6 Taxonomic compositions of gut microbial communities in different levels from Health groups and LDA groups. A: taxonomic distribution of gut microbial communities from each sample at the phylum level; B: taxonomic distribution of gut microbial communities from each sample at the family level; C: taxonomic distribution of gut microbial communities from each sample at the genus level. |
| 图选项 |
2.2.2 科分类学水平: 图 6-B展示了相对含量排名前10的菌科。瘤胃球菌科(Ruminococcaceae)相对丰度最高(Health组:48.46%,LDA组:41.95%)。毛螺菌科(Lachnospiraceae)、帕拉普氏菌科(Paraprevotellaceae)、拟杆菌科(Bacteroidaceae)、梭菌科(Clostridiaceae)、理研菌科(Rikenellaceae)、S24-7菌科、丹毒丝菌科(Erysipelotrichaceae)、p-2534-18B5菌科、艰难杆菌科(Mogibacteriaceae)在Health组分别为9.86%、2.35%、1.87%、2.64%、2.87%、2.51%、1.67%、0.76%、0.87%,在LDA组分别为12.56%、3.70%、3.19%、2.59%、1.74%、2.20%、1.55%、2.62%、1.92%。与Health相比,LDA组中拟杆菌科、p-2534-18B5菌科、艰难杆菌科的丰度显著升高(P < 0.05),瘤胃球菌科、理研菌科的丰度显著下降(P < 0.05)。
2.2.3 属分类学水平: 菌属水平研究显示(图 6-C),Health组和LDA组分别有87.93%、83.81%的序列reads未被分类。检测到丰度排名前10的菌属分别为:颤螺旋菌属(Oscillospira) (Health组:2.01%,LDA组:3.31%)、CF231菌属(Health组:2.05%,LDA组:3.28%)、梭菌属(Clostridium) (Health组:2.16%,LDA组:1.99%)、5-7N15菌属(Health组:1.10%,LDA组:2.42%)、瘤胃球菌属(Ruminococcus) (Health组:1.03%,LDA组:1.32%)、罗氏菌属(Roseburia) (Health组:0.95%,LDA组:1.16%)、粪球菌属(Coprococcus) (Health组:0.61%,LDA组:1.11%)、密螺旋体属(Treponema) (Health组:0.93%,LDA组:0.39%)、Dorea菌属(Health组:0.87%,LDA组:0.76%)、SMB53菌属(Health组:0.36%,LDA组:0.46%)。与Health相比,LDA组中颤螺旋菌属、5-7N15菌属的丰度显著升高(P < 0.01),密螺旋体属的丰度显著下降(P < 0.05)。
2.3 Health组和LDA组粪便微生物结构差异性分析 通过线性判别分析(linear discriminant analysis,LDA)方法分析多级物种的差异(图 7-A),试图发现健康奶牛和LDA奶牛差异显著物种。结合LDA效应值(linear discriminant analysis effect sizes,LEfSe)与P value比较各组粪便微生物物种丰度对差异效果影响大小,筛选出差异物种(LDA score≥3,P < 0.05)。结果显示,LDA奶牛变形菌门、梭杆菌门、梭杆菌科、拟杆菌科、艰难杆菌科、梭杆菌属、5-7N15菌属和颤螺旋菌属相对丰度显著增加;瘤胃球菌科、理研菌科的丰度显著下降。同时,由进化分支图(图 7-B)显示,发生显著变化的粪便微生物差异物种主要来源于梭杆菌门,以及拟杆菌科、理研菌科、瘤胃球菌科、艰难杆菌科、梭杆菌科这5个菌科。
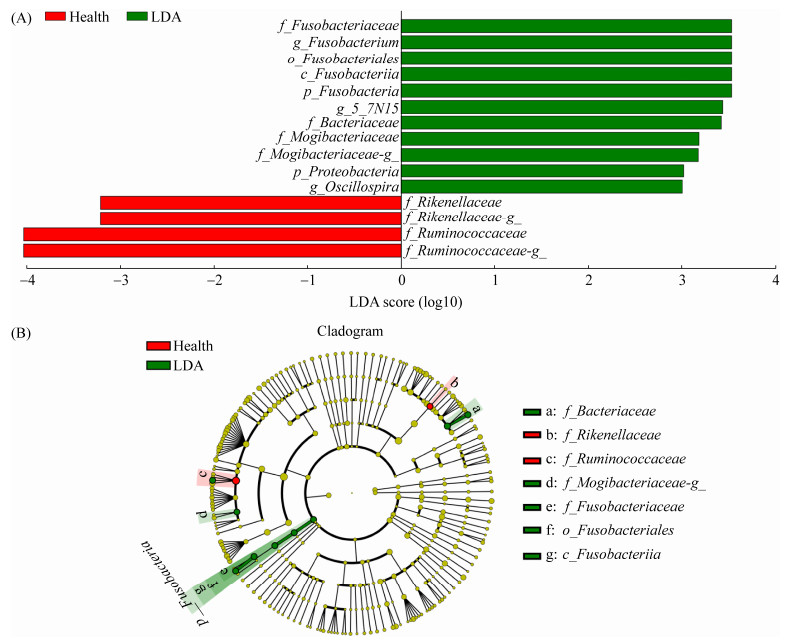 |
| 图 7 Health组和LDA组粪便微生物结构差异性分析 Figure 7 Analysis of the difference of fecal microbial communities between Health group and LDA group. A: linear discriminant analysis (LDA) effect size (LEfSe) analysis displayed significant bacterial differences in fecal microbial communities between Health group and LDA group. The LDA score (log10)≥3 and P < 0.05 are listed. B: cladogram made by LEfSe reflecting the phylogenetic distribution of fecal microbial communities between Health group and LDA group. |
| 图选项 |
2.4 奶牛罹患LDA后粪便中微生物菌群变化对机体代谢的影响 为进一步分析与真胃左方变位相关的差异菌群,本试验使用STAMP差异分析对属分类水平的显著差异菌群进行评估,筛选出28种菌属(图 8)。与健康组相比,LDA奶牛颤螺旋菌属、5-7N15菌属、阿克曼菌属(Akkermansia)、rc4-4菌属、普氏菌属(Prevotella)、考拉杆菌属(Phascolarctobacterium)、优杆菌属(Eubacterium)、Epulopiscium菌属、丁酸弧菌属(Butyrivibrio)、梭形杆菌属(Fusobacterium)、艰难杆菌属(Mogibacterium)、厌氧棒状菌属(Anaerostipes)、瘤胃杆菌属(Ruminobacter)、Moryella菌属、L7A_E11菌属、丁酸球菌属(Butyricicoccus)、低嗜盐细菌属(Dehalobacterium)、栖粪杆菌属(Faecalibacterium)、毛螺菌属(Lachnospira)、节杆菌属(Arthrobacter)、萨特菌属(Sutterella)、Trueperella菌属、奇异菌属(Atopobium)、脱硫弧菌属(Desulfovibrio)和施氏菌属(Schwartzia)的丰度显著升高(P < 0.05),厌氧原体属(Anaeroplasma)、甲烷短杆菌属(Methanobrevibacter)和RFN20菌属的丰度显著下降(P < 0.05)。
 |
| 图 8 Health组和LDA组粪便微生物在属水平上的STAMP差异分析 Figure 8 STAMP difference analysis of fecal microorganisms in the Health group and LDA group at the genus level. |
| 图选项 |
使用PICRUSt软件通过比对16S测序数据获得的菌群信息,推测样本中的功能基因组成,从而分析Health组和LDA组之间在功能上的差异。LEfSe分析根据分类学组成对样本按照不同的分组条件进行线型判别分析,找出对样本划分产生显著性差异影响的功能条目。基于KEGG功能预测的LEfSe分析结果表明,真位左方变位奶牛碳水化合物代谢(carbohydrate metabolism)和脂质代谢(lipid metabolism)通路相关功能基因显著上调,而遗传信息处理(genetic information processing)相关功能基因显著下调(图 9)。
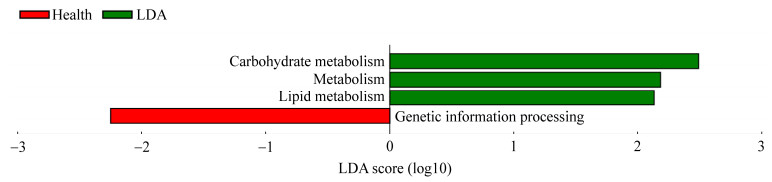 |
| 图 9 基于KEGG功能预测的LEfSe分析结果 Figure 9 LEfSe analysis results based on KEGG function prediction. The LDA score (log10)≥2 and P < 0.05 are listed. |
| 图选项 |
3 讨论 3.1 Health组和LDA组粪便中微生物群落的多样性比较 微生物群落的多样性往往由α多样性和β多样性进行评估。α多样性反映了样品中物种的多样性和丰富度,具体评价指标包括Chao1、ACE、Shannon和Simpsom指数。其中Chao1和ACE指数用于衡量物种丰富度即物种数量的多少;Shannon和Simpsom指数用于衡量物种多样性[10]。本研究为了解Health和LDA奶牛粪便微生物的多样性水平,对Chao1、ACE、Shannon和Simpsom指数进行计算评估。结果显示,LDA组的Chao1、ACE、Shannon、Simpson指数均高于Health组,表明LDA组奶牛粪便微生物群落丰富度和多样性均高于Health组。Song等[11]研究表明,与正常奶牛相比,患有LDA的荷斯坦奶牛的粪便微生物组成发生了变化,且多样性较低,这与本研究结果不同,有待进一步研究。微生物群落的β多样性主要用以评估样品间物种多样性差异,本研究中利用NMDS、PCoA法分析发现LDA组与Health组粪便微生物菌群能够明显地分开,说明这两者的菌群分别具有较高的稳定性且在微生物结构上具有较大差异。
3.2 Health组和LDA组粪便微生物在不同分类水平上的群落结构差异
3.2.1 门分类水平上的群落结构差异: 本试验结果显示,两组奶牛肠道内的优势菌群是厚壁菌门与拟杆菌门,占总菌群的90%以上,这与之前报道的一致[11-15]。两组奶牛肠道内优势菌群之间差异不显著(P > 0.05),其原因可能是奶牛消化道中存在核心菌群,且丰度始终稳定,一般不随日粮、生理时期和环境变化而改变[16]。厚壁菌门与拟杆菌门能消化动物所摄入的大部分碳水化合物与其他食物,对动物的营养代谢至关重要[17]。真胃运动主要受迷走神经控制,迷走神经功能障碍会降低真胃运动[9]。真胃运动迟缓已被证实为LDA的主要发病机制[9]。有研究报道,小鼠肠道神经系统与肠道菌群之间的相互作用有助于适当的胃肠蠕动[18]。小鼠肠道菌群能刺激迷走神经感觉神经元,可以将信息从胃肠道腔内物质传递到大脑,从而调节胃肠道的运动性[19-20]。研究发现,肠道微生物群落的失衡常源于变形菌门丰度的变化,因此,变形菌门可作为肠道菌群失调的微生物标志[21]。本试验通过LEfSe多级物种差异判别分析发现,LDA奶牛变形菌门相对丰度显著增加,说明LDA奶牛肠道微生物菌群已经紊乱。据此推断,失调的肠道菌群可能影响了奶牛胃肠道迷走神经的传导功能,引起真胃蠕动减弱,导致了LDA的发生。螺旋体门可以有效降解纤维素、果胶和磷酸酯,发酵形成挥发性脂肪酸,为动物机体提供能量[22]。Song等[11]报道健康奶牛和LDA奶牛螺旋体的比例在门水平上有显著差异(P < 0.01),在健康组的粪便中,平均有1.5%的菌群是螺旋体门而在LDA组的粪便中未检出螺旋体门。然而,本研究在LDA组的粪便中检测到了螺旋体门(Health组:0.93%,LDA组:0.39%,P < 0.01),这可能与饲喂饲料成分和检测方法不同有关。
3.2.2 属分类水平上的群落结构差异: 通过鉴定发病后发生变化的特定肠道菌株,不仅能为LDA提供早期诊断指标,还可以量身定制细菌混合物以调整肠道菌群,改善肠道微生态,达到早期预防的目的。本试验使用STAMP差异分析在属分类水平上筛选出了28种差异菌属。在所有检测到的菌属中,颤螺旋菌属丰度相对较高(Health组:2.01%,LDA组:3.31%),两组之间差异极显著(P < 0.01)。颤螺旋菌属多出现于草食动物的肠道,其通过利用动物来源的葡萄糖产生丁盐酸以供宿主的基本代谢[23]。有研究表明,大肠提供了一个类似于瘤胃-网胃的发酵活性部位[14]。粪便微生物中的一些细菌与纤维素和半纤维素降解有关,包括瘤胃球菌属、5-7N15菌属(拟杆菌科)、SMB53菌属(梭菌科)和梭菌属[14]。本试验显示,LDA组的5-7N15菌属和梭菌属显著升高(P < 0.05)。密螺旋体属被认为参与了精料的消化[24]。本试验中密螺旋体属丰度显著降低(P < 0.01),可能与奶牛罹患LDA后采食精料下降有关。LDA奶牛普氏菌属、丁酸弧菌属和丁酸球菌属的相对丰度较高。普氏菌属拥有能将半纤维素和淀粉降解成短链脂肪酸(short chain fatty acids, SCFAs) (如乙酸盐、丙酸盐、丁酸盐和琥珀酸盐)的酶[25-26]。丁酸弧菌属和丁酸球菌属的成员是丁酸盐生产者[27-28]。丁酸盐和丙酸盐是奶牛体内最重要的SCFAs。奶牛罹患LDA前可能经历了严重的能量负平衡和酮病,而产后碳水化合物摄入不足或代谢紊乱是发生严重能量负平衡和酮病的主要病因[3]。肠道内这些分解纤维素和产生SCFAs的菌属增加可能是“代偿性”的,以促进肠道内碳水化合物的充分利用,来补偿能量负平衡带来的危害,满足机体基本能量需要和产奶需求。与这一推断相对应的是Bo等[29]关于肠道菌群介导动物聚群行为节省能量的研究,他们发现,动物的聚群行为(适应寒冷)能够引起肠道菌群的丰富度和多样性升高,增加有益菌而降低有害菌的数量,提高代谢产物SCFAs的含量,因SCFAs可作为宿主的能量底物,从而降低宿主对外源性能量的需求。本研究结果表明,在属水平仍存在80%以上未成功注释到的基因序列,这就对总体结果的分析产生了一定影响,后续可以加入宏转录组等新兴微生物学分析技术,以实现对LDA奶牛发病前后肠道微生物的变化有准确的认知,从而更深入地揭示真胃移位的发病机理,为发病前微生态预防提供理论参考。
3.3 真胃变位奶牛发病前后粪便中微生物菌群变化对机体代谢的影响 通过LEfSe功能预测分析发现,LDA奶牛碳水化合物代谢和脂质代谢通路相关功能基因显著上调。而碳水化合物代谢与脂质代谢代谢紊乱后,会加重能量负平衡,进而引发酮病[30]。研究证实,能量负平衡、酮病、低血钙等营养代谢问题是LDA发生的主要诱因[3, 31-32]。发病前后代谢通路的变化,提示肠道微生物在促使奶牛罹患LDA方面发挥了一定的作用。
综上所述,奶牛肠道菌群发生紊乱后,一方面可能会影响奶牛胃肠道迷走神经的传导功能,引起真胃蠕动迟缓;另一方面可能会引发诸如碳水化合物代谢与脂质代谢等多种代谢通路失调。这两方面的原因,导致了LDA的发生。本研究进一步为LDA的致病机理、早期诊断和预防提供了理论依据与研究基础。
References
| [1] | Mueller K. Diagnosis, treatment and control of left displaced abomasum in cattle. In Practice, 2011, 33(9): 470-481. DOI:10.1136/inp.d6079 |
| [2] | McArt JAA, Nydam DV, Overton MW. Hyperketonemia in early lactation dairy cattle: A deterministic estimate of component and total cost per case. Journal of Dairy Science, 2015, 98(3): 2043-2054. DOI:10.3168/jds.2014-8740 |
| [3] | Caixeta LS, Herman JA, Johnson GW, McArt JAA. Herd-level monitoring and prevention of displaced abomasum in dairy cattle. Veterinary Clinics of North America: Food Animal Practice, 2018, 34(1): 83-99. DOI:10.1016/j.cvfa.2017.10.002 |
| [4] | Steinhoff U. Who controls the crowd? New findings and old questions about the intestinal microflora. Immunology Letters, 2005, 99(1): 12-16. DOI:10.1016/j.imlet.2004.12.013 |
| [5] | Hu XY, Li SM, Fu YH, Zhang NS. Targeting gut microbiota as a possible therapy for mastitis. European Journal of Clinical Microbiology & Infectious Diseases, 2019, 38(8): 1409-1423. DOI:10.1007/s10096-019-03549-4 |
| [6] | Pi Y, Gao K, Zhu WY. Advances in host-microbe metabolic axis. Acta Microbiologica Sinica, 2017, 57(2): 161-169. (in Chinese) 皮宇, 高侃, 朱伟云. 动物宿主——肠道微生物代谢轴研究进展. 微生物学报, 2017, 57(2): 161-169. |
| [7] | Ma C, Sun Z, Zeng BH, Huang S, Zhao J, Zhang Y, Su XQ, Xu J, Wei H, Zhang HP. Cow-to-mouse fecal transplantations suggest intestinal microbiome as one cause of mastitis. Microbiome, 2018, 6(1): 200. DOI:10.1186/s40168-018-0578-1 |
| [8] | 黄云飞. 基于肠道微生物测序和灌服原花青素对奶牛酮病的研究. 广西大学博士学位论文, 2018. |
| [9] | Doll K, Sickinger M, Seeger T. New aspects in the pathogenesis of abomasal displacement. The Veterinary Journal, 2009, 181(2): 90-96. DOI:10.1016/j.tvjl.2008.01.013 |
| [10] | Grice EA, Kong HH, Conlan S, Deming CB, Davis J, Young AC, Bouffard GG, Blakesley RW, Murray PR, Green ED, Turner ML, Segre JA. Topographical and temporal diversity of the human skin microbiome. Science, 2009, 324(5931): 1190-1192. DOI:10.1126/science.1171700 |
| [11] | Song ES, Jung SI, Park HJ, Seo KW, Son JH, Hong S, Shim M, Kim HB, Song KH. Comparison of fecal microbiota between german holstein dairy cows with and without left-sided displacement of the abomasum. Journal of Clinical Microbiology, 2016, 54(4): 1140-1143. DOI:10.1128/JCM.02442-15 |
| [12] | Mu?oz-Vargas L, Opiyo SO, Digianantonio R, Williams ML, Wijeratne A, Habing G. Fecal microbiome of periparturient dairy cattle and associations with the onset of Salmonella shedding. PLoS One, 2018, 13(5): e0196171. DOI:10.1371/journal.pone.0196171 |
| [13] | Pitta DW, Kumar S, Vecchiarelli B, Shirley DJ, Bittinger K, Baker LD, Ferguson JD, Thomsen N. Temporal dynamics in the ruminal microbiome of dairy cows during the transition period. Journal of Animal Science, 2014, 92(9): 4014-4022. DOI:10.2527/jas.2014-7621 |
| [14] | Huang S, Ji SK, Yan H, Hao YY, Zhang J, Wang YJ, Cao ZJ, Li SL. The day-to-day stability of the ruminal and fecal microbiota in lactating dairy cows. MicrobiologyOpen, 2020, 9(5): e990. |
| [15] | Shanks OC, Kelty CA, Archibeque S, Jenkins M, Newton RJ, McLellan SL, Huse SM, Sogin ML. Community structures of fecal bacteria in cattle from different animal feeding operations. Applied and Environmental Microbiology, 2011, 77(9): 2992-3001. DOI:10.1128/AEM.02988-10 |
| [16] | Jami E, Mizrahi I. Composition and similarity of bovine rumen microbiota across individual animals. PLoS One, 2012, 7(3): e33306. DOI:10.1371/journal.pone.0033306 |
| [17] | Turnbaugh PJ, Ley RE, Mahowald MA, Magrini V, Mardis ER, Gordon JI. An obesity-associated gut microbiome with increased capacity for energy harvest. Nature, 2006, 444(7122): 1027-1031. DOI:10.1038/nature05414 |
| [18] | Muller PA, Koscsó B, Rajani GM, Stevanovic K, Berres ML, Hashimoto D, Mortha A, Leboeuf M, Li XM, Mucida D, Stanley ER, Dahan S, Margolis KG, Gershon MD, Merad M, Bogunovic M. Crosstalk between muscularis macrophages and enteric neurons regulates gastrointestinal motility. Cell, 2014, 158(2): 300-313. DOI:10.1016/j.cell.2014.04.050 |
| [19] | Diaz Heijtz R, Wang SG, Anuar F, Qian Y, Bj?rkholm B, Samuelsson A, Hibberd ML, Forssberg H, Pettersson S. Normal gut microbiota modulates brain development and behavior. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(7): 3047-3052. DOI:10.1073/pnas.1010529108 |
| [20] | Schwartz GJ. The role of gastrointestinal vagal afferents in the control of food intake: current prospects. Nutrition, 2000, 16(10): 866-873. DOI:10.1016/S0899-9007(00)00464-0 |
| [21] | Shin NR, Whon TW, Bae JW. Proteobacteria: microbial signature of dysbiosis in gut microbiota. Trends in Biotechnology, 2015, 33(9): 496-503. DOI:10.1016/j.tibtech.2015.06.011 |
| [22] | Guo W, Guo XJ, Zhou X, Li SN, Zhu BC. Effect of corn stalk fermented by complex bacterial on rumen bacteria diversity in sheep. Acta Veterinaria Et Zootechnica Sinica, 2018, 49(4): 736-745. (in Chinese) 郭威, 郭晓军, 周贤, 李术娜, 朱宝成. 复合菌剂发酵玉米秸秆对绵羊瘤胃液细菌多样性的影响. 畜牧兽医学报, 2018, 49(4): 736-745. |
| [23] | Gophna U, Konikoff T, Nielsen HB. Oscillospira and related bacteria-from metagenomic species to metabolic features. Environmental Microbiology, 2017, 19(3): 835-841. DOI:10.1111/1462-2920.13658 |
| [24] | Bekele AZ, Koike S, Kobayashi Y. Phylogenetic diversity and dietary association of rumen Treponema revealed using group-specific 16S rRNA gene-based analysis. FEMS Microbiology Letters, 2011, 316(1): 51-60. DOI:10.1111/j.1574-6968.2010.02191.x |
| [25] | Flint HJ, Bayer EA. Plant cell wall breakdown by anaerobic microorganisms from the mammalian digestive tract. Annals of the New York Academy of Sciences, 2008, 1125(1): 280-288. DOI:10.1196/annals.1419.022 |
| [26] | Dodd D, Mackie RI, Cann IKO. Xylan degradation, a metabolic property shared by rumen and human colonic Bacteroidetes. Molecular Microbiology, 2011, 79(2): 292-304. DOI:10.1111/j.1365-2958.2010.07473.x |
| [27] | Mohammed R, Brink GE, Stevenson DM, Neumann AP, Beauchemin KA, Suen G, Weimer PJ. Bacterial communities in the rumen of Holstein heifers differ when fed orchardgrass as pasture vs. hay. Frontiers in Microbiology, 2014, 5: 689. |
| [28] | Geirnaert A, Steyaert A, Eeckhaut V, Debruyne B, Arends JBA, Van Immerseel F, Boon N, Van de Wiele T. Butyricicoccus pullicaecorum, a butyrate producer with probiotic potential, is intrinsically tolerant to stomach and small intestine conditions. Anaerobe, 2014, 30: 70-74. DOI:10.1016/j.anaerobe.2014.08.010 |
| [29] | Bo TB, Zhang XY, Wen J, Deng K, Qin XW, Wang DH. The microbiota-gut-brain interaction in regulating host metabolic adaptation to cold in male Brandt's voles (Lasiopodomys brandtii). The ISME Journal, 2019, 13(12): 3037-3053. DOI:10.1038/s41396-019-0492-y |
| [30] | Seifi HA, LeBlanc SJ, Leslie KE, Duffield TF. Metabolic predictors of post-partum disease and culling risk in dairy cattle. The Veterinary Journal, 2011, 188(2): 216-220. DOI:10.1016/j.tvjl.2010.04.007 |
| [31] | Liu ZX, Zhu XY, Wang JG, Wang XX, Li XW, Chen H, Yang WT, Liu GW. The research progress of dairy cow ketosis. China Animal Husbandry & Veterinary Medicine, 2012, 39(4): 204-206. (in Chinese) 刘兆喜, 朱晓岩, 王建国, 王晓旭, 李心慰, 陈灰, 杨文涛, 刘国文. 奶牛酮病的研究进展. 中国畜牧兽医, 2012, 39(4): 204-206. |
| [32] | LeBlanc SJ, Leslie KE, Duffield TF. Metabolic predictors of displaced abomasum in dairy cattle. Journal of Dairy Science, 2005, 88(1): 159-170. DOI:10.3168/jds.S0022-0302(05)72674-6 |
