 , 王海月, 毛念佳, 任洪强, 耿金菊, 许柯
, 王海月, 毛念佳, 任洪强, 耿金菊, 许柯

南京大学环境学院, 污染控制与资源再利用国家重点实验室, 南京 210023
收稿日期: 2020-07-21; 修回日期: 2020-09-18; 录用日期: 2020-09-18
基金项目: 国家水体污染控制与治理科技重大专项(No.2017ZX07202003)
作者简介: 彭玲(1997—), 女, E-mail: 13856041225@163.com
通讯作者(责任作者): 许柯, E-mail: kexu@nju.edu.cn
摘要:通过对照试验考察了添加MnO2对厌氧氨氧化污泥驯化和培养的影响,并采用16S rRNA基因高通量测序技术和定量PCR分析其机理.结果表明,添加MnO2粉末能够显著提高容积氮负荷及运行性能,其脱氮速率高达848.04 mg·L-1·d-1,几乎是对照组反应器的2倍.16S rRNA基因高通量测序技术和定量PCR结果表明,添加MnO2粉末促进了典型厌氧氨氧化菌Candidate Brocadia的富集,厌氧氨氧化菌16S rRNA基因和功能基因hzsB的丰度显著提高.
关键词:厌氧氨氧化生物脱氮启动MnO2
Cultivation of anammox sludge in an EGSB reactor with MnO2 powder addition
PENG Ling
 , WANG Haiyue, MAO Nianjia, REN Hongqiang, GENG Jinju, XU Ke
, WANG Haiyue, MAO Nianjia, REN Hongqiang, GENG Jinju, XU Ke

State Key Laboratory of Pollution Control and Resource Reuse, School of the Environment, Nanjing University, Nanjing 210023
Received 21 July 2020; received in revised from 18 September 2020; accepted 18 September 2020
Abstract: In this study, the effect of adding MnO2 on the acclimation and cultivation of anammox sludge was investigated through controlled experiments. High-throughput 16S rRNA gene sequencing and qPCR were used to clarify the mechanism. The results showed that the addition of MnO2 powder could increase the volume nitrogen load and performance, and the nitrogen removal efficiency reach up to 848.04 mg·L-1·d-1, which was almost twice as high as that of the control reactor. Moreover, the results of high-throughput 16S rRNA gene sequencing and qPCR revealed that the addition of MnO2 powder promoted the enrichment of typical anaerobic ammonium oxidation bacteria Candidate Brocadia, and the abundance of 16S rRNA gene and functional gene hzsB of anammox were significantly improved.
Keywords: anammoxbiological nitrogen removalstart-upMnO2
1 引言(Introduction)厌氧氨氧化(Anammox)是指在厌氧条件下, 厌氧氨氧化菌(AAOB)以氨氮为电子供体, 以亚硝酸盐为电子受体, 直接将其转化为氮气的脱氮过程(Mulder et al., 1995; Strous et al., 1997).厌氧氨氧化作为一种极具应用前景的生物脱氮新工艺, 已成为废水脱氮处理领域的研究热点(Ma et al., 2020).与传统硝化-反硝化过程相比, 厌氧氨氧化具有不需要有机碳、降低曝气、减少污泥产量等优势(Miao et al., 2019).
厌氧氨氧化菌是厌氧氨氧化工艺的核心, 是以氨和亚硝酸盐为基质的化能自养菌.目前发现的厌氧氨氧化菌共有6个属:Brocadia、Kuenenia、Jettenia、Scalindua、Anammoxoglobus、Anammoximicrobium, 19个种(Zhang et al., 2020).Liu等(2020)通过11项基于高通量测序技术的微生物群落物种丰富度的研究, 总结出Candidatus Brocadia、Candidatus Kuenenia、Candidatus Jettenia是厌氧氨氧化过程中最占优势的菌属.Kartal等(2011)在宏基因组及酶学研究基础上, 提出了厌氧氨氧化菌的分解代谢模型:在厌氧氨氧化体内, 首先NO2-在亚硝酸还原酶作用下被还原成NO;然后, NO与NH4+在联氨合成酶作用下缩合成中间产物N2H4;最后, N2H4在联氨脱氢酶的作用下被分解为N2.厌氧氨氧化菌增殖速率慢、世代周期长(Vega De Lille et al., 2015), 导致培养难、启动慢, 进而极大地限制了厌氧氨氧化工艺的工程应用.因此, 强化厌氧氨氧化菌的培养和富集一直是厌氧氨氧化研究领域关注的焦点.近年来, 许多研究人员采用不同措施以加快厌氧氨氧化菌培养, 包括选择合适的反应器类型(Du et al., 2017)、接种污泥(Ding et al., 2016; Kocamemi et al., 2018)、使用冲刷策略(Kieling et al., 2007)、添加载体(Lv et al., 2019; Gupta et al., 2019)、添加微量元素如Fe2+(Bi et al., 2014; Shu et al., 2016)等.郭蓓蓓(2019)通过添加Fe2+成功富集了厌氧氨氧化菌, qPCR结果表明, nirS基因的表达量增加近1倍, 亚硝酸盐还原酶的含量增加了26.4%.
锰是微生物多种生理生化反应所必需的微量元素(Srivastava et al., 2015), 对于微生物活性具有促进作用.研究表明, 锰通常参与细菌超氧化物歧化酶(SOD)的活动, 同时也是一些酶(PEP羧化酶、柠檬酸合酶)的辅助因子, 适量浓度的锰离子能够提高微生物的活性(Christianson, 1997).Huang等(2012)研究发现, 投加低浓度的锰离子可以促进好氧污泥颗粒化, 与对照组相比, 投加1 mg·L-1锰离子的实验组具有更高的COD和NH4+-N去除效率, 且好氧颗粒污泥的胞外聚合物具有更高的有机物多样性, 尤其是蛋白质的多样性.Wang等(2019)研究表明, 添加锰氧化物的MBBR反应器可以提高酚类污染物降解率, 在高苯酚浓度下实现了锰氧化菌(MnOB)的富集.此外, Kartal等(2007)研究发现, 厌氧氨氧化菌(Candidatus Kuenenia stuttgartiensis)具有多种代谢方式, 其中就有锰参与代谢的基因.锰氧化物也可以作为代谢过程中的替代电子受体(Mogollon et al., 2016), 参与厌氧氨氧化过程.然而, 有关锰氧化物对厌氧氨氧化工艺启动过程的影响及其生物学机理尚未见报道.
基于此, 本研究采用EGSB反应器, 接种反硝化污泥, 考察添加MnO2对厌氧氨氧化工艺启动过程的影响.同时, 采用荧光定量PCR技术研究厌氧氨氧化细菌丰度及与氮转化相关的功能基因的变化规律, 通过16S rRNA基因高通量测序技术研究菌群结构演替规律, 并利用扫描电镜(SEM)观察污泥形态.以期为强化厌氧氨氧化污泥的富集培养和工艺启动提供参考, 并为MnO2强化厌氧氨氧化工艺提供理论依据.
2 材料与方法(Materials and methods)2.1 接种污泥与模拟废水本实验接种污泥取自南京某市政污水厂氧化沟缺氧段, 为黄棕色絮状污泥, 反应器中接种污泥浓度为20.0 g·L-1.接种前用pH=7.2的K2HPO4/KH2PO4缓冲液冲洗接种污泥3次, 洗去污泥中的氨氮、有机物、亚硝酸盐及硝酸盐.实验采用模拟废水, 成分见表 1.模拟废水中添加1.25 mL·L-1的微量元素浓缩液(Van De Graaf et al., 1996), 成分见表 2.
表 1(Table 1)
| 表 1 模拟废水组分 Table 1 Composition of synthetic wastewater | |||||||||||||||||||||||||||
表 1 模拟废水组分 Table 1 Composition of synthetic wastewater
| |||||||||||||||||||||||||||
表 2(Table 2)
| 表 2 微量元素浓缩液组成 Table 2 Composition of trace elements solution | ||||||||||||||||||||||
表 2 微量元素浓缩液组成 Table 2 Composition of trace elements solution
| ||||||||||||||||||||||
2.2 实验装置与运行条件实验采用2个膨胀颗粒床反应器(Expanded Granular Sludge Bed, EGSB)(R1为对照组, R2为实验组), 由有机玻璃制成, 有效容积为0.8 L.反应器出水回流比为8, 上升流速为0.1 m·h-1, 反应器外裹锡纸以避免光的负面影响(van der Star et al., 2008; Van Hulle et al., 2010).实验装置示意图见图 1.
图 1(Fig. 1)
 |
| 图 1 实验装置示意图 Fig. 1Schematic diagram of the experimental device |
在R2反应器底部加入30 g MnO2粉末, 平均粒径为4~7 μm, 200 ℃活化6 h.两个反应器均连续运行, 水力停留时间均为24 h, 温度为(35±1) ℃, pH为7.5±0.2.进水用99.5% N2吹扫, 使水中溶解氧小于0.5 mg·L-1.进水中NH4+-N与NO2--N的浓度比设定为1:1.32, 初始NH4+-N浓度为30 mg·L-1, 然后根据反应器的运行情况逐步提高底物浓度.
2.3 样品采集与分析每天采集出水样品, 经0.45 μm滤膜过滤后测定.氨氮(NH4+-N)采用水杨酸分光光度法测定, 亚硝酸盐氮(NO2--N)采用N-(1-萘基)-乙二胺分光光度法测定, 硝酸盐氮(NO3--N)采用酚二磺酸分光光度法测定.溶解氧(DO)使用便携式溶解氧仪测定, pH值由pH计测定.
污泥样品进行SEM检测前的预处理方法如下(Araujo et al., 2003):从反应器中取适量污泥放于离心管中, 用超纯水清洗3次, 弃上清液;离心管中加入0.1 mmol·L-1的磷酸盐缓冲液(含2.5%戊二醛, pH=7.3), 淹没样品, 置于4 ℃冰箱中固定12 h;用0.1 mmol·L-1的磷酸盐缓冲液(pH=7.3)冲洗固定好的污泥3次, 每次10 min;用浓度为50%、70%、80%、90%、95%的乙醇对污泥样品进行脱水处理, 每次10 min;用100%的乙醇脱水处理3次, 每次10 min, 然后冷冻干燥;用离子溅射仪(ION SPUTTER, JFC-1100)喷金镀膜, 采用环境样品扫描电镜(QuantaTM 250 FEG, 美国FEI公司)观察.
2.4 实时荧光定量PCR分析2.4.1 DNA提取取适量污泥样品, 用FastDNA? SPIN Kit for soil试剂盒(MP, USA)提取DNA, 用NanoDrop 2000超微量紫外分光光度计定量后在-20 ℃下保存.
2.4.2 PCR扩增及质粒制备对Bacterial 16S rRNA(细菌16S rRNA基因)、Anammox bacterial 16S rRNA(厌氧氨氧化菌16S rRNA基因)及相关氮素转化功能基因进行定量PCR分析, 包括:amoA(编码氨单加氧酶)、napA(编码周质硝酸盐还原酶)、narG(编码膜结合硝酸盐还原酶)、nirK(编码铜型亚硝酸还原酶)、nirS(编码血红素cd1型亚硝酸还原酶)、hzsB(编码联胺合成酶b亚基).所用引物参考已发表的文献, 由南京金斯瑞生物科技有限公司合成, 使用前稀释至20 μmol·L-1.具体引物序列、目标基因片段长度及扩增条件见表 3.
表 3(Table 3)
| 表 3 定量PCR分析的引物及扩增程序 Table 3 Primers, protocols and parameters of target genes in qPCR analysis | |||||||||||||||||||||||||||||||||||||||||||||
表 3 定量PCR分析的引物及扩增程序 Table 3 Primers, protocols and parameters of target genes in qPCR analysis
| |||||||||||||||||||||||||||||||||||||||||||||
2.4.3 实时荧光定量PCR扩增采用实时荧光定量PCR仪AB7500(Life Technologies, 美国)进行定量PCR扩增, 反应体系25 μL, 包括12.5 μL 2×SYBR? Green PCR Master Mix(Vazyme, 中国)、2 μL DNA模板(稀释至20 ng·μL-1)、正反向引物各0.2 μL及10.1 μL ddH2O.具体扩增程序如表 3所示.实验中目标基因扩增效率均在90%~110%, 标准曲线的R2大于0.99.
2.5 16S rRNA基因高通量测序分析采用Illumina高通量测序平台(Miseq, Illumina Inc., USA)测定16S rRNA基因, 具体步骤包括DNA样品提取、PCR扩增、扩增产物验证与纯化、测序及后续数据处理.
采用细菌通用引物对污泥样品DNA进行16S rRNA基因扩增, 引物序列如下所示(Zhu et al., 2015):正向引物(5′- AGAGTTTGATYMTGGCTCAG-3′)和反向引物(5′- TGCTGCCTCCCGTAGGAGT-3′).扩增体系为50 μL, 包括2 μL DNA模板(稀释至20 ng·μL-1)、4 μL MgCl2、4 μL DNTP Mixture、0.25 μL Ex Taq DNA聚合酶、5 μL 10×PCR Buffer(TaKaRa, 日本)、正反引物各1 μL, 剩余体积以无菌超纯水补足.PCR反应于Veriti PCR仪(Life Technologies, 美国)进行, 扩增程序为:98 ℃预变性5 min, 扩增循环20次(98 ℃变性30 s, 50 ℃退火30 s, 72 ℃延伸40 s), 72 ℃延伸10 min. 取5 μL扩增产物, 用2%的琼脂糖凝胶做电泳验证, 用E.Z.N.A.TM Cycle-Pure Kit试剂盒进行纯化, 纯化后的样品送至江苏中宜金大分析检测有限公司, 基于Illumina Miseq测序平台进行高通量测序分析.数据经Sickle及Mothur软件处理后, 使用Heml(Version 1.0)绘制热图.
3 结果与讨论(Results and discussion)3.1 反应器运行性能两个反应器启动期间的氮素浓度变化、容积氮负荷及容积氮去除速率分别如图 2和图 3所示.根据反应器容积效能可将厌氧氨氧化启动过程分为4个阶段:内源反硝化阶段、活性迟滞阶段、活性提高阶段和稳定运行阶段(Tang et al., 2009; Bi et al., 2014).两个反应器R1和R2均能观察到4个阶段, 但由于R2中添加了MnO2粉末, 二者在不同阶段的运行效能存在一定差异.
图 2(Fig. 2)
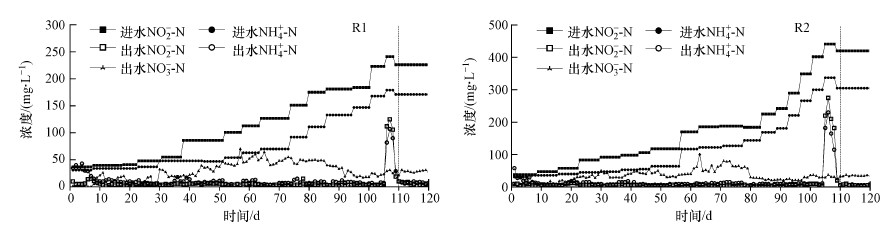 |
| 图 2 两个反应器运行期间的氮浓度分布 Fig. 2Nitrogen concentration profile during the operation of both reactors |
图 3(Fig. 3)
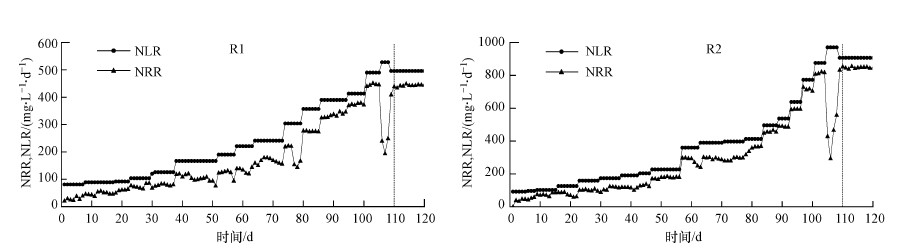 |
| 图 3 反应器R1和R2的容积氮负荷及容积氮去除速率 Fig. 3Nitrogen loading rate (NLR) and nitrogen removal rate (NRR) of R1 and R2 reactors |
如图 2所示, 在培养初期(0~8 d), 即内源反硝化阶段, 两个反应器的出水氨氮浓度均高于进水氨氮浓度.这是由于进水中不存在有机碳源, 部分异养微生物因缺乏底物发生自溶导致氨氮升高(Tang et al., 2009).此外, 两个反应器的出水中几乎没有亚硝酸盐, 这是由于反硝化菌以微生物自溶释放的有机物为底物进行反硝化从而将亚硝酸盐去除(van der Star et al., 2007).同时, 反应器内污泥由黄棕色变为黑色, 并有明显减量.
随着菌体自溶和反硝化作用的减弱, 两个反应器呈现出弱厌氧氨氧化作用, 进入活性迟滞阶段.R1和R2反应器的活性迟滞阶段分别持续了15 d(第9~23 d)和7 d(第9~15 d).在此阶段, 各反应器进水氨氮和亚硝酸盐浓度均稳定在45.1 mg·L-1和47.2 mg·L-1, R1和R2的平均氨氮去除率分别为31.5%和32.3%, 平均容积氨氮去除速率分别为17.6 mg·L-1·d-1和18.2 mg·L-1·d-1.在这一阶段两个反应器逐渐适应环境, 反硝化作用减弱, 出水水质变化特征相似, 无明显差异.
在活性提高阶段, 反应器中厌氧氨氧化作用不断增强, 成为主要脱氮途径.为避免NO2-累积抑制厌氧氨氧化, 将出水回流比设为8, 上流速度为0.8 m·h-1. 随着容积氮负荷(NLR)增加, 两个反应器均稳定运行, 容积氮去除速率(NRR)稳步提高.R1的活性提高阶段为第24~105 d, NLR由110.5 mg·L-1·d-1提升至495.6 mg·L-1·d-1, 氨和亚硝酸盐平均去除率分别为92.5%和96.7%.R2的活性提高阶段为第16~104 d, NLR由172.3 mg·L-1·d-1提升至847.2 mg·L-1·d-1, 氨和亚硝酸盐平均去除率分别为93.1%和97.4%, 容积氮负荷和脱氮效果均高于R1.闾刚等(2017)认为此过程中出水NO2--N浓度由高到低表明厌氧氨氧化菌的活性由弱到强, 并逐渐成为系统中主要菌种.
两个反应器在105 d时均出现了失稳现象, 出水NH4+-N和NO2--N浓度急剧升高, 亚硝酸盐浓度高于100 mg·L-1, NRR迅速下降.以往研究表明, 过量的游离氨、亚硝酸盐和硝酸盐对厌氧氨氧化有抑制作用(Aktan et al., 2012; Fernandez et al., 2012; Qi et al., 2018), 其中, 亚硝酸盐抑制作用最强.当亚硝酸盐浓度达到100 mg·L-1时, 可强烈抑制厌氧氨氧化菌的活性(Strous et al., 1999).反应器失稳后, 降低进水氮浓度, 增大回流比, 消除基质的抑制作用, 两个反应器均很快恢复正常, 进入稳定运行阶段, 此时R1进水NH4+-N为170.6 mg·L-1, R2进水NH4+-N为305.4 mg·L-1.R1的NLR和NRR分别为496.1 mg·L-1·d-1和441.5 mg·L-1·d-1, R2的NLR为907.6 mg·L-1·d-1, NRR为848.0 mg·L-1·d-1, 几乎是R1的2倍.表 4为两个反应器分别在4个阶段的时间分布.Chen等(2016b)在UASB反应器中接种污泥浓度(SS)为51.7 g·L-1的反硝化污泥, 经过151 d反应器运行稳定, NLR为700 mg·L-1·d-1, NRR为570 mg·L-1·d-1.钱俊伟等(2020)采用UAFB反应器, 接种MLSS为4500 mg·L-1的好氧活性污泥, 运行150 d后成功启动厌氧氨氧化反应器, 此时反应器容积负荷为610 mg·L-1·d-1.本实验结果表明, 在相对较低的污泥浓度下(20.0 g·L-1), MnO2可以强化富集厌氧氨氧化菌, 从而增加污泥负荷、提高脱氮效率, 加速厌氧氨氧化的启动.下文中的分子生物学检测结果也进一步证明了这一点.
表 4(Table 4)
| 表 4 反应器R1和R2每个阶段的时间 Table 4 Time of each stage of reactor R1 and R2 | |||||||||||||||
表 4 反应器R1和R2每个阶段的时间 Table 4 Time of each stage of reactor R1 and R2
| |||||||||||||||
3.2 功能基因及功能菌丰度变化两个反应器中16S rRNA基因和功能基因的丰度变化如图 4所示.与接种污泥相比, 两个反应器中细菌16S rRNA基因的绝对丰度均显著增加, 说明微生物富集成功.
amoA是氨单加氧酶的编码基因(Rotthauwe et al., 1997), 由于对进水溶解氧(DO)的严格控制, R1和R2中amoA的拷贝数均显著降低.反硝化功能基因narG(编码膜结合硝酸盐还原酶)、napA(编码周质硝酸盐还原酶)、nirK(编码铜型亚硝酸还原酶)、nirS(编码血红素cd1型亚硝酸还原酶)的丰度可用于表征反硝化作用的强弱(Throback et al., 2004; Kandeler et al., 2006; Bru et al., 2007).由于进水中不含有机碳源, 且严格限制溶解氧, 导致反硝化微生物受到抑制, 上述反硝化相关的功能基因丰度均显著降低(p < 0.05).接种污泥中厌氧氨氧化细菌16S rRNA基因的拷贝数为2.91×104 copies·ng-1 DNA, 稳定阶段时R1的厌氧氨氧化细菌16S rRNA基因拷贝数为4.18×106 copies·ng-1 DNA, R2中则达到8.47×106 copies·ng-1 DNA, 显著高于R1(p < 0.05), 这表明MnO2促进了厌氧氨氧化菌的富集.联胺合成酶是厌氧氨氧化代谢中的一种必需酶, 由hzsA、hzsB、hzsC基因编码的3个亚基组成(Strous et al., 2006; Kartal et al., 2011; Harhangi et al., 2012), 其中, hzsB基因是研究厌氧氨氧化菌丰度和生物多样性的合适分子标记.hzsB基因在接种污泥中的绝对丰度为1.88×103 copies·ng-1 DNA, 稳定阶段时hzsB基因在R1和R2中的拷贝数分别达到6.83×106和1.24×107 copies·ng-1 DNA, R2的hzsB基因丰度显著高于R1(p < 0.05), 这与厌氧氨氧化菌16S rRNA基因丰度的变化相一致, 表明MnO2富集厌氧氨氧化菌的同时也显著提高了厌氧氨氧化菌的活性.
图 4(Fig. 4)
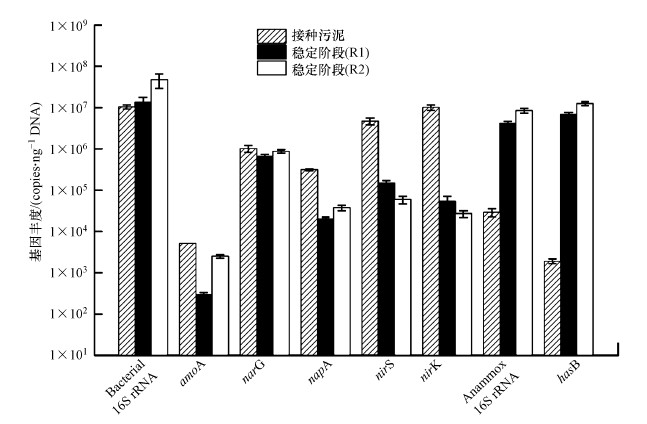 |
| 图 4 接种污泥和稳定阶段细菌、厌氧氨氧化菌及氮素转化功能基因丰度 Fig. 4Abundance of bacterial 16S rRNA, Anammox 16S rRNA and functional genes related to nitrogen transform in seed sludge and stationary phase |
3.3 微生物群落变化图 5a反映了接种污泥和稳定阶段两个反应器中污泥微生物群落门水平上的分布情况.在所有的样品中, 变形菌(Proteobacteria)和绿弯菌(Chloroflexi)是优势菌门, 这与Chen等(2016a)对COD/N为0的活性污泥群落分析结果一致.变形菌是一种典型的反硝化功能菌(Lu et al., 2014), 绿弯菌可以清除厌氧氨氧化细菌细胞衰老产生的有机化合物(Kindaichi et al., 2012).在接种的反硝化污泥中, 变形菌的比例为42.08%, 绿弯菌的比例为21.11%.当反应器启动成功后, 达到稳定阶段时, 厌氧氨氧化颗粒污泥中的菌落结构变化明显.随着反应器容积负荷的提高, 具有脱氮功能的变形菌所占比例逐渐降低, R1和R2中变形菌的相对丰度分别下降到26.72%和24.74%.由于变形菌消耗了反应器中大量的有机物, 使得绿弯菌可利用的有机物减少(姚芳等, 2017), 其相对丰度也相应地降低, 绿弯菌的相对丰度在R1和R2中分别下降到16.66%和14.25%.值得注意的是, 反应器中浮霉菌(Planctomycetes)有明显富集, 浮霉菌由接种污泥中的0.30%增加到稳定阶段中的14.10%(R1)和22.31%(R2).目前发现的厌氧氨氧化菌隶属于浮霉菌门(Gijs, 2008).Du等(2019)在成功启动厌氧氨氧化后, 浮霉菌的相对丰度显著提高至31.56%, 成为系统中最优势菌种.此外, 浮霉菌在R2中的相对丰度较R1中的高, 说明添加MnO2可以促进浮霉菌的富集.
图 5(Fig. 5)
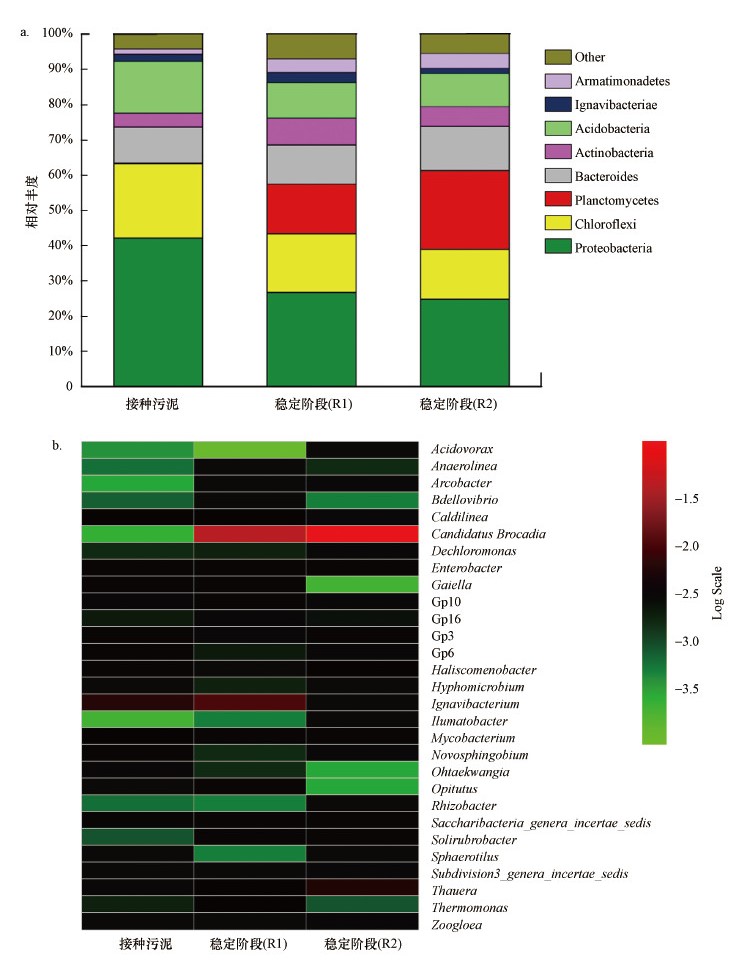 |
| 图 5 门水平上生物群落结构分布(a) 及污泥中属水平优势菌分布(相对丰度>0.1%)(b) Fig. 5Abundances of different phylum in seed sludge and sludge at stationary phase in both reactors(a) and heatmap of species abundance at the genus level (abundance>0.1%) in seed sludge and sludge at stationary phase in both reactors(b) |
图 5b为污泥微生物属水平物种丰度热图.可以发现, 在两个反应器中, 厌氧氨氧化菌Candidate Brocadia在稳定阶段污泥中所占比例较大.Candidate Brocadia是一种典型的厌氧氨氧化菌(AAOB)(Hu et al., 2010).在接种污泥中Candidate Brocadia的相对丰度为0.02%, 几乎检测不到.污泥经过富集培养后, 稳定阶段R1和R2中Candidate Brocadia的相对丰度分别达到5.1%和8.5%, 逐渐成为反应器内的优势菌属.姚芳等(2017)研究发现, 通过接种厌氧颗粒污泥, 反应器稳定运行后Candidate Brocadia的相对丰度为7.28%.上述结果表明, MnO2可以有效富集厌氧氨氧化菌Candidate Brocadia, 厌氧氨氧化是启动成功后反应器内主要的脱氮途径.
3.4 污泥形貌变化为探究MnO2添加对厌氧氨氧化污泥形貌的影响, 利用扫描电镜对启动前后污泥形貌进行对比分析, 结果如图 6所示.可以看到, 两个反应器的污泥形态都有显著变化.接种污泥表面不光滑, 这是由于各种微生物被胞外聚合物包裹, 且污泥表面为黑褐色.微生物主要为球菌(直径约为0.2 μm)和少量的芽孢杆菌, 污泥表面充满一定的空隙, 被认为是体内微生物能量代谢的重要通道(Pol et al., 2004).当厌氧氨氧化启动成功后, 两个反应器均达到稳定状态时, 污泥形态发生明显变化, 污泥生物量呈现砖红色细颗粒.Molinuevo等(2009)研究表明, 由于厌氧氨氧化菌中含有大量的血红素使得污泥呈红色.这表明启动过程中反应器中的厌氧氨氧化污泥不断形成与富集, 使得脱氮效率得到提高.图 6中球菌与丝状菌一起构成了污泥的大部分形态, 这与厌氧氨氧化菌的富集和污泥的颗粒化有关.R2中所培养出的厌氧氨氧化颗粒污泥的微生物相较对照反应器R1中更为丰富, 存在更多的球菌和丝状菌.Li等(2009)研究表明, 丝状菌在维持颗粒污泥形状和结构方面起着重要作用.这一现象与上述研究结果一致.结果表明, MnO2的添加对厌氧氨氧化的污泥形貌具有一定影响, 在反应器R2中可以看出明显更多的胞外聚合物分泌, 且厌氧氨氧化菌形态更好.
图 6(Fig. 6)
 |
| 图 6 接种污泥及稳定阶段R1和R2反应器污泥扫描电镜图 Fig. 6The SEM picture of seed sludge and sludges samples in R1 and R2 reactors in the stationary phase |
4 结论(Conclusions)1) 进水添加MnO2驯化和培养厌氧氨氧化污泥过程中, 实验组与对照组均成功完成厌氧氨氧化工艺的启动.实验组相比于对照组可承受更高的容积氮负荷.稳定运行阶段, R1和R2中容积氮负荷分别为496.1和907.6 mg·L-1·d-1, 容积氮去除速率分别为441.5和848.0 mg·L-1·d-1.添加MnO2对反应器容积氮负荷及容积氮去除速率具有促进作用.
2) 进水添加MnO2使得功能基因丰度和微生物群落结构发生显著变化.反硝化功能基因narG、napA、nirK、nirS的绝对丰度显著下降, 厌氧氨氧化功能基因hzsB的绝对丰度明显提高.厌氧氨氧化菌Candidatus Brocadia富集效果明显, 实验组R2中Candidatus Brocadia菌的相对丰度(8.5%)明显高于对照组R1(5.1%).
3) 添加MnO2对启动过程中污泥发育具有一定影响.稳定阶段实验组R2污泥中胞外聚合物分泌较多, 厌氧氨氧化菌富集效果较好.
参考文献
| Aktan C K, Yapsakli K, Mertoglu B. 2012. Inhibitory effects of free ammonia on Anammox bacteria[J]. Biodegradation, 23: 751-762. DOI:10.1007/s10532-012-9550-0 |
| Araujo J C, Teran F C, Oliveira R A, et al. 2003. Comparison of hexamethyidisilazane and critical point drying treatments for SEM analysis of anaerobic biofilms and granular sludge[J]. Journal of Electron Microscopy, 52: 429-433. DOI:10.1093/jmicro/52.4.429 |
| Bi Z, Qiao S, Zhou J, et al. 2014. Fast start-up of Anammox process with appropriate ferrous iron concentration[J]. Bioresour Technol, 170: 506-512. DOI:10.1016/j.biortech.2014.07.106 |
| Bru D, Sarr A, Philippot L. 2007. Relative abundances of proteobacterial membrane-bound and periplasmic nitrate reductases in selected environments[J]. Appl Environ Microbiol, 73: 5971-5974. DOI:10.1128/AEM.00643-07 |
| Chen C J, Sun F Q, Zhang H Q, et al. 2016a. Evaluation of COD effect on anammox process and microbial communities in the anaerobic baffled reactor (ABR)[J]. Bioresource Technology, 216: 571-578. DOI:10.1016/j.biortech.2016.05.115 |
| Chen H, Hu H Y, Chen Q Q, et al. 2016b. Successful start-up of the anammox process: Influence of the seeding strategy on performance and granule properties[J]. Bioresource Technology, 211: 594-602. DOI:10.1016/j.biortech.2016.03.139 |
| Christianson D W. 1997. Structural chemistry and biology of manganese metalloenzymes[J]. Progress in Biophysics & Molecular Biology, 67: 217-252. |
| Ding Z, Ventorino V, Panico A, et al. 2016. Enrichment of anammox biomass from different seeding sludge: Process strategy and microbial diversity[J]. Water, Air, & Soil Pollution: 228. DOI:10.1007/s11270-016-3181-8 |
| Du R, Cao S, Niu M, et al. 2017. Performance of partial-denitrification process providing nitrite for anammox in sequencing batch reactor (SBR) and upflow sludge blanket (USB) reactor[J]. International Biodeterioration & Biodegradation, 122: 38-46. |
| Du S, Li F, Li N, et al. 2019. Study on start-up and denitrification performance of anaerobic ammonium oxidation system[J]. Environmental Science and Technology, 42: 101-108. |
| Fernandez I, Dosta J, Fajardo C, et al. 2012. Short- and long-term effects of ammonium and nitrite on the Anammox process[J]. J Environ Manage, 95(Suppl): 170-174. |
| Francis C A, Roberts K J, Beman J M, et al. 2005. Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 102: 14683-14688. DOI:10.1073/pnas.0506625102 |
| Gijs K J. 2008. Anammox bacteria: from discovery to application[J]. Nature Reviews Microbiology, 6: 320-326. DOI:10.1038/nrmicro1857 |
| Gupta H P, Arora S, Verma S, et al. 2019. Evaluation of corncob as a bio-carrier for the enrichment of anammox bacteria using activated sludge as seed[J]. Biocatalysis and Agricultural Biotechnology, 19: 4. |
| 郭蓓蓓. 2019. 基于铁元素作用下强化厌氧氨氧化工艺研究[D]. 济南: 山东大学 |
| Harhangi H R, Le Roy M, Van Alen T, et al. 2012. Hydrazine synthase, a unique phylomarker with which to study the presence and biodiversity of anammox bacteria[J]. Applied and Environmental Microbiology, 78: 752-758. DOI:10.1128/AEM.07113-11 |
| He S, Gall D L, Mcmahon K D. 2007. "Candidatus accumulibacter" population structure in enhanced biological phosphorus removal sludges as revealed by polyphosphate kinase genes[J]. Applied and Environmental Microbiology, 73: 5865-5874. DOI:10.1128/AEM.01207-07 |
| Hu B L, Zheng P, Tang C J, et al. 2010. Identification and quantification of anammox bacteria in eight nitrogen removal reactors[J]. Water Research, 44: 5014-5020. DOI:10.1016/j.watres.2010.07.021 |
| Huang L H, Yang T, Wang W L, et al. 2012. Effect of Mn2+ augmentation on reinforcing aerobic sludge granulation in a sequencing batch reactor[J]. Applied Microbiology and Biotechnology, 93: 2615-2623. DOI:10.1007/s00253-011-3555-1 |
| Kandeler E, Deiglmayr K, Tscherko D, et al. 2006. Abundance of narG, nirS, nirK, and nosZ genes of denitrifying bacteria during primary successions of a glacier foreland[J]. Applied of the Environmental Microbiology, 72: 5957-5962. DOI:10.1128/AEM.00439-06 |
| Kartal B, Maalcke W J, De Almeida N M, et al. 2011. Molecular mechanism of anaerobic ammonium oxidation[J]. Nature, 479: 127-159. DOI:10.1038/nature10453 |
| Kartal B, Rattray J, Van Niftrik L A, et al. 2007. Candidatus "Anammoxoglobus propionicus" a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J]. Systematic and Applied Microbiology, 30: 39-49. DOI:10.1016/j.syapm.2006.03.004 |
| Kieling D D, Reginatto V, Schmidell W, et al. 2007. Sludge wash-out as strategy for Anammox process start-up[J]. Process Biochemistry, 42: 1579-1585. DOI:10.1016/j.procbio.2007.08.005 |
| Kindaichi T, Yuri S, Ozaki N, et al. 2012. Ecophysiological role and function of uncultured Chloroflexi in an anammox reactor[J]. Water Science and Technology, 66: 2556-2561. DOI:10.2166/wst.2012.479 |
| Kocamemi B A, Dityapak D, Semerci N, et al. 2018. Anammox start-up strategies: The use of local mixed activated sludge seed versus Anammox seed[J]. Water Science Technology, 78: 1901-1915. DOI:10.2166/wst.2018.431 |
| Li X R, Du B, Fu H X, et al. 2009. The bacterial diversity in an anaerobic ammonium-oxidizing (anammox) reactor community[J]. Systematic and Applied Microbiology, 32: 278-289. DOI:10.1016/j.syapm.2009.03.002 |
| Liu L J, Ji M, Wang F, et al. 2020. Insight into the influence of microbial aggregate types on nitrogen removal performance and microbial community in the anammox process-A review and meta-analysis[J]. Science of the Total Environment, 714: 8. |
| 闾刚, 徐乐中, 沈耀良, 等. 2017. 快速启动厌氧氨氧化工艺[J]. 环境科学, 38(3): 1116-1121. |
| Lu H J, Chandran K, Stensel D. 2014. Microbial ecology of denitrification in biological wastewater treatment[J]. Water Research, 64: 237-254. DOI:10.1016/j.watres.2014.06.042 |
| Lv Y, Pan J, Huo T, et al. 2019. Enhanced microbial metabolism in one stage partial nitritation-anammox system treating low strength wastewater by novel composite carrier[J]. Water Research, 163: 114872. DOI:10.1016/j.watres.2019.114872 |
| Ma W J, Li G F, Huang B C, et al. 2020. Advances and challenges of mainstream nitrogen removal from municipal wastewater with anammox-based processes[J]. Water Environment Research: 11. DOI:10.1002/wer.1342 |
| Miao L, Yang G Q, Tao T, et al. 2019. Recent advances in nitrogen removal from landfill leachate using biological treatments-A review[J]. Journal of Environmental Management, 235: 178-185. |
| Mogollon J M, Mewes K, Kasten S. 2016. Quantifying manganese and nitrogen cycle coupling in manganese-rich, organic carbon-starved marine sediments: Examples from the Clarion-Clipperton fracture zone[J]. Geophysical Research Letters, 43: 7114-7123. DOI:10.1002/2016GL069117 |
| Molinuevo B, Garcia M C, Karakashev D, et al. 2009. Anammox for ammonia removal from pig manure effluents: Effect of organic matter content on process performance[J]. Bioresource Technology, 100: 2171-2175. DOI:10.1016/j.biortech.2008.10.038 |
| Mulder A, Vandegraaf A A, Robertson L A, et al. 1995. Anaerobic ammonium oxidation discovered in a denitrifying fluidized-bed reactor[J]. Fems Microbiology Ecology, 16: 177-183. DOI:10.1111/j.1574-6941.1995.tb00281.x |
| Pol L W H, Lopes S I D, Lettinga G, et al. 2004. Anaerobic sludge granulation[J]. Water Research, 38: 1376-1389. DOI:10.1016/j.watres.2003.12.002 |
| Qi P, Li J, Dong H, et al. 2018. Performance of anammox process treating nitrogen-rich saline wastewater: Kinetics and nitrite inhibition[J]. Journal of Cleaner Production, 199: 493-502. DOI:10.1016/j.jclepro.2018.07.201 |
| 钱俊伟, 黎紫江, 王明明, 等. 2020. 升流式厌氧氨氧化流化床反应器脱氮效能研究[J]. 工业水处理, 40(1): 51-54. |
| Rotthauwe J H, Witzel K P, Liesack W. 1997. The ammonia monooxygenase structural gene amoA as a functional marker: Molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 63: 4704-4712. DOI:10.1128/AEM.63.12.4704-4712.1997 |
| Shu D, He Y, Yue H, et al. 2016. Effects of Fe(Ⅱ) on microbial communities, nitrogen transformation pathways and iron cycling in the anammox process: kinetics, quantitative molecular mechanism and metagenomic analysis[J]. RSC Advances, 6: 68005-68016. DOI:10.1039/C6RA09209H |
| Srivastava S, Agrawal S B, Mondal M K. 2015. A review on progress of heavy metal removal using adsorbents of microbial and plant origin[J]. Environmental Science and Pollution Research, 22: 15386-15415. DOI:10.1007/s11356-015-5278-9 |
| Strous M M, Kuenen J G J, Jetten M S M. 1999. Key physiology of anaerobic ammonium oxidation[J]. Applied and Environmental Microbiology: 7. DOI:10.3171/jns.2004.100.2.0332 |
| Strous M, Pelletier E, Mangenot S, et al. 2006. Deciphering the evolution and metabolism of an anammox bacterium from a community genome[J]. Nature, 440: 790-794. DOI:10.1038/nature04647 |
| Strous M, Vangerven E, Kuenen J G, et al. 1997. Effects of aerobic and microaerobic conditions on anaerobic ammonium-oxidizing (Anammox) sludge[J]. Applied and environmental microbiology, 63: 2446-2448. DOI:10.1128/AEM.63.6.2446-2448.1997 |
| Tang C J, Zheng P, Mahmood Q, et al. 2009. Start-up and inhibition analysis of the Anammox process seeded with anaerobic granular sludge[J]. J Ind Microbiol Biotechnol, 36: 1093-1100. DOI:10.1007/s10295-009-0593-0 |
| Throback I N, Enwall K, Jarvis A, et al. 2004. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE[J]. FEMS Microbiol Ecol, 49: 401-417. DOI:10.1016/j.femsec.2004.04.011 |
| Tsushima I, Ogasawara Y, Kindaichi T, et al. 2007. Development of high-rate anaerobic ammonium-oxidizing (anammox) biofilm reactors[J]. Water Research, 41: 1623-1634. DOI:10.1016/j.watres.2007.01.050 |
| Van De Graaf A A, De Bruijn P, Robertson L A, et al. 1996. Autotrophic growth of anaerobic ammonium-oxidizing micro-organisms in a fluidized bed reactor[J]. Microbiology (Reading), 142: 2187-2196. DOI:10.1099/13500872-142-8-2187 |
| Van Der Star W R L, Abma W R, Blommers D, et al. 2007. Startup of reactors for anoxic ammonium oxidation: Experiences from the first full-scale anammox reactor in Rotterdam[J]. Water Research, 41: 4149-4163. DOI:10.1016/j.watres.2007.03.044 |
| Van Der Star W R, Miclea A I, Van Dongen U G, et al. 2008. The membrane bioreactor: a novel tool to grow anammox bacteria as free cells[J]. Biotechnol Bioeng, 101: 286-294. DOI:10.1002/bit.21891 |
| Van Hulle S W H, Vandeweyer H J P, Meesschaert B D, et al. 2010. Engineering aspects and practical application of autotrophic nitrogen removal from nitrogen rich streams[J]. Chemical Engineering Journal, 162: 1-20. DOI:10.1016/j.cej.2010.05.037 |
| Vega De Lille M, Berkhout V, Fr ba L, et al. 2015. Ammonium estimation in an ANAMMOX SBR treating anaerobically digested domestic wastewater[J]. Chemical Engineering Science, 130: 109-119. DOI:10.1016/j.ces.2015.03.018 |
| Wang G C, Liu Y, Wu M H, et al. 2019. Coupling the phenolic oxidation capacities of a bacterial consortium and in situ-generated manganese oxides in a moving bed biofilm reactor (MBBR)[J]. Water Research, 166: 11. |
| Wang Y, Zhu G, Harhangi H R, et al. 2012. Co-occurrence and distribution of nitrite-dependent anaerobic ammonium and methane-oxidizing bacteria in a paddy soil[J]. Fems Microbiology Letters, 336: 79-88. DOI:10.1111/j.1574-6968.2012.02654.x |
| Yan T F, Fields M W, Wu L Y, et al. 2003. Molecular diversity and characterization of nitrite reductase gene fragments (nirK and nirS) from nitrate- and uranium-contaminated groundwater[J]. Environmental Microbiology, 5: 13-24. DOI:10.1046/j.1462-2920.2003.00393.x |
| 姚芳, 刘波, 王德朋, 等. 2017. 不同接种污泥的厌氧氨氧化反应器启动特性及菌群结构演替规律分析[J]. 环境科学学报, 37(7): 2543-2551. |
| Zhang L, Okabe S. 2020. Ecological niche differentiation among anammox bacteria[J]. Water Research, 171: 14. |
| Zhu G B, Wang S Y, Wang W D, et al. 2013. Hotspots of anaerobic ammonium oxidation at land-freshwater interfaces[J]. Nature Geoscience, 6: 103-107. DOI:10.1038/ngeo1683 |
| Zhu Y, Zhang Y, Ren H Q, et al. 2015. Physicochemical characteristics and microbial community evolution of biofilms during the start-up period in a moving bed biofilm reactor[J]. Bioresource Technology, 180: 345-351. DOI:10.1016/j.biortech.2015.01.006 |
