 , 严康1,2, 汪海珍1,2, 徐建明1,2
, 严康1,2, 汪海珍1,2, 徐建明1,2

1. 浙江大学土水资源与环境研究所, 杭州 310058;
2. 浙江省农业资源与环境重点实验室, 杭州 310058
收稿日期: 2019-08-06; 修回日期: 2019-09-06; 录用日期: 2019-09-06
基金项目: 国家自然科学基金(No.41771344);国家重点研发计划重点专项子课题(No.2016YFD0800207)
作者简介: 张维荣(1996—), 女, E-mail:figizhang@163.com
通讯作者(责任作者): 汪海珍, 女, 博士, 教授, 博士生导师, 主要研究方向为微生物与土水环境安全, 主持国家级和省部级科研项目10余项, 发表SCI科研论文50余篇. E-mail:wanghz@zju.edu.cn
摘要:为全面、系统地了解1983—2019年37年间多环芳烃降解基因研究领域的发文趋势、主要的研究国家和机构、研究热点及其变化趋势等,本文以Web of Science核心数据库为数据源,利用文献计量学方法对此进行分析,结果表明:①全球对多环芳烃降解基因研究的重视程度越来越高,而且分子生物学的技术手段在该研究领域中得到了广泛应用,发文主要集中于微生物学、生物技术与应用微生物学、环境科学等3个学科;②美国、中国、日本、德国等国家发文较多,相比其他国家,中国需进一步加强与其他研究机构间的交流合作;③1983—1995年该研究领域侧重于高效降解菌的筛选及其特性研究,1996—2019年则大规模展开了对其降解途径多样性的研究,近年来更侧重于运用多种生物信息技术来研究多环芳烃的代谢机理及构建生物降解调节机制;④目前已报道了nah、phd、nag、phn、nar和nid等6类多环芳烃降解菌的功能基因,为污染场地的生物修复提供了相应支撑.
关键词:多环芳烃降解基因Web of Science文献计量CiteSpaceHistCite
Bibliometric analysis of research progress on polycyclic aromatic hydrocarbons-degrading genes during 1983-2019
ZHANG Weirong1,2
 , YAN Kang1,2, WANG Haizhen1,2, XU Jianming1,2
, YAN Kang1,2, WANG Haizhen1,2, XU Jianming1,2

1. Institute of Soil and Water Resources and Environmental Science, College of Environmental and Resource Sciences, Zhejiang University, Hangzhou 310058;
2. Zhejiang Provincial Key Laboratory of Agricultural Resources and Environment, Hangzhou 310058
Received 6 August 2019; received in revised from 6 September 2019; accepted 6 September 2019
Abstract: This review employed bibliometric method to comprehensively and systematically analyze the trends, research hotspots and progress of polycyclic aromatic hydrocarbons (PAHs)-degrading genes based on the publications in the Web of Science Core Database during 1983-2019. Our analysis shows that ① research in this field has attracted more attention worldwide, and molecular biology technologies have been widely used in the studies. The top 3 categories of the publications are Microbiology, Biotechnology & Applied Microbiology, and Environmental Sciences; ②most of the publications in this field are from United States, China, Japan and Germany. The further cooperation between China and other countries or institutions needs to be strengthened; and ③ research on screening and characterizing highly-efficient bacteria for PAH degradation is mainly conducted during 1983-1995, and then attention is shifted to the pathways of PAHs degradation from 1996 to 2019. Recently, various bio-informatics technologies have been applied to determine the regulation mechanisms of PAHs metabolisms; and ④ six functional genes, nah, phd, nag, phn, nar, nid, have been identified and reported. These genes are useful for bioremediation of PAHs contaminated sites.
Keywords: polycyclic aromatic hydrocarbonsdegradation genesWeb of SciencebibliometricsCiteSpaceHistCite
1 引言(Introduction)多环芳烃(polycyclic aromatic hydrocarbons, PAHs)是一类典型持久性有机污染物, 广泛分布于大气、水体和土壤等自然环境中(Lu et al., 2011), 对人类、动植物及微生物具有潜在的生物富集性及致癌、致畸、致突变等高毒性(Obrista et al., 2015), 美国环保署(United States Environmental Protection Agency)和欧联(European Union)已将16种多环芳烃列为“优先控制的污染物”(Renner et al., 1999).过去数十年, 由于汽车尾气、家庭取暖和各项工业活动, 世界范围内的多环芳烃污染形势严峻, 而且多环芳烃具有随空气远距离易迁移、难降解和生物易积累等特性, 可在全球范围内各生态圈之间不断循环, 甚至在格陵兰岛和南极洲偏远原始的地方都检出了微量的多环芳烃(Obrista et al., 2015).因此, 多环芳烃污染一直是世界各国所面临的重大全球环境问题之一, 对其防治刻不容缓.
近年来, 随着各项生物技术的快速发展, 多环芳烃污染土壤的生物修复不仅仅关注于功能微生物的降解效应, 同时结合原位场地的生态环境, 土壤中微生物群落结构也得到了更为细致和深入的探究(Tuhuloula et al., 2019).此外, 多环芳烃的相关代谢途径和机理、功能基因的多样性及表达也得到了国内外的广泛关注(Teng et al., 2019).尤其是多环芳烃降解基因领域, 是近年来的研究热点之一, 与其他研究方向关联性较强, 可从分子层面更深入地揭示多环芳烃降解微生物的基因表达和功能信息.
CiteSpace是一个基于Java环境的科学可视化分析软件, 通过可视化的手段来呈现科学知识的结构、规律和分布状况等科学知识图谱(李杰等, 2017), 已被广泛应用于科学计量学领域.Histcite也是一种对可输出数据进行社交网络分析的计量软件, 可弥补CiteSpace在文献计量时的部分功能缺陷(Lucio-Arias et al., 2008).为了全面、系统地回顾与了解多环芳烃降解基因领域的研究进展和热点趋势, 本文对该领域内在Web of Science核心数据库中发表的研究论文进行了科学计量审查, 并使用CiteSpace软件进行可视化计量分析, 旨在为该领域的科研工作者提供理论参考.
2 数据来源与分析方法(Data sources and analysis methods)2.1 数据来源分析论文的数据检索源是Web of Science核心数据库, 该数据库被认为是大多数科研领域中最重要且使用频率最高的数据库, 文件记录内容包括作者、标题、来源出版物、摘要和引用的参考文献.主题关键词使用“PAHs degradation genes”或“polycyclic aromatic hydrocarbons degradation genes”或“polycyclic aromatic hydrocarbons dioxygenase genes”或“phenanthrene degradation genes”或“pyrene degradation genes”或“anthracene degradation genes”或“naphthalene degradation genes”或“fluorene degradation genes”, 共检索到1983—2019年发表的1710篇文献, 检索时间为2019年7月4日.
2.2 数据处理与分析使用引文网络分析工具CiteSpace V和HistCite 2.1进行文献数据挖掘和可视化分析.CiteSpace分析内容包括国家(Country)、机构(Institution)、学科分类(Category)、期刊(Journals)和关键词(Keyword)分析, 时间划分(Timing Slicing)设置为1983—2019(7月)年, 时间节点(Years Per Slice)设为5年, 术语来源(Term Source)依次勾选为标题(Title)、摘要(Abstract)、作者关键词(Author Keywords)、扩展关键词(Keywords Plus), 阈值设置分别为top=45和90(樊一阳等, 2015; 吴健等, 2016; 朱宇恩等, 2017).
CiteSpace V分析中的统计指标包括频数和中心度, 频数是指1710篇文献在某计量指标中出现的文献数量, 中心度是表示某计量分析内容中该指标节点与其他节点间的关联性强弱, 中心度越高, 该指标的关联性越强、重要性越大(李杰等, 2017);同时, 计量分析结果可以用可视化图谱呈现, 图中圆圈大小指某计量指标的频数, 连线数量则代表指标节点间的耦合强度, 不同颜色表示相关研究发文的时间, 其分析结果可在一定程度上反映多环芳烃降解基因的研究阶段、发展趋势以及热点时期.此外, 利用HistCite对期刊(Journal)进行计量分析, 参数为默认值(最小引用数10), 统计指标包括本地引用次数(Total local citation score, TLCS)和总引用次数(Total global citation score, TGCS), TLCS是指某篇文章在本次HistCite分析数据源(1710篇)中的被引次数, TGCS则表示该文章在Web of Science数据库中的总被引次数, 这两个指标可为分析研究领域主要的发文期刊提供重要参考(Lucio-Arias et al., 2008).
3 结果与讨论(Results and methods)3.1 文献产出趋势根据论文发表的年份进行分类统计, 继而归纳出每年的文献发文量随年份变化的趋势线(图 1).同时, 本研究分析了包括环境科学(Environmental Sciences)、环境工程(Engineering, Environmental)、环境研究(Environmental Studies)和公共环境及职业健康(Public, Environmental & Occupational Health)等环境领域学科的发文量变化趋势.分析可知, 从1983—2019年, 关于多环芳烃降解基因方面的发文量变化总体呈上升趋势, 表明国内外对该领域的关注度逐步提高.其中, 环境领域的发文量与总发文量的变化趋势大致相似.
图 1(Fig. 1)
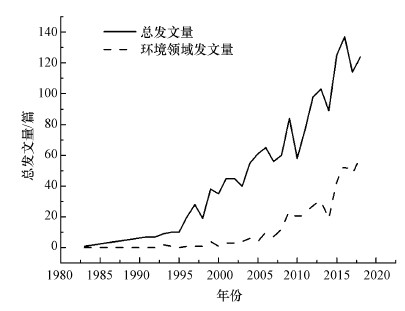 |
| 图 1 1983—2019年多环芳烃降解基因领域文献产出量 Fig. 1Publications on PAHs-degrading genes during 1983—2019 |
在36年的发展过程中, 该领域的研究整体上可以分为两个阶段, 1983—1995年为研究起步阶段, 该阶段发文量上升趋势较缓, 期间仅有45篇文献被收录, 占比总发文量的2.7%, 研究主要集中于生物技术与应用微生物学(Biotechnology & Applied Microbiology)和微生物学(Microbiology)学科(90%);1996—2019年多环芳烃降解基因的研究进入快速发展阶段, 发文量大致呈线性增长趋势, 发文量达到1665篇, 占比总发文量的95%以上, 远超研究起步阶段.在快速发展阶段, 生物技术与应用微生物学(Biotechnology & Applied Microbiology)和微生物学(Microbiology)学科的研究仍然占主体, 但与环境科学(Environmental Sciences)、环境工程(Engineering, Environmental)等环境领域学科的交叉联合逐渐增强(图 1).其中, 环境科学学科(Environmental Sciences)在多环芳烃降解基因领域的研究显著增多, 从1993年发表第1篇文献到2019年该学科的发文量占该领域1983—2019年总发文量的25.9%(443篇), 发文量仅次于微生物学科(Microbiology)、生物技术与应用微生物学科(Biotechnology & Applied Microbiology), 环境工程学科(Engineering, Environmental)的发文量和中心度也位于学科前10(表 1).即表明, 环境领域在多环芳烃降解基因方面的研究与生物学的相关领域联系紧密, 交叉程度较大.
表 1(Table 1)
| 表 1 1983—2019年多环芳烃降解基因领域发文量前十的学科 Table 1 Top 10 categories of publications on PAHs-degrading genes during 1983—2019 | ||||||||||||||||||||||||||||||||||||||||||||
表 1 1983—2019年多环芳烃降解基因领域发文量前十的学科 Table 1 Top 10 categories of publications on PAHs-degrading genes during 1983—2019
| ||||||||||||||||||||||||||||||||||||||||||||
3.2 主要的发文期刊使用HistCite软件对多环芳烃降解基因领域内发文量前10的期刊进行文献计量(表 2), 结果表明, 发文学科主要集中于微生物学(Microbiology)、生物技术与应用微生物学(Biotechnology & Applied Microbiology)、环境科学(Environmental Sciences)和环境工程(Engineering, Environmental), 与3.1节中结论相互印证;International Biodeterioration & Biodegradation(也属于生物技术与应用微生物学学科)、Journal of Hazardous Material、Environmental Science and Pollution Research和Chemosphere等环境领域期刊上的发文量排序分别为第3、5、7和9位.从本地引用次数(TLCS)和总引用次数(TGCS)的数值结果上看, Applied and Environmental Microbiology远高于其它期刊, 其次是Journal of Bacteriology和Applied Microbiology and Biotechnology(表 2).
表 2(Table 2)
| 表 2 1983—2019年多环芳烃降解基因研究领域发文量前10的期刊 Table 2 Top 10 journals of publications on PAHs-degrading genes during 1983—2019 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 2 1983—2019年多环芳烃降解基因研究领域发文量前10的期刊 Table 2 Top 10 journals of publications on PAHs-degrading genes during 1983—2019
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
3.3 主要的发文国家及机构表 3陈列了该领域发文量排名前10的国家, 美国(USA, count=409)和中国(Peoples R China, count=354)的发文量均远高于排名第3位的日本(Japan, count=119).在中国的发文机构中, 中国科学院(Chinese Acad Sci)的发文量有100篇, 远高于其他机构, 甚至高于前10位的部分国家, 如加拿大(Canada, count=95)、韩国(South Korea, count=87)、法国(France, count=73)等, 说明中国科学院在该领域研究中的研究成果显著.
表 3(Table 3)
| 表 3 1983—2019年多环芳烃降解基因领域发文量前十的国家 Table 3 Top 10 countries of publications on PAHs-degrading genes during 1983—2019 | ||||||||||||||||||||||||||||||||||||||||||||
表 3 1983—2019年多环芳烃降解基因领域发文量前十的国家 Table 3 Top 10 countries of publications on PAHs-degrading genes during 1983—2019
| ||||||||||||||||||||||||||||||||||||||||||||
另外, 从发文国家和机构的研究年限上来看, 美国(USA)是最早开始研究该领域的国家之一, 且中心度(表 3)和合作紧密度(图 2)远高于其他国家.日本(Japan)、德国(Germany)、加拿大(Canada)开始研究稍晚, 而中国(Peoples R China)的研究主要在快速发展阶段展开(1996—2019), 尤其是近10年来发展迅速, 发文量激增, 甚至超过日本(Japan)、德国(Germany)、加拿大(Canada)等(图 2), 但和世界各国的合作交流联系不紧密.而德国(Germany)的发文量远低于中国(Peoples R China), 但在全球范围内的合作交流联系仅次于美国(USA)和加拿大(Canada).从结果上看, 美国(USA)、日本(Japan)、德国(Germany)等经济发达体不仅研究起步时间较早, 且在该研究领域上的成果和创新性上都处于世界前列, 中国(Peoples R China)随着近年来经济的快速发展, 在该研究领域的发文量已超过部分发达国家, 但还需进一步加强与其他国家及机构的交流和合作.
图 2(Fig. 2)
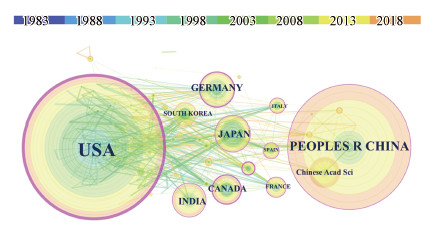 |
| 图 2 多环芳烃降解基因研究领域的主要发文国家和机构 Fig. 2The major countries and institutions of publications on PAHs-degrading genes |
3.4 关键词及研究热点关键词可视化是了解文献全文主旨和研究内容的一种快速有效的途径, 关键词的出现频数可以集中反映该领域关注的研究热点及其变化.基于CiteSpace可视化软件, 对1983—2019年多环芳烃降解基因领域中的关键词进行共被现分析, 并构建文献计量网络可视化图谱.由图 3可见, 近年来该领域的研究热点紧紧围绕多环芳烃污染物类型、污染介质、降解修复措施、降解功能菌群的种类和特性、降解基因这5个方面展开.另外, 表 4中统计整理了1983—2019年该领域排名前20的高频关键词, 按照每个聚集圈中出现的高频关键词的数量及研究时期对以上研究热点进一步展开概述.
图 3(Fig. 3)
 |
| 图 3 1983—2019年多环芳烃降解基因领域中关键词文献计量网络图谱 Fig. 3The keywords′ bibliometric network map of PAHs-degrading genes during 1983—2019 |
表 4(Table 4)
| 表 4 1983—2019年多环芳烃降解基因领域相关文献的高频关键词统计 Table 4 High frequency keywords of publications on PAHs-degrading genes during 1983—2019 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 4 1983—2019年多环芳烃降解基因领域相关文献的高频关键词统计 Table 4 High frequency keywords of publications on PAHs-degrading genes during 1983—2019
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
由污染物类型高频关键词统计结果可见(表 4), 该领域研究的几种主要污染物包括菲(phenanthrene, count=283)、萘(naphthalene, count=235)、芘(pyrene, count=112), 而荧蒽(fluoranthene, count=50)、蒽(anthracene, count=25)、苯并[a]芘(benzo[a]pyrene, count=10)等其他几种高环多环芳烃的出现频数较少.表明低分子量多环芳烃(low molecular weight PAHs, LMW PAHs)的研究显著多于高分子量多环芳烃(high molecular weight PAHs, HMW PAHs), 引起这样的差异原因可能是多环芳烃自身的化学结构会影响植物和微生物对其的生物降解速率, 苯环的数量越多降解难度越大、开展的研究较少(Teng et al., 2019).
从对污染介质的研究来看, 该领域排名前二十的相关高频关键词中分别出现了土壤(soil, count=221)、污染土壤(contaminated soil, count=134), 此外, 沉积物(sediment, count=113)出现次数也远高于其他环境介质.这表明, 从1983—2019年土壤和沉积物中多环芳烃降解基因的研究受到广泛关注.已有大量研究报道目前国内外多环芳烃污染主要集中在土壤和沉积物中, 如意大利的罗马、巴勒莫、那不勒斯等城市土壤中的多环芳烃总量整体偏高(59.52~755 μg·g-1), 环境健康风险较大(Ligaray et al., 2016);Grba等(2016)也报道在塞尔维亚共和国的伏伊丁那省境内某流域附近的沉积物和土壤样品中, 10个优先控制的多环芳烃浓度为47.6~577 μg·kg-1不等, 并与其他重金属污染形成协同作用, 危害当地的生物群落.另有研究表明, 土壤和水域沿岸的沉积物是环境中多环芳烃的主要载体, 土壤和沉积物中的多环芳烃含量(达到mg·kg-1数量级)要比水体中高出100倍以上(Li et al., 2015; Ma et al., 2015), 且具有更大的分布系数和更小的逸度(Huang et al., 2019), 使得多环芳烃长期储存在土壤和沉积物环境中, 造成严重的环境健康风险.
近年来, 多环芳烃生物降解修复得到了国内外的普遍关注, 该聚集圈中高频关键词有降解(degradation, count=680)、生物降解(biodegradation, count=667)、生物修复(bioremediation, count=365)等词.目前, 已报道多种具有降解修复能力的细菌、真菌、古菌及其他藻类植物, 甚至有研究者利用原生动物来进行降解修复(Haritash et al., 2009; 张银萍等, 2010; Ghosal et al., 2016).相比其他微生物, 细菌是一类更为积极参与污染场地有机污染物降解的微生物(Haritash et al., 2009), 利用CiteSpace可视化所得的聚集圈中, bacteria的出现频数也高达217次.早在1988年, Heitkamp等(1988)就从环境中分离出了第一株HMW PAHs的降解菌Mycobacterium vanbaalenii PYR-1.目前, 已分离出多株LMW PAHs的降解菌, 但HMW PAHs的降解菌相对较少.与细菌不同的是, 除小克银汉霉属(Cunninghamella)、侧耳属(Pleurotus)、青霉菌属(Penicillium)等真菌外, 大多数真菌不能以多环芳烃作为单一碳源和能源来生长, 只能通过与其他物质进行共代谢的方式降解多环芳烃(Ghosal et al., 2016), 该领域中相关研究内容也主要集中于通过微生物共培养体系进行共代谢降解研究.在该聚集圈中, 还出现了其他的高频关键词, 包括鉴定(identification, count=168)、菌株(strain, count=148)、恶臭假单胞菌(Pseudomonas Putida, count=120)、微生物群落(microbial community, count=117)等, 表明在过去近30年的研究中, 大量研究集中于多环芳烃降解菌株的筛选和分离鉴定.有报道表明, Pseudomonas putida即是一种常见的多环芳烃降解菌, 能将有毒的多环芳烃转化为环境友好的其他化合物(Tuhuloula et al., 2019).
在第5个高频关键词聚集圈中, 基因(gene, count=285)、代谢(metabolism, count=153)、双加氧酶(dioxygenase, count=135)、表达(expression, count=115)和核苷酸测序(nucleotide sequence, count=112)等词关联紧密, 该聚集圈中关键词的频数少于其他几个聚集圈, 但与其他4个聚集圈的共现强度和关联性很强.表明在筛选和分离高效多环芳烃降解菌并探究其种类与特性的基础上, 多环芳烃的相关代谢途径和机理、降解酶、降解基因的多样性及表达等也得到了国内外的广泛关注, 从分子层面更深入地揭示了多环芳烃降解微生物的基因表达和功能信息.据报道, 多环芳烃环羟化双加氧酶(PAHs-RHD)基因是该领域广泛关注的一个研究热点, 在多环芳烃降解过程中的一系列酶促反应中, 细菌通过编码双加氧酶(dioxygenase)来进行多环芳烃降解过程中最重要的一步——环裂解(DeSousa et al., 2017; Liang et al., 2019; Sun et al., 2019).
3.5 多环芳烃降解基因研究进展及其分类概述从1983—2019年多环芳烃降解基因领域的研究历程中, 国内外的研究者在研究起步阶段(1983—1995年)更多地关注降解功能菌株的筛选, 即从环境样品中筛选并通过人工富集培养, 分离得到降解性能较好的菌株, 进一步探究其菌属特性和最适降解条件(Ashok et al., 1995), 并初步对降解过程中的代谢中间产物进行测定.在快速发展阶段(1996—2019年), 微生物降解多环芳烃的降解路径及方式得到了国内外科研工作者的广泛关注(Guo et al., 2016), 代谢过程中的多种作用酶系被发现, 包括羟基化双加氧酶、脱氢酶、水合酶-醛缩酶等, 但对多环芳烃降解基因的认知还非常有限(Fang et al., 2016), 仅有部分LMW PAHs降解基因被报道(Ben et al., 2008);随着研究的深入, 在已探究到的多环芳烃生物降解途径的多样性基础上, 逐步展开对降解分子机制及其代谢机理的研究, 并且PCR技术、DNA分子杂交技术、基因探针等多种分子生物学的相关技术工具得到了大规模应用, 该领域中大量的多环芳烃降解基因被发现, 其作用和分子遗传机制得到深入认知, 这为环境中多环芳烃的高效生物降解提供了新的可能性和途径;近几年来, 随着生物技术的进步, 高通量测序技术、宏基因组学、转录组学、蛋白质代谢组学和基因芯片技术等生物信息技术迅速发展, 并被快速应用于各项科学研究, 从DNA、RNA和蛋白质水平上解析了微生物群落结构、相互作用机制及其对多环芳烃的代谢机理(Durate et al., 2017;Teng et al., 2019), 为更好地理解多环芳烃的生物降解调节机制以及开展应用可行性研究提供了支撑.在未来研究中, 进一步开发多环芳烃降解功能基因芯片, 可提升对土壤、水体等环境介质中多环芳烃降解菌相关功能标记物的全面认识.
本文对目前分别从革兰氏阴性细菌和革兰氏阳性细菌中发现的几类不同的代谢功能基因进行了归纳分析(图 4和表 5).在革兰氏阴性细菌中, 以同源性关系为依据对多环芳烃降解基因进行分类, 其中有一大部分是经典的nah-like基因, 这是一类多环芳烃降解通路的上游基因, 其组织和序列相似性都和nah基因相似, 以经典菌株Pseudomonas putida G7为例, 其NAH7质粒上的nah基因簇排列顺序是nahAaAbAcAdBFCQED (Hiroshi et al., 2003).nah-like中不同位点的基因分别编码降解菌作用过程中的不同酶系, 例如nahAa基因编码萘羟基化双加氧酶(NDO)的铁还原蛋白酶, nahAb基因编码NDO的铁还原蛋白, nahAc基因编码α亚基等等(Kamath et al., 2004).这类基因还包括ndo、dox、pah以及pdo基因等等.第二类基因是phd基因, 这是基因簇排序和同源性均不同于nah-like基因的一类基因, 且nah-like基因编码的部分酶的序列不在phd基因簇中.Comamonas teststeroni GZ39菌株发现可以在以菲为唯一碳源的培养基上生长, 含有phd基因来编码相关的多环芳烃分解酶, 但不含nah-like基因, 负责关键降解步骤的酶系即为phd基因, 基因簇排列顺序为phdAbAaBAcAdDE, 其中phdAb编码铁还原蛋白, phdAa编码铁还原蛋白酶, phdB编码脱氢酶, phdAc编码α亚基, phdAd编码β亚基, phdD编码异构酶, phdE编码水合酶-醛缩酶(郑乐等, 2007).第三类基因是nag基因, 该基因与假单胞菌属中经典的nah基因相似, 在已发现的菌株Ralstonia sp.U2中(Tondervik et al., 2011), 该基因簇排列顺序为nagAaGHAbAcAdBFCQED, 除了nagGH的插入外, 其余基因簇与nah基因顺序相同, 功能相近.第四类基因是phn基因, 该基因与nah-like基因的功能相同, 但不具同源性, 在基因排列顺序和DNA序列上均不相同, Burkholderia sp. RP007菌株中新发现的phn基因簇中的排列顺序是phnRSFECDAcAdB (Vanlaere et al., 2008), 其中phnE编码水合酶-醛缩酶, phnAc和phnAd分别编码双加氧酶的α亚基和β亚基.至此, 对以上功能基因特性的研究进一步加深了本研究对多环芳烃分解代谢遗传学的了解.
图 4(Fig. 4)
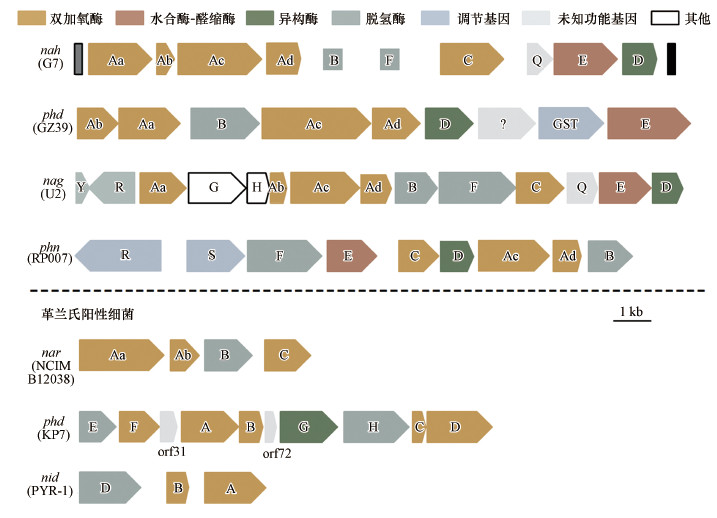 |
| 图 4 不同的多环芳烃降解菌株中的代谢功能基因结构 (Zylstra et al., 1997; Saito et al., 1999; Hiroshi et al., 2003; Kulakov et al., 2005; 郑乐等, 2007; 张丹等, 2010) Fig. 4PAH-catabolic genes′ organization of the different PAHs-degrading bacteria |
表 5(Table 5)
| 表 5 已有的PAHs降解基因 Table 5 Reported functional genes for PAHs degradation | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
表 5 已有的PAHs降解基因 Table 5 Reported functional genes for PAHs degradation
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
在革兰氏阳性细菌中, 多环芳烃降解基因主要有以下几类, 第一类是nar基因, 该类基因在Rhodococcus中较为常见, Rhodococcus是一种经典的可降解多环芳烃等多种外源化合物的革兰氏阳性菌, 目前已分离出多株具有降解功能的Rhodococcus, 例如Rhodococcussp. NCIMB12038、Rhodococcus imtechensis RKJ300等(Kulakov et al., 2005).在Rhodococcus sp. NCIMB12038菌株的基因组中, 双加氧酶基因簇的排列顺序为narAaAbBC (Kulakov et al., 2005), 与nah基因相似.第二类基因是phd基因, 在部分革兰氏阳性降解菌中含有phd降解基因, 例如Mycobacterium sp. SNP11 (Pagnout et al, 2007)、Nocardioides sp. KP7(张丹等, 2010)等.在Nocardioides sp. KP7菌株中, phd基因簇包括两个定位于染色体上的降解基因簇phdIJK和phdEFABGHCD(张丹等, 2010), 但不包含革兰氏阴性细菌的phd基因簇中存在的编码异构酶的基因, 且基因簇排列顺序完全不同(Hiroshi et al., 2003).第三类基因为nid基因, 该基因是在菌株Mycobacterium sp. PYR-1中被首先测序发现的, 这是一株可降解菲、芘、荧蒽、苯并(a)芘等多种多环芳烃污染物的降解菌株, 基因簇排列顺序为nidDBA, 其中nidB基因编码双加氧酶的β亚基, nidA基因编码双加氧酶的α亚基, 且其环羟化双加氧酶(PAHs-RHD)的α亚基和β亚基的基因排序在其他细菌的双加氧酶系统中很少发现(Kumari et al., 2017).环境中还存在着一些其他类型的降解基因簇, 如dbf/fln基因(主要存在于荧蒽降解菌中)、pht基因、fad基因等等(Stingley et al., 2004; Shintani et al., 2007; Eberlein et al., 2013).
与无法直接鉴定土壤中的多环芳烃降解微生物的16S rRNA基因测序方法相比, 多环芳烃降解基因可作为生物标记物更好地了解微生物对污染场地的修复潜力和应用可行性(Liang et al., 2019).在过去一段时间的研究中, 多环芳烃环羟化双加氧酶(PAHs-RHD)基因已被广泛应用为多环芳烃降解菌的生物标记物, 但越来越多研究者发现, 在PAHs-RHD基因中除nahAc和nidA基因的丰度及表达与萘、芘的降解成正相关外, 其他的多环芳烃底物降解与PAHs-RHD基因之间的关系是无规律的, 相关性一般较差(Cébron et al., 2009; Yergeau et al., 2009; Sawulski et al., 2014; Kumari et al., 2017).因此, 用PAHs-RHD基因评估多环芳烃降解潜力存在一定限制.基于此, 有人提出使用水合酶-醛缩酶(Hydratase-Aldolase)基因来作为一种新的多环芳烃降解菌功能性生物标志物, 经验证其具有更好的特异性且能扩增出多种菌株中的水合酶-醛缩酶基因(Liang et al., 2019), 但其应用可行性还有待进一步考察.
4 结论(Conclusions)从多个角度对1983—2019年Web of Science核心数据库中多环芳烃降解基因领域已发表文献的发文趋势、研究热点和进展方向等进行计量可视化分析, 结果表明:有关多环芳烃降解基因的研究领域中, 发文量整体呈持续增长趋势, 且在中后期增长速率加快, 发文主要集中在微生物学、生物技术与应用微生物学和环境科学等学科;在主要的研究国家中, 美国和中国的发文量远超世界其他国家和机构, 但美国研究起步较早, 且与全球各国的合作紧密度远高于中国;该领域近年来的研究热点主要聚集于多环芳烃污染物类型、污染介质、降解修复措施、降解功能菌群的种类和特性、降解基因等五方面的研究.其中, 降解基因聚集圈中出现的关键词频数均少于其他4个研究聚集圈, 但其间关联性很强, 与其他研究聚集圈都有较强的共现关系.降解基因的信息不仅从分子层面揭示了多环芳烃降解微生物的基因表达和功能信息, 还可作为生物标记物来推断和表征微生物对多环芳烃的降解潜力, 为多环芳烃的污染场地修复提供理论支撑.
参考文献
| Ashok B T, Saxena S. 1995. Biodegradation of polycyclic aromatic hydrocarbons-areview[J]. Journal of Scientific & Industrial Research, 54(8): 443-451. |
| Ben Said O, Goni-Urriza M S, El Bour M, et al. 2008. Characterization of aerobic polycyclic aromatic hydrocarbon-degrading bacteria from Bizerte lagoon sediments, Tunisia[J]. Journal of Applied Microbiology, 104(4): 987-997. DOI:10.1111/j.1365-2672.2007.03621.x |
| Cébron A, Beguiristain T, Faure P, et al. 2009. Influence of vegetation on the in situ bacterial community andpolycyclic aromatic hydrocarbon (PAH) degraders in aged PAH-contaminated or thermal-desorption-treated soil[J]. Applied and Environmental Microbiology, 75: 6322-6330. DOI:10.1128/AEM.02862-08 |
| 陈亚婷, 姜博, 邢弈, 等. 2018. 基于非培养手段的多环芳烃降解微生物解析[J]. 中国环境科学, 38(9): 3562-3575. DOI:10.3969/j.issn.1000-6923.2018.09.043 |
| Csiszar S A, Diamond M L, Daggupaty S M. 2014. The magnitude and spatial range of current-use urban PCB and PBDE emissions estimated using a coupled multimedia and air transport model[J]. Environmental Science & Technology, 48: 1075-1083. |
| De Sousa S T P, Cabral L, Lacerda G V, et al. 2017. Diversity of aromatic hydroxylating dioxygenase genes in mangrove microbiome and their biogeographic patterns across global sites[J]. Microbiology Open, 6: 4-18. |
| Duarte M, Nielsen A, Camarinha-Silva A, et al. 2017. Functional soil metagenomics:elucidation of polycyclic aromatic hydrocarbon degradation potential following 12 years of in situ bioremediation[J]. Environmental Microbiology, 19: 2992-3011. DOI:10.1111/1462-2920.13756 |
| Eberlein C, Estelmann S, Seifert J, et al. 2013. Identification and characterization of 2-naphthoyl-coenzyme a reductase, the prototype of a novel class of dearomatizing reductases[J]. Scientific Reports, 88(5): 1032-1039. |
| 樊一阳, 许京京. 2015. 基于CiteSpace文献计量法的石墨烯研究文献可视化图谱分析[J]. 现代情报, 35(8): 81-99. DOI:10.3969/j.issn.1008-0821.2015.08.017 |
| Fang T T, Pan R S, Jiang J, et al. 2016. Effect of salinity on community structure and naphthalene dioxygenase gene diversity of a halophilic bacterial consortium[J]. Frontiers of Environmental Science & Engineering, 10(6): 16. |
| Ghosal D, Ghosh S, Dutta T K, et al. 2016. Current state of knowledge in microbial degradation of polycyclic aromatic hydrocarbons (PAHs):A review[J]. Frontiers in Microbiology, 7(1369): 1-27. |
| Grba N, Kr?mar D, Isakovski M K, et al. 2016. Priority substances in sediments of the "Carska Bara" special nature reserve, a natural scientific research area on the UNESCO list[J]. Journal of Environmental Management, 182: 149-159. |
| Guo G, He F, Tian F, et al. 2016. Effect of salt contents on enzymatic activities and halophilic microbial community structure during phenanthrene degradation[J]. International Biodeterioration Biodegradation, 110: 8-15. DOI:10.1016/j.ibiod.2016.02.007 |
| Han X M, Liu Y R, Zheng Y M, et al. 2014. Response of bacterial pdo1, nah, and C12O genes to aged soil PAH pollution in a coke factory area[J]. Environmental Science & Pollution Research International, 21(16): 9754-9763. |
| Haritash A K, Kaushik C P. 2009. Biodegradation aspects of polycyclic aromatic hydrocarbons (PAHs):a review[J]. Journal of Hazardous Materials, 169(1/3): 1-15. |
| Heitkamp M A, Cerniglia C E. 1988. Mineralization of polycyclic aromatic hydrocarbons by a bacterium isolated from sediment below an oil-field[J]. Applied and Environmental Microbiology, 54: 1612-1614. DOI:10.1128/AEM.54.6.1612-1614.1988 |
| Hiroshi H, Toshio O. 2003. Genetics of polycyclic aromatic hydrocarbon metabolism in diverse aerobic bacteria[J]. Bioscience, Biotechnology, and Biochemistry, 67(2): 225-243. DOI:10.1271/bbb.67.225 |
| Huang Y P, Xun S, Min L, et al. 2019. A multimedia fugacity model to estimate the fate and transport of polycyclic aromatic hydrocarbons (PAHs) in a largely urbanized area, Shanghai, China[J]. Chemosphere, 217: 298-307. DOI:10.1016/j.chemosphere.2018.10.172 |
| Kamath R, Schnoor J L, Alvarez P J J. 2004. Effect of root-derived substrates on the expression of nah-lux genes in pseudomonas fluorescens HK44:Implications for PAH biodegradation in the rhizosphere[J]. Environmental Science and Technology, 38(6): 1740-1745. DOI:10.1021/es0306258 |
| Kulakov L A, Chen S C, Allen C C R, et al. 2005. Web-type evolution of Rhodococcus gene clusters associated with utilization of naphthalene[J]. Applied and Environmental Microbiology, 71(4): 1754-1764. DOI:10.1128/AEM.71.4.1754-1764.2005 |
| Kumari S, Regar R K, Bajaj A, et al. 2017. Simultaneous biodegradation of polyaromatic hydrocarbons by a Stenotrophomonas sp:characterization of nid genes and effect of surfactants on degradation[J]. Indian Journal of Microbiology, 57(1): 60-67. DOI:10.1007/s12088-016-0612-6 |
| 李杰, 陈超美. 2017. CiteSpace:科技文本挖掘及可视化[M]. 北京: 首都经济贸易大学出版社, 2-5. |
| Li Y X, Duan X Y. 2015. Polycyclic aromatic hydrocarbons in sediments of China Sea[J]. Environmental Science and Pollution Research, 22(20): 15432-15442. DOI:10.1007/s11356-015-5333-6 |
| Liang C Y, Huang Y, Wang H. 2019. pahE, a functional marker gene for polycyclic aromatic hydrocarbon-degrading bacteria[J]. Applied and Environmental Microbiology, 85: 3-21. |
| Ligaray M, Baek S S, Kwon H O, et al. 2016. Watershed-scale modeling on the fate and transport of polycyclic aromatic hydrocarbons (PAHs)[J]. Journal of Hazardous Materials, 320: 442-457. DOI:10.1016/j.jhazmat.2016.08.063 |
| Liu T T, Xu Y, Liu H, et al. 2011. Functional characterization of a gene cluster involved in gentisate catabolism in Rhodococcus sp. strain NCIMB 12038[J]. Applied Microbiology and Biotechnology, 90(2): 671-678. DOI:10.1007/s00253-010-3033-1 |
| Lu X Y, Zhang T, Fang H H. 2011. Bacteria-mediated PAH degradation in soil and sediment[J]. Applied Microbiology and Biotechnology, 89(5): 1357-1371. DOI:10.1007/s00253-010-3072-7 |
| Lucio-Arias D, Leydesdorff L. 2008. Main-path analysis and path-dependent transitions in HistCite-based historiograms[J]. Journal of the American Society for Information Science and Technology, 59(12): 1948-1962. DOI:10.1002/asi.20903 |
| Ma W L, Liu L Y, Tian C G, et al. 2015. Polycyclic aromatic hydrocarbons in Chinese surface soil:Occurrence and distribution[J]. Environmental Science and Pollution Research, 22: 4190-4200. DOI:10.1007/s11356-014-3648-3 |
| Obrista D, Zielinskaa B, Perlinger J A. 2015. Accumulation of polycyclic aromatic hydrocarbons (PAHs) and oxygenated PAHs (OPAHs) in organic and mineral soil horizons from four U.S. remote forests[J]. Chemosphere, 134: 98-105. DOI:10.1016/j.chemosphere.2015.03.087 |
| Ouyang W, Wang Y D, Lin C Y, et al. 2018. Heavy metal loss from agricultural watershed to aquatic system:A scientometrics review[J]. Science of the Total Environment, 637-638: 208-220. DOI:10.1016/j.scitotenv.2018.04.434 |
| Pagnout C, Frache G, Poupin P, et al. 2007. Isolation and characterization of a gene cluster involved in PAH degradation in Mycobacterium sp. strain SNP11:Expression in Mycobacterium smegmatismc2155[[J]. Research in Microbiology, 158(2): 175-186. DOI:10.1016/j.resmic.2006.11.002 |
| Renner R. 1999. EPA to strengthen persistent, bioaccumulative, and toxic pollutant controls-Mercury first to be targeted[J]. Environmental Science & Technology, 33(3): 62A. |
| Diamond M L, Hodge E. 2007. Urban contaminant dynamics:from source to effect[J]. Environmental Science & Technology, 41: 3796-3805. |
| Saito A, Iwabuchi T, Harayama S. 1999. Characterization of genes for enzymes involved in the phenanthrene degradation in Nocardioides sp. KP7[J]. Chemosphere, 38(6): 1331-1337. DOI:10.1016/S0045-6535(98)00534-7 |
| Sawulski P, Clipson N, Doyle E. 2014. Effects of polycyclic aromatic hydrocarbons on microbial community structure and PAH ring hydroxylating dioxygenase gene abundance in soil[J]. Biodegradation, 25: 835-847. DOI:10.1007/s10532-014-9703-4 |
| Schuler L, Jouanneau Y, Chadhain S M N, et al. 2009. Characterization of a ring-hydroxylating dioxygenase from phenanthrene-degrading Sphingomonas sp. strain LH128able to oxidize benz a anthracene[J]. Applied Microbiology and Biotechnology, 83(3): 465-475. DOI:10.1007/s00253-009-1858-2 |
| Shen H Z, Huang Y, Wang R, et al. 2013. Global atmospheric emissions of polycyclic aromatic hydrocarbons from 1960 to 2008 and future predictions[J]. Environmental Science & Technology, 47: 6415-6424. |
| Shintani M, Urata M, Inoue K, et al. 2007. The Sphingomonas plasmid pCAR3 is involved in complete mineralization of carbazole[J]. Journal of bacteriology, 189(5): 2007-2020. DOI:10.1128/JB.01486-06 |
| Stingley R L, Brezna B, Khan A A, et al. 2004. Novel organization of genes in a phthalate degradation operon of Mycobacterium vanbaalenii PYR-1[J]. Microbiology-SGM, 150: 3749-3761. DOI:10.1099/mic.0.27263-0 |
| Sun S S, Wang H Z, Chen Y Z, et al. 2019. Salicylate and phthalate pathways contributed differently on phenanthrene and pyrene degradations in Mycobacterium sp. WY10[J]. Journal of Hazardous Materials, 364: 509-518. DOI:10.1016/j.jhazmat.2018.10.064 |
| Teng Y, Chen W. 2019. Soil microbiomes-a promising strategy for contaminated soil remediation:A review[J]. Pedosphere, 29(3): 283-297. DOI:10.1016/S1002-0160(18)60061-X |
| Thiombane M, Albanese S, Di Bonito M, et al. 2019. Source patterns and contamination level of polycyclic aromatic hydrocarbons (PAHs) in urban and rural areas of Southern Italian soils[J]. Environmental Geochemistry and Health, 41: 507-528. DOI:10.1007/s10653-018-0147-3 |
| Tondervik A, Bruheim P, Berg L, et al. 2012. Ralstonia sp. U2 naphthalene dioxygenase and Comamonas sp. JS765 nitrobenzene dioxygenase show differences in activity towards methylated naphthalenes[J]. Journal of Bioscience and Bioengineering, 113(2): 173-178. DOI:10.1016/j.jbiosc.2011.10.001 |
| Tuhuloula A, Suprapto S, Altway A, et al. 2019. Biodegradation of extractable petroleum hydrocarbons by consortia Bacillus cereusand Pseudomonas putidain petroleum contaminated-soil[J]. Indonesian Journal of Chemistry, 19(2): 347-355. DOI:10.22146/ijc.33765 |
| Vanlaere E, van der Meer J R, Falsen E, et al. 2008. Burkholderia sartisoli sp., isolated from a polycyclic aromatic hydrocarbon-contaminated soil[J]. International Journal of Systematic and Evolutionary Microbiology, 58: 420-423. DOI:10.1099/ijs.0.65451-0 |
| Wang R F, Chen X Y, Yang Q X. 2018. Evolution of functional bacteria in a polycyclic aromatic hydrocarbon (PAH)-degrading bioreactor[J]. Water Environment Research, 90(12): 2022-2140. DOI:10.2175/106143017X15131012188259 |
| 吴健, 王敏, 靳志辉, 等. 2016. 基于Web of Science大数据的土壤环境中多环芳烃研究文献计量分析[J]. 上海环境科学, (1): 9-16. |
| Yergeau E, Arbour M, Brousseau R, et al. 2009. Microarray and real-time PCR analyses of theresponses of high-arctic soil bacteria to hydrocarbon pollution andbioremediation treatments[J]. Applied and Environmental Microbiology, 75: 6258-6267. DOI:10.1128/AEM.01029-09 |
| 张丹, 李兆格, 包新光, 等. 2010. 细菌降解萘、菲的代谢途径及相关基因的研究进展[J]. 生物工程学报, 26(6): 726-734. |
| Zhao B, Wang H, Mao X, et al. 2009. Biodegradation of phenanthrene by a halophilic bacterial consortium under aerobic conditions[J]. Current Microbiology, 58(3): 205-210. DOI:10.1007/s00284-008-9309-3 |
| 郑乐, 刘宛, 李培军. 2007. 多环芳烃微生物降解基因的研究进展[J]. 生物学杂志, 26(3): 449-454. |
| Zhu Q G, Wu Y C, Zeng J, et al. 2019. Influence of bacterial community composition and soil factors on the fate of phenanthrene and benzo[a]pyrene in three contrasting farmland soils[J]. Environment Pollution, 247: 229-237. DOI:10.1016/j.envpol.2018.12.079 |
| 朱宇恩, 张倩茹, 张维荣, 等. 2017. 基于文献计量分析的Cr污染土壤修复发展历程剖析(2001-2015年)[J]. 农业环境科学学报, 36(3): 409-419. |
| Zylstra G J, Kim E, Goyal A K. 1997. Comparative molecular analysis of genes for polycyclic aromatic hydrocarbon degradation[J]. Genetic Engineering, 19: 257. |
