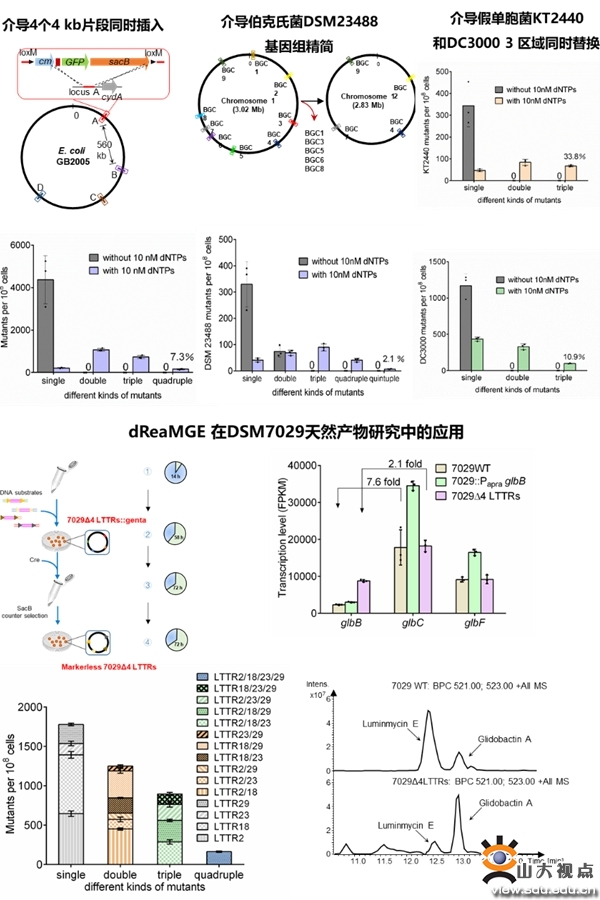
微生物基因组中多基因或途径的删除、替换和整合有助于释放微生物的巨大应用潜力。Red/ET重组工程是实现细菌基因组精确编辑的重要工具,该论文在过去工作基础之上首次发现细菌胞内dNTP水平对于重组工程的重要影响,并通过连续优化介导Red重组工程技术的重要参数,比如引物修饰、复苏时间、抗性标记使用策略、过表达核糖核苷酸还原酶以及添加外源dNTP等,建立了基于dsDNA重组工程的细菌多元基因组编辑方法(dReaMGE),该技术能在大肠杆菌、假单胞菌和伯克氏菌种中同时介导多个千碱基规模的序列替换、缺失和插入,与现有的多重基因组编辑技术相比具有不引发或者依赖双链断裂,无插入基因长度限制,不需要对宿主基因组进行预修饰等优势,能够高效地对多种细菌进行基因组精简、基因簇多转录单元激活以及代谢网络改造等工作。dReaMGE技术极大地简化了细菌基因组多元编辑的流程,是对现有基因组编辑技术的补充,扩展了原核基因组工程技术库,解决了dsDNA重组工程在多重基因组编辑技术中的缺失,对原核基因组工程具有广泛的意义。
卞小莹教授课题组主要从事微生物基因组编辑技术、天然产物与合成生物学研究,自2015年加入国重室以来,构建了富含基因簇非模式菌的基因组编辑技术,开发了新型天然产物底盘,解析了脂肽生物合成中的脂链选择机制并应用于结构理性改造,从而建立了以伯克氏菌为核心的基因簇挖掘与改造体系,推动了天然产物合成生物学的创新发展。近五年已在Nat. Commun.、PNAS.、Angewandte Chemie、Nucleic Acids Res等期刊上发表通讯作者论文20余篇。
张友明教授研发的Red/ET同源重组技术是世界上大片段基因克隆及改造研究领域的领跑者,也是具有知识产权的、在世界上具有国际竞争力的优势技术。他的团队在Red/ET重组工程发展及应用相关研究工作已经发表论文120余篇(其中有12篇Nature及其系列论文发表)。因此2018-2020年连续三年被评为中国高被引****,2019年被选为欧洲科学院院士,2020年被选为德国工程院院士。
该论文由山东大学和德国德累斯顿大学等单位的相关****合作完成。该项研究工作得到了国家重点研发计划、国家自然科学基金、国家博士后基金、山东省****基金与山东大学青年交叉科学创新群体等项目的资助。微生物技术国家重点实验室生命科学共性研究技术平台为本工作提供了重要技术支持。
论文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkab1076/6430846
