

中国药科大学 生命科学与技术学院, 江苏 南京 210000
收稿日期:2020-04-15;接收日期:2020-06-30;网络出版时间:2020-08-13
基金项目:国家自然科学基金(No. 81800512),江苏省自然科学基金(No. BK20180554)资助
摘要:长链非编码RNA (Long non-coding RNA, lncRNA)因参与多个层级上的生物进程而成为当下生命科学领域的研究热点。LncRNA可以与DNA、RNA和蛋白质等生物分子结合, 并进一步影响靶基因的转录、翻译以及翻译后修饰等过程, 从而发挥在细胞生理代谢过程中的调控作用。目前研究显示, lncRNA通过多种途径在肝脏代谢中发挥重要作用。文中以lncRNA的功能及其与肝脏能量代谢和相关疾病的关系为着眼点, 阐述了lncRNA发挥作用的机制以及未来的研究前景。
关键词:长链非编码RNA肝脏糖代谢脂代谢
lncRNA in hepatic glucose and lipid metabolism: a review
Xiaoxiao Chen, Chen Sun, Chang Liu, Jie Wu


School of Life Science & Technology, China Pharmaceutical University, Nanjing 210000, Jiangsu, China
Received: April 15, 2020; Accepted: June 30, 2020; Published: August 13, 2020
Supported by: National Natural Science Foundation of China (No. 81800512), Natural Science Foundation of Jiangsu Province, China (No. BK20180554)
Corresponding author: Jie Wu. Tel/Fax: +86-25-83271242; E-mail: wujie@cpu.edu.cn.
Abstract: In recent years, long non-coding RNA (lncRNA) has been proved to be involved in the regulation of biological processes at various levels, attracting research interests in life science. LncRNA possesses the unique capability and exert discrete effects on transcription, translation and post-translational modification of the target genes through interacting with DNA, RNA and protein. Current studies have revealed that lncRNA plays an important role in hepatic metabolism via diverse pathways. This review focuses on the function of lncRNA and its relationship with hepatic energy metabolism and the correlated diseases, to elucidate the underlying mechanisms and prospects of lncRNA researches.
Keywords: lncRNAliverglucose metabolismlipid metabolism
人类基因组中, 大约有93%的DNA可以被转录成为RNA, 但其中只有2%的RNA具有蛋白质编码功能, 其余98%的RNA均为非编码RNA (Non-coding RNA, ncRNA)[1]。根据长度不同, 大于200 nt的ncRNA被称为lncRNA, 短于200 nt的则被归为小非编码RNA (Small ncRNAs, sncRNA)。得益于全基因组测序技术和高通量测序技术高速发展, 大量lncRNA分子被发现, 科研工作者开始重新认识这一基因组中的“暗物质”[2]。相关报道显示, 与短链的sncRNA不同, lncRNA可以形成复杂的高级结构, 核苷酸长链中承载着更多的生物信息, 具有表观遗传学调控、转录调控、转录后调控、翻译调控及翻译后调控等多个层次的调控能力[3]。随着研究的深入, 科研工作者发现lncRNA在癌症、代谢性疾病、神经退行性疾病等多种疾病中发挥着重要的作用[4]。因此, lncRNA也逐渐成为生命科学领域内研究的热点。
近年来, lncRNA在代谢性疾病中发挥的作用逐渐受到重视。在糖尿病、高血脂症、非酒精性脂肪肝等疾病中, lncRNA表达模式与疾病的发展进程密切相关[5]。肝脏作为人体重要的代谢器官, 对人体稳态的维持极为重要, 研究lncRNA在肝脏代谢中起到的作用, 将为日后相关代谢性疾病的诊断、治疗和预后分析提供重要依据和全新方法。因此, 本文将对lncRNA的作用机制及其在肝脏糖脂代谢中的调控模式和相关靶点进行全面综述, 以期为后续****开展相关研究提供思路。
1 LncRNA的产生、分类及功能LncRNA曾被认为是基因组中的“暗物质”, 在发现之初并没有引起研究者们的重视, 例如H19和Xist在前基因组时代就被发现, 但直到21世纪初, 与之相关的研究才陆续见诸报道。在该领域的研究初始阶段, 研究者普遍认为lncRNA不具有编码蛋白质的能力, 故将这一类RNA称为非编码RNA, 但随着研究深入, 研究者开始发现, 一些lncRNA具有小的开放阅读框(Open reading frame, ORF), 可翻译出短肽, 这些来自lncRNA的短肽同样被认为具有调控其他蛋白的能力。LncRNA的来源主要包括以下5种:(1)由结构中断的编码基因形成的lncRNA;(2)染色质重组时产生的lncRNA;(3)在复制过程中反移位形成的lncRNA;(4)与局部复制子串联产生的lncRNA;(5)基因中插入转座子形成的lncRNA[6]。与mRNA相似, lncRNA的转录同样由RNA聚合酶Ⅱ完成, 经过剪切形成成熟的lncRNA, 具有“5′帽子结构”和“3′poly A尾巴”, 同一基因可以形成不同转录本的lncRNA。与mRNA比较而言, lncRNA种类更加繁多, 但其表达水平却远低于mRNA。目前研究发现, 多个物种基因组内均存在lncRNA, 但与mRNA相比, lncRNA在不同物种之间的保守性相对较差。
LncRNA的分类通常基于其功能和基因组中与附近基因的位置关系。其种类可被分为:(1)基因间lncRNA, 这类lncRNA位于两个蛋白质编码基因的中间, 其转录活性同样受到表观遗传的调控。(2)内含子lncRNA, 这类lncRNA来自某个基因的DNA序列内部, 与该基因内含子的正义链或反义链的部分序列相同。与基因间lncRNA相比, 内含子lncRNA的保守性更差。(3)正义lncRNA。(4)反义lncRNA。(3)和(4)这两类lncRNA转录自蛋白质编码基因的外显子, 既可以包括整个蛋白编码基因, 也可以与该基因部分重叠。(5)增强子lncRNA, 这一类lncRNA转录自蛋白质编码基因的启动子区域[7]。
与微小RNA (miRNA)不同, lncRNA具有复杂的二级结构和三级结构, 可以在组成蛋白复合物的过程中起到“支架”的作用, 实现不同信号通路之间信息的交叉和整合, 或者与单独的蛋白质进行结合, 在翻译后水平调控蛋白质的功能和(或)稳定性。目前, 随着高通量测序技术的发展, 对lncRNA的二级结构的检测也成为可能。RNA结构探针技术的应用揭示了部分较为重要的lncRNA的二级结构, 例如, HOX基因座转录而来的反义RNA (HOX tran antisense RNA, HOTAIR)具有4个结构域, 由36个螺旋、38个锥形、34个内部环和19个连接区组成, 进化上保守;核富集常染色体转录本1 (Nuclear-enriched abundant transcript 1, NEAT1)形成4个结构域, 并且NEAT1_L (NEAT1其中一个亚型)的5′末端和3′末端之间存在RNA-RNA相互作用[8]。上述研究结果表明, lncRNA通常折叠成多个结构域, 不同结构域对其功能的正常行使具有重要作用。LncRNA的结构生物学研究将有助于lncRNA的作用机制和调控过程的深入探究。
LncRNA与基因组DNA进行结合, 通常是通过与基因启动子上顺式作用元件的结合调控基因的转录, 这一类lncRNA通常与基因5′端边界的序列重复, 基本不包含基因的外显子序列。一方面, lncRNA可以与DNA的启动子区域结合, 形成DNA-RNA三链杂合片段, 阻止转录因子开启转录过程, 进而抑制基因的表达;另一方面, lncRNA在与DNA的结合过程中充当“连接工具”, 将具有转录调控功能的蛋白质复合物募集至DNA的启动子区段, 发挥起始染色质重构、激活或抑制转录起始以及通过组蛋白的修饰进行表观遗传学调控等功能[9]。
基于lncRNA本身的核酸结构, lncRNA与mRNA和miRNA之间的结合变得相对容易。通过与mRNA的结合, lncRNA可以调控mRNA的剪切、拼接、细胞内分布以及稳定性。而lncRNA与miRNA之间的结合通常更像是“海绵”的吸附作用, 减少miRNA与靶基因之间的结合, 降低miRNA对其靶基因的抑制效果[10]。
此外, 如前文所述, 部分lncRNA具有较短的ORF, 可以翻译出短肽。例如, LINC00961作为lncRNA, 可以翻译出由90个氨基酸构成的多肽SPAR, 具有抑制肌肉再生的功能[11]。
总而言之, lncRNA在多种不同层次的水平上参与基因表达的调控, 包括表观遗传的修饰、转录调控、RNA剪接、核穿梭、转录后水平、翻译水平以及翻译后水平, 基本贯穿体内目前已知的所有调控水平, 值得注意的是, 目前尚有多种未知调控机制我们仍不了解, 对lncRNA的研究更待进一步的深入。
2 LncRNA与肝脏能量代谢肝脏作为人体最重要的器官之一, 在维持机体能量代谢中发挥着重要作用。在糖代谢方面, 肝脏参与糖原分解、合成以及糖异生等过程以维持血糖水平。在脂代谢方面, 肝脏参与了脂质的合成和转运, 由于肝脏不属于体内脂质储存器官, 其合成的甘油三酯和磷脂、胆固醇与载脂蛋白形成极低密度脂蛋白, 通过血液被转运至周身器官或储存于脂肪组织之中。因此, 肝脏在维持机体糖脂代谢稳态中发挥着重要的作用。
2.1 糖代谢肝脏在维持机体血糖浓度中发挥重要作用, 空腹时, 机体血糖较低, 肝脏促进肝糖原分解和糖异生来提高血糖浓度;餐后, 血糖处于较高水平, 肝脏可促进糖原合成, 抑制糖原分解和糖异生来降低血糖。近年来发现, lncRNA在肝脏糖代谢中起到重要作用。
H19作为一个多功能的长链非编码RNA, 在细胞核和细胞质中均可行使功能, Goyal等[12]在对db/db小鼠进行转录组测序时检测出3种表达水平显著性下降的lncRNA, 其中H19的变化最为剧烈。在肝癌细胞HpG2和原代肝细胞中抑制H19可以促进糖异生基因葡萄糖-6-磷酸酶(Glucose-6-phosphatase, G6pc)、磷酸烯醇式丙酮酸羧激酶(Recombinant Phosphoenolpyruvate Carboxykinase 1, Pck)的表达并增加细胞的葡萄糖生成量。体内实验表明, 利用siRNA抑制小鼠肝脏中H19的表达会引起小鼠的高血糖症和高胰岛素血症[13]。更进一步的研究发现, 在HpG2中抑制H19的表达可以提高叉头转录因子(Forkhead transcription factor O1, FoxO1)的表达并影响其亚细胞移位, 促使更多的FoxO1转位入核, 调控糖异生相关基因的表达。而Zhang等[14]的研究发现, 在饥饿条件下, 小鼠组织中的H19的表达水平显著升高;此外, 在高脂饮食喂养(High-fat diet, HFD)小鼠和Ⅱ型糖尿病患者的肝脏组织中, H19表达都呈现上升趋势;体外实验显示, 在HpG2细胞中干扰H19的表达后, 细胞葡萄糖输出降低。这一结果与上述研究结果完全相反。相应的体内实验结果显示, 在肝脏特异性过表达H19会造成小鼠胰岛素抵抗进而影响体内的葡萄糖稳态。研究[14]发现, 这一现象的产生与H19对基因的甲基化调控相关, H19可以与S-腺苷高半胱氨酸水解酶结合, 抑制该酶的活性, 造成S-腺苷高半胱氨酸的积累, 进而抑制DNA甲基转移酶。在小鼠肝脏组织中, H19的升高会减少阻止肝细胞核因子-4α (Hepatocyte nuclear factor 1A, Hnf4α)启动子上CpG岛的甲基化, 促进其表达。随后, 被激活的HNF4a开启机体的糖异生过程。上述两项研究分别在不同的动物模型和相同的细胞模型中得到完全相反的实验结果, 造成这一矛盾的原因目前尚无法阐明。就目前的数据讨论, 二者研究的不同点在于, Zhang等[14]的研究中使用了包括饥饿小鼠模型、HFD小鼠模型在生理和病理条件下进行论证, 而Goyal等[13]的实验中使用db/db小鼠进行研究, 且其实验结果中H19的表达变化趋势与糖尿病患者体内H19的变化趋势相同, 具有更好的参考价值。
研究发现, 母本表达基因3 (Maternally expressed gene 3, MEG3)在HFD和ob/ob小鼠的肝脏组织以及使用棕榈酸、油酸和亚油酸刺激的原代肝脏细胞中表达显著升高。针对这一发现研究者从表观遗传学的角度进行研究, 发现组蛋白去乙酰化酶HDAC家族的成员HDAC1和HDAC3的表达在棕榈酸刺激的原代肝脏细胞中显著降低, 而使用HDAC的抑制剂Trichostatin A则剂量依赖性地提高MEG3的表达。而使用腺病毒在原代肝细胞中过表达MEG3促进FoxO1及其下游基因G6pc和Pck的表达。一系列的实验表明, 肝细胞中的MEG3受脂质增多的影响被激活, 促进糖异生基因的表达。在HFD小鼠和ob/ob小鼠中抑制MEG3的表达虽然没有改变小鼠的体重, 但缓解了肥胖小鼠葡萄糖耐量受损的状况[15]。另有研究表明, 胰高血糖素可诱导肝细胞中MEG3的表达。在胰高血糖素的刺激下, MEG3被环磷腺苷效应元件结合蛋白(cAMP- response element binding protein, CREB)激活, 行使内源竞争RNA (Competing endogenous RNA, ceRNA)的功能, 与miR-302a-3p结合并间接激活其靶基因CREB转录共激活因子(CREB- regulated transcription coactivator, Crtc)的家族成员Crtc2的表达, 进而促进肝脏糖异生相关的转录共激活因子Pgc-1α及其下游的糖异生相关基因G6pc和Pck的表达[16]。除此之外, MEG3作为ceRNA, 还可以与miR-214结合, 降低miR-214对活化转录因子4 (Activating transcription factor 4, Atf4)的抑制作用, 提高Atf4的转录。Atf4作为转录辅激活因子, 与FoxO1结合并提高FoxO1的转录活性, 进而激活机体的糖异生[17]。随着对MEG3研究的深入, Chen等[18]在最新的研究中发现, 除了对糖异生进程的调控作用外, MEG3还可以对肝脏组织的胰岛素敏感性进行调控, 影响胰岛素介导的肝细胞对葡萄糖的摄取。此外, MEG3可以结合miR-185-5p, 减弱miR-185-5p对早期生长反应蛋白2 (Early growth response proteins-2, Egr2) mRNA的抑制作用, EGR2通过对胰岛素受体底物的抑制, 促进了肝脏的胰岛素抵抗, 降低肝细胞对葡萄糖的吸收。
与MEG3相似, Gomafu也是一种在饥饿小鼠、HFD小鼠和db/db小鼠中表达量显著升高的长链非编码RNA。研究发现, 在肥胖小鼠中, NF-κB家族成员p65与Gomafu启动子区域的结合增多, 促进了Gomafu的转录, 造成Gomafu在肥胖小鼠肝脏组织中的表达升高。相反, 抑制肥胖小鼠肝脏中Gomafu的表达则可以降低小鼠的糖异生进程, 改善小鼠的胰岛素敏感性。与大部分lncRNA的功能相同, Gomafu在对糖异生进程的调控过程中同样起着ceRNA的作用, 通过与miR-139结合缓解miR-139对FoxO1的抑制作用[19]。
LncRNA抑制肝脏糖异生和脂代谢(LncRNA suppressor of hepatic gluconeogenesis and lipogenesis, LnsSHGL)是在肝脏组织中高表达lncRNA, Wang等[20]发现, 小鼠lncSHGL及其人类同源序列B4GALT1-AS1分别在肥胖小鼠(HFD和db/db小鼠)和非酒精性脂肪肝患者的肝脏组织中表达降低。进一步的实验表明, lncSHGL可以招募核不均一蛋白A1 (Heterogeneous nuclear ribonucleoprotein A1, hnRNPA1), 加速钙调蛋白(Calmodulin, CaM)的翻译, 以此增加CaM的蛋白表达。随着CaM蛋白水平上升, 磷脂酰肌醇3-激酶(PI3K)/Akt途径被激活, 抑制机体糖异生进程。
LncRNA葡萄糖激酶阻遏物(Liver GCK repressor, LncLGR)在小鼠全身的表达丰度较低, 但肝脏中的LGR在饥饿条件下被显著激活并随着饥饿后的再进食而恢复到正常的表达水平。进一步的研究发现, LGR主要分布于肝细胞核中, 在受到营养信号刺激后, 细胞核中的LGR在与hnRNPL结合的同时将其募集到葡萄糖激酶(Glucokinase, GCK)的启动子上, 抑制Gck的转录, 降低体内的糖酵解水平, 促进糖原的积累[21]。
LincIRS2是定位于细胞核的lncRNA。研究显示, 其表达水平可以积极响应外界营养信号的变化, 在饮食诱导的肥胖症(DIO)的小鼠肝脏组织中lincIRS2的表达显著下降, 而在野生型小鼠中, lincIRS2的表达随着小鼠禁食和再进食的处理而呈现先升后降的变化。进一步研究发现, 转录因子小Maf蛋白MAFG通过与lincIRS2启动子的结合抑制其表达, 在饥饿条件下, MAFG受到核因子红系-2相关因子(NRF1/NFE2L1)招募, 与其形成异二聚体, 从而减少MAFG在lincIRS2启动子区域富集, 促进lincIRS2的转录。而CRISPR/Cas9和RNAi介导的lincIRS2缺失会导致瘦小鼠血糖升高, 胰岛素抵抗和异常葡萄糖输出。相反, 当激活lincIRS2时, 小鼠即使体重增加但也同样保持健康的血糖水平。综上所述, MAFG-lncRNA轴在生理和病理条件下起到了调控肝葡萄糖代谢的作用[22]。
笔者课题组研究发现, 小鼠禁食16 h后, GM10768在肝脏中表达显著升高, 并随着小鼠再进食而恢复到正常的表达水平。此外, 研究发现GM10768在db/db小鼠肝脏组织中表达升高。进一步的研究发现, 在原代肝细胞中过表达GM10768后, 细胞的葡萄糖输出增加。相反, 在db/db小鼠中干扰GM10768可以抑制肝脏糖异生, 并缓解小鼠的高血糖症。实验结果证明, 与MEG3相同, GM10768在肝脏组织中发挥ceRNA的功能, 结合并抑制miR-214, 促进小鼠肝脏糖异生作用[23]。
哺乳动物中通过多种机制严格控制葡萄糖水平以满足全身能量需求, 目前研究已揭示了lncRNA在肝脏糖代谢中发挥重要作用。表 1列举了文中所提到的肝脏糖代谢相关lncRNA, 不难发现:FoxO1作为胰岛β细胞调控网络的一个关键因子, 可促进肝葡萄糖生成, 同时也是糖代谢中研究较多的miRNA的靶基因。而lncRNA在体内可作为RNA海绵, 吸附miRNA, 进一步影响miRNA靶基因的作用。另外, lncRNA可与RNA结合蛋白结合, 形成功能轴并作用于糖代谢相关基因, 从而调节糖原合成或分解。
表 1 肝脏中参与糖代谢的lncRNA及相关靶点和作用Table 1 Functional lncRNAs involved in hepatic glucose metabolism
| Name | Target | Function | Reference |
| H19 | FoxO1 | FoxO1↓, G6pase↓, Pck1↓, PC↓ | [12] |
| MEG3 | miR-302a-3p | miR-302a-3p↓, CRTC2↑, G6pase↑, Pck1↑ | [16] |
| miR-214 | miR214↓, ATF4↑, FoxO1↑, G6pase↑, Pck1↑, IR↑ | [17] | |
| miR-185-5p | miR-185-5p↓, Egr2↑, IR↑ | [18] | |
| Gomafu | miR-139-5p | miR-139-5p↓, FoxO1↑, G6pase↑, Pck1↑, IR↑ | [19] |
| SHGL | hnRNPA1 | hnRNPA1↓, CALM↑, (PI3K)/Akt/FoxO1↑, G6pase↓, Pck1↓ | [20] |
| LGR | hnRNPL | hnRNPL↓, GCK↓, G6pase↑, Pck1↑ | [21] |
| LincIRS2 | MAFG | G6pc↓, Pck1↓, FoxO1↓ | [22] |
| Gm10768 | miR-214 | miR214↓, ATF4↑, G6pase↑, Pepck↑ | [23] |
表选项
2.2 脂代谢肝脏在维持人体脂代谢稳态中行使重要功能, 肝脏脂代谢紊乱会引起非酒精性脂肪肝(NAFLD)、非酒精性肝炎, 甚至发展成为肝硬化和肝癌, 但轻度脂肪肝多无临床症状, 在临床上不易诊断, 而lncRNA在正常人和肝脏脂代谢异常者之间的差异性表达, 未来将lncRNA发展成为该类疾病的生物标志物或医学检测靶点前景可观。
Lnc uc372在NAFLD病人、HFD以及db/db小鼠的肝脏组织中表达升高。肝脏中的uc372主要分布于细胞核中, 通过与miR-195和miR-4668初级转录物(pri-miR-195和pri-miR-4668)结合抑制其剪切修饰而变为成熟的miRNA, 间接激活脂质合成相关基因胆固醇调节元件结合蛋白(Srebp1)、乙酰辅酶A羧化酶(Acetyl CoA carboxylase, Acc)和脂肪酸合成酶(Fatty acid synthase, Fas)以及脂质摄取相关蛋白CD36的表达, 造成肝脏脂质堆积[24]。
LncNEAT1调控肝脏脂质代谢的研究最早开始于2017年, Wang发现NEAT1通过激活雷帕霉素靶蛋白(Mammalian target of rapamycin, mTOR)/S6K1信号通路, 促进肝细胞中Acc和Fas的表达。相反, 利用慢病毒干扰NEAT1则缓解HFD引起的非酒精性脂肪肝[25]。另有研究表明, NEAT1与miR-140之间存在相互作用。体外抑制NEAT1可以下调miR-140的表达, 相反, 抑制miR-140同样可以降低NEAT1的表达, 但任意干扰二者之一都可以增加腺苷酸活化蛋白激酶(AMP activated protein kinase, AMPK)的磷酸化水平, 降低Srebp1、Fas以及Acc等基因的表达水平, 起到降脂的作用[26]。Chen等[27]发现, NEAT1还可以与miR-146a-5p结合, 解除miR-146-5p对Rho相关蛋白激酶1 (Rock1)的抑制作用。Rock1作为AMPK的上游基因可以抑制其活性, 促进机体的脂质合成。除了参与肝脏的脂质合成代谢, NEAT1还可以调控肝癌异常变化的脂质分解代谢。研究发现, 在肝癌细胞中, NEAT1的表达显著增加。NEAT1通过与miR-124-3p结合, 减弱miR-124-3p对Atgl的抑制作用, 间接促进Atgl的表达, 增强细胞的脂质水解进程[28]。
LncHR1 (HCV regulated 1)是一种可以被丙型肝炎病毒(Hepatitis C virus, HCV)激活的lncRNA。但在HCV促进肝脏脂质合成的过程中, HR1并没有起到介导这一进程发展的作用, 而是通过抑制SREBP-1C的表达来降低机体的脂质堆积[29]。此外, 体内和体外实验表明, HR1可以缓解由HFD引起的过度脂质堆积。Li等[30]进一步研究发现, HR1对SREBP-1C的抑制作用是由调控丙酮酸脱氢酶激酶1 (Pyruvate dehydrogenase kinase 1, PDK1)/AKT/FoxO1通路所介导的。结果显示, HR1抑制PDK1的磷酸化, 减弱了PDK1对AKT的磷酸化激活作用, 进而影响FoxO1的核质分布, 使FoxO1入核增多, 降低SREBP-1C的表达, 缓解肝脏脂质堆积。
LncRNA肝脏特异性调控甘油三脂(Liver- specific triglyceride regulator, LncLSTR)在24 h饥饿处理的小鼠肝脏组织中表达量显著降低, 并且其表达量随着再进食而快速恢复。体内实验表明, 肝脏特异性敲除LSTR增强小鼠血液中TG的清除率, 降低小鼠血液中TG含量。进一步研究发现, LSTR与核酸结合蛋白TDP-43结合, 减弱TDP-43对Cyp8b1的抑制作用。LSTR被抑制后Cyp8b1表达降低, 改变了体内胆汁酸与胆酸的比例, 使胆汁酸受体FXR被激活, 促进Apoc2的表达。Apoc2的表达升高增强了脂蛋白脂解酶的活性, 进而提高了血液中TG的清除率[31]。
棕色脂肪富集长链非编码RNA1 (Brown fat -enriched lncRNA 1, LncBlnc1)首先在脂肪组织中被发现, 可以调控褐色脂肪组织的分化和产热进程。随着研究的深入, 研究者发现Blnc1在肝脏中表达丰度较高。Blnc1在HFD小鼠、ob/ob小鼠以及db/db小鼠的肝脏组织中表达量显著上升。研究发现, Blnc1通过与EDF1结合, 促进肝X受体(Liver X receptors, LXR)转录复合体的装配。干扰Blnc1则减弱EDF1与LXR之间的结合以及LXR在SREBP-1C启动子上的募集, 降低脂质合成相关基因的表达[32]。
LXR是机体胆固醇代谢中重要的调控因子, 可促进胆固醇的外流和酯化, 抑制胆固醇的吸收。Sallam等[33]使用LXR的激动剂GW3965处理原代肝细胞, 而后进行转录组学分析, 筛选出表达升高最为剧烈的lncRNA并将其命名为肝脏LXR诱导表达的lncRNA (Liver-expressed LXR induced sequence, LeXis), 同时发现LXR除了参与上述调控途径外, 还可以抑制机体胆固醇的合成。研究显示, LeXis主要定位于细胞核, 被LXR诱导激活, 与异质核糖核蛋白RALY结合。RALY作为转录辅激活因子促进胆固醇合成相关基因的表达。LeXis与RALY的结合则抑制了RALY的转录调控功能, 进而对机体的胆固醇合成产生抑制作用。
LncRNA类固醇受体RNA激活剂(Steroid receptor RNA activator, LncSRA)是一种参与多种生理功能的lncRNA, SRA作为RNA转录辅激活物, 可同时增强类固醇和非类固醇核受体介导的基因表达, 在脂肪组织中发挥调控脂肪组织分化和葡萄糖吸收的作用。研究发现, 在肝脏组织中, SRA通过增强胰岛素信号引起的FoxO1的磷酸化水平升高来降低ATGL的表达。FoxO1的磷酸化水平通常决定着FoxO1的生物活性和核质分布, 而胰岛素缺乏时, 过表达SRA虽然增加了FoxO1的磷酸化水平却使FoxO1的入核显著增多。同时, 荧光素酶报告基因实验显示, 过表达SRA引起的FoxO1入核增多并没增强Atgl的启动子活性, 其结果与体内实验结果相一致, SRA过表达同样在细胞水平抑制ATGL的转录, 但是具体的分子机制尚不明确[34]。
除了在维持肝脏糖代谢稳态中发挥作用, H19也在肝脏脂质代谢中起着重要的作用。研究发现, H19的表达在高脂高蔗糖(High-fat/ high-sucrose, HFHS)饮食小鼠的肝脏中表达显著增高, 相应地, 干扰H19则可以缓解HFHS饮食引起的脂质堆积。进一步研究表明, 肝脏中的H19通过与hnRNP家族成员PTBP1结合, 一方面促进PTBP1与Srebp-1c的mRNA之间的结合, 提高Srebp-1c mRNA的稳定性;另一方面促进PTBP1与SREBP-1C的蛋白前体结合, 使SREBP-1C剪切入核增多的同时增强SREBP-1C的转录活性, 进而促进肝脏中的脂质合成[35]。过氧化物酶体增殖物激活受体γ (Peroxisome proliferator-activated receptor γ, PPARγ)是一种在脂肪组织中高表达的转录因子, 在脂质代谢的调节中起重要作用。据报道, NAFLD患者肝脏中PPARγ被诱导表达, 造成肝脏脂质堆积。Liu等[36]发现, H19可以通过结合miR-130a, 拮抗miR-130a对PPARγ的抑制作用, 进而促进肝脏脂质合成。另外的研究表明, 除了调控上述转录因子外, H19还可以通过促进碳水化合物反应元件结合蛋白(Carbohydrate response element binding protein, Chrebp)表达和激活mTOR信号通路促进肝脏的脂质合成。从目前的研究中不难看出, H19发挥“脂质合成感受器”的作用, 在血液中游离脂肪酸增加的情况下被诱导表达, 从代谢调控的不同层次和信号通路促进肝脏中的脂质合成, 降低血脂中游离脂肪酸的含量, 避免脂毒性的产生[37]。
LncAPOA1-AS被发现可以调控肝脏载脂蛋白A1 (Apolipoprotein A1, Apoa1)的表达, APOA1-AS作为Apoa1 DNA区段的反义序列, 与Apoa1的第4个外显子具有重叠部分。研究显示, 干扰APOA1-AS可以分别减弱Apoa1启动子上, 蛋白去甲基化酶(Lysine (K)-specific demethylase 1A, LSD1)在H3K4位点的结合与多梳抑制复合物2在H3K27位点的结合, 提高组蛋白第3亚基4号甲基化的赖氨酸(Monomethylated lysines 4 on histone H3, H3K4me)的水平同时降低H3K27me3的水平, 促进APOA1的表达[38]。随后APOA4-AS也被发现具有调控脂蛋白的作用, 与APOA1-AS相似, APOA4-AS是一段与Apoa4第3个外显子及3′端非编码区序列重叠的反义序列。不同的是, APOA4-AS与mRNA结合蛋白HuR形成复合物, 与Apoa4 mRNA结合并提高其稳定性。APOA4-AS和Apoa4在人脂肪肝患者和ob/ob小鼠肝脏中表达均呈现增多趋势, 抑制APOA4-AS可以显著降低ob/ob小鼠血液中TG和TC的含量[39]。
LncMEG3同样可以结合PTBP1, 并通过与PTBP1结合将PTBP1募集至小异源二聚体伴侣(Small heterodimer partner, SHP) mRNA上, 促进Shp mRNA的降解, MEG3在整个过程中并不直接介导mRNA的降解, 而是发挥着“引导”与“支架”的作用。SHP被证实是一种胆汁酸合成的阻遏分子, SHP被抑制后引起了胆固醇7a-羟化酶(Cholesterol 7-alpha hydroxylase, Cyp7a1)和胆固醇12α羟化酶(Sterol 12a-hydroxylase, Cyb8b1)的表达, 激活胆汁酸代谢。事实上, 在肝脏组织中, 胆汁酸代谢与脂质代谢之间存在密切的联系, 例如, FXR可以被胆汁酸代谢产物所激活, 在脂质代谢中发挥着重要的作用[40]。Huang等[41]利用非酒精性脂肪肝小鼠模型进行研究, 发现MEG3可以作为ceRNA, 结合miR-21并减弱miR-21对低密度脂蛋白受体相关蛋白6 (Low density lipoprotein receptor-related protein 6, Lrp6) mRNA的抑制作用。激活后的LRP6抑制AKT/mTOR信号通路, 减少肝脏脂质合成。
关于lncRNA对胆固醇代谢的调控, Lan等[42]发现lncHC在HFD小鼠肝脏中表达升高, 并参与肝脏中的胆固醇代谢。当肝脏中胆固醇含量增多时, LXRα被激活, 促进转录因子CCAAT增强子结合蛋白(CCAAT/enhancer-binding protein beta, C/EBPβ)的表达。随后HC被C/EBPβ激活, 与hnRNPA2B1结合并将其募集至HC的靶基因Cyp7a1和Abca1的mRNA上, 并降低靶基因mRNA的稳定性, 抑制胆汁酸代谢和高密度脂蛋白的合成。随后作者在另外的文章中指出, HC不仅可以调控胆汁酸代谢, 还可以调控肝脏脂质合成。实验证明, HC通过促进miR-130b-3p的表达, 抑制miR-130b-3p的靶基因PPARγ的表达, 降低肝脏中脂肪酸吸收相关基因和脂质合成相关基因的表达, 减少肝脏脂质堆积[43]。
LncKDM5D-4基因座位于Y染色体上, 在HepG2细胞中干扰KDM5D-4可以提高脂滴包被蛋白2的表达, 促进肝脏中脂滴的形成[44]。
LncRNA脂质相关的单核苷酸多态性区域(Lipid associated single nucleotide polymorphism region, LncLASER)可由LXR激活, 被激活的LASER可以直接与LSD1结合, Hnf1α启动子上H3K4me去甲基化的发生, 激活HNF-1α的表达。随后HNF-1α促进其下游基因前蛋白转化酶枯草杆菌蛋白酶/kexin9型(Proprotein convertase subtilisin/kexin type 9, Pcsk9)的表达。Pcsk9的升高减弱了肝脏对血液中胆固醇的清除能力, 造成高胆固醇血症[45]。
LncARSR被发现在高胆固醇患者肝脏内表达升高。动物实验显示, 过表达ARSR会提高胆固醇相关基因的表达, 例如, 羟甲基戊二酸单酰辅酶A还原酶(Hydroxymethylglutaryl CoA Reductase, Hmgcr)、Hmgcs、角鲨烯合酶(Squalene synthase, Sqs)等。进一步研究表明, ARSR通过激活PI3K/Akt信号通路提高Srebp-2的表达, 促进胆固醇的合成[46]。
Gm16551被发现在饥饿小鼠肝脏中表达降低, 随着再进食而恢复表达。Gm16551的表达受到SREBP1C的激活, Gm16551的表达与脂质合成相关基因呈负相关。实验显示, 在小鼠体内干扰Gm16551造成Acly、FasS以及Scd1的表达升高, 在干扰Gm16551的基础上同时敲低Srebp1c, 脂质合成相关基因的水平得到遏制, 反之, 同时过表达Srebp1c和Gm16551则使被SREBP1c激活的脂质合成相关基因表达降低。因此, Gm16551被认为是SREBP1c在促进肝脏脂质合成进程中的负反馈元件, 受SREBP1c的调控而表达, 并抑制SREBP1c激活的脂质合成[47]。
LncMALAT1被发现在ob/ob小鼠的肝脏组织以及使用棕榈酸处理的HepG2细胞和原代肝细胞中表达量显著升高。相反, 在体内和体外抑制MALAT1的表达则导致肝脏脂质堆积。研究发现, MALAT1可以与SREBP1C结合, 减少SREBP1c的泛素化降解, 提高SREBP1c的稳定性, 促进肝脏脂质堆积[48]。
表没食子儿茶素没食子酸酯(Epigallocatechin- 3-gallate, EGCG)是绿茶提取物的主要成分, 可以降低血液中胆固醇的含量。研究发现, 使用EGCG处理HepG2细胞可以激活lncRNA AT102202的表达。AT102202由303个氨基酸构成, 其序列与肝脏中胆固醇合成相关基因HmgcrDNA序列上第4–6个外显子序列相似度达100%。与此同时, 在HepG2细胞中干扰AT102202导致HMGCR表达量的降低[49]。
由表 2可知, lncRNA参与了脂肪分化、脂肪合成、脂肪分解和脂肪酸氧化等过程, 在肝脏脂肪代谢中起到了重要调控作用。LncRNA通过直接或间接的方式调控脂质代谢过程限速酶(ACC、HMGCR、SREBP1c、PPARγ和CYP7a1等), 而上述基因在转录或蛋白水平变化可以最直观反应肝脏脂质代谢状况, 成为****研究lncRNA下游生物学功能最主要的靶点。
表 2 肝脏中参与脂代谢的lncRNA及相关靶点和作用Table 2 Functional lncRNAs involved in hepatic lipid metabolism
| Name | Target | Function | Renference |
| SHGL | hnRNPA1 | hnRNPA1↓, CALM↑, mTOR/SREBP-1C↓, liver TG↓ | [20] |
| LincIRS2 | MAFG | Acox1↓, Cpt1a↓ | [22] |
| uc.372 | miR-195 | miR-195↑, AGO↓, ACC↓, FAS↓ | [24] |
| NEAT1 | mTOR/S6K1 | ACC↑, FAS↑ | [25] |
| miR-140 | miR-140↑, AMPK/SREBP1c↑, NAFLD↑ | [26] | |
| miR-146b-5p | miR-146b-5p↓, Rock↓, Ampk7↑, steatosis↓ | [27] | |
| miR-124-3p | miR-124-3p↓, ATGL↑, DAG+FFA↑, lipolysis↑ | [28] | |
| HR 1 | PDK1/AKT/FoxO1 | PDK↑, AKT↑, FoxO1↑, SREBP1C↑, TG↑ | [30] |
| LSTR | TDP43 | Cyp8b↑, MCA/CA↓, Apoc2↓ | [31] |
| Blnc1 | Srebp1c | Srebp1c↑, Fasn↑, AST↑, ALT↑ | [32] |
| Lexis | RALY | Srebf2↓, Pcsk9↓, cholesterol↓ | [33] |
| SRA | FoxO1 | FoxO1↓, ATGL↓, SREBP1C↓, FASN↓ | [34] |
| H19 | PTBP1 | PTBP1↑, SREBP1C↑, FASN↑ | [35] |
| miR-130a | miR-130a↓, PPARγ↑, FASN↑, ACC1↑, SCD↑ | [36] | |
| MLXLPL, mTOR | MLXLPL↑, mTOR↑, FASN↑, Acaca↑, Scd1↑ | [37] | |
| APOA1-AS | APOA1 | LSD↑, APOA1↓ | [38] |
| APOA4-AS | APOA4 | HUR↑, APOA4↑, plasm TC↑, TG↑ | [39] |
| MEG3 | PTBP1 | PTBP1↓, SHP↓, Cyp7a/8b↑, bile acid↑ | [40] |
| miR21 | LRP6↑, AKT↑, p-mTOR↑, TG↑ | [41] | |
| HC | hnRNPA2/B1 | HC↑, Cyp7a1↓, Abca1↓ | [42] |
| miR-130b-3p | HC↑, PPARγ↑, TG↓ | [43] | |
| KDM5D-4 | PLIN2 | PLIN2↓, Lipid droplet↓ | [44] |
| LASER | LSD | LSD↑, HNF-1α↓, PCSK9↑, cholesterol↑ | [45] |
| ARSR | Akt/SREBP-2/HMGCR | Akt↓, SREBP-2↑, HMGCR↑, TC↑ | [46] |
| GM16551 | Srebp1c | FAS↓, SCD↓, serumTG↓ | [47] |
| MALAT | SREBP1C | SREBP1C↑, ACC1↑, FAS↑ | [48] |
| AT102202 | HMGCR | HMGCR↓ | [49] |
表选项
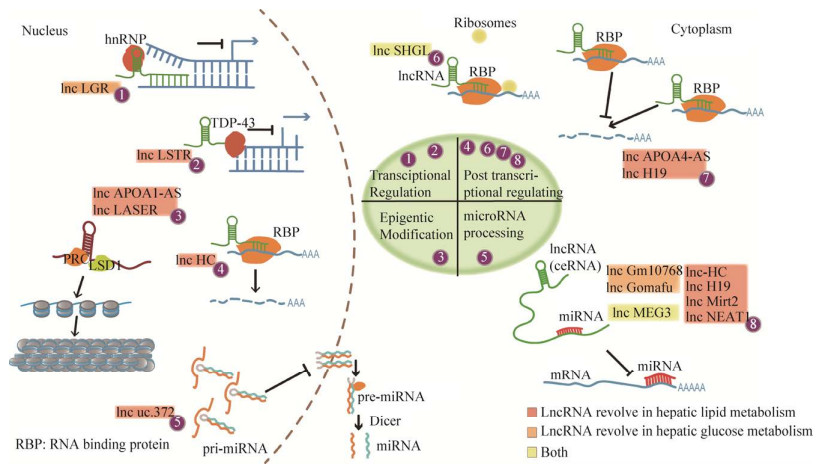
3 展望总而言之, lncRNA调控细胞代谢的不同生物过程, 并在肝脏代谢性疾病中具有显著的差异性表达。虽然lncRNA在最初被认为不具功能, 但lncRNA基因经转录剪切后成为lncRNA, 并在不同的细胞调节途径中发挥特定的功能。图 1为lncRNA作用机制模式图, lncRNA在细胞核和细胞质中分别执行不同功能。细胞核:(1)与核蛋白结合, 影响染色质状态。(2)与核酸结合蛋白结合, 调控基因表达。(3)影响miRNA成熟与释放。(4)与RNA结合蛋白结合, 改变转录后的RNA状态。细胞质:(1)与RNA结合蛋白结合, 影响mRNA翻译效率及稳定性。(2)与miRNA结合, 阻止其与靶基因mRNA相互作用。LncRNA在组织中的表达模式与疾病的不同进程相吻合, 由于这种特异性, lncRNA已逐渐成为糖脂紊乱疾病治疗中更有潜在价值的靶点。虽然这些研究仍处于初期阶段, 但研究人员开始利用lncRNA的这些特性并应用于临床工作中。研究表明, lncRNA在多种癌症中已经被用作生物标志物。但目前对lncRNA的研究仍处于起步阶段, 仍有许多需要深入探讨的问题:(1) lncRNA的二级结构、功能以及其功能行使的分子机制研究不够充分。(2)目前研究最多的依然是lncRNA对于肿瘤的作用, 但其在肝脏能量代谢方面的研究并不全面。(3)当下对于lncRNA的研究水平大多仅停留在实验阶段, 离临床应用还有很大距离。但是lncRNA在肝脏代谢性疾病中具有重大意义, 这意味着lncRNA将在该类疾病的诊断、治疗和预后中发挥重要作用。
 |
| 图 1 LncRNA作用机制模式图 Fig. 1 Mechanisms of lncRNA function. |
| 图选项 |
参考文献
| [1] | Johnsson P, Lipovich L, Grandér D, et al. Evolutionary conservation of long non-coding RNAs; sequence, structure, function. Biochim Biophys Acta, 2014, 1840(3): 1063-1071. DOI:10.1016/j.bbagen.2013.10.035 |
| [2] | Clark MB, Mercer TR, Bussotti G, et al. Quantitative gene profiling of long noncoding RNAs with targeted RNA sequencing. Nat Methods, 2015, 12(4): 339-342. DOI:10.1038/nmeth.3321 |
| [3] | Hu WQ, Alvarez-Dominguez JR, Lodish HF. Regulation of mammalian cell differentiation by long non-coding RNAs. EMBO Rep, 2015, 13(11): 971-983. |
| [4] | Kim J, Piao HL, Kim BJ, et al. Long noncoding RNA MALAT1 suppresses breast cancer metastasis. Nat Genet, 2018, 50(12): 1705-1715. DOI:10.1038/s41588-018-0252-3 |
| [5] | Batista PJ, Chang HY. Long noncoding RNAs: cellular address codes in development and disease. Cell, 2013, 152(6): 1298-1307. DOI:10.1016/j.cell.2013.02.012 |
| [6] | Kopp F, Mendell JT. Functional classification and experimental dissection of long noncoding RNAs. Cell, 2018, 172(3): 393-407. DOI:10.1016/j.cell.2018.01.011 |
| [7] | Pefanis E, Wang JG, Rothschild G, et al. RNA exosome-regulated long non-coding RNA transcription controls super-enhancer activity. Cell, 2015, 161(4): 774-789. DOI:10.1016/j.cell.2015.04.034 |
| [8] | Qian XY, Zhao JY, Yeung PY, et al. Revealing lncRNA structures and interactions by sequencing-based approaches. Trends Biochem Sci, 2019, 44(1): 33-52. DOI:10.1016/j.tibs.2018.09.012 |
| [9] | Lai F, Orom UA, Cesaroni M, et al. Activating RNAs associate with Mediator to enhance chromatin architecture and transcription. Nature, 2013, 494(7438): 497-501. DOI:10.1038/nature11884 |
| [10] | Lu YY, Zhao XD, Liu Q, et al. lncRNA MIR100HG-derived miR-100 and miR-125b mediate cetuximab resistance via Wnt/β-catenin signaling. Nat Med, 2017, 23(11): 1331-1341. DOI:10.1038/nm.4424 |
| [11] | Matsumoto A, Pasut A, Matsumoto M, et al. mTORC1 and muscle regeneration are regulated by the LINC00961-encoded SPAR polypeptide. Nature, 2017, 541(7636): 228-232. DOI:10.1038/nature21034 |
| [12] | Goyal N, Sivadas A, Shamsudheen KV, et al. RNA sequencing of db/db mice liver identifies lncRNA H19 as a key regulator of gluconeogenesis and hepatic glucose output. Sci Rep, 2017, 7: 8312. DOI:10.1038/s41598-017-08281-7 |
| [13] | Goyal N, Tiwary S, Kesharwani D, et al. Long non-coding RNA H19 inhibition promotes hyperglycemia in mice by upregulating hepatic FoxO1 levels and promoting gluconeogenesis. J Mol Med, 2019, 97(1): 115-126. DOI:10.1007/s00109-018-1718-6 |
| [14] | Zhang N, Geng TT, Wang ZS, et al. Elevated hepatic expression of H19 long noncoding RNA contributes to diabetic hyperglycemia. JCI Insight, 2018, 3(10): e120304. DOI:10.1172/jci.insight.120304 |
| [15] | Zhu X, Wu YB, Zhou J, et al. Upregulation of lncRNA MEG3 promotes hepatic insulin resistance via increasing FoxO1 expression. Biochem Biophys Res Commun, 2016, 469(2): 319-325. DOI:10.1016/j.bbrc.2015.11.048 |
| [16] | Zhu X, Li HQ, Wu YB, et al. CREB-upregulated lncRNA MEG3 promotes hepatic gluconeogenesis by regulating miR-302a-3p-CRTC2 axis. J Cell Biochem, 2019, 120(3): 4192-4202. DOI:10.1002/jcb.27706 |
| [17] | Zhu X, Li HQ, Wu YB, et al. LncRNA MEG3 promotes hepatic insulin resistance by serving as a competing endogenous RNA of miR-214 to regulate ATF4 expression. Int J Mol Med, 2019, 43(1): 345-357. |
| [18] | Chen DL, Shen DY, Han CK, et al. LncRNA MEG3 aggravates palmitate-induced insulin resistance by regulating miR-185–5p/Egr2 axis in hepatic cells. Eur Rev Med Pharmaco Sci, 2019, 23(12): 5456-5467. |
| [19] | Yan CF, Li J, Feng SY, et al. Long noncoding RNA Gomafu upregulates Foxo1 expression to promote hepatic insulin resistance by sponging miR-139-5p. Cell Death Dis, 2018, 9: 289. DOI:10.1038/s41419-018-0321-7 |
| [20] | Wang JP, Yang WL, Chen ZZ, et al. Long noncoding RNA lncSHGL recruits hnRNPA1 to suppress hepatic gluconeogenesis and lipogenesis. Diabetes, 2018, 67(4): 581-593. DOI:10.2337/db17-0799 |
| [21] | Ruan XB, Li P, Cangelosi A, et al. A long non-coding RNA, lncLGR, regulates hepatic glucokinase expression and glycogen storage during fasting. Cell Rep, 2016, 14(8): 1867-1875. DOI:10.1016/j.celrep.2016.01.062 |
| [22] | Pradas-Juni M, Hansmeier NR, Link JC, et al. A MAFG-lncRNA axis links systemic nutrient abundance to hepatic glucose metabolism. Nat Commun, 2020, 11: 644. DOI:10.1038/s41467-020-14323-y |
| [23] | Cui XW, Tan JM, Shi YJ, et al. The long non-coding RNA Gm10768 activates hepatic gluconeogenesis by sequestering microRNA-214 in mice. J Biol Chem, 2018, 293(11): 4097-4109. DOI:10.1074/jbc.M117.812818 |
| [24] | Guo J, Fang WW, Sun LB, et al. Ultraconserved element uc. 372 drives hepatic lipid accumulation by suppressing miR-195/miR4668 maturation. Nat Commun, 2018, 9: 612. DOI:10.1038/s41467-018-03072-8 |
| [25] | Wang X. Down-regulation of lncRNA-NEAT1 alleviated the non-alcoholic fatty liver disease via mTOR/S6K1 signaling pathway. J Cell Biochem, 2018, 119(2): 1567-1574. DOI:10.1002/jcb.26317 |
| [26] | Sun YF, Song Y, Liu CS, et al. LncRNA NEAT1-MicroRNA-140 axis exacerbates nonalcoholic fatty liver through interrupting AMPK/SREBP-1 signaling. Biochem Biophys Res Commun, 2019, 516(2): 584-590. DOI:10.1016/j.bbrc.2019.06.104 |
| [27] | Chen X, Tan XR, Li SJ, et al. LncRNA NEAT1 promotes hepatic lipid accumulation via regulating miR-146a-5p/ROCK1 in nonalcoholic fatty liver disease. Life Sci, 2019, 235: 116829. DOI:10.1016/j.lfs.2019.116829 |
| [28] | Liu XR, Liang YJ, Song RP, et al. Long non-coding RNA NEAT1-modulated abnormal lipolysis via ATGL drives hepatocellular carcinoma proliferation. Mol Cancer, 2018, 17: 90. DOI:10.1186/s12943-018-0838-5 |
| [29] | Li D, Cheng M, Niu YQ, et al. Identification of a novel human long non-coding RNA that regulates hepatic lipid metabolism by inhibiting SREBP-1c. Int J Biol Sci, 2017, 13(3): 349-357. DOI:10.7150/ijbs.16635 |
| [30] | Li D, Guo LW, Deng BG, et al. Long non-coding RNA HR1 participates in the expression of SREBP-1c through phosphorylation of the PDK1/AKT/FoxO1 pathway. Mol Med Rep, 2018, 18(3): 2850-2856. |
| [31] | Li P, Ruan XB, Yang L, et al. A liver-enriched long non-coding RNA, lncLSTR, regulates systemic lipid metabolism in mice. Cell Metab, 2015, 21(3): 455-467. DOI:10.1016/j.cmet.2015.02.004 |
| [32] | Zhao XY, Xiong XL, Liu TY, et al. Long noncoding RNA licensing of obesity-linked hepatic lipogenesis and NAFLD pathogenesis. Nat Commun, 2018, 9: 2986. DOI:10.1038/s41467-018-05383-2 |
| [33] | Sallam T, Jones MC, Gilliland T, et al. Feedback modulation of cholesterol metabolism by the lipid-responsive non-coding RNA LeXis. Nature, 2016, 534(7605): 124-128. DOI:10.1038/nature17674 |
| [34] | Chen G, Yu DS, Nian X, et al. LncRNA SRA promotes hepatic steatosis through repressing the expression of adipose triglyceride lipase (ATGL). Sci Rep, 2016, 6: 35531. DOI:10.1038/srep35531 |
| [35] | Liu CN, Yang ZH, Wu JG, et al. Long noncoding RNA H19 interacts with polypyrimidine tract-binding protein 1 to reprogram hepatic lipid homeostasis. Hepatology, 2018, 67(5): 1768-1783. DOI:10.1002/hep.29654 |
| [36] | Liu J, Tang T, Wang GD, et al. LncRNA-H19 promotes hepatic lipogenesis by directly regulating miR-130a/PPARγ axis in non-alcoholic fatty liver disease. Biosci Rep, 2019, 39(7): BSR20181722. DOI:10.1042/BSR20181722 |
| [37] | Wang H, Cao YD, Shu LQ, et al. Long non-coding RNA (lncRNA) H19 induces hepatic steatosis through activating MLXIPL and mTORC1 networks in hepatocytes. J Cell Mol Med, 2020, 24(2): 1399-1412. DOI:10.1111/jcmm.14818 |
| [38] | Halley P, Kadakkuzha BM, Ali Faghihi M, et al. Regulation of the apolipoprotein gene cluster by a long noncoding RNA. Cell Rep, 2014, 6(1): 222-230. DOI:10.1016/j.celrep.2013.12.015 |
| [39] | Qin WS, Li XZ, Xie LW, et al. A long non-coding RNA, APOA4-AS, regulates APOA4 expression depending on HuR in mice. Nucleic Acids Res, 2016, 44(13): 6423-6433. DOI:10.1093/nar/gkw341 |
| [40] | Zhang L, Yang ZH, Trottier J, et al. Long noncoding RNA MEG3 induces cholestatic liver injury by interaction with PTBP1 to facilitate shp mRNA decay. Hepatology, 2017, 65(2): 604-615. DOI:10.1002/hep.28882 |
| [41] | Huang P, Huang FZ, Liu HZ, et al. LncRNA MEG3 functions as a ceRNA in regulating hepatic lipogenesis by competitively binding to miR-21 with LRP6. Metabolism, 2019, 94: 1-8. DOI:10.1016/j.metabol.2019.01.018 |
| [42] | Lan X, Yan JD, Ren J, et al. A novel long noncoding RNA Lnc-HC binds hnRNPA2B1 to regulate expressions of Cyp7a1 and Abca1 in hepatocytic cholesterol metabolism. Hepatology, 2016, 64(1): 58-72. DOI:10.1002/hep.28391 |
| [43] | Lan X, Wu LT, Wu N, et al. Long noncoding RNA lnc-HC regulates PPARγ-mediated hepatic lipid metabolism through miR-130b-3p. molecular therapy. Nucleic Acids, 2019, 18: 954-965. DOI:10.1016/j.omtn.2019.10.018 |
| [44] | Molina E, Chew GS, Myers SA, et al. A novel Y-specific long non-coding RNA associated with cellular lipid accumulation in HepG2 cells and atherosclerosis-related genes. Sci Rep, 2017, 7: 16710. DOI:10.1038/s41598-017-17165-9 |
| [45] | Li CW, Hu ZX, Zhang W, et al. Regulation of cholesterol homeostasis by a novel long non-coding RNA LASER. Sci Rep, 2019, 9: 7693. DOI:10.1038/s41598-019-44195-2 |
| [46] | Huang JB, Chen SJ, Cai DL, et al. Long noncoding RNA lncARSR promotes hepatic cholesterol biosynthesis via modulating Akt/SREBP-2/HMGCR pathway. Life Sci, 2018, 203: 48-53. DOI:10.1016/j.lfs.2018.04.028 |
| [47] | Yang L, Li P, Yang WJ, et al. Integrative transcriptome analyses of metabolic responses in mice define pivotal LncRNA metabolic regulators. Cell Metab, 2016, 24(4): 627-639. DOI:10.1016/j.cmet.2016.08.019 |
| [48] | Yan CF, Chen JF, Chen NQ. Long noncoding RNA MALAT1 promotes hepatic steatosis and insulin resistance by increasing nuclear SREBP-1c protein stability. Sci Rep, 2016, 6: 22640. DOI:10.1038/srep22640 |
| [49] | Liu G, Zheng XX, Xu YL, et al. Long non-coding RNAs expression profile in HepG2 cells reveals the potential role of long non-coding RNAs in the cholesterol metabolism. Chinese Med J (English), 2015, 128(1): 91-97. DOI:10.4103/0366-6999.147824 |
