
 , 呼和巴特尔
, 呼和巴特尔

内蒙古农业大学 兽医学院 农业农村部动物疾病临床诊疗技术重点实验室,内蒙古 呼和浩特 010018
收稿日期:2019-06-07;接收日期:2019-09-02;网络出版时间:2019-09-18
基金项目:国家自然科学基金(No. 31760731),内蒙古自治区科技计划项目(No. 201702074)资助
通讯作者:Wenlong Wang. Tel:+86-471-4303726; E-mail: wwl.imau@163.com;
Huhebateer. Tel:+86-471-4303726; E-mail: hhbte@163.com.
摘要:分析捻转血矛线虫敏感株和耐药株中长链非编码RNA (Long non-coding RNA,lncRNA)的表达谱,探讨lncRNA与捻转血矛线虫丙硫咪唑耐药机制的关联性,为捻转血矛线虫耐药机理提供依据。文中对捻转血矛线虫敏感株和耐药株进行cDNA测序文库构建,使用Illumina HiSeq 4000平台进行双端测序,筛选出差异的lncRNA,基于顺式调控(cis)和反式调控(trans)对差异显著的lncRNA进行靶基因预测,并对靶基因进行过Gene Ontology (GO)功能注释和KEGG Pathway富集分析,利用FPKM法估计lncRNA和mRNA的表达水平。结果表明,敏感株和耐药株候选lncRNA分别为6 377和6 356个,两个文库中筛选出168个差异显著的lncRNA,其中在敏感株中表达上调有92个,表达下调76个。筛选得到的差异显著lncRNA候选靶基因416个,这些基因共注释到641条GO terms和92条信号通路;其中富集到耐药性相关的通路有药物代谢-其他酶、药物代谢-细胞色素P450、细胞色素P450对异生素的代谢等。综上表明,部分lncRNA介导的靶基因与捻转血矛线虫丙硫咪唑耐药性相关,lncRNA在捻转血矛线虫耐药性中具有潜在的重要的调节作用。探究了对于敏感虫株和耐药虫株中lncRNA的表达谱,发现了敏感虫株和耐药虫株中差异表达的lncRNA,有助于找出捻转血矛线虫如何抵抗丙硫咪唑的发生机制,为探讨捻转血矛线虫丙硫咪唑耐药机制提供科学的依据。
关键词:捻转血矛线虫丙硫咪唑耐药性转录组长链非编码RNA
Transcriptome sequencing and analysis of lncRNA related to albendazole resistant strain of Haemonchus contortus
Xueliang Zhao, Wenlong Wang

 , Huhebateer
, Huhebateer

Key Laboratory of Clinical Diagnosis and Treatment Technology in Animal Disease, Ministry of Agriculture and Rural Affairs, College of Veterinary Medicine, Inner Mongolia Agricultural University, Hohhot 010018, Inner Mongolia, China
Received: June 7, 2019; Accepted: September 2, 2019; Published: September 18, 2019
Supported by: National Natural Science Foundation of China (No. 31760731), Science and Technology Plan Project of Inner Mongolia Autonomous Region (No. 201702074)
Abstract: The objective of this study was to explore the link between long non-coding RNAs (lncRNAs) and resistance mechanism of albendazole in Haemonchus contortus by analyzing the gene expression profile of lncRNA in sensitive and resistant strains. This study provided the base for the resistance mechanism of Haemonchus contortus. In this experiment, the cDNA sequencing libraries were collected constructed for the sensitive and resistant strains of Haemonchus contortus, and the sequencing was performed using Illumina HiSeq 4000 platform, and differentially expressed lncRNAs (DEIncRNAs) were screened. Then the cis-targeted and trans-targeted genes of DElncRNAs were predicted, and the GO enrichment, KEGG pathway enrichment analysis were also made. The results displayed that 6 377 and 6 356 candidate lncRNA transcripts of sensitive and resistive strains were respectively obtained, 168 DElncRNAs of which were selected. Compared with resistive strains, there were 92 up-regulated and 76 down-regulated lncRNAs in sensitive strains. Meanwhile, 416 candidate target genes of DElncRNAs were obtained. The analytical results indicated that these genes participated in 641 GO terms and 92 signal pathways. The pathways involved in drug resistance are drug metabolism-other enzymes, drug metabolism-cytochrome P450, metabolism of xenobiotics by cytochrome P450 and so on. In conclusion, these findings inferred that some lncRNA-mediated target genes were associated with the resistance of Haemonchus contortus, and lncRNA may play an important role in the resistance of Haemonchus contortus. This study explored the expression profile of lncRNA in the Haemonchus contortus of sensitive and resistant strains. It was found that DElncRNAs in the sensitive and resistant strains, which helped to find out the Haemonchus contortus resistance mechanism of albendazole and provided a scientific basis for exploring the mechanism of resistance to Haemonchus contortus.
Keywords: Haemonchus contortusalbendazoledrug resistancetranscriptomelong non-coding RNA
捻转血矛线虫是反刍动物最常见的消化道线虫之一,由于缺乏有效的疫苗,长期以来对捻转血矛线虫病的防治几乎主要依靠丙硫咪唑、伊维菌素等化学药物。然而,随着该药物几十年以来的反复频繁使用,导致捻转血矛线虫耐药性的逐渐产生,给畜牧业造成严重的经济损失[1]。检测影响捻转血矛线虫丙硫咪唑耐药相关基因表达情况是了解耐药机制的基础,同时可以筛选与丙硫咪唑耐药相关的候选基因。1986年,在南非首次报道了寄生虫对丙硫咪唑的耐药性,由于驱虫药物频繁使用,使寄生虫多次与药物接触后,对药物敏感性下降甚至消失,导致药物对寄生虫无效[2]。国内外许多****在捻转血矛线虫耐药性流行病学调查、耐药性检测方法和耐药分子机制的研究中取得了大量的研究成果。但有关捻转血矛线虫耐药性组学的研究较少[3],尤其是关于长链非编码RNA (Long non-coding RNA,lncRNA)的研究仍属未知。
lncRNA是长度大于200 bp的非编码RNA,调控方式主要分为两类,一类为顺式调控,另一类为反式调控。研究表明,lncRNA在细胞周期、表观遗传和天然免疫等众多生命活动中发挥重要作用[4]。近年来,在人、绵羊、野猪上均鉴定到lncRNA,部分lncRNA与人类癌症耐药、肿瘤耐药有着密切的联系。Li等通过对lncRNA测序研究,揭示了lncARSR在肝脏癌症细胞中对阿霉素耐药性中的关键作用,表明lncARSR可作为肝癌预后生物标志物和药物治疗靶点[5]。相比于人类疾病耐药lncRNA,寄生虫耐药相关的lncRNA功能研究较滞后。本研究利用RNA-seq,对捻转血矛线虫丙硫咪唑敏感株和耐药株中lncRNA与mRNA的结构特征(长度分布、ORF长度、外显子数目)以及表达量进行比较,对差异表达lncRNA进行筛选,并基于反式调控机制进行靶基因预测以及功能注释分析,筛选出关键lncRNA。从lncRNA水平深入探索绵羊捻转血矛线虫对丙硫咪唑耐药机制,同时有望确定绵羊捻转血矛线虫丙硫咪唑耐药功能基因,为进一步从转录组水平阐释捻转血矛线虫丙硫咪唑耐药机制提供重要理论依据。
1 材料与方法1.1 材料本研究捻转血矛线虫丙硫咪唑敏感株和耐药株均采自内蒙古自治区乌兰察布市察右后旗(北纬41°3′–41°59′,东经112°42′–113°30′,平均海拔1 500 m左右),具体方法见参考文献[6]。
1.2 RNA提取及质控选取敏感株雌虫、敏感株雄虫、耐药株雌虫和耐药株雄虫各30条,参照Qiagen+TRK1002 (LC Science,Houston,TX)说明书分别对4个样本进行总RNA提取,用Bioanalyzer 2100和RNA 6000 Nano Lab Chip Kit进行总RNA质量和浓度检测,以RIN值>7.0为合格的标准。
1.3 去rRNA链特异性文库构建四个样本总RNA经过质检合格后,将敏感株雌虫及雄虫核苷酸等量混合,将耐药株的雌虫及雄虫核苷酸等量混合,然后进行敏感株和耐药株文库构建。利用Illumina公司的Ribo-ZeroTMrRNA Removal Kit试剂盒分别对rRNA进行捕获去除,剩余的RNA在高温条件下利用二价阳离子进行片段化。以此为模板,用六碱基随机引物合成第一链cDNA,随后加入大肠杆菌Escherichia coli DNA polymeraseⅠ与RNase H进行第二链cDNA合成,将这些DNA与RNA的复合双链转化成DNA双链,同时在二链中掺入dUTP,将双链DNA的末端补齐为平末端。再在其两端各加上一个A碱基,使其能够与末端带有T碱基的接头进行连接,利用磁珠对其片段大小进行筛选和纯化。以UDG酶消化二链,再通过PCR,使其形成片段大小为300 bp (±50 bp)的文库。最后使用Illumina HiSeq 4000 (LC Bio,China)测序平台按照标准操作对其进行双端测序,读长2×150 bp。
1.4 转录组数据分析首先使用Cutadapt[7]去除测序过程中测序接头及低质量序列,然后利用Bowtie2[8]和Tophat2[9]软件将测序得到的序列比对到捻转血矛线虫参考基因组上,再利用StringTie[10]对转录本进行组装。
识别lncRNA的关键是如何区分编码转录本和非编码转录本,本研究首先去除已知mRNA和具有编码潜能的未知mRNA,然后去除小于200 bp和reads覆盖度小于3的转录本,再利用CPC_Score (Score≤-1)[11]和CNCI_Score(Length≥200且exon≥1且score≤0)[12]软件综合预测lncRNA,能较好地从预测得到的新转录本中排除具蛋白编码能力的转录本,通过转录本含外显子数目≥1、已知lncRNA信息确定、与捻转血矛线虫非lncRNA序列比对等一系列过滤条件后,利用类代码(Class code)o (与参考转录本的外显子重叠)、I (内含子区域的转录本)、u (基因间转录本)、j (已知转录本的潜在亚型)及x (与参考转录本反义链的外显子重叠)作为新lncRNA转录本的候选。
对敏感株和耐药株lncRNA的表达量(FPMK值)进行差异分析,以|log2(差异倍数)|>1且P < 0.05作为显著差异表达基因筛选的阈值。将顺式调控定义为染色体上下游100 kb范围内存在差异表达的lncRNA与差异表达的mRNA,反式调控定义为lncRNA跨染色体调控基因的表达,主要基于lncRNA与mRNA序列之间形成二级结构所需要自由能的大小[13]。并然后利用GO和KEGG Pathway数据库对lncRNA靶向的差异mRNA进行功能注释和代谢通路分析。
2 结果与分析2.1 RNA-Seq测序质量评估对捻转血矛线虫敏感株和耐药株分别构建cDNA文库并进行转录组测序,将得到的数据进行质控和组装,去除低质量的数据,敏感株和耐药株分别获得86 162 364和89 454 054个读长,Q30质量值百分率分别达到了97.53%和97.38%,表明测序质量较高,符合后续分析要求。将有效序列比对到捻转血矛线虫参考基因组,结果显示,敏感虫株与参考基因组的比对率为85.97%,耐药虫株与参考基因组的比对率为94.56%。
2.2 lncRNA的鉴定和特征比较使用带有“u”、“i”、“x”、“j”和“o”的5类代码作为新lncRNA转录本的候选,共鉴定出6 505个新的lncRNA,其中敏感株6 377个,耐药株6 356个(表 1),同时鉴定出16 209个mRNA。对lncRNA与mRNA的结构特征(长度分布、和外显子数目)进行统计,结果表明,长度小于500 bp的lncRNA在总lncRNA中的分布密度较高,占58.4%,而mRNA的长度分布主要集中在大于1 000 bp,占总mRNA的44.0% (图 1)。外显子统计结果表明,鉴定出的lncRNA中有很大一部分仅含有1–2个外显子,占lncRNA总数的81.6%,mRNA中的外显子个数分布则较均匀(图 2)。利用FPKM值归一化转录本表达量,根据中位数可以看出mRNA表达水平高于lncRNA (图 3)。
表 1 lncRNA的鉴定Table 1 Identification of lncRNA
| Sample | Class code | Total | |||||
| i | j | o | u | x | = | ||
| HC | 509 | 450 | 85 | 4 952 | 379 | 0 | 6 377 |
| HC_BZ | 503 | 449 | 87 | 4 937 | 382 | 0 | 6 356 |
表选项
 |
| 图 1 敏感株与耐药株lncRNA和mRNA转录本长度分布 Fig. 1 Transcript length distribution of lncRNA and mRNA in sensitive and resistant strains. |
| 图选项 |
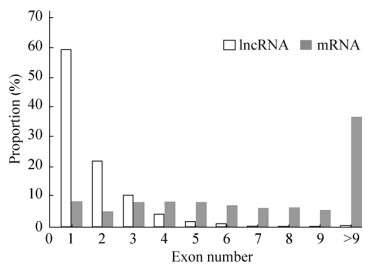 |
| 图 2 敏感株与耐药株lncRNA和mRNA外显子数目分布 Fig. 2 Exon number distribution of lncRNA and mRNA in sensitive and resistant strains. |
| 图选项 |
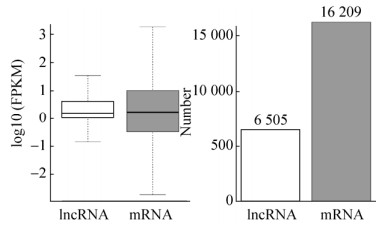 |
| 图 3 lncRNA和mRNA转录本表达水平及数量 Fig. 3 Expression level and quantity of lncRNA and mRNA transcript. |
| 图选项 |
2.3 lncRNA差异表达分析利用FPKM值对两样本进行归一化处理,以P < 0.05及log2 (fold change)>1或log2 (fold change) < -1为标准筛选差异lncRNA。结果表明,筛选条件下共获得168个差异显著的lncRNA,其中表达上调的有92个,表达下调的有76个(图 4)。
 |
| 图 4 样本间的差异lncRNA散点图 Fig. 4 Scatter plot of differentially expressed lncRNA. |
| 图选项 |
2.4 lncRNA靶基因预测通过皮尔森相关系数和自由能值对lncRNA进行靶基因预测,结果显示,共得到562个潜在靶基因,其中潜在顺式调控(cis)靶基因146个,潜在反式调控(trans)靶基因416个。以P < 0.05及log2(fold change)>1或log2(fold change) < -1为阈值,差异显著的靶基因416个。敏感株和耐药株差异倍数前10的lncRNA及靶基因如表 2所示。
表 2 差异表达基因最大的10个lncRNA及其上调和下调靶基因Table 2 Top 10 up and down-regulated lncRNA with the greatest fold changes and its targer genes
| LncRNA | Log2 (Sensitive/Resistant) | Predicted target genes |
| MSTRG.12738 | -9.84 | HCOI_01448700(Ribosomal protein L38e) |
| MSTRG.4271 | -10.59 | HCOI_00455500(Similar to piggyBac-derived 2) |
| MSTRG.20243 | -10.46 | HCOI_02146000(Cytochrome P450) |
| MSTRG.17393 | -7.71 | HCOI_01885300(C. briggsae CBR-LGC-18 protein) |
| MSTRG.17550 | -7.65 | HCOI_01723300(Zinc finger domain containing protein) |
| MSTRG.8198 | 10.58 | HCOI_00835800(Deoxynucleoside kinase) |
| MSTRG.9190 | 7.58 | HCOI_00938300(Protein Y57G11C.48) |
| MSTRG.10083 | 7.40 | HCOI_01020700(Protein UNC-89) |
| MSTRG.2723 | 6.75 | HCOI_00304700(Von Willebrand factor and C-type lectin) |
| MSTRG.13136 | 6.04 | HCOI_01486200(Fps Fes Fer CIP4 homology and RhoGAP) |
表选项
2.5 差异lncRNA靶基因GO和KEGG富集分析通过对鉴定出的所有lncRNA和mRNA的表达量情况进行相关性分析,而后从中筛选出与lncRNA的表达模式极显著相关的mRNA,从而对该lncRNA靶基因进行功能的预测和KEGG富集分析。
GO分析结果显示,差异显著lncRNA的靶基因共注释到641条GO terms,其中生物学过程317条,细胞组分113条,分子功能211条。主要注释到代谢过程、蛋白水解、线虫发育、虫卵孵化死亡;细胞膜、细胞器、细胞质;转移酶活性、核苷酸结合、ATP结合、蛋白结合等(图 5)。
 |
| 图 5 GO分析差异lncRNA靶基因 Fig. 5 GO analysis of the target genes of DElncRNAs. |
| 图选项 |
KEGG富集分析结果显示,捻转血矛线虫敏感株和耐药株差异表达lncRNA的靶基因共富集到92条通路,富集程度较高的前10条通路如表 3所示,其中与耐药性相关的通路有药物代谢-其他酶(Drug metabolism-other enzymes)、药物代谢-细胞色素P450 (Drug metabolism- cytochrome P450)、细胞色素P450对异生素的代谢(Metabolism of xenobiotics by cytochrome P450)等。
表 3 DElncRNA差异靶基因KEGG富集前10通路Table 3 10 enriched KEGG pathways of DElncRNA
| KEGG pathway | DEGs number | Total DEGs number | P-value |
| Drug metabolism-other enzymes | 4 | 70 | 3.24×10–4 |
| P450 Drug metabolism-cytochrome P450 | 4 | 70 | 1.16×10–4 |
| Metabolism of xenobiotics by cytochrome P450 | 4 | 70 | 1.32×10–3 |
| Glutathione metabolism | 4 | 70 | 6.47×10–3 |
| Apoptosis | 2 | 70 | 5.59×10–3 |
| Carbon metabolism | 12 | 70 | 2.74×10–7 |
| Endocytosis | 12 | 70 | 2.30×10–6 |
| Fatty acid metabolism | 8 | 70 | 1.25×10–4 |
| Valine, leucine and isoleucine degradation | 8 | 70 | 7.03×10–5 |
| MAPK signaling pathway | 6 | 70 | 1.42×10–5 |
表选项
3 讨论细菌的耐药机制较为简单,研究较成熟,而多细胞真核生物的耐药性显然要复杂得多,涉及真核生物和真核细胞双重的药物代谢、药效机制。随着高通量测序的发展,越来越多的lncRNA受到广大****的关注,目前已经鉴定的lncRNA主要与癌症、肿瘤耐药相关[14-15]。迄今为止,关于寄生虫耐药性的lncRNA,特别是有关捻转血矛线虫的研究未见报道。在本研究中以捻转血矛线虫耐药性为出发点,对敏感株和耐药株进行比较转录组学测序分析,鉴定出lncRNA并筛选出差异候选lncRNA的靶基因,在转录组层面为探讨捻转血矛线虫耐药性与lncRNA的关联提供有价值的数据。并且mRNA表达水平高于lncRNA,与组织中的表达趋势一致[15]。
在敏感株和耐药株中分别鉴定出6 377个和6 356个lncRNA,以log2 (差异倍数)>1或log2 (差异倍数) < -1且P < 0.05为阈值进行显著差异分析,共获得168个差异显著的lncRNA。并预测出差异靶基因562个,GO分析靶基因,共注释到3大功能中的641条GO terms,其中生物学过程317条,细胞组分113条,分子功能211条。在生物学过程中,差异的靶基因较多注释在代谢过程、转运过程等,除了这些常规的注释过程外,蛋白水解、线虫发育、虫卵孵化死亡等生物学过程也占了较大的比重,此结果可能是耐药株在药物的选择压力下,为响应药物选择压力的原因。在细胞组分中,差异的靶基因较多注释到细胞膜、细胞器和细胞质上。在细胞组分中,差异的靶基因除了较多注释到转移酶活性、核苷酸结合等常规的功能外,另外还富集到ATP结合、GTP结合、ATP酶活性、β-微管蛋白结合、微管结合、微管运动和G蛋白偶联GABA受体活性等。ATP蛋白属于转运蛋白的一种,研究发现该蛋白可以将药物泵出细胞外从而产生耐药性,据报道ATP结合蛋白在肿瘤耐药、癌症耐药中扮演了很重要的角色[16-17]。在本研究中该蛋白显著富集,推测该蛋白参与捻转血矛线虫丙硫咪唑耐药性的产生。丙硫咪唑以β-微管蛋白为靶点,阻止寄生虫微管蛋白聚合组装,从而导致虫体死亡[18],本研究中β-微管蛋白结合、微管结合、微管运动等在差异表达基因中均被富集,推测β-微管蛋白在耐药虫株中发挥重要作用,与前人研究结果一致[19]。
KEGG Pathway富集结果显示前10条通路中,包括药物代谢-其他酶(Drug metabolism-other enzymes)、药物代谢-细胞色素(P450 Drug metabolism-cytochrome P450)、细胞色素P450对异生素的代谢(Metabolism of xenobiotics by cytochrome P450)、谷胱甘肽代谢(Glutathione metabolism)等,这些信号通路均与耐药性有关,可能是DElncRNA共表达基因的重要KEGG途径,相关的lncRNA可能潜在地参与捻转血矛线虫丙硫咪唑耐药性的过程。该通路下的lncRNA可能在丙硫咪唑药物选择压力下,通过基因表达量的变化来发挥耐药性的作用。
在差异表达基因最大的10个lncRNA中,其靶基因细胞色素P450在敏感株中的表达水平显著低于耐药株。细胞色素P450是寄生虫体内药物代谢过程重要的系统,主要起着解毒等作用,目前在昆虫、按蚊对杀虫剂耐药性中研究较多[20-21]。Yilmaz等[22]通过荧光定量PCR对阿苯达唑不同敏感程度的捻转血矛线虫敏感株和耐药株P450家族进行检测,结果表明该家族基因在阿苯达唑耐药株中的表达量比敏感株高2.4–3.7倍,与本研究结果一致。差异表达lncRNA的靶基因除细胞色素P450与捻转血矛线虫阿苯达唑耐药性相关外,笔者未发现其他的靶基因与捻转血矛线虫耐药性有相关联系性的研究。但是锌指结构域含有蛋白(Zinc finger domain-containing protein)、脱氧核苷激酶(Deoxynucleoside kinase)等基因同样参与了细胞凋亡、多重耐药的过程。有关捻转血矛线虫阿苯达唑耐药性中部分信号通路及耐药基因的研究仍属未知,下一步可对样本间差异表达的
lncRNA进行生物功能注释分析及生物学功能验证,进一步对差异表达的lncRNA进行功能及代谢通路富集分析,以阐述捻转血矛线虫lncRNA对丙硫咪唑耐药性产生的分子调控机制。
4 结论本研究采用RNA-Seq测序技术对捻转血矛线虫敏感株和耐药株进行转录组学测序,筛选出差异的lncRNAs,并对差异显著的lncRNAs与其共表达的差异cis和trans调控靶基因进行生物信息学分析,推测lncRNA可能对捻转血矛线虫丙硫咪唑耐药性具有重要的调节作用,为从lncRNA角度分析捻转血矛线虫敏感株和耐药株的差异及丙硫咪唑耐药机制的进一步研究提供理论基础。
参考文献
| [1] | Wang CQ, Li FF, Zhang ZZ, et al. Recent research progress in China on Haemonchus contortus. Front Microbiol, 2017, 8: 1509. |
| [2] | Muchiut SM, Fernández AS, Steffan PE, et al. Anthelmintic resistance:management of parasite refugia for Haemonchus contortus through the replacement of resistant with susceptible populations. Vet Parasito, 2018, 254: 43-48. DOI:10.1016/j.vetpar.2018.03.004 |
| [3] | Zhao XL, Wang SY, Huhe BTE, et al. Comparative transcriptome analysis between before and after administration of albendazole resistant strain of Haemonchus contortus. Acta Vet Zoot Sin, 2019, 50(3): 637-644 (in Chinese). 赵学亮, 王姝懿, 呼和巴特尔, 等. 捻转血矛线虫阿苯达唑耐药株给药前后比较转录组学分析. 畜牧兽医学报, 2019, 50(3): 637-644. |
| [4] | Mowel WK, Kotzin JJ, McCright SJ, et al. Control of immune cell homeostasis and function by lncRNAs. Trends Immunol, 2018, 39(1): 55-69. |
| [5] | Li YL, Ye Y, Feng BM, et al. Long noncoding RNA lncARSR promotes doxorubicin resistance in hepatocellular carcinoma via modulating PTEN-PI3K/Akt pathway. J Cell Biochem, 2017, 118(12): 4498-4507. DOI:10.1002/jcb.26107 |
| [6] | Zhao XL, Liu XL, Su Q, et al. Epidemiological investigation on Haemonchus contortus in sheep in Chayouhouqi of Inner Mongolia and detection of its drug resistance. China Anim Health Inspect, 2019, 36(5): 6-10 (in Chinese). 赵学亮, 刘晓磊, 苏倩, 等. 2018年内蒙古察右后旗绵羊捻转血矛线虫流行病学调查及耐药性检测. 中国动物检疫, 2019, 36(5): 6-10. |
| [7] | Martin M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet J, 2011, 17(1): 10-12. DOI:10.14806/ej.17.1.200 |
| [8] | Langmead B, Salzberg SL. Fast gapped-read alignment with Bowtie 2. Nat Methods, 2012, 9(4): 357-359. DOI:10.1038/nmeth.1923 |
| [9] | Kim D, Pertea G, Trapnell C, et al. TopHat2:accurate alignment of transcriptomes in the presence of insertions, deletions and gene fusions. Genome Biol, 2013, 14(4). |
| [10] | Pertea M, Pertea GM, Antonescu CM, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads. Nat Biotechnol, 2015, 33(3): 290-295. |
| [11] | Kong L, Zhang Y, Ye ZQ, et al. CPC:assess the protein-coding potential of transcripts using sequence features and support vector machine. Nucleic Acids Res, 2007, 35(Web Server issue): W345-W349. |
| [12] | Sun L, Luo HT, Bu DC, et al. Utilizing sequence intrinsic composition to classify protein-coding and long non-coding transcripts. Nucleic Acids Res, 2013, 41(17): e166. DOI:10.1093/nar/gkt646 |
| [13] | Yan PX, Luo S, Lu JY, et al. Cis-and trans-acting lncRNAs in pluripotency and reprogramming. Curr Opin Genet Dev, 2017, 46: 170-178. |
| [14] | Chen XM, Shi YL, Zhou KW, et al. A bibliometric analysis of long non-coding RNA and chemotherapeutic resistance research. Oncotarget, 2019, 10(35): 3267-3275. |
| [15] | Xu YD, Shang J, Li M, et al. LncRNA DANCR accelerates the development of multidrug resistance of gastric cancer. Eur Rev Med Pharmacol Sci, 2019, 23(7): 2794-2802. DOI:10.26355/eurrev_201904_17554 |
| [16] | Wang L, Liu L, Chen YT, et al. Correlation between adenosine triphosphate (ATP)-binding cassette transporter G2(ABCG2) and drug resistance of esophageal cancer and reversal of drug resistance by artesunate. Pathol Res Pract, 2018, 214(9): 1467-1473. |
| [17] | Johnson ZL, Chen J. ATP binding enables substrate release from multidrug resistance protein 1. Cell, 2018, 172(1/2): 81-89. |
| [18] | Shen DD, Peng ZW, Hu M, et al. A detection of benzimidazole resistance-associated SNPs in the isotype-1 β-tubulin gene in Haemonchus contortus from wild blue sheep (Pseudois nayaur) sympatric with sheep in Helan Mountains, China. BMC Vet Res, 2019, 15: 89. DOI:10.1186/s12917-019-1838-4 |
| [19] | Atanásio-Nhacumbe A, Lambert SM, Da Silva Souza BMP, et al. Molecular detection of benzimidazole resistance levels associated with F167Y and F200Y polymorphisms in Haemonchus contortus of goats from Mozambique. Parasitol Res, 2019, 118(1): 245-253. DOI:10.1007/s00436-018-6162-6 |
| [20] | Su H, Gao Y, Liu Y, et al. Comparative transcriptome profiling reveals candidate genes related to insecticide resistance of Glyphodes pyloalis. Bull Entomol Res, 2020, 110(1): 57-67. DOI:10.1017/S0007485319000257 |
| [21] | Namountougou M, Soma DD, Kientega M, et al. Insecticide resistance mechanisms in Anopheles gambiae complex populations from Burkina Faso, West Africa. Acta Trop, 2019, 197: 105054. |
| [22] | Yilmaz E, Ramünke S, Demeler J, et al. Comparison of constitutive and thiabendazole-induced expression of five cytochrome P450 genes in fourth-stage larvae of Haemonchus contortus isolates with different drug susceptibility identifies one gene with high constitutive expression in a multi-resistant isolate. Int J Parasitol Drugs Drug Resist, 2017, 7(3): 362-369. DOI:10.1016/j.ijpddr.2017.10.001 |
