

华南师范大学 生命科学学院 广东省植物发育与生物工程重点实验室,广东 广州 510631
收稿日期:2016-10-18;接收日期:2017-01-03 基金项目:国家自然科学基金(No. 31571759) 资助
摘要: CRISPR/Cas9是新兴的基因组编辑技术,该技术操作简单、效率高,已成为目前最主流的基因组编辑技术。利用该技术进行突变体创制,对基因功能研究和育种应用具有重要的意义,而快速、高效、低成本的基因编辑个体鉴定是其中的重要环节。本研究对影响CELⅠ粗提物鉴定CRISPR/Cas9介导的水稻基因编辑个体的条件,包括蛋白用量及作用时间、PCR反应缓冲液等条件进行了探索,并将整个检测体系集成于一管操作。同时,采用CELⅠ粗提物检测了CRISPR/Cas9介导水稻stn1突变的T0代植株及后代,对杂合突变、纯合突变及双等位突变的鉴定策略进行了探讨;该方法检测正确率经测序验证达100%。上述结果表明,采用CELⅠ粗提物检测突变体与已有方法比较具有廉价、快速和高效的特点。
关键词: CELⅠ粗提物 CRISPR/Cas9 突变体 鉴定 stn1
Efficient identification of gene knockout mutant mediated by CRISPR/Cas9 by CELⅠ crude extracts
Wanbing Luo, Qiupeng Lin, Xiaoxia Li, Zejiao Zhou, Huiyong Yang, Rongyu Du, Hongqing Li


Guangdong Key Laboratory of Biotechnology for Plant Development, School of Life Sciences, South China Normal University, Guangzhou 510631, Guangdong, China
Received: October 18, 2016; Accepted: January 3, 2017
Supported by: National Natural Science Foundation of China (No. 31571759)
Corresponding author:Hongqing Li. Tel/Fax: +86-20-85211375; E-mail: hqli@scnu.edu.cn
Abstract: CRISPR/Cas9, emerged as an efficient and powerful gene editing technology, has become the mainstream genome editing technology. Constructing mutants using CRISPR/Cas9 system is of great significance to the functional study and breeding application of useful genes. As the basis of the technology, a method for identification of mutation with efficiency and lower cost is needed. In this report, we studied the factors influencing mutation detected by CEL Ⅰ crude extracts, such as the amount of protein, enzyme incubation time, PCR buffers. Under the optimized conditions, we can integrate the mutation detection steps into one-tube reaction. We used this system to examine the mutation types and frequency of rice stn1 mediated by CRISPR/Cas9. We also used this method to identify different mutation types including homozygous, heterozygous and bi-allelic mutations. The accuracy of this method reached 100% verified by sequencing. Altogether, our results showed that using CELⅠ crude extracts was an efficient and low cost method for identification of CRISPR/Cas9 mediated mutation.
Key words: CELⅠ crude extracts CRISPR/Cas9 mutant identification stn1
随着测序技术的发展,许多物种的全基因组测序工作已经完成。阐明基因的功能及构建基因调控和互作网络成为现阶段的主要任务。构建基因突变个体是十分有效的基因功能研究手段。基因组编辑技术的出现,使得快速、高效地定点突变目的基因成为可能。
CRISPR/Cas9系统是细菌的免疫系统,其主要作用是防御外来质粒或噬菌体DNA的入侵。科学家在大肠杆菌发现了特殊的成簇的规律间隔的短回文重复序列[1],并于2002年正式将其命名为CRISPR (Clustered regularly interspaced short palindromic repeats)[2]。2013年,Science杂志同期刊登了两篇关于CRISPR/Cas9介导的基因敲除的文献,首次报道了CRISPR/Cas9系统可用于人类和小鼠细胞的基因组编辑,证明了CRISPR技术可用于编辑高等真核基因组[3-4]。此后,CRISPR技术也被应用到植物基因组编辑中,并在模式植物拟南芥、烟草以及小麦、水稻、高粱、玉米等重要农作物中获得成功[5-10],并且证明了产生的编辑可以在植物中稳定遗传[11]。此外,基因编辑过的植物可以通过自交、回交等手段,获得不带CRISPR元件的基因编辑株[12]。CRISPR/Cas9引入突变的方式是通过sgRNA识别靶位点,利用Cas9的核酸内切酶活性使靶位点DNA的双链发生断裂(Double-strand breaks,DSBs),从而激活个体自身修复机制。编辑个体可以通过非同源末端连接介导的修复方式(Non-homologous end joining,NHEJ)和同源重组介导的修复方式(Homology-directed repair,HDR)对双链断裂处进行修复[13]。通过NHEJ介导的修复方式进行修复时,断裂的DNA末端重新连接,这种非精确的修复会导致在断裂位置上产生少量核苷酸的插入或删除,进而产生突变[14]。CRISPR技术与传统基因打靶技术相比突变效率更高,与ZFN、TALEN等基因组编辑技术相比具有耗时更短、操作简单等特点,因此已被广泛用于突变体的构建。由于编辑效率的限制,通过CRISPR技术产生的大量编辑个体仍需通过进一步的筛选鉴定,因此建立快速、高效和低成本的鉴定体系尤为重要。
CELⅠ酶是芹菜中发现的可以特异识别DNA双链中的错配位点并切割双链的核酸内切酶[15]。研究表明,芹菜CEL Ⅰ酶粗提物可以用于点突变的检测[16-17]。利用该酶的特点,可以有效鉴定CRISPR突变体。本文以水稻为材料构建了dep1基因和stn1基因的突变体,利用CEL Ⅰ酶粗提物进行鉴定,并对鉴定条件进行了优化,将PCR反应、产物变性、复性处理和CEL Ⅰ酶切整合在一管中进行,结合琼脂糖凝胶电泳,建立了一种快速、高效和廉价的突变体检测系统。
1 材料与方法1.1 材料水稻“中花11” (Oryza sativa L.)由本实验室保存。水稻dep1突变株由本实验室创制[18]。rTaq聚合酶、LA Taq聚合酶缓冲液购自大连TaKaRa生物有限公司。B Taq聚合酶、KOD聚合酶均购自东洋纺(上海)生物科技有限公司。大肠杆菌感受态菌株JM109、根癌农杆菌感受态菌株EHA105为本实验室保存。CRISPR载体由华南农业大学刘耀光教授提供[19]。限制性内切酶和T4 DNA连接酶购自大连TaKaRa生物有限公司。质粒抽提试剂盒为Axygen公司产品。引物合成由上海生工生物技术有限公司完成。测序由北京睿博兴科生物技术有限公司完成。蛋白浓度测定BCA试剂盒由碧云天生物技术公司提供。
1.2 方法1.2.1 CEL Ⅰ酶粗提及蛋白质浓度测定蒸馏水冲洗芹菜后,用无菌吸水纸擦干,去掉根部,用榨汁机榨取并收集汁液,在4 ℃的条件下用Bradley等的方法[20]进行CELⅠ酶的粗提取,提取物保存于–20 ℃备用。蛋白测定的方法采用BCA蛋白浓度测定试剂盒进行测定,调整蛋白浓度至1 μg/μL。
1.2.2 构建水稻stn1突变用的CRISPR/Cas9载体stn1为端粒结合蛋白CST复合体中的组分,是染色体末端的保护蛋白[21-22]。为了突变水稻中stn1基因,首先设计用于构建CRISPR载体的spacer序列,其gRNA靶位点位于stn1基因CDS序列的第100 bp (图 1),两条接头互补序列见表 1。
 |
| 图 1 CRISPR载体图谱和stn1的CRISPR靶位点设计 Figure 1 Physical map of CRISPR vector and CRISPR target site of stn1. |
| 图选项 |
表 1 本研究所用的引物Table 1 Primers used in this research
| Primer name | Primer sequence (5'–3') |
| stn1CR-Fw | GCCGTTCGCTCGCTGCGGCCGTA |
| stn1CR-Re | CCCATACGGCCGCAGCGAGCGAA |
| dep1CR-Fw | GCCGATCTTGAAGCAGCTGTACA |
| dep1CR-Re | AAACTGTACAGCTGCTTCAAGAT |
| stn1-Fw | AGAGGGAGACCTGCAGGAGCGAA |
| stn1-Re | CGTTGAGGATGTCAGAGGTGGTGAG |
| dep1-Fw | GCCTCTAGTCCCTTTGTATG |
| dep1-Re | GAGCAGGAGAAGCAGTCC |
| U6-Fw | GAGCTTGGAGTGGATGGAAT |
| U6-Re | AGCACCGACTCGGTGCCACT |
表选项
将所需接头引物等量混合,经变、复性处理,与Bsa Ⅰ酶切的载体相连接,转化大肠杆菌,提取阳性菌株的质粒,测序。将确认无误的质粒转入农杆菌EHA105,获得含有用于水稻stn1突变的农杆菌菌株EHA105/CRISPRstn1。
1.2.3 农杆菌介导转化水稻参照Li等[18]的方法,利用农杆菌EHA105/ CRISPRstn1转化水稻品种中花11的愈伤组织,用潮霉素筛选获得再生组培苗。组培苗经炼苗后,移栽盆钵,即为T0代植株。按转基因水稻种植要求严格管理。
1.2.4 扩增dep1及stn1突变位点所在片段提取水稻叶片的基因组DNA,以引物组合dep1-Fw/dep1-Re和stn1-Fw/stn1-Re进行PCR扩增,理论产物长度约为948 bp和491 bp。反应体系包括基因组DNA 1μL,10×PCR缓冲液2.5 μL,dNTPs (2 mmol/L) 2.5 μL,上游引物(10 μmol/L) 1 μL,下游引物(10 μmol/L) 1 μL,DNA polymerase 1 U,补去离子水到25 μL。PCR反应的参数设置如下:94 ℃预变性5 min,然后进行35个循环的PCR扩增(94 ℃,30 s;退火温度,30 s;72 ℃,1 min),最后72 ℃延伸10 min。其中stn1退火温度为65 ℃,dep1的退火温度为55 ℃。
1.2.5 CEL Ⅰ酶粗提物切割PCR产物转化株DNA经PCR扩增后,将产物(或加入等量的野生型DNA扩增产物)进行变、复性处理,再向其中加入CEL Ⅰ酶,42 ℃温育,用EDTA终止反应。取10 μL样品进行电泳检测。
2 结果与讨论2.1 CEL Ⅰ酶粗提物用于突变体的检测dep1是水稻直立密穗控制基因[23],本论文dep1-29突变株由本实验室创制,测序结果表明,该植株为双位点突变(–2/–5) (图 2)。为检测CEL Ⅰ酶粗提物是否能切割异源双链DNA,取1 μL CEL Ⅰ酶粗提物直接加入到经变性、复性处理的PCR样品中,与不加CEL Ⅰ酶的样品进行对比。电泳结果显示CEL Ⅰ酶可以切割异源双链DNA,切出条带符合预期大小,证明CELⅠ酶粗提物适用于点突变的检测。
 |
| 图 2 dep1-29突变株突变位点的测序结果和CEL Ⅰ酶切检测结果(dep1-29 a和dep1-29 b表示dep1-29突变株包含双等位突变的两种突变类型) Figure 2 Sequence of dep1-29 mutant and CEL Ⅰ analysis. dep1-29 a and dep1-29 b showed the target sequences of bi-alleles mutation of dep1-29. |
| 图选项 |
2.2 CEL Ⅰ酶用量及作用时间的优化酶的用量是影响酶切效果的重要因素。因此,采用不同CEL Ⅰ酶粗提物的量对来源于dep1-29的PCR产物进行酶切(图 3),结果表明,粗提物总蛋白用量在0.1 μg时,酶切效果达到最佳。增加粗提物的用量,酶切效果不会有显著提升。
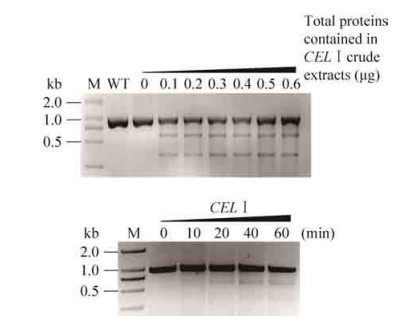 |
| 图 3 CEL Ⅰ酶粗提物用量及酶切时间对突变鉴定的影响 Figure 3 Effects of the amount of CELⅠ crude extracts and digestion time on mutation identification. |
| 图选项 |
酶切时间是影响酶切效果的另一重要因素。采用0.1 μg CEL Ⅰ酶对dep1-29突变体材料的PCR产物进行酶切(图 3)。结果表明,CEL Ⅰ酶作用10 min时可以见到酶切的条带,随着时间增加,CEL Ⅰ酶的切割效率逐渐提升,在40 min时达到最佳。继续增加时间并不能提升CEL Ⅰ酶的切割效率。
2.3 PCR体系对CEL Ⅰ切割效率的影响简便、快捷的突变体鉴定方法要求采用CEL Ⅰ粗提物在合适的PCR缓冲体系中进行酶切。因此,首先选择目前普遍使用的聚合酶和缓冲系统(r Taq聚合酶、LA Taq聚合酶、Blend Taq聚合酶、KOD聚合酶)进行实验(图 4)。结果表明,KOD酶的扩增效果最佳,但CEL Ⅰ酶在该体系中的切割效果不理想。B Taq酶体系和r Taq酶体系中PCR扩增效果差,电泳检测时不能看见1 000 bp处的条带。LA Taq酶体系能够扩增到dep1片段,且CEL Ⅰ酶在该体系中表现出了切割活性。进一步通过采用LA buffer与不同的Taq酶组合,发现B Taq与LA buffer组合可以提高PCR的效率和保证CEL Ⅰ的酶切活性。
 |
| 图 4 不同PCR体系对CEL Ⅰ粗提物酶切效率的影响 Figure 4 Effect of digestion using CELⅠ crude extracts in different PCR systems. |
| 图选项 |
2.4 CEL Ⅰ酶对CRISPR介导的stn1基因敲除株突变进行分型采用农杆菌介导的方法,我们将stn1-CRISPR转化水稻,获得35个独立的转基因株系。由于转基因株中可能含有无突变、杂合突变、纯合突变及双位点突变等多种类型,我们根据CEL Ⅰ酶能切割异源DNA双链的特点,设计了两种方法对上述类型进行区分:1) 将PCR产物直接变性、复性、酶切;2) 将PCR产物与野生型植株的PCR产物等量混合,变性、复性,再用CEL Ⅰ酶切。stn1-1和stn1-4是通过的stn1-CRISPR转化获得的两个突变株系。其中stn1-1的PCR产物变、复性后可以被CEL Ⅰ酶切割,其PCR产物与野生型植株PCR产物混合变、复性后也可以被CEL Ⅰ酶切割,说明突变株为杂合突变或双位点突变。stn1-4的PCR产物变、复性后不能被CEL Ⅰ酶切割,其PCR产物与野生型植株PCR产物混合变、复性后可以被CEL Ⅰ酶切割,说明该突变株为纯合突变。对上述两个突变株PCR产物进行克隆,转化大肠杆菌并挑单克隆进行测序,结果表明stn1-1突变株存在两种突变类型(–3 bp/–1 bp),而stn1-4为单碱基缺失的纯合突变(–1 bp/–1 bp)。以上结果表明,CEL Ⅰ酶不仅可以快速鉴定突变体,而且可以对不同的类型进行有效区分(图 5)。
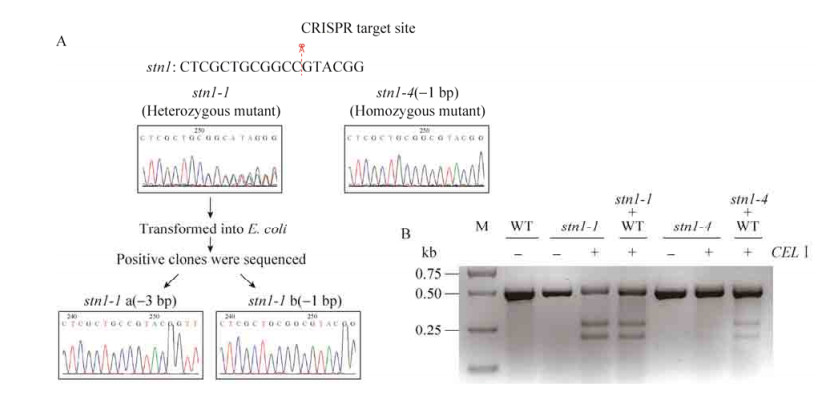 |
| 图 5 CEL Ⅰ酶检测两种不同突变类型的突变株(A:stn1-1和stn1-4两种突变株的测序结果;B:利用CEL Ⅰ酶策略检测杂合和纯合突变株) Figure 5 Identification of two types of mutations using CELⅠ enzyme. (A) Sequences of stn1-1 and stn1-4. (B) Identification of heterozygous and homozygous mutants using CELⅠ strategy. |
| 图选项 |
2.5 CEL Ⅰ酶策略检测CRISPR介导的stn1基因突变株及验证为验证CEL Ⅰ酶策略检测的正确率,利用该策略对CRISPR介导的stn1突变的T0代植株(已测序)进行酶切检测。结果表明,其中#4、#9、#17为纯合突变植株,#8为野生型植株,其余植株为杂合体或双等位突变体或嵌合体(图 6)。CEL Ⅰ酶检测结果与测序结果对比完全一致。以上结果表明利用CEL Ⅰ酶策略在本次实验中的正确率达100%,表明该方法是一种非常理想的检测CRISPR突变株的方法。
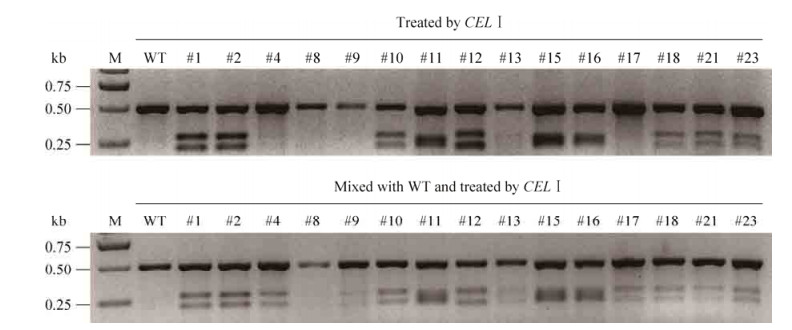 |
| 图 6 CEL Ⅰ酶策略检测CRISPR介导的stn1基因突变株 Figure 6 Identification of stn1 knockout mutant mediated by CRISPR/Cas9 using CEL Ⅰ strategy. |
| 图选项 |
3 讨论3.1 CEL Ⅰ酶策略与其他方法用于鉴定CRISPR基因敲除株的比较CEL Ⅰ酶具有切割异源双链中非配对碱基位点的特性,该特性已被广泛用于突变体的鉴定,本研究采用CEL Ⅰ的粗提物,通过优化酶的用量、作用时间、PCR反应系统及采用检测方法,建立了一个廉价、高效的检测CRISPR方法创制的突变体,并且所有集成于一管的检测方法,其正确率达100%。该方法可以广泛用于CRISPR基因敲除个体初筛及后代鉴定。
商品化的产品CEL Ⅰ价格较高,单个样品检测成本大于3元人民币,而CEL Ⅰ粗提物来源于廉价的芹菜,从600 g材料就能得到15–20 mL的蛋白粗提物,每次用量也只需0.5 μL,一次提取的量至少可用于30 000个样品的鉴定。与采用聚丙烯酰胺凝胶电泳的方法相比[24],采用CEL Ⅰ酶的方法,只需要使用常规的琼脂糖凝胶电泳,操作更加方便、省时。而且,采用CEL Ⅰ酶粗提物时,我们扩增PCR产物一般为1 kb左右,有利于检测大片段的缺失。
T7E1法[25]与CEL Ⅰ策略法非常相似,其工作原理与CEL Ⅰ法相同,都是通过错配酶识别并切割异源双链DNA,从而达到基因型分型的目的。该方法也是目前广泛应用的CRISPR突变个体检测方法。相比于CEL Ⅰ而言,该方法商品化程度更高,应用面更广。然而在大量样品的检测筛选方面,CEL Ⅰ酶策略只需要获得芹菜粗提物,大大降低了成本,更能适应大量样品的筛选,表现出比T7E1更高的性价比。
PCR-SSCP是一种简单、快速的点突变筛查手段[26],其技术原理是PCR扩增后的DNA片段经变性成单链DNA,单链DNA在中性聚丙烯酰胺凝胶中电泳时形成不同的立体构象,其构象直接影响泳动速率,相同长度的DNA单链其核苷酸序列仅有单个碱基的差别,就可以产生立体构象的不同,产生不同的泳动带。PCR-SSCP的缺点是存在假阴性和假阳性,对长度不超过300 bp的待测片段的检出率为70%–80%。与PCR-SSCP相比,CEL Ⅰ粗提物检测法只需要简单的琼脂糖凝胶电泳,而且可检测片段不受长度限制,检出突变的效率高。
PCR/RE (PCR/restriction enzyme)检测法是将靶点切割位点设计在限制性内切酶识别位点上,CRISPR/Cas9诱导的突变会改变该酶切位点,被突变的靶序列不能被限制性内切酶切断,从而可以通过对酶切基因组DNA进行PCR扩增来鉴定突变。其检测特定是:操作简单,突变检出效率高。缺点是CRISPR系统作用的靶点选择受到限制,尤其是需要对特定碱基进行突变时。各种酶的使用也需要一定的费用。与采用PCR/RE (PCR/restriction enzyme)检测法鉴定CRISPR突变个体相比[27],使用CEL Ⅰ酶鉴定的方法可以灵活选择靶位点,使用的是能针对所有位点的廉价的酶。
DNA序列测定法(Direct sequencing,DS)是PCR产物经克隆后测序或直接对PCR产物进行测序,可以直接提供突变的位点和序列。在采用CRISPR系统获得高效率突变的情况下,可以采用PCR产物直接测序的方法进行鉴定。但在突变效率不高或进行子代突变体遗传规律分析时,与DNA序列测定法相比,CEL Ⅰ酶策略具有廉价的特点。
3.2 不同PCR体系对CEL Ⅰ酶策略效果有影响采用CEL Ⅰ酶粗提物进行突变体的鉴定,除廉价外,还需要做到快速和高效。为此,通过整合检测过程的各个环节包括PCR、产物变复性和酶切,达到在同一管中操作。我们发现,不同的PCR体系对基因的扩增效果有影响,且不同的缓冲液对CEL Ⅰ酶的粗提物的切割效率也有影响。其中Blend Taq聚合酶在LA Taq聚合酶所配套的LA buffer中能表现出更加优越的PCR效率以及切割效率。由于不同缓冲液的组成成分不同,对聚合酶和CEL Ⅰ酶切的作用效率存在差异。因此在进行CEL Ⅰ酶策略鉴定的时候应重视PCR体系的选择,建议先通过预实验挑选最适合的PCR体系。
3.3 CEL Ⅰ酶策略对CRISPR突变材料进行鉴定和分型采用CRISPR创制的突变个体多数表现为少量的碱基缺失或增加,且创制的个体在T0代可能为野生型、杂合突变、纯合突变或双位点突变。CEL Ⅰ具有可以切割异质双链DNA的特点,可以对DNA双链中不配对的核苷酸所在的位置进行切割。利用CEL Ⅰ酶的这一特点,可以对CRISPR突变动植物材料进行鉴定。采用该方法还可以对突变类型进行区分。其原理是:杂合突变和双位点突变个体的PCR产物变性、复性后能被CEL Ⅰ酶识别。纯合突变个体PCR产物与野生型个体PCR产物混合后经变、复性处理后可以被CEL Ⅰ酶识别。
参考文献
| [1] | Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product.J Bacteriol, 1987, 169(12): 5429–5433.DOI: 10.1128/jb.169.12.5429-5433.1987 |
| [2] | Zhang BX, Sun QX, Li HF. Advances in genetic modification technologies.Chin J Biotech, 2015, 31(8): 1162–1174.(in Chinese). 张白雪, 孙其信, 李海峰. 基因修饰技术研究进展.生物工程学报, 2015, 31(8): 1162-1174. |
| [3] | Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9.Science, 2013, 339(6121): 823–826.DOI: 10.1126/science.1232033 |
| [4] | Cong L, Ran FA, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems.Science, 2013, 339(6121): 819–823.DOI: 10.1126/science.1231143 |
| [5] | Shan QW, Wang YP, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system.Nat Biotechnol, 2013, 31(8): 686–688.DOI: 10.1038/nbt.2650 |
| [6] | Miao J, Guo DS, Zhang JZ, et al. Targeted mutagenesis in rice using CRISPR-Cas system.Cell Res, 2013, 23(10): 1233–1236.DOI: 10.1038/cr.2013.123 |
| [7] | Feng ZY, Zhang BT, Ding WN, et al. Efficient genome editing in plants using a CRISPR/Cas system.Cell Res, 2013, 23(10): 1229–1232.DOI: 10.1038/cr.2013.114 |
| [8] | Nekrasov V, Staskawicz B, Weigel D, et al. Targeted mutagenesis in the model plant Nicotiana benthamiana using Cas9 RNA-guided endonuclease.Nat Biotechnol, 2013, 31(8): 691–693.DOI: 10.1038/nbt.2655 |
| [9] | Jiang WZ, Zhou HB, Bi HH, et al. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in Arabidopsis, tobacco, sorghum and rice.Nucleic Acids Res, 2013, 41(20): e188.DOI: 10.1093/nar/gkt780 |
| [10] | Liang Z, Zhang K, Chen KL, et al. Targeted mutagenesis in Zea mays using TALENs and the CRISPR/Cas system.J Genet Genomics, 2014, 41(2): 63–68.DOI: 10.1016/j.jgg.2013.12.001 |
| [11] | Feng ZY, Mao YF, Xu NF, et al. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in Arabidopsis.Proc Natl Acad Sci USA, 2014, 111(12): 4632–4637.DOI: 10.1073/pnas.1400822111 |
| [12] | Qu LJ, Guo DS, Zhang JZ, et al. The application of CRISPR/Cas system in plant genome editing.Chinese Bulletin of Life Sciences, 2015, 27(1): 64–70.(in Chinese). 瞿礼嘉, 郭冬姝, 张金喆, 等. CRISPR/Cas系统在植物基因组编辑中的应用.生命科学, 2015, 27(1): 64-70.DOI:10.3785/j.issn.1008-9209.2014.02.241 |
| [13] | Symington LS, Gautier J. Double-strand break end resection and repair pathway choice.Annu Rev Genet, 2011, 45(1): 247–271.DOI: 10.1146/annurev-genet-110410-132435 |
| [14] | Shan QW, Gao CX. Research progress of genome editing and derivative technologies in plants.Hereditas (Beijing), 2015, 37(10): 953–973.(in Chinese). 单奇伟, 高彩霞. 植物基因组编辑及衍生技术最新研究进展.遗传, 2015, 37(10): 953-973. |
| [15] | Till BJ, Burtner C, Comai L, et al. Mismatch cleavage by single-strand specific nucleases.Nucleic Acids Res, 2004, 32(8): 2632–2641.DOI: 10.1093/nar/gkh599 |
| [16] | Han SY, Yang ML, Gai JY, et al. Crude extraction and activity assay of CELⅠ.Hereditas (Beijing), 2006, 28(9): 1112–1116.(in Chinese). 韩锁义, 杨玛丽, 盖钧镒, 等. CELⅠ酶的粗提取及其活性检测.遗传, 2006, 28(9): 1112-1116. |
| [17] | Cui HR, Li CN, Wu DX, et al. A detection method for point mutations based on CEL Ⅰ crude extract.J Nucl Agric Sci, 2011, 25(1): 37–42.(in Chinese). 崔海瑞, 李春楠, 吴殿星, 等. CELⅠ粗提物用于点突变检测的技术.核农学报, 2011, 25(1): 37-42.DOI:10.11869/hnxb.2011.01.0037 |
| [18] | Li MR, Li XX, Zhou ZJ, et al. Reassessment of the four yield-related genes Gn1a, DEP1, GS3, and IPA1 in rice using a CRISPR/Cas9 system.Front Plant Sci, 2016, 7: 377. |
| [19] | Ma XL, Zhang QY, Zhu QL, et al. A robust CRISPR/Cas9 system for convenient, high-efficiency multiplex genome editing in monocot and dicot plants.Mol Plant, 2015, 8(8): 1274–1284.DOI: 10.1016/j.molp.2015.04.007 |
| [20] | Till BJ, Zerr T, Comai L, et al. A protocol for TILLING and ecotilling in plants and animals.Nat Protoc, 2006, 1(5): 2465–2477.DOI: 10.1038/nprot.2006.329 |
| [21] | Martín V, Du LL, Rozenzhak S, et al. Protection of telomeres by a conserved Stn1-Ten1 complex.Proc Natl Acad Sci USA, 2007, 104(35): 14038–14043.DOI: 10.1073/pnas.0705497104 |
| [22] | Song XY, Leehy K, Warrington RT, et al. STN1 protects chromosome ends in Arabidopsis thaliana.Proc Natl Acad Sci USA, 2008, 105(50): 19815–19820.DOI: 10.1073/pnas.0807867105 |
| [23] | Huang XZ, Qian Q, Liu ZB, et al. Natural variation at the DEP1 locus enhances grain yield in rice.Nat Genet, 2009, 41(4): 494–497.DOI: 10.1038/ng.352 |
| [24] | Zhu XX, Xu YJ, Yu SS, et al. An efficient genotyping method for genome-modified animals and human cells generated with CRISPR/Cas9 system.Sci Rep, 2014, 4: 6420. |
| [25] | Mashal RD, Koontz J, Sklar J. Detection of mutations by cleavage of DNA heteroduplexes with bacteriophage resolvases.Nat Genet, 1995, 9(2): 177–183.DOI: 10.1038/ng0295-177 |
| [26] | Orita M, Iwahana H, Kanazawa H, et al. Detection of polymorphisms of human DNA by gel electrophoresis as single-strand conformation polymorphisms.Proc Natl Acad Sci USA, 1989, 86(8): 2766–2670.DOI: 10.1073/pnas.86.8.2766 |
| [27] | Gao X, Ma N, Wang DY, et al. Characterize CRISPR/Cas9 established Chrm3 gene knockout mice by enzyme restriction.Chin J Biochem Mol Biol, 2016, 32(4): 404–409.(in Chinese). 高旭, 马宁, 王大勇, 等. 限制性内切酶策略鉴定CRISPR/Cas9介导的Chrm3基因敲除小鼠.中国生物化学与分子生物学报, 2016, 32(4): 404-409. |
