

1 南京工业大学 生物与制药工程学院 材料化学工程国家重点实验室,江苏 南京 211816;
2 扬州工业职业技术学院 化学工程学院,江苏 扬州 225127;
3 南京工业大学 食品与轻工学院 材料化学工程国家重点实验室,江苏 南京 211816;
4 南京工业大学 药学院,江苏 南京 211816
收稿日期:2016-08-30;接收日期:2016-11-15 基金项目:国家自然科学基金 (No. 21406111),江苏省自然科学基金 (No. BK20130917),大学生创新与实验开放基金 (No. 2016DC365) 资助
摘要: 作为类异戊二烯化合物中四萜的代表性产品番茄红素,在生物体中是典型的多酶参与催化的反应产物,在2-甲基-D-赤藓糖醇-4-磷酸盐 (MEP) 和甲羟戊酸 (MVA) 合成途径中起着至关重要的作用。从番茄红素在原核和真核中的多酶合成途径出发,针对番茄红素合成途径的各种优化策略,首先介绍了多酶合成中的常规调控方法,包括多基因共表达质粒构建、基因顺序调控、启动子与核糖体结合位点调控及基因敲除和替换等方法。之后介绍了一些新型的多酶调控方法,包括多片段组装技术、人工支架自组装方法等。最后重点介绍了这些多酶调控方法在番茄红素合成中的应用。这些多酶合成调控方法为构建高产番茄红素菌株提供了极大的启发和研究基础。
关键词: 番茄红素 多酶调控 支架 基因工程 多片段组装
Research progress in multi-enzyme regulation of genetically engineered bacteria producing lycopene
Jiali Xu1, Siyi Zuo1, Chengjia Xie2, Ling Jiang3, Shuang Li1, He Huang4, Xian Xu4


1 State Key Laboratory of Materials-Oriented Chemical Engineering, College of Biotechnology and Pharmaceutical Engineering, Nanjing Tech University, Nanjing 211816, Jiangsu, China;
2 College of Chemical Engineering, Yangzhou Polytechnic Institute, Yangzhou 225127, Jiangsu, China;
3 State Key Laboratory of Materials-Oriented Chemical Engineering, College of Food Science and Light Industry, Nanjing Tech University, Nanjing 211816, Jiangsu, China;
4 College of Pharmacy, Nanjing Tech University, Nanjing 211816, Jiangsu, China
Received: August 30, 2016; Accepted: November 15, 2016
Supported by: National Natural Science Foundation of China (No. 21406111), Natural Science Foundation of Jiangsu Province, China (No. BK20130917), College Students' Innovation and Experimental Open Fund (No. 2016DC365)
Corresponding author:Xian Xu. Tel: +86-25-58139942; E-mail: xuxian@njtech.edu.cn
Abstract: Lycopene plays a crucial role in the biosynthesis pathway of 2-methyl-derythritol-4-phosphate (MEP) and mevalonic acid (MVA). It is a representative product of isoprenoid family, and a typical product of multi-enzyme catalytic reaction in organism. In this paper, we first introduced the general regulation methods in multi-enzyme synthesis reaction, including the construction of multi gene co-expression plasmid, gene order regulation, promoter and ribosome binding site regulation, gene knockout and replacement, aiming at the optimization strategies of multi-enzyme catalytic reaction in lycopene synthesis pathway. Meanwhile, we introduced several new regulation methods in multi-enzyme reaction, including multi-fragment assembly technology, artificial scaffold self-assembly methods and so on. At last, we summarized the application of these multi-enzyme regulation methods in lycopene synthesis. These methods provide a great inspiration and research foundation for the construction of lycopene-producing strains with high yield.
Key words: lycopene multi-enzyme regulation scaffold genetic engineering multi-fragment assembly
番茄红素是一类C40的异戊二烯类化合物,其研究起始于1873年,由Hartsen首次分离得到番茄红素的晶体。1910年,Willstaller和Escher首次确定番茄红素分子式C40H56,分子量为536.85[1],番茄红素具有11个共扼双键及2个非共扼双键的非环状平面多不饱和脂肪烃链,经过环化后可形成β-胡萝卜素[2]。作为自然界中被发现的最强抗氧化剂之一,番茄红素具有预防心脑血管疾病、提高免疫力、延缓衰老等功效,在癌症的防治方面效果显著[3-4],被广泛应用于食品、药剂、保健品以及化妆品等行业中[5]。
目前已报道的番茄红素生产方法有化学合成法、天然提取法和微生物发酵法[6]。化学合成法步骤繁琐、存在毒副作用,制约了番茄红素的规模化生产。从天然植物中提取番茄红素可利用常规溶剂提取、超声辅助提取、有机溶剂浸提、酶反应、微波辐射萃取及超临界CO2萃取等方法[7]。这些方法中以有机溶剂浸提法提取番茄红素最为普遍,但这种方法提取效率低,工艺复杂且成本高。除此之外,天然提取法提取番茄红素还易受番茄红素产量及季节性变化的影响。
现阶段随着基因工程技术的迅速发展,以及代谢工程和合成生物学方法的引入,人们越来越关注利用微生物发酵法生产番茄红素。迄今为止,已有噬夏孢欧文氏菌Erwinia uredovora、草生欧文氏杆菌Erwinia herbicola、卷枝毛霉Mucor circinelloides、布氏须霉Phycomyces blakesleeanus、产朊假丝酵母Candida utilis、三孢布拉氏霉菌Blakeslea trispora、藻类Dunaliellasalina和基因工程改造的酵母和细菌等微生物被应用于番茄红素的生产[6, 8-9]。随着番茄红素合成途径的阐明和相关酶基因的克隆,基因重组、基因敲除等技术手段被广泛应用于番茄红素合成过程中关键酶的改造、调节和修饰,再结合番茄红素代谢过程的调控,以此构建的番茄红素基因工程菌,生产番茄红素的能力大大提升[10-12]。利用微生物发酵法生产番茄红素安全无毒,并能解决番茄红素产量和产率低、提取成本高等一系列问题,具有巨大的实用价值和开发前景,是未来的研究热点[13]。
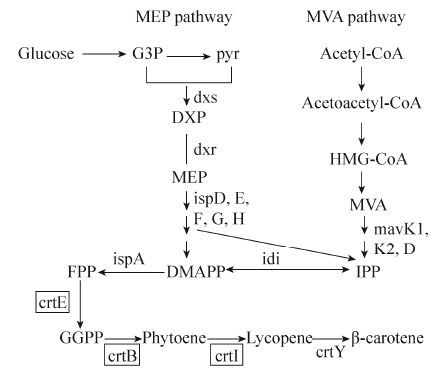
1 番茄红素的生物合成途径番茄红素属于异戊二烯类化合物,在生物体中是一个多酶参与的复杂代谢产物,它有两种合成途径,分别是2-甲基-D-赤藓糖醇-4-磷酸盐 (MEP) 途径和甲羟戊酸 (MVA) 途径[14] (图 1)。MEP途径存在于原核生物中,由丙酮酸和甘油醛-3-磷酸经催化生成1-脱氧-D-木酮糖-5-磷酸 (DXP),后转化为MEP,再经过一系列酶的催化,生成番茄红素的合成前体异戊二烯焦磷酸 (IPP) 和二甲基丙烯焦磷酸酯 (DMAPP)。MVA途径存在于真核生物和植物的内质网中,起始于乙酰辅酶A,生成乙酰乙酰辅酶A后再经羟甲基戊二酸单酰辅酶A (HMG-COA) 生成MVA,之后再经几步激酶催化反应生成IPP。在这两种途径中,直接前体IPP和DMAPP在法尼基焦磷酸合成酶的催化下生成法尼基焦磷酸 (FPP),之后通过牻牛儿基二磷酸合成酶 (GGPS)、八氢番茄红素合酶 (PSY) 和八氢番茄红素脱氢酶 (PDS) 这3个关键酶合成番茄红素,这3个酶分别由基因crtE、crtB和crtI编码。之后番茄红素可以继续进行环化、加氧和脱氢等修饰反应形成多种结构独特的类胡萝卜素及其衍生物。
 |
| 图 1 生物体中番茄红素的合成途径 Figure 1 Biosynthesis pathway of lycopene. |
| 图选项 |
2 多酶调控技术与方法2.1 多酶合成体系常规调控方法多酶体系指催化机体内的一些连续反应的酶互相联系在一起,形成的反应链体系。类似于番茄红素这种萜类大分子,合成路线长,涉及代谢途径众多,因此许多原始番茄红素产生菌并不是理想的生产宿主,目前研究主要是以传统基因工程手段对番茄红素合成途径中的关键酶进行鉴定和过表达,并优化MEP或者MVA途径,增加前体途径的供给和限速酶的表达量,这主要通过体外构建多基因共表达质粒导入到表达宿主中进行关键基因的异源表达来实现。现在有很多成熟的表达系统用于基因的过表达,例如大肠杆菌和酵母[15]。但是在这些微生物体内引入多个基因甚至整条或者多条代谢途径的过程中,难以保持各步代谢流量的平衡,中间有毒代谢物质的大量积累会严重影响宿主细胞的生长,导致代谢流量失衡,从而影响产物产量。因此在共表达质粒基础上对多基因表达进行多层次的控制是必要的,主要策略包括基因顺序调控及上游调控元件优化。基因顺序的调控方式运用在多基因表达产物中很常见,通过改变关键基因的顺序影响基因表达量,能够提高表达蛋白的催化效率,增加产物单位产量。上游调控元件的优化包括启动子和核糖体结合位点 (RBS) 调控。启动子在代谢工程中所起的作用非常重要,不同系统中采用的启动子完全不同,启动子强弱影响基因的转录水平,从而影响到蛋白的表达。对于共表达质粒,一般策略是使用强启动子 (如T7、T5和Trc等) 替换原载体上的弱启动子,增强基因的转录,同时将基因的表达置于严密控制的条件下,从而提高目标产物的产量。从翻译水平方面,目的基因上游的核糖体结合位点可以影响翻译的起始水平和翻译速率,改变或调整RBS序列通常也是对多基因表达调控比较有效的方法。
除了通过过表达代谢途径中关键基因外,增加最终产物的另一种方式是阻断竞争性分支途径,类异戊二烯化合物番茄红素的合成会受到宿主内源代谢途径的干扰,除了合成途径下游的FPP、牻牛儿基牻牛儿基二磷酸 (GGPP) 存在分支代谢外,还有相应的其他代谢途径如胞质乙酰CoA的合成途径,会与番茄红素竞争合成前体。阻断分支途径的方法一般是基因的敲除或替换。在敲除或替换基因的方法中,λ-Red重组技术是近年来应用较广的DNA同源重组技术,该技术可以简单、快速地对任意大小的DNA分子进行插入、敲除和突变等多种修饰,碱基突变率很小。此外基因编辑方法——CRISPR/Cas9技术也应用到了类异戊二烯的代谢途径改造中,CRISPR/Cas9技术是一种由向导RNA引导Cas9蛋白对靶序列DNA进行切割的技术手段,该系统以接近100%的编辑效率对基因进行插入、敲除和替换,可引入多种类型的基因修饰,方便快捷。与传统基因敲除技术相比,CRISPR/Cas9技术在具有完整的错配修复系统的细胞中有较高编辑效率,有利于细胞基因组稳定遗传。
2.2 多酶合成体系调控技术的新策略上述内容已经介绍了在转录水平、翻译水平、阻断竞争性分支代谢途径等层面对多酶合成进行调控的方法,随着生物技术的进步,一些新的调控策略应运而生。
2.2.1 多片段组装技术以番茄红素为代表的复杂天然产物都是多酶参与的顺序合成产物,前一个酶催化反应的产物 (中间产物) 是下一歩酶催化反应的底物,但在细胞工厂中,这些酶分子之间的距离很难控制,通常情况下处于游离状态,从而制约了其催化效率。通过Linker将两个或多个蛋白连接起来的融合蛋白技术[16]可以很好地解决这类问题。在构建基因工程菌的过程中,多个片段的连接是非常重要的,Linker在其中起到了关键作用,这关系到基因片段的高效连接,以及融合蛋白中的不同蛋白是否能够分别形成正确的空间结构、发挥各自的生物学活性。
目前新兴起来的Gibson Assembly?组装方法是另一种简单的等温一步拼接法,由Gibson[17]开发,对于多片段组装,只需要对待克隆的DNA片段 (包括载体) 进行PCR扩增,片段两端添加15 bp到30 bp的同源臂。之后在Gibson Assembly的孵育体系中,依靠同源臂之间的重叠序列退火,即可按正确的顺序进行拼接,形成完整的双链DNA分子,实现无痕拼接。该方法能直接使用PCR片段,不需要回收步骤,并且可以同时快速处理多个片段,因此在多片段组装中广泛使用。
随着多片段组装技术的进步,娄春波等[18]开发出了一种新型的DNA片段组装技术Oligo-linker mediated assembly (OLMA) 方法,此方法引入多个寡核苷酸双链片段作为linker,依靠这样的短片段,可以将DNA片段按照预先设计好的顺序进行组装,该技术在模块化组装多个片段以及额外添加调控元件如RBS及启动子片段方面颇具成效。
2.2.2 支架介导的多酶组装技术近年来随着生物技术与材料科学的快速发展,新一批的多酶组装方法随之产生,这就是支架介导的多酶组装方法。这些方法是通过人工合成的支架固定催化反应中的酶,在缩短酶与酶的空间距离,以及实现底物通道效应的基础上,还可通过调整支架对酶结合数量进行精确控制,从而平衡整体代谢流量[19]。人工合成支架的构建形式包括蛋白支架、RNA支架及DNA支架等。
蛋白支架通常是相互作用的蛋白对,或是蛋白质多聚物,生物体中存在着许多具备自组装性质的蛋白,因此可以按照实验设计所需合理地将这些蛋白组装起来,构建蛋白支架。在人类基因组中,RNA也占据了很大的份额,高达90%的基因组会被转录成RNA,在生物活动中起着重要作用。近年来,RNA也被应用于胞内组装多酶,合成RNA支架与RNA配基Aptamer结合使用,其中RNA配基Aptamer是一种能与RNA结合的适配体蛋白,通过适配体蛋白与目标蛋白或目标酶结合,将多酶体系中的不同酶定位至RNA上,实现酶与酶之间空间距离的缩减。DNA支架的构建形式不同于其他两种支架,是将目的蛋白与锌指蛋白融合表达,以锌指蛋白的锌指结构为结合域特异性识别DNA支架上碱基序列并与之结合,实现目的蛋白按照底物-产物的催化顺序在支架上的“自组装” (图 2)。相较于蛋白支架和RNA支架,DNA支架不会存在错误折叠、容易聚集及降解等问题。DNA支架可以通过碱基互补配对实现精准控制,因此可被广泛应用于固定化、自组装目的蛋白等方面。
 |
| 图 2 DNA支架的构建模式 Figure 2 Construction mode of DNA scaffold. |
| 图选项 |
3 多酶调控技术在番茄红素合成中的应用3.1 常规方法构建番茄红素工程菌在利用共表达质粒异源表达番茄红素关键基因的方法中,番茄红素合成基因crtE、crtB和crtI大多来源于噬夏孢欧文氏菌Erwinia uredovora、草生欧文氏杆菌Erwinia herbicola和泛菌属等微生物,将这3个基因构建共表达质粒后导入到酵母或者大肠杆菌中表达生产番茄红素。如Yoon等[20]分别从成团泛菌Pantoea agglomerans和凤梨泛菌Pantoea ananatis两株菌中扩增出番茄红素合成的关键基因crtE、crtB和crtI,整合到质粒pTrc99A上并导入到大肠杆菌中。在2YT培养基中,无IPTG诱导剂情况下,携带来源于Pantoea agglomerans crt基因的菌株番茄红素的产量为27 mg/L。加入0.1 mmol/L的IPTG诱导剂时,携带来源于Pantoea agglomerans crt基因的菌株,番茄红素的产量是12 mg/L,是携带Pantoea ananatis crt基因的2倍多。Jin等[21]将来源于耐辐射奇异球菌Deinococcus wulumiqiensis R12的crtE、crtB、crtI基因克隆至表达载体pET22b上,获得重组质粒pET-EBI,导入大肠杆菌后番茄红素的产量为49 mg/g DCW,发酵液中产量为417 mg/L。刘天罡等[22]从Pantoea ananatis中扩增出crtE、crtB和crtI基因,构建了6个质粒和3个菌株,其中菌株L3获得的番茄红素最高产量为1 440 mg/L。Miura等[23]在产朊假丝酵母Candida utilis中导入了外源Erwinia uredovora的crtE、crtB、crt I基因,获得重组菌株的番茄红素产量达到758 μg/g DCW。Matth?us等[24]将Pantoea ananatis的crtB、crtI和解脂耶氏酵母的TEF1启动子、终止子控制元件插入质粒pUCBM21,转入到解脂耶氏酵母Yarrowia lipolytica中产生了16 mg/g DCW的番茄红素。Araya-Garay等[25]将来源于Erwinia uredovora的crtE、crtB、crtI基因插入到质粒pGAPZB中,构建了表达番茄红素合成过程中3个关键酶质粒pGAPZB-EBI,将此质粒导入毕赤酵母Pichia pastoris X33后番茄红素的产量为1 141 μg/g DCW。Bhataya等[26]也以毕赤酵母Pichia pastoris X33作为宿主,从不同细菌中扩增出番茄红素的关键基因构建到pGAPZB质粒上,所构建的酵母工程菌在甲醇等有机物条件下高密度培养,菌体番茄红素产量达4.6 mg/g DCW,发酵液中番茄红素产量达73.9 mg/L。Sun等[27]从三孢布拉霉Blakeslea trispora中克隆出β-类胡萝卜素合成途径中的关键基因ipi和carG基因,两者分别表达异戊烯焦磷酸异构酶和牻牛儿基二磷酸合成酶,在大肠杆菌中异源表达后,β-类胡萝卜素的产量由0.5 mg/g DCW上升到0.95 mg/g DCW。以上例子都说明,通过基因工程技术构建多顺反子表达番茄红素合成基因,使得原本不产生色素的大肠杆菌、酵母等菌株产生色素,从而生产发酵得到番茄红素,利用不同来源的番茄红素合成基因构建的基因工程菌及其产量总结见表 1。
多酶合成体系中多基因表达的协同作用非常必要,因此基因顺序的调控在番茄红素合成体系中也至关重要。Jin等[31]构建了24个含有番茄红素合成关键基因顺序不同的重组质粒,讨论基因顺序对番茄红素合成的影响,结果发现基因顺序的不同对番茄红素的合成具有很大的影响,筛选获得的最高产菌株IEB11,其番茄红素的产量是菌株EBI11的2倍多。Zhang等[18]构建了11个番茄红素合成基因顺序不同的菌株,其中菌株BIE的番茄红素产量是菌株EIB的3倍,证明了基因顺序对番茄红素的合成有较大影响。Xie等[34]在酿酒酵母中异源表达合成β-胡萝卜素,通过对上下游及FPP竞争途径的顺序调控,以预定的顺序排列萜类化合物生物合成过程中的关键代谢节点的基因,由此发酵液中类胡萝卜素的产量为1 156 mg/L (20.79 mg/g DCW)。Ye等[35]通过改变基因的顺序构造了10个高效的多顺反子的质粒,通过蛋白电泳分析得出其中酶活最高的是质粒pET-P-B。
表 1 重组大肠杆菌和酵母生产番茄红素能力的比较Table 1 Comparison of lycopene production capacity between recombinant Escherichia coli and yeast
| Gene sources | Recombinant strains | Lycopene production | Fermentation type | References | |
| (mg/L) | (mg/g) | ||||
| Pantoea agglomerans | E. coli | 60 | - | Shake-flask fermentation | [20] |
| Pantoea ananatis | E. coli | 35 | - | Shake-flask fermentation | [20] |
| Pantoea agglomerans | E. coli | 1 350 | 32.0 | Fed-batch fermentation | [28] |
| Erwinia herbicola | E. coli | - | 7.6 | Shake-flask fermentation | [10] |
| Erwinia herbicola | E. coli | 102 | 22.0 | Shake-flask fermentation | [12] |
| Pantoea agglomerans | E. coli | 1 050 | - | Shake-flask fermentation | [29] |
| Erwinia herbicola | E. coli | 78 | 33.4 | Shake-flask fermentation | [30] |
| Deinococcus wulumiqiensis R12 | E. coli | 1 288 | 157.1 | Fed-batch fermentation | [31] |
| Pantoea agglomerans | E. coli | 3 520 | 50.6 | Fed-batch fermentation | [32] |
| Pantoea ananatis | E. coli | 1 440 | 32.1 | Fed-batch fermentation | [22] |
| Erwinia uredovora | Candida utilis | - | 0.8 | Shake-flask fermentation | [23] |
| Erwinia uredovora | Pichia pastoris | - | 1.1 | Shake-flask fermentation | [25] |
| Pantoea ananatis | Yarrowia lipolytica | - | 16.0 | Fed-batch fermentation | [24] |
| Erwinia uredovora | Saccharomyces cerevisiae | - | 3.3 | Shake-flask fermentation | [33] |
| Different bacterial species | Pichia pastoris X33 | 74 | 4.6 | Fed-batch fermentation | [26] |
表选项
启动子的置换是调控多基因表达体系的常见手段,启动子的强度对目的基因在工程菌中的表达发挥了重大作用,由此影响整个代谢流。翁志明等[11]将dxs基因的天然启动子置换为T5启动子,导致重组大肠杆菌中番茄红素的产量提高了103%。Bahieldin等[33]在酿酒酵母中外源表达来自欧文氏菌的crtE、crtB、crtI三个关键基因,并在此之前加入了葡萄糖阻遏型启动子ADH2,重组菌株番茄红素产量达到了3.3 mg/g DCW,远远高于异源表达同样3个基因的其余菌株。Suh等[36]用强启动子T5调控MEP途径中的关键基因dxs、idi和ispDF后,类胡萝卜素产量比对照提高了4.5倍。Zhao等[14]采用6个不同强度的人工启动子调控元件对大肠杆菌MEP途径中的7个基因进行调控,最终使β-胡萝卜素产量提高3.5倍,并提出替换强启动子对于获得目的产物的最大代谢流量来说并不一定是最优的。这些例子说明,不同启动子对于产物的合成是具有显著影响的,但并不表示使用强启动子就能获得最高的酶活和最大的产物产量,因此在后期研究中,往往需要考察不同启动子及宿主的适配性,从而选择出最优的启动子。
对RBS序列进行优化能够提高蛋白的表达量,将此方法应用到番茄红素多酶合成调控中,能够有效地提高番茄红素的产量,如Kang等[37]从Erwinia herbicola基因组中扩增出crtE、crtB、crtI和ipi基因,由此构建质粒pAC-LYCO4,在质粒pAC-LYCO4基础上使用强RBS序列调控,经调控优化后番茄红素的产量是未调控的3倍。Wang等[38]采用“多重自动基因组改造技术” (Multiplex automated genome engineering, MAGE),定向改造大肠杆菌中番茄红素合成过程中的20个基因的RBS序列,最终筛选得到高产菌株,番茄红素产量提高了5倍。Jin等[21]在基因crtB和crtI之前插入了两种不同的RBS调控序列,导入大肠杆菌经优化后番茄红素的最高产量为88 mg/g DCW,比未插入RBS调控序列重组菌的番茄红素产量提高了2倍。Ye等[35]在一个高效的多顺反子质粒上构建多级酶联反应,通过改变RBS序列及其与起始密码子之间的序列aligned spacing (AS),在最佳RBS及AS序列条件下,质粒pET-B-SD2-AS1-P所表达酶的酶活比原始质粒高4 U/mg。戴冠苹等[39]通过RBS文库对MEP合成途径中关键基因dxs、idi和crt进行调控,使β-胡萝卜素产量相对于出发菌株提高了35%,并且发现通过RBS调控,可调控范围较广,这种调控方法从某种程度上比使用多个固定强度调控元件更有利于类胡萝卜素的生产,同时也为基因表达的精确调控提供一种新的思路。
在萜类物质的合成过程中,IPP转化为DMAPP是限速步骤,通过对此环节关键基因的过表达,可以显著提高番茄红素的产量,如Zhu[22, 40]在质粒pFZ110的基础上插入idi片段,由此获得菌株L3的番茄红素产量是原菌株L2的2倍。Zhao等[14]过表达MEP途径中限速步骤中的idi、dxs和ispDF基因,使得番茄红素产量从0.89 mg/g DCW提高到5.39 mg/g DCW。
在利用λ-Red重组技术阻断番茄红素合成途径中的中间产物流向分支途径的方法中,翁志明等[11]利用λ-Red重组技术敲除谷氨酸脱氢酶编码基因gdhA、丙酮酸脱氢酶基因aceE、甲醛脱氢酶编码基因fdhF,阻断了丙酮酸分解支路,增加了MEP途径中起始物丙酮酸的积累,并提高了NADPH供给,促进了产物番茄红素的合成,产物产量提高了103%。Lu等[41]利用λ-Red重组技术将lacZ基因的启动子用文库中的启动子和信使RNA替换掉,通过测定β-半乳糖苷酶的产量确定了最强启动子,之后将此启动子应用到番茄红素的合成之中。Sun等[32]敲除了β-胡萝卜素生产菌中的玉米黄素糖基转移酶 (crtX) 和番茄红素环化酶基因 (crt Y),阻断了β-胡萝卜素的合成,敲除后番茄红素产量达到50.6 mg/g DCW。Zhou等[10]将中心碳代谢途径中葡萄糖-6-磷酸脱氢酶基因 (zwf) 进行敲除后,番茄红素的产量提高了130%。
最新的基因编辑方法CRISPR/Cas9技术也已被应用于番茄红素及其相关产物合成途径的优化中,如Li等[42]将来源于Pantoea agglomerans菌株的番茄红素和β-类胡萝卜素合成关键基因crtE、crtB、crtI、crtY整合到大肠杆菌染色体上,通过CRISPR/Cas9技术对MEP途径、产物合成途径以及中心碳代谢途径中的33处进行优化来改变代谢通路,获得了100多个突变体,最终获得了能产生2 g/L β-类胡萝卜素的高产菌株。EauClaire等[43]应用CRISPR/Cas9技术从斯氏泛菌Pantoea stewartii DC413菌株中扩增出tHMG1、crtE、crtB、crtI和crtY基因,导入到酿酒酵母Saccharomyces cerevisiae中,作为过表达番茄红素合成基因的另一途径。Ronda等[44]通过CRISPR/Cas9技术将β-胡萝卜素合成的相关3条途径的基因精确而高效地整合到酿酒酵母基因组上的相应位点,超过84%的都是正确克隆。Tadas等[45]应用此技术以一步转化的方法对基因组上5个不同的位点同时进行编辑,成功构建了MVA高产菌株,相较于野生型酿酒酵母,高产菌株的产量提升了41倍。这些相关研究的最新进展表明这项高效的多重基因组编辑技术的应用渐趋成熟,在功能基因组学和代谢工程中具有极大的应用潜力。
3.2 多酶调控新技术在番茄红素合成体系中的应用多片段组装技术在番茄红素多酶体系中常用于酶与酶的连接及调控元件的引入,Linker序列的设计和选择十分重要。Arai等[46]在两个不同功能的蛋白结构域之间插入了不同类型和不同长度的linker序列,研究结果表明随着linker长度的增加,会形成更多的螺旋,这些螺旋linker会控制两个结构域之间的距离并且能够减小两者之间的干扰。Heider等[47]在构建重组质粒时,使用Gibson assembly方法将MEP途径中的多个基因,2个操纵子片段和RBS调控片段同时组装到质粒上,方便快捷。Zhang等[18]运用独特的OLMA方法 (Oligo-linker mediated assembly method),将番茄红素合成途径中的4个基因和不同强度的RBS调控片段,以及不同的基因顺序进行DNA模块化组装,无需测序即可一步构建至载体上。
而一些由支架介导的多酶调控技术也应用到番茄红素合成途径中,主要是增加MVA途径中MVA的产量:Dueber等[48]设计了一个蛋白支架,利用支架与目标蛋白相结合的能力,将MVA途径中的3个关键酶硫解酶 (AtoB)、HMG-CoA合成酶 (HMGS) 和HMG-CoA还原酶 (HMGR) 按一定的比例组装固定在蛋白支架上,形成了可控的代谢途径,成功将番茄红素合成前体甲羟戊酸的产量提高了77倍,并减少了宿主的代谢负荷。Conrado等[49]利用DNA支架,在大肠杆菌中进行了甲羟戊酸的途径优化。从酶的配比、酶结合序列之间的碱基数等方面进行研究,将MVA途径中的3种关键酶AtoB、HMGS和HMGR进行顺序调控,并通过改变支架碱基序列从而调整代谢途径中酶的化学计量数比 (图 3),最终MVA的产量达到1.7 g/L,比无支架体系提高了3-5倍,解决了酶和代谢物的简单扩散和随机碰撞导致的低效率问题。目前,利用支架应用在番茄红素、β-胡萝卜素等类胡萝卜素产物合成体系中的研究还鲜见报道。
 |
| 图 3 DNA支架中锌指蛋白的作用机理 Figure 3 Mechanism of zinc finger protein in DNA scaffold. |
| 图选项 |
除此之外,这些支架体系也应用于一些其他的典型多酶合成体系中:Mingardon等[50]将来源于解纤维素梭菌Clostridium cellulolyticum中的各种纤维素酶 (CelA/E/F/G/M) 和来源于新美鞭菌Neocallimastix patriciarum的纤维素酶 (CelA/D) 组装在蛋白支架上,体外高效降解纤维素的活力是未组装的游离酶系统的2.6倍,实验数据表明蛋白支架能够有效提高酶的催化效率。Rahman等[51]在正烷烃的生产中引入了DNA支架,在考察了脂酰-ACP还原酶 (AARs) 和醛脱甲酰基氧化酶 (ADOs) 两个酶的配比后,在两种酶数量以3:1比例结合到DNA支架上时,产生正烷烃44 mg/L,与对照相比,提高了8.8倍。Funabashi等[52]将丙酮酸磷酸双激酶 (PPDK) 和荧光素酶 (Fluc) 分别连接到锌指蛋白上,考察了DNA支架上锌指蛋白结合序列分别间隔0、10、20和30 bp的碱基数目对ATP循环的影响,结果证实当间隔为10 bp时效果最好。在此情况下,两个酶在支架上的空间距离较近,由于邻近效应提高了ATP从PPKD到Fluc的扩散效率。Lee等[53]在大肠杆菌中将L-苏氨酸合成途径中的关键酶采用DNA支架进行固定,考察了锌指蛋白结合区之间的距离和酶的化学计量配比对催化效率的影响,在最终优化过的支架体系中,酶反应的速率显著提高,L-苏氨酸的生产时间减少了50%,并且支架的引入减少了有毒中间代谢物的积累,提高了宿主细胞生长的速率。Liu等[54]首次将DNA支架运用到枯草芽孢杆菌生产乙酰氨基葡糖体系中,产物的产量由原先的1.83 g/L提升至4.55 g/L。哈佛大学的Delebecque等[55]选用PP7、MS2两种锚定蛋白分别与氢化酶、铁氧还蛋白融合,以RNA支架为载体催化质子还原成氢分子,结果氢分子的输出量相比改造前分别提高了11倍和48倍,酶的催化效率显著提高。
以上研究表明,支架介导的调控技术在一些多酶合成体系中的应用得到了较好的效果,虽然在番茄红素等类胡萝卜素的合成中还鲜有应用,但这些研究对类似于番茄红素多酶合成途径的调控具有一定的借鉴意义,为类似的多酶合成体系中多基因表达协同作用机制奠定了研究基础。基于支架构建的优点,蛋白、DNA等支架的构建模式可作为一个平台技术应用到多酶催化大分子化合物的模块化合成的精确调控之中,为解决多酶催化合成复杂大分子化合物中的共性问题提供参考及示范,为其他生物基化工产品代谢途径的构建及优化提供强而有力的基础。
4 总结目前在针对番茄红素等多酶催化复杂生物大分子合成途径的常规改造方法中,一方面是通过替换合成途径中关键酶调控元件,增强酶的活力及产物合成途径;另一方面是通过基因敲除技术,降低支路合成效率,削弱代谢支流,确保番茄红素合成途径中有足够的前体和能量供应。但这些传统的基因工程及代谢调控技术往往需要引入多基因表达甚至整套的代谢途径,从而忽略了“底物通道”在多酶合成体系中不可或缺的作用,导致细胞中游离酶之间距离较远,制约了酶的催化效率。在研究多酶调控的新策略后,多酶自组装、人工支架的构建等新兴方法为提高这类生物大分子的产量以及多酶合成的效率提供了新的研究技术手段,极大地提升了整个多酶合成体系的能动性和精确性。将多酶自组装、人工支架等方法引入到以番茄红素为代表的烯萜类大分子的合成之中,可建立起多酶合成调控的新方法,以实现催化体系中目的蛋白顺次线性化的自组装,提高多酶的催化效率。同时为阐明番茄红素多酶合成体系中多基因表达协同作用机制奠定理论研究基础,也对其他多酶催化复杂大分子化合物的高效快速合成具有一定的指导借鉴作用。
参考文献
| [1] | Shi J, Mazza G, Le Maguer M. Lycopene from tomatoes//Functional Foods: Biochemical and Processing Aspects, Volume 2. Boca Raton: CRC Press, 2002: 135-167. |
| [2] | Nguyen ML, Schwartz SJ. Lycopene: chemical and biological properties.Food Technol, 1999, 53(2): 38–45. |
| [3] | Kong KW, Khoo HE, Prasad KN, et al. Revealing the power of the natural red pigment lycopene.Molecules, 2010, 15(2): 959–987.DOI: 10.3390/molecules15020959 |
| [4] | Aggarwal BB, Shishodia S. Molecular targets of dietary agents for prevention and therapy of cancer.Biochem Pharmacol, 2006, 71(10): 1397–1421.DOI: 10.1016/j.bcp.2006.02.009 |
| [5] | Moise AR, Al-Babili S, Wurtzel ET. Mechanistic aspects of carotenoid biosynthesis.Chem Rev, 2014, 114(1): 164–193.DOI: 10.1021/cr400106y |
| [6] | Liu XJ, Liu RS, Li HM, et al. Lycopene production from synthetic medium by Blakeslea trispora NRRL 2895 (+) and 2896 (-) in a stirred-tank fermenter.Bioprocess Biosyst Eng, 2012, 35(5): 739–749.DOI: 10.1007/s00449-011-0654-4 |
| [7] | Liao YQ, Xie YQ, Huang B. Production methods of lycopene.Subtrop Agric Res, 2007, 3(1): 64–68.(in Chinese). 廖益强, 谢拥群, 黄彪. 番茄红素的生产方法.亚热带农业研究, 2007, 3(1): 64-68. |
| [8] | Alper H, Miyaoku K, Stephanopoulos G. Construction of lycopene-overproducing E. coli strains by combining systematic and combinatorial gene knockout targets.Nat Biotechnol, 2005, 23(5): 612–616.DOI: 10.1038/nbt1083 |
| [9] | Hohmann HP, Pasamontes L, Tessier M, et al. Fermentative carotenoid production: US, 6124113. 2000-09-26. |
| [10] | Zhou Y, Nambou K, Wei LJ, et al. Lycopene production in recombinant strains of Escherichia coli is improved by knockout of the central carbon metabolism gene coding for glucose-6-phosphate dehydrogenase.Biotechnol Lett, 2013, 35(12): 2137–2145.DOI: 10.1007/s10529-013-1317-0 |
| [11] | Weng ZM, Wang Y, Liu JZ. Overproduction of lycopene by metabolic engineering Escherichia coli.Bioprocess, 2012, 2(2): 51–57.(in Chinese). 翁志明, 王玥, 刘建忠. 代谢工程大肠杆菌过量生产番茄红素.生物过程, 2012, 2(2): 51-57. |
| [12] | Yoon SH, Lee YM, Kim JE, et al. Enhanced lycopene production in Escherichia coli engineered to synthesize isopentenyl diphosphate and dimethylallyl diphosphate from mevalonate.Biotechnol Bioeng, 2006, 94(6): 1025–1032.DOI: 10.1002/(ISSN)1097-0290 |
| [13] | Wu JL, Wu QP, Zhang JM, et al. Advances on microbial biosynthesis and fermentation production of lycopene.Food Sci, 2013, 34(19): 336–340.(in Chinese). 吴军林, 吴清平, 张菊梅, 等. 番茄红素的微生物合成及发酵生产研究进展.食品科学, 2013, 34(19): 336-340.DOI:10.7506/spkx1002-6630-201319069 |
| [14] | Zhao J, Li QY, Sun T, et al. Engineering central metabolic modules of Escherichia coli for improving beta-carotene production.Metab Eng, 2013, 17: 42–50.DOI: 10.1016/j.ymben.2013.02.002 |
| [15] | Jiang TY, Li LX, Ma CQ, et al. Strategies for regulating multiple genes in microbial cell factories.Chin J Biotech, 2010, 26(10): 1419–1425.(in Chinese). 姜天翼, 李理想, 马翠卿, 等. 微生物细胞工厂中多基因表达的控制策略.生物工程学报, 2010, 26(10): 1419-1425. |
| [16] | Gustavsson M, Lehti? J, Denman S, et al. Stable linker peptides for a cellulose-binding domain-lipase fusion protein expressed in Pichia pastoris.Protein Eng, 2001, 14(9): 711–715.DOI: 10.1093/protein/14.9.711 |
| [17] | Gibson DG, Young L, Chuang RY, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases.Nat Methods, 2009, 6(5): 343–345.DOI: 10.1038/nmeth.1318 |
| [18] | Zhang SS, Zhao XJ, Tao Y, et al. A novel approach for metabolic pathway optimization: oligo-linker mediated assembly (OLMA) method.J Biol Eng, 2015, 9: 23.DOI: 10.1186/s13036-015-0021-0 |
| [19] | Zhang YHP. Substrate channeling and enzyme complexes for biotechnological applications.Biotechnol Adv, 2011, 29(6): 715–725.DOI: 10.1016/j.biotechadv.2011.05.020 |
| [20] | Yoon SH, Kim JE, Lee SH, et al. Engineering the lycopene synthetic pathway in E. coli by comparison of the carotenoid genes of Pantoea agglomerans and Pantoea ananatis.Appl Microbiol Biotechnol, 2007, 74(1): 131–139.DOI: 10.1007/s00253-006-0623-z |
| [21] | Jin WY, Xu X, Jiang L, et al. Putative carotenoid genes expressed under the regulation of Shine-Dalgarno regions in Escherichia coli for efficient lycopene production.Biotechnol Lett, 2015, 37(11): 2303–2310.DOI: 10.1007/s10529-015-1922-1 |
| [22] | Zhu FY, Lu L, Fu S, et al. Targeted engineering and scale up of lycopene overproduction in Escherichia coli.Proc Biochem, 2015, 50(3): 341–346.DOI: 10.1016/j.procbio.2014.12.008 |
| [23] | Miura Y, Kondo K, Shimada H, et al. Production of lycopene by the food yeast, Candida utilis that does not naturally synthesize carotenoid.Biotechnol Bioeng, 1998, 58(2/3): 306–308. |
| [24] | Matth?us F, Ketelhot M, Gatter M, et al. Production of lycopene in the non-carotenoid-producing yeast Yarrowia lipolytica.Appl Environ Microbiol, 2014, 80(5): 1660–1669.DOI: 10.1128/AEM.03167-13 |
| [25] | Araya-Garay JM, Feijoo-Siota L, Rosa-dos-Santos F, et al. Construction of new Pichia pastoris X-33 strains for production of lycopene and β-carotene.Appl Microbiol Biotechnol, 2012, 93(6): 2483–2492.DOI: 10.1007/s00253-011-3764-7 |
| [26] | Bhataya A, Schmidt-Dannert C, Lee PC. Metabolic engineering of Pichia pastoris X-33 for lycopene production.Proc Biochem, 2009, 44(10): 1095–1102.DOI: 10.1016/j.procbio.2009.05.012 |
| [27] | Sun J, Sun XX, Tang PW, et al. Molecular cloning and functional expression of two key carotene synthetic genes derived from Blakeslea trispora into E scherichia coli for increased β-carotene production.Biotechnol Lett, 2012, 34(11): 2077–2082.DOI: 10.1007/s10529-012-1004-6 |
| [28] | Kim YS, Lee JH, Kim NH, et al. Increase of lycopene production by supplementing auxiliary carbon sources in metabolically engineered Escherichia coli.Appl Microbiol Biotechnol, 2011, 90(2): 489–497.DOI: 10.1007/s00253-011-3091-z |
| [29] | Zhang TC, Nakajima M. Increase of the lycopene production in the recombinant strains of Escherichia coli by supplementing with fructose//Advances in Applied Biotechnology.Berlin Heidelberg: Springer, 2015: 29–35. |
| [30] | Chen YY, Shen HJ, Cui YY, et al. Chromosomal evolution of Escherichia coli for the efficient production of lycopene.BMC Biotechnol, 2013, 13: 6.DOI: 10.1186/1472-6750-13-6 |
| [31] | Xu X, Jin WY, Jiang L, et al. A high-throughput screening method for identifying lycopene-overproducing E. coli strain based on an antioxidant capacity assay.Biochem Eng J, 2016, 112: 277–284.DOI: 10.1016/j.bej.2016.04.032 |
| [32] | Sun T, Miao LT, Li QY, et al. Production of lycopene by metabolically-engineered Escherichia coli.Biotechnol Lett, 2014, 36(7): 1515–1522.DOI: 10.1007/s10529-014-1543-0 |
| [33] | Bahieldin A, Gadalla NO, Al-Garni SM, et al. Efficient production of lycopene in Saccharomyces cerevisiae by expression of synthetic crt genes from a plasmid harboring the ADH2 promoter.Plasmid, 2014, 72: 18–28.DOI: 10.1016/j.plasmid.2014.03.001 |
| [34] | Xie WP, Ye LD, Lv XM, et al. Sequential control of biosynthetic pathways for balanced utilization of metabolic intermediates in Saccharomyces cerevisiae.Metab Eng, 2015, 28: 8–18.DOI: 10.1016/j.ymben.2014.11.007 |
| [35] | Ye Q, Cao H, Yan M, et al. Construction and co-expression of a polycistronic plasmid encoding carbonyl reductase and glucose dehydrogenase for production of ethyl (S)-4-chloro-3-hydroxybutanoate.Bioresour Technol, 2010, 101(17): 6761–6767.DOI: 10.1016/j.biortech.2010.03.099 |
| [36] | Suh W, Cheng Q. High isoprenoid flux Escherichia coli as a host for carotenoids production. Microbial Metabolic Engineering: Methods and Protocols.New York: Springer, 2012: 49-62. |
| [37] | Kang MJ, Yoon SH, Lee YM, et al. Enhancement of lycopene production in Escherichia coli by optimization of the lycopene synthetic pathway.J Microbiol Biotechnol, 2005, 15(4): 880–886. |
| [38] | Wang HH, Isaacs FJ, Carr PA, et al. Programming cells by multiplex genome engineering and accelerated evolution.Nature, 2009, 460(7257): 894–898.DOI: 10.1038/nature08187 |
| [39] | Dai GP, Sun T, Miao LT, et al. Modulating expression of key genes within β-carotene synthetic pathway in recombinant Escherichia coli with RBS library to improve β-carotene production.Chin J Biotech, 2014, 30(8): 1193–1203.(in Chinese). 戴冠苹, 孙涛, 苗良田, 等. RBS文库调控重组大肠杆菌β-胡萝卜素合成途径关键基因提高β-胡萝卜素合成能力.生物工程学报, 2014, 30(8): 1193-1203. |
| [40] | Zhu FY, Zhong XF, Hu MZ, et al. In vitro reconstitution of mevalonate pathway and targeted engineering of farnesene overproduction in Escherichia coli.Biotechnol Bioeng, 2014, 111(7): 1396–1405.DOI: 10.1002/bit.25198 |
| [41] | Lu J, Tang JL, Liu Y, et al. Combinatorial modulation of galP and glk gene expression for improved alternative glucose utilization.Appl Microbiol Biotechnol, 2012, 93(6): 2455–2462.DOI: 10.1007/s00253-011-3752-y |
| [42] | Li YF, Lin ZQ, Huang C, et al. Metabolic engineering of Escherichia coli using CRISPR-Cas9 meditated genome editing.Metab Eng, 2015, 31: 13–21.DOI: 10.1016/j.ymben.2015.06.006 |
| [43] | EauClaire SF, Zhang JZ, Rivera CG, et al. Combinatorial metabolic pathway assembly in the yeast genome with RNA-guided Cas9.J Indust Microbiol Biotechnol, 2016, 43(7): 1001–1015.DOI: 10.1007/s10295-016-1776-0 |
| [44] | Ronda C, Maury J, Jako?iūnas T, et al. CrEdit: CRISPR mediated multi-loci gene integration in Saccharomyces cerevisiae.Microb Cell Fact, 2015, 14: 97.DOI: 10.1186/s12934-015-0288-3 |
| [45] | Jako?iūnas T, Bonde I, Herrg?rd M, et al. Multiplex metabolic pathway engineering using CRISPR/Cas9 in Saccharomyces cerevisiae.Metab Eng, 2015, 28: 213–222.DOI: 10.1016/j.ymben.2015.01.008 |
| [46] | Arai R, Ueda H, Kitayama A, et al. Design of the linkers which effectively separate domains of a bifunctional fusion protein.Protein Eng, 2001, 14(8): 529–532.DOI: 10.1093/protein/14.8.529 |
| [47] | Heider SA, Wolf N, Hofemeier A, et al. Optimization of the IPP precursor supply for the production of lycopene, decaprenoxanthin and astaxanthin by Corynebacterium glutamicum.Front Bioeng Biotechnol, 2014, 2: 28. |
| [48] | Dueber JE, Wu GC, Malmirchegini GR, et al. Synthetic protein scaffolds provide modular control over metabolic flux.Nat Biotechnol, 2009, 27(8): 753–759.DOI: 10.1038/nbt.1557 |
| [49] | Conrado RJ, Wu GC, Boock JT, et al. DNA-guided assembly of biosynthetic pathways promotes improved catalytic efficiency.Nucleic Acids Res, 2012, 40(4): 1879–1889.DOI: 10.1093/nar/gkr888 |
| [50] | Mingardon F, Chanal A, López-Contreras AM, et al. Incorporation of fungal cellulases in bacterial minicellulosomes yields viable, synergistically acting cellulolytic complexes.Appl Environ Microbiol, 2007, 73(12): 3822–3832.DOI: 10.1128/AEM.00398-07 |
| [51] | Rahman Z, Sung BH, Yi JY, et al. Enhanced production of n-alkanes in Escherichia coli by spatial organization of biosynthetic pathway enzymes.J Biotechnol, 2014, 192(Part A): 187–191. |
| [52] | Funabashi H, Yanagi S, Suzuki S, et al. Assembly of zinc finger motif-fused enzymes on a dsDNA scaffold for catalyzing consecutive reactions with a proximity effect.Biotechnol Lett, 2015, 37(1): 109–114.DOI: 10.1007/s10529-014-1644-9 |
| [53] | Lee JH, Jung SC, Bui LM, et al. Improved production of L-threonine in Escherichia coli by use of a DNA scaffold system.Appl Environ Microbiol, 2013, 79(3): 774–782.DOI: 10.1128/AEM.02578-12 |
| [54] | Liu YF, Zhu YQ, Ma WL, et al. Spatial modulation of key pathway enzymes by DNA-guided scaffold system and respiration chain engineering for improved N-acetylglucosamine production by Bacillus subtilis.Metab Eng, 2014, 24: 61–69.DOI: 10.1016/j.ymben.2014.04.004 |
| [55] | Delebecque CJ, Lindner AB, Silver PA, et al. Organization of intracellular reactions with rationally designed RNA assemblies.Science, 2011, 333(6041): 470–474.DOI: 10.1126/science.1206938 |
