马建荣1, 刘安娜2, 张文彬2, 毛雅慧3, 余永红1


1. 广东食品药品职业学院, 广东 广州 510520;
2. 华南农业大学生命科学学院, 广东省农业生物蛋白质功能与调控重点实验室, 广东 广州 510642;
3. 湖北工程学院, 特色果蔬质量安全控制湖北省重点实验室, 湖北 孝感 432000
收稿日期:2020-01-07;修回日期:2020-02-12;网络出版日期:2020-03-23
基金项目:广州市科技计划项目(202002030422);广东省医学科学技术研究基金(A2018409);广东省农业生物蛋白质功能与调控重点实验室开放课题基金(PFRAO201804);广东食品药品职业学院院级课题(2019ZR13,2019ZR17)
*通信作者:余永红, Tel:+86-20-28194616;E-mail:yuyh@gdyzy.edu.cn.
摘要:[目的] 硫辛酸是细胞内重要的辅因子,参与多种基础代谢过程。野油菜黄单胞菌(Xcc)是十字花科植物黑腐病的病原菌,在全球范围内引起植物病害,引起重大经济损失。为此研究Xcc中硫辛酸的合成途径,为防治黑腐病提供新思路。[方法] 利用大肠杆菌硫辛酸合成关键酶LipA和LipB序列,同源比对发现Xcc基因组中XC_0713(XccLipA)和XC_0712(XccLipB)具有较高的同源性。采用PCR方法分别扩增XccLipA和XccLipB基因,并连入表达载体pBAD24M后分别互补大肠杆菌突变株,并检测转化子生长表型。利用同源重组方法,获得替换突变株,分析其生长性状,并利用剪叶法检测替换突变株对寄主植物甘蓝的致病力。[结果] XcclipA和XcclipB能分别恢复大肠杆菌lipA和lipB突变株在基础培养上生长。XcclipA和XcclipB都是菌体生长的必需基因,不能直接被敲除。但导入pSRK-EclplA后,成功分别获得XcclipA和XcclipB敲除突变株。两种EclplA替换后的敲除突变株在基础培养上都不能生长,添加硫辛酸后能恢复生长表型。在丰富培养基上,XcclipB敲除突变株能正常生长,而XcclipA敲除突变株不能生长,添加硫辛酸后生长也能恢复。分别测定不同培养条件下生长曲线,也得到同样的结果。寄主植物侵染结果显示,与野生菌相比,XcclipA敲除突变株致病性几乎丧失,而XcclipB敲除突变株的致病性与野生菌无显著性差异。[结论] Xcc中lipA编码硫辛酸合成酶,lipB编码辛酰转移酶,两者都是必需基因。Xcc中LipB-LipA途径是唯一的硫辛酰化途径,而没有外源性的硫辛酸途径。lipA敲除后显著影响Xcc的致病性,可作为抗菌药物筛选的靶点。
关键词:野油菜黄单胞菌硫辛酸合成硫辛酸合成酶LipA致病性分析
LipB-LipA is the only lipoyl acylation pathway in Xanthomonas campestris
Jianrong Ma1, Anna Liu2, Wenbin Zhang2, Yahui Mao3, Yonghong Yu1


1. Guangdong Food and Drug Vocational College, Guangzhou 510520, Guangdong Province, China;
2. Guangdong Provincial Key Laboratory of Protein Function and Regulation in Agricultural Organisms, College of Life Sciences, South China Agricultural University, Guangzhou 510642, Guangdong Province, China;
3. Hubei Key Laboratory of Quality Control of Characteristic Fruits and Vegetables, Hubei Engineering University, Xiaogan 432000, Hubei Province, China
Received: 7 January 2020; Revised: 12 February 2020; Published online: 23 March 2020
*Corresponding author: Yonghong Yu, Tel:+86-20-28194616;E-mail:yuyh@gdyzy.edu.cn.
Foundation item: Supported by the Guangzhou Science and Technology Plan Project (202002030422), by the Medical Science and Technology of Guangdong Provence (A2018409), by the Open fund of Guangdong Provincial Key Laboratory of Protein Function and Regulation in Agricultural Organisms (PFRAO201804) and Science Foundation of Guangdong Food & Drug Vocational College (2019ZR13, 2019ZR17)
Abstract: [Objective] Cofactor lipoic acid participates in many basic cellular metabolic processes. Xanthomonas campestris pv. campestris (Xcc) is the pathogen of black rot of cruciferous plants, causing plant disease all over the world. Studies on the synthetic mechanism of lipoic acid in Xcc will facilitate the discovery of novel methods to control the disease. [Methods] XC_0713 (XccLipA) and XC_0712 (XccLipB) in Xcc were found based on the sequence alignments with E. coli LipA and LipB, the key enzymes for lipoic acid synthesis. For complementation analysis, XcclipA and XcclipB were amplified and ligated into expression vector pBAD24M, which were further transferred into E. coli mutants, respectively. The growth of transformants was analyzed. With the aid of E. coli lplA expression, XcclipA and XcclipB were deleted in the genome by homologous recombination, respectively. The growth and virulence of the two mutants were analyzed. [Results] XcclipA and XcclipB complemented the growth of E.coli fabA and fabB mutants on the minimal media, respectively. Both XcclipA and XcclipB are essential genes, which cannot be deleted directly. However, XcclipA and XcclipB could be deleted when pSRK-EclplA was introduced. The two mutants could not grow on the minimal medium, but grew well when lipoic acid supplemented. XcclipB mutant could grow on the nutritional medium. XcclipA mutant almost abolished the virulence to the host plant, but XcclipB mutant showed similar virulence with wild-type strain. [Conclusion] XcclipA codes lipoyl synthase, and XcclipB codes octanoyl transferrase, both are essential genes for Xcc growth. The LipB-LipA is the only lipoic acylation pathway in Xcc. XcclipA deletion mutant showed no virulence to host plant, indicating XccLipA is a promising target for anti-bacteria agent selection.
Keywords: Xanthomonas campestris pv. campestris (Xcc)lipoic acid synthesislipoyl synthase LipAvirulence analysis
硫辛酸(lipoic acid)是在6位和8位碳原子之间插入硫原子的八碳饱和脂肪酸,其化学名称为1, 2-二硫代戊环-3-戊酸[1]。硫辛酸属于维生素B类化合物,作为天然的抗氧化剂广泛分布于动植物体内,能直接清除活性氧,对人类糖尿病、肝损伤、动脉粥样硬化治疗以及抗衰老等方面都具有较好的应用前景[2]。硫辛酸在细胞内作为辅因子参与2-酮酸脱氢酶(OGDH)复合体的催化反应,包括丙酮酸脱氢酶(PDH)、α-酮戊二酸脱氢酶(KGDH)、分支酮酸脱氢酶(BCDH)、3-羟基丁酮脱氢酶(ADH)和甘氨酸裂解系统(GCV)等5种复合酶体,参与生物体的能量代谢和一碳单位形成,并为脂肪酸和氨基酸的合成提供前体物质[3-4]。2-酮酸脱氢酶复合体都含有三种亚基:E1,E2和E3,每种亚基都催化部分反应,而硫辛酸的羧基与E2亚基上的赖氨酸残基的ε-氨基共价结合,催化反应中硫辛酸起在多酶复合体的各个活性中心之间传递中间产物的作用[5]。
在模式生物大肠杆菌中,研究表明存在两种互补的硫辛酰化途径:①依赖于硫辛酰蛋白连接酶(LplA)的外源性途径。该途径分两步进行,首先LplA催化硫辛酸与ATP结合生成硫辛酰-AMP,然后催化硫辛酰基团从硫辛酰-AMP上转移到E2亚基中保守的赖氨酸残基的ε-氨基上,并伴随AMP的释放[6]。②内源性硫辛酰化途径。从细菌脂肪酸合成途径来源的辛酰ACP,在辛酰转移酶(LipB)的催化下,将辛酰基团转移到2-酮酸脱氢酶E2亚基中的硫辛酸结构域上,进一步在硫辛酸合成酶(LipA)的催化下插入硫原子形成硫辛酰化蛋白亚基[7-9]。但不同细菌的硫辛酸合成途径具有多样性。在革兰氏阳性细菌枯草芽孢杆菌中,脂肪酸途径来源的辛酰ACP首先在辛酰转移酶LipM的催化下,将辛酰基团转移至甘氨酸裂解系统H亚基中的赖氨酸残基上,而后再在酰胺转移酶LipL的催化下,将辛酰基团转移到E2亚基中的硫辛酸结构域上,最后在LipA催化下生成硫辛酰化蛋白亚基[10-11]。而在金黄色葡萄球菌中,辛酰基团在H亚基上被LipA催化生成硫辛酰化,而后LipL再将硫辛酰基团转移到E2亚基上[12-13]。
由于硫辛酸作为重要的辅因子和抗氧化剂,其合成途径可作为潜在的抗菌药物作用靶点,用于新型抗菌药物的筛选[14]。此外,细菌的硫辛酸合成途径与致病性还紧密相关[12]。野油菜黄单胞菌(Xanthomonas campestris pv. campestris,Xcc)是引起十字花科植物黑腐病的病原菌,侵染寄主包括甘蓝、白菜、花椰菜、萝卜、芥菜等多种重要蔬菜,全球范围内引起重大经济损失,迫切需要开发新的针对性防控措施[15-17]。但Xcc中硫辛酸合成途径还未见报道。因此,本研究通过生物信息学分析、异体遗传互补和体内基因敲除等方法,研究了Xcc中硫辛酸的合成途径以及在植物致病中的功能,为开发新型抗菌药物、防治黑腐病病害提供新的思路。
1 材料和方法 1.1 材料
1.1.1 菌株,质粒和培养基: 本研究使用的大肠杆菌(Escherichia coli)菌株有MG1655、DH-5α、S17-1、BL21 (DE3)、Tm131和Tm136,野油菜黄单胞菌Xcc8004。质粒载体有pK18mobsacB、pSRK-Gm[18]和pBAD24m[19],其他载体均为上述质粒的衍生质粒(具体构建见下文),具体的菌株和质粒见表 1。LB用作培养大肠杆菌的丰富培养基,培养条件37 ℃。NYG和XOG分别作为野油菜黄单胞菌及突变株的丰富培养基和基础培养基。抗生素的使用浓度如下:50 mg/L利福平(Rif)、100 mg/L氨苄青霉素(Amp)、30 mg/L卡那霉素(Km)、10 mg/L庆大霉素(Gm)。诱导剂L-阿拉伯糖(Ara)使用浓度为0.02%,异丙基-β-D-硫代吡喃半乳糖苷(IPTG)使用浓度为1 mmol/L。
表 1. 菌株及质粒 Table 1. Strains and plasmids
| Strains/Plasmids | Characteristics | Source |
| E. coli strains | ||
| MG1655 | Wild type | |
| DH-5α | φ80d lacZΔM15 endA1recA1hsdR17(rK-, mK+) | Lab collection |
| S17-1 | TcrSmr, RP4::2(Tc::Mu) (Km::Tn7), pro, recA, thi, hsdR-M+, λpir | Lab collection |
| TM131 | rpsLlipA150::Tn1000dKnlplA148::Tn10dTc fadE | [6] |
| TM136 | rpsLlipB150::Tn1000dKnlplA148::Tn10dTc fadE | [6] |
| Xcc Strains | ||
| 8004 | Rif R, wild type | Lab collection |
| L1 | RifR, KmR, pL3 integrated in Xcc8004 genome | This study |
| L2 | RifR, KmR, pL4 integrated in Xcc8004 genome | This study |
| L3 | Rif R, ΔlipA/pSRK-EclplA | This study |
| L4 | Rif R, ΔlipB/pSRK-EclplA | This study |
| Plasmids | ||
| pBAD24M | Ampr, expression vector | Lab collection |
| pK18mobsacB | Kmr, conjugation vector | Lab collection |
| pSRK-Gm | Gmr, expression vector | Lab collection |
| pSRK-EclplA | GmR, EclplA in pSRK-Gm | Lab collection |
| pL1 | AmpR, XcclipA in pBAD24M | This study |
| pL2 | AmpR, XcclipB in pBAD24M | This study |
| pL3 | KmR, XcclipA in-frame deletion fragment inserted to pK18mobsacB between EcoR I/Hind III sites | This study |
| pL4 | KmR, XcclipB in-frame deletion fragment inserted to pK18mobsacB between EcoR I/Hind III sites | This study |
表选项
1.1.2 试剂: 核酸限制性内切酶、T4 DNA连接酶、DNA聚合酶、Marker DL2000等试剂,T-载体克隆、质粒提取和DNA凝胶回收等试剂盒均购自大连TaKaRa公司;利福平、氨苄青霉素、卡那霉素、庆大霉素、IPTG、硫辛酸、阿拉伯糖、蔗糖等试剂购自Sigma公司;PCR扩增引物的合成以及核酸序列测定由上海Sangon公司完成。
1.2 互补质粒构建 以野油菜黄单胞菌Xcc8004的总DNA为模板,以表 2中XcclipANde I和XcclipAHind III引物PCR扩增XcclipA基因,以XcclipBNde I和XcclipBHind III引物扩增XcclipB基因。两种扩增产物纯化并经Nde I和Hind III酶切后,分别连入pBAD24M表达载体上,转化并筛选阳性菌落,经测序验证后获得互补质粒pL1和pL2。
表 2. 所用引物列表 Table 2. Sequences of the PCR primers
| Primer name | Sequence (5′→3′) |
| XcclipA P1 | AATTGAATTCGGAAGTGAAGTGGACGCTGT |
| XcclipA P2 | ACACCGATGCAGTTGGGCGACGGTACGGGCGCTCA |
| XcclipA P3 | TGAGCGCCCGTACCGTCGCCCAACTGCATCGGTGT |
| XcclipA P4 | AATTAAGCTTAGTTGCTTGTCGTCGGCT |
| XcclipA checkup | AGTTCGACTCCGCGCATCA |
| XcclipA checkdown | GCAGCCAGTCGTTCATCACC |
| XcclipA Nde I | TATATACCATATGACCCAGCCTATCGCCC |
| XcclipA Hind III | AATTAAGCTTCACACACCGATGCAGTTGG |
| XcclipB P1 | AATTGAATTCACACCGATATCGTCAGCCAGT |
| XcclipB P2 | GTGGGCTGCAACACGAGTCTAGACACCGGGGCCACGACAG |
| XcclipB P3 | CGTGGCCCCGGTGTCTAGACTCGTGTTGCAGCCCACC |
| XcclipB P4 | AATTAAGCTTGGATCGCCGAGATGCAGTC |
| XcclipB checkup | GCTGGCGTATCTGCAGGC |
| XcclipB checkdown | CCGGGGTCAGGATCTCGATC |
| XcclipB Nde I | TATATTACATATGGTGGACGCTGTAGCGGC |
| XcclipB Hind III | AATTAAGCTTGGTCATGACGGTACGGGC |
| Note: The underlined sequences are the introduced restriction sites. | |
表选项
1.3 构建基因替换突变株 以Xcc8004基因组为模板,分别以表 2中的引物P1、P2和P3、P4分别扩增XcclipA基因和XcclipB基因的上下游片段(P1P2片段和P3P4片段),电泳回收后通过融合PCR方法将上下游片段融合为P1P4片段,并利用EcoR I和Hind III酶切后克隆到自杀性载体pK18mobsacB的相同位点上,经测序验证后分别获得XcclipA基因的敲除质粒pL3和XcclipB基因的敲除质粒pL4。
将pL3和pL4转化大肠杆菌S17-1后,分别与Xcc8004在NYG平板上30 ℃共培养36 h后,稀释涂布于含有Rif和Km的NYG平板,继续培养后获得单菌落。进一步提取总DNA,分别用XcclipA P1和XcclipA P4引物以及XcclipB P1和XcclipB P4引物进行PCR检测,确认获得XcclipA敲除的一次重组菌株Xcc L1和XcclipB敲除的一次重组菌株Xcc L2,而后在添加Rif的NYG中培养后,涂布于含有10%蔗糖的NYGS平板,筛选Km敏感的菌落。
将质粒pSRK-EclplA导入大肠杆菌S17-1后,分别与一次重组菌株Xcc L1和Xcc L2接合,涂布于含有Rif、Km和Gm的NYG平板上,30 ℃培养后获得单菌落,进一步PCR验证后分别获得Xcc L1/pSRK-EclplA和Xcc L2/pSRK-EclplA菌株。再将其分别在含有Rif和Gm的NYG中培养后,涂布于含有10%蔗糖的NYGS平板,用checkup和checkdown引物PCR筛选后分别获得XcclipA被敲除的Xcc L3 (ΔXcclipA/pSRK-EclplA)以及XcclipB被敲除的Xcc L4 (ΔXcclipB/pSRK-EclplA)。
1.4 替换突变株生长表型分析 通过平板接种和生长曲线两种方法分析替换突变株的生长表型。将野生菌和突变株分别接种到NYG液体培养基中(突变株培养添加5 mmol/L硫辛酸),30 ℃摇床培养24 h后离心收集菌体,并用相同体积的XO盐溶液(不含有碳源)悬浮后,30 ℃摇床继续培养24 h,再次离心后重悬菌体浓度至OD600均约为1.0。而后等量菌液划线到不同平板上继续培养观察生长,同时按1%接种量分别接种到不同的液体培养基中,并测定生长曲线。
1.5 替换突变株致病性分析 Xcc菌株侵染寄主植物参考文献[20-21]进行。将不同菌株接种到NYG中(突变株培养添加5 mmol硫辛酸),30 ℃摇床培养24 h后,调整不同菌株浓度至OD600均约为1.0,剪叶法分别接种到4片叶龄的甘蓝叶片上,继续培养2周后,测定并统计叶片病斑长度。
2 结果和分析 2.1 生物信息学分析 为研究野油菜黄单胞菌(Xcc)中硫辛酸的合成机制,根据模式生物大肠杆菌中硫辛酸合成的关键酶硫辛酸合成酶(EcLipA)[9]和辛酰转移酶(EcLipB)[8],分别同源性比对Xcc基因组,发现同源性较高的XC_0713和XC_0712。其中XC_0713编码蛋白与EcLipA的氨基酸序列一致性为60%,注释为硫辛酸合成酶(lipoic acid synthetase),还具有保守的的铁-硫簇结合位点CXXXCXXC和对底物结合具有辅助功能的CX4CX5C簇[4] (图 1-A),因此将其命名为XccLipA。而XC_0712编码的氨基酸序列与EcLipB的一致性为56%,注释为硫辛酰转移酶(lipoyl transferase),因此将其命名为XccLipB (图 1-B)。根据生物信息学分析推测,XccLipA和XccLipB是Xcc中硫辛酸合成的关键途径。但同源性比对在Xcc基因组中没有发现大肠杆菌硫辛酰蛋白连接酶(EcLplA)的同源基因,推测Xcc不具有硫辛酸利用的外源性途径。为验证以上观点,本课题组对野油菜黄单胞菌中XcclipA和XcclipB作了以下研究。
 |
| 图 1 XccLipA和XccLipB序列比对分析 Figure 1 Sequences alignments of XccLipA and XccLipB. A: the Fe-S cluster binding site CXXXCXXC and substrate binding site CX4CX5C are underlined. B: the key site cysteine (C) asterisked. |
| 图选项 |
2.2 异体遗传互补 大肠杆菌可以利用LipB-LipA途径内源性合成硫辛酸,也可以通过LplA利用外源性硫辛酸。大肠杆菌Tm131菌株是lipA和lplA基因双敲除突变株,而Tm136菌株是lipB和lplA基因的双敲除菌株,因此两个菌株不能从头合成硫辛酸,也不能利用外源硫辛酸,只能在添加乙酸盐(acetate)和琥珀酸盐(succinate)的培养基上生长[6]。为研究XcclipA的功能,我们首先将其遗传互补大肠杆菌Tm131菌株。结果显示,在基础培养基M9中添加乙酸盐和琥珀酸盐时,大肠杆菌野生菌MG1655、Tm131菌株以及互补XcclipA的转化子都能正常生长,但不添加乙酸盐和琥珀酸盐时,Tm131由于不能合成硫辛酸,不能正常生长,而互补XcclipA的转化子能微弱生长,添加诱导剂Ara后恢复正常生长,初步显示XcclipA编码蛋白具有硫辛酸合成酶活性(图 2-A)。
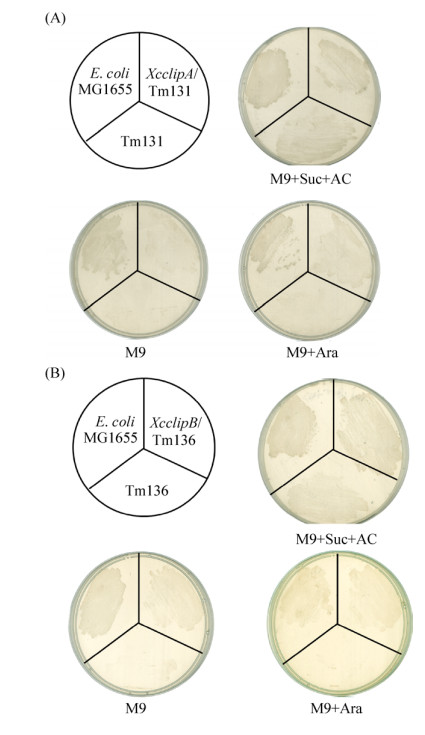 |
| 图 2 XcclipA和XcclipB基因分别互补大肠杆菌突变株Tm131 (A)和Tm136 (B) Figure 2 Complementation of E. coli mutants Tm131 and Tm136 with XcclipA and XcclipB, respectively. Suc: sucrose; Ac: acetate salt; Ara: arabinose; M9: minima medium. |
| 图选项 |
按照类似策略,将XcclipB遗传互补大肠杆菌Tm136菌株。结果也显示Tm136菌株不能在基础培养基M9上生长,而只能在添加有乙酸盐和琥珀酸盐的培养基上生长,但互补XcclipB的转化子则能恢复Tm136菌株在M9上生长,初步显示XcclipB编码蛋白具有辛酰转移酶活性(图 2-B)。
2.3 XcclipA和XcclipB是生长的必需基因 为进一步研究XcclipA和XcclipB在细胞内的生理功能,我们首先构建了含有XcclipA基因上下游片段的自杀性载体pL3,以及含有XcclipB上下游片段的自杀性载体pL4。分别转化大肠杆菌S17-1后,利用接合转移方法将pL3和pL4分别导入到野生菌Xcc8004中,在NYG (Rif和Km)平板上分别筛选获得一次重组菌株Xcc L1和Xcc L2。但将一次重组菌株在含有10%蔗糖的NYGS平板培养后,通过多次筛选都不能获得XcclipA或XcclipB基因的敲除突变株,而在培养基中添加硫辛酸也同样不能获得基因敲除突变株,推测XcclipA和XcclipB都是Xcc生长的必需基因,XccLipB-XccLipA参与的硫辛酸从头合成途径是Xcc获得硫辛酸的唯一途径,而不能吸收外源性硫辛酸。这一推测与生物信息学分析结果相吻合。
为验证以上推测,将pSRK-EclplA表达质粒分别导入一次重组菌株Xcc L1和Xcc L2,而后在含有Rif和Gm抗性并添加硫辛酸的NYGS平板上,筛选出对Km敏感的菌株,并利用验证引物Check up和Check down进一步PCR筛选,并对扩增片段进行测序验证。结果显示,染色体上的XcclipA和XcclipB分别被删除,获得基因替换突变株Xcc L3 (ΔXcclipA/pSRK-EclplA)和Xcc L4 (ΔXcclipB/pSRK-EclplA)。这一结果证明Xcc中硫辛酸从头合成途径是获得硫辛酸的唯一途径,只有导入外源途径的硫辛酰蛋白连接酶基因lplA后,才能敲除XcclipA或XcclipB基因。
2.4 基因替换突变株生长性状分析 首先分析了两个替换突变株Xcc L3和Xcc L4在营养丰富培养基NYG上的生长情况。结果显示,Xcc L3 (ΔXcclipA/pSRK-EclplA)在NYG平板上不能生长,而Xcc L4 (ΔXcclipB/pSRK-EclplA)能生长,但与野生菌Xcc8004相比生长较弱,而在添加硫辛酸(lip)的NYG培养上时,两种替换突变株都能恢复到野生菌的生长状态(图 3-A)。进一步测定了不同菌株在两种培养条件下的生长曲线,结果也显示,Xcc L3在NYG中不能生长,而Xcc L4能生长(图 3-B),但在添加硫辛酸后,Xcc L3、Xcc L4和野生菌的生长无差异(图 3-C)。
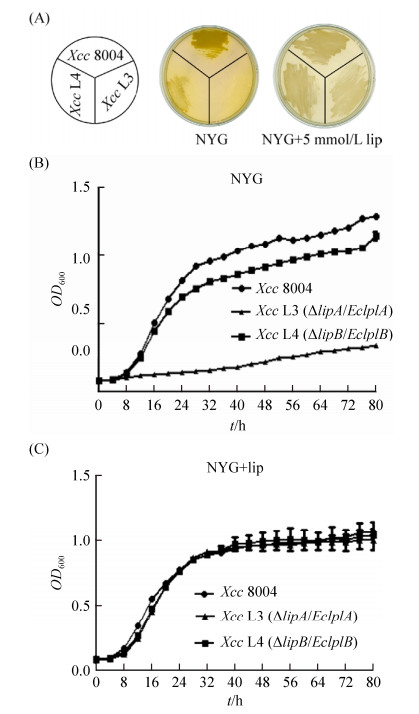 |
| 图 3 Xcc L3和Xcc L4替换突变株在丰富培养基上的生长表型检测 Figure 3 Growth phenotype of Xcc L3 and Xcc L4 on nutritional media. A: growth on NYG plates; B: growth curves in NYG. C: growth curves in NYG with lipoic aicd. lip: lipoic acid. Data are mean±one standard deviation of three independent assays. |
| 图选项 |
其次,分析了两个替换突变株Xcc L3和Xcc L4在基础培养基XOG上的生长情况。结果显示,野生菌Xcc 8004能正常生长,但Xcc L3和Xcc L4突变株都不能生长(图 4-A)。但在XOG培养基中添加50 nmol/L硫辛酸(lip)后,Xcc L3和Xcc L4替换突变株都能恢复到野生菌的生长水平(图 4-A)。进一步测定不同条件下的生长曲线,结果也显示类似结果,Xcc L3和Xcc L4只能在添加有硫辛酸的XOG培养基中才能恢复生长(图 4-B和C)。以上结果说明,XcclipA或XcclipB基因被大肠杆菌lplA基因替换后,都直接影响了菌株的生长,但在不同培养条件下,两种替换突变株出现了不同的生长性状。
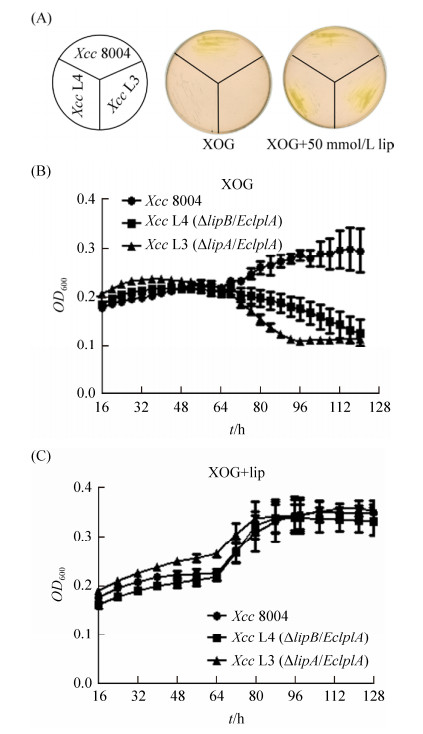 |
| 图 4 Xcc L3和Xcc L4替换突变株在基础培养基上的生长表型检测 Figure 4 Growth phenotype of Xcc L3 and Xcc L4 on minimal media. A: Growth on XOG plates. B: Growth curves in XOG. C: Growth curves in XOG with lipoic aicd. lip: lipoic acid. Data are mean± one standard deviation of three independent assays. |
| 图选项 |
2.5 基因替换突变株致病性分析 为了解硫辛酸合成关键基因被替换后是否影响Xcc的致病性,本研究进一步利用剪叶法,分析了野生菌株和两种替换突变株对寄主植物甘蓝的致病性。结果显示,野生菌Xcc8004能引起寄主植物病斑,但两种替换突变株出现不同的侵染结果,其中Xcc L4 (ΔXcclipB/pSRK-EclplA)菌株也能引起植物病斑,而Xcc L3 (ΔXcclipA/pSRK- EclplA)几乎不能导致植物病斑(图 5-A)。进一步统计叶片病斑长度显示,Xcc8004引起的平均病斑长度为6.6 mm,而Xcc L3引起的平均病斑长度仅为0.3 mm,与野生菌有极其显著性差异,但Xcc L4引起的平均病斑长度达到4.5 mm,与野生菌的病斑长度无显著性差异(图 5-B)。
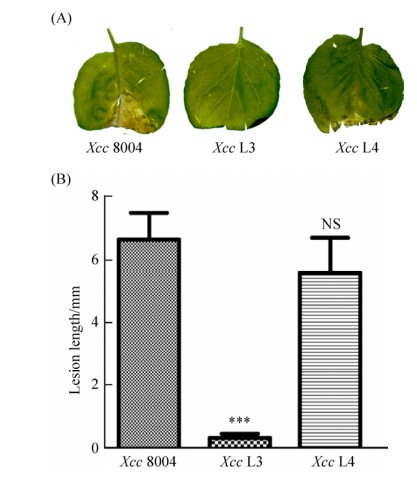 |
| 图 5 Xcc L3和Xcc L4替换突变株致病力检测 Figure 5 Virulence assay of Xcc L3 and Xcc L4. A: black rot symptoms caused by different strains on cabbage leaves inoculated by leaf-clip method. B: average lesion lengths caused by different strains. Values are expressed as the means and standard deviations of triplicate measurements, each comprised of 10 leaves. The asterisk (***) indicates P < 0.001, NS means no significant difference. |
| 图选项 |
3 讨论 B族维生素硫辛酸作为一种抗氧化剂被广泛使用,其医用价值和应用领域不断扩展。而在细菌中硫辛酸作为辅因子参与2-酮酸脱氢酶复合体的催化反应,涉及细胞的能量代谢、一碳单位生成以及脂肪酸和氨基酸合成等许多重要的代谢过程,因此干扰硫辛酸合成或吸收可作为新型抗菌药物的筛选策略[22-24]。野油菜黄单胞菌(Xcc)能引起几乎所有的十字花科植物黑腐病,包括许多的经济类蔬菜等。黑腐病不仅影响产量,也影响品质,在全球范围内引起重大经济损失,当前的抗性品种选育或化学药物处理方法都效果不佳,亟需寻找新的黑腐病防控策略。但Xcc的硫辛酸合成机制还未见报道。
本研究首先通过生物信息学分析的方法,通过同源性比对在Xcc基因组中发现XcclipA和XcclipB基因,但没有发现与大肠杆菌硫辛酰蛋白连接酶(EcLplA)具有同源性的基因,推测Xcc中LipA-LipB合成途径是其获得硫辛酸的唯一途径。而后分别将XcclipA和XcclipB分别异体遗传互补大肠杆菌lipA和lipB基因突变株,结果显示都能恢复突变株在基础培养基上的生长,初步证明XcclipA编码蛋白具有硫辛酸合成酶活性,而XccLipB具有辛酰转移酶活性。进一步尝试在基因组中分别敲除XcclipA和XcclipB,但在多种条件下都不能获得基因敲除突变株,而在导入大肠杆菌lplA基因并添加硫辛酸后成功获得敲除突变株,证明XcclipA和XcclipB都是Xcc生长的必需基因,Xcc不能利用环境中的硫辛酸而只能从头合成。
本研究还分析了两种EclplA替换突变株的生长性状。在丰富培养基中,XcclipA敲除菌株不能生长,但XcclipB敲除菌株能生长,而在基础培养基中,XcclipA和XcclipB敲除菌株都不能生长。但在培养基中添加硫辛酸后两种替换突变株均能恢复到野生菌的生长表型。由于大肠杆菌EcLplA不仅可将硫辛酸转移到脱氢酶的E2亚基上,也可以将辛酸连接到脱氢酶的E2亚基上,即可部分取代LipB的功能[6]。因此在丰富培养基中,可能含有少量的辛酸,EclplA替换的XcclipB敲除菌株可以生长,而在贫瘠的基础培养上由于没有辛酸,XcclipB突变株也不能生长,只有添加硫辛酸后才能恢复生长。进一步分析两种EclplA替换突变株对寄主植物甘蓝的致病力,结果显示XcclipA敲除株几乎丧失了致病力,而XcclipB突变株侵染的病斑长度与野生菌无显著性差异。
硫辛酸作为辅因子在基础代谢中发挥重要作用,但细胞对硫辛酸的需求浓度并不高。本研究中在基础培养基中添加50 nmol/L硫辛酸,即能完全满足突变菌株的生长,而提高硫辛酸含量至1 μmol/L时显著抑制Xcc的生长。此现象与文献报道类似,极低浓度即可满足菌株生长[4, 25-26]。但在丰富培养基中,硫辛酸浓度即使提高到5 mmol/L时也不抑制Xcc的生长,但具体原因还有待继续探索。
不同来源的细菌中硫辛酸合成及吸收途径都具有较高的多样性,也为开发特异性的抗菌药物提供了可能性。大肠杆菌同时具有LipB-LipA合成途径和LplA参与的外源硫辛酸吸收途径,而枯草芽孢杆菌具有LipM-LipL-LipA合成途径和LplJ吸收途径[10],金黄色葡萄球菌具有LipM-LipA-LipL合成途径和LplA1/LplA2共参与的吸收途径,而单增李斯特菌(Listeria monocytogenes)不具有硫辛酸合成能力,只能利用LplA1从寄主细胞中吸收硫辛酸[27]。本研究发现Xcc只具有LipB-LipA合成途径,不能直接吸收利用外源硫辛酸,而XcclipA敲除株的致病力几乎丧失,表明XccLipA可能是一个良好的防控黑腐病药物的筛选靶点。
References
| [1] | Reed KE, Cronan JE Jr. Lipoic acid metabolism in Escherichia coli: sequencing and functional characterization of the lipA and lipB genes. Journal of Bacteriology, 1993, 175(5): 1325-1336. DOI:10.1128/JB.175.5.1325-1336.1993 |
| [2] | Sun YR, Zhang WB, Ma JC, Pang HS, Wang HH. Overproduction of α-lipoic acid by gene manipulated Escherichia coli. PLoS One, 2017, 12(1): e0169369. DOI:10.1371/journal.pone.0169369 |
| [3] | Cronan JE Jr. Advances in synthesis of biotin and assembly of lipoic acid. Current Opinion in Chemical Biology, 2018, 47: 60-66. DOI:10.1016/j.cbpa.2018.08.004 |
| [4] | Cronan JE Jr. Assembly of lipoic acid on its cognate enzymes: an extraordinary and essential biosynthetic pathway. Microbiology and Molecular Biology Reviews, 2016, 80(2): 429-450. DOI:10.1128/MMBR.00073-15 |
| [5] | Spalding MD, Prigge ST. Lipoic acid metabolism in microbial pathogens. Microbiology and Molecular Biology Reviews, 2010, 74(2): 200-228. DOI:10.1128/MMBR.00008-10 |
| [6] | Morris TW, Reed KE, Cronan JE Jr. Lipoic acid metabolism in Escherichia coli: the lplA and lipB genes define redundant pathways for ligation of lipoyl groups to apoprotein. Journal of Bacteriology, 1995, 177(1): 1-10. DOI:10.1128/JB.177.1.1-10.1995 |
| [7] | Cronan JE Jr. Biotin and lipoic acid: synthesis, attachment and regulation. Ecosal Plus, 2014, 6(1). DOI:10.1128/ecosalplus.ESP-0001-2012 |
| [8] | Jordan SW, Cronan JE Jr. The Escherichia coli lipB gene encodes lipoyl (octanoyl)-acyl carrier protein: protein transferase. Journal of Bacteriology, 2003, 185(5): 1582-1589. DOI:10.1128/JB.185.5.1582-1589.2003 |
| [9] | Cronan JE Jr. The structure of lipoyl synthase, a remarkable enzyme that performs the last step of an extraordinary biosynthetic pathway. Biochemical Journal, 2014, 464(1): e1-3. |
| [10] | Martin N, Christensen QH, Mansilla MC, Cronan JE Jr, de Mendoza D. A novel two-gene requirement for the octanoyltransfer reaction of Bacillus subtilis lipoic acid biosynthesis. Molecular Microbiology, 2011, 80(2): 335-349. DOI:10.1111/j.1365-2958.2011.07597.x |
| [11] | Martin N, Lombardia E, Altabe SG, de Mendoza D, Mansilla MC. A lipA (yutB) mutant, encoding lipoic acid synthase, provides insight into the interplay between branched-chain and unsaturated fatty acid biosynthesis in Bacillus subtilis. Journal of Bacteriology, 2009, 191(24): 7447-7455. DOI:10.1128/JB.01160-09 |
| [12] | Zorzoli A, Grayczyk JP, Alonzo F Ⅲ. Staphylococcus aureus tissue infection during sepsis is supported by differential use of bacterial or host-derived lipoic acid. PLoS Pathogens, 2016, 12(10): e1005933. DOI:10.1371/journal.ppat.1005933 |
| [13] | Mashruwala AA, Roberts CA, Bhatt S, May KL, Caroll RK, Shaw LN, Boyd JM. Staphylococcus aureus SufT: an essential iron-sulphur cluster assembly factor in cells experiencing a high-demand for lipoic acid. Molecular Microbiology, 2016, 102(6): 1099-1119. DOI:10.1111/mmi.13539 |
| [14] | Lanz ND, Lee KH, Horstmann AK, Pandelia ME, Cicchillo RM, Krebs C, Booker SJ. Characterization of lipoyl synthase from Mycobacterium tuberculosis. Biochemistry, 2016, 55(9): 1372-1383. DOI:10.1021/acs.biochem.5b01216 |
| [15] | Yu YH, Ma JC, Guo QQ, Ma JC, Wang HH. A novel 3-oxoacyl-ACP reductase (FabG3) is involved in the xanthomonadin biosynthesis of Xanthomonas campestris pv. campestris. Molecualr Plant Pathology, 2019, 20(12): 1696-1709. DOI:10.1111/mpp.12871 |
| [16] | Hu Z, Dong HJ, Ma JC, Yu YH, Li KH, Guo QQ, Zhang C, Zhang WB, Cao XY, Cronan JE, Wang HH. Novel Xanthomonas campestris long-chain-specific 3-oxoacyl-acyl carrier protein reductase involved in diffusible signal factor synthesis. mBio, 2018, 9(3): e00596-18. |
| [17] | Yu YH, Hu Z, Dong HJ, Ma JC, Wang HH. Xanthomonas campestris FabH is required for branched-chain fatty acid and DSF-family quorum sensing signal biosynthesis. Scientific Reports, 2016, 6: 32811. DOI:10.1038/srep32811 |
| [18] | Khan SR, Gaines J, Roop RM Ⅱ, Farrand SK. Broad-host-range expression vectors with tightly regulated promoters and their use to examine the influence of TraR and TraM expression on Ti plasmid quorum sensing. Applied and Environmental Microbiology, 2008, 74(16): 5053-5062. DOI:10.1128/AEM.01098-08 |
| [19] | Guzman LM, Belin D, Carson MJ, Bechwith J. Tight regulation, modulation, and high-level expression by vectors containing the arabinose PBAD promoter. Journal of Bacteriology, 1995, 177(14): 4121-4130. DOI:10.1128/JB.177.14.4121-4130.1995 |
| [20] | Wei K, Tang DJ, He YQ, Feng JX, Jiang BL, Lu GT, Chen B, Tang JL. hpaR, a putative marR family transcriptional regulator, is positively controlled by HrpG and HrpX and involved in the pathogenesis, hypersensitive response, and extracellular protease production of Xanthomonas campestris pathovar campestris. Journal of Bacteriology, 2007, 189(5): 2055-2062. |
| [21] | Liu YF, Liao CT, Chiang YC, Li CE, Hsiao YM. WxcX is involved in bacterial attachment and virulence in Xanthomonas campestris pv. campestris. Journal of Basic Microbiology, 2018, 58(5): 403-413. DOI:10.1002/jobm.201700591 |
| [22] | Grayczyk JP, Harvey CJ, Laczkovich I, Alonzo F Ⅲ. A lipoylated metabolic protein released by Staphylococcus aureus suppresses macrophage activation. Cell Host & Microbe, 2017, 22(5): 678-687.e9. |
| [23] | Tort F, Ferrer-Cortes X, Ribes A. Differential diagnosis of lipoic acid synthesis defects. Journal of Inherited Metabolic Disease, 2016, 39(6): 781-793. DOI:10.1007/s10545-016-9975-4 |
| [24] | Lu WF, Cao JJ, Guo YJ, Zhong K, Zha GM, Wang LF, Yang GY. Expression of the porcine lipoic acid synthase (LIAS) gene in Escherichia coli. Genetics and Molecular Research, 2014, 13(3): 5369-5377. DOI:10.4238/2014.July.24.16 |
| [25] | Zhang HM, Luo QX, Gao HC, Feng YJ. A new regulatory mechanism for bacterial lipoic acid synthesis. MicrobiologyOpen, 2015, 4(2): 282-300. DOI:10.1002/mbo3.237 |
| [26] | Hermes FA, Cronan JE Jr. The role of the Saccharomyces cerevisiae lipoate protein ligase homologue, Lip3, in lipoic acid synthesis. Yeast, 2013, 30(10): 415-427. |
| [27] | Christensen QH, Hagar JA, O'riordan MXD, Cronan JE. A complex lipoate utilization pathway in Listeria monocytogenes. Journal of Biological Chemistry, 2011, 286(36): 31447-31456. DOI:10.1074/jbc.M111.273607 |
