陶嫦立

 , 张琴, 曾文静, 谭有为, 邵红伟
, 张琴, 曾文静, 谭有为, 邵红伟 广东药科大学生命科学与生物制药学院, 广东省生物技术候选药物研究重点实验室, 广东 广州 510006
收稿日期:2019-10-06;修回日期:2020-01-08;网络出版日期:2020-06-10
基金项目:国家自然科学基金(81703053)
*通信作者:陶嫦立, Te/Fax:+86-20-39352201;E-mail:taochangli@126.com.
摘要:[方法] 选择50只健康的10周龄BALB/c小鼠,随机分为2组,其中15只为正常对照组,余下35只饮用水中含4种抗生素连续喂养7 d,诱导小鼠肠道菌群严重失调后,再随机分为长链菊粉恢复组(饮用水中添加5%(W/W)长链菊粉)和自发恢复组(饮用水为无菌水),连续处理21 d。受试小鼠在抗生素治疗后的第7天以及恢复喂养的第7、14和21天,取结肠组织进行切片然后进行HE染色分析,无菌取粪便进行16S rRNA测序分析,观察小鼠肠道组织及菌群恢复情况。[结果] 抗生素处理7 d后,小鼠结肠组织有轻微炎症,但肠道菌群严重失调。组织学分析表明,在补充长链菊粉或自发恢复21 d后,结肠炎症逐渐减轻;但相比于自发恢复,长链菊粉干预延迟了结肠组织的恢复。16S rRNA基因V3-V4区扩增子测序分析显示无论是长链菊粉补充还是自发恢复都无法在属水平上恢复肠道菌群组成。尤其是长链菊粉的补充,反而导致了某些机会致病菌的选择性扩增,并提高了与肠道菌群相关的疾病途径。[结论] 抗生素诱导肠道菌群严重失调后补充长链菊粉会延迟肠道菌群的重建,可能会导致潜在的不良影响。
关键词:长链菊粉抗生素肠道菌群菌群重建
Effects of long-chain inulin on the reconstruction of gut microbiota in mice after antibiotics treatment
Tao Changli

 , Zhang Qin, Zeng Wenjing, Tan Youwei, Shao Hongwei
, Zhang Qin, Zeng Wenjing, Tan Youwei, Shao Hongwei Guangdong Province Key Laboratory of Biotechnology Drug Candidates, School of Biosciences and Biopharmaceutics, Guangdong Pharmaceutical University, Guangzhou 510006, Guangdong Province, China
Received: 6 October 2019; Revised: 8 January 2020; Published online: 10 June 2020
*Corresponding author: Changli Tao, Tel/Fax: +86-20-39352201; E-mail: taochangli@126.com.
Foundation item: Supported by the National Natural Science Foundation of China (81703053)
Abstract: [Objective] To investigate the effects of long-chain inulin supplementation on the gut microbiota recovery after antibiotics treatment induced dysbiosis. [Methods] Fifty healthy 10-week-old BALB/c mice were randomly divided into 2 groups, 15 mice were used as controls (the Ctrl group), and the remaining 35 mice were provided water containing 4 antibiotics (the ABx group) ad libitum for 7 days. On the 8th day, the antibiotic-induced dysbiosis mice were randomly divided into 2 groups. One group was provided drinking water containing 5% inulin as the inulin recovery group (the Ire group), and the other group received normal drinking water as the spontaneous recovery group (the Sre group). The treatment continued for 21 continuous days. On the 7th day after ABx treatment and on the 7th, 14th and 21th day of the recovery treatment, the colon samples were collected and subjected to histological analysis; the feacal samples were collected and subjected to 16S rRNA V3-V4 region sequencing and analyzed with bioinformatic softwares. [Results] The antibiotics treatment induced slight colonic inflammation but serious gut microbiota dysbiosis. Histological analysis showed that the colonic inflammation gradually decreased after 21 days of either long chain inulin supplementation or spontaneous recovery. However long-chain inulin intervention delayed the recovery of colon tissue compared to spontaneous recovery. Neither inulin supplementation nor spontaneous recovery could restore gut microbiota composition at the genus level. In particular, long-chain inulin supplementation might result in selective expansion of some opportunistic pathogens and elevated the pathways associated with diseases linked to gut microbiota function. [Conclusion] Long-chain inulin supplementation, after antibiotic-induced severe gut microbiota dysbiosis, delayed the reconstruction of the gut microbiota and might led to potential adverse effects.
Keywords: long-chain inulinantibioticsgut microbiotamicrobiota reconstitution
人体肠道微生态是一个包含了1000多种微生物的复杂的生态系统,作为人体的特殊“器官”,这些种类繁多的肠道菌群具有非常重要的生理功能[1]。不仅能够参与宿主代谢、促进物质消化吸收、抑制肠道致病菌生长,而且还在胃肠道系统的发育和自稳过程中起关键作用,对心血管系统和免疫系统亦具有重要影响[2]。然而长期大量使用广谱抗生素后,大多数敏感细菌与正常菌群被抑制或杀灭,但耐药菌以及一些外来的机会致病菌则获得生存优势并大量繁殖从而引起多种疾病的发生[3]。一直以来,人们普遍认为采用益生菌或益生元可调节抗生素引起的菌群失调,促进机体内有益菌的定植和繁殖,从而达到恢复肠道菌群的目的[4–5]。然而目前的研究显示益生菌或益生元对肠道菌群紊乱的调节并不都是有益的[6],例如最近有研究表明缺失了鞭毛蛋白受体TLR5并因此引起肠道菌群严重失调的基因敲除小鼠(T5KO)在补充菊粉6个月后诱发了胆汁淤积性肝癌[7]。因此益生元的补充可能受到宿主的状态等多方面因素的影响。对于由抗生素引起的肠道微生态严重失调后,长链菊粉对肠道菌群重建的影响尚不清楚。因此本研究采用4种抗生素鸡尾酒诱导小鼠肠道菌群严重失调,在补充长链菊粉和自发恢复3周后,通过检测结肠组织切片、肠道菌群组成结构和功能的变化,阐明在抗生素引起的肠道菌群严重失调后采用长链菊粉干预对肠道菌群重建的影响。
1 材料和方法 1.1 试剂 抗生素:氨苄青霉素(Biofroxx,货号:1146GR001),甲硝唑(Sigma-Aldrich,货号:M1547),新霉素(Sigma-Aldrich,货号:N6386),万古霉素(Sigma-Aldrich,货号:V2002)。长链菊粉(Orafti HP):比利时ORAFTI公司进口分装(纯度≥95%,平均聚合度 > 23)。
1.2 实验动物和实验设计 50只健康的8周龄BALB/c小鼠,适应喂养2周后,随机分为两组,其中15只为正常对照组(Ctrl组),余下35只为抗生素诱导的菌群失调模型组(ABx组)。菌群失调模型组饮用水中含有4种广谱抗生素[氨苄青霉素(1 mg/mL),甲硝唑(1 mg/mL),新霉素(1 mg/mL),万古霉素(0.5 mg/mL)],连续饮用7 d,每隔2 d更换1次含抗生素的饮用水。建模成功后将菌群失调模型组小鼠再随机分为2组,其中一组为长链菊粉恢复组Ire组[饮用水中添加5% (W/W)长链菊粉],另一组为自发恢复组Sre组(饮用水为无菌水)。连续恢复喂养21 d (图 1-A)。在实验开始后每2 d记录体重,每天记录食物摄入量和耗水量。
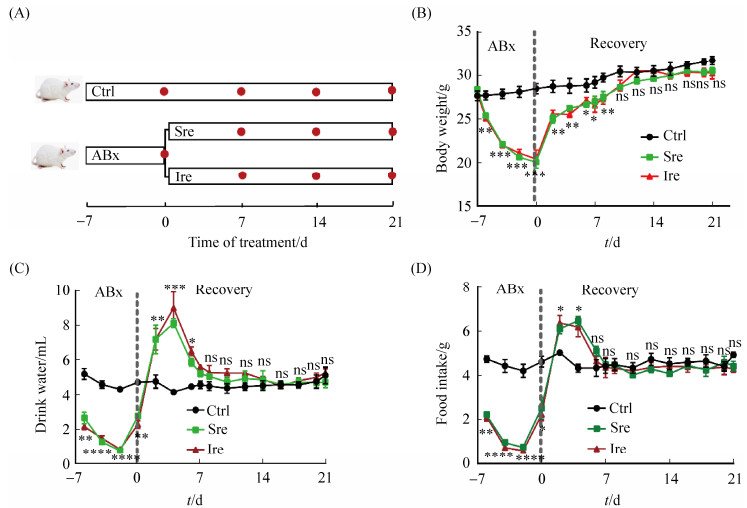 |
| 图 1 小鼠实验方案及各组小鼠体重及饮食变化趋势 Figure 1 Mice experimental protocol and the trend of body weight and food and drink change in different groups. A: Study design; B: The trend of body weight changes; C: The trend of drink changes; D: The trend of food changes. Ctrl: n=15, ABx: n=35, Sre: n=15, Ire: n=15. Data are expressed as mean±SEM. *: P < 0.05; **: P < 0.01; ***: P < 0.001; ****: P < 0.0001. |
| 图选项 |
1.3 样品采集 分别在抗生素处理后第7天,恢复喂养第7、14和21天收集粪便样品,立即快速冷冻于液氮中保存。另外各个采样时间点使用CO2对小鼠实施安乐死,然后断颈,小心地取出结肠组织并立即保存于4%多聚甲醛溶液中。
1.4 组织化学分析 新鲜的近端结肠组织在4%多聚甲醛溶液中固定24 h后,脱水并包埋入石蜡中,然后制片(4 μm)并用苏木精和曙红(H & E)染色。在光学显微镜NIKON ECLIPSE Ci-S(NIKON,Japan)下观察并描述,接着采用成像系统NIKON digital sight DS-FI2 (NIKON,Japan)对主要描述部位进行拍照(拍摄倍数:100×、200×等)。组织学评分评价结肠炎症程度,样本的上皮损伤、淋巴细胞聚集和浆膜层与肌层的水肿的评分为0–4 (其中0=无,1=轻度,2=中度,3=严重,4=非常严重)。
1.5 肠道微生物测序及其菌群分析 采用QIAamp DNA Mini Kit (QIAGEN公司,德国)提取小鼠粪便样品中总DNA,用微量核酸蛋白分析仪(NanoDrop Technologies Inc,美国)测定DNA浓度和纯度。使用通用引物338F (5′-ACTC CTACGGGAGGCAGCA-3′)和806R (5′-GGACTA CHVGGGTWTCTAAT-3′)扩增16S rRNA基因V3–V4可变区,在Illuminate Hiseq 2500 (北京百迈克生物科技有限公司)平台完成测序。使用Trimmomatic软件[8]对测序数据进行过滤,通过FLASH软件[9]将有overlap的一对reads进行拼接。使用Usearch[10]对Tags在97%的相似度水平下进行聚类、获得OTU,并基于Silva (细菌)数据库采用RDP classifier对OTU进行物种分类学注释,统计每个样品在各分类水平上的构成。利用Mothur软件[11]计算97%相似度的OTU的Alpha多样性指数(ACE、Chao1、Shannon和Simpson指数),利用R语言制作稀释曲线图和香农指数曲线图。利用QIIME软件[12]在属水平上进行Beta多样性分析,基于Bray-Curtis距离算法进行principal coordinates analysis (PCoA)统计分析和作图。利用QIIME软件基于Weight unifrac距离进行ANOSIM及Adonis组间相似性分析,检验组间差异的显著性。利用LEfSe软件[13]通过非参数因子Kruskal-Wallis秩和检验检测不同样本间物种的丰度差异的显著性水平,获得ABx、Ctrl、Sre组和Ire组间显著性差异物种。
原始测序数据已上传至NCBI的Sequence Read Archive (SRA)数据库,BioProject登录号为PRJNA575094,BioSample登录号为SAMN12877561- SAMN12877593。
1.6 基于16S rRNA测序的菌群基因预测分析 以已知的微生物基因作为参考,使用PICRUSt软件[14]预测16S rRNA测序数据功能基因的组成,从而获得不同样本或分组之间在功能上的差异。简言之通过每个OTU对应的greengene ID,获得OTU对应的KEGG和COG家族信息,计算该KEGG和COG的丰度并从KEGG数据库的信息中获得Pathway、EC信息、OTU丰度,然后计算各功能类别的丰度。使用STAMP软件[15]计算不同样本间的物种基因丰度并作图,对不同组间进行两两T-test检验,P < 0.05表示两组间有显著性差异。
1.7 统计分析 采用SPSS 20.0软件进行统计学分析,造模前数据用非配对t检验比较Ctrl组和ABx组之间差异;造模后数据使用两因素方差分析进行各组之间的比较,多重比较采用Student-Newman-Keuls进行检验,P < 0.05表示差异显著。所有结果均表示为平均值±SEM。使用GraphPad Prism 6.0程序(GraphPad,Inc)进行作图。
2 结果和分析 2.1 小鼠体重、饮食指标检测 与对照组相比,ABx造模组小鼠饮水与饲料摄入在前5 d急剧下降,相应的体重也逐渐下降(P > 0.05) (图 1-B、C、D)。在第8天停止抗生素处理并在饮用水中加入菊粉(Ire组)或饮用水不做其他干预(Sre组)后,小鼠的饮水与饲料摄入突然升高且远高于对照组并持续4 d (P > 0.05) (图 1-C、D);小鼠体重也逐渐恢复,至恢复喂养的第12天已经与对照组的体重无统计学差异(P < 0.05) (图 1-B)。
2.2 抗生素处理引发肠道炎症并能在停止抗生素处理后逐渐恢复 结肠组织经切片后HE染色,结果显示Ctrl组肠粘膜层上皮完整,肠腺排列整齐规则,多见皱襞结构,粘膜下层紧密。而ABx组明显充血、上皮严重脱落且不连续、肠黏膜水肿(绿色箭头),浆膜层与肌层有不同程度的水肿(红色箭头);血管内可见少量淋巴细胞聚集(黑色箭头) (图 2-A)。
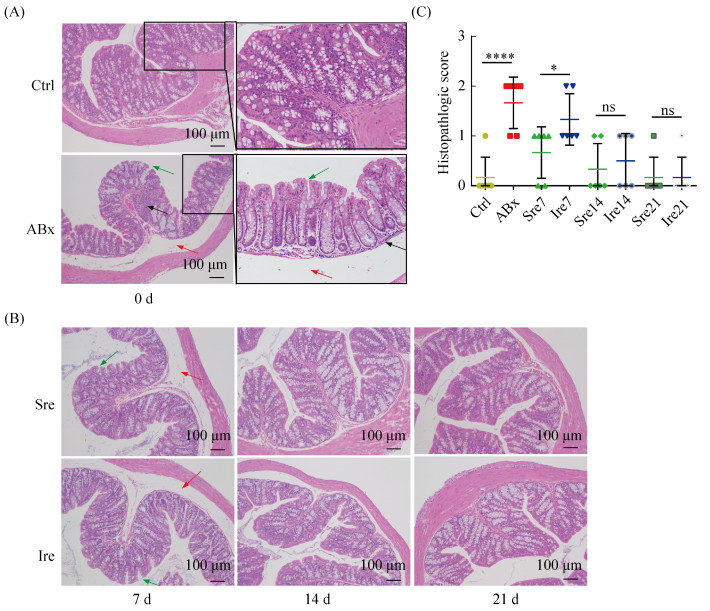 |
| 图 2 菊粉干预恢复和自发恢复后结肠组织切片分析 Figure 2 Histological analysis of the colon after spontaneous recovery and inulin-supplemented recovery. A: ABx-induced colon inflammation; B: Spontaneous recovery and inulin-supplemented recovery at 7, 14, 21 days after ABx treatment; C: Statistical analysis of histopathologic scores in each group (n=5–6). *: P < 0.05; ****: P < 0.0001. |
| 图选项 |
在恢复喂养7 d后即Sre7组或是Ire7组,肠黏膜仍然水肿、肠间质明显充血,而Ire7组相比于Sre7组,结肠上皮依然可见有脱落且不连续;恢复喂养14 d后,Sre14组和Ire14组结肠炎症均不同程度减轻且无统计学差异(P < 0.05);恢复喂养21 d后,Sre21组和Ire21组结肠均与对照组无异且无统计学差异(P < 0.05),说明结肠组织炎症基本恢复(图 2-B、C)。
2.3 菊粉干预延迟了抗生素处理后小鼠肠道菌群多样性的恢复 在各个采样点取小鼠粪便进行16S rRNA基因V3–V4可变区测序以分析肠道菌群组成。各组小鼠肠道细菌alpha多样性指数如表 1所示,ACE和Chao1指数可反映微生物丰富度,Shannon和Simpson指数可反映微生物的多样性,ABx组样本细菌的丰富度和多样性相比于Ctrl组急剧降低,随着恢复喂养时间的延长,Ire组与Sre组细菌的丰富度和多样性逐渐升高,然而Ire组始终低于Sre组。值得注意的是,恢复喂养1周后Ire7组的OTU (78个)反而少于ABx组,而Sre7组OTU已经上升至144个。Rarefaction曲线和Shannon曲线表明测序深度和覆盖率足以反映样本中菌群的多样性信息(图 3-A、B),虽然可能还有少量细菌未被检测到,但现有序列已能满足后续分析要求。
表 1. 菊粉干预恢复和自发恢复后肠道菌群的alpha多样性 Table 1. Alpha diversity of gut bacteria after spontaneous recovery and inulin-supplemented recovery
| Sample | OTU | ACE | Chao1 | Simpson | Shannon |
| ABx | 130 | 299.3090 | 240.6250 | 0.9720 | 0.1151 |
| Ctrl0 | 310 | 315.8480 | 317.3333 | 0.0381 | 4.1254 |
| Ctrl7 | 329 | 332.8039 | 331.7692 | 0.0699 | 3.8441 |
| Ctrl14 | 323 | 332.7765 | 337.2500 | 0.0669 | 3.6876 |
| Ctrl21 | 319 | 324.3903 | 325.8750 | 0.1034 | 3.6851 |
| Sre7 | 144 | 254.3380 | 195.0000 | 0.1172 | 2.7556 |
| Sre14 | 226 | 239.1321 | 252.2500 | 0.0475 | 3.7006 |
| Sre21 | 250 | 258.6374 | 261.3750 | 0.0405 | 3.9729 |
| Ire7 | 78 | 186.3115 | 109.6250 | 0.1868 | 2.1891 |
| Ire14 | 118 | 128.5021 | 126.2500 | 0.1718 | 2.4751 |
| Ire21 | 189 | 213.2672 | 207.4000 | 0.0760 | 3.2289 |
表选项
 |
| 图 3 菊粉干预恢复和自发恢复后肠道微生物丰富度和多样性的变化 Figure 3 Changes in gut microbial abundance and diversity after spontaneous recovery and inulin-supplemented recovery. A: Rarefaction curve; B: Shannon index curve; C: PCoA on Genus level; D: Anosim_distances on Genus level. Data are expressed as the mean±SEM (n=3). |
| 图选项 |
另一种多样性度量方法——基于Bray curtis算法的PCoA分析表明,抗生素处理后的ABx组样本聚集在一起且与对照组Ctrl组样本之间距离较大,而对照组Ctrl0、Ctrl7、Ctrl14和Ctrl21的各样本均聚集在一起。随着恢复喂养时间的延长,Ire组和Sre组各组样本逐渐与ABx组样本分离,并且逐渐向Ctrl组靠近;其中恢复喂养至21 d后,Sre21组相比于Ire21组明显更靠近Ctrl组(图 3-C)。基于Weight unifrac算法的ANOSIM分析显示R值为0.713 (P < 0.001),说明组间差异明显大于组内差异,因此各组之间的差异具有显著意义(图 3-D)。以上结果说明在抗生素导致肠道微生物群严重失调后采用菊粉干预相比于自发恢复反而延迟了菌群多样性的恢复。
2.4 菊粉干预改变了抗生素处理后小鼠肠道菌群在属水平上的组成 各组样本门水平菌群组成柱形图显示,来自对照Ctrl0组小鼠的菌群主要以Firmicutes和Bacteriodetes菌门为主(图 4-A)。而抗生素处理后ABx组小鼠菌群中的Firmicutes和Bacteriodetes菌门相对丰度急剧下降,取而代之的是Proteobacteria菌门(98.60%±0.18% vs. 6.08%±1.61%,P < 0.0001) (图 4-B)。停止抗生素并恢复喂养1周后,Firmicutes和Bacteriodetes菌门的相对丰度很快恢复,然而Ire21组的Proteobacteria菌门的相对丰度高于Sre21组(11.06%±1.77% vs. 2.04%±0.25%,P < 0.05)和Ctrl21组(11.06%±1.77% vs. 2.79%±0.46%,P < 0.05)。值得注意的是,恢复至第21天,Saccharibacteria菌门细菌在Ire组和Sre组都难以恢复甚至被消除(图 4-C)。
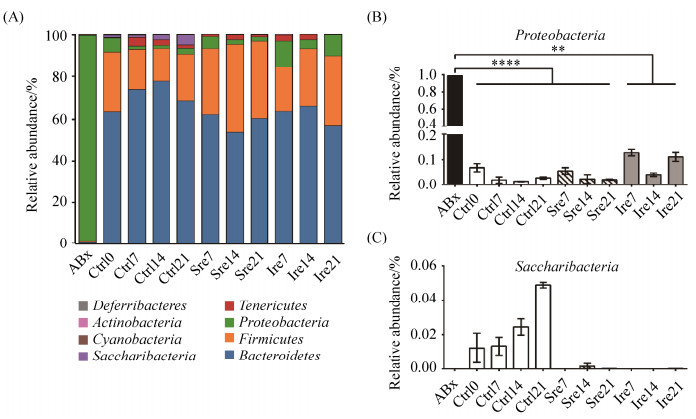 |
| 图 4 菊粉干预恢复与自发恢复后门水平的肠道菌群差异分析 Figure 4 Analysis of the difference between inulin-supplemented recovery and spontaneous recovery of gut bacterial composition at the phylum level. A: Gut bacterial composition at the phylum level in each group; B: Relative abundance of Proteobacteria in each group; C: Relative abundance of Saccharibacteria in each group. Data are expressed as mean±SEM (n=3). *: P < 0.05; **: P < 0.01; ***: P < 0.001; ****: P < 0.0001. |
| 图选项 |
在属的水平上,ABx组主要以Proteobacteria的Escherichia-Shigella为主,相对丰度为98.6% (P < 0.0001) (图 5-A)。停止抗生素后,在Ire组和Sre组中,Bacteroide、uncultured_bacterium_f_Lachnospiraceae和Lachnospiraceae_NK4A136_ group被大大扩增(P < 0.05)(图 5-B)。而在Ctrl组中占优势的细菌如Candidatus_Saccharimonas和Ruminococcaceae_UCG-014在Sre和Ire中依然保持减少,一些在Ctrl组中相对丰富的细菌例如Prevotella_1、Rikenella和Alistipes被消除而无法恢复(图 5-B)。值得注意的是,与Sre组相比,长链菊粉能促进Bifidobacterium的扩增,在恢复喂养的第7天,Ire7组中Bifidobacterium的相对丰度突然增高,有意思的是Proteus也在长链菊粉补充的第7天恢复并突然增多。虽然停止抗生素后条件致病菌Escherichia-Shigella的相对丰度大大降低,但是在Ire7组中的Escherichia-Shigella反而高于Sre7组(P < 0.05)。另外变形杆菌门细菌Helicobacter在Sre组中不能被恢复,而在Ire组中被恢复且随着喂养时间延长其相对丰度逐渐增加最后高于Ctrl组(P < 0.05)(图 5-B)。
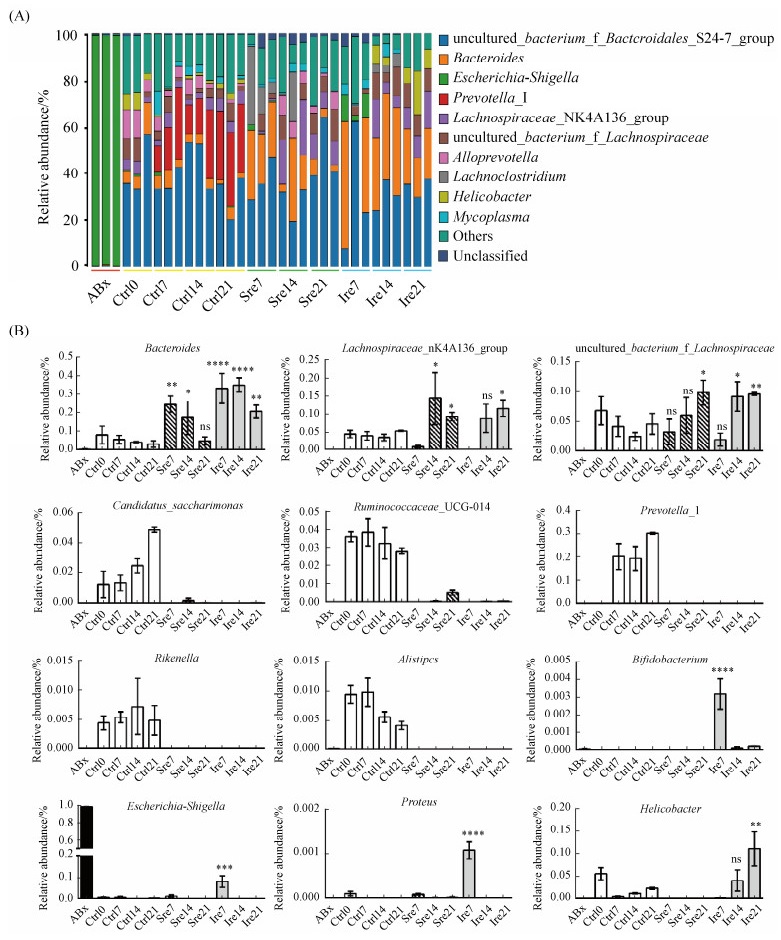 |
| 图 5 菊粉干预恢复与自发恢复后属水平的肠道菌群差异分析 Figure 5 Analysis of the difference between inulin-supplemented recovery and spontaneous recovery of gut bacterial composition at the genus level. A: Gut bacterial composition at the genus level in each sample; B: Relative abundance of bacterial taxa at the genus level in each group. Data are expressed as mean±SEM (n=3). *: P < 0.05; **: P < 0.01; ***: P < 0.001; ****: P < 0.0001. |
| 图选项 |
根据多级物种差异判别(LEfSe)分析显示了四组间丰度差异显著的物种(图 6),Ctrl组中菌群结构主要以Bacteroidetes菌门中的未分类的Bacteroidales_S24_7属、Prevotella_1属和Alloprevotella属以及Firmicutes菌门中的Ruminococcaceae_UCG_014属为代表;而在抗生素处理后的ABx组中菌群结构只剩Proteobacteria菌门中的Escherichia_Shigella属占优势;在Ire组菌群结构以Bacteroidaceae科中的Bacteroides属、Clostridiales目中uncultured_bacterium_f_Lachnospiraceae属和Peptoclostridium属以及Epsilonproteobacteria纲中的Helicobacter属为代表;Sre组中菌群结构则以Firmicutes菌门Clostridia纲中Lachnoclostridium属、Clostridium__innocuum_group属和uncultured_bacterium_f_Ruminococcaceae属为代表。
 |
| 图 6 菊粉干预恢复和自发恢复在不同水平的LEfSe分析 Figure 6 LEfSe analysis of inulin-supplemented recovery and spontaneous recovery on different levels. |
| 图选项 |
因此,抗生素对宿主肠道菌群的消耗导致肠道微生态严重失调后,采用长链菊粉干预相比于自发恢复反而延迟了肠道菌群多样性及组成结构的恢复;长链菊粉补充在刺激益生菌生长的同时,也选择性地促进了某些机会致病菌的生长。
2.5 菊粉干预提高了肠道菌群与疾病相关的功能途径 肠道微生物的组成和多样性变化也反映了功能上的差别。我们用已知微生物基因组作为参考,基于16S rRNA基因测序数据使用PICRUSt软件预测其功能基因含量,以评估各组微生物之间潜在的功能差异。在ABx组中,一些二级KEGG途径,包括细胞膜运输(membrane transport)、细胞运动(cell motility)、信号转导(signal transduction)以及疾病相关的途径如感染性疾病(infectious diseases)、癌症(cancers)、神经退行性疾病(neurodegenerative diseases)与Ctrl组相比被显著上调(P < 0.05)(图 7-A)。而与Ctrl组相比,ABx组中许多下调的KEGG功能差异都集中在代谢途径中,包括氨基酸代谢(amino acid metabolism)、能量代谢(energy metabolism)、核酸代谢(nucleotide metabolism)和其他次级代谢产物的生物合成(biosynthesis of other secondary metabolites)等;另外遗传过程和细胞过程相关途径包括折叠、分类和降解途径(folding, sorting and degradation)、转录途径(transcription)、复制和修复途径(replication and repair)以及细胞生长和死亡途径(cell growth and death)在ABx组中也下调(P < 0.05) (图 7-A)。在经过长链菊粉恢复和自发恢复21 d后,许多代谢途径恢复。然而,值得注意的是,Ire21组和Sre21组都没能恢复复制和修复途径(replication and repair),与Ctrl21组相比依然下调(P < 0.05) (图 7-B、C)。特别是经过21 d恢复喂养,Ire21组小鼠的脂代谢(lipid metabolism)和聚糖生物合成和代谢(glycan biosynthesis and metabolism)途径高于Sre21组小鼠,但是出乎意料的是,Ire21组中与疾病相关的途径例如感染性疾病(infectious diseases)和癌症(cancers)均明显高于Sre21组(P < 0.05)(图 7-D)。总之,抗生素处理导致肠道微生物几乎被耗竭,尤其是Firmicutes和Bacteriodetes菌门物种的相对丰度急剧降低,同时微生物多样性也急剧降低,菌群的潜在功能也被彻底改变。即使通过长链菊粉干预或自发恢复21 d后,肠道菌群的功能也依然不能恢复,并且长链菊粉的补充反而提高了小鼠肠道菌群与疾病相关途径的相对水平。
 |
| 图 7 菊粉干预恢复和自发恢复后16S rRNA功能基因预测及差异分析 Figure 7 Prediction and differential analysis of 16S rRNA functional genes after inulin-supplemented recovery and spontaneous recovery. A: Comparison of the Ctrl0 group with ABx group; B: Comparison of the Sre21 group with the Ctrl21 group; C: Comparison of the Ire21 group with the Ctrl21 group; D: Comparison of the Ire21 group with the Sre21 group. |
| 图选项 |
3 讨论 菊粉是一种天然果糖聚合物,属于可溶性膳食纤维,根据链的长短可分为短链菊粉(平均聚合度≤9)和长链菊粉(平均聚合度 > 23),由于其特殊的β-(2, 1)-糖苷键结构,使得菊粉在口腔、胃、小肠中均不会发生消化分解,只能在结肠中被肠道微生物作为底物分解产生少量的热量,因此菊粉作为一种益生元可以调节肠道菌群,改善宿主健康。许多随机对照试验表明,服用菊粉可以改善许多疾病,如体重过重和肥胖[16]、2型糖尿病[17]、代谢综合征和血脂异常[18]、炎症性肠病(IBD)[19]和便秘[20]。然而菊粉对肠道菌群的影响非常复杂,受到多种宿主和环境因素的影响,包括分娩方式和早期喂养[21]、抗生素[22]、疾病状况和成人饮食[23]。例如,最近一项研究表明,TLR5受体基因敲除小鼠连续补充菊粉6个月后促进了胆汁淤积,进而导致肝癌[7]。因此,补充菊粉并不是在任何情况下都是对宿主有益的,某些情况下可能会产生可怕的后果。而对广谱抗生素诱导肠道菌群严重失调后的宿主采用长链菊粉干预后的影响目前尚不清楚。
因此,我们在广谱抗生素治疗后肠道菌群严重失调的小鼠模型中评估了补充长链菊粉对其肠道菌群重建的影响。我们的研究证实即使只是短短7 d的广谱抗生素处理也会导致肠道菌群严重失调。16S rRNA基因测序分析表明,厚壁菌门和拟杆菌门的细菌几乎被消耗殆尽,取而代之的是的变形杆菌门细菌,其相对丰度达98%。在长链菊粉干预和自发恢复喂养21 d后,小鼠肠道微生物群的多样性依然没能恢复至对照组水平,特别是菊粉的补充反而使菌群在属水平的组成与初始组成完全相反(前50)。虽然,双歧杆菌的相对丰度在补充长链菊粉后恢复并高于自发恢复,但是我们观察到长链菊粉干预相比于自发恢复明显促进了一些瞬时性微生物的显著扩增,如变形杆菌门细菌Escherichia-Shigella和Helicobacter,然而它们并不是对照小鼠肠道微生物群中的优势物种,尤其Escherichia-Shigella是一种机会致病菌。因此我们推测,在抗生素引起的微生物群被耗竭的情况下,长链菊粉的补充不仅刺激了益生菌的生长,还可能选择性地促进一些致病菌的生长。因此长链菊粉干预相比于自发恢复反而延迟了结肠炎的恢复。
通过进一步基于16S rRNA测序的菌群基因预测分析发现,抗生素治疗导致的微生物群的耗竭,彻底改变了肠道菌群的潜在功能。KEGG二级代谢通路显示,相比于对照组,抗生素处理组的许多代谢途径下调,特别是复制和修复途径(replication and repair)以及细胞生长和死亡途径(cell growth and death)的下调说明抗生素对肠道菌群的直接作用严重影响了肠道微生物的生长,而与疾病相关的代谢途径如感染性疾病(infectious diseases)、癌症(cancers)、神经退行性疾病(neurodegenerative diseases)升高,也反映了抗生素提高了机会致病菌的相对丰度。在停止抗生素后,肠道菌群及其功能也逐渐恢复,然而长链菊粉恢复组中的脂质代谢(lipid metabolism)和聚糖生物合成和代谢(glycan biosynthesis and metabolism)途径的丰度比自发恢复组和对照组的要高得多,这与之前的研究显示菊粉能被肠道细菌代谢并产生短链脂肪酸(SCFA)的结果一致[24],正如我们的研究显示长链菊粉的补充刺激了双歧杆菌的生长,而双歧杆菌降解菊粉产生的单糖可通过交叉饲喂(cross-feeding)的方式促进产丁酸盐细菌的相对丰度[25]进而提高脂质代谢及多糖类的生物代谢。但是值得注意的是,一些疾病相关的途径,如传染病、癌症在长链菊粉恢复组依然高于自发恢复组。这一观察表明,在抗生素治疗后导致的肠道微生态严重失调的情况下,长链菊粉补充虽然促进了某些有益菌的生长,但是也刺激了某些机会致病菌使其获得生存优势并大量繁殖,进而改变微生物的潜在功能,导致各种疾病的发生。
综上所述,在抗生素处理后致肠道菌群严重失调的情况下,长链菊粉的补充反而延迟了肠道菌群多样性的恢复而且不利于肠道菌群的重建,使肠道菌群的组成与初始结构相反,甚至刺激某些条件致病菌在肠道内重新生长,提高了与肠道菌群相关的疾病途径进而延迟肠道炎症的恢复。然而,在肠道菌群严重失调的情况下,长链菊粉是如何刺激这些条件致病菌在肠道内的生长依然不清楚,需要进一步的研究。
References
| [1] | Kurilshikov A, Wijmenga C, Fu JY, Zhernakova A. Host genetics and gut microbiome:challenges and perspectives. Trends in Immunology, 2017, 38(9): 633-647. DOI:10.1016/j.it.2017.06.003 |
| [2] | Feng QQ, Chen WD, Wang YD. Gut microbiota:an integral moderator in health and disease. Frontiers in Microbiology, 2018, 9: 151. DOI:10.3389/fmicb.2018.00151 |
| [3] | Lichtman JS, Ferreyra JA, Ng KM, Smits SA, Sonnenburg JL, Elias JE. Host-microbiota interactions in the pathogenesis of antibiotic-associated diseases. Cell Reports, 2016, 14(5): 1049-1061. DOI:10.1016/j.celrep.2016.01.009 |
| [4] | Li J, Sung CYJ, Lee N, Ni YQ, Pihlajam?ki J, Panagiotou G, El-Nezami H. Probiotics modulated gut microbiota suppresses hepatocellular carcinoma growth in mice. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(9): E1306-E1315. DOI:10.1073/pnas.1518189113 |
| [5] | Chunchai T, Thunapong W, Yasom S, Wanchai K, Eaimworawuthikul S, Metzler G, Lungkaphin A, Pongchaidecha A, Sirilun S, Chaiyasut C, Pratchayasakul W, Thiennimitr P, Chattipakorn N, Chattipakorn SC. Decreased microglial activation through gut-brain axis by prebiotics, probiotics, or synbiotics effectively restored cognitive function in obese-insulin resistant rats. Journal of Neuroinflammation, 2018, 15: 11. DOI:10.1186/s12974-018-1055-2 |
| [6] | Suez J, Zmora N, Zilberman-Schapira G, Mor U, Dori-Bachash M, Bashiardes S, Zur M, Regev-Lehavi D, Ben-Zeev Brik R, Federici S, Horn M, Cohen Y, Moor AE, Zeevi D, Korem T, Kotler E, Harmelin A, Itzkovitz S, Maharshak N, Shibolet O, Pevsner-Fischer M, Shapiro H, Sharon I, Halpern Z, Segal E, Elinav E. Post-antibiotic gut mucosal microbiome reconstitution is impaired by probiotics and improved by autologous FMT. Cell, 2018, 174(6): 1406-1423. DOI:10.1016/j.cell.2018.08.047 |
| [7] | Singh V, Yeoh BS, Chassaing B, Xiao X, Saha P, Aguilera Olvera R, Lapek Jr JD, Zhang LM, Wang WB, Hao SJ, Flythe MD, Gonzalez DJ, Cani PD, Conejo-Garcia JR, Xiong N, Kennett MJ, Joe B, Patterson AD, Gewirtz AT, Vijay-Kumar M. Dysregulated microbial fermentation of soluble fiber induces cholestatic liver cancer. Cell, 2018, 175(3): 679-694. DOI:10.1016/j.cell.2018.09.004 |
| [8] | Bolger AM, Lohse M, Usadel B. Trimmomatic:a flexible trimmer for illumina sequence data. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [9] | Mago? T, Salzberg S L. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [10] | Edgar RC. UPARSE:highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [11] | Schloss PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, van Horn DJ, Weber CF. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [12] | Caporaso JG, Kuczynski J, Stombaugh J, Bittinger K, Bushman FD, Costello EK, Fierer N, Pe?a AG, Goodrich JK, Gordon JI, Huttley GA, Kelley ST, Knights D, Koenig JE, Ley RE, Lozupone CA, McDonald D, Muegge BD, Pirrung M, Reeder J, Sevinsky JR, Turnbaugh PJ, Walters WA, Widmann J, Yatsunenko T, Zaneveld J, Knight R. QIIME allows analysis of high-throughput community sequencing data. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [13] | Segata N, Izard J, Waldron L, Gevers D, Miropolsky L, Garrett WS, Huttenhower C. Metagenomic biomarker discovery and explanation. Genome Biology, 2011, 12(6): R60. DOI:10.1186/gb-2011-12-6-r60 |
| [14] | Langille MGI, Zaneveld J, Caporaso JG, McDonald D, Knights D, Reyes JA, Clemente JC, Burkepile DE, Vega Thurber RL, Knight R, Beiko RG, Huttenhower C. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nature Biotechnology, 2013, 31(9): 814-821. DOI:10.1038/nbt.2676 |
| [15] | Parks DH, Tyson GW, Hugenholtz P, Beiko RG. STAMP:statistical analysis of taxonomic and functional profiles. Bioinformatics, 2014, 30(21): 3123-3124. DOI:10.1093/bioinformatics/btu494 |
| [16] | Fernandes R, do Rosario VA, Mocellin MC, Kuntz MGF, Trindade EBSM. Effects of inulin-type fructans, galacto-oligosaccharides and related synbiotics on inflammatory markers in adult patients with overweight or obesity:a systematic review. Clinical Nutrition, 2017, 36(5): 1197-1206. DOI:10.1016/j.clnu.2016.10.003 |
| [17] | Roshanravan N, Mahdavi R, Alizadeh E, Jafarabadi MA, Hedayati M, Ghavami A, Alipour S, Alamdari NM, Barati M, Ostadrahimi A. Effect of butyrate and inulin supplementation on glycemic status, lipid profile and glucagon-like peptide 1 level in patients with type 2 diabetes:a randomized double-blind, placebo-controlled trial. Hormone and Metabolic Research, 2017, 49(11): 886-891. DOI:10.1055/s-0043-119089 |
| [18] | Liu F, Prabhakar M, Ju J, Long H, Zhou HW. Effect of inulin-type fructans on blood lipid profile and glucose level:a systematic review and meta-analysis of randomized controlled trials. European Journal of Clinical Nutrition, 2017, 71(1): 9-20. DOI:10.1038/ejcn.2016.156 |
| [19] | Ghouri YA, Richards DM, Rahimi EF, Krill JT, Jelinek KA, DuPont AW. Systematic review of randomized controlled trials of probiotics, prebiotics, and synbiotics in inflammatory bowel disease. Clinical and Experimental Gastroenterology, 2014, 7: 473-487. |
| [20] | Closa-Monasterolo R, Ferré N, Castillejo-DeVillasante G, Luque V, Gispert-Llaurado M, Zaragoza-Jordana M, Theis S, Escribano J. The use of inulin-type fructans improves stool consistency in constipated children. A randomised clinical trial:pilot study. International Journal of Food Sciences and Nutrition, 2017, 68(5): 587-594. DOI:10.1080/09637486.2016.1263605 |
| [21] | Matsuki T, Yahagi K, Mori H, Matsumoto H, Hara T, Tajima S, Ogawa E, Kodama H, Yamamoto K, Yamada T, Matsumoto S, Kurokawa K. A key genetic factor for fucosyllactose utilization affects infant gut microbiota development. Nature Communications, 2016, 7: 11939. DOI:10.1038/ncomms11939 |
| [22] | Blaser MJ. Antibiotic use and its consequences for the normal microbiome. Science, 2016, 352(6285): 544-545. DOI:10.1126/science.aad9358 |
| [23] | Griffin NW, Ahern PP, Cheng JY, Heath AC, Ilkayeva O, Newgard CB, Fontana L, Gordon JI. Prior dietary practices and connections to a human gut microbial metacommunity alter responses to diet interventions. Cell Host & Microbe, 2017, 21(1): 84-96. |
| [24] | Schroeder BO, Birchenough GMH, St?hlman M, Arike L, Johansson MEV, Hansson GC, B?ckhed F. Bifidobacteria or fiber protects against diet-induced microbiota-mediated colonic mucus deterioration. Cell Host & Microbe, 2018, 23(1): 27-40. |
| [25] | Scott KP, Martin JC, Duncan SH, Flint HJ. Prebiotic stimulation of human colonic butyrate-producing bacteria and bifidobacteria, in vitro. FEMS Microbiology Ecology, 2014, 87(1): 30-40. DOI:10.1111/1574-6941.12186 |
