温崇庆1, 何瑶瑶1, 薛明1

 , 梁华芳1, 董俊德2
, 梁华芳1, 董俊德21. 广东海洋大学水产学院, 广东湛江 524025;
2. 中国科学院热带海洋生物资源与生态重点实验室, 中国科学院南海海洋研究所, 广东广州 510301
收稿日期: 2015-04-08; 修回日期: 2015-04-08
资助课题: 国家自然科学基金(31372536);中国科学院热带海洋生物资源与生态重点实验室开放基金(LMB131009);广东海洋大学引进人才启动项目(E10046)
通讯作者: 薛明,E-mail:xuemtc@163.com
摘要: 【目的】通过高通量测序技术,评价不同DNA试剂盒提取引起的对虾肠道菌群结构偏差,了解健康凡纳滨对虾肠道菌群结构特征。【方法】分别以细菌、粪便和组织DNA试剂盒3次重复提取凡纳滨对虾肠道总DNA(分别编号为SIB, SIS和SIT),检测DNA含量、纯度及其16S rDNA V4区可扩增性,进一步采用Illumina MiSeq高通量测序比较SIB和SIS样品菌群组成和多样性。【结果】细菌试剂盒提取的虾肠总DNA效果最好,粪便试剂盒次之,而组织试剂盒所提DNA含量低且难以被扩增。从SIB和SIS样品分别获得52151±5085和55296±5147条有效序列,同一(46800条)测序深度下, SIS样品OTU(operationaltaxonomic unit)数量和Shannon多样性指数均显著高于SIB的,而SIB样品间OTU重复性则优于SIS样品间的。从SIB和SIS样品鉴定的优势门一致,均包括变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、浮霉菌门(Planctomycetes)、放线菌门(Actinobacteria)和蓝细菌门(Cyanobacteria),但不同分类水平上绝大多数优势菌群丰度在两种样品间差异明显。【结论】高通量测序分析表明对虾肠道菌群结构因DNA提取方法不同而呈现显著偏差;本研究健康凡纳滨对虾肠道核心菌群主要由发光杆菌属(Photobacterium),乳球菌属(Lactococcus),弧菌属(Vibrio), Aliivibrio和3个分类未定属构成。
关键词: DNA提取商品化试剂盒凡纳滨对虾肠道菌群高通量测序
Biases on community structure during DNA extraction of shrimp intestinal microbiota revealed by high-throughput sequencing
Chongqing Wen1, Yaoyao He1, Ming Xue1

 , Huafang Liang1, Junde Dong2
, Huafang Liang1, Junde Dong21. Fisheries College, Guangdong Ocean University, Zhanjiang 524025, Guangdong Province, China;
2. Key Laboratory of Tropical Marine Bio-resources and Ecology, South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, Guangdong Province, China
Abstract:[Objectives] High-throughput sequencing technology is increasingly applied in intestinal microbiota of aquatic animals including shrimp. However, there is a lack of standard method or kit for DNA isolation from shrimp intestinal microbiota, and little is known about the effectiveness and biases regarding DNA extraction based on high-throughput sequencing. The aim of this study was to study the biases of different DNA extraction kits on community structure of shrimp intestinal microbiota through high-throughput sequencing, and to better understand the structure and composition of bacterial flora associated with healthy Litopenaeus vannamei.[Methods] We extracted the total DNA of intestinal microbiota from L. vannamei with three commercial kits designed for DNA extraction from bacteria, stool and tissue(Omega, USA). DNA quality was evaluated based on the absorbance ratios of 260/280 nm by NanoDrop, while DNA concentration was quantified using PicoGreen. Then Illumina MiSeq high-throughput sequencing was used to examine the intestinal bacterial communities following PCR amplification of 16S rDNA V4 region.[Results] The yield and purity of the DNA from the Bacterial Kit(SIB) were superior to those from the Stool Kit(SIS), whereas the DNA from Tissue Kit(SIT) presented too small amount to be amplified efficiently. The average sequence reads obtained from SIB and SIS samples were 52151±5085 and 55296±5147 respectively. After resampling at the same depth of 46800 reads, the operational taxonomic unit(OTU) number and Shannon diversity index of SIS samples were significantly higher than those of SIB samples. By contrast, the reproducibility of OTU among SIB replicates was higher than that among SIS replicates. The dominant phyla of SIS and SIB samples were identical, including Proteobacteria, Firmicutes, Bacteroidetes, Planctomycetes, Actinobacteria, and Cyanobacteria. However, the relative abundances of almost all the dominant groups at various taxonomic levels differed greatly between these two samples.[Conclusion] Significant biases on community structure of shrimp intestinal microbiota were detected which originated from DNA extraction. And the core microbiota of the healthy L. vannamei in this study was mainly composed of genera Photobacterium, Lactococcus, Aliivibrio, Vibrio, as well as three other unclassified groups.
Key words: DNA extractioncommerical kitLitopenaeus vannameiintestinal microbiotahigh-throughput sequencing
凡纳滨对虾(Litopenaeus vannamei),又称南美白对虾,俗称白虾,目前是全球尤其是我国对虾养殖与消费的最主要品种。近年来凡纳滨对虾养殖过程中病害尤其是细菌性病害频发,严重影响其产量与质量[1]。肠道微生物在水产动物营养吸收、生长代谢和免疫抗病等方面发挥着重要作用,其结构组成与多样性是影响宿主健康的重要因素[2, 3, 4]。对对虾肠道菌群研究有助于了解宿主与菌群间相互作用关系,并可为对虾养殖用益生菌的开发与应用提供依据[5, 6, 7]。
二十一世纪之前对对虾肠道菌群研究主要限于可培养微生物,其后16S rRNA基因克隆文库测序和变性梯度凝胶电泳等常规分子生态学技术陆续用于虾肠菌群结构和多样性研究[3, 7, 8, 9, 10, 11],一定程度上克服了分离培养技术局限。但常规分子生态学技术也只能检测少数优势种群,难以深入认识菌群结构组成与多样性。最近几年高通量测序技术飞速进展,突破了这种局限。高通量测序技术是相对于第一代测序(Sanger法)技术,又称新一代或第二代测序技术,具有测序通量高、准确度高、性价比高等优点[12],可实现宏基因组水平上多重样品间的平行比较,能更灵敏探测包括稀少种群在内的微生物多样性,显著拓展了微生物生态学研究广度和深度[13],是当前和今后微生物多样性及群落结构研究中最强有力技术之一。最近,以Roche 454和Illumina MiSeq为代表的高通量测序平台也开始用于包括对虾在内的水产动物肠道菌群研究,如Rungrassamee等[5, 14]用454测序研究了斑节对虾(Penaeus monodon)野生与养殖亲本以及不同养殖阶段虾肠菌群组成。同样用454测序,Huang等[15]分析了凡纳滨对虾仔虾不同阶段肠道菌群结构变化。通过Illumina MiSeq测序,Zhang等[16]比较了不同脂肪源饲料对凡纳滨对虾肠道菌群结构组成影响,Xiong等[17]则分析了不同健康状态下凡纳滨对虾肠道菌群差异。
高通量测序技术的出现和应用,使得免培养微生物多样性研究中测序技术不再是瓶颈,而实验方法尤其是DNA提取作为第一步引起的潜在偏差更显突出[18]。动物肠道样品不仅含有微生物和宿主细胞,且肠道中食物残渣及其代谢物复杂多样,从中获得高质量有代表性的DNA极具挑战。目前,对肠道菌群DNA的提取主要采用各种商品化试剂盒,但不同试剂盒或方法获得的DNA可能对菌群结构和多样性分析产生显著差异,这在人肠道菌群研究中已有证实[19]。迄今,DNA提取引起的对虾肠道菌群结构偏差尚未见报道。本文在研究不同试剂盒对凡纳滨对虾肠道菌群总DNA提取效果基础上,进一步通过高通量测序比较DNA提取引起的菌群结构差异,为对虾肠道菌群多样性研究时DNA的提取提供参考和依据。同时,本研究将有助于认识健康凡纳滨对虾肠道菌群结构特征及其蕴含的潜在益生菌。
1 材料和方法1.1 材料1.1.1 对虾肠道样品:从广东湛江东海岛选择一个放养2个月的凡纳滨对虾海水虾塘,养殖期间无病害发生且采样前1个月内未使用过微生物制剂和抗生素等化学药物。从虾塘不同位点随机捕捞体表无损伤、活力好的对虾于冰中保存,带回实验室处理。共取45条虾[体长(7.6±0.6) cm,体重(8.2±1.2) g],无菌操作取出完整虾肠,用70%乙醇漂洗3次,-20 °C冻存备用。
1.1.2 主要试剂和仪器:EZNA Bacterial DNA Kit,Stool DNA Kit,Tissue DNA Kit (Omega,美国);AccuPrime Taq DNA Polymerase High Fidelity,Quant-iT PicoGreen dsDNA Assay Kit (Invitrogen,美国);QIAquick Gel Extraction Kit (Qiagen,美国);MiSeq Reagent Kit v2 (500-cycle),PhiX Control (Illumina,美国);Gene Amp PCR-System 9700 (Applied Biosystems,美国);GeneGenius凝胶成像系统(SynGene,英国);NanoDrop ND-1000 Spectrophotometer (NanoDrop,美国);FLUOstar Optima (BMG Labtech,德国);MiSeq Desktop Sequencer (Illumina,美国)。
1.2 对虾肠道总DNA提取和检测将虾肠加少量无菌水在冰浴上匀浆并定容至5mL,充分混匀后从中取9份300 μL匀桨液,6000×g离心5 min,去上清,再分别用细菌、粪便和组织DNA试剂盒按操作说明提取总DNA。每种试剂盒各做3个技术重复提取,最后以100 μL TE缓冲液洗脱DNA,-20 °C保存。以NanoDrop所测260 nm与280 nm吸光度比值(260/280)评估所提DNA纯度,采用FLUOstar Optima以PicoGreen法[20]检测DNA含量。为便于描述,分别以SIB、SIS和SIT代表细菌(Bacteria)、粪便(Stool)和组织(Tissue)试剂盒提取的虾肠(Shrimp Intestine)总DNA,另以1-3表示各试剂盒3个重复提取样。
1.3 16S rDNA序列扩增和MiSeq测序根据PicoGreen定量结果,将各样品DNA稀释至2 ng/μL作为模板DNA,通过细菌16S rDNA V4区引物扩增各样品。扩增用V4区引物是在常规引物515F (5′-GTGCCAGCMGCCGCGGTAA-3′)和806R (5′-GGACTACHVGGGTWTCTAAT-3′)上加有接头和测序引物序列,以适合MiSeq测序平台用,同时反向引物上另接有12个不同碱基的标签(barcode)序列以区分不同样品[21]。PCR反应体系为25 μL反应液含10×AccuPrime buffer II 2.5 μL,AccuPrime Taq DNA Polymerase 0.1 μL,正反向引物(10 μmol/L)各1 μL,模板DNA 5 μL (10 ng),补充超纯水至25 μL,每份DNA做3个重复扩增。PCR反应条件为94 °C 3 min; 94 °C 20 s,53 °C 25 s,68 °C 45 s,28个循环;68 °C 10 min。将3个重复PCR的阳性扩增产物混合为一个样品,PicoGreen进行定量。各样品PCR产物以等摩尔量混合后通过QIAquick试剂盒切胶回收目的片段,进一步定量后与其它样品以及PhiX Control一同构成测序样品文库,在美国俄克拉荷马大学环境基因组研究所由Illumina MiSeq测序仪采用MiSeq Reagent Kit v2 (500-cycle)完成测序。
1.4 序列处理和分析对测序原始数据先通过BLAST程序[22]去除PhiX序列并通过barcode分配样品reads,由Btrim程序[23]去除低质量(Window size 5 bp时Quality score< 20)正反向reads,再运用FLASH程序[24]以≥50 bp重叠且错误率<5%拼接正反向序列,拼接后得到序列长度(253±10) bp的Fastq序列文件,最后由UPARSE程序[25]在97%相似性水平上生成去除singleton的OTU表及对应序列文件。
通过Mouther程序[26]对OTU表再抽样(subsample)使各样品获得一致序列数,做出基于OTU的稀释曲线(Rarefaction curve),计算Chao1值和Shannon多样性指数。分别以Jaccard、Sørensen 和Bray-Curtis相似性指数比较同一和不同试剂盒所提DNA样品间OTU的β多样性。对OTU覆盖度(Coverage,C)按公式C=1-n1/N计算,其中N代表某样品获得的有效序列数,n1表示在该样品中仅有一条序列的OTU数。通过CANOCO 4.5运用除趋势对应分析(Detrended correspondence analysis,DCA)比较样品间细菌群落结构组成差异。
以序列丰度≥0.5%定义优势门、科、属和OTU,并对优势OTU序列,通过EzTaxon (http://www.ezbiocloud.net/eztaxon)调出相似性最高的模式菌株序列作为参考序列,若与模式菌株序列相似性小于97%,则通过GenBank (http://www.ncbi.nlm.nih.gov/)进行同源性比对,获取相似性最高的代表序列作为参考序列。将优势OTU序列及其参考序列通过MEGA 5软件[27]以ClustalW进行序列比对,以Kimura-2参数模型估算系统进化矩阵,采用邻接法(Neighbor-joining)构建优势OTU序列系统进化树,Bootstrap 1000次评估进化树的拓扑稳定性。
1.5 统计分析数据以平均值±标准偏差表示,通过Excel独立样本t检验比较成对样本间差异显著性。运用SPSS 15.0统计软件进行数据的单因素方差分析(ANOVA),当差异达显著性水平时,以Duncan’s多重比较进行不同处理间的显著性分析,检验标准为P<0.05或P<0.01。
2 结果和分析2.1 不同试剂盒提取虾肠总DNA比较3种试剂盒所提虾肠总DNA纯度与产量如表1所示。单从OD260/OD280比值上看,细菌和组织试剂盒所提DNA纯度都很高,而粪便试剂盒所提DNA OD260/OD280值低于理想值(1.9)。从DNA产量上看,细菌试剂盒所提DNA (SIB)含量(PicoGreen检测)分别是SIS和SIT含量的10倍和25倍。通过16S rDNA V4引物对各样品PCR扩增后取5 μL电泳检测显示,从SIB和SIS模板都很容易扩增出目的条带,而SIT的3个重复样都不能被扩增(图1)。这表明,从DNA纯度、产量和可扩增性上看,细菌试剂盒提取效果最好,尽管粪便试剂盒所提DNA可能有少量蛋白污染,但并不影响其扩增效果,而组织试剂盒所提DNA却不能被扩增,因此只对SIB和SIS扩增产物做高通量测序分析。
表1 3种试剂盒提取对虾肠道DNA的产量和纯度Table 1 Yield and purity of shrimp intestinal DNA extracted by three kinds of DNA kits
| Sample/DNA Kit | OD260/OD280 | DNA concentration/(ng/μL) | |
| NanoDrop | PicoGreen | ||
| SIB/Bacteria | 1.90±0.03a | 175.61±8.66a | 115.66±6.15a |
| SIS/Stool | 1.67±0.02b | 28.39±8.90b | 11.36±2.92b |
| SIT/Tissue | 1.93±0.12a | 40.94±8.51b | 4.56±1.10b |
| Means with different superscript letters in the same column are significantly different (P < 0.05). | |||
表选项
 |
| 图1 16S rDNA V4区引物对虾肠DNA的PCR扩增 Figure 1 PCR amplification of 16S rDNA V4 region with DNA templates extracted from shrimp intestines by various DNA kits. M: DNA marker; lane 1: Negative control; lane 2-4: SIB1-SIB3; 5-7: SIS1-SIS3; lane 8-10: SIT1-SIT3. SIB1-SIB3, SIS1-SIS3, and SIT1-SIT3 indicated DNA samples extracted in triplicate from shrimp intestines by Bacterial, Stool, and Tissue DNA Kits, respectively. |
| 图选项 |
2.2 MiSeq测序概况及虾肠菌群多样性FLASH程序拼接正反向序列后,SIB1-SIB3和SIS1-SIS3 6个样品共获得352279条有效序列。通过UPRASE生成去除singleton的OTU表共有322339条序列,SIB和SIS样品平均序列数分别为52151±5085与55296±5147,其中序列数最多和最少样品分别是SIS1(60100)和SIB3(46834)。对OTU表再抽样,使每个样品获得一致序列数(46800),同样测序深度下SIB和SIS样品都获得约99.7%的高覆盖度,且各样品OTU稀释曲线趋于饱和(图2),表明46800条的测序深度能比较全面反应本研究虾肠菌群多样性。
 |
| 图2 虾肠菌群OTU稀释曲线 Figure 2 Rarefaction curves for the OTU numbers of shrimp intestinal microbiota. SIB1-SIB3 and SIS1-SIS3 indicated DNA samples extracted in triplicate from shrimp intestines by Bacterial and Stool DNA Kits, respectively. |
| 图选项 |
从门到属的水平上,从SIS样品检测的已知分类单元数均高于SIB的,但两者间无显著差异(表2)。而OTU水平上,SIS样品OTU数量和Shannon多样性指数均显著高于SIB,且Chao1所预测SIS的OTU数也高于SIB,可见粪便试剂盒较细菌试剂盒所提虾肠DNA经MiSeq测序后检测的菌群多样性更高。
表2 MiSeq高通量测序分析虾肠菌群多样性Table 2 Analysis of diversity of shrimp intestinal microbiota by MiSeq high-throughput sequencing
| Sample/DNA Kit | Number | OTU | ||||||||
| Phylum | Class | Order | Family | Genus | OTU | Coverage/% | Chao1 | Shannon index | ||
| SIB/Bacteria | 12±1 | 22±2 | 42±3 | 97±3 | 184±1 | 462±8 | 99.69±0.02 | 589±15 | 1.52±0.02 | |
| SIS/Stool | 15±2 | 27±4 | 47±3 | 104±5 | 197±12 | 542±47* | 99.72±0.02 | 641±51* | 2.01±0.12* | |
| * indicates the means in the same column are significantly different (P<0.05). | ||||||||||
表选项
2.3 虾肠菌群结构组成分析对6个样品再抽样后的OTU表进行除趋势对应分析(DCA)结果如图3所示,SIB和SIS的3个技术重复样都各自聚为一类,且两者菌群间adonis Jaccard和adonis Bray差异都达极显著(P<0.01),这表明细菌和粪便试剂盒提取的虾肠DNA经高通量测序后菌群结构组成差异显著。另一方面,SIB 3个重复样紧密聚集在一起,而SIS重复样虽在横轴上相近但在纵轴上比较分散,说明SIB重复样间菌群结构组成更相似即重复性相对SIS的更高。各样品间β多样性比较显示,SIB重复样品间3个群落结构相似性指数均高于SIS (表3),尤其是定量序列丰度的Bray-Curtis指数差异显著,进一步证实SIB样品重复性相对更高。此外,SIB或SIS重复间相似性指数都显著高于SIB和SIS间的相似性(表3),说明不同试剂盒引起的菌群结构差异明显高于同一试剂盒技术重复间产生的波动。
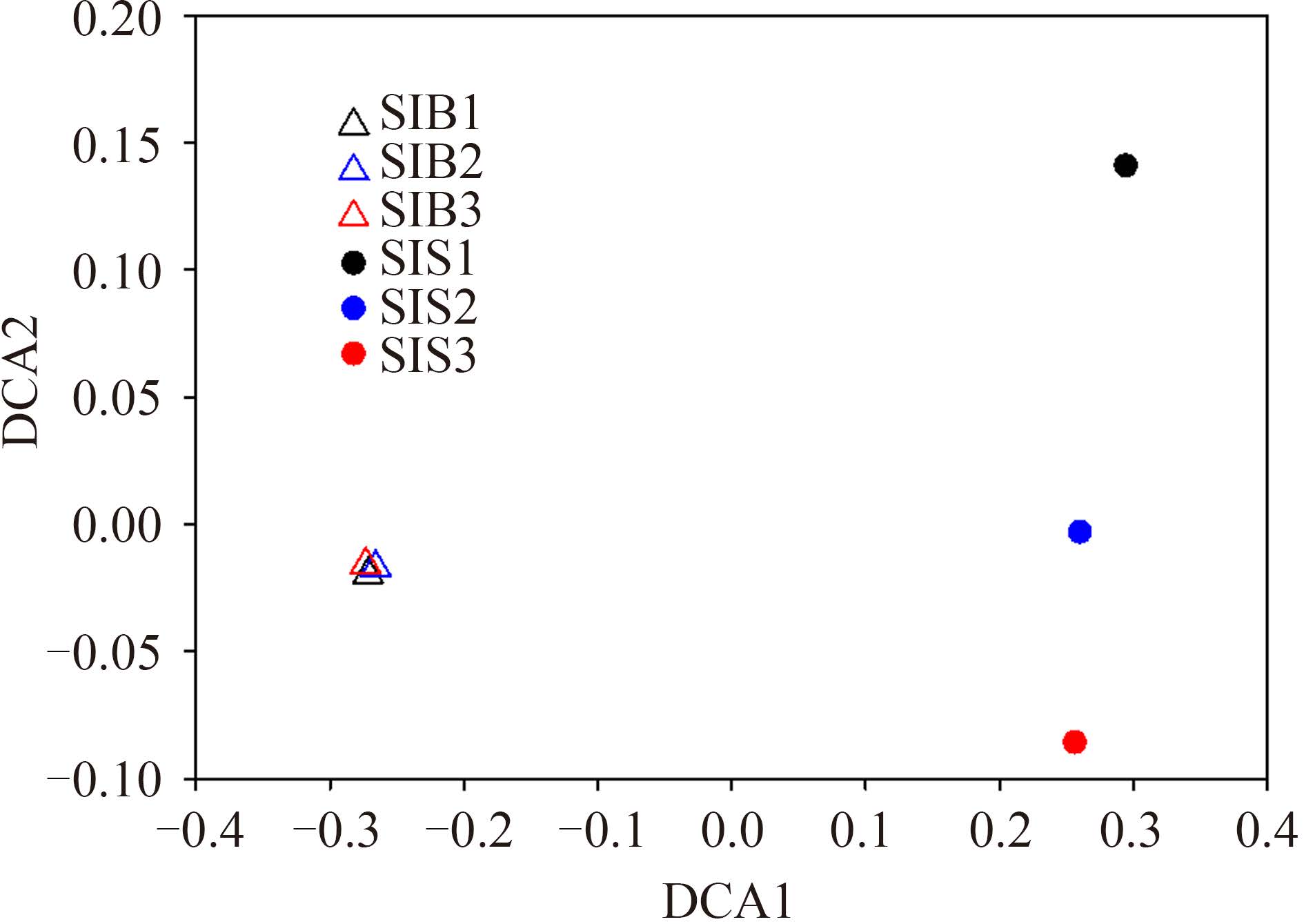 |
| 图3 除趋势对应分析虾肠菌群结构 Figure 3 Detrended correspondence analysis (DCA) of community structure of shrimp intestinal microbiota. SIB1-SIB3 and SIS1-SIS3 indicated DNA samples extracted in triplicate from shrimp intestines by Bacterial and Stool DNA Kits, respectively. |
| 图选项 |
表3 SIB和SIS样品间及其重复样品间菌群结构β多样性比较Table 3 β-diversity comparisons among replicates (SIB or SIS) and between treatments (SIB and SIS) based on OTU table
| Level | Data size* | Similarity index | ||
| Jaccard | S?rensen | Bray-Curtis | ||
| SIB | 3 | 0.5830±0.0266a | 0.7360±0.0211a | 0.9767±0.0029a |
| SIS | 3 | 0.5527±0.0272a | 0.7117±0.0229a | 0.9537±0.0134b |
| SIB and SIS | 9 | 0.5119±0.0246b | 0.6770±0.0215b | 0.8096±0.0153c |
| *Data sizes are the number of data points of the pairwise comparisons within the replicates or treatments; Means with different superscript letters in the same column are significantly different (P<0.05). | ||||
表选项
SIB和SIS样品分别共检测到19和13个已知门,各样品优势门序列丰度见图4。其中变形菌门(Proteobacteria)在所有样品中均以绝对优势(>80%)存在。变形菌中又以γ-变形菌纲最优势,其次为α-变形菌纲,两者分别占SIB的78.35%和3.00%,分别占SIS的75.52%和5.14%。SIB各样品第2到第6优势门分别是厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、浮霉菌门(Planctomycetes)和蓝细菌门(Cyanobacteria)。SIS优势门次序与SIB的总体相似,只是浮霉菌门在SIS中比放线菌门(Actinobacteria)更优势。以上6个优势门中,除变形菌门和蓝细菌门在SIB和SIS中丰度差异不显著(P>0.05)外,其余4个门丰度在两种样品间都呈极显著差异(P<0.01),即厚壁菌门、拟杆菌门和浮霉菌门在SIS中以及放线菌门在SIB中都显著更高。此外,除总序列数占极少的非优势已知门外,SIB和SIS中分别尚有12.36%和1.94%的序列属于细菌域中Unclassified门。以上表明,细菌和粪便试剂盒所提虾肠菌群DNA经PCR扩增和MiSeq测序后所得优势门是一致的,但两者对大多数优势门以及Unclassified门的丰度检测结果差异显著。
 |
| 图4 虾肠菌群优势门相对丰度 Figure 4 Relative abundance (%) of dominant bacterial phyla in shrimp intestinal microbiota. SIB1-SIB3 and SIS1-SIS3 indicated DNA samples extracted in triplicate from shrimp intestines by Bacterial and Stool DNA Kits, respectively. |
| 图选项 |
从SIB和SIS获得序列丰度≥0.5%的优势科、属和OTU结果如表4所示。SIS有11个优势科,其中有7个与SIB优势科共享,另4个只在SIS中为优势;而SIB只有8个优势科,其中仅肉杆菌科(Carnobacteriaceae)为SIB独有优势科。弧菌科(Vibrionaceae)以显著优势存在于所有样品,并由发光杆菌属(Photobacterium)、弧菌属(Vibrio)和Aliivibrio 3个优势属构成,其中几乎完全由OTU1序列构成的发光杆菌属为绝对优势型。链球菌科(Streptococcaceae)在SIS中是仅次于弧菌科的第二优势科,但在SIB中平均丰度只有0.02%,其序列主要由属于乳球菌属(Lactococcus)的OTU3和OTU123组成,两者均为SIS的优势OTU。SIB的第二优势科完全由Unclassified的OTU2构成,但其在SIS中仅排第五位。SIB和SIS第三和第四优势科都分别为红细菌科(Rhodobacteraceae)和由OTU5构成的Unclassified科。从SIB和SIS中共鉴定12个优势科,除OTU5和OTU6这两个Unclassified科在SIB和SIS中丰度相当,以及弧菌科在SIB中的丰度显著高于其在SIS中的外,其余9个科的丰度在两类样品间都呈极显著差异。相似地,绝大多数优势属或优势OTU的丰度在SIB和SIS间也呈显著或极显著差异。以上表明两种试剂盒对虾肠绝大多数优势菌群DNA提取效率,无论在门、科、属还是OTU水平上都有明显差异,各有其偏好。另一方面,若以占两种样品平均丰度≥1%的属作为核心菌群,本研究虾肠核心菌群按丰度大小依次由发光杆菌属、OTU2 (Unclassified)、乳球菌属、Aliivibrio、弧菌属、OTU5 (Unclassified)和OTU6 (Unclassified)组成。
表4 SIB和SIS样品间优势科、属和OTU丰度比较Table 4 Abundance comparisons of dominant Families, Genera and OTUs between SIB and SIS samples
| Dominant Family | Dominant Genus | Dominant OTU | ||||||||
| Family | SIB/% | SIS/% | Genus | SIB/% | SIS/% | OTU | SIB/% | SIS/% | ||
| Vibrionaceae | 75.38±0.45 | 70.16±2.00* | Photobacterium | 70.81±0.53 | 65.70±2.04* | OTU1 | 70.79±0.53 | 65.67±2.05* | ||
| Aliivibrio | 2.65±0.06 | 2.20±0.04** | OTU4 | 2.65±0.06 | 2.20±0.04** | |||||
| Vibrio | 1.93±0.05 | 2.27±0.09** | OTU7 | 1.42±0.07 | 1.60±0.05* | |||||
| OTU60 | 0.51±0.04 | 0.67±0.05 | ||||||||
| Unclassified | 11.78±0.16 | 1.81±0.28** | Unclassified | 11.78±0.16 | 1.81±0.28** | OTU2 | 11.78±0.16 | 1.81±0.28 | ||
| Rhodobacteraceae | 2.72±0.06 | 4.71±0.60** | Paracoccus | 0.50±0.03 | 0.30±0.05** | NA | NA | NA | ||
| Marivita | 0.27±0.02 | 0.55±0.14* | OTU115 | 0.27±0.02 | 0.55±0.14* | |||||
| Unclassified | 1.83±0.11 | 1.83±0.03 | Unclassified | 1.83±0.11 | 1.83±0.03 | OTU5 | 1.83±0.11 | 1.83±0.03 | ||
| Unclassified | 1.13±0.05 | 1.14±0.06 | Unclassified | 1.13±0.05 | 1.14±0.06 | OTU6 | 1.13±0.05 | 1.14±0.06 | ||
| Flavobacteriaceae | 0.92±0.08 | 1.61±0.15** | NA | NA | NA | NA | NA | NA | ||
| Carnobacteriaceae | 0.70±0.05 | 0.14±0.03** | Carnobacterium | 0.70±0.05 | 0.14±0.03** | OTU8 | 0.61±0.07 | 0.07±0.03** | ||
| Planctomycetaceae | 0.51±0.03 | 1.39±0.05** | NA | NA | NA | NA | NA | NA | ||
| Alteromonadaceae | 0.35±0.04 | 0.56±0.02** | Haliea | 0.33±0.04 | 0.54±0.01** | NA | NA | NA | ||
| Moraxellaceae | 0.27±0.01 | 1.70±0.14** | Psychrobacter | 0.24±0.01 | 0.90±0.10** | OTU19 | 0.02±0.01 | 0.50±0.08** | ||
| Streptococcaceae | 0.02±0.00 | 9.47±0.36** | Lactococcus | 0.01±0.00 | 9.19±0.32** | OTU3 | 0.01±0.00 | 8.29±0.25** | ||
| OTU123 | <0.01 | 0.90±0.07** | ||||||||
| Leuconostocaceae | <0.01 | 0.61±0.07** | Leuconostoc | <0.01 | 0.61±0.07** | OTU9 | <0.01 | 0.50±0.06** | ||
| *, ** indicate the means in the same row from the same taxon are significantly (P<0.05) and very significantly (P<0.01) different, respectively; NA: not available, as the abundance of any single Genus or OTU is below 0.5%. | ||||||||||
表选项
2.4 优势OTU序列系统进化分析如表4所示,SIB和SIS样品总共有13个优势OTU,其序列在GenBank中的收录号为KP765749- KP765761。EzTaxon序列比对分析进一步确证了其中10个OTU在属水平上的分类,它们与其最相近模式菌株的序列相似性均为100%或99.6%;另3个OTU如前所述,为Unclassified科(OTU5,OTU6)甚至Unclassified门(OTU2),与其最相近模式菌株序列同源性都低于94%,而与GenBank中未培养细菌克隆序列相似性100%。优势OTU的 16S rDNA序列系统发育树如图5所示。除OTU2属Unclassified门外,有7个OTU属于变形菌门,另5个归厚壁菌门。变形菌门中除OTU115属于α-变形菌纲Marivita属外,其余均为γ-变形菌纲成员,分别隶属于发光杆菌属(OTU1)、弧菌属(OTU7,OTU60)、Aliivibrio (OTU4)和嗜冷杆菌属(Psychrobacter)(OTU19)以及一个Unclassified科(OTU5)。厚壁菌门中除OTU6为梭菌目(Clostridiales)Unclassified科外,其它4个OTU分别属于乳球菌属(OTU3,OTU123)、明串珠菌属(Leuconostoc) (OTU9)和肉杆菌属(Carnobacterium) (OTU8)。
 |
| 图5 虾肠菌群优势OTU序列系统进化树 Figure 5 Phylogenetic tree of the dominant OTUs sequences of shrimp intestinal microbiota by using Neighbor-joining method. Numbers at branch points indicate bootstrap values above 80%; the sequences accession number in GenBank were showen in parentheses. The scale bar corresponds to 1% sequence divergence; the dominant OTUs from this study are indicated by filled circles. |
| 图选项 |
3 讨论目前对对虾肠道菌群DNA的提取尚无统一标准,也未见有专门针对虾肠菌群DNA提取的试剂盒,而是沿用哺乳动物肠道或微生物或土壤等DNA提取用商品化试剂盒[3, 5, 8, 9, 10, 11, 14, 16, 17, 28],或采用人工CTAB法[7, 15, 28]。无论哪种试剂盒或方法,其DNA提取都包括细胞裂解和核酸纯化两个步骤,对肠道微生物DNA的提取,关键是如何有效裂解细胞[28]。通常采用物理、化学或酶解作用裂解微生物细胞,使DNA得以释放,再进一步纯化。本研究3种试剂盒所提虾肠DNA产量间差异明显(表1),除试剂盒本身针对对象不同而提取步骤和提取液有所差异外,很可能与它们裂解细胞方式有关。细菌试剂盒所提虾肠DNA得率高质量好,除与细菌占肠道生物量高有关外,与细菌试剂盒既使用溶菌酶也使用玻璃粉破裂细胞有关;粪便试剂盒通过玻璃粉结合温度差(90 °C)裂解细胞,其DNA (SIS)产量虽然只有SIB的1/10但却是SIT的2.5倍;而组织试剂盒则完全依赖其提取液裂解细胞,DNA产量最少。
对以PCR扩增为基础的微生物多样性研究,因扩增所需模板DNA量很少(10-100 ng),样品DNA产量通常并不影响下游操作,而DNA质量尤其是扩增效率则是关键。3种试剂盒所提DNA中,SIB和SIS模板都很容易被扩增,即使SIS中可能含有少量蛋白污染,也不影响其扩增。相反,尽管NanoDrop检测SIT的OD260/OD280显示其纯度很高,极少有蛋白污染,但其模板却不能被扩增,即使增加或减少模板量,或者增加PCR循环数也不见效(结果未显示)。本研究NanoDrop检测的SIB、SIS和SIT含量分别约是PicoGreen定量结果的1.5、2.5和9倍(表1)。NanoDrop对DNA含量的检测是建立在260 nm吸收峰基础上,包含了相同或相近吸收峰的非DNA成分,准确度不高,而PicoGreen结果则代表真正双链DNA含量[20]。可见,SIT中含有大量未知成分,很可能因此抑制了PCR扩增。因此,从DNA产量、纯度和可扩增性上看,细菌试剂盒优于粪便试剂盒,而组织试剂盒则不适合虾肠菌群DNA提取。
对免培养微生物多样性研究而言,更重要的是DNA经扩增测序后能多大程度反映实际菌群多样性。高通量测序技术具有测序深度高和样品间平行比较性好等优点,能更真实反映不同样品间菌群结构差异[13]。本研究采用MiSeq高通量测序,表明两种试剂盒所提虾肠DNA反映的菌群结构组成差异显著,其中粪便试剂盒较细菌试剂盒能更多反映菌群多样性,但细菌试剂盒的重复性更好。Zhou等[13]以土壤样品为对象评估454测序16S rDNA扩增测序重复性,发现OTU水平上两个技术重复样品间Jaccard平均相似性只有26.6%,而本研究则显示这种相似性在SIB和SIS技术重复间分别高达58.3%和55.3%。这种重复性显著提高,除与土壤和虾肠样品细菌多样性不同有关外,很大程度得益于本研究测序深度显著提高,OTU覆盖度接近饱和,而Zhou等[13]平均每个样品有效序列数不到2000,OTU覆盖度不足10%。可见测序深度的提高显著增加了扩增测序重复性。
两种试剂盒所提DNA分析得到的虾肠菌群结构组成差异显著,这在优势菌群序列丰度上更易体现。在优势门水平上,厚壁菌门、拟杆菌门和浮霉菌门在SIS中丰度都显著高于它们在SIB中的,而放线菌门和Unclassified门在SIB中丰度则显著高于其在SIS中的。同样,绝大多数情况下,同一科、属或OTU丰度在SIB和SIS样品间也呈显著差异(表4)。特别是,厚壁菌门中乳球菌和明串珠菌分别占SIS总序列的9.19%和0.61%,但在SIB中比例都不超过0.01%,差异极其明显。相似地,单个OTU2 (Unclassified)序列占SIB总序列高达11.78%,但在SIS中只有1.81%。序列丰度差异很大程度上代表着相应菌群的DNA被提取效率,与试剂盒采用的提取策略和微生物细胞壁结构成分等有关,不同试剂盒间可能产生明显差异,由此造成PCR扩增不同菌群DNA效率及后续高通量测序检测上的偏差,因此在肠道菌群多样性研究中选用何种试剂盒或采用哪种DNA提取方式非常关键。理想情况下,采用一种试剂盒或方法若能提取肠道所有代表菌群DNA最为简便高效;其次是对不同方法比较后选用一种最合理方法或进一步加以优化,或者同时采用两种或两种以上方法提取DNA并分别加以研究再做综合分析。由于虾肠菌群实际组成并不知晓,本研究两种试剂盒又各有所长,尚难判断哪种方法更可靠。因此,今后对对虾肠菌群多样性研究时,可考虑同时选用这两种试剂盒,或者选用其中一种试剂盒并对其提取程序加以优化。如细菌试剂盒所提DNA (SIB)产量高质量好,测序后菌群结构重演性高且对诸如OTU2这样的Unclassified优势菌更有效,但其多样性相对SIS的少,说明其对某些菌群尤其是厚壁菌门中由乳球菌和明串珠菌为主的乳酸菌类DNA提取不足,因此对这类菌细胞裂解效率进行研究和优化将有助于提高细菌试剂盒对虾肠菌群DNA提取的适用性。
李可等[7]通过克隆文库测序研究显示凡纳滨对虾肠道菌群最优势门为厚壁菌门,其次为变形菌门,但因其检测克隆子数有限,只鉴定到这两个门。Huang等[15]以454测序研究发现养殖2个月的凡纳滨对虾肠道菌群中拟杆菌门最为优势,其次分别为变形菌门、Unclassified门和放线菌门,而厚壁菌门含量很少。对养殖时间同样约是2个月左右的凡纳滨对虾,本研究与Zhang等[16]运用MiSeq测序,平均每个样测序深度分别约是Huang等[15]454样品的20倍和10倍,在门水平上检测到的分类单元数显著增加,分别达19和18个已知门,且最优势者均为变形菌门。本研究第二优势门是厚壁菌门,尤其在SIS中丰度高达12%以上,而在Huang等[15]和Zhang等[16]的报道中厚壁菌门丰度都很低。除与所研究对虾来源和养殖条件等差异有关外,如前所述,DNA提取方法不同很可能影响厚壁菌门检出率。
因两种试剂盒所提DNA测序后菌群结构差异明显,本研究综合SIB和SIS结果,以占两者平均丰度≥1%的属作为虾肠核心菌群,按丰度大小依次由发光杆菌属、OTU2 (Unclassified)、乳球菌属、Aliivibrio、弧菌属、OTU5(Unclassified)和OTU6(Unclassified)组成。发光杆菌属、Aliivibrio和弧菌属均属于弧菌科,其中发光杆菌属和/或弧菌属也普遍存在于对虾肠道中[3, 5, 7, 8, 9, 10, 11, 14, 15]。值得注意的是,属于发光杆菌属的OTU1占SIB和SIS丰度分别高达70.79%和65.67%,并与美人鱼发光杆菌(P. damselae)两个亚种模式菌株序列相似性达100%。美人鱼发光杆菌一方面可能会引起海产动物病害,另一方面也是健康对虾正常群菌并可作为对虾益生菌,与该菌可能存在不同基因型有关[29]。考虑到OTU1是本研究健康对虾肠道中绝对优势OTU,因此美人鱼发光杆菌很可能在其中发挥有益作用。单个OTU4构成本研究Aliivibrio,其下有4个模式菌株与OTU4序列完全一致(图5)。Aliivibrio是从弧菌属中独立出来的,其成员如A. fischeri等经常与海洋动物互利共生,有的种也可能致病[30]。弧菌属由OTU7和OTU60构成,两者分别与5种和1种弧菌模式菌株序列相似性达99.6% (图5)。尽管弧菌是引起对虾细菌病害的最常见病原,但与OTU7和OTU60最相近的这6种弧菌尚未见它们引起对虾病害的报道。OTU2属于细菌域中Unclassified门,GenBank中与其最相似序列几乎都源自健康斑节对虾肠道未培养细菌(收录号如JN399968等)或中华绒鳌蟹肠道未培养细菌(如HG792233等),可见它们很可能是水产甲壳动物肠道正常菌群。乳球菌属由OTU3和OTU123构成,前者与植物乳球菌(L. plantarum)和L. piscium 序列完全一致,后者与乳酸乳球菌(L. lactis)和台湾乳球菌(L. taiwanensis)序列99.6%相似,这些乳酸菌对水产和陆生动物均未见有致病性,其中有的种如乳酸乳球菌可用作水产动物益生菌[6],可见乳球菌在本研究虾肠中应发挥有益作用。OTU5属于γ-变形菌纲假交替单胞菌目,与来自海洋沉积物2个未培养细菌序列(KF268912和KF268884)最相似,其对水产动物的影响尚不清楚。OTU6是厚壁菌门梭菌目中Unclassified科,与OTU2相似,与其最相似序列几乎都来自健康斑节对虾肠道未培养细菌(如JN399977等)或中华绒鳌蟹肠道未培养细菌(如HG792238等)。上述核心菌群总丰度分别占SIB和SIS的90.12%和84.14%,对维持凡纳滨对虾健康生长可能起重要作用。
本研究表明不同试剂盒对对虾肠道菌群DNA的提取效率、DNA质量和可扩增性差异明显,细菌和粪便试剂盒所提DNA经高通量测序分析得到的虾肠菌群结构组成差异显著。此外,本研究揭示健康凡纳滨对虾肠道核心菌群主要包括发光杆菌属、乳球菌属、弧菌属、Aliivibrio和3个分类未定属构成。本文结果为对虾肠道菌群多样性研究中DNA提取策略提供了一定参考和依据,同时有助于认识健康凡纳滨对虾肠道菌群结构特征,为对虾肠道有益菌的开发和应用提供参考。
致谢
感谢美国俄克拉荷马大学环境基因组研究所周集中教授为本研究提供的技术平台,吴力游教授和秦愈佳博士分别对本研究测序实验和测序数据处理提供了帮助。
参考文献
| [1] | FAO. Report of the FAO/MARD technical workshop on early mortality syndrome(EMS) or acute hepatopancreatic necrosis syndrome(AHPNS) of cultured shrimp(under TCP/VIE/3304). Hanoi, Viet Nam, 25-27 June 2013. FAO Fisheries and Aquaculture Report. No. 1053. Rome:54. |
| [2] | Harris JM. The presence, nature, and role of gut microflora in aquatic invertebrates:a synthesis. Microbial Ecology. 1993, 25(3):195-231. |
| [3] | Chaiyapechara S, Rungrassamee W, Suriyachay I, Kuncharin Y, Klanchui A, Karoonuthaisiri N, Jiravanichpaisal P. Bacterial community associated with the intestinal tract of P. monodon in commercial farms. Microbial Ecology, 2012, 63(4):938-953. |
| [4] | De Schryver P, Vadstein O. Ecological theory as a foundation to control pathogenic invasion in aquaculture. ISME Journal, 2014, 8(12), 2360-2368. |
| [5] | Rungrassamee W, Klanchui A, Maibunkaew S, Chaiyapechara S, Jiravanichpaisal P, Karoonuthaisiri N. Characterization of intestinal bacteria in wild and domesticated adult black tiger shrimp(Penaeus monodon). PLoS One, 2014, 9(3):e91853. |
| [6] | Newaj-Fyzul A, Al-Harbi AH, Austin B. Review, developments in the use of probiotics for disease control in aquaculture. Aquaculture, 2014, 431:1-11. |
| [7] | Li K, Zheng TL, Tian Y, Yuan JJ. Bacterial community structure in intestine of the white shrimp, Litopenaeus vannamei. Acta Microbiologica Sinica, 2007, 47(4):649-653.(in Chinese) 李可, 郑天凌, 田蕴, 袁建军. 南美白对虾肠道微生物群落的分子分析. 微生物学报, 2007, 47(4):649-653. |
| [8] | Johnson CN, Barnes S, Ogle J, Grimes DJ, Chang YJ, Peacock AD, Kline L. Microbial community analysis of water, foregut, and hindgut during growth of pacific white shrimp, Litopenaeus vannamei, in closed-system aquaculture. Journal of the World Aquaculture Society, 2008, 39(2):251-258. |
| [9] | Liu HD, Wang L, Liu M, Wang BJ, Jiang KY, Ma SS, Li QF. The intestinal microbial diversity in Chinese shrimp(Fenneropenaeus chinensis) as determined by PCR-DGGE and clone library analyses. Aquaculture, 2011, 317(1-4):32-36. |
| [10] | Li YH, Chai PC, Hu XG, Sun Y, Huang J, Song XL. Analysis of intestinal microecology of Litopenaeus vannamei in industrial aquaculture by RFLP and DGGE techniques. Progress in Fishery Sciences, 2014, 35(2):83-89.(in Chinese) 李玉宏, 柴鹏程, 胡修贵, 孙艳, 黄倢, 宋晓玲. 应用RFLP和DGGE技术分析工厂化养殖凡纳滨对虾肠道微生物群落特征. 渔业科学进展, 2014, 35(2):83-89. |
| [11] | Wang CZ, Ling GR, Yan T, Zheng ZP, Ou QF, Chen B, Sun FL. Microbial community in the shrimp(Penaeus penicillatus) intestine and its culture environment. Journal of Fisheries of China, 2014, 38(5):706-712.(in Chinese) 王春忠, 林国荣, 严涛, 郑志鹏, 欧清峰, 陈斌, 孙富林. 长毛对虾海水养殖环境以及虾肠道微生物群落结构研究. 水产学报, 2014, 38(5):706-712. |
| [12] | Qin N, Li DF, Yang RF. Next-generation sequencing technologies and the application in microbiology-a review. Acta Microbiologica Sinica, 2011, 51(4):445-457.(in Chinese) 秦楠, 栗东芳, 杨瑞馥. 高通量测序技术及其在微生物学研究中的应用. 微生物学报, 2011, 51(4):445-457. |
| [13] | Zhou JZ, Wu LY, Deng Y, Zhi XY, Jiang YH, Tu QC, Xie JP, Van Nostrand JD, He ZL, Yang YF. Reproducibility and quantitation of amplicon sequencing-based detection. ISME Journal,. 2011, 5(8):1303-1313. |
| [14] | Rungrassamee W, Klanchui A, Chaiyapechara S, Maibunkaew S, Tangphatsornruang S, Jiravanichpaisal P, Karoonuthaisiri N. Bacterial population in intestines of the black tiger shrimp(Penaeus monodon) under different growth stages. PLoS ONE. 2013, 8(4):e60802. |
| [15] | Huang ZB, Li XY, Wang LP, Shao ZZ. Changes in the intestinal bacterial community during the growth of white shrimp, Litopenaeus vannamei. Aquaculture Research, doi:10.1111/are.12628. |
| [16] | Zhang ML, Sun YH, Chen K, Yu N, Zhou ZG, Chen LQ, Du ZY, Li EC. Characterization of the intestinal microbiota in Pacific white shrimp, Litopenaeus vannamei, fed diets with different lipid sources. Aquaculture. 2014, 434:449-455. |
| [17] | Xiong JB, Wang K, Wu JF, Qiuqian LL, Yang KJ, Qian YX, Zhang DM. Changes in intestinal bacterial communities are closely associated with shrimp disease severity. Applied Microbiology and Biotechnology, 2015, 99(16):6911-6919. |
| [18] | Rubin BER, Sanders JG, Hampton-Marcell J, Owens SM, Gilbert JA, Moreau CS. DNA extraction protocols cause differences in 16S rRNA amplicon sequencing efficiency but not in community profile composition or structure. MicrobiologyOpen, 2014, 3(6):910-921. |
| [19] | Kennedy NA, Walker AW, Berry SH, Duncan SH, Farquarson FM, Louis, P, Thomson, JM, Satsangi, J, Flint, HJ, Parkhill, J, Lees, CW, Hold, GL. The impact of different DNA extraction kits and laboratories upon the assessment of human gut microbiota composition by 16S rRNA gene sequencing. PLoS One, 2014, 9(2):e88982. |
| [20] | Ahn SJ, Costa J, Emanuel JR. PicoGreen quantitation of DNA:effective evaluation of samples pre-or post-PCR. Nucleic Acids Research. 1996. 24(13):2623-2625. |
| [21] | Caporaso JG, Lauber CL, Walters WA, Berg-Lyons D, Huntley J, Fierer N, Owens SM, Betley J, Fraser L, Bauer M, Gormley N, Gilbert JA, Smith G, Knight R. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms. ISME Journal, 2012, 6(8):1621-1624. |
| [22] | Altschul SF, Gish W, Miller W, Myers EW, Lipman DJ. Basic local alignment search tool. Journal of Molecular Biology, 1990, 215(3):403-410. |
| [23] | Kong Y. Btrim:a fast, lightweight adapter and quality trimming program for next-generation sequencing technologies. Genomics, 2011, 98(2):152-153. |
| [24] | Magoc T, Salzberg SL. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 2011, 27(21):2957-2963. |
| [25] | Edgar RC. UPARSE:highly accurate OTU sequences from microbial amplicon reads. Nature Methods, 2013, 10(10):996-998. |
| [26] | Schloss, PD, Westcott SL, Ryabin T, Hall JR, Hartmann M, Hollister EB, Lesniewski RA, Oakley BB, Parks DH, Robinson CJ, Sahl JW, Stres B, Thallinger GG, Van Horn DJ, Weber CF. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities. Appllied and Environmental Microbiology, 2009, 75(23):7537-7541. |
| [27] | Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA 5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance and maximum parsimony methods. Molcular Biology and Evolution, 2011, 28(10):2731-2739. |
| [28] | Xu DF, Li CH, Sun LJ, Wang YL, Ye RY. Comparative analysis of extraction method in genome DNA from intestinal microflora of penaeus vannamei. Biotechnology Bulletin, 2013,(12):119-122.(in Chinese) 徐德峰, 李彩虹, 孙力军, 王雅玲, 叶日英. 南美白对虾肠道微生物宏基因组提取方法的比较. 生物技术通报, 2013,(12):119-122. |
| [29] | Liu J, Song XL, Liu L, Chai PL, Huang J. Effects of digestive tract probiotics on immune enzyme activity and anti-WSSV ability of Litopenaeus vannamei. Journal of Fisheries of China, 2012, 36(3):444-450.(in Chinese) 刘君,宋晓玲, 刘莉, 柴鹏程, 黄倢. 2株消化道优势菌对凡纳滨对虾免疫酶活性和抗白斑综合征病毒感染力的影响. 水产学报, 2012, 36(3):444-450. |
| [30] | Urbanczyk H, Ast JC, Higgins MJ, Carson J. Dunlap PV. Reclassification of Vibrio fischeri, Vibrio logei, Vibrio salmonicida and Vibrio wodanis as Aliivibrio fischeri gen. nov., comb. nov., Aliivibrio logei comb. nov., Aliivibrio salmonicida comb. nov. and Aliivibrio wodanis comb. nov.. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(12):2823-2829. |
